Bu içeriği görüntülemek için JoVE aboneliği gereklidir. Oturum açın veya ücretsiz deneme sürümünü başlatın.
Method Article
Histoflow Sitometri ile Yüksek Parametreli Histoloji Görüntülerinin Oluşturulması ve Analiz Edilmesi
Bu Makalede
Özet
Burada açıklanan, immünofloresan mikroskobu ile beş veya daha fazla floresan parametreyi görüntülemek için kullanılabilecek bir yöntemdir. Bu görüntülerden tek hücrelerin çıkarılması ve doku bölümlerindeki hücre alt kümelerini tanımlayabilen akış sitometrisi benzeri geçit stratejileri yoluyla tek hücre analizi yapmak için bir analiz boru hattı ana hatlarıyla belirtilmiştir.
Özet
Merkezi sinir sisteminden (CNS) türetilenler gibi doku kesitlerinde bağışıklık hücresi çeşitliliğini araştırmak için histolojinin kullanımı, tek bir seferde görüntülenebilen floresan parametrelerinin sayısı ile kritik bir şekilde sınırlıdır. Çoğu bağışıklık hücresi alt kümesi, protein belirteçlerinin karmaşık kombinasyonları kullanılarak akış sitometrisi kullanılarak tanımlanmıştır, bu genellikle kesin olarak tanımlamak için dört veya daha fazla parametre gerektirir, bu da çoğu geleneksel mikroskobun yeteneklerinin ötesindedir. Akış sitometrisi dokuları ayrıştırdığından ve uzamsal bilgiyi kaybettiğinden, karmaşık hücre tiplerinin rollerini sorgularken uzamsal bilgiyi koruyabilen tekniklere ihtiyaç vardır. Bu sorunlar burada, spektral olarak örtüşen floroforların sinyallerini toplayarak ve her bir ayrı floroforun sinyallerini ayırmak için spektral karıştırma kullanarak görüntülenebilen floresan parametrelerinin sayısını genişletmek için bir yöntem oluşturularak ele alınmaktadır. Bu görüntüler daha sonra yüksek parametreli histoloji görüntüleri almak ve bu görüntülerden tek hücreleri çıkarmak için bir analiz hattı kullanılarak işlenir, böylece her hücrenin benzersiz floresan özellikleri tek hücre düzeyinde analiz edilebilir. Akış sitometrisi benzeri geçit stratejileri kullanılarak, hücreler daha sonra alt kümelere ayrılabilir ve yalnızca bolluklarını ölçmekle kalmayıp aynı zamanda doku ortamıyla nasıl etkileşime girdiklerini belirlemek için histoloji bölümlerine geri eşlenebilir. Genel olarak, histoloji bölümlerinde karmaşık immün popülasyonları incelemek için histoflow sitometri kullanmanın basitliği ve potansiyeli gösterilmiştir.
Giriş
Bağışıklık sistemi hücreleri ve glial hücreler tarafından yönlendirilen iltihaplanma, her bir popülasyonun diğer 1,2,3'ün aktivitesini teşvik edebileceği CNS'nin kronik bozukluklarına katkıda bulunabilir. Bağışıklık sisteminin, CNS inflamasyonunu teşvik etmek için CNS'nin bu unsurları ile nasıl etkileşime girdiğini anlamak şu anda önemli bir ilgi konusudur ve tek hücreli RNA dizilimi gibi yüksek parametreli tekniklerle büyük ölçüde kolaylaştırılmıştır. Tek hücreli RNA dizilimi sayesinde, çeşitli CNS bozukluklarındaglial hücreler ve bağışıklık sistemi arasında geniş bir iletişim olduğunu keşfettik 4,5,6. Bu etkileşimlerin bu bozuklukları nasıl etkilediğini anlamak, bu hastalıkların biyolojisini aydınlatmak için çok önemli olacaktır.
Tek hücreli dizileme analizleriyle ilgili bir sorun, bu tekniklerin tek hücreler veya çekirdekler elde etmek için dokunun bozulmasını gerektirmesi ve bunun da tam bir uzamsal bilgi kaybına neden olmasıdır. Bir hücrenin bir dokuda nerede bulunduğunu bilmek, hücrenin iltihabı yönlendirmedeki rolünü anlamak için kritik öneme sahiptir. Örneğin, B hücreleri gibi bağışıklık hücreleri, nöroinflamasyon sırasında CNS'de konsantre olabilir; bununla birlikte, nadiren CNS parankimine girerler ve bunun yerine CNS bariyerlerine7 konsantre olurlar. Lokalizasyonları göz önüne alındığında, bu hücrelerin CNS parankimindeki glial hücrelerle fiziksel olarak etkileşime girerek CNS inflamasyonuna katkıda bulunmaları olası değildir, bu da glial hücrelerle sahip olabilecekleri herhangi bir etkileşimin salgılanan faktörler yoluyla gerçekleşeceğini düşündürür. Ek olarak, CNS bozukluklarında ortaya çıkan patoloji genellikle 8,9 yapısına sahiptir, öyle ki bir hücrenin dokudaki lokalizasyonu, bozukluğa aktif olarak katkıda bulunup bulunmadığını veya bir seyirci olup olmadığını kritik olarak belirleyebilir. Bu nedenle, bir hücrenin patolojideki rolünü değerlendirmek için uzamsal yönelimin kullanılması esastır.
Dokudaki hücrelerin incelenmesi tipik olarak immünohistokimya veya immün floresan mikroskobu kullanılarak gerçekleştirilmiştir. Bu tekniklerle ilgili bir sorun, tipik olarak aynı anda en fazla dört parametreyi görüntüleyebilmeleridir. Akış sitometrisi ve tek hücreli RNA dizileme analizlerinden bildiğimiz gibi, birçok hücre popülasyonunun tanımlanması için iki veya daha fazla parametreye ihtiyaç duyduğunu bildiğimiz için, bu teknikler için önemli bir sınırlamadır; Ayrıca, bir hücre tipi10'un belirli alt kümelerini ararken gereken parametrelerin sayısı tipik olarak artar. Bu nedenle, hücre alt kümelerinin bir doku içinde nasıl etkileşime girebileceğini incelemek için standart görüntüleme tekniklerini kullanmak pratik değildir.
Bu sorun, uzamsal RNA dizilimi11 ve görüntüleme kütle sitometrisi12 gibi uzamsal bilgileri koruyabilen daha yeni yüksek parametreli yöntemlerle kısmen aşılmıştır. Bu teknikler değerli olsa da, yaygın olarak bulunamama, üç boyutlu verileri iki boyuta indirgeme ve yürütme konusunda önemli uzmanlık gerektirme gibi çeşitli sorunları vardır. Dokuların bir antikor seti ile boyandığı ve ardından başka bir antikor seti ile boyanmadan önce önceki antikor setinin inaktive edildiği sıralı boyama olarak bilinen başka bir teknik, özel ekipman veya uzmanlığa ihtiyaç duymadan yüksek parametreli histoloji elde edebilir 13,14,15. Bununla birlikte, sıralı boyama son derece emek yoğun olabilir ve büyük miktarda mikroskopi süresi gerektirir, bu da kişisel mikroskobu olmayan laboratuvarlar için pratik olmayabilir. Bu nedenle, yaygın olarak bulunan mikroskoplarda bir seferde görüntülenebilen floresan parametrelerinin sayısını ve zamanında genişletilebilen tekniklere ihtiyaç vardır.
Yüksek parametreli veriler elde edildikten sonra, başka bir sorun ortaya çıkar: geleneksel görüntü analizi yöntemlerinin verileri başarılı bir şekilde analiz etmesi pek olası değildir. Manuel sayma veya eşikleme gibi teknikler, yalnızca analiz tek bir parametreden oluşuyorsa veya yalnızca çakışan sinyallerin sayıldığı birden fazla işaretleyici aynı lokalizasyona sahipse geçerlidir. Bu sınırlama, geleneksel analizi yüksek parametreli veri kümeleriyle çalışmak için yetersiz hale getirir. Bu veri kümelerinin başarılı analizi, histoloji görüntülerinden tek hücrelerin bölümlere ayrılması ve ardından hücre tiplerini tanımlamak için akış sitometrisi benzeri geçit stratejileri yürütülmesiyle elde edilmiştir16,17. Bununla birlikte, bu analizleri etkileyen bir diğer konu da, yalnızca ilgilenilen tüm hücrelerin fiziksel olarak birbirinden ayrıldığı veri kümeleri için çalışmalarıdır, çünkü bu teknikler fiziksel temas halinde olan hücreleri doğru bir şekilde ayırabilecek yöntemler kullanmaz. Bu nedenle, hücreler fiziksel temas halinde olsa bile histoloji kesitleri üzerinde tek hücreli analizler yapabilen daha yeni bir yöntem gereklidir.
Bu makalede, daha önce tanıtılanve yaygın olarak bulunan mikroskoplar kullanılarak aynı anda görüntülenebilen floresan parametrelerinin sayısını genişleten histoflow sitometri adı verilen basit bir protokol açıklanmaktadır 18. Bu protokol, dokuları spektral olarak örtüşen boyalarla boyayarak ve daha sonra net tek lekeler elde etmek için üst üste binen kanallardan kanamayı gidermek için spektral kompanzasyon kullanarak çalışır. Yüksek parametreli histoloji görüntülerinin analizini kolaylaştırmak için, akış sitometrisi benzeri geçit stratejileri kullanarak hücreleri farklı popülasyonlara ayırmak amacıyla doku bölümlerinden tek hücreleri çıkaran ayrıntılı bir analiz hattı açıklanmaktadır. Bu protokol, hücrelerin yaygın olarak bulunduğu dokularda ve hücrelerin birbirine sıkı sıkıya sıkıştırıldığı dokularda çalışır ve bu tekniği hem homeostaz hem de nöroinflamasyonda CNS gibi dokuların incelenmesi için çok yönlü hale getirir. Bu nedenle, histoflow sitometri, uzamsal bilgileri korurken hücreleri tanımlamak için birden fazla hücre işaretleyicisi gerektiren karmaşık hücre tipleri arasındaki etkileşimleri incelemek için yararlı bir tekniktir.
Protokol
Bu protokol histoloji için dokuların kesitini kapsamaz; histoloji için dokuların nasıl kesileceğine dair açıklamalar için lütfen Jain ve ark.18 veya19'a bakınız. Bu protokol, cam slaytlar üzerindeki herhangi bir kesitli doku ile kullanılabilir. Bu makale, daha önce tarif edildiği gibi aşılanmış bir hayvandan izole edilen kasık lenf nodlarını kullanır18. Bu protokolün prosedürü ve zaman çizelgesi Şekil 1'de özetlenmiştir. Bu çalışmada kullanılan reaktiflerin ve ekipmanların detayları Malzeme Tablosunda listelenmiştir.
1. Boyama dokusu bölümleri
NOT: Araştırmacının ilgilendiği tüm antikor etiketleriyle dokuları boyamaya ek olarak, araştırmacı ayrıca bitişik veya başka bir şekilde aynı doku slaytları üzerinde tek renk kontrolleri (kullanılması amaçlanan her floresan parametresi için bir tane) hazırlamalıdır, burada her bir renk kontrolü, bir floresan parametresinin sinyalini üretmek için gereken reaktiflerle boyanır.
- Perma ve blokaj tamponu ve Perma ve boyama tamponu hazırlayın (kompozisyon için Tablo 1'e bakınız).
- Cam slaytlardaki doku bölümlerini oda sıcaklığında çözün ve bir mendil kullanarak yoğuşmayı silin. Lekelenmesi gereken doku(lar)ın sınırını belirlemek için hidrofobik bir işaretleyici kullanın ve işaretlerin kurumasını bekleyin.
- Doku bölümlerini 1 dakika boyunca fosfat tamponlu salin (PBS) içinde bekleterek dokuları yeniden sulandırın. PBS'yi sallayın ve dokuya doğrudan dokunmadan fazla PBS'yi çıkarmak için bir mendil kullanın.
- Dokuyu tamamen kaplayacak kadar %4 paraformaldehit (PFA) ekleyerek dokuları sabitleyin. Oda sıcaklığında 20 dakika inkübe edin.
DİKKAT: PFA toksik olabilir ve çeker ocak içinde kullanılmalıdır.
NOT: Bu adım yalnızca taze donmuş doku için gereklidir. Doku zaten sabitse, bu adımı göz ardı edin. PFA fiksasyonu burada açıklanmıştır, ancak doku, deneyin ihtiyaçlarına bağlı olarak diğer yöntemler kullanılarak sabitlenebilir. - Mendilleri bir PBS banyosunda 1 dakika boyunca hafifçe sallayarak yıkayın ve ardından slaytı banyodan çıkarın. PBS'yi değiştirin ve yıkamayı 3 kez daha tekrarlayın.
- Fazla PBS'yi silkeleyin ve dokuya doğrudan dokunmadan fazla PBS'yi çıkarmak için bir mendil kullanın.
- Protokolün 1.12. adımında hangi ikincil antikorların kullanılması gerektiğini ve ikincil antikorların türetildiği hayvan türlerini belirleyin. Bu hayvanlardan Perm'e serum ekleyerek ve tamponu her tür için% 5'lik bir nihai konsantrasyona kadar bloke ederek Perma'yı ve bloke edici tamponu güçlendirin.
NOT: İkincil antikorların kullanılması, sinyal yoğunluğunu yükseltmeye yardımcı olur. Bu nedenle, zayıf sinyaller üreten birincil antikorların, sinyallerini yükseltmek için ikincil antikorlarla eşleştirilmesi önerilir. Boyanan tüm antikor hedefleri ikincil antikorlara ihtiyaç duymayacak kadar parlaksa, adım 1.7-1.12 ve adım 1.15 atlanabilir. - Her dokuyu kaplayacak kadar güçlendirilmiş Perm ve bloke edici tampon ekleyerek antikorların spesifik olmayan bağlanmasını bloke edin. Slaytları ışıktan korunan nemlendirilmiş bir odada oda sıcaklığında 1-8 saat inkübe edin.
- Deneysel olarak belirlenen seyreltmelerde Perm ve boyama tamponuna konjuge olmayan birincil antikorlar ekleyin (birincil antikorların türleri veya immünoglobulin izotiplerinin tümü farklı olmalı ve araştırmacının adım 1.12'de kullanmayı planladığı ikincil antikorlar tarafından ayırt edilebilir olmalıdır).
- Perma ve bloke edici tamponu boşaltın ve dokuya doğrudan dokunmadan fazla tamponu çıkarmak için bir mendil kullanın. Dokuyu kaplayacak kadar Perma ve primer antikorlar içeren boyama tamponu ekleyin ve slaytı 4 ° C'de 12-48 saat inkübe edin.
NOT: Araştırmacının antikor inkübasyonu için en uygun zamanı belirlemesi gerekecektir. - Engelleme tamponunu ve boyama tamponunu hazırlayın (Tablo 1).
- Deneysel olarak belirlenen seyreltmelerde adım 1.11'de hazırlanan boyama tamponuna ikincil antikorlar ekleyin. Kullanılan ikincil antikorlar, adım 1.9'da kullanılan aynı türleri veya antikor izotiplerini hedeflemelidir.
- Söndürme tamponunu hazırlayın ( Tablo 1'deki gibi).
NOT: Bu adım, dokudaki otofloresanı azaltmaya yardımcı olur. Bu gerekli değilse, 1.13 ve 1.14 adımları atlanabilir. - Mendilleri adım 1.5-1.6'da anlatıldığı gibi yıkayın. Daha sonra slaytların üzerindeki dokuları kaplayacak kadar su verme tamponu ekleyin ve slaytları oda sıcaklığında 1-2 dakika inkübe edin.
- Mendili 1.5-1.6 adımlarında anlatıldığı gibi yıkayın. Daha sonra, adım 1.12'de hazırlanan boyama tamponunda seyreltilen ikincil antikorları, dokular tamamen boyama tamponu ile kaplanacak şekilde dokulara ekleyin. Slaytları 4 ° C'de 2-8 saat inkübe edin.
- Adım 1.18'de kullanılacak antikor türlerinden (her türün %5'i) 50 μL normal serumu, adım 1.11'de hazırlanan bloke edici tampona ekleyerek blokaj solüsyonunu hazırlayın.
- Mendilleri adım 1.5-1.6'da anlatıldığı gibi yıkayın. Daha sonra, adım 1.16'da hazırlanan normal serum ile blokaj solüsyonunu dokulara tamamen boyama tamponu ile kaplanacak şekilde ekleyin. Slaytları oda sıcaklığında 1-8 saat inkübe edin.
- Adım 1.11'de hazırlanan boyama tamponuna deneysel olarak belirlenmiş seyreltmelerde florofor konjuge primer antikorlar ekleyerek boyama solüsyonunu hazırlayın. Ayrıca, deneysel olarak belirlenmiş bir seyreltmede boyama çözeltisine nükleer sarı gibi bir nükleer leke ekleyin.
NOT: Adım 1.18-1.19, yalnızca araştırmacının sinyal amplifikasyonu için ikincil antikorlar gerektirmeyen ek antikor etiketleri kullanması gerekiyorsa gereklidir. Birincil konjuge antikorlar kullanılmıyorsa, adım 1.12'de çekirdek boyaları eklenebilir. - Bloke edici tamponu sallayın ve dokuya doğrudan dokunmadan fazla tamponu çıkarmak için bir mendil kullanın. Adım 1.18'de hazırlanan boyama solüsyonunu dokuya kaplayacak kadar ekleyin ve 4 ° C'de 12-48 saat inkübe edin.
- Mendilleri adım 1.5-1.6'da anlatıldığı gibi yıkayın. Kabarcıklar oluşturmadan her slayta 5 damla mountant ekleyin ve dokuyu tamamen mountant ile kaplamak ve tüm hava kabarcıklarını dışlamak için dokunun üzerine nazikçe ve yavaşça bir cam lamel eğin. Slaytları görüntülenmeye hazır olana kadar karanlıkta 4 °C'de saklayın.
2. Doku kesitlerinin görüntülenmesi
NOT: Bu aşamada, doku kesitleri, görüntülenmesi amaçlanan tüm durumlar için ilgilenilen tüm antikorlarla boyanır ve tek renkli kontroller, her bölümde bir floresan parametre ile boyanır. Görüntüleme, belirli ışık aralıklarını tespit etmek için ayarlanabilen birden fazla dedektöre sahip bir mikroskopta yapılmalı ve ideal olarak, uyarma için mümkün olduğunca çok sayıda lazer çizgisine erişimi olmalıdır.
- Tamamen lekeli slaytlardan birini mikroskoba yerleştirin ve mikroskobu, veri toplamak için kullanılması amaçlanan hedefi kullanarak bölümü görüntüleyecek şekilde ayarlayın.
- Dokunun boyandığı floroforlara dayanarak, bu floroforları uyarmak için gereken uyarma lazer çizgilerini belirleyin ve en fazla miktarda sinyal içeren her bir florofor için emisyon aralıklarını belirleyin.
NOT: Çevrimiçi floresan spektrum görüntüleme araçları (örneğin, Malzeme Tablosuna bakın), floroforların en uygun uyarma dalga boylarını belirlemek ve florofor tespiti için en uygun aralıkları belirlemek için yararlı olabilir. Histoflow sitometrisi için kullanılan floroforlar, her bir floroforun, floroforun uyarıldığı optimal lazer hattında farklılık göstereceği veya optimum emisyon dalga boyları aralığına göre farklılık göstereceği şekilde seçilmelidir. Daha yakın uyarma/emisyon dalga boylarına sahip floroforların ayrılması daha zor olacaktır. Bu çalışma için kullanılan floroforlar sonuçlar bölümünde detaylandırılmıştır. - Yazılım 1'i (Malzeme Tablosuna bakın) konfokal mikroskopta açın ve üst kısımdaki konfigürasyon sekmesini seçin. Ardından sol taraftaki donanım düğmesini seçin ve bit derinliğini 16 bit olarak değiştirin.
NOT: Spektral telafisinin doğruluğunu artıracağı için veri toplama için 16 bit veri gibi daha yüksek bit derinliklerinin kullanılması önerilir. Ek olarak, daha yüksek piksel yoğunlukları, satır ortalamaları için daha büyük sayılar ve z-yığınında daha fazla adım, spektral ayrımın kalitesini artıracaktır. - Ekranın üst kısmındaki Al sekmesini seçin ve görüntünün piksel yoğunluğunu dahil etmek için biçim ayarını girin (genellikle daha yüksek çözünürlükler daha iyi sonuçlar verir).
- Varsa, bu özelliğin yeterince kalibre edilmiş olması koşuluyla, görüntüleme süresini hızlandırmak için çift yönlü X alımını seçin.
- Konfokal mikroskop için bir iğne deliği değeri seçin (tipik olarak 400-600 nm arasında), daha küçük değerlerin tipik olarak daha yüksek kaliteli histoflow sitometri sonuçları üreteceği yerde, daha küçük iğne deliği değerleri görüntü elde etmeyi zorlaştıracak ve ek görüntüleme süresi gerektirebilecek veya daha yüksek uyarma yoğunlukları üretebilir.
- Sıralı tarama seçeneğini açmak için sol menünün üst kısmındaki Sıralı Tarama Panelini Göster'i seçin. Ek diziler oluşturmak için '+' işaretini kullanın, burada her bir dizi, adım 2.2'de kararlaştırılan bir uyarma dalga boyunu içerecektir.
- Ekranın ortasındaki ilgilenilen lazerlerle ilişkili AÇIK düğmelerine tıklayarak ihtiyaç duyulan tüm lazerleri AÇIK duruma getirin. Her bir özel diziye gidin ve ekranın ortasındaki lazer gücünü, her dizinin bir uyarma dalga boyunun kullanılacağı istenen değere yükseltin.
- Bu lazer çizgisinde görüntülenecek tüm floroforları görüntülemek için her dizide ekranın alt kısmında gerektiği kadar dedektör açın . Dedektörlerin aralıklarını , aralıklarına çift tıklayarak ve toplanması gereken üst ve alt dalga boyu aralıklarını girerek ayarlayın.
NOT: Mümkün olduğunda yüksek hassasiyetli dedektörlerin kullanılması önerilir ve ideal olarak, bu gerekli olmasa da tüm kanalların aynı tip dedektörlerde toplanması gerekir. - Canlı düğmesine basın, ardından örneğe odaklanın. Kanalların sinyale yeterince doygun olup olmadığını değerlendirmek için yazılımdaki görüntünün solundaki Otomatik Ölçek düğmesini kullanın. Kullanıcı, otomatik ölçeklendirme maksimum değere (16 bit veriler için 65535) ulaşmayana ancak tüm kanallar için doygunluğa çok yakın olana kadar her dizideki kazanç değerlerini ve lazer yoğunluklarını ayarlamalıdır.
NOT: Farklı floroforların parlaklıkları ve farklı antikor hedefleri bollukları açısından farklılık gösterebileceğinden, her iki florofor da aynı lazer tarafından uyarılırsa, dedektörlerin hassasiyetinin parlak sinyaller için düşük hassasiyet ve loş sinyaller için yüksek hassasiyet olacak şekilde ayarlanması gerekecektir (bu senaryoda her iki florofor da etkileneceğinden lazer yoğunluğu değişemez). En iyi sonuçlar için, deneydeki her kanaldan gelen floresan sinyali, her kanalın kabaca eşdeğer miktarda sinyale sahip olması için dengelenmelidir, bu da diğer kanallara spektral sızıntıyı azaltacak ve daha güçlü sinyallerle sonuçlanacaktır. Farklı numuneler, boyanan ilgilenilen belirteçlerin bolluğu açısından farklılık gösterebileceğinden, deney koşullarının hiçbirinde ağartmanın meydana gelmediğini doğrulamak için mikroskop ayarlarının birden fazla deney koşulunda test edilmesi önerilir. - Mikroskop ayarları tamamlandıktan sonra, mikroskobu tek renk kontrol slaytlarıyla yükleyin ve her florofor için temsili boyama bulun. Örneğin altına odaklanarak ve z yığını menüsündeki başlat düğmesine basarak, ardından örneğin üzerine odaklanarak ve sonlandır düğmesine basarak bir Z yığını ayarlayın.
- Adım sayısını ve adımlar arasındaki mesafeyi seçin (ve tamamen lekeli slaytları görüntülemek için kullanılacak piksel yoğunlukları ve bit derinliği gibi aynı sayıda adım ve parametre kullanın), ardından başlat düğmesine basın.
NOT: İdeal olarak, bu görüntüler slaytta bulunabilecek gerçek boyamadan gelen en parlak sinyalleri içermelidir. - Tek renk kontrolleri görüntülendikten sonra, tamamen lekeli tüm numuneleri görüntülemek için aynı mikroskop ayarlarını kullanmaya devam edin.
3. Floresan telafisi
- Yazılım 1'de .lif dosyalarını açın ve işlem sekmesine tıklayın.
- Boya Ayırma modülünü seçin ve ardından Otomatik Boya Ayırma seçeneğini seçin. Boya Ayrımı için El ile yöntemini seçin ve yeniden ölçeklendirme yok'u seçin.
- Tek renkli kontrol görüntülerinden birini açın. Uygun olmayan kanallarda bir sinyal kanıtı olup olmadığını görmek için her kanal için gri tonlamalı pencereleri gözlemleyerek bir kanaldan diğerine spektral taşma için tek renk kontrollerini manuel olarak inceleyin. Zayıf taşmayı görmek için gerekirse Otomatik Ölçeklendir düğmesini kullanın.
- Ekrandaki matrise manuel olarak sayılar girerek (genellikle 0-1, burada 0 taşma olmamasını ve 1 %100 taşma payını temsil eder) taşma payını kaldırmaya başlayın ve ardından taşma payının yeterince kaldırılıp kaldırılmadığını test etmek için uygula düğmesine basın. Bu, ilgilenilen kanal dışındaki diğer tüm kanallardaki sinyaller arka plan seviyelerine düşürülene kadar tekrarlanmalıdır.
NOT: Aşırı kompanzasyon yanlış sonuçlara yol açacaktır, bu nedenle floresan arka plan seviyelerine düşürülmeli, ancak bunların altına düşürülmemelidir. Aşırı kompanzasyonlu veriler, bir kanalın bir sinyale sahip olması ve diğer kanalların ilgilenilen sinyalin olduğu yerde kara deliklere sahip gibi görünmesi olarak görünecektir (Şekil 2). - Tek renkli denetimlerden biri tamamlandığında, değerleri matrise kaydedin ve ardından matrisi sıfırlayın ve bir sonraki tek renkli denetime geçin. Her bir renk kontrolü için bu işlemi tekrarlayın.
NOT: İlk boya Boya 1 satırında olacak ve Boya 1'den çıkarılması gereken floresan miktarı bitişik sütunlarda olacaktır. - Her kontrolden elde edilen tüm değerleri tek bir matriste birleştirin ve bu matrisi tamamen lekeli bir numuneye uygulayın . Renk ayrımı doğru görünüyorsa, ayarları deneydeki tüm görüntülere uygulayın, aksi takdirde tek renk kontrollerini yeniden değerlendirin.
NOT: Etkili kompanzasyon, Şekil 3'te özetlendiği ve gösterildiği gibi, kanallar arasındaki taşma payının çoğunu ortadan kaldırmalıdır. - Telafi edilen tüm dosyaları ayrı bir .lif dosyasına kaydedin.
4. ilastik kullanarak çekirdek ve hücrelerin tanımlanması
- Telafi edilmiş .lif dosyalarını FIJI20'de açın. Yığınları bir hiper yığında görüntülemeyi etkinleştirin ve bir görüntü seçin.
- (İsteğe bağlı) Görüntünün analiz edilecek kısımlarını dahil etmek ve analiz edilmeyecek alanlarını dışarıda bırakmak için dikdörtgen aracı gibi bir seçim aracı kullanın. Görüntü > kırpma işlevini kullanarak görüntüyü kırpın.
NOT: Görüntünün boyutunu küçültmek, rasgele erişimli bellek (RAM) tüketimini azaltmak ve işlem hızını artırmak için sonraki adımlarda yararlıdır. - Görüntüyü .tif dosyası olarak kaydedin. Ayrıca, ilastik > Eklentiler > Dışa Aktar hdf5'e tıklayarak FIJI için ilastik eklentisini kullanarak görüntüyü bir hdf5 dosyası olarak kaydedin. Dosyayı 0 sıkıştırma ile "tercih edilen ad".h5 olarak kaydedin.
- Ilastik21'i açın ve bir piksel sınıflandırma projesi yapın. Yeni giriş verileri eklemeye giderek en az 3 temsili görüntü eklemek için Yeni Ekle > Ayrı Görüntü Ekle işlevini kullanın.
- Görüntülerden birine sağ tıklayın, Özellikleri düzenle'yi seçin ve Görüntü Modunu gri tonlamalı olarak değiştirin.
- Ekranın sol tarafında, özellik seçimi sekmesini seçin. Özellikleri seç'e tıklayın ve programın kullanmanıza izin vereceği en yüksek sigma değerine kadar olan tüm özellikleri seçin, ardından Tamam'a basın.
- Şimdi Eğitim sekmesini seçin. İlgilenilen hücreleri (Şekil 4) iç kısım (sitoplazma ve çekirdek) ve hücre zarı vurguya dahil edilecek şekilde vurgulamak için boya fırçası aracını (birinci etiket seçiliyken) kullanın.
- Bunu, ilgilenilen tüm hücre tiplerinin seçildiği tüm eğitim örneklerinde tekrarlayın ve her örnekten dokuda farklı yoğunluklarda, morfolojilerde ve lokalizasyonlarda birden fazla hücre seçilmelidir.
NOT: Tek bir leke seti için seçilen belirteçlere bağlı olarak, ilgilenilen hücreler için hücre içi lekeler veya hücre zarı lekeleri olabilir veya olmayabilir. Hangi işaretleyiciler kullanılırsa kullanılsın, vurgunun ilgilenilen hücreler üzerindeki en dıştaki lekeyi içerdiğinden ve hücre içi bir leke kullanılıp kullanılmadığına bakılmaksızın hücre içini içerdiğinden emin olun. - Ardından, ilgilenilen hücreler olmayan her şeyi vurgulamak için ikinci etiketi kullanın (Şekil 4). Bu, boş alanı, etiketlenmemiş çekirdekleri veya ilgilenilen belirteçleri olmayan dokuları içerebilir. Kesin hücre sınırlarının öğrenilmesini teşvik etmek için ilgilenilen hücrelerin hemen yanındaki alanı dahil ettiğinizden emin olun.
- İlgilenilen görüntülerle ilgili eğitimin kalitesini değerlendirmek için Canlı Güncelleme işlevini kullanın. Eğitim yetersizse, 4.7 ila 4.9 arasındaki adımlarda eğitime devam edin. Aksi takdirde, tatmin ediciyse, Tahmin Dışa Aktarma sekmesine tıklayın.
- Kaynağı olasılıklar olarak ayarlayın. Görüntü Ayarlarını Dışa Aktar Seç düğmesine tıklayın, ardından c parametresini 0-1'i dışa aktaracak şekilde ayarlayın (etiket 1'in ilgilenilen parametreleri vurguladığı varsayılarak).
- Veri türünü 16 bitlik bir Tamsayıya dönüştürün ve ilgilendiğiniz adla ilgilendiğiniz dizine bir hdf5 dosyası olarak dışa aktarın ({takma ad} mask.h5 kullanmanızı öneririz), ardından Tamam'a basın.
- Toplu İşleme sekmesine tıklayın. Ham Veri Dosyalarını Seç düğmesine basın, işlenmesi gereken tüm .h5 dosyalarını seçin ve ardından Aç'a basın. Tüm dosyaları işle'ye tıklayın.
- 4.4 ila 4.13 arasındaki adımları tekrarlayın, ancak hücreleri tanımlamak yerine, şimdi çekirdekleri tanımlayın ve diğer her şeyi hariç tutun (Şekil 4). Bunu, hücre çekirdeklerini tanımlamak için ayrı bir tahmin olarak dışa aktarın.
- İlastik'i açın ve yeni bir Veri Dönüştürme projesi oluşturun. Yeni Ekle > Ayrı Görüntü Ekle işlevini kullanarak Giriş verileri sekmesine temsili bir hdf5 dosyası ekleyin.
- Veri Dışa Aktarma sekmesine tıklayın ve Kaynak giriş olarak ayarlanmış olarak Görüntü Ayarlarını Dışa Aktar Ayarlarını Seç düğmesini seçin. Formatı çok sayfalı tiff olarak değiştirin, select işleviyle bir kaydetme konumu seçin ve dosyanın adını {nickname} olarak ayarlayın. Tamam'a basın.
- Toplu işleme sekmesine tıklayın ve Giriş Veri Dosyalarını Seç düğmesini kullanarak tüm hdf5 dosyalarınızı seçin ve Tüm dosyaları işle düğmesine basın.
- Yazılım 2'yi açın (Malzeme Tablosuna bakın) ve Dosya Ekle düğmesini kullanarak adım 4.3 ve 4.17'de oluşturulan tüm tif/tiff dosyalarını ekleyin. Dosya çıktıları için bir konum seçin ve tüm dosyaları .ims dosyalarına dönüştürmek için Tümünü Başlat düğmesine basın. Dosyalar artık Yazılım 3'te analiz için hazırdır (bkz. Malzeme Tablosu).
5. Yazılımda Analiz 3
- Tüm floresan lekelerini içeren resimlerden birini açın. Düzenle-> Kanal Ekle'yi seçin ve çekirdekler ve hücre gövdeleri üzerinden yapılan maskelere karşılık gelen .ims dosyalarını ekleyin. Bunu tüm numuneler için tekrarlayın.
- Yeni Hücre Ekle düğmesini seçin. Çekirdeği ve hücreyi algıla seçeneğini seçin ve ilerleyin.
- Çekirdekleri tespit etmek için kaynak kanal olarak çekirdekler üzerindeki maskeyi seçin ve çekirdekleri tohum noktalarına bölmek için gelişmiş seçeneği belirleyin. Çekirdek çapı için bir değer seçin) ve ilerleyin.
NOT: Çekirdek çapı için en uygun değerin, simülasyonu çalıştırarak ve programın yeterince çekirdeği bölmediğini (birden fazla çekirdek birlikte gruplandırılmıştır) veya aşırı bölünme olup olmadığını (bir çekirdek birden fazla parçaya bölünmüş) manuel olarak değerlendirerek test edilmesi gerekecektir. - Kalite değerlendirmesi için bir değer seçin ve devam edin. Tipik olarak, tüm çekirdekleri dahil etmek en iyisidir, çünkü çekirdeklerin ihmal edilmesi birden fazla hücrenin tek bir hücrede kaynaşmasına yol açabilir.
- Nükleer yüzeyler oluşturmak için bir eşik değeri seçin ve ardından devam edin. Daha sönük olanlar da dahil olmak üzere tüm çekirdeklerin hala eşiğe dahil edildiğinden emin olun. Eşiği çok düşük yapmayın, çünkü bu, çekirdeğin boyutunu abartabilir veya potansiyel olarak nükleer lekelenme olarak döküntü içerebilir.
- Çekirdek başına minimum Voksel sayısı için bir değer seçin ve devam edin. Tipik olarak, bu parametre kullanılarak enkaz veya çekirdek olmayanlar hariç tutulmadıkça her şeyi saklamak en iyisidir.
- Hücre sınırı ve sitoplazma seçeneğini seçin, hücre gövdelerini algılamak için kaynak kanal olarak hücreler üzerindeki maskeyi seçin ve ardından devam edin.
- İlgilenilen tüm hücreleri doğru bir şekilde yakalayan, ancak arka planı veya hücrelerin spesifik olmayan herhangi bir algılamasını içerecek kadar düşük olmayan bir Hücre eşiği seçin. Dokunan hücreleri, hücre başına bir çekirdek olacak şekilde bölme seçeneğini belirleyin ve bunlar çekirdeğe olan mesafeye göre ayrılır. Ayrıca, çekirdeklerdeki hücreleri genişletmeyi seçin ve ardından devam edin.
- Hücre başına minimum voksel sayısı için bir değer seçin ve devam edin. Tipik olarak, bu parametre kullanılarak enkaz veya hücre olmayanlar hariç tutulmadıkça her şeyi saklamak en iyisidir.
- Birden çok hücrenin birbirine kaynaşıp kaynaşmadığına veya hücrelerin eksik olup olmadığına bakarak hücrelerin üzerinde oluşturulan yüzeylerin doğru olup olmadığını değerlendirin. Yüzeyler yetersizse, adım 5.2'ye geri dönün ve en uygun ayarlar belirlenene kadar yeni parametreler deneyin.
- En iyi ayarlar oluşturulduktan sonra, soldaki menüden oluşturulan hücre nesnesine tıklayın, oluşturma sekmesine tıklayın ve kullanılan ayarları kaydetmek için Toplu İş için Parametreleri Sakla düğmesine tıklayın.
- Yazılım 3'ün birden çok penceresini açın ve her pencerede ayrı görüntüler açın. Her pencerede yeni Hücreler düğmesine basın, Favori Oluşturma Parametreleri menüsünde kaydedilen ayarları seçin ve her pencereye tamamlanana kadar çalışmasını söyleyin. Tüm numuneler analiz edilene kadar bunu gerektiği kadar tekrarlayın.
NOT: Yazılım 3'ün birden çok örneğini çalıştırmak bu analizin daha hızlı ilerlemesini sağlar, ancak paralel olarak çalıştırılabilecek pencere sayısı, merkezi işlem birimi (CPU) işlem hızına ve analizin çalıştırıldığı bilgisayarın RAM miktarına bağlı olacaktır. - Tüm örnekler tamamlandıktan sonra, her görüntüde sol menüde oluşturulan hücre nesnesine tıklayın ve İstatistikler sekmesine tıklayın. Görünür İstatistik Değerleri listesini yapılandır düğmesini tıklayın, 'Hücre Yoğunluğu Ortalaması' değeri dışındaki tüm istatistiklerin seçimini kaldırın ve ardından Tamam'a basın.
- Analiz edilmesi gereken her görüntü için, istatistikler sekmesindeki Tüm istatistikleri dosyaya aktar düğmesini tıklayın ve dosyaları bir konuma kaydedin.
6. Histoflow sitometri analizi ve popülasyonların doku kesitlerine haritalanması
- Anaconda ve JupyterLab'ı Anaconda içinde açın. Ardından, Ek Kodlama Dosyası 1'de sağlanan komut dosyasını açın. Çalıştır menüsünü kullanarak kodu çalıştırın ve Tüm Hücreleri Çalıştır'ı seçin veya Seçili Hücreleri Çalıştır'ı seçerek hücreleri sırayla tek tek çalıştırın.
- Kaynak dosyayı girmeniz istendiğinde, adım 5.14'te oluşturulan dışa aktarılan floresan değerleri için dosya dizinini girin. Dosya dizini, dışa aktarılan .csv dosyalarından birinin özelliklerine bakılarak ve dosya yolunu komut istemine kopyalayarak elde edilebilir.
- Ardından, program işlenen dosyalar için bir çıktı konumu isteyecektir. İşlenen dosyaları kaydetmek için uygun olan herhangi bir konum için dosya dizinini girin. Program, klasörde bulunan floresan kanallarının sayısını otomatik olarak algılayacaktır.
- Verilere her kanalın adlarıyla açıklama eklemek için kodun sonraki bölümünü çalıştırın. Bu kod, "numberOfChannels" parametresini adım 6.3'te algılanan kanal sayısıyla eşleşecek şekilde değiştirmek için gerektiği gibi değiştirilebilir. Ek olarak, kanalların adları, 'ChX':'Marker' satırını gerektiği kadar tekrarlayarak ve işaretçilerin adlarını değiştirerek gerektiği kadar kanal için değiştirilebilir.
- Sonraki kod satırını çalıştırmak, belirtilen konumda açıklamalı ve veri kümesindeki tüm floresan parametrelerini içeren bir .csv dosyası oluşturur. Tüm veriler için .csv dosyaları oluşturmak üzere her Yazılım 3 dışa aktarma kümesi için 6.1-6.5 arasındaki adımları yineleyin.
- Yazılım 4'ü açarak (Malzeme Tablosuna bakın) ve .csv dosyalarını program penceresine sürükleyerek .csv dosyalarını .fcs dosyalarına dönüştürün. Yazılım 4, dosyaları otomatik olarak .fcs dosyalarına dönüştürmeye başlayacaktır.
- Yazılım 4'teki örneklerden birine çift tıklayarak bu örnekte bir pencere açın. Bir geçit stratejisi oluşturmak için Yazılım 4'teki geçit araçlarını kullanın. İlgilenilen tüm hücreleri içeren soy tanımlayıcı bir işaretleyici üzerinde geçit oluşturarak başlamanız, ardından önce daha nadir alt kümeleri kaldıran kapılara ilerlemeniz ve ardından daha bol hücre tiplerinde bitirmeniz önerilir.
- Bir geçit stratejisi oluşturulduktan sonra, menüde o popülasyona sağ tıklayarak ve dışa aktarmayı seçerek geçitin doğruluğunu test edin. Tüm parametreleri, başlıklar dahil olmak üzere bir .csv dosyası olarak dışa aktarın.
- Anaconda'da JupyterLab'ı açın. Ardından, Ek Kodlama Dosyası 2'de sağlanan komut dosyasını açın ve kodu çalıştırın. Yazılım 4 dosyasının konumunu girmeniz istendiğinde, önceki adımda oluşturulan dışa aktarılan .csv dosyasının dosya dizinini girin. Dosya dizini, dışa aktarılan .csv dosyasının özelliklerine bakılarak ve dosya yolunu komut istemine kopyalayarak elde edilebilir.
- Çıktı konumunu eklemeniz istendiğinde, tercih ettiğiniz herhangi bir dosya dizinini seçin ve dosya dizininin sonunda dosya adını içerdiğinden emin olun ('dosya dizini'\filename.txt). Kodun geri kalanını çalıştırın.
- .txt dosyasındaki metni kopyalayın ve dışa aktarılan verilerin temel aldığı aynı örnek için ilgili .ims dosyasını Yazılım 3'te açın. Dosyadaki Hücre nesnesine tıklayın, istatistikler sekmesine geçin, metni arama çubuğuna yapıştırın ve aramayı başlatın. İlgilenilen tüm hücreler vurgulanacaktır.
- İlgilenilen tüm hücrelerin geçit stratejisi kullanılarak yakalanıp yakalanmadığını manuel olarak inceleyin. Hücre sayıları az veya fazla tahmin ediliyorsa ( Şekil 5'te gösterilen örnekler), adım 6.7'ye geri dönün, geçidi ayarlayın ve hücrelerin yeni geçitle doğru bir şekilde yakalanıp yakalanmadığını yeniden değerlendirin. Bunu geçit stratejisindeki tüm kapılarla tekrarlayın ve geçitlemeyi birden fazla örnek üzerinde değerlendirin.
- Tüm geçitler doğrulandıktan sonra, tablo düzenleyici işlevini kullanarak verileri Yazılım 4'ten dışa aktarın ve tercih edilen analiz yöntemini kullanarak verileri analiz edin.
NOT: Hücre popülasyonları, 6.8-6.11 adımlarındaki gibi kapılı popülasyonu tanımlayarak ve ardından seçilen hücrelere dayalı bir yüzey oluşturarak Yazılım 3'teki doku bölümlerine geri eşlenebilir. Bu, Şekil 6 ve Şekil 7'de gösterildiği gibi, her bir kapılı nüfus için yapılabilir.
Sonuçlar
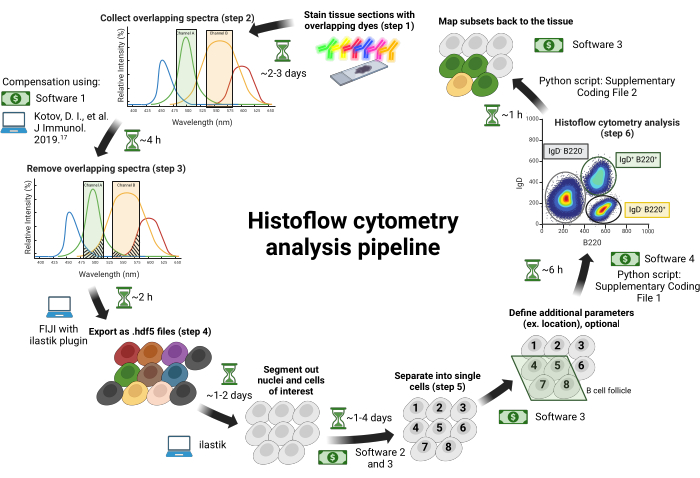
Şekil 1: Histoflow sitometri iş akışı. Doku kesitleri spektral olarak örtüşen boyalarla boyanır (adım 1). Görüntüler, floroforlar arasındaki spektral sızıntıyı en aza indirmek için ayarlanabilir bant geçiren filtrelerle eşleştirilmiş bireysel uyarma lazerleri arasında toplanır (adım 2). Kanallar arasındaki spektral kanama,...
Tartışmalar
Burada, daha önce doğrulanmış bir teknik olan histoflow sitometrisinin kullanımı açıklanmaktadır18. Doku kesitlerini spektral olarak örtüşen boyalarla boyarken, kanallar arasındaki kanamanın spektral kompanzasyon kullanılarak giderilebileceği ve bunun sonucunda normalde geleneksel yöntemlerle mümkün olandan daha fazla sayıda floresan parametresinin net bir şekilde çözüldüğü gösterilmiştir. Yüksek parametreli histoloji görüntülerinin geleneksel yöntemler kullanıla...
Açıklamalar
Yazarların herhangi bir finansal çıkar çatışması yoktur.
Teşekkürler
Görüntüleme altyapısı ve uzmanlığı için Hotchkiss Beyin Enstitüsü Gelişmiş Mikroskopi Platformu'na teşekkür ederiz. RWJ, Calgary Üniversitesi Göz Lisesi programından doktora sonrası burs fonu ve Kanada Multipl Skleroz Derneği ve Roche Kanada sınırsız eğitim bursu tarafından desteklendi. VWY, Kanada Araştırma Koltuğu Tier 1 programından maaş desteği aldı. Bu çalışma, Kanada Sağlık Araştırmaları Enstitüleri Hibe 1049959, Kanada Multipl Skleroz Derneği Hibe 3236 ve Kongre Tarafından Yönetilen Multipl Skleroz Araştırma Programı ABD Savunma Bakanlığı'nın işletme fonları ile desteklenmiştir. Şekil 1 BioRender.com ile oluşturulmuştur. Bu yayında uyarlanan rakamlar ilk olarak The Journal of Immunology'de yayınlandı. Rajiv W. Jain, David A. Elliott ve V. Wee Yong. 2023. Histoflow Sitometri Kullanılarak Yüksek Parametreli Histoloji Görüntülerinin Tek Hücre Analizi. J. İmmünol. 210: 2038-2049. Telif Hakkı © [2023]. Amerikan İmmünologlar Derneği, Inc.
Malzemeler
| Name | Company | Catalog Number | Comments |
| 100% Ethanol | Sigma | 676829-1L | |
| 4% PFA | Electron Microscopy Sciences | 157-4 | |
| Anaconda | N/A | N/A | https://www.anaconda.com/download |
| Bovine Serum Albumin | Sigma | A4503-50G | |
| Cold fish stain gelatin | Sigma | G7765 | |
| Collating multichannel data from Imaris.ipynb script | N/A | N/A | https://github.com/elliottcalgary/Histoflow-Cytometry-Analysis- |
| Convert FlowJo output to txt file for Cell selection in Imaris.ipynb script | N/A | N/A | https://github.com/elliottcalgary/Histoflow-Cytometry-Analysis- |
| Donkey anti-rat Alexa Fluor 647 | JacksonImmunoResearch | 712-605-153 | 1:300 concentration |
| Donkey anti-rat DyLight 405 | Jackson ImmunoResearch | 712-475-153 | 1:200 concentration |
| Donkey Serum | JacksonImmunoResearch | 017-000-001 | |
| F(ab')2-Goat anti-Mouse IgG PerCP-eFluor 710 | Thermofisher | 46-4010-82 | 1:25 concentration |
| FIJI | N/A | N/A | https://imagej.net/software/fiji/ |
| FlowJo | FlowJo LLC | Software 4 | |
| Fluorescence spectraviewer | https://www.thermofisher.com/order/fluorescence-spectraviewer/#!/ | ||
| Fluoromount-G | Southern Biotech | 0100-01 | |
| Fresh frozen human tonsil sections | amsbio | HF-707 | |
| Glass coverslip | VWR | 48393 106 | |
| Goat anti-human IgA Alexa Fluor 488 | JacksonImmunoResearch | 109-546-011 | 1:400 concentration |
| Goat anti-human IgG Cy3 | JacksonImmunoResearch | 709-166-098 | 1:400 concentration |
| Goat anti-human IgM Dylight 405 | JacksonImmunoResearch | 109-476-129 | 1:300 concentration |
| Goat anti-rabbit A546 | Thermo Fisher Scientific | A-11035 | 1:250 concentration |
| Goat anti-rabbit IgG PE-Alexa Fluor 610 | Thermofisher | A-20981 | 1:250 concentration |
| Horse Serum | Sigma | H1138 | |
| Ilastik | N/A | N/A | https://www.ilastik.org/ |
| Ilastik FIJI plugin | N/A | N/A | https://www.ilastik.org/documentation/fiji_export/plugin |
| Imaris File Converter | Oxford Instruments | Software 2 | |
| Imaris with cell module | Oxford Instruments | Software 3 | |
| kimwipe | Kimtech | 34155 | |
| LasX Life Science software | Leica | Software 1 | |
| Mouse anti-human CD20 | VWR | CA95024-322 | 1:40 concentration |
| Mouse anti-human CD38 APC-R700 | BD Biosciences | 564980 | 1:20 concentration |
| Normal Goat Serum | JacksonImmunoResearch | 005-000-001 | |
| Normal Mouse Serum | JacksonImmunoResearch | 015-000-001 | |
| Normal Rabbit Serum | JacksonImmunoResearch | 011-000-001 | |
| Normal Rat Serum | JacksonImmunoResearch | 012-000-120 | |
| Nuclear Yellow | Abcam | ab138903 | Dissolve in DMSO at a concentration of 2 mg/ml and store at 4°C in the dark |
| PAP pen | Cedarlane | MU22 | |
| PBS | Gibco | 10010-023 | |
| Rabbit anti-human Ki67 | Abcam | ab15580 | 1:500 concentration |
| Rabbit anti-mouse Iba1 | Wako | 019-19741 | 1:500 concentration |
| Rat anti-human Blimp1 | Thermofisher | 14-5963-82 | 1:40 concentration |
| Rat anti-mouse B220 Alexa Fluor 647 | BioLegend | 103226 | 1:250 concentration |
| Rat anti-mouse CD138 | Biolegend | 142502 | 1:200 concentration |
| Rat anti-mouse CD3 PE-eFluor 610 | Thermo Fisher Scientific | 61-0032-82 | 1:40 concentration |
| Rat anti-mouse CD4 Alexa Fluor 488 | BioLegend | 100529 | 1:200 concentration |
| Rat anti-mouse CD45 allophycocyanin-R700 | BD Biosciences | 565478 | 1:50 concentration |
| Rat anti-mouse IgD PerCP-eFluor 710 | Thermo Fisher Scientific | 46-5993-82 | 1:50 concentration |
| SP8 Confocal microscope | Leica | ||
| Triton X-100 | Sigma | X100-500ml | |
| Trueblack | Biotium | 23007 | |
| Tween-20 | Sigma | P7949-500ml | |
| Ultracomp ebeads | Thermofisher | 01-2222-42 |
Referanslar
- Bar-Or, A., Li, R. Cellular immunology of relapsing multiple sclerosis: interactions, checks, and balances. Lancet Neurol. 20 (6), 470-483 (2021).
- Borst, K., Dumas, A. A., Prinz, M. Microglia: Immune and non-immune functions. Immunity. 54 (10), 2194-2208 (2021).
- Han, R. T., Kim, R. D., Molofsky, A. V., Liddelow, S. A. Astrocyte-immune cell interactions in physiology and pathology. Immunity. 54 (2), 211-224 (2021).
- Absinta, M., et al. A lymphocyte-microglia-astrocyte axis in chronic active multiple sclerosis. Nature. 597 (7878), 709-714 (2021).
- Piwecka, M., Rajewsky, N., Rybak-Wolf, A. Single-cell and spatial transcriptomics: Deciphering brain complexity in health and disease. Nat Rev Neurol. 19 (6), 346-362 (2023).
- Schirmer, L., et al. Neuronal vulnerability and multilineage diversity in multiple sclerosis. Nature. 573 (7772), 75-82 (2019).
- Jain, R. W., Yong, V. W. B cells in central nervous system disease: Diversity, locations and pathophysiology. Nat Rev Immunol. 22 (8), 513-524 (2022).
- Lassmann, H. Multiple sclerosis pathology. Cold Spring Harb Perspect Med. 8 (3), (2018).
- Yong, V. W. Microglia in multiple sclerosis: Protectors turn destroyers. Neuron. 110 (21), 3534-3548 (2022).
- Sharma, S., Boyer, J., Teyton, L. A practitioner's view of spectral flow cytometry. Nat Methods. 21 (5), 740-743 (2024).
- Longo, S. K., Guo, M. G., Ji, A. L., Khavari, P. A. Integrating single-cell and spatial transcriptomics to elucidate intercellular tissue dynamics. Nat Rev Genet. 22 (10), 627-644 (2021).
- Ramaglia, V., et al. Multiplexed imaging of immune cells in staged multiple sclerosis lesions by mass cytometry. Elife. 8, e48051 (2019).
- Bolognesi, M. M., et al. Multiplex staining by sequential immunostaining and antibody removal on routine tissue sections. J Histochem Cytochem. 65 (8), 431-444 (2017).
- Radtke, A. J., et al. IBEX: An iterative immunolabeling and chemical bleaching method for high-content imaging of diverse tissues. Nat Protoc. 17 (2), 378-401 (2022).
- Radtke, A. J., et al. IBEX: A versatile multiplex optical imaging approach for deep phenotyping and spatial analysis of cells in complex tissues. Proc Natl Acad Sci U S A. 117 (52), 33455-33465 (2020).
- Gerner, M. Y., Kastenmuller, W., Ifrim, I., Kabat, J., Germain, R. N. Histo-cytometry: A method for highly multiplex quantitative tissue imaging analysis applied to dendritic cell subset microanatomy in lymph nodes. Immunity. 37 (2), 364-376 (2012).
- Kotov, D. I., Pengo, T., Mitchell, J. S., Gastinger, M. J., Jenkins, M. K. Chrysalis: A new method for high-throughput histo-cytometry analysis of images and movies. J Immunol. 202 (1), 300-308 (2019).
- Jain, R. W., Elliott, D. A., Yong, V. W. Single-cell analysis of high-parameter histology images using histoflow cytometry. J Immunol. 210 (12), 2038-2049 (2023).
- JoVE Science Education Database. General Laboratory Techniques. Histological Sample Preparation for Light Microscopy. JoVE. , (2023).
- Schindelin, J., et al. Fiji: An open-source platform for biological-image analysis. Nat Methods. 9 (7), 676-682 (2012).
- Berg, S., et al. ilastik: Interactive machine learning for (bio)image analysis. Nat Methods. 16 (12), 1226-1232 (2019).
- Reichard, A., Asosingh, K. Best practices for preparing a single cell suspension from solid tissues for flow cytometry. Cytometry A. 95 (2), 219-226 (2019).
- Ruhlandt, D., et al. Absolute quantum yield measurements of fluorescent proteins using a plasmonic nanocavity. Commun Biol. 3 (1), 627 (2020).
- Combs, C. A., Shroff, H. Fluorescence microscopy: A concise guide to current imaging methods. Curr Protoc Neurosci. 79, 1-25 (2017).
- Elliott, A. D. Confocal microscopy: Principles and modern practices. Curr Protoc Cytom. 92 (1), e68 (2020).
- Jonkman, J., Brown, C. M., Wright, G. D., Anderson, K. I., North, A. J. Tutorial: Guidance for quantitative confocal microscopy. Nat Protoc. 15 (5), 1585-1611 (2020).
- McNally, J. G., Karpova, T., Cooper, J., Conchello, J. A. Three-dimensional imaging by deconvolution microscopy. Methods. 19 (3), 373-385 (1999).
- Stringer, C., Wang, T., Michaelos, M., Pachitariu, M. Cellpose: A generalist algorithm for cellular segmentation. Nat Methods. 18 (1), 100-106 (2021).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiThis article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır