需要订阅 JoVE 才能查看此. 登录或开始免费试用。
Method Article
使用 Histoflow 流式细胞术生成和分析高参数组织学图像
摘要
这里描述的是一种可用于通过免疫荧光显微镜对五个或更多荧光参数进行成像的方法。概述了从这些图像中提取单细胞并通过类似流式细胞术的门控策略进行单细胞分析的分析管道,该管道可以识别组织切片中的细胞亚群。
摘要
使用组织学来研究组织切片(例如来自中枢神经系统 (CNS) 的组织切片)中的免疫细胞多样性受到一次可以成像的荧光参数数量的严重限制。大多数免疫细胞亚群是使用流式细胞术通过使用蛋白质标志物的复杂组合来定义的,通常需要四个或更多参数才能最终识别,这超出了大多数传统显微镜的能力。由于流式细胞术会解离组织并丢失空间信息,因此需要能够在询问复杂细胞类型的作用的同时保留空间信息的技术。这些问题在这里通过创建一种方法来解决,该方法通过收集光谱重叠荧光团的信号并使用光谱解混来分离每个单独荧光团的信号来扩展可以成像的荧光参数的数量。然后使用分析管道处理这些图像,以拍摄高参数组织学图像并从这些图像中提取单细胞,以便可以在单细胞水平上分析每个细胞的独特荧光特性。然后,使用类似流式细胞术的设门策略,可以将细胞分析成亚群并定位回组织学切片,不仅可以量化它们的丰度,还可以确定它们如何与组织环境相互作用。总体而言,证明了使用组织流式细胞术在组织学切片中研究复杂免疫群体的简单性和潜力。
引言
由免疫系统细胞和神经胶质细胞驱动的炎症会导致 CNS 的慢性疾病,其中每个群体都可以促进其他群体的活动 1,2,3。了解免疫系统如何与 CNS 的这些元素相互作用以促进 CNS 炎症是目前感兴趣的主要话题,并且单细胞 RNA 测序等高参数技术极大地促进了这一主题。通过单细胞 RNA 测序,我们发现在几种 CNS 疾病中,神经胶质细胞和免疫系统之间存在广泛的通信 4,5,6。了解这些相互作用如何影响这些疾病对于阐明这些疾病的生物学至关重要。
单细胞测序分析的一个问题是,这些技术需要破坏组织以获得单细胞或细胞核,从而导致空间信息的完全丢失。了解细胞在组织中的位置对于了解细胞在驱动炎症中的作用至关重要。例如,B 细胞等免疫细胞在神经炎症期间可以集中在 CNS 中;然而,它们很少进入 CNS 薄壁组织,而是集中在 CNS 屏障7。鉴于它们的定位,这些细胞不太可能通过与 CNS 薄壁细胞中的神经胶质细胞发生物理相互作用来促进 CNS 炎症,这表明它们可能与神经胶质细胞发生的任何相互作用都会通过分泌因子发生。此外,中枢神经系统疾病中发生的病理通常具有结构 8,9,因此细胞在组织中的定位可以关键地确定它是积极导致疾病还是旁观者。因此,使用空间定向来评估细胞在病理学中的作用是必不可少的。
研究组织中的细胞通常是通过使用免疫组织化学或免疫荧光显微镜来完成的。这些技术的一个问题是,它们通常只能同时对最多四个参数进行成像。这是这些技术的主要限制,因为我们从流式细胞术和单细胞 RNA 测序分析中了解到,许多细胞群需要两个或多个参数进行鉴定;此外,在查找 Cell Type10 的特定子集时,所需的参数数量通常会增加。因此,使用标准成像技术来研究细胞亚群如何在组织内相互作用是不切实际的。
通过可以保留空间信息的较新的高参数方法,例如空间 RNA 测序11 和成像质谱流式细胞术12,已经部分解决了这个问题。虽然这些技术很有价值,但它们确实存在一些问题,例如没有广泛使用、将 3D 数据简化为二维以及需要相当多的专业知识来执行。另一种称为顺序染色的技术,其中组织用一组抗体染色,然后灭活前一组抗体,然后再用另一组抗体染色,无需专用设备或专业知识即可实现高参数组织学 13,14,15.然而,顺序染色可能非常耗费人力,并且需要大量的显微镜检查时间,这对于没有个人显微镜的实验室来说可能不切实际。因此,需要能够扩大荧光参数数量的技术,这些参数可以在广泛可用且及时的显微镜上一次成像。
一旦采集了高参数数据,另一个问题就出现了:传统的图像分析方法不太可能成功分析数据。手动计数或阈值等技术仅在分析由单个参数组成或多个标记具有相同的定位(仅计数重叠信号)时才可行。此限制使传统分析不足以处理高参数数据集。通过从组织学图像中分割单个细胞,然后进行类似流式细胞术的门控策略来识别细胞类型,可以成功分析这些数据集16,17。然而,影响这些分析的另一个问题是,它们仅适用于所有感兴趣的细胞都彼此物理分离的数据集,因为这些技术没有采用可以准确分离物理接触细胞的方法。因此,需要一种更新的方法,即使细胞处于物理接触状态,也可以对组织学切片进行单细胞分析。
在本文中,描述了一种称为组织流式细胞术的简单方案,该方案之前已经介绍过18 ,它扩展了可以使用广泛使用的显微镜同时成像的荧光参数的数量。该方案的工作原理是用光谱重叠染料对组织进行染色,然后使用光谱补偿去除重叠通道的渗出,以获得清晰的单一染色。为了便于分析高参数组织学图像,描述了一个详细的分析管道,该管道从组织切片中提取单个细胞,以便使用类似流式细胞术的门控策略将细胞分选为不同的群体。该方案适用于细胞弥散存在的组织和细胞紧密压实在一起的组织,使该技术可用于研究稳态和神经炎症中的 CNS 等组织。因此,组织流式细胞术是研究复杂细胞类型之间相互作用的有用技术,这些细胞类型需要多个细胞标志物来定义细胞,同时保持空间信息。
研究方案
该方案不包括用于组织学的组织切片;请参阅 Jain 等人 18 或19 有关如何为组织学切片的描述。该方案可用于载玻片上的任何切片组织。本文使用从免疫动物中分离的腹股沟淋巴结,如前所述18。该协议的程序和时间表总结在 图 1 中。本研究中使用的试剂和设备的详细信息列在 材料表中。
1. 组织切片染色
注:除了用研究人员感兴趣的所有抗体标记对组织进行染色外,研究人员还应在相邻或其他相同的组织载玻片上准备单色对照(每个要使用的荧光参数一个),其中每个单色对照都用产生一个荧光参数信号所需的试剂进行染色。
- 准备 Perm 和封闭缓冲液以及 Perm 和染色缓冲液(组合物见 表 1 )。
- 在室温下解冻载玻片上的组织切片,然后用抹布擦去冷凝水。使用疏水记号笔勾勒出需要染色的组织边界,然后让标记干燥。
- 通过将组织切片浸泡在磷酸盐缓冲盐水 (PBS) 中 1 分钟来重新水化组织。甩掉 PBS 并使用湿巾去除多余的 PBS,而无需直接接触组织。
- 通过添加足够的 4% 多聚甲醛 (PFA) 以完全覆盖组织来固定组织。在室温下孵育 20 分钟。
注意:PFA 可能有毒,应在通风橱内处理。
注意:此步骤仅适用于新鲜的冷冻组织。如果组织已经固定,则忽略此步骤。这里描述了 PFA 固定,但可以根据实验的需要使用其他方法固定组织。 - 在 PBS 浴中轻轻摇动洗涤纸巾 1 分钟,然后从浴中取出载玻片。更换 PBS 并再重复洗涤 3 次。
- 甩掉多余的 PBS,然后用湿巾去除多余的 PBS,不要直接接触组织。
- 确定方案的步骤 1.12 中需要使用哪些二抗以及来源二抗的动物种类。通过将这些动物的血清添加到 Perm 中,并将每种物种的封闭缓冲液终浓度为 5%,强化 Perm 和封闭缓冲液。
注:使用二抗有助于放大信号强度。因此,建议将产生弱信号的一抗与二抗配对以放大其信号。如果所有被染色的抗体靶标都足够明亮,不需要二抗,则可以跳过步骤 1.7-1.12 和步骤 1.15。 - 通过添加足够的强化 Perm 和封闭缓冲液来覆盖每个组织,从而阻断抗体的非特异性结合。将载玻片在室温下在避光的加湿室中孵育 1-8 小时。
- 将未偶联的一抗以实验确定的稀释度添加到 Perm 和染色缓冲液中(一抗的物种或免疫球蛋白同种型都应该不同,并且可以通过研究人员打算在步骤 1.12 中使用的二抗来区分)。
- 倾倒 Perm 和封闭缓冲液,并使用抹布去除多余的缓冲液,而无需直接接触组织。向组织中加入足够的 Perm 和带有一抗的染色缓冲液以覆盖它,并将载玻片在 4 °C 下孵育 12-48 小时。
注意:研究人员需要确定抗体孵育的最佳时间。 - 准备封闭缓冲液和染色缓冲液(表 1)。
- 将二抗添加到步骤 1.11 中制备的染色缓冲液中,以实验确定的稀释度添加。使用的二抗应靶向步骤 1.9 中使用的相同物种或抗体同种型。
- 准备淬灭缓冲液(如 表 1 所示)。
注意:此步骤有助于减少组织的自发荧光。如果不需要,则可以跳过步骤 1.13 和 1.14。 - 如步骤 1.5-1.6 中所述洗涤组织。然后,加入足够的淬灭缓冲液以覆盖载玻片上的组织,并在室温下孵育载玻片 1-2 分钟。
- 按照步骤 1.5-1.6 中的说明洗涤组织。然后,将步骤 1.12 中制备的染色缓冲液中稀释的二抗添加到组织中,使组织完全被染色缓冲液覆盖。将载玻片在 4 °C 下孵育 2-8 小时。
- 通过将 50 μL 来自步骤 1.18 中将使用的任何抗体种类的正常血清(每种种类的 5%)添加到步骤 1.11 中制备的封闭缓冲液中来制备封闭溶液。
- 如步骤 1.5-1.6 中所述洗涤组织。然后,将步骤 1.16 中制备的含有正常血清的封闭溶液添加到组织中,使其完全被染色缓冲液覆盖。在室温下孵育载玻片 1-8 小时。
- 通过将荧光团偶联的一抗以实验确定的稀释度添加到步骤 1.11 中制备的染色缓冲液中来制备染色溶液。此外,将细胞核染色剂(如细胞核黄)以实验确定的稀释度添加到染色溶液中。
注:仅当研究人员需要使用不需要二抗进行信号放大的额外抗体标记时,才需要步骤 1.18-1.19。如果不使用一抗偶联抗体,可以在步骤 1.12 添加细胞核染色。 - 甩掉封闭缓冲液,用抹布去除多余的缓冲液,不要直接接触组织。将步骤 1.18 中制备的足够染色溶液添加到组织中以覆盖它,并在 4 °C 下孵育 12-48 小时。
- 如步骤 1.5-1.6 中所述洗涤组织。向每张载玻片中加入 5 滴封固剂,不引入气泡,然后轻轻缓慢地将玻璃盖玻片倒在组织上,以将组织完全覆盖在封固剂中并排除所有气泡。将载玻片在 4 °C 下避光储存,直到它们准备好进行成像。
2. 组织切片成像
注:在此阶段,组织切片用所有要成像的条件的所有感兴趣抗体染色,单色对照在每个切片上用一个荧光参数染色。成像应在具有多个检测器的显微镜上进行,这些检测器可以调整以检测特定范围的光,理想情况下,它应该可以使用尽可能多的激光线进行激发。
- 将一张完全染色的载玻片插入显微镜中,并设置显微镜以使用用于收集数据的物镜对切片进行成像。
- 根据组织染色的荧光基团,确定刺激这些荧光基团所需的激发激光线,并确定包含最大信号量的每个荧光基团的发射范围。
注:在线荧光光谱查看工具(例如,参见 材料表)可用于确定荧光团的最佳激发波长和确定荧光团检测的最佳范围。选择用于组织流式细胞术的荧光团时,应使每个荧光团在荧光团受到刺激的最佳激光线中不同,或在发射波长的最佳范围内有所不同。具有更接近激发/发射波长的荧光基团将更难分离。用于本研究的荧光团在结果部分有详细说明。 - 在共聚焦显微镜中打开软件 1(参见 材料表),然后选择顶部的 配置选项卡 。然后选择左侧的 硬件按钮 并将 位深度 更改为 16 位。
注意:建议使用更高的位深度(例如 16 位数据)进行数据收集,因为这将提高频谱补偿的精度。此外,更高的像素密度、更大的线平均值数字以及 z 堆栈中的更多步骤将提高光谱分离的质量。 - 选择屏幕顶部的 Acquire 选项卡 ,然后输入格式设置以包含图像的像素密度(通常,较高的分辨率会产生更好的结果)。
- 如果可用,请选择 双向 X 采集以加快成像时间,前提是此功能已充分校准。
- 为共聚焦显微镜选择一个 针孔值 (通常在 400-600 nm 之间),其中较小的值通常会产生更高质量的组织流式细胞术结果,尽管较小的针孔值会使获取图像变得更加困难,并且可能需要额外的成像时间或更高的激发强度才能产生图像。
- 选择左侧菜单顶部的 Show Sequential Scan Panel(显示顺序扫描面板 )以打开顺序扫描选项。使用 “+” 号设置其他序列,其中每个序列将包含一个在步骤 2.2 中确定的激发波长。
- 通过单击屏幕中央与感兴趣激光器关联的 ON 按钮,打开所有需要的激光器,将它们变为 ON 状态。进入每个特定的序列,将屏幕中心的激光功率增加到所需的任何值,其中每个序列将使用一个激发波长。
- 在每个序列中,根据需要在屏幕底部 打开 任意数量的检测器,以对将在该激光线上成像的所有荧光团进行成像。通过双击检测器的范围并输入需要收集的波长的上限和下限来调整检测器 的范围 。
注意:建议尽可能使用高灵敏度探测器,理想情况下,所有通道都应在相同类型的探测器上收集,尽管这不是必需的。 - 按下 Live 按钮,然后专注于样本。使用软件中图像左侧的 Auto Scale 按钮 来评估通道是否有足够的信号饱和。用户应调整每个序列中的增益值和激光强度,直到自动缩放未达到最大值(16 位数据为 65535),但所有通道都非常接近饱和。
注:由于不同的荧光基团的亮度可能不同,而不同的抗体靶标的丰度可能不同,因此如果两个荧光基团都受到同一激光的刺激,则需要将检测器的灵敏度调整为对明亮信号的低灵敏度和对暗暗信号的高灵敏度(在这种情况下,激光强度不能改变,因为两个荧光基团都会受到影响)。为获得最佳结果,实验中每个通道的荧光信号应保持平衡,以便每个通道具有大致相等的信号量,这将减少光谱渗入其他通道并产生更强的信号。由于不同的样品在被染色的目标标记物的丰度上可能有所不同,因此建议在多个实验条件下测试显微镜设置,以确认在任何实验条件下都不会发生漂白。 - 一旦显微镜设置完成,用单色对照玻片加载显微镜,并找到每个荧光团的代表性染色。通过聚焦在样本下方并点击 z 堆栈菜单中的 开始 按钮,然后聚焦在样本上方并按下 结束 按钮来设置 Z 堆栈。
- 选择步骤数和步骤之间的距离(并使用将用于对完全染色的玻片进行成像的相同步骤数和参数,例如像素密度和位深度),然后按 开始按钮。
注意:理想情况下,这些图像应包括载玻片上可以找到的来自真实染色的最亮信号。 - 对单色对照进行成像后,继续使用相同的显微镜设置对所有完全染色的样品进行成像。
3. 荧光补偿
- 在软件 1 中打开 .lif 文件,然后单击 process 选项卡。
- 选择 Dye Separation 模块 ,然后选择 Automatic Dye Separation 选项。为 Dye Separation (染料分离) 选择 Manual method (手动 方法),然后选择 no rescaling (不缩放)。
- 打开其中一个单色控件图像。通过观察每个通道的灰度窗口,手动检查从一个通道到下一个通道的频谱溢出的单色控件,以寻找信号在不适当通道中的证据。如果需要,请使用 Auto Scale 按钮查看弱渗透。
- 通过在屏幕上的矩阵中手动输入数字(通常为 0-1,其中 0 表示无渗透,1 表示 100% 渗透)来开始删除渗透,然后按 应用 以测试是否充分去除了渗透。应重复此操作,直到除目标通道之外的所有其他通道中的信号都降低到背景电平。
注意:过度补偿会导致结果不准确,因此必须将荧光降低到背景水平,但不能低于背景水平。过补偿数据将显示为一个通道具有信号,而其他通道在目标信号所在的位置似乎有黑洞(图 2)。 - 完成其中一个单色控件后,在矩阵中记录值,然后 重置 矩阵,并移动到下一个单色控件。对每个单个颜色控件重复此过程。
注意:第一种染料将位于染料 1 行中,需要从染料 1 中去除的荧光量将位于相邻列中。 - 将从每个对照获得的所有值组装到一个基质中,并将该基质 应用于 完全染色的样品。如果分色看起来准确,请将设置应用于实验中的所有图像,否则,请重新评估单色控件。
注意:有效的补偿应消除通道中的大部分渗漏,如图 3 所示。 - 将所有补偿文件保存到单独的 .lif 文件中。
4. 使用 ilastik 识别细胞核和细胞
- 在 FIJI20 中打开补偿的 .lif 文件。 启用查看 hyperstack 中的堆栈并选择图像。
- (可选)使用选择工具(如矩形工具)包括将要分析的图像部分,并排除将不分析的区域。使用 image > crop 函数裁剪图像。
注意:减小图像的大小在后续步骤中非常有用,可以减少随机存取内存 (RAM) 消耗并提高处理速度。 - 将图像另存为 .tif 文件。此外,使用适用于 FIJI 的 ilastik 插件将图像另存为 hdf5 文件,方法是单击 ilastik >导出 hdf5 >插件。将文件另存为 “name of choice”.h5,压缩率为 0。
- 打开 Ilastik21 并制作像素分类项目。使用 Add New > Add Separate images 功能,通过添加新的输入数据来添加至少 3 个代表性图像。
- 右键单击其中一个图像,选择 Edit properties,然后将 Display Mode 更改为 greyscale 。
- 在屏幕左侧,选择 Feature selection 选项卡。单击选择特征并选择程序允许您使用的最高 sigma 值之前的所有特征,然后按 OK。
- 现在选择 Training 选项卡。使用画笔工具(选择 标签 1 时)突出显示感兴趣的单个细胞(图 4),以便内部(细胞质和细胞核)和细胞膜包含在突出显示中。
- 在所有选择了所有感兴趣细胞类型的训练样本中重复此操作,并且应从每个样本中选择组织中不同强度、形态和定位的多个细胞。
注:根据为一组染色剂选择的标记物,目标细胞可能存在也可能不存在细胞内染色或细胞膜染色。无论使用什么标记物,无论是否使用细胞内染色剂,都要确保高光包括目标细胞上最外层的染色剂,并包括细胞内部。 - 接下来,使用 标签 2 突出显示不是感兴趣单元格的所有内容(图 4)。这可能包括空白、未标记的细胞核或没有感兴趣标志物的组织。确保包括紧邻感兴趣单元格的空间,以促进对确切单元格边界的学习。
- 使用 Live Update 函数评估感兴趣图像的训练质量。如果训练不足,请继续执行步骤 4.7 到 4.9 中的训练。否则,如果满意,请单击 Prediction Export 选项卡。
- 将源设置为 probability。单击 Choose Export Image Settings 按钮,然后将 c 参数设置为 export 0-1(假设标签 1 突出显示感兴趣的参数)。
- 将数据类型转换为 16 位 Integer,并作为 hdf5 文件导出到具有感兴趣名称的感兴趣目录(我们建议使用 {nickname} mask.h5),然后按 OK。
- 单击 Batch Processing 选项卡。按 Select Raw Data files 按钮,选择所有需要处理的感兴趣的 .h5 文件,然后按 Open.单击 Process all files(处理所有文件)。
- 重复步骤 4.4 到 4.13,除了现在不是识别细胞,而是识别细胞核并排除其他所有内容(图 4)。将其导出为用于识别细胞核的单独预测。
- 打开 Ilastik 并创建一个新的 Data Conversion 项目。将具有代表性的 hdf5 文件添加到 Input data 选项卡中 添加新> 添加单独图像 功能。
- 点击 数据导出 选项卡,然后选择 选择导出图像设置 按钮,并将源设置为 input。将格式更改为多页 tiff,使用 select 功能选择保存位置,并将文件名设置为 {nickname}。按 OK。
- 点击 批处理 选项卡并使用 选择输入数据文件 按钮,然后按 处理所有文件 按钮。
- 打开软件 2(请参阅 材料表)并使用 Add Files(添加文件 )按钮添加在步骤 4.3 和 4.17 中生成的所有 tif/tiff 文件。选择文件输出的位置,然后按 全部启动(Start All ) 按钮将所有文件转换为 .ims 文件。这些文件现在可以在软件 3 中进行分析了(参见 材料表)。
5. 软件分析 3
- 打开包含所有荧光染色剂的图像之一。选择 Edit-> Add Channels,然后添加与在细胞核和细胞体上制作的遮罩相对应的 .ims 文件。对所有样品重复此操作。
- 选择 Add new Cells 按钮。选择 detect nucleus and cell 选项并继续。
- 选择细胞 核遮罩 作为检测细胞核的源通道,然后选择 按种子点分割细胞核的高级选项。为 nucleus diameter 选择一个值),然后继续前进。
注意:需要通过运行模拟并手动评估程序是否没有分裂足够的细胞核(多个细胞核组合在一起)或是否存在过度分裂(一个细胞核分裂成多个细胞核)来测试细胞核直径的最佳值。 - 为质量评估选择一个值,然后继续。通常,最好包括所有细胞核,因为缺失细胞核会导致多个细胞融合成一个。
- 选择用于创建核表面的阈值,然后继续。确保所有细胞核(即使是较暗的细胞核)仍包含在阈值中。不要将阈值设置得太低,因为这可能会高估细胞核的大小或可能包括碎片作为细胞核染色。
- 为每个细胞核的最小体素数选择一个值,然后继续。通常,最好保留所有内容,除非可以使用此参数排除碎片或非原子核。
- 选择 Cell boundary and cytoplasm 选项,选择 细胞遮罩 作为检测细胞体的源通道,然后继续。
- 选择可准确捕获所有目标细胞的细胞阈值,但不要太低以至于包括背景或任何非特异性细胞检测。选择选项以拆分接触单元格,以便 每个单元格有一个细胞核,并且它们与 细胞核的距离相隔。此外,选择 扩增细胞核上的细胞 ,然后继续。
- 为每个单元格的最小体素数选择一个值,然后继续。通常,最好保留所有内容,除非可以使用此参数排除碎片或非细胞。
- 通过查看多个像元是否融合在一起或是否缺少像元,来评估在像元上创建的表面是否准确。如果表面不足,请返回步骤 5.2 并尝试新参数,直到确定最佳设置。
- 建立最佳设置后,单击 左侧菜单中创建的单元格对象 ,单击 创建选项卡,然后单击 Store Parameters for Batch 按钮保存使用的设置。
- 打开 Software 3 的多个窗口,并在每个窗口中打开单独的图像。在每个窗口中按新的 Cells 按钮,在 Favorite Creation Parameters 菜单中选择保存的设置,并指示每个窗口运行直到完成。根据需要重复此操作多次,直到分析完所有样品。
注意:运行软件 3 的多个实例将使此分析进行得更快,但可以并行运行的窗口数量将取决于中央处理器 (CPU) 处理速度和运行分析的计算机的 RAM 量。 - 完成所有样本后,单击每个图像左侧菜单中 创建的单元格对象 ,然后单击 统计 选项卡。单击 Configure list of visible Statistics Values 按钮,取消选择除 Cell Intensity Mean(单元格强度平均值)值之外的所有统计数据,然后按 OK(确定)。
- 对于需要分析的每张图像,请单击 Export all statistics to file 按钮,然后将文件保存到某个位置。
6. 组织流式细胞术分析并将群体定位到组织切片上
- 在 Anaconda 中打开 Anaconda 和 JupyterLab 。接下来,打开 Supplementary Coding File 1 中提供的脚本。使用 Run 菜单运行代码,然后选择 Run All Cells,或者通过选择 Run Selected Cells 按顺序单独运行单元格。
- 当提示输入源文件时,输入步骤 5.14 中生成的导出荧光值的文件目录。通过查看其中一个导出的 .csv 文件的属性并将文件路径复制到提示符中,可以获取文件目录。
- 接下来,程序将询问已处理文件的输出位置。输入适合保存已处理文件的任何位置的文件目录。该程序将自动检测文件夹中包含的荧光通道数。
- 运行代码的下一部分,使用每个通道的名称对数据进行注释。可以根据需要修改此代码,以更改 “numberOfChannels” 参数以匹配步骤 6.3 中检测到的通道数。此外,可以根据需要修改任意数量的通道的通道名称,方法是根据需要多次重复 'ChX':'Marker' 行并修改标记的名称。
- 运行下一行代码将在指定位置创建一个 .csv 文件,该文件带有注释,并包含数据集中的所有荧光参数。对每组 Software 3 导出重复步骤 6.1-6.5,为所有数据创建 .csv 文件。
- 通过打开软件 4(请参阅 材料表)并将.csv文件拖动到程序窗口中,将 .csv 文件转换为 .fcs 文件。软件 4 将自动开始将文件转换为 .fcs 文件。
- 双击 Software 4 中的一个 采样 以打开该采样的窗口。使用软件 4 中的门控工具创建门控策略。建议从包含所有目标细胞的谱系定义标记物上开始设门,然后首先去除更稀有亚群的设门,最后在更丰富的细胞类型上完成。
- 建立门控策略后,通过右键单击菜单中的 该群体 并选择 export 来测试门控的准确性。将所有参数导出为包含标题的 .csv 文件。
- 在 Anaconda 中打开 JupyterLab。接下来,打开 Supplementary Coding File 2 中提供的脚本,并运行代码。当系统提示输入 Software 4 文件的位置时,输入上一步生成的导出 .csv 文件的文件目录。通过查看导出的 .csv 文件的属性并将文件路径复制到提示符中,可以获取文件目录。
- 当系统提示添加输出位置时,选择所需的任何文件目录,并确保文件目录的末尾包含文件名 ('file directory'\filename.txt)。运行代码的其余部分。
- 复制 .txt 文件中的文本,并在软件 3 中打开导出数据所基于的相同样品的相应 .ims 文件。单击文件中的 Cell 对象 ,切换到 statistics 选项卡,将文本粘贴到搜索栏中,然后开始搜索。所有感兴趣的单元格都将突出显示。
- 手动检查是否使用设门策略捕获了所有感兴趣的细胞。如果细胞数量被低估或高估( 图 5 中所示的示例),请返回步骤 6.7,调整门,并重新评估是否使用新门准确捕获了细胞。对设门策略中的所有门重复此操作,并评估多个样品的门控。
- 验证所有门后,使用 表格编辑器 功能从软件 4 导出数据,并使用首选分析方法分析数据。
注意:通过按照步骤 6.8-6.11 识别门控群体,然后根据所选细胞创建表面,可以将细胞群定位回软件 3 中的组织切片。这可以针对每个门控群体进行,如图 6 和 图 7 所示。
结果
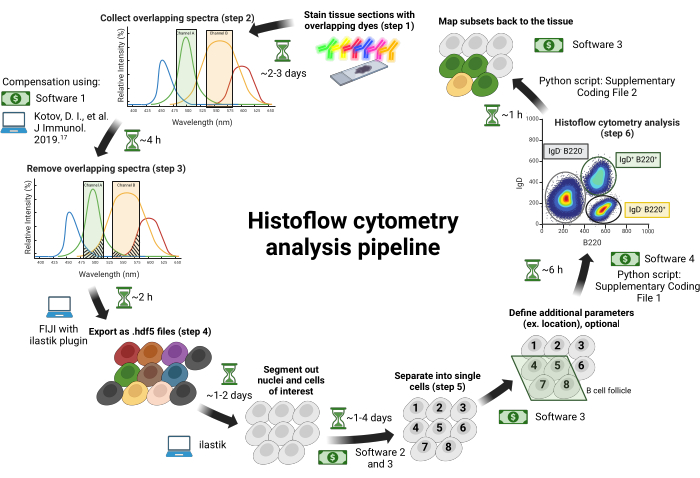
图 1:组织流式细胞术工作流程。 组织切片用光谱重叠染料染色(步骤 1)。使用与可调谐带通滤光片配对的单个激发激光器收集图像,以最大限度地减少荧光团之间的光谱渗漏(步骤 2)。通道之间的光谱渗出根据使用单一颜色染色组织对照生成的补偿矩阵(步骤 3...
讨论
这里描述了组织流式细胞术的使用,这种技术之前已经验证了18.结果表明,当使用光谱重叠染料对组织切片进行染色时,可以使用光谱补偿去除跨通道的渗漏,从而比通常通过传统方法清晰分辨更多的荧光参数。由于使用传统方法难以分析高参数组织学图像,因此提供了一个分析管道来从这些图像中分割单个细胞。通过导出这些单个细胞的荧光特征,证明像在流式细胞术中进行?...
披露声明
作者没有财务利益冲突。
致谢
我们感谢 Hotchkiss 脑研究所高级显微镜平台提供的成像基础设施和专业知识。RWJ 得到了卡尔加里大学 Eyes High 计划的博士后奖学金资助以及加拿大多发性硬化症协会和加拿大罗氏无限制教育奖学金的支持。VWY 获得了加拿大研究主席 Tier 1 计划的薪资支持。这项工作得到了加拿大卫生研究院研究资助 1049959、加拿大多发性硬化症协会资助 3236 和美国国防部国会指导的多发性硬化症研究计划的运营资金的支持。 图 1 是使用 BioRender.com 创建的。本出版物中改编的数字最初发表在 The Journal of Immunology 上。Rajiv W. Jain、David A. Elliott 和 V. Wee Yong。2023. 使用 Histoflow Cytometry 对高参数组织学图像进行单细胞分析。 免疫学杂志。210: 2038-2049.版权所有 © [2023]。美国免疫学家协会
材料
| Name | Company | Catalog Number | Comments |
| 100% Ethanol | Sigma | 676829-1L | |
| 4% PFA | Electron Microscopy Sciences | 157-4 | |
| Anaconda | N/A | N/A | https://www.anaconda.com/download |
| Bovine Serum Albumin | Sigma | A4503-50G | |
| Cold fish stain gelatin | Sigma | G7765 | |
| Collating multichannel data from Imaris.ipynb script | N/A | N/A | https://github.com/elliottcalgary/Histoflow-Cytometry-Analysis- |
| Convert FlowJo output to txt file for Cell selection in Imaris.ipynb script | N/A | N/A | https://github.com/elliottcalgary/Histoflow-Cytometry-Analysis- |
| Donkey anti-rat Alexa Fluor 647 | JacksonImmunoResearch | 712-605-153 | 1:300 concentration |
| Donkey anti-rat DyLight 405 | Jackson ImmunoResearch | 712-475-153 | 1:200 concentration |
| Donkey Serum | JacksonImmunoResearch | 017-000-001 | |
| F(ab')2-Goat anti-Mouse IgG PerCP-eFluor 710 | Thermofisher | 46-4010-82 | 1:25 concentration |
| FIJI | N/A | N/A | https://imagej.net/software/fiji/ |
| FlowJo | FlowJo LLC | Software 4 | |
| Fluorescence spectraviewer | https://www.thermofisher.com/order/fluorescence-spectraviewer/#!/ | ||
| Fluoromount-G | Southern Biotech | 0100-01 | |
| Fresh frozen human tonsil sections | amsbio | HF-707 | |
| Glass coverslip | VWR | 48393 106 | |
| Goat anti-human IgA Alexa Fluor 488 | JacksonImmunoResearch | 109-546-011 | 1:400 concentration |
| Goat anti-human IgG Cy3 | JacksonImmunoResearch | 709-166-098 | 1:400 concentration |
| Goat anti-human IgM Dylight 405 | JacksonImmunoResearch | 109-476-129 | 1:300 concentration |
| Goat anti-rabbit A546 | Thermo Fisher Scientific | A-11035 | 1:250 concentration |
| Goat anti-rabbit IgG PE-Alexa Fluor 610 | Thermofisher | A-20981 | 1:250 concentration |
| Horse Serum | Sigma | H1138 | |
| Ilastik | N/A | N/A | https://www.ilastik.org/ |
| Ilastik FIJI plugin | N/A | N/A | https://www.ilastik.org/documentation/fiji_export/plugin |
| Imaris File Converter | Oxford Instruments | Software 2 | |
| Imaris with cell module | Oxford Instruments | Software 3 | |
| kimwipe | Kimtech | 34155 | |
| LasX Life Science software | Leica | Software 1 | |
| Mouse anti-human CD20 | VWR | CA95024-322 | 1:40 concentration |
| Mouse anti-human CD38 APC-R700 | BD Biosciences | 564980 | 1:20 concentration |
| Normal Goat Serum | JacksonImmunoResearch | 005-000-001 | |
| Normal Mouse Serum | JacksonImmunoResearch | 015-000-001 | |
| Normal Rabbit Serum | JacksonImmunoResearch | 011-000-001 | |
| Normal Rat Serum | JacksonImmunoResearch | 012-000-120 | |
| Nuclear Yellow | Abcam | ab138903 | Dissolve in DMSO at a concentration of 2 mg/ml and store at 4°C in the dark |
| PAP pen | Cedarlane | MU22 | |
| PBS | Gibco | 10010-023 | |
| Rabbit anti-human Ki67 | Abcam | ab15580 | 1:500 concentration |
| Rabbit anti-mouse Iba1 | Wako | 019-19741 | 1:500 concentration |
| Rat anti-human Blimp1 | Thermofisher | 14-5963-82 | 1:40 concentration |
| Rat anti-mouse B220 Alexa Fluor 647 | BioLegend | 103226 | 1:250 concentration |
| Rat anti-mouse CD138 | Biolegend | 142502 | 1:200 concentration |
| Rat anti-mouse CD3 PE-eFluor 610 | Thermo Fisher Scientific | 61-0032-82 | 1:40 concentration |
| Rat anti-mouse CD4 Alexa Fluor 488 | BioLegend | 100529 | 1:200 concentration |
| Rat anti-mouse CD45 allophycocyanin-R700 | BD Biosciences | 565478 | 1:50 concentration |
| Rat anti-mouse IgD PerCP-eFluor 710 | Thermo Fisher Scientific | 46-5993-82 | 1:50 concentration |
| SP8 Confocal microscope | Leica | ||
| Triton X-100 | Sigma | X100-500ml | |
| Trueblack | Biotium | 23007 | |
| Tween-20 | Sigma | P7949-500ml | |
| Ultracomp ebeads | Thermofisher | 01-2222-42 |
参考文献
- Bar-Or, A., Li, R. Cellular immunology of relapsing multiple sclerosis: interactions, checks, and balances. Lancet Neurol. 20 (6), 470-483 (2021).
- Borst, K., Dumas, A. A., Prinz, M. Microglia: Immune and non-immune functions. Immunity. 54 (10), 2194-2208 (2021).
- Han, R. T., Kim, R. D., Molofsky, A. V., Liddelow, S. A. Astrocyte-immune cell interactions in physiology and pathology. Immunity. 54 (2), 211-224 (2021).
- Absinta, M., et al. A lymphocyte-microglia-astrocyte axis in chronic active multiple sclerosis. Nature. 597 (7878), 709-714 (2021).
- Piwecka, M., Rajewsky, N., Rybak-Wolf, A. Single-cell and spatial transcriptomics: Deciphering brain complexity in health and disease. Nat Rev Neurol. 19 (6), 346-362 (2023).
- Schirmer, L., et al. Neuronal vulnerability and multilineage diversity in multiple sclerosis. Nature. 573 (7772), 75-82 (2019).
- Jain, R. W., Yong, V. W. B cells in central nervous system disease: Diversity, locations and pathophysiology. Nat Rev Immunol. 22 (8), 513-524 (2022).
- Lassmann, H. Multiple sclerosis pathology. Cold Spring Harb Perspect Med. 8 (3), (2018).
- Yong, V. W. Microglia in multiple sclerosis: Protectors turn destroyers. Neuron. 110 (21), 3534-3548 (2022).
- Sharma, S., Boyer, J., Teyton, L. A practitioner's view of spectral flow cytometry. Nat Methods. 21 (5), 740-743 (2024).
- Longo, S. K., Guo, M. G., Ji, A. L., Khavari, P. A. Integrating single-cell and spatial transcriptomics to elucidate intercellular tissue dynamics. Nat Rev Genet. 22 (10), 627-644 (2021).
- Ramaglia, V., et al. Multiplexed imaging of immune cells in staged multiple sclerosis lesions by mass cytometry. Elife. 8, e48051 (2019).
- Bolognesi, M. M., et al. Multiplex staining by sequential immunostaining and antibody removal on routine tissue sections. J Histochem Cytochem. 65 (8), 431-444 (2017).
- Radtke, A. J., et al. IBEX: An iterative immunolabeling and chemical bleaching method for high-content imaging of diverse tissues. Nat Protoc. 17 (2), 378-401 (2022).
- Radtke, A. J., et al. IBEX: A versatile multiplex optical imaging approach for deep phenotyping and spatial analysis of cells in complex tissues. Proc Natl Acad Sci U S A. 117 (52), 33455-33465 (2020).
- Gerner, M. Y., Kastenmuller, W., Ifrim, I., Kabat, J., Germain, R. N. Histo-cytometry: A method for highly multiplex quantitative tissue imaging analysis applied to dendritic cell subset microanatomy in lymph nodes. Immunity. 37 (2), 364-376 (2012).
- Kotov, D. I., Pengo, T., Mitchell, J. S., Gastinger, M. J., Jenkins, M. K. Chrysalis: A new method for high-throughput histo-cytometry analysis of images and movies. J Immunol. 202 (1), 300-308 (2019).
- Jain, R. W., Elliott, D. A., Yong, V. W. Single-cell analysis of high-parameter histology images using histoflow cytometry. J Immunol. 210 (12), 2038-2049 (2023).
- JoVE Science Education Database. General Laboratory Techniques. Histological Sample Preparation for Light Microscopy. JoVE. , (2023).
- Schindelin, J., et al. Fiji: An open-source platform for biological-image analysis. Nat Methods. 9 (7), 676-682 (2012).
- Berg, S., et al. ilastik: Interactive machine learning for (bio)image analysis. Nat Methods. 16 (12), 1226-1232 (2019).
- Reichard, A., Asosingh, K. Best practices for preparing a single cell suspension from solid tissues for flow cytometry. Cytometry A. 95 (2), 219-226 (2019).
- Ruhlandt, D., et al. Absolute quantum yield measurements of fluorescent proteins using a plasmonic nanocavity. Commun Biol. 3 (1), 627 (2020).
- Combs, C. A., Shroff, H. Fluorescence microscopy: A concise guide to current imaging methods. Curr Protoc Neurosci. 79, 1-25 (2017).
- Elliott, A. D. Confocal microscopy: Principles and modern practices. Curr Protoc Cytom. 92 (1), e68 (2020).
- Jonkman, J., Brown, C. M., Wright, G. D., Anderson, K. I., North, A. J. Tutorial: Guidance for quantitative confocal microscopy. Nat Protoc. 15 (5), 1585-1611 (2020).
- McNally, J. G., Karpova, T., Cooper, J., Conchello, J. A. Three-dimensional imaging by deconvolution microscopy. Methods. 19 (3), 373-385 (1999).
- Stringer, C., Wang, T., Michaelos, M., Pachitariu, M. Cellpose: A generalist algorithm for cellular segmentation. Nat Methods. 18 (1), 100-106 (2021).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。