É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Gerando e analisando imagens histológicas de alto parâmetro com citometria de histofluxo
Neste Artigo
Resumo
Aqui está descrito um método que pode ser usado para obter imagens de cinco ou mais parâmetros fluorescentes por microscopia imunofluorescente. Um pipeline de análise para extrair células únicas dessas imagens e conduzir análises de células únicas por meio de estratégias de gating semelhantes à citometria de fluxo é delineado, o que pode identificar subconjuntos de células em seções de tecido.
Resumo
O uso da histologia para investigar a diversidade de células imunes em seções de tecido, como as derivadas do sistema nervoso central (SNC), é criticamente limitado pelo número de parâmetros fluorescentes que podem ser visualizados de uma só vez. A maioria dos subconjuntos de células imunes foi definida usando citometria de fluxo usando combinações complexas de marcadores de proteínas, muitas vezes exigindo quatro ou mais parâmetros para serem identificados de forma conclusiva, o que está além das capacidades da maioria dos microscópios convencionais. Como a citometria de fluxo dissocia os tecidos e perde informações espaciais, há necessidade de técnicas que possam reter informações espaciais enquanto interrogam os papéis de tipos de células complexas. Essas questões são abordadas aqui criando um método para expandir o número de parâmetros fluorescentes que podem ser visualizados coletando os sinais de fluoróforos espectralmente sobrepostos e usando a separação espectral para separar os sinais de cada fluoróforo individual. Essas imagens são então processadas usando um pipeline de análise para obter imagens histológicas de alto parâmetro e extrair células únicas dessas imagens para que as propriedades fluorescentes exclusivas de cada célula possam ser analisadas em um nível de célula única. Usando estratégias de gating semelhantes à citometria de fluxo, as células podem ser perfiladas em subconjuntos e mapeadas de volta para as seções de histologia para não apenas quantificar sua abundância, mas também estabelecer como elas interagem com o ambiente do tecido. No geral, a simplicidade e o potencial do uso da citometria de histofluxo para estudar populações imunes complexas em seções de histologia são demonstradas.
Introdução
A inflamação impulsionada por células do sistema imunológico e células gliais pode contribuir para distúrbios crônicos do SNC, onde cada população pode promover a atividade da outra 1,2,3. Compreender como o sistema imunológico interage com esses elementos do SNC para promover a inflamação do SNC é atualmente um grande tópico de interesse e tem sido muito facilitado por técnicas de alto parâmetro, como o sequenciamento de RNA de célula única. Por meio do sequenciamento de RNA de célula única, descobrimos que há uma extensa comunicação ocorrendo entre as células gliais e o sistema imunológico em vários distúrbios do SNC 4,5,6. Entender como essas interações estão afetando esses distúrbios será crucial para elucidar a biologia dessas doenças.
Um problema com as análises de sequenciamento de célula única é que essas técnicas exigem que o tecido seja interrompido para obter células ou núcleos únicos, resultando em uma perda completa de informações espaciais. Saber onde uma célula existe em um tecido é fundamental para entender o papel da célula na condução da inflamação. Por exemplo, células imunes, como células B, podem se concentrar no SNC durante a neuroinflamação; no entanto, raramente entram no parênquima do SNC e, em vez disso, concentram-se nas barreiras do SNC7. Dada a sua localização, é improvável que essas células contribuam para a inflamação do SNC interagindo fisicamente com as células gliais no parênquima do SNC, sugerindo que quaisquer interações que possam estar tendo com as células gliais ocorreriam por meio de fatores secretados. Além disso, a patologia que ocorre nos distúrbios do SNC geralmente tem estrutura 8,9 de modo que a localização de uma célula no tecido pode determinar criticamente se ela está contribuindo ativamente para o distúrbio ou se é um espectador. Assim, o uso da orientação espacial para avaliar o papel de uma célula na patologia é essencial.
O estudo de células no tecido geralmente é realizado usando imuno-histoquímica ou microscopia de imunofluorescência. Um problema com essas técnicas é que elas normalmente só podem gerar imagens de até quatro parâmetros simultaneamente. Esta é uma grande limitação para essas técnicas, pois sabemos por citometria de fluxo e análises de sequenciamento de RNA de célula única que muitas populações de células requerem dois ou mais parâmetros para sua identificação; Além disso, o número de parâmetros necessários normalmente aumenta ao procurar subconjuntos específicos de um tipo de célula10. Assim, é impraticável usar técnicas de imagem padrão para estudar como subconjuntos de células podem estar interagindo dentro de um tecido.
Esse problema foi parcialmente superado por meio de métodos mais recentes de alto parâmetro que podem reter informações espaciais, como sequenciamento espacial de RNA11 e citometria de massa de imagem12. Embora essas técnicas sejam valiosas, elas têm vários problemas, como não estarem amplamente disponíveis, reduzir os dados tridimensionais em duas dimensões e exigir uma experiência considerável para serem executadas. Outra técnica conhecida como coloração sequencial, em que os tecidos são corados com um conjunto de anticorpos seguido pela inativação do conjunto anterior de anticorpos antes da coloração com outro conjunto de anticorpos, pode atingir histologia de alto parâmetro sem a necessidade de equipamento especializado ou experiência 13,14,15. No entanto, a coloração sequencial pode ser extremamente trabalhosa e requer uma grande quantidade de tempo de microscopia, o que pode ser impraticável para laboratórios que não possuem um microscópio pessoal. Assim, há uma necessidade de técnicas que possam expandir o número de parâmetros fluorescentes que podem ser visualizados de uma só vez em microscópios amplamente disponíveis e em tempo hábil.
Uma vez que os dados de alto parâmetro tenham sido adquiridos, surge outro problema: é improvável que os métodos convencionais de análise de imagem analisem os dados com sucesso. Técnicas como contagem manual ou limiar só são viáveis se a análise consistir em um único parâmetro ou se vários marcadores tiverem a mesma localização onde apenas sinais sobrepostos são contados. Essa limitação torna a análise tradicional inadequada para trabalhar com conjuntos de dados de alto parâmetro. A análise bem-sucedida desses conjuntos de dados foi alcançada segmentando células únicas de imagens histológicas e, em seguida, conduzindo estratégias de bloqueio semelhantes à citometria de fluxo para identificar tipos de células16,17. No entanto, outra questão que afeta essas análises é que elas funcionam apenas para conjuntos de dados em que todas as células de interesse estão fisicamente separadas umas das outras, pois essas técnicas não empregam métodos que possam separar com precisão as células que estão em contato físico. Assim, é necessário um método mais recente que possa realizar análises de células únicas em seções histológicas, mesmo que as células estejam em contato físico.
Neste artigo, é descrito um protocolo simples chamado citometria de histofluxo, que foi introduzido anteriormente18 , que expande o número de parâmetros fluorescentes que podem ser visualizados simultaneamente usando microscópios amplamente disponíveis. Este protocolo funciona colorindo tecidos com corantes espectralmente sobrepostos e, em seguida, usando compensação espectral para remover o sangramento de canais sobrepostos para obter manchas únicas claras. Para facilitar a análise de imagens histológicas de alto parâmetro, é descrito um pipeline de análise detalhado que extrai células únicas de seções de tecido com o objetivo de classificar células em populações distintas usando estratégias de gating semelhantes à citometria de fluxo. Este protocolo funciona em tecidos onde as células estão difusamente presentes e em tecidos onde as células estão compactadas, tornando esta técnica versátil para o estudo de tecidos como o SNC tanto na homeostase quanto na neuroinflamação. A citometria de histofluxo é, portanto, uma técnica útil para estudar interações entre tipos de células complexas que requerem vários marcadores celulares para definir as células, mantendo as informações espaciais.
Protocolo
Este protocolo não cobre a secção de tecidos para histologia; consulte Jain et al.18 ou19 para obter descrições de como seccionar tecidos para histologia. Este protocolo pode ser usado com qualquer tecido seccionado em lâminas de vidro. Este artigo utiliza linfonodos inguinais isolados de um animal imunizado, conforme descrito anteriormente18. O procedimento e o cronograma para este protocolo estão resumidos na Figura 1. Os detalhes dos reagentes e dos equipamentos utilizados neste estudo estão listados na Tabela de Materiais.
1. Coloração de seções de tecido
NOTA: Além de corar os tecidos com todos os rótulos de anticorpos nos quais o pesquisador está interessado, o pesquisador também deve preparar controles de cor única (um para cada parâmetro fluorescente que se destina a ser usado) em lâminas de tecido adjacentes ou idênticas, em que cada controle de cor única é corado com os reagentes necessários para produzir o sinal de um parâmetro fluorescente.
- Prepare Perm e tampão de bloqueio e Perm e tampão de coloração (consulte a Tabela 1 para a composição).
- Descongele as seções de tecido em lâminas de vidro em temperatura ambiente e limpe a condensação com um pano. Use um marcador hidrofóbico para delinear a borda do(s) tecido(s) que precisa(m) ser corado(s) e deixe as marcas secarem.
- Reidrate os tecidos embebendo as seções de tecido em solução salina tamponada com fosfato (PBS) por 1 min. Sacuda o PBS e use um lenço para remover o excesso de PBS sem tocar diretamente no tecido.
- Fixe os tecidos adicionando paraformaldeído (PFA) a 4% suficiente para cobrir completamente o tecido. Incubar à temperatura ambiente durante 20 min.
CUIDADO: O PFA pode ser tóxico e deve ser manuseado dentro de uma capela de exaustão.
NOTA: Esta etapa é necessária apenas para tecido fresco congelado. Se o tecido já estiver fixado, ignore esta etapa. A fixação do PFA é descrita aqui, mas o tecido pode ser fixado usando outros métodos, dependendo das necessidades do experimento. - Lave os lenços em um banho PBS por 1 min com um balanço suave e, em seguida, remova a lâmina do banho. Substitua o PBS e repita a lavagem mais 3 vezes.
- Sacuda o excesso de PBS e use um lenço para remover o excesso de PBS sem tocar diretamente no tecido.
- Determinar quais os anticorpos secundários que devem ser utilizados na etapa 1.12 do protocolo e as espécies de animais das quais derivam os anticorpos secundários. Fortifique o Perm e o tampão de bloqueio adicionando soro desses animais ao Perm e bloqueando o tampão até uma concentração final de 5% para cada espécie.
NOTA: O uso de anticorpos secundários ajuda a amplificar a intensidade do sinal. Assim, recomenda-se que os anticorpos primários que produzem sinais fracos sejam emparelhados com anticorpos secundários para amplificar seus sinais. Se todos os alvos de anticorpos que estão sendo corados forem brilhantes o suficiente para não exigir anticorpos secundários, as etapas 1.7-1.12 e 1.15 podem ser ignoradas. - Bloqueie a ligação não específica de anticorpos adicionando Perm fortificado suficiente e tampão de bloqueio para cobrir cada tecido. Incube as lâminas por 1-8 h em temperatura ambiente em uma câmara umidificada protegida da luz.
- Adicione anticorpos primários não conjugados ao Perm e ao tampão de coloração em diluições determinadas experimentalmente (as espécies ou isotipos de imunoglobulina dos anticorpos primários devem ser diferentes e distinguíveis pelos anticorpos secundários que o pesquisador pretende usar na etapa 1.12).
- Despeje o Perm e o tampão de bloqueio e use um lenço para remover o excesso de tampão sem tocar diretamente no tecido. Adicione Perm e tampão de coloração suficientes com anticorpos primários ao tecido para cobri-lo e incube a lâmina a 4 °C por 12-48 h.
NOTA: O pesquisador precisará determinar o momento ideal para a incubação de anticorpos. - Prepare o tampão de bloqueio e o tampão de coloração (Tabela 1).
- Adicionar anticorpos secundários ao tampão de coloração preparado na etapa 1.11 em diluições determinadas experimentalmente. Os anticorpos secundários utilizados devem ter como alvo as mesmas espécies ou isotipos de anticorpos que foram utilizados no passo 1.9.
- Prepare o tampão de têmpera (como na Tabela 1).
NOTA: Esta etapa ajuda a reduzir a autofluorescência do tecido. Se isso não for necessário, as etapas 1.13 e 1.14 podem ser ignoradas. - Lave os lenços conforme descrito nas etapas 1.5-1.6. Em seguida, adicione tampão de têmpera suficiente para cobrir os tecidos nas lâminas e incube as lâminas em temperatura ambiente por 1-2 min.
- Lave o lenço conforme descrito nas etapas 1.5-1.6. Em seguida, adicione os anticorpos secundários diluídos no tampão de coloração preparado na etapa 1.12 aos tecidos, de modo que os tecidos fiquem completamente cobertos pelo tampão de coloração. Incubar as lâminas a 4 °C durante 2-8 h.
- Preparar a solução de bloqueio adicionando 50 μL de soro normal de qualquer espécie de anticorpos que será utilizada no passo 1.18 (5% de cada espécie) ao tampão de bloqueio preparado no passo 1.11.
- Lave os lenços conforme descrito nas etapas 1.5-1.6. Em seguida, adicione a solução de bloqueio com soro normal preparado na etapa 1.16 aos tecidos para que fiquem completamente cobertos com tampão de coloração. Incube as lâminas em temperatura ambiente por 1-8 h.
- Preparar a solução de coloração adicionando anticorpos primários conjugados com fluoróforo em diluições determinadas experimentalmente ao tampão de coloração preparado no passo 1.11. Além disso, adicione uma coloração nuclear, como amarelo nuclear, à solução de coloração em uma diluição determinada experimentalmente.
NOTA: As etapas 1.18-1.19 só são necessárias se o pesquisador precisar usar rótulos de anticorpos adicionais que não exijam anticorpos secundários para amplificação de sinal. Se não estiver usando anticorpos conjugados primários, as colorações de núcleos podem ser adicionadas na etapa 1.12. - Sacuda o tampão de bloqueio e use um lenço para remover o excesso de tampão sem tocar diretamente no tecido. Adicione ao tecido uma solução de coloração suficiente preparada na etapa 1.18 para cobri-lo e incube a 4 °C por 12-48 h.
- Lave os lenços conforme descrito nas etapas 1.5-1.6. Adicione 5 gotas de mountant a cada lâmina sem introduzir bolhas e, suave e lentamente, incline uma lamínula de vidro sobre o tecido para cobrir o tecido completamente com mountant e excluir todas as bolhas de ar. Armazene os slides a 4 ° C no escuro até que estejam prontos para serem fotografados.
2. Imagem das seções de tecido
NOTA: Nesta fase, as seções de tecido são coradas com todos os anticorpos de interesse para todas as condições que se destinam a ser visualizadas, e os controles de cor única são corados com um parâmetro fluorescente em cada seção. A imagem deve ser feita em um microscópio que tenha vários detectores que podem ser ajustados para detectar faixas específicas de luz e, idealmente, deve ter acesso ao maior número possível de linhas de laser para excitação.
- Insira uma das lâminas totalmente coradas no microscópio e configure o microscópio para obter imagens da seção usando a objetiva que se destina a ser usada para coletar dados.
- Com base nos fluoróforos com os quais o tecido é corado, identifique as linhas de laser de excitação necessárias para estimular esses fluoróforos e identifique as faixas de emissão para cada fluoróforo que contém a maior quantidade de sinal.
NOTA: As ferramentas de visualização de espectros de fluorescência on-line (por exemplo, consulte a Tabela de Materiais) podem ser úteis para identificar os comprimentos de onda de excitação ideais dos fluoróforos e identificar as faixas ideais para a detecção de fluoróforos. Os fluoróforos usados para citometria de histofluxo devem ser escolhidos de forma que cada fluoróforo difira na linha de laser ideal pela qual o fluoróforo é estimulado ou difere pela faixa ideal de comprimentos de onda de emissão. Os fluoróforos que têm comprimentos de onda de excitação/emissão mais próximos serão mais difíceis de separar. Os fluoróforos usados para este estudo são detalhados na seção de resultados. - Abra o Software 1 (consulte a Tabela de Materiais) em um microscópio confocal e selecione a guia de configuração na parte superior. Em seguida, selecione o botão de hardware no lado esquerdo e altere a profundidade de bits para 16 bits.
NOTA: Recomenda-se usar profundidades de bits mais altas, como dados de 16 bits, para coleta de dados, pois isso melhorará a precisão da compensação espectral. Além disso, densidades de pixels mais altas, números maiores para médias de linha e mais etapas na pilha z melhorarão a qualidade da separação espectral. - Selecione a guia Adquirir na parte superior da tela e insira a configuração de formato para incluir a densidade de pixels da imagem (normalmente, resoluções mais altas produzem melhores resultados).
- Se disponível, selecione a aquisição X bidirecional para acelerar o tempo de imagem, desde que esse recurso tenha sido calibrado adequadamente.
- Escolha um valor de orifício para o microscópio confocal (normalmente entre 400-600 nm) onde valores menores normalmente produzirão resultados de citometria de histofluxo de maior qualidade, embora valores de orifício menores dificultem a aquisição de imagens e possam exigir tempo de imagem adicional ou intensidades de excitação mais altas para produzir imagens.
- Selecione Mostrar painel de verificação sequencial na parte superior do menu à esquerda para abrir a opção de verificação sequencial. Use o sinal '+' para configurar sequências adicionais, onde cada sequência conterá um comprimento de onda de excitação que foi decidido na etapa 2.2.
- Ligue todos os lasers necessários clicando nos botões ON associados aos lasers de interesse no centro da tela para transformá-los em um estado ON. Vá para cada sequência específica e aumente a potência do laser no centro da tela para qualquer valor desejado, onde cada sequência terá um comprimento de onda de excitação sendo usado.
- Ligue quantos detectores forem necessários na parte inferior da tela em cada sequência para obter imagens de todos os fluoróforos que serão fotografados nessa linha de laser. Ajuste os intervalos dos detectores clicando duas vezes em seus intervalos e inserindo os intervalos superior e inferior dos comprimentos de onda que precisam ser coletados.
NOTA: Recomenda-se que detectores de alta sensibilidade sejam usados sempre que possível e, idealmente, todos os canais devem ser coletados nos mesmos tipos de detectores, embora isso não seja obrigatório. - Pressione o botão ao vivo e concentre-se na amostra. Use o botão Auto Scale à esquerda da imagem no software para avaliar se os canais estão adequadamente saturados com sinal. O usuário deve ajustar os valores de ganho e as intensidades do laser em cada sequência até que o dimensionamento automático não atinja o valor máximo (65535 para dados de 16 bits), mas esteja muito próximo da saturação para todos os canais.
NOTA: Como diferentes fluoróforos podem diferir em seu brilho e diferentes alvos de anticorpos podem diferir em sua abundância, a sensibilidade dos detectores precisará ser ajustada para ser de baixa sensibilidade para sinais brilhantes e alta sensibilidade para sinais escuros se ambos os fluoróforos forem estimulados pelo mesmo laser (a intensidade do laser não pode mudar neste cenário, pois ambos os fluoróforos seriam afetados). Para obter melhores resultados, o sinal de fluorescência de cada canal no experimento deve ser balanceado de modo que cada canal tenha quantidades aproximadamente equivalentes de sinal, o que reduzirá o sangramento espectral para outros canais e resultará em sinais mais fortes. Como amostras diferentes podem diferir na abundância dos marcadores de interesse que estão sendo corados, recomenda-se que as configurações do microscópio sejam testadas em várias condições experimentais para confirmar que o branqueamento não está ocorrendo em nenhuma das condições experimentais. - Assim que as configurações do microscópio forem finalizadas, carregue o microscópio com as lâminas de controle de cor única e encontre a coloração representativa para cada fluoróforo. Configure uma pilha Z concentrando-se abaixo da amostra e pressionando o botão Iniciar no menu da pilha z, depois focando acima da amostra e pressionando o botão Final .
- Escolha o número de etapas e a distância entre as etapas (e use o mesmo número de etapas e parâmetros, como densidades de pixels e profundidade de bits, que serão usados para criar imagens dos slides totalmente manchados) e pressione o botão Iniciar.
NOTA: Idealmente, essas imagens devem incluir os sinais mais brilhantes da coloração real que podem ser encontrados na lâmina. - Depois que os controles de cor única forem criados, continue usando as mesmas configurações do microscópio para obter imagens de todas as amostras totalmente coradas.
3. Compensação de fluorescência
- Abra os arquivos .lif no Software 1 e clique na guia do processo.
- Escolha o módulo Separação de corantes e, em seguida, escolha a opção Separação automática de corantes . Selecione o método Manual para Separação de Tinta e escolha sem redimensionamento.
- Abra uma das imagens de controle de cor única. Inspecione manualmente os controles de cor única para sangramento espectral de um canal para o próximo, observando janelas em escala de cinza para cada canal em busca de evidências de um sinal em canais inadequados. Use o botão Auto Scale , se necessário, para ver o sangramento fraco.
- Comece a remover a sangria inserindo manualmente números (normalmente 0-1, onde 0 representa nenhuma sangria e 1 representa 100% de sangria) na matriz na tela e, em seguida, pressione aplicar para testar se a sangria foi removida adequadamente. Isso deve ser repetido até que os sinais em todos os outros canais, exceto o canal de interesse, tenham sido reduzidos a níveis de fundo.
NOTA: A supercompensação levará a resultados imprecisos, portanto, a fluorescência deve ser reduzida a níveis de fundo, mas não abaixo deles. Os dados supercompensados aparecerão como um canal com um sinal e outros canais parecendo ter buracos negros onde está o sinal de interesse (Figura 2). - Depois que um dos controles de cor única for concluído, registre os valores na matriz e, em seguida, redefina a matriz e passe para o próximo controle de cor única. Repita esse processo para cada controle de cor único.
NOTA: O primeiro corante estará na linha Corante 1 e a quantidade de fluorescência que precisa ser removida do Corante 1 estará nas colunas adjacentes. - Reúna todos os valores adquiridos de cada controle em uma matriz e aplique essa matriz a uma amostra totalmente corada. Se a separação parecer precisa, aplique as configurações a todas as imagens no experimento, caso contrário, reavalie os controles de cor única.
NOTA: A compensação efetiva deve remover a maior parte do sangramento entre os canais, conforme resumido e mostrado na Figura 3. - Salve todos os arquivos compensados em um arquivo .lif separado.
4. Identificação de núcleos e células usando ilastik
- Abra os arquivos .lif compensados no FIJI20. Habilite a visualização de pilhas em uma hiperpilha e selecione uma imagem.
- (Opcional) Use uma ferramenta de seleção, como a ferramenta retângulo, para incluir as partes da imagem que serão analisadas e excluir áreas que não serão analisadas. Corte a imagem usando a função de corte de imagem >.
NOTA: Reduzir o tamanho da imagem é útil nas etapas subsequentes para reduzir o consumo de memória de acesso aleatório (RAM) e aumentar a velocidade de processamento. - Salve a imagem como um arquivo .tif. Além disso, salve a imagem como um arquivo hdf5 usando o plug-in ilastik para FIJI clicando em Plug-ins > ilastik > Exportar hdf5. Salve o arquivo como "nome de escolha".h5 com compactação 0.
- Abra o Ilastik21 e faça um projeto de classificação de pixels. Use a função Adicionar novo > Adicionar imagens separadas para adicionar pelo menos 3 imagens representativas adicionando novos dados de entrada.
- Clique com o botão direito do mouse em uma das imagens, selecione Editar propriedades e altere o Modo de exibição para escala de cinza.
- À esquerda da tela, selecione a guia de seleção de recursos. Clique em Selecionar recursos e selecione todos os recursos até o valor sigma mais alto que o programa permitirá que você use e pressione OK.
- Agora selecione a guia Treinamento. Use a ferramenta pincel (enquanto o rótulo um estiver selecionado) para destacar células individuais (Figura 4) de interesse, de modo que o interior (citoplasma e núcleo) e a membrana celular sejam incluídos no destaque.
- Repita isso em todas as amostras de treinamento onde todos os tipos de células de interesse são selecionados, e várias células de intensidades, morfologias e localizações variadas no tecido devem ser selecionadas de cada amostra.
NOTA: Dependendo dos marcadores escolhidos para um conjunto individual de colorações, pode ou não haver colorações intracelulares ou colorações de membrana celular para as células de interesse. Quaisquer que sejam os marcadores usados, certifique-se de que o destaque inclua a coloração mais externa nas células de interesse e inclua o interior da célula, independentemente de uma coloração intracelular ser usada. - Em seguida, use o rótulo dois para destacar tudo o que não são as células de interesse (Figura 4). Isso pode incluir espaço em branco, núcleos não marcados ou tecido que não possui marcadores de interesse. Certifique-se de incluir o espaço imediatamente adjacente às células de interesse para promover o aprendizado dos limites exatos das células.
- Use a função Live Update para avaliar a qualidade do treinamento em imagens de interesse. Se o treinamento for inadequado, continue o treinamento nas etapas 4.7 a 4.9. Caso contrário, se satisfatório, clique na guia Exportação de previsão .
- Defina a origem como probabilidades. Clique no botão Escolher configurações de imagem de exportação e defina o parâmetro c para exportar 0-1 (supondo que o rótulo 1 esteja destacando os parâmetros de interesse).
- Converta o tipo de dados em um inteiro de 16 bits e exporte como um arquivo hdf5 para o diretório de interesse com o nome de interesse (recomendamos usar {nickname} mask.h5) e pressione OK.
- Clique na guia Processamento em lote . Pressione o botão Selecionar arquivos de dados brutos , selecione todos os arquivos .h5 de interesse que exigem processamento e pressione Abrir. Clique em Processar todos os arquivos.
- Repita as etapas 4.4 a 4.13, exceto que, em vez de identificar as células, agora identifique os núcleos e exclua todo o resto (Figura 4). Exporte isso como uma previsão separada para identificar núcleos celulares.
- Abra o Ilastik e crie um novo projeto de conversão de dados. Adicione um arquivo hdf5 representativo à guia Dados de entrada usando a função Adicionar novo > Adicionar imagem separada .
- Clique na guia Exportação de dados e escolha o botão Escolher configurações de imagem de exportação com a fonte definida para entrada. Altere o formato para tiff de várias páginas, escolha um local de salvamento com a função select e defina o nome do arquivo como {nickname}. Pressione OK.
- Clique na guia Processamento em lote e selecione todos os seus arquivos hdf5 usando o botão Selecionar arquivos de dados de entrada e pressione o botão Processar todos os arquivos .
- Abra o Software 2 (consulte Tabela de Materiais) e adicione todos os arquivos tif/tiff gerados nas etapas 4.3 e 4.17 usando o botão Adicionar Arquivos . Escolha um local para as saídas de arquivo e pressione o botão Iniciar tudo para converter todos os arquivos em arquivos .ims. Os arquivos agora estão prontos para análise no Software 3 (consulte Tabela de Materiais).
5. Análise em Software 3
- Abra uma das imagens que contém todas as manchas fluorescentes. Selecione Edit-> Adicionar canais e adicione os arquivos .ims correspondentes às máscaras feitas sobre núcleos e corpos celulares. Repita isso para todas as amostras.
- Selecione o botão Adicionar novas células . Selecione a opção detectar núcleo e célula e prossiga.
- Selecione a máscara sobre os núcleos para ser o canal de origem para detectar núcleos e selecione a opção avançada para dividir núcleos por pontos de semente. Escolha um valor para o diâmetro do núcleo) e prossiga.
NOTA: O valor ideal para o diâmetro do núcleo precisará ser testado executando a simulação e avaliando manualmente se o programa não está dividindo núcleos suficientes (vários núcleos são agrupados) ou se há divisão excessiva (um núcleo é dividido em mais de um). - Escolha um valor para a avaliação de qualidade e prossiga. Normalmente, é melhor incluir todos os núcleos, pois a omissão de núcleos pode levar à fusão de várias células em uma.
- Escolha um valor limite para criar superfícies nucleares e prossiga. Certifique-se de que todos os núcleos, mesmo aqueles que são mais escuros, ainda estejam incluídos no limite. Não torne o limite muito baixo, pois isso pode superestimar o tamanho dos núcleos ou potencialmente incluir detritos como coloração nuclear.
- Selecione um valor para o número mínimo de Voxels por núcleo e prossiga. Normalmente, é melhor manter tudo, a menos que detritos ou não núcleos possam ser excluídos usando este parâmetro.
- Escolha a opção Limite celular e citoplasma , selecione a máscara sobre as células para ser o canal de origem para detectar corpos celulares e prossiga.
- Escolha um limite de célula que capture com precisão todas as células de interesse, mas não seja tão baixo que inclua o plano de fundo ou qualquer detecção não específica de células. Selecione a opção para dividir as células em contato de forma que haja um núcleo por célula e elas sejam separadas pela distância dos núcleos. Além disso, opte por expandir as células nos núcleos e prossiga.
- Selecione um valor para o número mínimo de voxels por célula e prossiga. Normalmente, é melhor manter tudo, a menos que detritos ou não células possam ser excluídos usando este parâmetro.
- Avalie se as superfícies criadas sobre as células são precisas, observando se várias células foram fundidas ou se as células estão faltando. Se as superfícies forem inadequadas, volte para a etapa 5.2 e tente novos parâmetros até que as configurações ideais sejam determinadas.
- Depois que as melhores configurações forem estabelecidas, clique no objeto de células criadas no menu à esquerda, clique na guia de criação e clique no botão Armazenar parâmetros para lote para salvar as configurações usadas.
- Abra várias janelas do Software 3 e abra imagens separadas em cada janela. Pressione o novo botão Células em cada janela, escolha as configurações salvas no menu Parâmetros de criação favoritos e instrua cada janela a ser executada até a conclusão. Repita isso quantas vezes forem necessárias até que todas as amostras sejam analisadas.
NOTA: A execução de várias instâncias do Software 3 fará com que essa análise prossiga mais rapidamente, mas o número de janelas que podem ser executadas em paralelo dependerá da velocidade de processamento da unidade central de processamento (CPU) e da quantidade de RAM que o computador em que a análise está sendo executada. - Depois que todas as amostras forem concluídas, clique no objeto de células criadas no menu à esquerda em cada imagem e clique na guia Estatísticas. Clique no botão Configurar lista de valores de estatísticas visíveis, desmarque todas as estatísticas, exceto o valor 'Média de intensidade da célula' e pressione OK.
- Para cada imagem que precisa ser analisada, clique no botão Exportar todas as estatísticas para o arquivo na guia de estatísticas e salve os arquivos em um local.
6. Análise de citometria de histofluxo e mapeamento de populações em seções de tecido
- Abra o Anaconda e o JupyterLab no Anaconda. Em seguida, abra o script fornecido no Arquivo de Codificação Suplementar 1. Execute o código usando o menu Executar e selecione Executar Todas as Células ou execute as células individualmente em ordem selecionando Executar Células Selecionadas.
- Quando solicitado a inserir o arquivo de origem, insira o diretório de arquivos para os valores fluorescentes exportados gerados na etapa 5.14. O diretório de arquivos pode ser obtido examinando as propriedades de um dos arquivos .csv exportados e copiando o caminho do arquivo para o prompt.
- Em seguida, o programa solicitará um local de saída para os arquivos processados. Insira o diretório de arquivos para qualquer local adequado para salvar os arquivos processados. O programa detectará automaticamente o número de canais fluorescentes contidos na pasta.
- Execute a próxima seção do código para anotar os dados com os nomes de cada canal. Esse código pode ser modificado conforme necessário para alterar o parâmetro "numberOfChannels" para corresponder ao número de canais detectados na etapa 6.3. Além disso, os nomes dos canais podem ser modificados para quantos canais forem necessários, repetindo a linha 'ChX':'Marker' quantas vezes forem necessárias e modificando os nomes dos marcadores.
- A execução da próxima linha de código criará um arquivo .csv no local especificado que é anotado e contém todos os parâmetros fluorescentes no conjunto de dados. Repita as etapas 6.1 a 6.5 para cada conjunto de exportações do Software 3 para criar arquivos .csv para todos os dados.
- Converta os arquivos .csv em arquivos .fcs abrindo o Software 4 (consulte Tabela de materiais) e arrastando os arquivos .csv para a janela do programa. O Software 4 começará automaticamente a converter os arquivos em arquivos .fcs.
- Clique duas vezes em uma das amostras no Software 4 para abrir uma janela nessa amostra. Use as ferramentas de gating no Software 4 para criar uma estratégia de gating. Recomenda-se começar com um marcador definidor de linhagem contendo todas as células de interesse, depois prosseguir para portas que removem subconjuntos mais raros primeiro e, em seguida, terminar nos tipos de células mais abundantes.
- Depois que uma estratégia de gating for estabelecida, teste a precisão do gating clicando com o botão direito do mouse nessa população no menu e escolhendo exportar. Exporte todos os parâmetros como um arquivo .csv com cabeçalhos incluídos.
- Abra o JupyterLab no Anaconda. Em seguida, abra o script fornecido no Arquivo de Codificação Suplementar 2 e execute o código. Quando solicitado a inserir o local do arquivo Software 4, insira o diretório de arquivos para o arquivo de .csv exportado gerado na etapa anterior. O diretório de arquivos pode ser obtido examinando as propriedades do arquivo .csv exportado e copiando o caminho do arquivo para o prompt.
- Quando solicitado a adicionar o local de saída, escolha qualquer diretório de arquivo de sua escolha e certifique-se de que o diretório de arquivos inclua o nome do arquivo no final ('diretório de arquivos'\filename.txt). Execute o restante do código.
- Copie o texto no arquivo .txt e abra o arquivo .ims correspondente no Software 3 para o mesmo exemplo no qual os dados exportados se baseiam. Clique no objeto Cell no arquivo, alterne para a guia de estatísticas, cole o texto na barra de pesquisa e inicie a pesquisa. Todas as células de interesse serão destacadas.
- Inspecione manualmente se todas as células de interesse foram capturadas usando a estratégia de gating. Se o número de células for subestimado ou superestimado (exemplos mostrados na Figura 5), volte para a etapa 6.7, ajuste a porta e reavalie se as células são capturadas com precisão com a nova porta. Repita isso com todas as portas na estratégia de gating e avalie o gating em várias amostras.
- Quando todas as portas forem validadas, exporte os dados do Software 4 usando a função de editor de tabelas e analise os dados usando o método de análise preferido.
NOTA: As populações de células podem ser mapeadas de volta para as seções de tecido no Software 3, identificando a população fechada como nas etapas 6.8-6.11 e, em seguida, criando uma superfície com base nas células selecionadas. Isso pode ser feito para cada população fechada, conforme mostrado na Figura 6 e na Figura 7.
Resultados
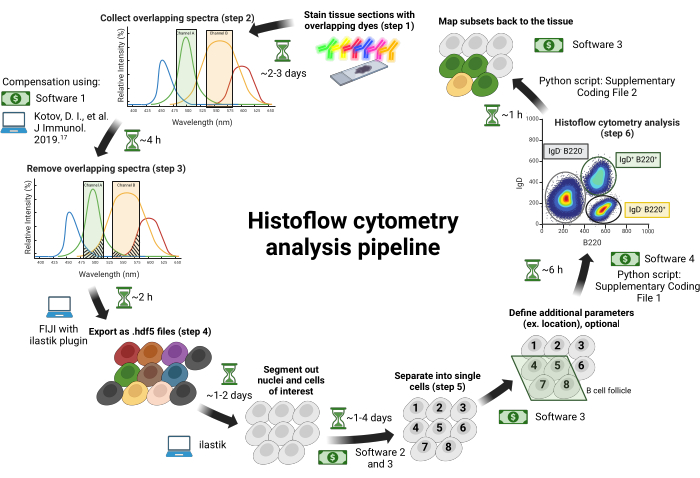
Figura 1: Fluxo de trabalho de citometria de histofluxo. As seções de tecido são coradas com corantes espectralmente sobrepostos (etapa 1). As imagens são coletadas em lasers de excitação individuais emparelhados com filtros passa-banda sintonizáveis para minimizar o sangramento espectral entre os fluoróforos (etapa 2). O sangramento espectr...
Discussão
Descreve-se aqui o uso da citometria de histofluxo, técnica validada anteriormente18. Está demonstrado que, ao corar seções de tecido com corantes espectralmente sobrepostos, esse sangramento através dos canais pode ser removido usando compensação espectral, resultando em um número maior de parâmetros fluorescentes sendo claramente resolvidos do que normalmente seria possível através de métodos convencionais. Como as imagens histológicas de alto parâmetro são difíceis de analisar u...
Divulgações
Os autores não têm conflitos de interesse financeiros.
Agradecimentos
Agradecemos à Plataforma de Microscopia Avançada do Hotchkiss Brain Institute pela infraestrutura e experiência em imagens. A RWJ foi apoiada por financiamento de bolsas de pós-doutorado do programa Eyes High da Universidade de Calgary e por uma bolsa educacional irrestrita da Sociedade de Esclerose Múltipla do Canadá e da Roche Canadá. A VWY recebeu apoio salarial do programa Canada Research Chair Tier 1. Este trabalho foi apoiado por fundos operacionais do Canadian Institutes of Health Research Grant 1049959, da Multiple Sclerosis Society of Canada Grant 3236 e do Departamento de Defesa dos EUA do Programa de Pesquisa de Esclerose Múltipla Dirigido pelo Congresso. A Figura 1 é criada com BioRender.com. Os números adaptados nesta publicação foram originalmente publicados no The Journal of Immunology. Rajiv W. Jain, David A. Elliott e V. Wee Yong. 2023. Análise de célula única de imagens histológicas de alto parâmetro usando citometria de histofluxo. J. Imunol. 210: 2038-2049. Direitos autorais © [2023]. A Associação Americana de Imunologistas, Inc.
Materiais
| Name | Company | Catalog Number | Comments |
| 100% Ethanol | Sigma | 676829-1L | |
| 4% PFA | Electron Microscopy Sciences | 157-4 | |
| Anaconda | N/A | N/A | https://www.anaconda.com/download |
| Bovine Serum Albumin | Sigma | A4503-50G | |
| Cold fish stain gelatin | Sigma | G7765 | |
| Collating multichannel data from Imaris.ipynb script | N/A | N/A | https://github.com/elliottcalgary/Histoflow-Cytometry-Analysis- |
| Convert FlowJo output to txt file for Cell selection in Imaris.ipynb script | N/A | N/A | https://github.com/elliottcalgary/Histoflow-Cytometry-Analysis- |
| Donkey anti-rat Alexa Fluor 647 | JacksonImmunoResearch | 712-605-153 | 1:300 concentration |
| Donkey anti-rat DyLight 405 | Jackson ImmunoResearch | 712-475-153 | 1:200 concentration |
| Donkey Serum | JacksonImmunoResearch | 017-000-001 | |
| F(ab')2-Goat anti-Mouse IgG PerCP-eFluor 710 | Thermofisher | 46-4010-82 | 1:25 concentration |
| FIJI | N/A | N/A | https://imagej.net/software/fiji/ |
| FlowJo | FlowJo LLC | Software 4 | |
| Fluorescence spectraviewer | https://www.thermofisher.com/order/fluorescence-spectraviewer/#!/ | ||
| Fluoromount-G | Southern Biotech | 0100-01 | |
| Fresh frozen human tonsil sections | amsbio | HF-707 | |
| Glass coverslip | VWR | 48393 106 | |
| Goat anti-human IgA Alexa Fluor 488 | JacksonImmunoResearch | 109-546-011 | 1:400 concentration |
| Goat anti-human IgG Cy3 | JacksonImmunoResearch | 709-166-098 | 1:400 concentration |
| Goat anti-human IgM Dylight 405 | JacksonImmunoResearch | 109-476-129 | 1:300 concentration |
| Goat anti-rabbit A546 | Thermo Fisher Scientific | A-11035 | 1:250 concentration |
| Goat anti-rabbit IgG PE-Alexa Fluor 610 | Thermofisher | A-20981 | 1:250 concentration |
| Horse Serum | Sigma | H1138 | |
| Ilastik | N/A | N/A | https://www.ilastik.org/ |
| Ilastik FIJI plugin | N/A | N/A | https://www.ilastik.org/documentation/fiji_export/plugin |
| Imaris File Converter | Oxford Instruments | Software 2 | |
| Imaris with cell module | Oxford Instruments | Software 3 | |
| kimwipe | Kimtech | 34155 | |
| LasX Life Science software | Leica | Software 1 | |
| Mouse anti-human CD20 | VWR | CA95024-322 | 1:40 concentration |
| Mouse anti-human CD38 APC-R700 | BD Biosciences | 564980 | 1:20 concentration |
| Normal Goat Serum | JacksonImmunoResearch | 005-000-001 | |
| Normal Mouse Serum | JacksonImmunoResearch | 015-000-001 | |
| Normal Rabbit Serum | JacksonImmunoResearch | 011-000-001 | |
| Normal Rat Serum | JacksonImmunoResearch | 012-000-120 | |
| Nuclear Yellow | Abcam | ab138903 | Dissolve in DMSO at a concentration of 2 mg/ml and store at 4°C in the dark |
| PAP pen | Cedarlane | MU22 | |
| PBS | Gibco | 10010-023 | |
| Rabbit anti-human Ki67 | Abcam | ab15580 | 1:500 concentration |
| Rabbit anti-mouse Iba1 | Wako | 019-19741 | 1:500 concentration |
| Rat anti-human Blimp1 | Thermofisher | 14-5963-82 | 1:40 concentration |
| Rat anti-mouse B220 Alexa Fluor 647 | BioLegend | 103226 | 1:250 concentration |
| Rat anti-mouse CD138 | Biolegend | 142502 | 1:200 concentration |
| Rat anti-mouse CD3 PE-eFluor 610 | Thermo Fisher Scientific | 61-0032-82 | 1:40 concentration |
| Rat anti-mouse CD4 Alexa Fluor 488 | BioLegend | 100529 | 1:200 concentration |
| Rat anti-mouse CD45 allophycocyanin-R700 | BD Biosciences | 565478 | 1:50 concentration |
| Rat anti-mouse IgD PerCP-eFluor 710 | Thermo Fisher Scientific | 46-5993-82 | 1:50 concentration |
| SP8 Confocal microscope | Leica | ||
| Triton X-100 | Sigma | X100-500ml | |
| Trueblack | Biotium | 23007 | |
| Tween-20 | Sigma | P7949-500ml | |
| Ultracomp ebeads | Thermofisher | 01-2222-42 |
Referências
- Bar-Or, A., Li, R. Cellular immunology of relapsing multiple sclerosis: interactions, checks, and balances. Lancet Neurol. 20 (6), 470-483 (2021).
- Borst, K., Dumas, A. A., Prinz, M. Microglia: Immune and non-immune functions. Immunity. 54 (10), 2194-2208 (2021).
- Han, R. T., Kim, R. D., Molofsky, A. V., Liddelow, S. A. Astrocyte-immune cell interactions in physiology and pathology. Immunity. 54 (2), 211-224 (2021).
- Absinta, M., et al. A lymphocyte-microglia-astrocyte axis in chronic active multiple sclerosis. Nature. 597 (7878), 709-714 (2021).
- Piwecka, M., Rajewsky, N., Rybak-Wolf, A. Single-cell and spatial transcriptomics: Deciphering brain complexity in health and disease. Nat Rev Neurol. 19 (6), 346-362 (2023).
- Schirmer, L., et al. Neuronal vulnerability and multilineage diversity in multiple sclerosis. Nature. 573 (7772), 75-82 (2019).
- Jain, R. W., Yong, V. W. B cells in central nervous system disease: Diversity, locations and pathophysiology. Nat Rev Immunol. 22 (8), 513-524 (2022).
- Lassmann, H. Multiple sclerosis pathology. Cold Spring Harb Perspect Med. 8 (3), (2018).
- Yong, V. W. Microglia in multiple sclerosis: Protectors turn destroyers. Neuron. 110 (21), 3534-3548 (2022).
- Sharma, S., Boyer, J., Teyton, L. A practitioner's view of spectral flow cytometry. Nat Methods. 21 (5), 740-743 (2024).
- Longo, S. K., Guo, M. G., Ji, A. L., Khavari, P. A. Integrating single-cell and spatial transcriptomics to elucidate intercellular tissue dynamics. Nat Rev Genet. 22 (10), 627-644 (2021).
- Ramaglia, V., et al. Multiplexed imaging of immune cells in staged multiple sclerosis lesions by mass cytometry. Elife. 8, e48051 (2019).
- Bolognesi, M. M., et al. Multiplex staining by sequential immunostaining and antibody removal on routine tissue sections. J Histochem Cytochem. 65 (8), 431-444 (2017).
- Radtke, A. J., et al. IBEX: An iterative immunolabeling and chemical bleaching method for high-content imaging of diverse tissues. Nat Protoc. 17 (2), 378-401 (2022).
- Radtke, A. J., et al. IBEX: A versatile multiplex optical imaging approach for deep phenotyping and spatial analysis of cells in complex tissues. Proc Natl Acad Sci U S A. 117 (52), 33455-33465 (2020).
- Gerner, M. Y., Kastenmuller, W., Ifrim, I., Kabat, J., Germain, R. N. Histo-cytometry: A method for highly multiplex quantitative tissue imaging analysis applied to dendritic cell subset microanatomy in lymph nodes. Immunity. 37 (2), 364-376 (2012).
- Kotov, D. I., Pengo, T., Mitchell, J. S., Gastinger, M. J., Jenkins, M. K. Chrysalis: A new method for high-throughput histo-cytometry analysis of images and movies. J Immunol. 202 (1), 300-308 (2019).
- Jain, R. W., Elliott, D. A., Yong, V. W. Single-cell analysis of high-parameter histology images using histoflow cytometry. J Immunol. 210 (12), 2038-2049 (2023).
- JoVE Science Education Database. General Laboratory Techniques. Histological Sample Preparation for Light Microscopy. JoVE. , (2023).
- Schindelin, J., et al. Fiji: An open-source platform for biological-image analysis. Nat Methods. 9 (7), 676-682 (2012).
- Berg, S., et al. ilastik: Interactive machine learning for (bio)image analysis. Nat Methods. 16 (12), 1226-1232 (2019).
- Reichard, A., Asosingh, K. Best practices for preparing a single cell suspension from solid tissues for flow cytometry. Cytometry A. 95 (2), 219-226 (2019).
- Ruhlandt, D., et al. Absolute quantum yield measurements of fluorescent proteins using a plasmonic nanocavity. Commun Biol. 3 (1), 627 (2020).
- Combs, C. A., Shroff, H. Fluorescence microscopy: A concise guide to current imaging methods. Curr Protoc Neurosci. 79, 1-25 (2017).
- Elliott, A. D. Confocal microscopy: Principles and modern practices. Curr Protoc Cytom. 92 (1), e68 (2020).
- Jonkman, J., Brown, C. M., Wright, G. D., Anderson, K. I., North, A. J. Tutorial: Guidance for quantitative confocal microscopy. Nat Protoc. 15 (5), 1585-1611 (2020).
- McNally, J. G., Karpova, T., Cooper, J., Conchello, J. A. Three-dimensional imaging by deconvolution microscopy. Methods. 19 (3), 373-385 (1999).
- Stringer, C., Wang, T., Michaelos, M., Pachitariu, M. Cellpose: A generalist algorithm for cellular segmentation. Nat Methods. 18 (1), 100-106 (2021).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados