このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
Histoflow Cytometryによるハイパラメーター組織像の生成と解析
要約
ここで説明するのは、免疫蛍光顕微鏡法により5つ以上の蛍光パラメータを画像化するために使用できる方法である。これらの画像から単一細胞を抽出し、フローサイトメトリーのようなゲーティング戦略を通じて単一細胞解析を実施するための解析パイプラインについて概説し、これにより組織切片の細胞サブセットを同定することができます。
要約
中枢神経系(CNS)に由来する組織切片などの免疫細胞の多様性を調査するための組織学の使用は、一度に画像化できる蛍光パラメータの数によって非常に制限されます。ほとんどの免疫細胞サブセットは、タンパク質マーカーの複雑な組み合わせを使用してフローサイトメトリーを使用して定義されており、最終的に同定するには4つ以上のパラメーターが必要になることが多く、これは従来のほとんどの顕微鏡の能力を超えています。フローサイトメトリーは組織を解離し、空間情報を失うため、複雑な細胞タイプの役割を調査しながら空間情報を保持できる技術が求められています。ここでは、スペクトル的に重なり合う蛍光色素のシグナルを収集し、スペクトルアンミキシングを使用して個々の蛍光色素分子のシグナルを分離することで、イメージングできる蛍光パラメーターの数を増やす方法を作成することで、これらの問題に対処します。次に、これらの画像を解析パイプラインを使用して処理し、ハイパラメーターの組織像を撮影し、これらの画像から単一細胞を抽出して、各細胞のユニークな蛍光特性を単一細胞レベルで分析できるようにします。フローサイトメトリーのようなゲーティング戦略を使用して、細胞をサブセットにプロファイリングし、組織学セクションにマッピングして、その存在量を定量化するだけでなく、組織環境とどのように相互作用するかを確立することができます。全体として、組織学切片の複雑な免疫集団を研究するためにヒストフローサイトメトリーを使用することの単純さと可能性が実証されています。
概要
免疫系の細胞とグリア細胞によって引き起こされる炎症は、各集団が他の1,2,3の活性を促進することができるCNSの慢性障害に寄与する可能性があります。免疫系がCNSのこれらの要素とどのように相互作用してCNSの炎症を促進するかを理解することは、現在主要な関心事であり、シングルセルRNAシーケンシングなどのハイパラメーター技術によって大幅に促進されています。シングルセルRNAシーケンシングを通じて、いくつかのCNS疾患において、グリア細胞と免疫系との間に広範なコミュニケーションが起こっていることを発見しました4,5,6。これらの相互作用がこれらの疾患にどのように影響しているかを理解することは、これらの疾患の生物学を解明するために非常に重要です。
シングルセルシーケンシング解析の問題点の1つは、これらの技術では、シングルセルまたは核を得るために組織を破壊する必要があり、その結果、空間情報が完全に失われることです。細胞が組織内のどこに存在するかを知ることは、炎症を引き起こす細胞の役割を理解するために重要です。例えば、B細胞などの免疫細胞は、神経炎症中にCNSに集中することができる。しかし、彼らはめったにCNS実質に入ることはなく、代わりにCNSバリア7に集中します。その局在性を考えると、これらの細胞がCNS実質のグリア細胞と物理的に相互作用することによってCNS炎症に寄与している可能性は低く、グリア細胞との相互作用は分泌因子を介して起こることが示唆されています。さらに、CNS障害で発生する病理は、多くの場合、組織内での細胞の局在化が、それが障害に積極的に寄与しているのか、それとも傍観者なのかを決定的に判断できるように、構造8,9を持っています。したがって、病理学における細胞の役割を評価するために空間的オリエンテーションを使用することが不可欠である。
組織中の細胞の研究は、通常、免疫組織化学または免疫蛍光顕微鏡を使用して行われてきました。これらの手法の問題は、通常、同時に最大4つのパラメータしか画像化できないことです。フローサイトメトリーやシングルセルRNAシーケンシング解析から、多くの細胞集団が同定のために2つ以上のパラメータを必要とすることがわかっているため、これはこれらの技術の大きな制限です。また、必要なパラメータの数は、通常、セルタイプ10の特定のサブセットを探すときに増加します。したがって、細胞のサブセットが組織内でどのように相互作用しているかを研究するために、標準的なイメージング技術を使用することは現実的ではありません。
この問題は、空間的RNAシーケンシング11やイメージングマスサイトメトリー12など、空間情報を保持できる新しいハイパラメータ法によって部分的に克服されています。これらの手法は価値がありますが、広く利用されていない、3 次元データを 2 次元に縮小する、実行するにはかなりの専門知識が必要になるなど、いくつかの問題があります。逐次染色として知られる別の技術は、組織を1セットの抗体で染色し、続いて前の抗体セットを不活性化してから別の抗体セットで染色するというもので、特殊な機器や専門知識を必要とせずにハイパラメーターの組織型検査を達成できる13,14,15.しかし、逐次染色は非常に手間がかかり、顕微鏡観察に長時間かかるため、個人用顕微鏡を所有していない研究室では実用的ではありません。そのため、広く利用可能な顕微鏡で一度にイメージングできる蛍光パラメータの数をタイムリーに拡大できる技術が求められています。
ハイパラメータデータを取得すると、従来の画像解析方法ではデータの解析がうまくいかないという別の問題が発生します。手動カウントやしきい値処理などの手法は、解析が 1 つのパラメーターで構成されている場合、または複数のマーカーが同じ位置を特定し、重複する信号のみがカウントされる場合にのみ実行可能です。この制限により、従来の分析では高パラメーターのデータセットを操作できなくなりました。これらのデータセットの解析は、組織像から単一細胞をセグメント化し、次いで細胞型を同定するためのフローサイトメトリー様ゲーティング戦略を実施することによって達成された16,17。しかし、これらの解析に影響を与える別の問題は、これらの手法では物理的に接触している細胞を正確に分離できる方法を採用していないため、対象のすべての細胞が物理的に分離されているデータセットでのみ機能することです。そのため、細胞が物理的に接触している場合でも、組織切片のシングルセル解析を行うことができる新しい方法が求められています。
この記事では、以前に紹介された18 、広く利用可能な顕微鏡を使用して同時にイメージングできる蛍光パラメータの数を増やす、ヒストフローサイトメトリーと呼ばれる簡単なプロトコルについて説明します。このプロトコールは、スペクトル的に重なり合う色素で組織を染色し、スペクトル補償を使用して重なり合うチャネルからブリードスルーを除去し、明確な単一染色を得ることによって機能します。ハイパラメータ組織像の解析を容易にするために、フローサイトメトリーのようなゲーティング戦略を用いて細胞を異なる集団に選別する目的で、組織切片から単一細胞を抽出する詳細な解析パイプラインが記載されている。このプロトコルは、細胞がびまん性して存在する組織や、細胞が密接に圧縮されている組織で機能するため、この技術は、恒常性と神経炎症の両方におけるCNSのような組織の研究に汎用性があります。したがって、Histoflow Cytometryは、空間情報を維持しながら細胞を定義するために複数の細胞マーカーを必要とする複雑な細胞タイプ間の相互作用を研究するための有用な技術です。
プロトコル
このプロトコルは、組織学のための切片組織を対象としていません。組織学のための組織切片の作り方の説明については、Jain et al.18 または19 を参照してください。このプロトコールは、スライドガラス上の任意の切片組織に使用できます。この記事では、前に説明したように、免疫化された動物から分離された鼠径リンパ節を使用します18。このプロトコルの手順とタイムラインを 図 1 にまとめます。本試験で使用した試薬および装置の詳細は、 材料表に記載されています。
1. 組織切片の染色
注:研究者が関心のあるすべての抗体標識で組織を染色することに加えて、研究者は、隣接するまたはそれ以外の同一の組織スライド上に単一カラーコントロール(使用を意図した蛍光パラメータごとに1つ)も調製する必要があります。そこでは、各単一カラーコントロールが1つの蛍光パラメータのシグナルを生成するために必要な試薬で染色されます。
- PermおよびブロッキングバッファーおよびPermおよび染色バッファーを調製します(組成については 表1 を参照)。
- スライドガラス上のティッシュ切片を室温で解凍し、ワイプで結露を拭き取ります。疎水性マーカーを使用して、染色する必要のある組織の境界の輪郭を描き、マークを乾かします。
- 組織切片をリン酸緩衝生理食塩水(PBS)に1分間浸して、組織を再水和します。PBSを振り落とし、ティッシュに直接触れずにワイプを使用して余分なPBSを取り除きます。
- 組織を完全に覆うのに十分な4%パラホルムアルデヒド(PFA)を追加して組織を固定します。室温で20分間インキュベートします。
注意:PFAは有毒である可能性があるため、ドラフト内で取り扱う必要があります。
注:この手順は、新鮮な凍結組織にのみ必要です。組織がすでに固定されている場合は、この手順を無視してください。ここではPFA固定について説明しますが、実験のニーズに応じて他の方法を使用して組織を固定することができます。 - ティッシュをPBSバスで1分間静かに揺らしながら洗い、スライドをバスから取り出します。PBSを交換し、さらに3回洗浄を繰り返します。
- 余分なPBSを振り落とし、ティッシュに直接触れずにワイプを使用して余分なPBSを取り除きます。
- プロトコールのステップ1.12で使用する必要がある二次抗体と、二次抗体が由来する動物種を決定します。これらの動物からの血清をパーマに加え、各種の最終濃度5%までブロッキングバッファーを添加することにより、パーミとブロッキングバッファーを強化します。
注:二次抗体を使用すると、シグナル強度を増幅するのに役立ちます。したがって、弱いシグナルを産生する一次抗体は、二次抗体と組み合わせてシグナルを増幅することが推奨されます。染色対象のすべての抗体ターゲットが、二次抗体を必要としないほど明るい場合は、ステップ1.7-1.12およびステップ1.15をスキップできます。 - 抗体の非特異的結合をブロックします:十分な強化Permを添加し、各組織を覆うようにバッファーをブロッキングします。スライドを室温で1〜8時間、光から保護された加湿チャンバー内でインキュベートします。
- 実験的に決定された希釈率で、非標識一次抗体をPermおよび染色バッファーに添加します(一次抗体の種または免疫グロブリンアイソタイプはすべて異なり、ステップ1.12で使用する予定の二次抗体によって区別できる必要があります)。
- Permとブロッキングバッファーを捨て、ワイプを使用して、ティッシュに直接触れずに余分なバッファーを取り除きます。組織を覆うのに十分な量のPermと一次抗体を含む染色バッファーを組織に加え、スライドを4°Cで12〜48時間インキュベートします。
注:研究者は、抗体インキュベーションの最適な時間を決定する必要があります。 - ブロッキングバッファーと染色バッファーを調製します(表1)。
- ステップ1.11で調製した染色バッファーに、実験的に決定した希釈倍率で二次抗体を添加します。使用する二次抗体は、ステップ1.9で使用したものと同じ種または抗体アイソタイプを標的とする必要があります。
- クエンチング・バッファーを準備します ( 表 1 を参照)。
注:このステップは、組織からの自家蛍光を減らすのに役立ちます。これが不要な場合は、手順 1.13 と 1.14 をスキップできます。 - 手順1.5〜1.6の説明に従ってティッシュを洗浄します。次に、スライド上の組織を覆うのに十分なクエンチングバッファーを追加し、スライドを室温で1〜2分間インキュベートします。
- 手順1.5〜1.6の説明に従ってティッシュを洗浄します。次に、ステップ1.12で調製した染色バッファーで希釈した二次抗体を組織に添加し、組織が染色バッファーで完全に覆われるようにします。スライドを4°Cで2〜8時間インキュベートします。
- ステップ1.11で調製したブロッキングバッファーに、ステップ1.18で使用する抗体種(各種の5%)から採取した正常血清50 μLを添加して、ブロッキング溶液を調製します。
- 手順1.5〜1.6の説明に従ってティッシュを洗浄します。次に、ステップ1.16で調製した正常血清を含むブロッキング溶液を組織に加えて、染色バッファーで完全に覆われるようにします。スライドを室温で1〜8時間インキュベートします。
- ステップ1.11で調製した染色バッファーに、実験的に決定された希釈倍率で蛍光色素標識一次抗体を添加して、染色溶液を調製します。また、核イエローなどの核染色剤を、実験的に決定された希釈で染色液に加えます。
注:ステップ1.18-1.19は、研究者がシグナル増幅に二次抗体を必要としない追加の抗体標識を使用する必要がある場合にのみ必要です。一次標識抗体を使用しない場合は、ステップ1.12で核染色剤を追加できます。 - ブロッキングバッファーを振り落とし、ワイプを使用して、ティッシュに直接触れずに余分なバッファーを取り除きます。ステップ1.18で調製した十分な染色溶液を組織に添加して覆い、4°Cで12〜48時間インキュベートします。
- 手順1.5〜1.6の説明に従ってティッシュを洗浄します。気泡を出さずに各スライドに5滴の封入剤を加え、ガラスカバースリップをティッシュの上に優しくゆっくりと傾けて、ティッシュを封入剤で完全に覆い、気泡をすべて排除します。スライドを4°Cの暗所で、イメージングする準備ができるまで保管します。
2. 組織切片のイメージング
注:この段階では、組織切片を、イメージングを意図するすべての条件について関心のあるすべての抗体で染色し、単色コントロールを各切片に1つの蛍光パラメータで染色します。イメージングは、特定の範囲の光を検出するように調整できる複数の検出器を備えた顕微鏡で行う必要があり、理想的には、励起のためにできるだけ多くのレーザーラインにアクセスできる必要があります。
- 完全に染色されたスライドの1つを顕微鏡に挿入し、データ収集に使用することを目的とした対物レンズを使用して切片を画像化するように顕微鏡をセットアップします。
- 組織が染色された蛍光色素に基づいて、それらの蛍光色素を刺激するために必要な励起レーザーラインを特定し、シグナルの量が最も多い各蛍光色素の発光範囲を特定します。
注:オンライン蛍光スペクトル表示ツール( 例えば、材料の表を参照)は、蛍光色素の最適な励起波長を特定し、蛍光色素検出の最適な範囲を特定するのに役立ちます。ヒストフローサイトメトリーに使用される蛍光色素は、各蛍光色素分子が刺激を受ける最適なレーザーラインが異なるか、または蛍光波長の最適な範囲が異なるように選択する必要があります。励起/発光波長が近い蛍光色素は、分離が難しくなります。この研究に使用した蛍光色素については、結果セクションで詳しく説明しています。 - 共焦点顕微鏡でソフトウェア1( 材料の表を参照)を開き、上部にある 設定タブ を選択します。次に、左側の ハードウェア ボタン を選択し、 ビット深度 を 16 ビットに変更します。
注:データ収集には、16ビットデータなどの高いビット深度を使用することをお勧めします。これにより、スペクトル補正の精度が向上します。さらに、ピクセル密度が高く、ライン平均の数が多くなり、Z スタックのステップ数が増えると、スペクトル分離の品質が向上します。 - 画面上部の [取得] タブ を選択し、画像のピクセル密度を含む形式設定を入力します (通常、解像度が高いほど良い結果が得られます)。
- 可能な場合は、 双方向X 取得を選択してイメージング時間を短縮します(この機能が適切にキャリブレーションされている場合)。
- 共焦点顕微鏡の ピンホール値 (通常は400〜600 nm)を選択すると、値が小さいほど通常、より高品質の組織フローサイトメトリーの結果が得られますが、ピンホール値が小さいと画像の取得が難しくなり、画像を生成するために追加のイメージング時間またはより高い励起強度が必要になる場合があります。
- 左側のメニューの上部にある[ シーケンシャルスキャンパネルの表示 ]を選択して、シーケンシャルスキャンオプションを開きます。 「+」 記号を使用して追加のシーケンスを設定し、各シーケンスにはステップ2.2で決定した1つの励起波長が含まれます。
- 画面の中央にある目的のレーザーに関連付けられている ON ボタンをクリックして、必要なすべてのレーザーをオンにします。各特定のシーケンスに移動し、画面の中央にあるレーザー出力を任意の値まで増やし、各シーケンスに1つの励起波長が使用されます。
- 各シーケンスの画面下部で必要な数の検出器 をオンにして 、そのレーザーラインでイメージングされるすべての蛍光色素をイメージングします。検出器 の範囲 を調整するには、検出器の範囲をダブルクリックし、収集する必要のある波長の上限範囲と下限範囲を入力します。
注:可能な限り高感度検出器を使用することを推奨し、理想的には、すべてのチャネルを同じタイプの検出器で収集する必要がありますが、これは必須ではありません。 - ライブボタンを押してから、サンプルにフォーカスを合わせます。ソフトウェアの画像の左側にある[Auto Scale]ボタンを使用して、チャンネルが信号で十分に飽和しているかどうかを評価します。ユーザーは、オートスケーリングが最大値(16ビットデータの場合は65535)に達しないが、すべてのチャンネルで飽和に非常に近づくまで、各シーケンスのゲイン値とレーザー強度を調整する必要があります。
注:蛍光色素が異なれば、その輝度も異なり、抗体ターゲットも存在量が異なる可能性があるため、両方の蛍光色素が同じレーザーによって刺激される場合、検出器の感度を明るい信号に対しては低く、暗い信号に対しては高感度に調整する必要があります(このシナリオでは、両方の蛍光色素が影響を受けるため、レーザー強度は変化しません)。最良の結果を得るには、実験の各チャンネルからの蛍光シグナルのバランスを取り、各チャンネルのシグナル量がほぼ同じになるようにすることで、他のチャンネルへのスペクトルのブリードスルーを減らし、より強力なシグナルを得ることができます。サンプルが異なれば、染色する対象マーカーの量も異なる場合があるため、顕微鏡の設定を複数の実験条件でテストして、どの実験条件下でも漂白が発生していないことを確認することをお勧めします。 - 顕微鏡の設定が完了したら、顕微鏡に単色制御スライドをロードし、各蛍光色素の代表的な染色を見つけます。Zスタックを設定するには、サンプルの下に焦点を合わせてZスタックメニュー の開始 ボタンを押し、次にサンプルの上にフォーカスして 終了 ボタンを押します。
- ステップ数とステップ間の距離を選択し(また、完全に染色されたスライドの画像化に使用されるピクセル密度やビット深度と同じステップ数とパラメータを使用します)、 スタートボタンを押します。
注:理想的には、これらの画像には、スライド上に見られる実際の染色からの最も明るい信号が含まれている必要があります。 - 単一カラーコントロールのイメージングが完了したら、同じ顕微鏡設定を使用して、完全に染色されたすべてのサンプルをイメージングします。
3.蛍光補償
- ソフトウェア1で.lifファイルを開き、[ プロセス]タブをクリックします。
- Dye Separationモジュールを選択し、次にAutomatic Dye Separationオプションを選択します。Dye Separationの方法として「Manual」を選択し、「no rescaling」を選択します。
- 単色制御画像の 1 つを開きます。1 つのチャンネルから次のチャンネルへのスペクトル ブリードスルーについて、各チャンネルのグレースケール ウィンドウを観察して、不適切なチャンネルの信号の証拠がないか、単色コントロールを手動で検査します。必要に応じて[ Auto Scale ]ボタンを使用して、弱いにじみを確認します。
- ブリードスルーの除去を開始するには、画面上のマトリックスに数字(通常は0-1、0はブリードスルーなし、1は100%ブリードスルー)を手動で入力し、 次に[適用 ]を押してブリードスルーが適切に除去されているかどうかをテストします。これは、対象のチャネルを除く他のすべてのチャネルの信号がバックグラウンドレベルに減少するまで繰り返す必要があります。
注:過度な補正は不正確な結果につながるため、蛍光はバックグラウンドレベルまで減らす必要がありますが、それ以下ではないようにする必要があります。過剰に補正されたデータは、1つのチャンネルに信号があり、他のチャンネルには目的の信号がある場所にブラックホールがあるように見えます(図2)。 - 単色コントロールの 1 つが完了したら、マトリックスに値を記録し、マトリックスを リセット して、次の単色コントロールに移動します。このプロセスを 1 つのカラーコントロールごとに繰り返します。
注:最初の色素はDye 1の行にあり、Dye 1から除去する必要がある蛍光の量は隣接する列にあります。 - 各コントロールから取得したすべての値を 1 つのマトリックスに組み立て、このマトリックスを完全に染色したサンプル に適用 します。色分解が正確であるように見える場合は、実験内のすべての画像に設定を適用し、そうでない場合は、単色コントロールを再評価します。
注:効果的な補正を行うと、 図3にまとめられているように、チャネル間のブリードスルーの大部分が除去されます。 - 補正されたすべてのファイルを別の .lif ファイルに保存します。
4. ilastikを用いた核・細胞の同定
- 補正された.lifファイルをFIJI20で開きます。ハイパースタックでの スタックの表示を有効にし 、画像を選択します。
- (オプション)四角形ツールなどの選択ツールを使用して、解析する画像の部分を含め、解析しない領域を除外します。 画像>切り抜き機能を使用して画像を切り抜きます。
注: イメージのサイズを小さくすると、ランダム アクセス メモリ (RAM) の消費量を減らし、処理速度を上げるために、後続の手順で役立ちます。 - 画像を.tifファイルとして保存します。また、ilastik plugin for FIJIを使用して、 Plugins > ilastik > Export hdf5をクリックして、画像をhdf5ファイルとして保存します。ファイルを "name of choice".h5 として保存し、圧縮率 0 にします。
- Ilastik21 を開き、ピクセル分類プロジェクトを作成します。 [新しい画像の追加] > [個別の画像の追加 ] 機能を使用して、新しい入力データを追加することで、少なくとも 3 つの代表的な画像を追加します。
- 画像の 1 つを右クリックし、[ プロパティの編集] を選択し、[ 表示モード ] をグレースケールに変更します。
- 画面の左側で、 機能選択 タブを選択します。 [機能の選択 ]をクリックし、プログラムが使用できる最高のシグマ値までのすべての機能を選択して、[ OK]を押します。
- 次に、[ トレーニング]タブを選択します。ペイントブラシツール( ラベル1 が選択されている間)を使用して、内部(細胞質と核)と細胞膜がハイライトに含まれるように、関心のある個々の細胞(図4)をハイライトします。
- これを、対象のすべての細胞タイプが選択されているすべてのトレーニングサンプルで繰り返し、各サンプルから組織内のさまざまな強度、形態、および局在の複数の細胞を選択する必要があります。
注:個々の染色セットに対して選択されたマーカーに応じて、目的の細胞の細胞内染色または細胞膜染色がある場合とない場合があります。どのマーカーを使用する場合でも、細胞内染色が使用されているかどうかに関係なく、ハイライトに目的の細胞の最も外側の染色が含まれていることと、細胞内部が含まれていることを確認してください。 - 次に、 ラベル 2 を使用して、対象のセル以外のすべてのセルを強調表示します (図 4)。これには、空白スペース、標識されていない核、または目的のマーカーがない組織が含まれる場合があります。正確なセル境界の学習を促進するために、関心のあるセルのすぐ隣のスペースを含めるようにしてください。
- ライブアップデート機能を使用して、関心のある画像のトレーニングの品質を評価します。トレーニングが不十分な場合は、ステップ 4.7 から 4.9 のトレーニングを続行します。それ以外の場合は、問題がなければ、[予測エクスポート]タブをクリックします。
- ソースを 確率に設定します。 [Choose Export Image Settings ] ボタンをクリックし、 c パラメータを 0-1 でエクスポートするように設定します (ラベル 1 が目的のパラメータを強調表示していると仮定します)。
- データ型を 16 ビット整数に変換し、関心のあるディレクトリに hdf5 ファイルとして目的の名前でエクスポートし ({nickname} mask.h5 を使用することをお勧めします)、 OK を押します。
- [バッチ処理]タブをクリックします。[生データファイルの選択]ボタンを押し、処理が必要なすべての.h5ファイルを選択して、[開く]を押します。[すべてのファイルを処理]をクリックします。
- 手順4.4から4.13を繰り返しますが、細胞を同定する代わりに、核を同定し、他のすべてを除外します(図4)。これを細胞核を同定するための別の予測としてエクスポートします。
- Ilastik を開き、新しいデータ変換プロジェクトを作成します。「新規追加」>「別の画像を追加」機能を使用して、代表的な hdf5 ファイルを「入力データ」タブに追加します。
- [Data Export] タブをクリックし、ソースを入力に設定した [Choose Export Image Settings] ボタンを選択します。フォーマットを複数ページのtiffに変更し、select機能で保存場所を選択し、ファイル名を{nickname}に設定します。OKを押します。
- バッチ処理タブをクリックし、入力データファイルの選択ボタンを使用してすべてのhdf5ファイルを選択し、すべてのファイルを処理ボタンを押します。
- ソフトウェア 2 ( 資料の表を参照) を開き、手順 4.3 と 4.17 で生成したすべての tif/tiff ファイルを [ファイルの追加 ] ボタンを使用して追加します。ファイル出力の場所を選択し、[ すべて開始 ]ボタンを押して、すべてのファイルを.imsファイルに変換します。これで、ファイルをソフトウェア3で分析する準備が整いました( 資料の表を参照)。
5. ソフトウェア3での分析
- すべての蛍光染色を含む画像の1つを開きます。Edit-> Add Channelsを選択し、核と細胞体に対して作成されたマスクに対応する.imsファイルを追加します。これをすべてのサンプルに対して繰り返します。
- [新しいセルの追加] ボタンを選択します。「detect nucleus and cell」オプションを選択し、先に進みます。
- 核を検出するためのソースチャネルとなる 核上のマスク を選択し、 シードポイントで核を分割するための詳細オプションを選択します。原子核の直径の値を選択してください)そして先に進みます。
注:原子核の直径の最適値は、シミュレーションを実行し、プログラムが十分な原子核を分割していないか(複数の原子核がグループ化されている)、または過剰に分裂している(1つの原子核が複数の原子核に分割されている)かを手動で評価してテストする必要があります。 - 品質評価の値を選択し、続行します。通常、核が欠落すると複数の細胞が1つに融合する可能性があるため、すべての核を含めるのが最善です。
- 核表面を作成するためのしきい値を選択してから、次に進みます。すべての原子核が、暗い原子核も含めて、閾値に含まれていることを確認してください。閾値を低くしすぎると、原子核のサイズが過大評価されたり、デブリが核染色として含まれる可能性があるため、低すぎないようにしてください。
- 原子核あたりの最小ボクセル数の値を選択し、続行します。通常、このパラメータを使用して破片や非原子核を除外できない限り、すべてを保持するのが最善です。
- [Cell boundary and cytoplasm] オプションを選択し、細胞体を検出するためのソースチャネルとなる [Mask over Cells] を選択してから、次に進みます。
- セルのしきい値は、関心のあるすべてのセルを正確にキャプチャしますが、バックグラウンドやセルの非特異的検出が含まれるほど低くはありません。接触している細胞を分割して、 細胞ごとに 1 つの核があり、 核からの距離によって分離されるようにするオプションを選択します。また、 核上の細胞を増殖 させてから進めることを選択します。
- セルあたりのボクセルの最小数の値を選択し、続行します。通常、このパラメータを使用して破片や非セルを除外できない限り、すべてを保持するのが最善です。
- セル上に作成されたサーフェスが正確かどうか、複数のセルが融合しているかどうか、またはセルが欠落していないかどうかを調べて評価します。サーフェスが不適切な場合は、手順5.2に戻り、最適な設定が決定されるまで新しいパラメータを試してください。
- 最適な設定が確立されたら、左側のメニューで 作成したセルオブジェクト をクリックし、 作成タブをクリックして、[ バッチのパラメーターを保存 ]ボタンをクリックして、使用した設定を保存します。
- ソフトウェア3の複数のウィンドウを開き、各ウィンドウで別々の画像を開きます。各ウィンドウで新しい セル ボタンを押し、 保存した設定をお気に入り作成パラメータ メニューで選択し、各ウィンドウに完了まで実行するように指示します。これを必要な回数だけ繰り返して、すべてのサンプルが分析されるまで繰り返します。
注: ソフトウェア 3 の複数のインスタンスを実行すると、この解析の進行は速くなりますが、並行して実行できるウィンドウの数は、中央処理装置 (CPU) の処理速度と、解析を実行するコンピュータの RAM の量によって異なります。 - すべてのサンプルが完了したら、各画像の左側のメニューにある 作成されたセルオブジェクト をクリックし、[ 統計]タブをクリックします。 [表示される統計値のリストを構成 ] ボタンをクリックし、[Cell Intensity Mean] 値を除くすべての統計の選択を解除して、[ OK] を押します。
- 分析が必要な画像ごとに、統計タブの [ すべての統計情報をファイルにエクスポート ] ボタンをクリックして、ファイルを場所に保存します。
6. Histoflow サイトメトリー解析と組織切片への集団のマッピング
- Anaconda 内で Anaconda と JupyterLab を開きます。次に、Supplementary Coding File 1 に記載されているスクリプトを開きます。[実行] メニューを使用してコードを実行し、[すべてのセルを実行] を選択するか、[選択したセルを実行] を選択してセルを個別に順番に実行します。
- ソースファイルの入力を求められたら、ステップ 5.14 で生成したエクスポートされた蛍光値のファイルディレクトリを入力します。ファイルディレクトリを取得するには、エクスポートされた.csvファイルの1つのプロパティを確認し、ファイルパスをプロンプトにコピーします。
- 次に、プログラムは処理されたファイルの出力場所を求めます。処理されたファイルの保存に適した場所のファイルディレクトリを入力します。プログラムは、フォルダに含まれる蛍光チャネルの数を自動的に検出します。
- コードの次のセクションを実行して、各チャネルの名前でデータに注釈を付けます。このコードは、ステップ6.3で検出されたチャネルの数と一致するように「numberOfChannels」パラメータを変更するために、必要に応じて変更できます。さらに、チャンネルの名前は、'ChX':'Marker' 行を必要な回数だけ繰り返し、マーカーの名前を変更することで、必要な数のチャンネルで変更できます。
- 次のコード行を実行すると、指定した場所に注釈が付けられ、データセット内のすべての蛍光パラメーターを含む .csv ファイルが作成されます。Software 3 エクスポートのセットごとに手順 6.1 から 6.5 を繰り返して、すべてのデータの .csv ファイルを作成します。
- ソフトウェア4( 資料の表を参照)を開き、.csvファイルをプログラムウィンドウにドラッグして、.csvファイルを.fcsファイルに変換します。ソフトウェア4は、ファイルから.fcsファイルへの変換を自動的に開始します。
- Software 4 のサンプルの 1 つをダブルクリックして、そのサンプルのウィンドウを開きます。Software 4 のゲーティング ツールを使用して、ゲーティング戦略を作成します。まず、目的のすべての細胞を含む系統定義マーカーでゲーティングを行い、次に希少なサブセットを最初に除去するゲートに進み、次により豊富な細胞タイプで終了することをお勧めします。
- ゲーティング戦略が確立されたら、メニューでその 母集団 を右クリックし、[ エクスポート]を選択して、ゲーティングの精度をテストします。すべてのパラメータを、ヘッダーを含む.csvファイルとしてエクスポートします。
- アナコンダでJupyterLabを開きます。次に、Supplementary Coding File 2 に記載されているスクリプトを開き、コードを実行します。Software 4 ファイルの場所を入力するように求められたら、前の手順で生成したエクスポートされた .csv ファイルのファイル ディレクトリを入力します。ファイルディレクトリは、エクスポートされた.csvファイルのプロパティを確認し、ファイルパスをプロンプトにコピーすることで取得できます。
- 出力場所を追加するように求められたら、任意のファイルディレクトリを選択し、ファイルディレクトリの末尾にファイル名 ('file directory'\filename.txt) が含まれていることを確認します。残りのコードを実行します。
- .txtファイル内のテキストをコピーし、エクスポートされたデータの基になっているのと同じサンプルの対応する.imsファイルをソフトウェア3で開きます。ファイル内の Cellオブジェクト をクリックし、 統計タブに切り替えて、検索バーにテキストを貼り付けて、検索を開始します。関心のあるすべてのセルが強調表示されます。
- ゲーティング戦略を使用して、目的のすべての細胞が捕捉されたかどうかを手動で検査します。セルの数が過小評価または過大評価されている場合 ( 図 5 に示す例)、ステップ 6.7 に戻り、ゲートを調整して、新しいゲートでセルが正確に捕捉されているかどうかを再評価します。ゲーティング戦略のすべてのゲートでこれを繰り返し、複数のサンプルでゲーティングを評価します。
- すべてのゲートが検証されたら、 テーブルエディタ 機能を使用してSoftware 4からデータをエクスポートし、推奨される分析方法を使用してデータを分析します。
注:ステップ6.8〜6.11のようにゲート集団を特定し、選択した細胞に基づいて表面を作成することにより、細胞集団をソフトウェア3の組織切片にマッピングし直すことができます。これは、 図 6 と 図 7 に示すように、ゲート化された母集団ごとに実行できます。
結果
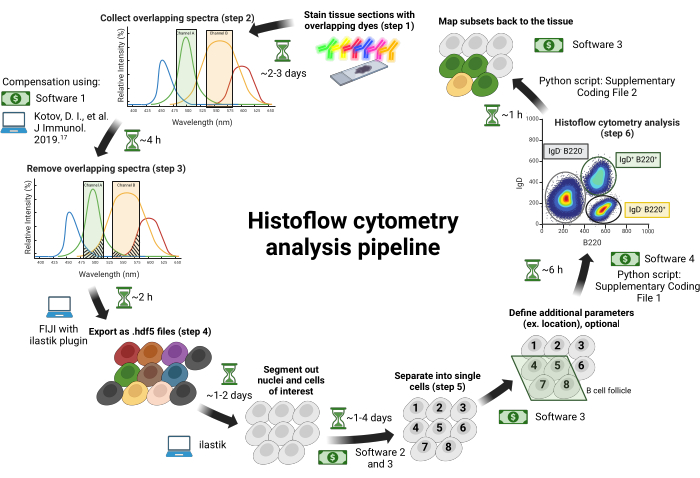
図1:Histoflowサイトメトリーのワークフロー。 組織切片は、スペクトル的に重なり合う色素で染色されます(ステップ1)。画像は、蛍光色素間のスペクトルブリードスルーを最小限に抑えるために、調整可能なバンドパスフィルターと組み合わせた個々の励起レーザ...
ディスカッション
ここでは、以前に検証された技術である組織流サイトメトリーの使用について説明します18。スペクトル的に重なり合う色素で組織切片を染色すると、スペクトル補償を使用してチャネルをまたいだブリードスルーを除去できるため、従来の方法よりも多くの蛍光パラメータが明確に分離されることが実証されています。従来の方法ではハイパラメータの組織像の解析が難?...
開示事項
著者には金銭的な利益相反はありません。
謝辞
Hotchkiss Brain Institute Advanced Microscopy Platformのイメージングインフラストラクチャと専門知識に感謝します。RWJは、カルガリー大学アイズハイプログラムからのポスドクフェローシップの資金提供と、カナダの多発性硬化症協会およびロシュカナダの無制限の教育フェローシップによって支援されました。VWYは、Canada Research Chair Tier 1プログラムから給与支援を受けました。この研究は、Canadian Institutes of Health Research Grant 1049959、Canada Multiple Sclerosis Society of Canada Grant 3236、および米国国防総省のCongressionally Directed Multiple Sclerosis Research Programからの運営資金によって支援されました。 図 1 は BioRender.com を使用して作成されています。この出版物で採用された数値は、もともと The Journal of Immunologyに掲載されました。ラジブ・W・ジェイン、デビッド・A・エリオット、V・ウィー・ヨン。2023. Histoflow Cytometryを使用したハイパラメータ組織像のシングルセル解析。 J.イムノール。210: 2038-2049.著作権 © [2023]。米国免疫学会
資料
| Name | Company | Catalog Number | Comments |
| 100% Ethanol | Sigma | 676829-1L | |
| 4% PFA | Electron Microscopy Sciences | 157-4 | |
| Anaconda | N/A | N/A | https://www.anaconda.com/download |
| Bovine Serum Albumin | Sigma | A4503-50G | |
| Cold fish stain gelatin | Sigma | G7765 | |
| Collating multichannel data from Imaris.ipynb script | N/A | N/A | https://github.com/elliottcalgary/Histoflow-Cytometry-Analysis- |
| Convert FlowJo output to txt file for Cell selection in Imaris.ipynb script | N/A | N/A | https://github.com/elliottcalgary/Histoflow-Cytometry-Analysis- |
| Donkey anti-rat Alexa Fluor 647 | JacksonImmunoResearch | 712-605-153 | 1:300 concentration |
| Donkey anti-rat DyLight 405 | Jackson ImmunoResearch | 712-475-153 | 1:200 concentration |
| Donkey Serum | JacksonImmunoResearch | 017-000-001 | |
| F(ab')2-Goat anti-Mouse IgG PerCP-eFluor 710 | Thermofisher | 46-4010-82 | 1:25 concentration |
| FIJI | N/A | N/A | https://imagej.net/software/fiji/ |
| FlowJo | FlowJo LLC | Software 4 | |
| Fluorescence spectraviewer | https://www.thermofisher.com/order/fluorescence-spectraviewer/#!/ | ||
| Fluoromount-G | Southern Biotech | 0100-01 | |
| Fresh frozen human tonsil sections | amsbio | HF-707 | |
| Glass coverslip | VWR | 48393 106 | |
| Goat anti-human IgA Alexa Fluor 488 | JacksonImmunoResearch | 109-546-011 | 1:400 concentration |
| Goat anti-human IgG Cy3 | JacksonImmunoResearch | 709-166-098 | 1:400 concentration |
| Goat anti-human IgM Dylight 405 | JacksonImmunoResearch | 109-476-129 | 1:300 concentration |
| Goat anti-rabbit A546 | Thermo Fisher Scientific | A-11035 | 1:250 concentration |
| Goat anti-rabbit IgG PE-Alexa Fluor 610 | Thermofisher | A-20981 | 1:250 concentration |
| Horse Serum | Sigma | H1138 | |
| Ilastik | N/A | N/A | https://www.ilastik.org/ |
| Ilastik FIJI plugin | N/A | N/A | https://www.ilastik.org/documentation/fiji_export/plugin |
| Imaris File Converter | Oxford Instruments | Software 2 | |
| Imaris with cell module | Oxford Instruments | Software 3 | |
| kimwipe | Kimtech | 34155 | |
| LasX Life Science software | Leica | Software 1 | |
| Mouse anti-human CD20 | VWR | CA95024-322 | 1:40 concentration |
| Mouse anti-human CD38 APC-R700 | BD Biosciences | 564980 | 1:20 concentration |
| Normal Goat Serum | JacksonImmunoResearch | 005-000-001 | |
| Normal Mouse Serum | JacksonImmunoResearch | 015-000-001 | |
| Normal Rabbit Serum | JacksonImmunoResearch | 011-000-001 | |
| Normal Rat Serum | JacksonImmunoResearch | 012-000-120 | |
| Nuclear Yellow | Abcam | ab138903 | Dissolve in DMSO at a concentration of 2 mg/ml and store at 4°C in the dark |
| PAP pen | Cedarlane | MU22 | |
| PBS | Gibco | 10010-023 | |
| Rabbit anti-human Ki67 | Abcam | ab15580 | 1:500 concentration |
| Rabbit anti-mouse Iba1 | Wako | 019-19741 | 1:500 concentration |
| Rat anti-human Blimp1 | Thermofisher | 14-5963-82 | 1:40 concentration |
| Rat anti-mouse B220 Alexa Fluor 647 | BioLegend | 103226 | 1:250 concentration |
| Rat anti-mouse CD138 | Biolegend | 142502 | 1:200 concentration |
| Rat anti-mouse CD3 PE-eFluor 610 | Thermo Fisher Scientific | 61-0032-82 | 1:40 concentration |
| Rat anti-mouse CD4 Alexa Fluor 488 | BioLegend | 100529 | 1:200 concentration |
| Rat anti-mouse CD45 allophycocyanin-R700 | BD Biosciences | 565478 | 1:50 concentration |
| Rat anti-mouse IgD PerCP-eFluor 710 | Thermo Fisher Scientific | 46-5993-82 | 1:50 concentration |
| SP8 Confocal microscope | Leica | ||
| Triton X-100 | Sigma | X100-500ml | |
| Trueblack | Biotium | 23007 | |
| Tween-20 | Sigma | P7949-500ml | |
| Ultracomp ebeads | Thermofisher | 01-2222-42 |
参考文献
- Bar-Or, A., Li, R. Cellular immunology of relapsing multiple sclerosis: interactions, checks, and balances. Lancet Neurol. 20 (6), 470-483 (2021).
- Borst, K., Dumas, A. A., Prinz, M. Microglia: Immune and non-immune functions. Immunity. 54 (10), 2194-2208 (2021).
- Han, R. T., Kim, R. D., Molofsky, A. V., Liddelow, S. A. Astrocyte-immune cell interactions in physiology and pathology. Immunity. 54 (2), 211-224 (2021).
- Absinta, M., et al. A lymphocyte-microglia-astrocyte axis in chronic active multiple sclerosis. Nature. 597 (7878), 709-714 (2021).
- Piwecka, M., Rajewsky, N., Rybak-Wolf, A. Single-cell and spatial transcriptomics: Deciphering brain complexity in health and disease. Nat Rev Neurol. 19 (6), 346-362 (2023).
- Schirmer, L., et al. Neuronal vulnerability and multilineage diversity in multiple sclerosis. Nature. 573 (7772), 75-82 (2019).
- Jain, R. W., Yong, V. W. B cells in central nervous system disease: Diversity, locations and pathophysiology. Nat Rev Immunol. 22 (8), 513-524 (2022).
- Lassmann, H. Multiple sclerosis pathology. Cold Spring Harb Perspect Med. 8 (3), (2018).
- Yong, V. W. Microglia in multiple sclerosis: Protectors turn destroyers. Neuron. 110 (21), 3534-3548 (2022).
- Sharma, S., Boyer, J., Teyton, L. A practitioner's view of spectral flow cytometry. Nat Methods. 21 (5), 740-743 (2024).
- Longo, S. K., Guo, M. G., Ji, A. L., Khavari, P. A. Integrating single-cell and spatial transcriptomics to elucidate intercellular tissue dynamics. Nat Rev Genet. 22 (10), 627-644 (2021).
- Ramaglia, V., et al. Multiplexed imaging of immune cells in staged multiple sclerosis lesions by mass cytometry. Elife. 8, e48051 (2019).
- Bolognesi, M. M., et al. Multiplex staining by sequential immunostaining and antibody removal on routine tissue sections. J Histochem Cytochem. 65 (8), 431-444 (2017).
- Radtke, A. J., et al. IBEX: An iterative immunolabeling and chemical bleaching method for high-content imaging of diverse tissues. Nat Protoc. 17 (2), 378-401 (2022).
- Radtke, A. J., et al. IBEX: A versatile multiplex optical imaging approach for deep phenotyping and spatial analysis of cells in complex tissues. Proc Natl Acad Sci U S A. 117 (52), 33455-33465 (2020).
- Gerner, M. Y., Kastenmuller, W., Ifrim, I., Kabat, J., Germain, R. N. Histo-cytometry: A method for highly multiplex quantitative tissue imaging analysis applied to dendritic cell subset microanatomy in lymph nodes. Immunity. 37 (2), 364-376 (2012).
- Kotov, D. I., Pengo, T., Mitchell, J. S., Gastinger, M. J., Jenkins, M. K. Chrysalis: A new method for high-throughput histo-cytometry analysis of images and movies. J Immunol. 202 (1), 300-308 (2019).
- Jain, R. W., Elliott, D. A., Yong, V. W. Single-cell analysis of high-parameter histology images using histoflow cytometry. J Immunol. 210 (12), 2038-2049 (2023).
- JoVE Science Education Database. General Laboratory Techniques. Histological Sample Preparation for Light Microscopy. JoVE. , (2023).
- Schindelin, J., et al. Fiji: An open-source platform for biological-image analysis. Nat Methods. 9 (7), 676-682 (2012).
- Berg, S., et al. ilastik: Interactive machine learning for (bio)image analysis. Nat Methods. 16 (12), 1226-1232 (2019).
- Reichard, A., Asosingh, K. Best practices for preparing a single cell suspension from solid tissues for flow cytometry. Cytometry A. 95 (2), 219-226 (2019).
- Ruhlandt, D., et al. Absolute quantum yield measurements of fluorescent proteins using a plasmonic nanocavity. Commun Biol. 3 (1), 627 (2020).
- Combs, C. A., Shroff, H. Fluorescence microscopy: A concise guide to current imaging methods. Curr Protoc Neurosci. 79, 1-25 (2017).
- Elliott, A. D. Confocal microscopy: Principles and modern practices. Curr Protoc Cytom. 92 (1), e68 (2020).
- Jonkman, J., Brown, C. M., Wright, G. D., Anderson, K. I., North, A. J. Tutorial: Guidance for quantitative confocal microscopy. Nat Protoc. 15 (5), 1585-1611 (2020).
- McNally, J. G., Karpova, T., Cooper, J., Conchello, J. A. Three-dimensional imaging by deconvolution microscopy. Methods. 19 (3), 373-385 (1999).
- Stringer, C., Wang, T., Michaelos, M., Pachitariu, M. Cellpose: A generalist algorithm for cellular segmentation. Nat Methods. 18 (1), 100-106 (2021).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved