A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
הפקה וניתוח של תמונות היסטולוגיה בעלות פרמטרים גבוהים באמצעות ציטומטריה של Histoflow
In This Article
Summary
המתוארת כאן היא שיטה שניתן להשתמש בה כדי לצלם חמישה פרמטרים פלואורסצנטיים או יותר על ידי מיקרוסקופ immunofluorescent. מתואר צינור ניתוח לחילוץ תאים בודדים מתמונות אלה ולביצוע אנליזה של תא בודד באמצעות אסטרטגיות gating דמויות ציטומטריה של זרימה, אשר יכולות לזהות תת-קבוצות תאים במקטעי רקמות.
Abstract
השימוש בהיסטולוגיה כדי לחקור את מגוון תאי החיסון בחלקי רקמות, כגון אלה הנגזרים ממערכת העצבים המרכזית (CNS), מוגבל באופן קריטי על ידי מספר הפרמטרים הפלואורסצנטיים שניתן לצלם בו זמנית. רוב תת-הקבוצות של תאי מערכת החיסון הוגדרו באמצעות ציטומטריית זרימה באמצעות שילובים מורכבים של סמני חלבון, שלעתים קרובות דורשים ארבעה פרמטרים או יותר כדי לזהות באופן חד-משמעי, וזה מעבר ליכולות של רוב המיקרוסקופים הקונבנציונליים. כאשר ציטומטריית זרימה מנתקת רקמות ומאבדת מידע מרחבי, יש צורך בטכניקות שיכולות לשמור מידע מרחבי תוך חקירת תפקידיהם של סוגי תאים מורכבים. בעיות אלה מטופלות כאן על ידי יצירת שיטה להרחבת מספר הפרמטרים הפלואורסצנטיים שניתן לצלם על ידי איסוף האותות של פלואורופורים חופפים ספקטרלית ושימוש בפירוק ספקטרלי כדי להפריד את האותות של כל פלואורופור בודד. תמונות אלה מעובדות לאחר מכן באמצעות צינור ניתוח כדי לצלם תמונות היסטולוגיה בעלות פרמטרים גבוהים ולחלץ תאים בודדים מתמונות אלה, כך שניתן יהיה לנתח את התכונות הפלואורסצנטיות הייחודיות של כל תא ברמת תא יחיד. באמצעות שימוש באסטרטגיות gating דמויות ציטומטריית זרימה, ניתן ליצור פרופיל של תאים לתת-קבוצות ולמפות אותם בחזרה לקטעי ההיסטולוגיה כדי לא רק לכמת את השפע שלהם, אלא גם לקבוע כיצד הם מתקשרים עם סביבת הרקמה. בסך הכל, הודגמה הפשטות והפוטנציאל של שימוש בציטומטריה של היסטופלואו לחקר אוכלוסיות חיסוניות מורכבות במדורי היסטולוגיה.
Introduction
דלקת המונעת על ידי תאים של מערכת החיסון ותאי גלייה יכולה לתרום להפרעות כרוניות של מערכת העצבים המרכזית, כאשר כל אוכלוסייה יכולה לקדם את הפעילות של 1,2,3 האחרים. הבנת האופן שבו מערכת החיסון מתקשרת עם אלמנטים אלה של מערכת העצבים המרכזית כדי לקדם דלקת במערכת העצבים המרכזית היא כיום נושא מרכזי של עניין, וזה כבר הקל מאוד על ידי טכניקות פרמטרים גבוהים כגון ריצוף RNA תא יחיד. באמצעות ריצוף RNA חד-תאי, גילינו כי קיימת תקשורת נרחבת בין תאי גלייה לבין מערכת החיסון במספר הפרעות במערכת העצבים המרכזית 4,5,6. הבנת האופן שבו אינטראקציות אלה משפיעות על הפרעות אלה תהיה חיונית להבהרת הביולוגיה של מחלות אלה.
בעיה אחת עם ניתוחי ריצוף של תא בודד היא שטכניקות אלה דורשות לשבש את הרקמה כדי להשיג תאים בודדים או גרעינים, וכתוצאה מכך אובדן מוחלט של מידע מרחבי. הידיעה היכן תא קיים ברקמה היא קריטית להבנת תפקידו של התא בהנעת דלקת. לדוגמה, תאים חיסוניים כגון תאי B יכולים להתרכז במערכת העצבים המרכזית במהלך דלקת עצבית; עם זאת, לעתים רחוקות הם נכנסים לפרנכימה של מערכת העצבים המרכזית ובמקום זאת מתרכזים במחסומי CNS7. בהתחשב בלוקליזציה שלהם, אין זה סביר כי תאים אלה תורמים דלקת CNS על ידי אינטראקציה פיזית עם תאי גליה בפרנכימה CNS, דבר המצביע על כך שכל אינטראקציה שהם עשויים לקיים עם תאי גליה תתרחש באמצעות גורמים מופרשים. בנוסף, הפתולוגיה המתרחשת בהפרעות CNS לעתים קרובות יש מבנה 8,9 כך המיקום של התא ברקמה יכול לקבוע באופן קריטי אם הוא תורם באופן פעיל להפרעה או הוא צופה מהצד. לפיכך, השימוש באוריינטציה מרחבית כדי להעריך את תפקידו של התא בפתולוגיה הוא חיוני.
חקר תאים ברקמה נעשה בדרך כלל באמצעות אימונוהיסטוכימיה או מיקרוסקופ פלואורסצנטי של מערכת החיסון. בעיה עם טכניקות אלה היא שהן בדרך כלל יכולות לדמיין רק עד ארבעה פרמטרים בו זמנית. זוהי מגבלה משמעותית לטכניקות אלה, כפי שאנו יודעים מציטומטריית זרימה ומניתוחי ריצוף RNA של תא בודד כי אוכלוסיות תאים רבות דורשות שני פרמטרים או יותר לזיהוין; כמו כן, מספר הפרמטרים הנדרשים בדרך כלל גדל כאשר מחפשים תת-קבוצות ספציפיות של סוג תא10. לכן, אין זה מעשי להשתמש בטכניקות הדמיה סטנדרטיות כדי לחקור כיצד תת-קבוצות של תאים עשויות לקיים אינטראקציה בתוך רקמה.
בעיה זו נפתרה חלקית באמצעות שיטות חדשות יותר בעלות פרמטרים גבוהים שיכולות לשמור מידע מרחבי, כגון ריצוף RNA מרחבי11 וציטומטריית מסה12. בעוד טכניקות אלה הן בעלות ערך, יש להם כמה בעיות, כגון לא להיות זמין באופן נרחב, צמצום נתונים תלת מימדיים לשני ממדים, ודורש מומחיות רבה לבצע. טכניקה נוספת המכונה צביעה רציפה, שבה רקמות מוכתמות עם קבוצה אחת של נוגדנים ואחריו השבתה של קבוצה קודמת של נוגדנים לפני צביעה עם קבוצה אחרת של נוגדנים, יכול להשיג היסטולוגיה פרמטרים גבוהים ללא צורך בציוד מיוחד או מומחיות 13,14,15. עם זאת, צביעה רציפה יכולה להיות אינטנסיבית מאוד עבודה ודורשת כמות גדולה של זמן מיקרוסקופיה, אשר עשוי להיות לא מעשי עבור מעבדות שאין בבעלותן מיקרוסקופ אישי. לכן, יש צורך בטכניקות שיכולות להרחיב את מספר הפרמטרים הפלואורסצנטיים שניתן לצלם בו זמנית על מיקרוסקופים הזמינים באופן נרחב ובזמן.
לאחר רכישת הנתונים בעלי הפרמטרים הגבוהים, מתעוררת בעיה נוספת: שיטות ניתוח תמונה קונבנציונליות לא צפויות לנתח בהצלחה את הנתונים. טכניקות כגון ספירה ידנית או סף הן בנות קיימא רק אם הניתוח מורכב מפרמטר יחיד או אם לסמנים מרובים יש לוקליזציה זהה שבה נספרים רק אותות חופפים. מגבלה זו הופכת את הניתוח המסורתי לבלתי מספק לעבודה עם מערכי נתונים בעלי פרמטרים גבוהים. ניתוח מוצלח של מערכי נתונים אלה הושג על ידי פילוח תאים בודדים מתמונות היסטולוגיה ולאחר מכן ביצוע אסטרטגיות gating דמויות ציטומטריית זרימה כדי לזהות סוגי תאים16,17. עם זאת, בעיה נוספת המשפיעה על ניתוחים אלה היא שהם עובדים רק עבור מערכי נתונים שבהם כל התאים המעניינים מופרדים פיזית זה מזה, שכן טכניקות אלה אינן משתמשות בשיטות שיכולות להפריד במדויק תאים הנמצאים במגע פיזי. לכן, נדרשת שיטה חדשה יותר שיכולה לבצע אנליזות של תא בודד על קטעי היסטולוגיה גם אם התאים נמצאים במגע פיזי.
במאמר זה מתואר פרוטוקול פשוט שנקרא ציטומטריה היסטופלואו שהוצג בעבר18 המרחיב את מספר הפרמטרים הפלואורסצנטיים שניתן לצלם בו זמנית באמצעות מיקרוסקופים זמינים באופן נרחב. פרוטוקול זה פועל על ידי צביעת רקמות בצבעים חופפים ספקטרלית ולאחר מכן שימוש בפיצוי ספקטרלי כדי להסיר דימום מתעלות חופפות לקבלת כתמים בודדים ברורים. כדי להקל על ניתוח תמונות היסטולוגיה בעלות פרמטרים גבוהים, מתואר צינור ניתוח מפורט המחלץ תאים בודדים מקטעי רקמה לצורך מיון תאים לאוכלוסיות נפרדות באמצעות אסטרטגיות gating דמויות ציטומטריה של זרימה. פרוטוקול זה פועל ברקמות שבהן תאים נמצאים באופן מפוזר וברקמות שבהן תאים דחוסים זה לזה, מה שהופך את הטכניקה הזו לרב-תכליתית לחקר רקמות כמו מערכת העצבים המרכזית הן בהומאוסטזיס והן בדלקת עצבית. הציטומטריה של היסטופלואו היא, אם כן, טכניקה שימושית לחקר אינטראקציות בין סוגי תאים מורכבים הדורשים סמני תאים מרובים כדי להגדיר תאים תוך שמירה על מידע מרחבי.
Protocol
פרוטוקול זה אינו מכסה חיתוך רקמות להיסטולוגיה; אנא ראה Jain et al.18 או19 לתיאורים כיצד לחתוך רקמות להיסטולוגיה. פרוטוקול זה יכול לשמש עם כל רקמות חתך על שקופיות זכוכית. מאמר זה משתמש בבלוטות לימפה מפשעתיות שבודדו מבעל חיים מחוסן כפי שתואר קודם לכן18. ההליך וציר הזמן עבור פרוטוקול זה מסוכמים באיור 1. פרטי הריאגנטים והציוד ששימש במחקר זה מפורטים בטבלת החומרים.
1. צביעת קטעי רקמות
הערה: בנוסף להכתמת רקמות עם כל תוויות הנוגדנים שהחוקר מעוניין בהן, על החוקר להכין גם פקדי צבע בודדים (אחד לכל פרמטר פלואורסצנטי המיועד לשימוש) על שקופיות רקמה סמוכות או זהות אחרות, שבהן כל פקד צבע יחיד מוכתם בריאגנטים הדרושים להפקת האות של פרמטר פלואורסצנטי אחד.
- הכינו את חיץ הפרם והחסימה ואת חיץ הפרם והצביעה (ראו טבלה 1 להרכב).
- הפשירו מקטעי רקמות על מגלשות זכוכית בטמפרטורת החדר ונגבו את העיבוי באמצעות מגבון. השתמש בטוש הידרופובי כדי לשרטט את גבול הרקמה (ים) שיש להכתים ולתת לסימנים להתייבש.
- יש לייבש מחדש את הרקמות על ידי השריית חלקי הרקמה במי מלח חוצצי פוספט (PBS) למשך דקה אחת. נערו את ה-PBS והשתמשו במגבון כדי להסיר עודפי PBS מבלי לגעת ישירות ברקמה.
- תקן את הרקמות על ידי הוספת מספיק 4% paraformaldehyde (PFA) כדי לכסות את הרקמה לחלוטין. יש לדגור בטמפרטורת החדר למשך 20 דקות.
זהירות: PFA עלול להיות רעיל ויש לטפל בו בתוך מכסה מנוע.
הערה: שלב זה נדרש רק עבור רקמות קפואות טריות. אם הרקמה כבר קבועה, התעלם משלב זה. קיבוע PFA מתואר כאן, אך ניתן לתקן את הרקמה בשיטות אחרות בהתאם לצרכי הניסוי. - שטפו את הרקמות באמבט PBS למשך דקה אחת בנדנדה עדינה, ולאחר מכן הסירו את המגלשה מהאמבטיה. החלף את PBS וחזור על הכביסה 3 פעמים נוספות.
- נערו את עודפי PBS והשתמשו במגבון כדי להסיר עודפי PBS מבלי לגעת ישירות ברקמה.
- לקבוע אילו נוגדנים משניים יש להשתמש בשלב 1.12 של הפרוטוקול ואת מיני בעלי החיים שמהם נגזרים הנוגדנים המשניים. ביצור הפרם וחסימת החיץ על ידי הוספת נסיוב מבעלי חיים אלה לפרם וחסימת החיץ לריכוז סופי של 5% לכל מין.
הערה: שימוש בנוגדנים משניים מסייע להגביר את עוצמת האות. לכן, מומלץ כי נוגדנים ראשוניים המייצרים אותות חלשים צריכים להיות מזווגים עם נוגדנים משניים כדי להגביר את האותות שלהם. אם כל מטרות הנוגדנים המוכתמות בהירות מספיק כדי לא לדרוש נוגדנים משניים, ניתן לדלג על שלבים 1.7-1.12 ושלב 1.15. - חסום קשירה לא ספציפית של נוגדנים על ידי הוספת מספיק פרם מועשר וחסימת חיץ כדי לכסות כל רקמה. דגרו על המגלשות במשך 1-8 שעות בטמפרטורת החדר בתא לח המוגן מפני אור.
- הוסף נוגדנים ראשוניים לא מצומדים לפרם וחיץ כתמים בדילולים שנקבעו בניסוי (המינים או איזוטיפים האימונוגלובולינים של הנוגדנים הראשוניים צריכים להיות שונים ומובחנים על ידי הנוגדנים המשניים שהחוקר מתכוון להשתמש בהם בשלב 1.12).
- השליכו את החיץ החוסם ואת הפרם והשתמשו במגבון כדי להסיר את החיץ העודף מבלי לגעת ישירות ברקמה. הוסף מספיק חיץ פרם ומכתים עם נוגדנים ראשוניים לרקמה כדי לכסות אותה ולדגור על המגלשה ב 4 מעלות צלזיוס במשך 12-48 שעות.
הערה: החוקר יצטרך לקבוע את הזמן האופטימלי לדגירת נוגדנים. - הכינו חיץ חוסם ומאגר כתמים (טבלה 1).
- הוסף נוגדנים משניים למאגר הצביעה שהוכן בשלב 1.11 בדילולים שנקבעו בניסוי. הנוגדנים המשניים המשמשים צריכים להתמקד באותו מין או איזוטיפים נוגדנים שהיו בשימוש בשלב 1.9.
- הכינו את מאגר המרווה (כמו בטבלה 1).
הערה: שלב זה מסייע להפחית את הפלואורסצנטיות העצמית מהרקמה. אם אין צורך בכך, ניתן לדלג על שלבים 1.13 ו- 1.14. - לשטוף את הרקמות כמתואר בשלבים 1.5-1.6. לאחר מכן, הוסיפו מספיק חיץ מרווה כדי לכסות את הרקמות במגלשות ולדגור על המגלשות בטמפרטורת החדר למשך 1-2 דקות.
- שטפו את הרקמה כמתואר בשלבים 1.5-1.6. לאחר מכן, הוסף את הנוגדנים המשניים המדוללים במאגר הצביעה שהוכן בשלב 1.12 לרקמות כך שהרקמות מכוסות לחלוטין במאגר הצביעה. דגירה על שקופיות ב 4 ° C במשך 2-8 שעות.
- הכן את תמיסת החסימה על ידי הוספת 50 μL של סרום רגיל מכל מין של נוגדנים שישמשו בשלב 1.18 (5% מכל מין) למאגר החוסם שהוכן בשלב 1.11.
- לשטוף את הרקמות כמתואר בשלבים 1.5-1.6. לאחר מכן, הוסף את הפתרון החוסם עם סרום רגיל מוכן בשלב 1.16 לרקמות, כך שהם מכוסים לחלוטין במאגר מכתים. יש לדגור על מגלשות בטמפרטורת החדר למשך 1-8 שעות.
- הכינו את תמיסת הצביעה על ידי הוספת נוגדנים ראשוניים מצומדים פלואורופורים בדילול שנקבע בניסוי למאגר הצביעה שהוכן בשלב 1.11. כמו כן, הוסף כתם גרעיני, כגון צהוב גרעיני, לתמיסת הצביעה בדילול שנקבע בניסוי.
הערה: שלבים 1.18-1.19 נדרשים רק אם החוקר צריך להשתמש בתוויות נוגדנים נוספות שלא ידרשו נוגדנים משניים להגברת אותות. אם לא משתמשים בנוגדנים מצומדים ראשוניים, ניתן להוסיף כתמי גרעינים בשלב 1.12. - נערו את המאגר החוסם והשתמשו במגבון כדי להסיר את המאגר העודף מבלי לגעת ישירות ברקמה. הוסיפו לרקמה מספיק תמיסת צביעה שהוכנה בשלב 1.18 כדי לכסות אותה ולדגור בטמפרטורה של 4°C למשך 12-48 שעות.
- לשטוף את הרקמות כמתואר בשלבים 1.5-1.6. הוסיפו 5 טיפות של תושבת לכל מגלשה מבלי להכניס בועות, ובעדינות ובאיטיות הטו כיסוי זכוכית מעל הרקמה כדי לכסות את הרקמה לחלוטין בתושבת ולא לכלול את כל בועות האוויר. אחסן את השקופיות בטמפרטורה של 4°C בחושך עד שהן מוכנות לצילום.
2. הדמיה של חלקי הרקמה
הערה: בשלב זה, קטעי הרקמה מוכתמים בכל הנוגדנים המעניינים לכל התנאים המיועדים לצילום, ופקדים בצבע יחיד מוכתמים בפרמטר פלואורסצנטי אחד בכל קטע. הדמיה צריכה להיעשות על מיקרוסקופ שיש לו גלאים מרובים שניתן לכוונן כדי לזהות טווחים ספציפיים של אור, ובאופן אידיאלי, צריכה להיות לו גישה לכמה שיותר קווי לייזר לעירור.
- הכנס את אחת השקופיות המוכתמות במלואן למיקרוסקופ והגדר את המיקרוסקופ כך שיצלם את המקטע באמצעות המטרה המיועדת לאיסוף נתונים.
- בהתבסס על הפלואורופורים שהרקמה מוכתמת בהם, זהה את קווי לייזר העירור הדרושים כדי לעורר את אותם פלואורופורים וזהה את טווחי הפליטה עבור כל פלואורופור המכילים את כמות האות הגדולה ביותר.
הערה: כלים מקוונים לצפייה בספקטרום פלואורסצנטי (לדוגמה, עיין בטבלת החומרים) יכולים להיות שימושיים לזיהוי אורכי גל העירור האופטימליים של פלואורופורים ולזיהוי הטווחים האופטימליים לזיהוי פלואורופורים. יש לבחור פלואורופורים המשמשים לציטומטריית היסטופלואו כך שכל פלואורופור שונה בקו הלייזר האופטימלי שהפלואורופור מגורה על ידי או שונה בטווח האופטימלי של אורכי גל פליטה. פלואורופורים בעלי אורכי גל עירור / פליטה קרובים יותר יהיו קשים יותר להפרדה. הפלואורופורים המשמשים למחקר זה מפורטים בסעיף התוצאות. - פתח את תוכנה 1 (ראה טבלת חומרים) במיקרוסקופ קונפוקלי ובחר את כרטיסיית התצורה בחלק העליון. לאחר מכן בחר את לחצן החומרה בצד שמאל ושנה את עומק הסיביות ל- 16 סיביות.
הערה: מומלץ להשתמש בעומקי סיביות גבוהים יותר, כגון נתונים של 16 סיביות, לאיסוף נתונים, מכיוון שהדבר ישפר את דיוק הפיצוי הספקטרלי. בנוסף, צפיפות פיקסלים גבוהה יותר, מספרים גדולים יותר עבור ממוצעי קווים ושלבים רבים יותר בערימת z ישפרו את איכות ההפרדה הספקטרלית. - בחרו בכרטיסייה 'רכישה ' בחלק העליון של המסך והזינו את הגדרת העיצוב כדי לכלול את צפיפות הפיקסלים של התמונה (בדרך כלל, רזולוציות גבוהות יותר מפיקות תוצאות טובות יותר).
- אם אפשרות זו זמינה, בחר רכישת X דו-כיוונית כדי להאיץ את זמן ההדמיה, בתנאי שתכונה זו כוילתה כראוי.
- בחר ערך חור סיכה עבור המיקרוסקופ הקונפוקלי (בדרך כלל בין 400-600 ננומטר) שבו ערכים קטנים יותר יפיקו בדרך כלל תוצאות ציטומטריה היסטופלומית באיכות גבוהה יותר, אם כי ערכים קטנים יותר של חורי סיכה יקשו על קבלת תמונות ועשויים לדרוש זמן הדמיה נוסף או עוצמות עירור גבוהות יותר כדי להפיק תמונות.
- בחר בחלונית Show Sequential Scan בראש התפריט השמאלי כדי לפתוח את אפשרות הסריקה הרציפה. השתמש בסימן '+' כדי להגדיר רצפים נוספים, כאשר כל רצף יכיל אורך גל עירור אחד שהוחלט עליו בשלב 2.2.
- הפעל את כל הלייזרים הדרושים על ידי לחיצה על לחצני ON המשויכים ללייזרים בעלי עניין במרכז המסך כדי להפוך אותם למצב ON. היכנס לכל רצף ספציפי והגדל את עוצמת הלייזר במרכז המסך לכל ערך רצוי, כאשר לכל רצף יהיה אורך גל עירור אחד שישמש.
- הפעל גלאים רבים ככל הנדרש בחלק התחתון של המסך בכל רצף כדי לצלם את כל הפלואורופורים שיצולמו על קו לייזר זה. התאם את טווחי הגלאים על ידי לחיצה כפולה על הטווחים שלהם והזנת הטווחים העליונים והתחתונים של אורכי הגל שיש לאסוף.
הערה: מומלץ להשתמש בגלאים בעלי רגישות גבוהה במידת האפשר, ובאופן אידיאלי, יש לאסוף את כל הערוצים על אותם סוגי גלאים, אם כי הדבר אינו נדרש. - לחצו על כפתור השידור החי והתמקדו בדגימה. השתמש בלחצן קנה מידה אוטומטי משמאל לתמונה בתוכנה כדי להעריך אם ערוצים רוויים במידה מספקת באות. על המשתמש להתאים את ערכי הרווח ואת עוצמות הלייזר בכל רצף עד שקנה המידה האוטומטי אינו מגיע לערך המרבי (65535 עבור נתוני 16 סיביות) אך קרוב מאוד לרוויה עבור כל הערוצים.
הערה: מכיוון שפלואורופורים שונים יכולים להיות שונים בבהירותם ומטרות נוגדנים שונות יכולות להיות שונות בשפע שלהם, יהיה צורך להתאים את רגישות הגלאים כך שתהיה בעלת רגישות נמוכה לאותות בהירים ורגישות גבוהה לאותות עמומים אם שני הפלואורופורים מגורים על ידי אותו לייזר (עוצמת הלייזר אינה יכולה להשתנות בתרחיש זה מכיוון ששני הפלואורופורים יושפעו). לקבלת התוצאות הטובות ביותר, יש לאזן את האות הפלואורסצנטי מכל ערוץ בניסוי כך שלכל ערוץ יהיו כמויות אות שוות ערך, מה שיפחית את הדימום הספקטרלי לערוצים אחרים ויביא לאותות חזקים יותר. מכיוון שדגימות שונות עשויות להיות שונות בשפע הסמנים המעניינים המוכתמים, מומלץ לבדוק את הגדרות המיקרוסקופ על תנאי ניסוי מרובים כדי לאשר שהלבנה אינה מתרחשת באף אחד מתנאי הניסוי. - לאחר השלמת הגדרות המיקרוסקופ, טען את המיקרוסקופ עם שקופיות בקרת הצבע היחיד ומצא צביעה מייצגת עבור כל פלואורופור. הגדר ערימת Z על-ידי התמקדות מתחת לדגימה ולחיצה על לחצן התחל בתפריט ערימת Z, ולאחר מכן התמקדות מעל הדגימה ולחיצה על לחצן הסיום .
- בחר את מספר השלבים ואת המרחק בין השלבים (והשתמש באותו מספר שלבים ופרמטרים, כגון צפיפות פיקסלים ועומק סיביות שישמשו לצילום השקופיות המוכתמות במלואן) ולאחר מכן לחץ על לחצן התחל.
הערה: באופן אידיאלי, תמונות אלה צריכות לכלול את האותות הבהירים ביותר מצביעה אמיתית שניתן למצוא בשקופית. - לאחר צילום פקדי הצבע הבודדים, המשיכו להשתמש באותן הגדרות מיקרוסקופ כדי לצלם את כל הדגימות המוכתמות במלואן.
3. פיצוי פלואורסצנטי
- פתח קבצי .lif בתוכנה 1 ולחץ על כרטיסיית התהליך.
- בחרו במודול הפרדת הצבע ולאחר מכן בחרו באפשרות 'הפרדת צבע אוטומטית '. בחרו בשיטה הידנית להפרדת צבע ובחרו ללא שינוי גודל.
- פתח אחת מתמונות הבקרה בצבע יחיד. בדוק ידנית את בקרי הצבע הבודדים לאיתור גלישה ספקטרלית מערוץ אחד למשנהו על-ידי התבוננות בחלונות עם גווני אפור עבור כל ערוץ, כדי לראות עדות לאות בערוצים לא מתאימים. השתמש בלחצן קנה מידה אוטומטי במידת הצורך כדי לראות גלישה חלשה.
- התחל להסיר גלישה על-ידי הזנה ידנית של מספרים (בדרך כלל 0-1, כאשר 0 מייצג ללא גלישה ו- 1 מייצג 100% גלישה) למטריצה שעל המסך, ולאחר מכן הקש החל כדי לבדוק אם הגלישה הוסרה כראוי. יש לחזור על כך עד שהאותות בכל הערוצים האחרים למעט ערוץ העניין יצומצמו לרמות רקע.
הערה: פיצוי יתר יוביל לתוצאות לא מדויקות, ולכן יש להפחית את הפלואורסצנטיות לרמות רקע אך לא מתחתן. נתונים עם פיצוי יתר יופיעו כערוץ אחד שיש לו אות, וערוצים אחרים ייראו כבעלי חורים שחורים שבהם נמצא האות המעניין (איור 2). - לאחר השלמת אחד מהפקדים בצבע יחיד, רשום את הערכים במטריצה ולאחר מכן אפס את המטריצה ועבור לפקד הצבע היחיד הבא. חזור על תהליך זה עבור כל פקד צבע בודד.
הערה: הצבע הראשון יהיה בשורה Dye 1, וכמות הפלואורסצנטיות שיש להסיר מ- Dye 1 תהיה בעמודות הסמוכות. - רכז את כל הערכים המתקבלים מכל פקד למטריצה אחת והחל מטריצה זו על מדגם מוכתם במלואו. אם הפרדת הצבע נראית מדויקת, החילו את ההגדרות על כל התמונות בניסוי, אחרת, העריכו מחדש את בקרי הצבע הבודדים.
הערה: פיצוי אפקטיבי אמור להסיר את רוב הדימום בין הערוצים, כפי שמסוכם ומוצג באיור 3. - שמור את כל הקבצים המפצים בקובץ .lif נפרד.
4. זיהוי גרעינים ותאים באמצעות אילסטיק
- פתח את קבצי .lif המפצים ב- FIJI20. הפעל הצגת אוספי תמונות בהיפר-ערימה ובחר תמונה.
- (אופציונלי) השתמשו בכלי בחירה, כגון הכלי מלבן, כדי לכלול את חלקי התמונה שינותחו ולא לכלול אזורים שלא ינותחו. חתוך את התמונה באמצעות התמונה > פונקציית החיתוך.
הערה: הקטנת גודל התמונה שימושית בשלבים הבאים כדי להפחית את צריכת זיכרון הגישה האקראית (RAM) ולהגדיל את מהירות העיבוד. - שמור את התמונה כקובץ .tif. כמו כן, שמור את התמונה כקובץ hdf5 באמצעות תוסף ilastik עבור FIJI על ידי לחיצה על תוספים > ilastik > ייצוא hdf5. שמור את הקובץ כ"שם הבחירה".h5 עם דחיסה 0.
- פתח את Ilastik21 ועשה פרויקט סיווג פיקסלים. השתמש בפונקציה Add New > Add Separate images כדי להוסיף לפחות 3 תמונות מייצגות על-ידי הוספת נתוני קלט חדשים.
- לחץ לחיצה ימנית על אחת התמונות, בחר ערוך מאפיינים ושנה את מצב התצוגה לגווני אפור.
- בצד ימין של המסך, בחר בכרטיסיה בחירת תכונות . לחץ על בחר תכונות ובחר את כל התכונות עד לערך הסיגמא הגבוה ביותר שהתוכנית תאפשר לך להשתמש בו, ולאחר מכן לחץ על אישור.
- כעת בחר בכרטיסיה הדרכה. השתמשו בכלי מברשת צבע ( כשנבחרה תווית ראשונה ) כדי להדגיש תאים בודדים (איור 4) בעלי עניין, כך שהחלק הפנימי (ציטופלסמה וגרעין) וקרום התא ייכללו באור.
- חזור על כך בכל דגימות האימון שבהן נבחרים כל סוגי התאים המעניינים, ויש לבחור תאים מרובים בעוצמות, מורפולוגיות ולוקליזציה שונות ברקמה מכל דגימה.
הערה: בהתאם לסמנים שנבחרו עבור קבוצת כתמים בודדת, ייתכנו או לא יהיו כתמים תוך-תאיים או כתמי קרום התא עבור התאים המעניינים. לא משנה מה הסמנים משמשים, ודא כי האור כולל את הכתם החיצוני ביותר על התאים המעניינים וכולל את פנים התא ללא קשר אם כתם תוך תאי משמש. - לאחר מכן, השתמשו בתווית שתיים כדי להדגיש את כל מה שאינו התאים המעניינים (איור 4). זה יכול לכלול שטח ריק, גרעינים לא מסומנים, או רקמה שאין לה סמנים של עניין. הקפד לכלול את החלל הסמוך לתאים מעניינים כדי לקדם את הלמידה של גבולות התא המדויקים.
- השתמש בפונקציה Live Update כדי להעריך את איכות האימון בתמונות מעניינות. אם ההכשרה אינה מספקת, המשך את האימון בשלבים 4.7 עד 4.9. אחרת, אם משביע רצון, לחץ על הכרטיסייה ייצוא חיזוי .
- הגדר את המקור כהסתברויות. לחץ על הלחצן Choose Export Image Settings ולאחר מכן הגדר את הפרמטר c לייצוא 0-1 (בהנחה שתווית 1 מדגישה פרמטרים מעניינים).
- המר את סוג הנתונים למספר שלם של 16 סיביות, וייצא כקובץ hdf5 לספריית תחומי העניין עם שם העניין (מומלץ להשתמש ב- {nickname} mask.h5) ולאחר מכן הקש OK.
- לחץ על הכרטיסייה עיבוד אצווה . לחץ על לחצן בחר קבצי נתונים גולמיים , בחר את כל קבצי ה- .h5 המעניינים הדורשים עיבוד ולאחר מכן הקש פתח. לחץ על עבד את כל הקבצים.
- חזרו על שלבים 4.4 עד 4.13, אלא שבמקום לזהות תאים, כעת זהו את הגרעינים ולא כללו את כל השאר (איור 4). ייצא זאת כחיזוי נפרד לזיהוי גרעיני התא.
- פתח את Ilastik וצור פרויקט המרת נתונים חדש. הוסף קובץ HDF5 מייצג לכרטיסייה נתוני קלט באמצעות הפונקציה הוסף תמונה > חדשה והוסף תמונה נפרדת .
- לחץ על הכרטיסייה ייצוא נתונים ובחר את בחר הגדרות תמונה כפתור עם המקור מוגדר קלט. שנה את התבנית ל- tiff מרובה עמודים, בחר מיקום שמירה באמצעות פונקציית הבחירה והגדר את שם הקובץ ל- {nickname}. לחץ על אישור.
- לחץ על עיבוד אצווה הכרטיסייה ובחר את כל קבצי hdf5 שלך באמצעות בחר קבצי נתוני קלט כפתור ולחץ על עבד את כל הקבצים לחצן.
- פתח את תוכנה 2 (ראה טבלת חומרים) והוסף את כל קובצי tif/tiff שנוצרו בשלבים 4.3 ו- 4.17 באמצעות הלחצן הוסף קבצים . בחר מיקום עבור פלטי קבצים ולחץ על לחצן התחל הכל כדי להמיר את כל הקבצים לקבצי .ims. הקבצים מוכנים כעת לניתוח בתוכנה 3 (ראה טבלת חומרים).
5. ניתוח בתוכנה 3
- פתח את אחת התמונות המכילה את כל כתמי הפלואורסצנט. בחרו 'עריכה' >'הוסף ערוצים' והוסיפו את קובצי ה- .ims המתאימים למסיכות שנוצרו מעל גרעינים וגופי תאים. חזור על פעולה זו עבור כל הדגימות.
- בחר בלחצן הוסף תאים חדשים . בחר באפשרות זהה גרעין ותא והמשך קדימה.
- בחרו במסיכה שמעל גרעינים כערוץ המקור לגילוי גרעינים ובחרו באפשרות המתקדמת לפיצול גרעינים לפי נקודות זרע. בחר ערך לקוטר הגרעין) והמשך קדימה.
הערה: הערך האופטימלי לקוטר הגרעין יצטרך להיבדק על ידי הפעלת הסימולציה והערכה ידנית אם התוכנית אינה מפצלת מספיק גרעינים (גרעינים מרובים מקובצים יחד) או אם יש פיצול יתר (גרעין אחד מפוצל ליותר מאחד). - בחר ערך עבור הערכת האיכות ולאחר מכן המשך. בדרך כלל, עדיף לכלול את כל הגרעינים, שכן השמטת גרעינים יכולה להוביל לאיחוי תאים מרובים לאחד.
- בחר ערך סף ליצירת משטחים גרעיניים ולאחר מכן המשך. ודא שכל הגרעינים, גם אלה שהם עמומים יותר, עדיין כלולים בסף. אל תהפכו את הסף לנמוך מדי, מכיוון שהדבר עלול להעריך יתר על המידה את גודל הגרעינים או לכלול פסולת ככתמים גרעיניים.
- בחר ערך עבור מספר המספר המינימלי Voxels לכל גרעין והמשך. בדרך כלל, עדיף לשמור הכל אלא אם כן פסולת או שאינם גרעינים ניתן להוציא באמצעות פרמטר זה.
- בחרו באפשרות 'גבול תא' ו'ציטופלסמה ', בחרו במסיכה מעל התאים כערוץ המקור לזיהוי גופי תאים ולאחר מכן המשך.
- בחר סף תא אשר לוכד במדויק את כל התאים המעניינים אך אינו נמוך עד כדי כך שהוא כולל רקע או כל זיהוי לא ספציפי של תאים. בחר באפשרות לפצל תאים נוגעים כך שיהיה גרעין אחד לכל תא, והם מופרדים על ידי המרחק מגרעינים. כמו כן, בחר להרחיב תאים על גרעינים ולאחר מכן להמשיך.
- בחר ערך עבור המספר המינימלי של ווקסלים לכל תא והמשך. בדרך כלל, עדיף לשמור הכל אלא אם כן פסולת או שאינם תאים ניתן להוציא באמצעות פרמטר זה.
- הערך אם המשטחים שנוצרו מעל התאים מדויקים על-ידי התבוננות אם תאים מרובים אוחדו יחד או אם חסרים תאים. אם המשטחים אינם מספקים, חזור לשלב 5.2 ונסה פרמטרים חדשים עד לקביעת הגדרות אופטימליות.
- לאחר קביעת ההגדרות הטובות ביותר, לחץ על אובייקט התאים שנוצרו בתפריט השמאלי, לחץ על הכרטיסייה יצירה ולחץ על הלחצן אחסן פרמטרים עבור אצווה כדי לשמור את ההגדרות בשימוש.
- פתח חלונות מרובים של תוכנה 3 ופתח תמונות נפרדות בכל חלון. לחץ על לחצן התאים החדש בכל חלון, בחר את ההגדרות שנשמרו בתפריט פרמטרים של יצירה מועדפת והנחה כל חלון לפעול עד להשלמתו. חזור על פעולה זו פעמים רבות ככל הנדרש עד שכל הדגימות מנותחות.
הערה: הפעלת מופעים מרובים של Software 3 תגרום לניתוח זה להתקדם מהר יותר, אך מספר החלונות שניתן להפעיל במקביל יהיה תלוי במהירות העיבוד של יחידת העיבוד המרכזית (CPU) ובכמות ה- RAM שעליה פועל המחשב שבו הניתוח מופעל. - לאחר השלמת כל הדגימות, לחץ על אובייקט התאים שנוצרו בתפריט השמאלי בכל תמונה ולחץ על הכרטיסייה סטטיסטיקה. לחץ על הלחצן קבע תצורה של רשימת ערכי סטטיסטיקה גלויים , בטל את הבחירה בכל הנתונים הסטטיסטיים למעט הערך 'ממוצע עוצמת התא' ולאחר מכן לחץ על אישור.
- עבור כל תמונה שיש לנתח, לחץ על הלחצן יצא את כל הנתונים הסטטיסטיים לקובץ בכרטיסייה סטטיסטיקה ושמור את הקבצים במיקום.
6. ניתוח ציטומטריה היסטופלופלית ומיפוי אוכלוסיות על גבי מקטעי רקמה
- פתח את אנקונדה ואת JupyterLab בתוך אנקונדה. לאחר מכן, פתח את הסקריפט שסופק בקובץ קידוד משלים 1. הפעל את הקוד באמצעות תפריט הפעלה ובחר הפעל את כל התאים, או הפעל את התאים בנפרד לפי סדר על-ידי בחירה באפשרות הפעל תאים נבחרים.
- כאשר תתבקש להזין את קובץ המקור, הזן את ספריית הקבצים עבור הערכים הפלואורסצנטיים המיוצאים שנוצרו בשלב 5.14. ניתן להשיג את ספריית הקבצים על-ידי עיון במאפיינים של אחד מקבצי .csv המיוצאים והעתקת נתיב הקובץ לבקשה.
- לאחר מכן, התוכנית תבקש מיקום פלט עבור הקבצים המעובדים. הזן את ספריית הקבצים לכל מיקום המתאים לשמירת הקבצים המעובדים. התוכנית תזהה באופן אוטומטי את מספר ערוצי הפלואורסצנט הכלולים בתיקיה.
- הפעל את המקטע הבא של הקוד כדי להוסיף ביאורים לנתונים עם השמות של כל ערוץ. ניתן לשנות קוד זה לפי הצורך כדי לשנות את הפרמטר "numberOfChannels" כך שיתאים למספר הערוצים שזוהו בשלב 6.3. בנוסף, ניתן לשנות את שמות הערוצים עבור ערוצים רבים ככל שיידרש על ידי חזרה על השורה 'ChX':'Marker' כמה פעמים שצריך ועל ידי שינוי שמות הסמנים.
- הפעלת שורת הקוד הבאה תיצור קובץ .csv במיקום שצוין עם הערות ומכיל את כל הפרמטרים הפלואורסצנטיים בערכת הנתונים. חזור על שלבים 6.1-6.5 עבור כל קבוצה של ייצוא תוכנה 3 כדי ליצור קבצי .csv עבור כל הנתונים.
- המר את קבצי .csv לקבצי .fcs על-ידי פתיחת תוכנה 4 (ראה טבלת חומרים) וגרירת קבצי .csv לחלון התוכנית. תוכנה 4 תתחיל באופן אוטומטי להמיר את הקבצים לקבצי .fcs.
- לחץ פעמיים על אחת הדוגמאות בתוכנה 4 כדי לפתוח חלון במדגם זה. השתמש בכלי gating בתוכנה 4 כדי ליצור אסטרטגיית gating. מומלץ להתחיל בסימון מגדיר שושלת המכיל את כל התאים המעניינים, לאחר מכן להמשיך לשערים המסירים תחילה תת-קבוצות נדירות יותר, ולאחר מכן לסיים בסוגי התאים הנפוצים יותר.
- לאחר שנקבעה אסטרטגיית gating, בדוק את דיוק ה- gating על ידי לחיצה ימנית על אוכלוסייה זו בתפריט ובחירת ייצוא. יצא את כל הפרמטרים כקובץ .csv עם כותרות כלולות.
- פתח את JupyterLab באנקונדה. לאחר מכן, פתח את הסקריפט שסופק בקובץ קידוד משלים 2 והפעל את הקוד. כאשר תתבקש להזין את המיקום של קובץ Software 4, הזן את ספריית הקבצים עבור קובץ .csv המיוצא שנוצר בשלב הקודם. ניתן להשיג את ספריית הקבצים על-ידי עיון במאפיינים של קובץ .csv המיוצא והעתקת נתיב הקובץ לבקשה.
- כאשר תתבקש להוסיף את מיקום הפלט, בחר ספריית קבצים לפי בחירתך, וודא שספריית הקבצים כוללת את שם הקובץ בסוף ('file directory'\filename.txt). הפעל את שאר הקוד.
- העתק את הטקסט בקובץ .txt ופתח את קובץ ה- .ims המתאים בתוכנה 3 עבור אותה דוגמה שעליה מבוססים הנתונים המיוצאים. לחץ על אובייקט תא בקובץ, עבור לכרטיסייה סטטיסטיקה, הדבק את הטקסט בסרגל החיפוש והתחל את החיפוש. כל התאים המעניינים יודגשו.
- בדוק ידנית אם כל התאים המעניינים נלכדו באמצעות אסטרטגיית gating. אם מספר התאים אינו מוערך כראוי או מוערך יתר על המידה (דוגמאות מוצגות באיור 5), חזור לשלב 6.7, התאם את השער והערך מחדש אם התאים נלכדו במדויק באמצעות השער החדש. חזור על כך עם כל השערים באסטרטגיית ה- gating והערך את ה- gating על מספר דגימות.
- כאשר כל השערים מאומתים, יצא נתונים מתוכנה 4 באמצעות פונקציית עורך הטבלה ונתח את הנתונים באמצעות שיטת הניתוח המועדפת.
הערה: ניתן למפות אוכלוסיות תאים בחזרה למקטעי הרקמה בתוכנה 3 על-ידי זיהוי האוכלוסייה המגודרת כמו בשלבים 6.8-6.11 ולאחר מכן יצירת משטח המבוסס על התאים שנבחרו. ניתן לעשות זאת עבור כל אוכלוסייה מגודרת, כפי שניתן לראות באיור 6 ובאיור 7.
תוצאות
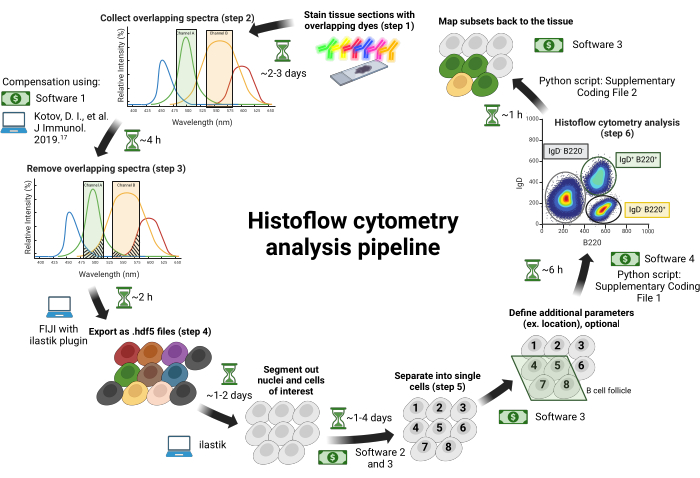
איור 1: זרימת עבודה של ציטומטריה של Histoflow. חלקי הרקמה מוכתמים בצבעים חופפים ספקטרלית (שלב 1). התמונות נאספות בלייזרים נפרדים של עירור בשילוב עם מסנני פסים מתכווננים כדי למזער את הדימום הספקטרלי ב?...
Discussion
כאן מתואר השימוש בציטומטריה של היסטופלו, טכניקה שאומתה בעבר18. הוכח כי בעת צביעת קטעי רקמה בצבעים חופפים ספקטרלית, ניתן להסיר את הדימום דרך ערוצים באמצעות פיצוי ספקטרלי, וכתוצאה מכך מספר רב יותר של פרמטרים פלואורסצנטיים נפתרים בבירור מאשר בדרך כלל מתאפשר בשיטות קונבנציונליות...
Disclosures
למחברים אין ניגודי עניינים כספיים.
Acknowledgements
אנו מודים לפלטפורמה המתקדמת למיקרוסקופיה של מכון המוח הוצ'קיס על תשתית הדימות והמומחיות. RWJ נתמך על ידי מימון מלגות פוסט-דוקטורט מתוכנית Eyes High של אוניברסיטת קלגרי ועל ידי האגודה לטרשת נפוצה של קנדה ומלגת רוש קנדה ללא הגבלה. VWY קיבלה תמיכה בשכר מתוכנית Canada Research Chair Tier 1. עבודה זו נתמכה על ידי קרנות תפעול של מענק מחקר הבריאות של המכונים הקנדיים 1049959, מענק 3236 של האגודה לטרשת נפוצה של קנדה, ומשרד ההגנה האמריקאי של תוכנית המחקר לטרשת נפוצה המכוונת על ידי הקונגרס. איור 1 נוצר באמצעות BioRender.com. הנתונים המותאמים בפרסום זה פורסמו במקור בכתב העת Journal of Immunology. רג'יב ו. ג'יין, דייוויד א. אליוט וו. ווי יונג. 2023. ניתוח תא בודד של תמונות היסטולוגיה בעלות פרמטרים גבוהים באמצעות ציטומטריה של Histoflow. י. אימונול. 210: 2038-2049. זכויות יוצרים © [2023]. האגודה האמריקאית לאימונולוגים, Inc.
Materials
| Name | Company | Catalog Number | Comments |
| 100% Ethanol | Sigma | 676829-1L | |
| 4% PFA | Electron Microscopy Sciences | 157-4 | |
| Anaconda | N/A | N/A | https://www.anaconda.com/download |
| Bovine Serum Albumin | Sigma | A4503-50G | |
| Cold fish stain gelatin | Sigma | G7765 | |
| Collating multichannel data from Imaris.ipynb script | N/A | N/A | https://github.com/elliottcalgary/Histoflow-Cytometry-Analysis- |
| Convert FlowJo output to txt file for Cell selection in Imaris.ipynb script | N/A | N/A | https://github.com/elliottcalgary/Histoflow-Cytometry-Analysis- |
| Donkey anti-rat Alexa Fluor 647 | JacksonImmunoResearch | 712-605-153 | 1:300 concentration |
| Donkey anti-rat DyLight 405 | Jackson ImmunoResearch | 712-475-153 | 1:200 concentration |
| Donkey Serum | JacksonImmunoResearch | 017-000-001 | |
| F(ab')2-Goat anti-Mouse IgG PerCP-eFluor 710 | Thermofisher | 46-4010-82 | 1:25 concentration |
| FIJI | N/A | N/A | https://imagej.net/software/fiji/ |
| FlowJo | FlowJo LLC | Software 4 | |
| Fluorescence spectraviewer | https://www.thermofisher.com/order/fluorescence-spectraviewer/#!/ | ||
| Fluoromount-G | Southern Biotech | 0100-01 | |
| Fresh frozen human tonsil sections | amsbio | HF-707 | |
| Glass coverslip | VWR | 48393 106 | |
| Goat anti-human IgA Alexa Fluor 488 | JacksonImmunoResearch | 109-546-011 | 1:400 concentration |
| Goat anti-human IgG Cy3 | JacksonImmunoResearch | 709-166-098 | 1:400 concentration |
| Goat anti-human IgM Dylight 405 | JacksonImmunoResearch | 109-476-129 | 1:300 concentration |
| Goat anti-rabbit A546 | Thermo Fisher Scientific | A-11035 | 1:250 concentration |
| Goat anti-rabbit IgG PE-Alexa Fluor 610 | Thermofisher | A-20981 | 1:250 concentration |
| Horse Serum | Sigma | H1138 | |
| Ilastik | N/A | N/A | https://www.ilastik.org/ |
| Ilastik FIJI plugin | N/A | N/A | https://www.ilastik.org/documentation/fiji_export/plugin |
| Imaris File Converter | Oxford Instruments | Software 2 | |
| Imaris with cell module | Oxford Instruments | Software 3 | |
| kimwipe | Kimtech | 34155 | |
| LasX Life Science software | Leica | Software 1 | |
| Mouse anti-human CD20 | VWR | CA95024-322 | 1:40 concentration |
| Mouse anti-human CD38 APC-R700 | BD Biosciences | 564980 | 1:20 concentration |
| Normal Goat Serum | JacksonImmunoResearch | 005-000-001 | |
| Normal Mouse Serum | JacksonImmunoResearch | 015-000-001 | |
| Normal Rabbit Serum | JacksonImmunoResearch | 011-000-001 | |
| Normal Rat Serum | JacksonImmunoResearch | 012-000-120 | |
| Nuclear Yellow | Abcam | ab138903 | Dissolve in DMSO at a concentration of 2 mg/ml and store at 4°C in the dark |
| PAP pen | Cedarlane | MU22 | |
| PBS | Gibco | 10010-023 | |
| Rabbit anti-human Ki67 | Abcam | ab15580 | 1:500 concentration |
| Rabbit anti-mouse Iba1 | Wako | 019-19741 | 1:500 concentration |
| Rat anti-human Blimp1 | Thermofisher | 14-5963-82 | 1:40 concentration |
| Rat anti-mouse B220 Alexa Fluor 647 | BioLegend | 103226 | 1:250 concentration |
| Rat anti-mouse CD138 | Biolegend | 142502 | 1:200 concentration |
| Rat anti-mouse CD3 PE-eFluor 610 | Thermo Fisher Scientific | 61-0032-82 | 1:40 concentration |
| Rat anti-mouse CD4 Alexa Fluor 488 | BioLegend | 100529 | 1:200 concentration |
| Rat anti-mouse CD45 allophycocyanin-R700 | BD Biosciences | 565478 | 1:50 concentration |
| Rat anti-mouse IgD PerCP-eFluor 710 | Thermo Fisher Scientific | 46-5993-82 | 1:50 concentration |
| SP8 Confocal microscope | Leica | ||
| Triton X-100 | Sigma | X100-500ml | |
| Trueblack | Biotium | 23007 | |
| Tween-20 | Sigma | P7949-500ml | |
| Ultracomp ebeads | Thermofisher | 01-2222-42 |
References
- Bar-Or, A., Li, R. Cellular immunology of relapsing multiple sclerosis: interactions, checks, and balances. Lancet Neurol. 20 (6), 470-483 (2021).
- Borst, K., Dumas, A. A., Prinz, M. Microglia: Immune and non-immune functions. Immunity. 54 (10), 2194-2208 (2021).
- Han, R. T., Kim, R. D., Molofsky, A. V., Liddelow, S. A. Astrocyte-immune cell interactions in physiology and pathology. Immunity. 54 (2), 211-224 (2021).
- Absinta, M., et al. A lymphocyte-microglia-astrocyte axis in chronic active multiple sclerosis. Nature. 597 (7878), 709-714 (2021).
- Piwecka, M., Rajewsky, N., Rybak-Wolf, A. Single-cell and spatial transcriptomics: Deciphering brain complexity in health and disease. Nat Rev Neurol. 19 (6), 346-362 (2023).
- Schirmer, L., et al. Neuronal vulnerability and multilineage diversity in multiple sclerosis. Nature. 573 (7772), 75-82 (2019).
- Jain, R. W., Yong, V. W. B cells in central nervous system disease: Diversity, locations and pathophysiology. Nat Rev Immunol. 22 (8), 513-524 (2022).
- Lassmann, H. Multiple sclerosis pathology. Cold Spring Harb Perspect Med. 8 (3), (2018).
- Yong, V. W. Microglia in multiple sclerosis: Protectors turn destroyers. Neuron. 110 (21), 3534-3548 (2022).
- Sharma, S., Boyer, J., Teyton, L. A practitioner's view of spectral flow cytometry. Nat Methods. 21 (5), 740-743 (2024).
- Longo, S. K., Guo, M. G., Ji, A. L., Khavari, P. A. Integrating single-cell and spatial transcriptomics to elucidate intercellular tissue dynamics. Nat Rev Genet. 22 (10), 627-644 (2021).
- Ramaglia, V., et al. Multiplexed imaging of immune cells in staged multiple sclerosis lesions by mass cytometry. Elife. 8, e48051 (2019).
- Bolognesi, M. M., et al. Multiplex staining by sequential immunostaining and antibody removal on routine tissue sections. J Histochem Cytochem. 65 (8), 431-444 (2017).
- Radtke, A. J., et al. IBEX: An iterative immunolabeling and chemical bleaching method for high-content imaging of diverse tissues. Nat Protoc. 17 (2), 378-401 (2022).
- Radtke, A. J., et al. IBEX: A versatile multiplex optical imaging approach for deep phenotyping and spatial analysis of cells in complex tissues. Proc Natl Acad Sci U S A. 117 (52), 33455-33465 (2020).
- Gerner, M. Y., Kastenmuller, W., Ifrim, I., Kabat, J., Germain, R. N. Histo-cytometry: A method for highly multiplex quantitative tissue imaging analysis applied to dendritic cell subset microanatomy in lymph nodes. Immunity. 37 (2), 364-376 (2012).
- Kotov, D. I., Pengo, T., Mitchell, J. S., Gastinger, M. J., Jenkins, M. K. Chrysalis: A new method for high-throughput histo-cytometry analysis of images and movies. J Immunol. 202 (1), 300-308 (2019).
- Jain, R. W., Elliott, D. A., Yong, V. W. Single-cell analysis of high-parameter histology images using histoflow cytometry. J Immunol. 210 (12), 2038-2049 (2023).
- JoVE Science Education Database. General Laboratory Techniques. Histological Sample Preparation for Light Microscopy. JoVE. , (2023).
- Schindelin, J., et al. Fiji: An open-source platform for biological-image analysis. Nat Methods. 9 (7), 676-682 (2012).
- Berg, S., et al. ilastik: Interactive machine learning for (bio)image analysis. Nat Methods. 16 (12), 1226-1232 (2019).
- Reichard, A., Asosingh, K. Best practices for preparing a single cell suspension from solid tissues for flow cytometry. Cytometry A. 95 (2), 219-226 (2019).
- Ruhlandt, D., et al. Absolute quantum yield measurements of fluorescent proteins using a plasmonic nanocavity. Commun Biol. 3 (1), 627 (2020).
- Combs, C. A., Shroff, H. Fluorescence microscopy: A concise guide to current imaging methods. Curr Protoc Neurosci. 79, 1-25 (2017).
- Elliott, A. D. Confocal microscopy: Principles and modern practices. Curr Protoc Cytom. 92 (1), e68 (2020).
- Jonkman, J., Brown, C. M., Wright, G. D., Anderson, K. I., North, A. J. Tutorial: Guidance for quantitative confocal microscopy. Nat Protoc. 15 (5), 1585-1611 (2020).
- McNally, J. G., Karpova, T., Cooper, J., Conchello, J. A. Three-dimensional imaging by deconvolution microscopy. Methods. 19 (3), 373-385 (1999).
- Stringer, C., Wang, T., Michaelos, M., Pachitariu, M. Cellpose: A generalist algorithm for cellular segmentation. Nat Methods. 18 (1), 100-106 (2021).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved