A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
تجارب الاسترخاء NMR 15N للتحقيق في الديناميكيات الهيكلية للبروتينات بيكو ثانية إلى نانو ثانية
In This Article
Summary
يمكن أن يميز التحليل الطيفي بالرنين المغناطيسي النووي (NMR) ديناميكيات البروتين الهيكلية بطريقة خاصة بالبقايا. نحن نقدم بروتوكولا عمليا لتسجيل استرخاء الرنين المغناطيسي النووي 15و N R1 و R2 و {1H}-15N تجارب تأثير Overhauser غير المتجانسة (hetNOE) ، الحساسة للمقياس الزمني للبيكو ثانية إلى النانو ثانية.
Abstract
يسمح التحليل الطيفي بالرنين المغناطيسي النووي (NMR) بدراسة البروتينات في المحلول وتحت درجات الحرارة الفسيولوجية. في كثير من الأحيان ، يتم استخدام مجموعات الأميد من العمود الفقري للبروتين أو مجموعات الميثيل في السلاسل الجانبية كمراسلين للديناميكيات الهيكلية في البروتينات. عادة ما تعمل دراسة الديناميكيات الهيكلية للعمود الفقري للبروتين للبروتينات الكروية على عينات 15نيوتن مصنفة وبروتونية بالكامل بشكل جيد للبروتينات التي يصل وزنها الجزيئي إلى 50 كيلو دالتون. عند تطبيق التحليل الطيفي المحسن للاسترخاء العرضي (TROSY) ، يمكن تمديد هذا الحد حتى 200 كيلو دالتون للبروتينات الكروية وما يصل إلى 1 MDa عندما يكون التركيز على السلاسل الجانبية. عندما يتم فحص البروتينات المضطربة جوهريا (IDPs) أو البروتينات ذات المناطق المضطربة جوهريا (IDRs) ، فإن قيود الوزن هذه لا تنطبق ولكن يمكن أن تتجاوز ذلك بكثير. والسبب هو أن النازحين داخليا أو IDRs، الذين يتميزان بالمرونة الداخلية العالية، غالبا ما يتم فصلهم ديناميكيا. تقدم طرق الرنين المغناطيسي النووي المختلفة رؤى ذات دقة ذرية لديناميكيات البروتين الهيكلية عبر مجموعة واسعة من المقاييس الزمنية ، من بيكو ثانية إلى ساعات. نظرة عامة على قياسات الاسترخاء القياسية 15N المرونة الداخلية للبروتين وتميز ديناميكيات العمود الفقري للبروتين التي يتم اختبارها على المقياس الزمني السريع من بيكو إلى نانو ثانية. تقدم هذه المقالة بروتوكولا عمليا لإعداد وتسجيل تجارب الرنين المغناطيسي النووي 15N R1 و R2 وتأثير Overhauser غير المتجانس (hetNOE). نعرض بيانات نموذجية ونشرح كيفية تفسيرها ببساطة نوعيا قبل أي تحليل أكثر تعقيدا.
Introduction
لا يتم تحديد وظيفة البروتين من خلال هيكله ثلاثي الأبعاد فحسب ، بل أيضا من خلال ديناميكياته الهيكلية ، بما في ذلك مرونته الداخلية والتحولات الهيكلية بين المطابقات المختلفة التي سيتبناها البروتين. يمكن للتحليل الطيفي بالرنين المغناطيسي النووي (NMR) التحقيق في الديناميكيات الهيكلية للبروتينات في المحلول1،2،3. تسمح التطورات الحديثة في الرنين المغناطيسي النووي للحالة الصلبة المكتشفة بالبروتون أيضا بتوصيف ديناميكيات البروتين في حالة أقل قابلية للذوبان ، مثل ، على سبيل المثال ، غشاء ثنائي الطبقة دهني4،5،6. في محلول الرنين المغناطيسي النووي ، يمكن دراسة الديناميكيات الهيكلية للعمود الفقري للبروتين والسلاسل الجانبية للبروتين. بالنسبة للبروتين الكروي ، يمكن تحقيق دراسة ديناميكية هيكلية للعمود الفقري للبروتين حتى 50 كيلو دالتون بمجرد أن يتم تصنيف البروتين 15نيوتن نظائريا. عند استخدام التحليل الطيفي المحسن للاسترخاء العرضي (TROSY) ، يمكن تمديد هذا الحد حتى 200 كيلو دالتون 7,8. عندما ينصب التركيز على ديناميكيات السلسلة الجانبية ، يمكن تمديد نطاق البروتينات والمجمعات التي يمكن الوصول إليها حتى 1 MDa2،9.
لا تنطبق قيود الوزن المسماة على البروتينات المضطربة جوهريا (IDPs) ، والتي تظهر في كثير من الأحيان ديناميكيات جوهرية عالية. أكثر من 30٪ من البروتين حقيقيات النواة يتألف من النازحين داخليا أو المناطق المضطربة جوهريا (IDRs) 10،11،12،13. يلعبون أدوارا مركزية في العديد من العمليات الخلوية ، مثل نقل الإشارات والنسخ1 ، وغالبا ما يشاركون في فصل الطور داخل الخلايا14،15،16،17. يفتقر النازحون داخليا إلى بنية أصلية ثلاثية الأبعاد (ثلاثية الأبعاد) محددة جيدا في ظل الظروف الفسيولوجية ولديهم مشهد طاقة ضعيف أو متين17،18. نظرا للكراهية المنخفضة للماء والتنافر الكهروستاتيكي القوي الموزع على العمود الفقري للنازحين داخليا أو IDRs ، فإن القوة الدافعة للطي إلى هيكل صلبمفقودة 19. وكثيرا ما يتبنى المشردون داخليا شكلا مطويا عندما يكون معقدا مع شركاء ملزمينآخرين 10،20،21. كما أن التعديلات اللاحقة للترجمة (PTMs) توسع إمكانيات طي النازحين داخليا أوIDRs 22،23. تم تحديد سوء التفكية للنازحين داخليا كسبب لأمراض مختلفة ، بما في ذلك الأمراض التنكسيةالعصبية 15 ، 24 ، 25 ، 26.
يظهر النازحون داخليا وIDRs مرونة داخلية عالية21،27،28. تم اشتقاق المجموعات التوافقية التي تعرض تباين المواضع الذرية والزوايا ثنائية السطوح من محاكاة الديناميكيات الجزيئية والقيود التي تم الحصول عليها من البياناتالتجريبية 29،30،31،32. نظرا للديناميكيات والاضطراب الناتج في الحالة المتجمدة ، فإن كثافة الإلكترون المنتشرة تجعل من الصعب توصيفها هيكليا باستخدام أحدث الأساليب في علم الأحياء الهيكلي ، مثل cryo-EM أو علم البلورات بالأشعة السينية. كما أن ظروف التبلور أو تقنيات تحضير العينات للتجارب في درجات حرارة مبردة قد تؤثر على المساحة التوافقية التي يعيشها النازحون داخليا. ومع ذلك ، فإن الحل بالرنين المغناطيسي النووي يعمل بشكل جيد مع البروتينات عالية الديناميكية ، وبالتالي فهو مناسب تماما للتحقيق في النازحينداخليا 16،20،22،28،29،30،31،32،33،34،35،36،37،38.
كما هو موضح أعلاه ، يقدم الرنين المغناطيسي النووي للحل تقنيات مختلفة لدراسة ديناميكيات البروتين الداخلية عبر مجموعة واسعة من المقاييس الزمنية (الشكل 1) ، بناء بشكل أساسي على استرخاء الدوران31،33،38،39،40،41،42.
يتم إحداث استرخاء الدوران لنوى 15N في مجموعات الأميد للعمود الفقري للبروتين من خلال تغيرات زاوية اتجاه رابطة 1 H-15N بسبب ديناميكيات البروتين الداخلية والحركات الجماعية (بما في ذلك ، عند الاقتضاء ، الانتشار الدوراني) 27،43،44،45،46،47،48،49،50،51. في النطاقات الزمنية الأقصر من وقت الارتباط الدوراني τR (الوقت الذي يحتاج فيه الجزيء إلى تدوير مادة مشعة واحدة ، يطلق عليه أيضا اسم وقت ارتباط الهبوط الكلي) ، يكون تباين التحول الكيميائي (CSA) والاقتران ثنائي القطب (D) نشطين ولا يتم قياسه في المتوسط من خلال الانتشار الدوراني للبروتين. تؤدي الديناميكيات الداخلية للعمود الفقري للبروتين ، والتي تشتمل على اختلافات في زوايا الرابطة ، وإعادة توجيه الروابط ، والهبوط الدوراني ، إلى حدوث تقلبات عشوائية في CSA وموتر اقتران ثنائي القطب ، مما يؤدي إلى تباين في المجال المغناطيسي المحلي ، مما يؤدي في النهاية إلى استرخاء دوران الرنين المغناطيسيالنووي 47،48،52،53. يمكن وصف هذه التقلبات من خلال دالة ارتباط شاملة. يسمى تحويل فورييه لدالة الارتباط الكلي دالة الكثافة الطيفية. في نظرية استرخاء ريدفيلد شبه الكلاسيكية ، يمكن وصف ثوابت معدل استرخاء الرنين المغناطيسي النووي من خلال مجموعات خطية من وظائف الكثافة الطيفية54.
تشتمل تجارب استرخاء العمود الفقري 15N NMR التي تم تطويرها في أوائل التسعينيات من 15N R1 و R1ρ و {1H}-15N تجارب تأثير Overhauser النووي ، الحساسة للمقياس الزمني السريع بيكو ثانية (ps) نانو ثانية (ns) ، أسرع من وقت الارتباط الدوراني τR للبروتين45،55،56،57. لتوصيف ديناميكيات العمود الفقري أبطأ من وقت الارتباط الدوراني τR ، يتم استخدام ما يسمى بتجارب تشتت الاسترخاء ، R1ρ ، و Carr-Purcell-Meiboom-Gill (CPMG) الحساسة لديناميكيات الميكروثانية (μs) - مللي ثانية (مللي ثانية)44،46،58،59،60،61 . يمكن التقاط الديناميكيات الأبطأ من الميكروثانية عن طريق نقل تشبع التبادل الكيميائي 15نيوتن (CEST) NMR62 ، أو التحليل الطيفي للتبادل (EXSY ، مللي ثانية إلى ثواني) ، أو الرنين المغناطيسي النووي في الوقت الحقيقي (RT) (من ثوان إلى ساعات) 63،64. يمكن استخدام تأثير PRE (تعزيز الاسترخاء المغناطيسي) للمجسات المغناطيسية ، بالإضافة إلى أدوات التوصيل ثنائية القطب المتبقية (RDCs) ، لتقييم النطاق الكامل لديناميكيات ps إلى ms65،66،67،68.
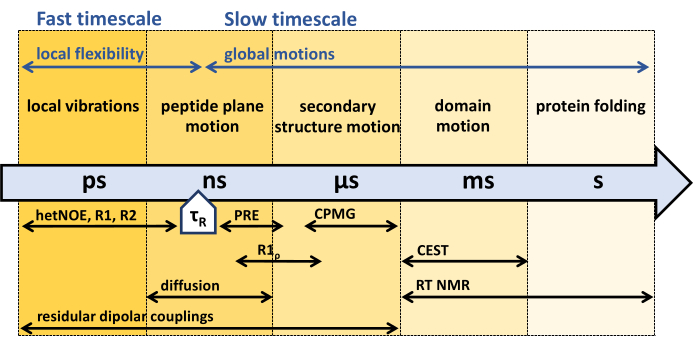
الشكل 1: المقاييس الزمنية لديناميكيات العمود الفقري للبروتين والنافذة الزمنية الحساسة لتجارب ديناميكيات الرنين المغناطيسي النووي المختلفة. يقدم الرنين المغناطيسي النووي مجموعة متنوعة من الطرق لتوصيف ديناميكيات العمود الفقري للبروتين عبر مجموعة واسعة من النطاقات الزمنية. يشار إلى الحركات المختلفة التي يتعرض لها العمود الفقري للبروتين في الجداول الزمنية الخاصة بها. وقت الارتباط الدوراني للبروتين ، τR ، هو الوقت الذي يحتاجه البروتين للدوران الكلي (بواسطة مشع واحد). يمكن أن ترتبط الحركات الأسرع من وقت الارتباط الدوراني للبروتين ، τR ، بالمرونة الداخلية للبروتين. يشار إلى تجارب الرنين المغناطيسي النووي المختلفة وحساسيتها للجداول الزمنية المعنية أسفل السهم. الرجاء النقر هنا لعرض نسخة أكبر من هذا الرقم.
يصف البروتوكول أدناه إعداد تجارب استرخاء الرنين المغناطيسي النووي بواسطة Lakomek et al.69 و Stief et al.70 ، باستخدام مخطط الكشف عن التماسك الكمي الأحادي غير المتجانس (HSQC) المعزز بالحساسية. قبل الشروع في التنفيذ التجريبي ، يتم تقديم نظرة عامة موجزة جدا على تجارب استرخاء الدوران بالرنين المغناطيسي النووي واسترخاء الرنين المغناطيسي النووي. نظرا لقيود الحجم وللحفاظ على هذا البروتوكول مفهوما ، يجب أن تظل هذه النظرة العامة مبسطة (وبالتالي غير مكتملة).
يصف الاسترخاء الطولي أو الشبكي الدوراني ، الذي يتميز بالوقت T1 أو ثابت معدل R1 = 1 / T1 ، عودة المغنطة إلى توازن بولتزمان. في حالة التوازن ، تتم محاذاة المغنطة على طول محور المجال المغناطيسي الخارجي ، والذي يحدد المحور z لإطار المختبر. تساهم الكثافة الطيفية عند ترددات Larmor العالية (1H) والصغيرة (15N) (ترددات رنين الرنين النووي ، على سبيل المثال ، 600 ميجاهرتز ل 1ساعة لمغناطيس 14.1 تسلا) والمجموعات الخطية لترددات Larmor هذه في استرخاء 15N R1 ، والذي يتميز بثوابت معدل 15N R1 المقاسة في rad·s-1. الحركات على المقاييس الزمنية معكوسة لترددات لارمور هذه. وبالتالي ، فإن الحركات على المقياس الزمني بيكو ثانية إلى نانو ثانية تساهم في ثابت معدل الاسترخاء R1. بالنسبة للجزيئات التي تظهر الهبوط الكلي وحيث يمكن تحديد وقت الارتباط الدوراني ، يظهر منحنى R1 (T1) الحد الأقصى (الحد الأدنى) ل ωτR = 1 ، مع الارتباط الدوراني τR وتردد Larmor ω للدوران قيد الدراسة. إذا ساهمت ترددات Larmor المتعددة ، فإن التردد الأقل هو التردد السائد ، على سبيل المثال ، ωN في حالة 15N R1. ينطبق نظام الحركة السريعة (ωτR أصغر بكثير من 1) على الجزيئات الصغيرة التي تتعثر بسرعة كبيرة وللمجالات المغناطيسية المنخفضة واللزوجة المنخفضة. نظام الحركة البطيئة (ωτR أكبر بكثير من 1) صالح للجزيئات الأكبر حجما التي تتعثر بشكل أبطأ وللمجالات المغناطيسية العالية واللزوجة العالية.
تظهر البروتينات المطوية الكروية الهبوط الكلي في المحلول ، ويمكن تعيين وقت ارتباط دوراني. ومع ذلك ، فإن مفهوم الهبوط الكلي لم يعد صالحا للبروتينات المضطربة جوهريا وغالبا ما يختلف عن تعيين وقت ارتباط دوراني واحد. هنا ، يصبح وقت الارتباط الداخلي الخاص بالبقايا أكثر أهمية.
يعتمد تسلسل النبض الموصوف الذي يقيس معدلات الاسترخاء 15N R1 (الشكل 2) على تجربة قراءة HSQC المحسنة بالحساسية مع اكتشاف الصدى / المضاد للصدى للكشف عن التربيع69،70،71. تستخدم التدرجات القصيرة ذات القوة والطول المتغيرين لاختيار التماسك وتحسين قمع المياه70. خلال ذلك الوقت ، سوف يرتاح الاستقطاب الطولي 15نيوتن. تؤدي أوقات الاضمحلال الأطول إلى انخفاض الشدة في المستويات ثنائية الأبعاد المرتبطة بهذا الطيف ثلاثي الأبعاد الزائف (يتم تسجيل نقاط بيانات التأخير في البعد الثالث). يتم تنفيذ عنصر الحلقة ، الموضح أدناه ، لعدد متزايد من المرات لأوقات الاسترخاء الأطول. نظرا لأن الاسترخاء المترابط بين تباين التحول الكيميائي 15نيوتن (CSA) والاقتران ثنائي القطب 1H و 15N (D) نشط أيضا أثناء تأخير الاسترخاء ، فإن نبضة I-BURP-2 180 درجةالمركزية 72 ، انتقائية على بروتونات الأميد ، ضرورية لإعادة تركيز المساهمة عن طريق الاسترخاء المترابط (والذي إذا لم يتم إعادة التركيز عليه سيؤدي إلى ثوابت معدل 15N R1 منحرفة وخاطئة).
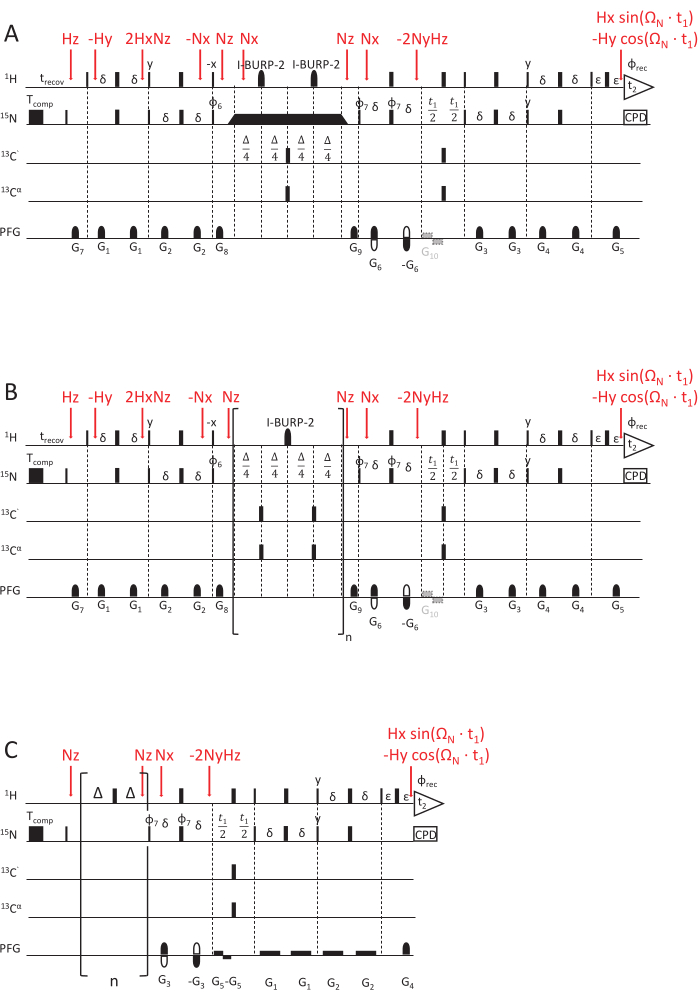
الشكل 2: مخططات تسلسل نبضات الرنين المغناطيسي النووي لتحديد ثوابت معدل استرخاء الرنين المغناطيسي النووي. (أ) 15N R1ρ ، (B) 15N R1 ، و (C) تجربة hetNOE ، باستخدام مخطط قراءة HSQC المعزز بالحساسية69،70. يتم تصور نبضات 90 درجة (x) بواسطة مستطيلات ضيقة ونبضات 180 درجة (x) بواسطة مستطيلات عريضة ، ما لم يذكر خلاف ذلك. يتم تطبيق دورة المرحلة التالية: φ6 = y ، y ، -y ، -y ؛ φ7 = y ، -y ، φrec = y ، -y ، -y ، y. يتم تحقيق الكشف عن التربيع عن طريق عكس قطبية التدرج G5 ودورة الطور φ7 (الكشف عن الصدى / المضاد للصدى). (أ) تجربة 15N R1ρ : يمثل المستطيل الأسود قفل الدوران ، والذي تختلف مدته للحصول على تأخيرات استرخاء مختلفة. تشير المثلثات قبل وبعد قفل الدوران إلى نبضات الشكل الثابت التي تقوم بمحاذاة المغنطة على طول محور المجال المغناطيسي الفعال Beff. G10 هو تدرج اختياري لمنع التخميد الإشعاعي لمغنطة الماء أثناء مرحلة التطور. (ب) تجربة 15NR 1 : يظهر الجزء الموضوع بين قوسين عنصر حلقة التسلسل ، يتكرر عدد n من المرات لمطابقة تأخير الاسترخاء المطلوب. (ج) يشبه مخطط نبض hetNOE النصف الثاني من مخططات النبض R1 و R1ρ ، أي وقت تطور t1 وعنصر الكشف عن HSQC. ومع ذلك ، فإن مغنطة 15نيوتن متحمسة مباشرة دون أي INEPT. يتم تحقيق تشبع مغنطة البروتون (لتحقيق الاسترخاء المتقاطع بين 1H و 15N) بواسطة قطار من 180 (1H) نبضة مطبقة لمدة 5 ثوان على الأقل. يتم تطبيق تأخير خامل بنفس الأطوال (هنا ، 5 ثوان) بدون أي قطار نبضي للتجربة المرجعية. G5 هو تدرج اختياري لمنع التخميد الإشعاعي ، والانعكاس في قطبية التدرج G4 ، بالاشتراك مع المرحلة φ7 = y ، -y ، -y ، يحقق الكشف التربيعي. يتم تمييز خطوات نقل المغنطة ، التي يمثلها مشغلو المنتج ، باللون الأحمر. الرجاء النقر هنا لعرض نسخة أكبر من هذا الرقم.
يصف ثابت معدل الاسترخاء R2 استرخاء الاستقطاب العرضي (في المستوى xy المتعامد مع المجال المغناطيسي الخارجي) بسبب فقدان تماسك الطور بين الدوران ، مما يؤدي إلى اضمحلال المغنطة القابلة للاكتشاف53،54. تساهم وظيفة الكثافة الطيفية عند الترددات العالية والصغيرة في R2 ، على غرار R1. ومع ذلك ، فإن أكبر مساهمة في R2 تنشأ من الكثافة الطيفية عند التردد الصفر. لهذا السبب ، R2 حساس جدا للهبوط الدوراني ، الموصوف بوقت الارتباط الدوراني τR ، والذي يكون في ترتيب بضعة نانوثانيات لبروتين كروي صغير في درجة حرارة الغرفة. وبالتالي ، فإن حركات العمود الفقري البطيئة في مئات ps إلى نظام ns المنخفض تساهم أكثر من غيرها. ديناميكيات التبادل للعمود الفقري التي تسبب تعديلا للجزء المتناحي من موتر التحول الكيميائي للنوى 15N ، من حيث المبدأ ، تضيف مساهمة التبادل R2ex إلى ثوابت معدل R2 43،44،49،60،61. في التجارب الموصوفة ، يتم قمع مساهمة R2ex من خلال قفل الدوران الذي يعيد تركيز الديناميكيات بشكل أبطأ من التردد الدائري العكسي لقفل الدوران. قفل الدوران عبارة عن نبضة ترددات راديوية طويلة ذات موجة مستمرة تحافظ على محاذاة المغنطة على طول محور المجال المغناطيسي الفعال Beff (مجموع المتجه لحقل قفل الدوران ω1 وإزاحة التحول الكيميائي من تردد الموجة الحاملة 15نيوتن (انظر أدناه)). يطلق على استرخاء المغنطة المحاذاة على طول المحور B1 ، eff استرخاء R1ρ ، والذي يحتوي على مكون R1 ومكون R2. باستخدام الصيغة (1) ، يمكن حساب R2 من R1ρ و R144,73:
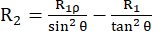 (1).
(1).
الزاوية بين محور المجال المغناطيسي الفعال Beff والمجال المغناطيسي الخارجي B0 هي 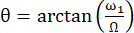 . ω1 هي سعة التردد اللاسلكي لقفل الدوران Ω إزاحة التحول الكيميائي بين التحول الكيميائي 15نيوتن للبقايا المقابلة وتردد الموجة الحاملة 15نيوتن 44،73.
. ω1 هي سعة التردد اللاسلكي لقفل الدوران Ω إزاحة التحول الكيميائي بين التحول الكيميائي 15نيوتن للبقايا المقابلة وتردد الموجة الحاملة 15نيوتن 44،73.
مخطط النبض R1ρ (الشكل 2 أ ، 70) مشابه جدا لمخطط 15NR 1 باستثناء تأخير الاسترخاء. لقياس معدلات الاسترخاء 15N R1ρ ، يجب أن يكون قفل الدوران نشطا بعد محاذاة المغنطة على طول محور المجال الفعال Beff بواسطة نبضة ثابتة اللهجة بنفس سعة التردد الراديوي (RF) مثل قفل الدوران. سيتم تغيير طول قفل الدوران للحصول على تأخيرات الاسترخاء المختلفة.
الحالة المستقرة {1H}-15N تأثير Overhauser النووي (1 H-15N NOE) ، الذي يطلق عليه اسم hetNOE في ما يلي ، هو نسبة معدل الاسترخاء المتقاطع ومعدل الاسترخاء الطولي 15نيوتن. يؤدي إلى تقليل استقطاب الحالة المستقرة على 15نيوتن بسبب الاسترخاء المتبادل مع البروتون عند تشبع استقطاب البروتون45،53،54،74،75. يعتمد الاسترخاء المتقاطع على دالات الكثافة الطيفية لمجموع وفرق ترددات 1H و 15N Larmor. لذلك ، فإن hetNOE حساس لكل من ديناميكيات بيكو ثانية السريعة (< 100 ثانية) وأيضا لديناميكيات ps-ns (بسبب اعتمادها على R1 ). يعتمد التسلسل69 (الشكل 2 ج) على قراءة HSQC المحسنة بالحساسية مع تدرجات الصدى / المضادة للصدى للكشف عن التربيع. لتشبع مغنطة البروتون و hetNOE الناتج ، يتم عكس مغنطة البروتون المتوازنة ثم تشبعها لاحقا عن طريق نبضات 180 درجة بسرعة لحوالي 5 أضعاف 15 N T1. بالنسبة للتجربة المرجعية ، فإن تأخير الاسترداد يساوي تأخير التشبع ولكن بدون قطار النبض 1H 180 درجة. يضاف تأخير إضافي قدره D1 = 2 ثانية للتجربة المرجعية والتجربة ذات التشبع 1H. يتم تسجيل كلتا التجربتين متتاليتين وتختلف فقط في تطبيق نبضات 1ساعة 180 درجة (التشبع) أو لا (مرجع). نسبة الشدة الطيفية المسجلة في التجربة مع تشبع 1H مقسومة على شدة التجربة المرجعية (بدون قطار نبض البروتون 180 درجة) تعطي قيمة {1H}-15N NOE (hetNOE).
يصف البروتوكول التالي إعداد تجارب استرخاء الرنين المغناطيسي النووي بواسطة Lakomek et al.69 و Stief et al.70. نحن نركز على تسلسل نبضات الرنين المغناطيسي النووي باستخدام مخطط الكشف عن HSQC المعزز بالحساسية. يتم تنفيذ تجارب 15N R1 و R1ρ كما هو موضح بالتفصيل بواسطة Stief et al.70 ، وتم وصف تجربة hetNOE بواسطة Lakomek et al.69.
Access restricted. Please log in or start a trial to view this content.
Protocol
1. تحضير عينة الرنين المغناطيسي النووي
ملاحظة: يتم إجراء وضع العلامات على النظائر للبروتينات لتجارب الرنين المغناطيسي النووي ذات الأبعاد الأعلى والرنين المغناطيسي النووي المتقدمة. عندما يتم إنشاء تعبير البروتين في الإشريكية القولونية وتنقية البروتين باستخدام الوسائط الغنية (على سبيل المثال ، Luria-Bertani [LB] أو 2x مستخلص الخميرة وسط التربتون [2YT]) بعائد عدة ملليغرام لكل لتر ، فإن تحضير عينة الرنين المغناطيسي النووي النظيرية عادة ما يكون بسيطا نسبيا.
- لوضع العلامات على النظائر ، استخدم وسائط M9 الدنيا للتعبير الجيني ، حيث يكون كلوريد الأمونيوم 15نيوتن هو المصدر الوحيد للنيتروجين و 13الجلوكوز المخصب ب C هو مصدر الكربون الوحيد.
- لتنقية البروتين ، اتبع البروتوكول القياسي المعمول به في المختبر للبروتين المعني.
ملاحظة: يعرض قسم النتائج نتائج تمثيلية ل IDP ، بروتين SNARE الأحادي Synaptobrevin-2 (1-96). تم إجراء وسم نظائر الرنين المغناطيسي النووي ل Synaptobrevin-2 كما هو موضح في70. بالنسبة للقياسات عالية الدقة جدا ، يوصى باستخدام تركيزات D2O منخفضة ، أقل من 5٪. هذا بسبب تأثير نظير الديوتيريوم ، والذي يمكن ملاحظته في Hahn-Echo 15N T2 وفي تجارب تشتت الاسترخاء منخفضة التردد 15N CPMG (لم تتم مناقشتها هنا) ولكن ليس له تأثير ضئيل فقط في تجارب 15N R1ρ (حيث يتم إعادة تركيزها بشكل أساسي) 76.
2. الاستعدادات لإجراء تجارب استرخاء الرنين المغناطيسي النووي على مقياس الطيف
ملاحظة: تجارب استرخاء الرنين المغناطيسي النووي الموصوفة خاصة بمطياف بروكر. تم اختبارها على مجسات الرنين الثلاثي المبردة ودرجة حرارة الغرفة 1ساعة و 15نيوتن و 13درجة مئوية ووحدات تحكم Avance III و Avance Neo التي يديرها برنامج Bruker Topsin 3.6 أو أعلى.
- اتبع الاحتياطات.
- قم بعمل إحاطة السلامة قبل العمل في مطياف الرنين المغناطيسي النووي.
- تحقق من مواصفات طاقة المسبار لمقياس الطيف المستخدم واختر طول النبضة وقوة النبضة المناسبة لكل نبضة ، على سبيل المثال ، من خلال مقارنة الاتساق مع إدخالات جدول Topspin "prosol".
- استيراد تسلسلات النبض.
- قم بتنزيل برامج نبض الرنين المغناطيسي النووي إما من موقع الويب الموضح أدناه أو من مستودع بنك الرنين المغناطيسي البيولوجي الموسع (BMRB) (bmrbig102) إلى الدليل /opt/topspin4.1.4/exp/stan/nmr/lists/pp/user للكمبيوتر الذي يقوم بتشغيل مطياف الرنين المغناطيسي النووي. يرجى ضبط إصدار topspin (هنا: topspin4.1.4 إلى الإصدار المعني المثبت على مقياس الطيف).
- قم بتنزيل النبضات على شكل رنين مغناطيسي إما من موقع الويب المشار إليه أدناه أو من مستودع بنك الرنين المغناطيسي البيولوجي الموسع (BMRB) (bmrbig102) إلى الدليل /opt/topspin4.1.4/exp/stan/nmr/lists/pp/user للكمبيوتر الذي يقوم بتشغيل مطياف الرنين المغناطيسي النووي. يرجى ضبط إصدار topspin (هنا: topspin 4.1.4 إلى الإصدار المعني المثبت على مقياس الطيف).
- معايرة مقياس الطيف والعينة.
- اضبط درجة حرارة الاهتمام. لمعايرة درجة الحرارة ، قم بتطبيق بروتوكولات المنشأة. على سبيل المثال ، استخدم عينة الميثانول d4 وقم بتفسير فرق التحول الكيميائي المرصود باستخدام مقياس حرارة الرنين المغناطيسي النووي لتطبيق الويب (https://chem.ch.huji.ac.il/nmr/software/thermometer.html) ؛ انظر أيضا77.
- أدخل العينة (بالإضافة إلى 5٪ D2O إضافية للقفل) وانتظر حوالي 10 دقائق للسماح للعينة بموازنة درجة حرارتها. قم بقفل إشارة الديوتيريوم (2H) (الأمر: قفل ، حدد H2O + D2O).
- قم بمطابقة وضبط القنوات 1H و 15N و 13C ، على سبيل المثال ، باستخدام روتين atma أو atmm. إذا لم يتم تثبيت وحدة مطابقة وضبط تلقائية، فقم بمطابقة المسبار وضبطه يدويا باستخدام الأمر wobb.
- قم بأداء الحشوة لحقل B0 متجانس ، على سبيل المثال ، باستخدام روتين topshim tg.
- معايرة طول النبضة 90 درجة (1ساعة).
- استخدم تجربة zg. استخدم getprosol لاستيراد معلمات المسبار. اختر مدة قصيرة تبلغ 5 ميكرو ثانية ل P1. تحقق من مستوى الطاقة المحدد للمسبار وصححه ل PLW1 إذا لزم الأمر.
- ابدأ التجربة مع zg. معالجة البيانات باستخدام EFP. اضبط المرحلة باستخدام.درجة الحموضة بحيث يكون خط الأساس متساويا وتكون إشارة الرنين المغناطيسي النووي لرنين الماء في طور
- قم بتقدير نبضة 360 درجة (1ساعة) عن طريق أخذ نبضة شائعة أو تقريبية 90 درجة (1ساعة) (على سبيل المثال ، من مواصفات مقياس الطيف المدرجة) وضربها في معامل أربعة. قم بتشغيل التجربة ومعالجتها.
ملاحظة: المدة المختارة طويلة جدا إذا كانت الإشارة موجبة وقصيرة جدا إذا كانت سالبة. ستكون النبضة 360 درجة (1ساعة) هي العبور الصفر. - اضبط طول النبضة وفقا لذلك. افعل ذلك بشكل متكرر لإيجاد تقاطع الصفر. اقسم القيمة التي تم الحصول عليها على أربعة. سيكون هذا هو النبض 90 درجة (1ساعة).
ملاحظة: بديل لطريقة 360 درجة الموصوفة هو روتين pulscal ، والذي يمكن أن يبدأ من سطر الأوامر ويسمح بمعايرة نبضة 1H الآلية.
- معايرة تردد الموجة الحاملة O1 إلى رنين الماء.
- انسخ تجربة zg عن طريق كتابة edc في سطر الأوامر إلى رقم تجربة جديد. اكتب rpar لفتح مجموعات المعلمات (الشكل التكميلي 1 أ).
- ابحث عن مجموعة المعلمات ZGPR. انقر فوق مجموعة المعلمات ZGPR (الشكل التكميلي 1 أ) ، وانقر فوق قراءة ، واختر تنفيذ "getprosol" وانقر فوق موافق (الشكل التكميلي 1 ب).
- قم بتغيير P1 إلى طول النبضة المعاير 90 درجة (1ساعة). اختر طول التشبع D1 = 2 ثانية وقوة نبضة PLW9 تبلغ 50 ديسيبل. اضبط كسب جهاز الاستقبال (rg) على 16. بدلا من ذلك ، قم بتنفيذ ضبط كسب المستقبل عن طريق كتابة rga وضبط كسب المستقبل على القيمة المحددة (ولكن تجنب "تجاوز كسب المستقبل").
- اكتب gs في سطر الأوامر لبدء الاستحواذ المتكرر والمستمر. انتقل إلى نافذة الاستحواذ وقم بتغيير O1 لتقليل منطقة FID التي تهيمن عليها إشارة الماء (الشكل التكميلي 2).
- انقر أسفل العلامة لتقليل O1 ، وانقر فوق العلامة لزيادتها. استخدم زر الحساسية لتغيير حجم الخطوة. حفظ O1 الحالي ؛ يوقف Close الاستحواذ المتكرر (الشكل التكميلي 2).
- معايرة طول النبضة 90 درجة (15نيوتن).
- انسخ تجربة zg إلى رقم تجربة جديد (اكتب edc). اكتب rpar لقراءة مجموعة المعلمات HSQCFPF3GPPHWG لتغيير برنامج النبض إلى تجربة 1 H-15N HSQC. استخدم خيار الاحتفاظ بالمعلمات P1 و O1 و PLW1 (الشكل التكميلي 1).
- اكتب getprosol 1H <معاير 90 درجة (1ساعة) طول النبضة في μs> <90 درجة (1H) قوة النبض بوحدة ديسيبل> في سطر الأوامر لاستيراد معلمات المسبار. اضبط عدد عمليات الفحص (NS) على 8 وعمليات المسح الوهمية (DS) إلى 2.
ملاحظة: يحدد < > هنا القيم المراد تعيينها. على سبيل المثال: getprosol 1H 8.5 -11.79. - قم بتشغيل الزيادة الأولى للتجربة عن طريق كتابة zg في سطر الأوامر. انسخ الزيادة الأولى إلى رقم تجربة جديد باستخدام rser 1 <رقم التجربة الجديدة>. معالجة البيانات باستخدام efp ووضع الطيف بشكل أساسي باستخدام.درجة الحموضة. استخدم الزيادة الأولى (تجربة 1D) للتجربة للخطوات التالية.
ملاحظة: تحدد < > الرقم المختار للتجربة الجديدة، على سبيل المثال، rser 1 101. - اكتب ased في سطر الأوامر لفتح معلمات تسلسل النبض لتجربة 1D. انقر فوق E في سطر PULPROG (انظر الشكل التكميلي 3 أ) لفتح محرر برنامج النبض.
- استخدم سير العمل التالي لحفظ تسلسل النبض تحت اسم جديد: انقر فوق ملف (الزاوية العلوية اليسرى)، حفظ باسم، أدخل الاسم الجديد، ثم انقر فوق موافق. ثم انقر فوق تعيين PULPROG إلى مجموعة البيانات (انظر الشكل التكميلي 3 ب) وانقر فوق موافق.
- قم بتعديل تسلسل 1 H-15N HSQC الأصلي (المحفوظ تحت اسم جديد ، انظر 2.6.4.) بضرب 90 درجة (15نيوتن) بعد فترة تطور t1 (قبل النقل الخلفي) بعامل 2 (النبضة 90 درجة (15نيوتن) هي الآن 180 درجة (15نيوتن)). اضغط على Ctrl S لحفظ تسلسل النبض. اضبط طول النبضة على 90 درجة (15نيوتن) (P21) بحيث تختفي إشارة الرنين المغناطيسي النووي للزيادة الأولى (عند معبر الصفر) (الشكل التكميلي 3 ب).
- افتح تجربة 1 H-15N HSQC (تجربة ثنائية الأبعاد) وأدخل طول النبضة المعاير 90 درجة (15نيوتن) (P21). اكتب getprosol 1H <معايرة 90 درجة (1ساعة) طول النبضة بقوة النبضة μs> <90 درجة (1H) بقوة النبضة بوحدة ديسيبل> 15N <معايرة 90 درجة (15نيوتن) بقوة النبضة بوحدة ديسيبل >> <90 درجة (15نيوتن ) في سطر الأوامر.
- تحديد العرض الطيفي وتخصيص أوقات الاستحواذ.
- في نافذة معلمة الاستحواذ لتجربة 1 H-15N HSQC (اكتب eda في سطر الأوامر) ، اضبط العرض الطيفي (معلمة الاستحواذ SW) وعدد زيادات الأبعاد بالأبعاد المباشرة وغير المباشرة (معلمة الاستحواذ TD).
- اختر SW ، 16 صفحة في الدقيقة في البعد المباشر (1ساعة) و 30 صفحة في الدقيقة في البعد غير المباشر (15نيوتن). اختر TD ، 2048 في البعد المباشر (1ساعة) و 512 في البعد غير المباشر (15نيوتن).
- احصل على تجربة 1 H-15N HSQC.
- تحقق من اكتشاف جميع الإشارات ذات الأهمية وعدم وجود عناصر مستعارة في الطيف. قم بتغيير O3P إذا كان مركز الطيف لا يتناسب مع مركز توزيع إشارة البروتين على طول المحور 15N.
- قم بزيادة SW إذا كانت النافذة الطيفية صغيرة جدا. قم بزيادة TD للحصول على دقة FID أعلى (تحقق من مواصفات المسبار لمعرفة أقصى مدة للفصل).
ملاحظة: هذا هو الإعداد العام ونقطة البداية لإعداد تجارب استرخاء الرنين المغناطيسي النووي. يوصى بتسجيل تجربة 1 H-15N HSQC في بداية ونهاية مجموعة التجارب بأكملها ، وبالنسبة للأوقات التجريبية الممتدة ، أيضا بينهما لمراقبة سلامة العينة.
- في نافذة معلمة الاستحواذ لتجربة 1 H-15N HSQC (اكتب eda في سطر الأوامر) ، اضبط العرض الطيفي (معلمة الاستحواذ SW) وعدد زيادات الأبعاد بالأبعاد المباشرة وغير المباشرة (معلمة الاستحواذ TD).
3. تنفيذ تجارب استرخاء الرنين المغناطيسي النووي
ملاحظة: تتوفر تسلسلات نبضات استرخاء الرنين المغناطيسي النووي (الشكل 2) في https://www.ipb.hhu.de/en/teams/team-lakomek/pulsesequences أو في مستودع بنك الرنين المغناطيسي البيولوجي الموسع (BMRB) (bmrbig102).
- قم بإعداد 15ن ر1ρ التجربه.
- انسخ تجربة HSQC (اكتب edc) إلى دليل جديد. اكتب ased وقم بتغيير برنامج النبض إلى تجربة 15N R1ρ من خلال النقر على النقاط الثلاث لخط المعلمة PULPROG في معلمات تسلسل النبض (ased ، انظر الشكل التكميلي 3 أ). انقر نقرا مزدوجا فوق برنامج النبض ، وانقر فوق تعيين PULPROG على مجموعة البيانات (انظر الشكل التكميلي 3 ب) ، وانقر فوق موافق.
ملاحظة: قد تظهر بعض رسائل الخطأ. أغلق هذه الرسائل. - اكتب gpnam في سطر الأوامر لإدراج جميع أشكال التدرج المفقودة (gpnam1- gpnam9) كما هو موضح في تسلسل النبض (الشكل التكميلي 4).
- اكتب vplist في سطر الأوامر. انقر فوق النقاط الثلاث (الشكل التكميلي 5 أ) ، والملف (الزاوية اليسرى العليا) ، والجديد (الشكل التكميلي 5 ب).
- اكتب اسما ل vplist وهمي جديد ، واضغط على موافق. قم بإنشاء vplist الوهمي (5 م ، 65 م ، 25 م ، 45 م ، 15 م ، 55 م ، 35 م ، 10 م ؛ صف جديد لكل إدخال vplist). اضغط على Ctrl S لحفظ القائمة وإغلاق نافذة vplist (الشكل التكميلي 5C).
- انقر نقرا مزدوجا فوق اسم vplist الذي تم إنشاؤه حديثا (الشكل التكميلي 5 ب).
ملاحظة: هذا ضروري لتجنب أخطاء التجميع في تسلسل النبض. سيتم تحديد الإدخالات المناسبة ل vplist أدناه (النقطة 3.1.29). - اكتب ased لفتح معلمات تسلسل النبض (الشكل التكميلي 6). أدخل جميع نقاط قوة التدرج وأطوال التدرج المفقودة كما هو موضح في تسلسل النبض (الشكل التكميلي 6 د).
- أدخل عداد الحلقة L3 (عدد النقاط المعقدة في البعد غير المباشر) 1/2 من إدخال 15N TD في نافذة معلمة الاكتساب (eda). أدخل في عداد الحلقة L6 عدد نقاط بيانات الاسترخاء المسجلة المستخدمة لتركيب منحنى الاسترخاء.
ملاحظة: يتوافق هذا الرقم L6 مع عدد إدخالات vplist ، هنا: 8 (الشكل التكميلي 6 أ). - اضبط نبضة TanhTan_half.nl الثابتة، التي تم تنزيلها من موقع الويب المحدد أعلاه، على شكل نبضة 15N SP8. اضبط نبضة ثابت الحرارة TanhTan_2nd.nl من موقع الويب على شكل نبضة 15N SP9 (الشكل التكميلي 6C).
- اضبط مدخل P8 على طول نبضة TanhTan الثابت للحرارة - طويلة بما يكفي لضمان سدادية النبض ؛ هنا يتم اختيار P8 = 3000 ميكرو ثانية (الشكل التكميلي 6C).
- اضبط فصل 15نيوتن (CPDPRG 3) على مخطط فصل garp. استخدم لطول فصل PCPD3 200 ميكرو ثانية (الشكل التكميلي 6C).
- اضبط تأخير الاسترداد بين المسح الضوئي على D1 على الأقل = 2 ثانية أو أكثر.
ملاحظة: لاختيار تأخير الاسترداد المناسب، راجع قسم المناقشة. - اضبط عمليات المسح الوهمية (DS) على 64 على الأقل للتجربة النهائية (أثناء الإعداد ، DS = 4 كافية لأسباب زمنية). كنقطة انطلاق ، استخدم عددا من عمليات المسح (NS) من 4 ؛ استخدم مضاعفات 4 إذا كانت الإشارة إلى ضوضاء (SNR) منخفضة جدا (الشكل التكميلي 6 أ).
- اضبط O1 على تردد الموجة الحاملة المعايرة للخطوة 2.5. اضبط O2P على 176 صفحة في المليون وانسخ O3P من تجربة 1 H-15N HSQC (الشكل التكميلي 6A-C).
- اضبط طول النبضة P7 على طول النبضة 90 درجة (15نيوتن) الذي تمت معايرته في الخطوة 2.6. أيضا ، انسخ مستوى طاقة النبضة للنبضة 90 درجة (15نيوتن) من الخطوة 2.6 إلى PLW3 و PLW7 (الشكل التكميلي 6C).
- اضبط طول النبضة P1 و P19 على طول النبضة 90 درجة (1ساعة) المحسوب في الخطوة 2.4. (الشكل التكميلي 6 ب).
- قم بتعيين عدد الزيادات في البعد غير المباشر TD = l3 * 2 * l6 (eda أو اكتب td في سطر الأوامر).
ملاحظة: يتم إضافة تعليقات توضيحية إلى جميع معلمات تسلسل النبض الموضحة هنا في تسلسل النبض. - اضبط نبضة الشكل SP5 على شكل I-BURP2 (Iburp2.1000). اضبط طول النبضة P15 على 2000 ميكرو ثانية. افتح شاشة أداة الشكل بالنقر فوق E بجوار نبضة الشكل I-BURP2 في نافذة معلمة تسلسل نبض Topspin (ased) (الشكل التكميلي 6 ب).
- قم بمحاكاة نبضة الشكل بالطول الصحيح بالنقر فوق الزر بدء محاكاة الرنين المغناطيسي النووي. تحقق من طول النبضة المشكل [μs] (= 2000 ميكرو ثانية) وزاوية الدوران [°] (= 180 درجة) في نافذة المحاكاة. انقر فوق بدء الرنين المغناطيسي النووي (الشكل التكميلي 7 أ).
- تحقق من نطاق الإثارة واختر طول نبضة I-BURP2 المناسب لتغطية التشتت الطيفي للبروتين 1H ولكن تجنب إثارة الماء في نفس الوقت. اضبط P15 على طول النبضة على شكل نافذة المحاكاة مع محاكاة أفضل نبضة IBURP-2 (الشكل التكميلي 7 ب).
ملاحظة: يتم اختيار الطول المناسب إذا كان I-BURP2 يثير (فعالية > 95٪) منطقة الأميد بأكملها. لمحاكاة نبضة I-BURP2 بطول مختلف ، أغلق نافذة ملف تعريف الإثارة وقم بتحرير طول النبضة المشكل في نافذة المحاكاة. انقر فوق بدء تشغيل NMR-SIM. النبضات ذات الطول الأقصر لها نطاق إثارة أوسع بالهرتز. - اضبط SPOFFS5 لتغيير تردد الموجة الحاملة لنبضة I-BURP2 وتحويل نطاق الإثارة إلى اليسار أو اليمين لتجنب ملامسة نبضة I-BURP2 وإزعاج مغنطة الماء (الشكل التكميلي 6 ب).
- لتحديد مستوى الطاقة المناسب للنبضة المشكلة، افتح أداة الشكل Bruker، وانقر فوق الزر بدء محاكاة الرنين المغناطيسي النووي، واضبط طول النبضة I-BURP2 على طول النبضة على شكل طول النبضة [μs]. تذكر الطول المقابل لنبضة مستطيلة ناعمة 90 درجة (1ساعة) ، مرئية في نافذة المحاكاة عند النبضة المربعة المقابلة 90 درجة [μs] (الشكل التكميلي 7 أ).
- اكتب calcpowlev. استخدم النبضة الصلبة 90 درجة (1ساعة) (P1) بالميكروثانية كطول مرجعي (الشكل التكميلي 8 ب) والنبضة المستطيلة الناعمة 90 درجة (1ساعة) المقابلة لنبضة I-BURP2 بالميكروثانية كطول جديد (الشكل التكميلي 8 أ). تذكر فرق القدرة Δ في ديسيبل بين النبضة الصلبة 90 درجة (1ساعة) والنبضة المستطيلة الناعمة (الشكل التكميلي 8C). انسخ قوة النبضة للنبضة الصلبة 90 درجة (1ساعة) في ديسيبل إلى SPW5 وأضف الفرق المحفوظ Δ في ديسيبل (الشكل التكميلي 6 ب).
- لتحديد مستوى طاقة قفل الدوران ، احسب طول النبضة المقابل 90 درجة (15نيوتن) لقفل الدوران. استخدم الصيغة: 90 درجة (15نيوتن) طول النبضة في s = 0.25 / (قوة قفل الدوران بالهرتز).
- استخدم calcpowlev لحساب فرق القدرة Δ بالديسيبل بين قوة قفل الدوران والنبضة الصلبة 90 درجة (15نيوتن) (P7). جديد: 90 درجة (15نيوتن) طول النبضة لقفل الدوران في μs (الشكل التكميلي 8 أ). المرجع: طول النبضة الصلبة 90 درجة (15نيوتن) بميكرو ثانية (الشكل التكميلي 8 ب). انسخ مستوى القدرة PLW7 للنبضة الصلبة 90 درجة (15نيوتن) بوحدة ديسيبل إلى مستوى طاقة قفل الدوران PLW8 (الشكل التكميلي 6C) ، وأضف فرق القدرة المحسوب Δ بوحدة ديسيبل (الشكل التكميلي 8C).
- انسخ مستوى الطاقة لقفل الدوران PLW8 إلى مستويات الطاقة SPW8 و SPW9. (الشكل التكميلي 6C).
- لتحديد مستوى القدرة لفصل 15نيوتن ، استخدم calcpowlev لحساب فرق القدرة Δ بين قوة نبضة الفصل 90 درجة (15نيوتن) (PCPD3) والنبضة الصلبة 90 درجة (15نيوتن) (P7). جديد: PCPD3 في μs (200) (الشكل التكميلي 8 أ). المرجع: طول النبضة الصلبة 90 درجة (15نيوتن) بميكرو ثانية (الشكل التكميلي 8 ب). انسخ مستوى القدرة PLW7 للنبضة الصلبة 90 درجة (15نيوتن) بوحدة ديسيبل إلى مستوى الفصل PLW31 (الشكل التكميلي 6C) وأضف Δ بوحدة ديسيبل (الشكل التكميلي 8C).
- لاستخدام تعويض درجة الحرارة، قم بتضمين #define TEMP_COMPENSATION (عن طريق حذف الفاصلة المنقوطة في تسلسل النبض). اضبط P18 على المدة القصوى لقفل الدوران المستخدم في تجربة 15N R1ρ .
ملاحظة: يوصى بإيقاف تشغيل تعويض درجة الحرارة للتجربة الأولى من التجربة. - في حالة العينات المسماة 15N 13C ، قم بتضمين # لتحديد LABEL_CN في برنامج النبض (عن طريق حذف الفاصلة المنقوطة في تسلسل النبض). اضبط P4 على طول النبضة المحسوب بالصيغةفي 78. على سبيل المثال ، يقابل 23.70 ميكرو ثانية عند 600 ميجاهرتز و 11.85 ميكرو ثانية عند 1200 ميجاهرتز. اكتب calcpowlev لحساب فرق القدرة بالديسيبل بين P4 والنبضة الصلبة (13درجة مئوية). انسخ طاقة النبضة الصلبة (13درجة مئوية) بوحدة ديسيبل إلى PLW4 وأضف Δ بوحدة ديسيبل. انسخ PLW4 إلى PLW2 (الشكل التكميلي 6 ب).
- تحديد تأخيرات الاسترخاء لأخذ العينات المناسبة.
- قم بتشغيل أول ثمانية FIDs (في حالة L6 = 8) وقم بمعالجتها (rser <رقم FID> < رقم التجربة الجديدة>). اختر الإدخالات في vplist التي تنخفض فيها كثافة ذروة التجربة ذات التأخير الأطول إلى 1 / e على الأقل (حوالي 37٪) ، ولكن ليس أقل من 25٪ مقارنة بالتجربة ذات التأخير الأقصر (والتي تعمل كمرجع).
- اضبط الإدخالات المتبقية من vplist لتحقيق أخذ عينات متساوية من انخفاض الشدة بين FID مع أطول تأخير و FID مع أقصر تأخير. اقرأ الملاحظة ومواصفات الطاقة الخاصة بالمسبار.
ملاحظة: ستهيمن البقايا التي تسترخي ببطء على أول FID. اضبط الإدخالات المتبقية من vplist لتحقيق أخذ عينات متساوية من انخفاض الشدة بين FID الأول والثاني. لا تتجاوز الحد الأقصى المسموح به لقوة قفل الدوران والطول المسبار (راجع دليل المسبار ومواصفات المسبار). في حالة تعويض درجة الحرارة ، تحقق من P18 (الحد الأقصى لمدة قفل الدوران) ، حيث سيتم تطبيق المدة الإجمالية لقفل الدوران P18 ، مقسمة بين تأخير الاسترخاء وتعويض درجة الحرارة. إنه يحمل الوقت (تأخير الاسترخاء) + الوقت (تعويض درجة الحرارة) = const = P18. بالنسبة لقفل الدوران 2 كيلو هرتز ، نفضل عدم تجاوز مدة قفل الدوران التي تزيد عن 65 مللي ثانية ، باستخدام تأخير الاسترداد (D1) البالغ 2 ثانية. في هذه الحالة ، يجب اتخاذ حل وسط بين الحد الأقصى لمدة قفل الدوران وأطول تأخير للاسترخاء لصالح الحد الأقصى لمدة قفل الدوران المسموح بها. في حالة تأخير استرداد D1 لفترة أطول، قد تكون قوى قفل الدوران أطول قابلة للتطبيق. ما يهم هو ترسيب الطاقة الكلي في المسبار بمرور الوقت. إذا كانت هناك حاجة إلى فترات أطول لقفل الدوران ، فقم فقط بزيادة أطوال قفل الدوران في خطوات صغيرة وراقب إشارة القفل. كقاعدة عامة ، اضبط مستوى القفل في الثلث العلوي من شاشة القفل. إذا انخفض مستوى القفل بأكثر من مربعين أثناء تنفيذ تسلسل النبض ، فإن قوة قفل الدوران المودعة في المسبار تكون أكثر من اللازم.
- اكتب rga في سطر الأوامر لتحديد كسب جهاز الاستقبال. للحصول على نسبة إشارة إلى ضوضاء جيدة ، استهدف قيمة كسب المستقبل (rg) بين 128 و 256 ، مما يثبت قمع الماء بشكل جيد.
ملاحظة: لا يمكن الوصول إلى كسب جهاز الاستقبال بين 128 و 256 يشير إلى إشارة مائية قوية جدا. وفي هذه الحالة، تعدل قدرة التدرج والمدد بلطف حتى يتم الوصول إلى كسب المستقبل. انظر أيضا قسم المناقشة. - ابدأ تشغيلا تجريبيا لتجربة 15N R1ρ عن طريق كتابة zg في سطر الأوامر. تأكد من أن إشارة الماء مكبوتة جيدا لجميع التأخيرات (من 1 إلى 8 ، في حالة حدوث 8 تأخيرات في vplist). تحقق أيضا من الزيادة 9 ، وهي الزيادة الثانية في مخطط الكشف التربيعي (Echo / Anti-Echo).
- انسخ تجربة HSQC (اكتب edc) إلى دليل جديد. اكتب ased وقم بتغيير برنامج النبض إلى تجربة 15N R1ρ من خلال النقر على النقاط الثلاث لخط المعلمة PULPROG في معلمات تسلسل النبض (ased ، انظر الشكل التكميلي 3 أ). انقر نقرا مزدوجا فوق برنامج النبض ، وانقر فوق تعيين PULPROG على مجموعة البيانات (انظر الشكل التكميلي 3 ب) ، وانقر فوق موافق.
- قم بإعداد تجربة 15NR 1 .
- انسخ تجربة 15N R1ρ وقم بتغيير برنامج النبض إلى تجربة 15N R1 .
- اكتب vclist ، وقم بإنشاء vclist ، وأدخل vclist الوهمي الموصوف في70: 0 ، 24 ، 6 ، 20 ، 4 ، 16 ، 8 ، 12 ؛ صف جديد لكل إدخال VCLIST).
- فتح ased. تحقق من أطوال التدرج ونقاط القوة. قم بتغيير GPZ9 إلى 14٪.
- تحقق من أطوال النبضة وأشكال النبض وقوة النبض (يجب نسخ القيم الصحيحة من تجربة 15N R1ρ ).
- في حالة 15N13C العينات المسماة ، استخدم #define LABEL_CN في برنامج النبض.
- لاستخدام تعويض درجة الحرارة ، قم بتضمين #define TEMP_COMPENSATION.
- قم بتشغيل أول ثمانية FIDs (في حالة L6 = 8) وقم بمعالجتها (rser <رقم FID> < رقم التجربة الجديدة>). اختر الإدخالات في vclist التي تنخفض فيها ذروة شدة التجربة ذات التأخير الأطول إلى 1 / e على الأقل (حوالي 37٪) ولكن ليس أقل من 25٪ مقارنة بالتجربة ذات الأقصر تأخير. اضبط الإدخالات المتبقية من vplist لتحقيق أخذ عينات متساوية من انخفاض الشدة بين FID مع أطول تأخير و FID مع أقصر تأخير.
ملاحظة: يسمح فقط بالأرقام الزوجية لإدخالات vclist.
- قم بإعداد تجربة {1H}-15N NOE.
- انسخ تجربة الاسترخاء 15N R1 وقم بتغيير تسلسل النبض إلى مخطط نبض {1H}-15N NOE.
- اضبط طول فترة التشبع باستخدام عداد الحلقة L8. التباعد بين نبضات 180 درجة (1ساعة) هو 22 مللي ثانية79 (2 * 1 / JNH). لتحقيق أدق النتائج ، استخدم فترة تشبع تبلغ حوالي 5 أضعاف البروتين 15N T1 (15N). هنا: 5 ث. هذا يتوافق مع L8 = 230.
ملاحظة: يقدم قسم المناقشة نصائح إضافية حول اختيار فترة التشبع / تأخير الاسترداد. - قم بتغيير طول وقوة التدرجات كما هو موضح في مخطط النبض. استخدم sine.20 أشكال متدرجة.
- اضبط عدد الزيادات في البعد غير المباشر يساوي TD = l3 * 2 * 2
- في حالة العينات المسماة 15N13C ، استخدم #define LABEL_CN و -DLABEL_CN ك ZGOPTNS.
- قم بتشغيل مخططات النبض عن طريق كتابة zg في سطر الأوامر.
4. معالجة وتحليل تجارب الرنين المغناطيسي النووي المسجلة
ملاحظة: تم تسجيل الأطياف باستخدام نظام بروكر. تتم المعالجة باستخدام نظام تشغيل Unix أو Linux. تم إجراء معالجة الأطياف وتحليل البيانات باستخدام NMRPipe80 و python3. يمكن تنزيل برنامج NMRPipe من https://www.ibbr.umd.edu/nmrpipe/index.html. يتم تنزيل البرامج النصية للمعالجة المستندة إلى NMRPipe من موقع الويب: https://www.ipb.hhu.de/en/teams/team-lakomek/pulsesequences أو من مستودع بنك الرنين المغناطيسي البيولوجي الموسع (BMRB) (bmrbig102). يوصى باستخدام NMRPipe. إذا لم يكن NMRPipe متاحا أو مرغوبا فيه ، فيمكن استخدام بدائل مثل CCPN81 أو SPARKY (SPARKY 3 ، أو خلفائها NMRFAM-SPARKY82 أو POKY83 ).
- المعالجة والتحليل 15ن ر1 التجربه.
ملاحظة: يتم تنفيذ الأوامر التالية في وحدة Unix الطرفية.- انسخ الملفات الموجودة في الدليل الذي تم تنزيله R1_processing إلى الدليل الذي يحتوي على بيانات R1 التجريبية (دليل العمل).
- اجعل الملفات قابلة للتنفيذ بواسطة الأمر الطرفي: > chmod u + x * .com
- قم بتنفيذ > bruker لفتح NMRPipe Conversion Utility وإنشاء ملف fid.com. انقر فوق قراءة المعلمات. قم بتغيير عدد الأبعاد إلى اثنين إذا تم حساب ثلاثة أبعاد، ثم انقر فوق قراءة المعلمات مرة أخرى. ثم انقر فوق مسح البرنامج النصي وتحديث البرنامج النصي وحفظ البرنامج النصي (الشكل التكميلي 9).
ملاحظة: إذا كان تردد الموجة الحاملة غير صحيح، فقم بتغيير الموضع المركزي PPM بعد النقر فوق قراءة المعلمات في المرة الثانية. - تنفيذ > ./fid.com
- قم بتحويل vclist في vplist مع إدخالات ms : تأخير في ms = 40 ms * إدخال vclist. استخدم > cp vclist vplist وقم بتحرير vplist.
- استخدم > ./split.com لتقسيم البيانات ثلاثية الأبعاد الزائفة إلى ثمانية ملفات ثنائية الأبعاد.
- قم بتحرير tauValues (قيم vplist بترتيب vplist) وقيم idxExpmt (ترتيب tauValues فيما يتعلق بالوقت) في nmrproc.com (الشكل التكميلي 10).
- استخدم > ./nmrproc.com لمعالجة البيانات الأولية لجميع الأطياف ثنائية الأبعاد الثمانية.
- استخدم > nmrDraw لرسم الأطياف.
- استخدم الطيف المرجعي (الإدخال الأول في vplist) لهذه الخطوات. قم بالتبديل إلى هذا الطيف عن طريق تغيير البعد z إلى واحد (الزاوية اليسرى العليا). اضبط مستوى الكفاف باستخدام + و - بجوار زر العامل . أعد رسم الطيف باستخدام الزر رسم.
- استخدم مستوى كفاف حيث يمكن التعرف على جميع الإشارات ذات الأهمية مع عدد قليل من الإشارات القادمة من الضوضاء (مستوى الكفاف عند حافة مستوى الضوضاء) (الشكل التكميلي 11).
- قم بتصحيح طور الطيف ، من النوع v لإسقاط إشارة 1D الرأسي و h لإسقاط إشارة 1D الأفقية. اضبط المرحلة باستخدام P0 و P1 (الشكل التكميلي 11) ، وتذكر فرق الطور ، وقم بتصحيح المرحلة في ملف nmrproc.com (الشكل التكميلي 10).
ملاحظة: إذا انقلب الطيف في البعد 15نيوتن ، فقم بإزالة -neg في السطر: | nmrPipe -fn FT -neg \ (الشكل التكميلي 10). - أغلق nmrDraw وقم بمعالجته مرة أخرى باستخدام > nmrproc.com. بالنسبة لانتقاء الذروة (بعد أن يتم تقسيم الطيف بشكل صحيح وعرض الطيف المرجعي بمستوى كفاف حيث توجد جميع القمم) ، اكتب K (shift + k) ، واضغط على اكتشاف ، وانقر فوق حفظ. (الشكل التكميلي 11).
- انسخ قائمة الذروة إلى دليل العمل وقم بتغيير -assName في ass.com إلى اسم قائمة الذروة (الشكل التكميلي 12).
ملاحظة: يتوفر مثال على قائمة الذروة بالتنسيق الصحيح على https://www.ipb.hhu.de/en/teams/team-lakomek/pulsesequences
وفي مستودع بنك الرنين المغناطيسي البيولوجي الموسع (BMRB) (bmrbig102). - قم بتنفيذ > ./ass.com لتعيين الأطياف. تحقق من التعيين الذي يقترحه البرنامج النصي للقمم المختلفة. استخدم زر الماوس الأيسر للتعيين وزر الماوس الأيمن لإلغاء تعيين القمم. استخدم الزر التالي للانتقال إلى الحمض الأميني التالي و Prev للانتقال إلى الحمض الأميني السابق (الشكل التكميلي 13).
ملاحظة: يمكن تغيير مستوى الكفاف باستخدام + و - في الزاوية اليمنى العليا. استخدم الزر رسم لإعادة رسم الطيف بمستوى الكفاف الجديد (الشكل التكميلي 13). - بعد الانتهاء من الواجب، انقر فوق حفظ ثم حفظ مرة أخرى. بعد ذلك ، انقر فوق إنهاء وإلغاء وإنهاء (الشكل التكميلي 13).
- قم بتنفيذ > python3 NoneDeleter.py لإنشاء relax.tab ، والذي يحتوي فقط على معلومات حول القمم المعينة.
- قم بتنفيذ > ./relax.com لإنشاء البرنامج النصي autofit.com.
- قم بتنفيذ > ./autoFit.com لإجراء استيفاء فورييه وإنشاء نسب زائفة لأوقات التأخير المختلفة لكل حمض أميني (يتم إنشاء axt.tab و nlin.tab ).
- قم بتنفيذ > ./model.com لإنشاء البرنامج النصي modelExp.com استنادا إلى البيانات الموجودة في nlin.tab.
- قم بتنفيذ > ./modelExp.com لتناسب منحنى التطور لكل بقايا باستخدام نموذج تحسين المربعات الصغرى غير الخطي.
- قم بتنفيذ > bash summary.tcl -in relax.tab > t1.tab. تجاهل رسالة خطأ محتملة: إشعار GDB: يمكن أن تحدث 21 قيمة مفقودة في ملف الجدول .
- انسخ t1.tab إلى دليل تم إنشاؤه حديثا R1R2.
- معالجة وتحليل تجربة 15N R1ρ .
- انسخ الملفات الموجودة في R1rho_processing الدليل الذي تم تنزيله إلى الدليل الذي يحتوي على بيانات R1ρ التجريبية (دليل العمل).
- تنفيذ > chmod u + x * .com
- قم بتنفيذ > bruker لفتح NMRPipe Conversion Utility وإنشاء ملف fid.com. انظر أيضا معالجة أطياف 15N R1 .
- تنفيذ > ./fid.com
- قم بتنفيذ > ./split.com لتقسيم البيانات ثلاثية الأبعاد الزائفة إلى ثمانية ملفات ثنائية الأبعاد.
- قم بتنفيذ > ./nmrproc.com (قم بتحرير tauValues [قيم vplist] و idxExpmt مسبقا) لمعالجة البيانات الأولية لجميع الأطياف ثنائية الأبعاد الثمانية (الشكل التكميلي 10).
- قم بتنفيذ > nmrDraw لرسم الأطياف واختيار القمم. استخدم الطيف المرجعي (الإدخال الأول في vplist) لهذه الخطوات. اتبع نفس الإجراء المتبع في تجربة 15NR 1 .
- انسخ قائمة الذروة إلى دليل العمل وقم بتغيير -assName في ass.com إلى اسم قائمة الذروة (الشكل التكميلي 12).
- قم بتنفيذ > ./ass.com واتبع نفس الإجراء المتبع في تجربة 15NR 1 .
- قم بتنفيذ > python3 NoneDeleter.py لإنشاء relax.tab ، والذي يحتوي فقط على معلومات حول القمم المعينة.
- تنفيذ > ./relax.com
- تنفيذ > ./autoFit.com
- تنفيذ > ./model.com
- تنفيذ > ./modelExp.com
- قم بتنفيذ > bash summary.tcl -in relax.tab > t1rho. علامة التبويب. تجاهل رسالة الخطأ المحتملة: إشعار GDB: قد تحدث 21 قيمة مفقودة في ملف الجدول .
- انسخ الرأس التالي في ملف t1rho.tab (يمكن الحصول على القيم من ملف fid.com) (الشكل التكميلي 14):
ملاحظة 600 ميجا هرتز ل T1rho
ملاحظة -yOBS = 60.818 (ضع هنا القيمة الموجودة في fid.com)
ملاحظة -yCAR = 119.06 (ضع هنا القيمة الموجودة في fid.com) - قم بتنفيذ > ./extract_PPM.com لإنشاء جدول يسمى fusionOffset.tab يحتوي على إزاحة ذروة تردد 15نيوتن بالنسبة لتردد الموجة الحاملة (والتي ستكون هناك حاجة لحساب الزاوية θ لاستخراج بيانات R2 من بيانات R1 و R1ρ ).
- لخص بيانات الاسترخاء.
- انسخ t1rho.tab و fusionOffset.tab في الدليل R1R2.
- انسخ البرنامج النصي crtR1p.tcl في الدليل R1R2 ، واضبط تردد مقياس الطيف وقوة قفل الدوران ، وقم بتنفيذ bash crtR1p.tcl | sort -n > R1R2.tab. (الشكل التكميلي 15). سيحتوي R1R2.tab على ثوابت معدل R1ρ المجهزة ، وزاوية ثيتا ، وثوابت معدل R2 ، وثوابت معدل R1 .
- معالجة وتحليل تجربة hetNOE.
- انسخ الملفات الموجودة في الدليل الذي تم تنزيله NOE_processing إلى دليل بيانات NOE التجريبي (دليل العمل).
- تنفيذ > chmod u + x * .com
- قم بتنفيذ > bruker لفتح NMRPipe Conversion Utility وإنشاء ملف fid.com. انقر فوق قراءة المعلمات. قم بتغيير عدد الأبعاد إلى اثنين إذا تم حساب ثلاثة أبعاد واضغط على قراءة المعلمات مرة أخرى. ثم اضغط على مسح البرنامج النصي وتحديث البرنامج النصي وحفظ البرنامج النصي.
- تنفيذ > ./fid.com
- تنفيذ > ./split.com (يجب إنشاء vplist مع إدخالين (0 ، 1) من قبل)
- تنفيذ > ./ft2.com
- قم بتنفيذ > nmrDraw لرسم الطيف واختيار القمم في الطيف المرجعي (z-dimension = 1). استخرج الضوضاء وتذكرها (اضغط على T = shift + t). يتبع تصحيح الطور وانتقاء الذروة نفس الإجراء مثل تجربة 15NR 1 .
- انسخ قائمة الذروة إلى دليل العمل وقم بتغيير -assName في ass.com إلى اسم قائمة الذروة (الشكل التكميلي 12)
- قم بتنفيذ > ./ass.com لتعيين الأطياف. اتبع نفس الإجراء كما هو مفصل لتجربة 15NR 1 .
- قم بتنفيذ > python3 NoneDeleter.py لإنشاء relax.tab.
- تنفيذ > ./script.com
- تنفيذ > ./autoFit.com
- قم بتغيير التشويش في error_hetnoe.tcl.
- تنفيذ > bash error_hetnoe.tcl |sort -n > hetNOE.tab
Access restricted. Please log in or start a trial to view this content.
النتائج
يوضح ما يلي بعض بيانات استرخاء الرنين المغناطيسي النووي النموذجية المسجلة على بروتين SNARE الحويصلي Synaptobrevin-2 (1-96) ، والذي يطلق عليه غالبا VAMP2 (البروتين المرتبط بالحويصلة 2). لتسجيل بيانات الرنين المغناطيسي النووي ، استخدمنا عينة 171 ميكرومتر 15نيوتن Synaptobrevin-2 (1-96) (يطلق عل...
Access restricted. Please log in or start a trial to view this content.
Discussion
وصف هذا البروتوكول إعداد تجارب الاسترخاء NMR 15N بواسطة Lakomek et al.69 و Stief et al.70. ركزنا على تسلسل نبضات الرنين المغناطيسي النووي باستخدام مخطط الكشف عن HSQC المعزز بالحساسية. يتم تنفيذ تجارب 15N R1 و R1ρ كما هو موضح بالتفصيل بواسطة Stief...
Access restricted. Please log in or start a trial to view this content.
Disclosures
يعلن المؤلفون أنه ليس لديهم مصالح مالية متنافسة معروفة أو علاقات شخصية يمكن أن يبدو أنها تؤثر على العمل المبلغ عنه في هذه الورقة.
Acknowledgements
نشكر ميليندا جاسبرت وكيفن بوتشينسكي على المناقشات المفيدة. تشكر N.L. مؤسسة العلوم الألمانية على التمويل من خلال برنامج هايزنبرغ (منحة DFG رقم 433700474). يتم دعم هذا العمل أيضا من خلال مشروع "المحددات الفيروسية والمناعية للتسبب في COVID-19 - دروس للاستعداد للأوبئة المستقبلية (KA1-Co-02 "COVIPA") ، وهي منحة من صندوق المبادرات والشبكات التابع لجمعية هيلمهولتز. نحن نقر بالوصول السخي إلى مركز الرنين المغناطيسي النووي الجزيئي الحيوي في جوليش دوسلدورف ، الذي يديره بشكل مشترك Forschungszentrum Jülich وجامعة Heinrich Heine في دوسلدورف (HHU).
Access restricted. Please log in or start a trial to view this content.
Materials
| Name | Company | Catalog Number | Comments |
| Bruker 600 MHz AVANCE III HD spectrometer | Bruker | https://www.bruker.com/en/products-and-solutions/mr/nmr/avance-nmr-spectrometer.html | NMR experiments conducted |
References
- Sekhar, A., Kay, L. E. An NMR view of protein dynamics in health and disease. Annu Rev Biophys. 48, 297-319 (2019).
- Rosenzweig, R., Kay, L. E. Bringing dynamic molecular machines into focus by methyl-TROSY NMR. Annu Rev Biochem. 83, 291-315 (2014).
- Palmer, A. G. 3rd Enzyme dynamics from NMR spectroscopy. Acc Chem Res. 48 (2), 457-465 (2015).
- Reif, B. Ultra-high resolution in MAS solid-state NMR of perdeuterated proteins: implications for structure and dynamics. J Magn Reson. 216, 1-12 (2012).
- Schanda, P., Ernst, M. Studying dynamics by magic-angle spinning solid-state NMR spectroscopy: Principles and applications to biomolecules. Prog Nucl Magn Reson Spectrosc. 96, 1-46 (2016).
- Le Marchand, T., et al. (1)H-H-detected biomolecular NMR under fast magic-angle spinning. Chem Rev. 122 (10), 9943-10018 (2022).
- Pervushin, K., Riek, R., Wider, G., Wuthrich, K. Attenuated T2 relaxation by mutual cancellation of dipole-dipole coupling and chemical shift anisotropy indicates an avenue to NMR structures of very large biological macromolecules in solution. Proc Natl Acad Sci U S A. 94 (23), 12366-12371 (1997).
- Tzakos, A. G., Grace, C. R., Lukavsky, P. J., Riek, R. NMR techniques for very large proteins and RNAs in solution. Annu Rev Biophys Biomol Struct. 35, 319-342 (2006).
- Sprangers, R., Kay, L. E. Quantitative dynamics and binding studies of the 20S proteasome by NMR. Nature. 445 (7128), 618-622 (2007).
- Wright, P. E., Dyson, H. J. Intrinsically disordered proteins in cellular signalling and regulation. Nat Rev Mol Cell Biol. 16 (1), 18-29 (2015).
- van der Lee, R., et al. Classification of intrinsically disordered regions and proteins. Chem Rev. 114 (13), 6589-6631 (2014).
- Tompa, P. Intrinsically unstructured proteins. Trends Biochem Sci. 27 (10), 527-533 (2002).
- Dyson, H. J., Wright, P. E. Coupling of folding and binding for unstructured proteins. Curr Opin Struct Biol. 12 (1), 54-60 (2002).
- Uversky, V. N. Intrinsically disordered proteins in overcrowded milieu: Membrane-less organelles, phase separation, and intrinsic disorder. Current Opinion in Structural Biology. 44, 18-30 (2017).
- Patel, A., et al. A liquid-to-solid phase transition of the ALS protein FUS accelerated by disease mutation. Cell. 162 (5), 1066-1077 (2015).
- Abyzov, A., Blackledge, M., Zweckstetter, M. Conformational dynamics of intrinsically disordered proteins regulate biomolecular condensate chemistry. Chem Rev. 122 (6), 6719-6748 (2022).
- Uversky, V. N. Unusual biophysics of intrinsically disordered proteins. Biochim Biophys Acta. 1834 (5), 932-951 (2013).
- Papoian, G. A. Proteins with weakly funneled energy landscapes challenge the classical structure-function paradigm. Proc Natl Acad Sci U S A. 105 (38), 14237-14238 (2008).
- Uversky, V. N., Gillespie, J. R., Fink, A. L. Why are "natively unfolded" proteins unstructured under physiologic conditions. Proteins. 41 (3), 415-427 (2000).
- Bah, A., et al. Folding of an intrinsically disordered protein by phosphorylation as a regulatory switch. Nature. 519 (7541), 106-109 (2015).
- Tompa, P., Schad, E., Tantos, A., Kalmar, L. Intrinsically disordered proteins: emerging interaction specialists. Curr Opin Struct Biol. 35, 49-59 (2015).
- Dyson, H. J., Wright, P. E. Perspective: the essential role of NMR in the discovery and characterization of intrinsically disordered proteins. J Biomol NMR. 73 (12), 651-659 (2019).
- Adamski, W., et al. A unified description of intrinsically disordered protein dynamics under physiological conditions using NMR spectroscopy. J Am Chem Soc. 141 (44), 17817-17829 (2019).
- Dobson, C. M. Protein folding and misfolding. Nature. 426 (6968), 884-890 (2003).
- Bertoncini, C. W., et al. Release of long-range tertiary interactions potentiates aggregation of natively unstructured alpha-synuclein. Proc Natl Acad Sci U S A. 102 (5), 1430-1435 (2005).
- Buell, A. K., et al. Solution conditions determine the relative importance of nucleation and growth processes in alpha-synuclein aggregation. Proc Natl Acad Sci U S A. 111 (21), 7671-7676 (2014).
- Salvi, N., Abyzov, A., Blackledge, M. Atomic resolution conformational dynamics of intrinsically disordered proteins from NMR spin relaxation. Prog Nucl Magn Reson Spectrosc. 102, 43-60 (2017).
- Schneider, R., Blackledge, M., Jensen, M. R. Elucidating binding mechanisms and dynamics of intrinsically disordered protein complexes using NMR spectroscopy. Curr Opin Struct Biol. 54, 10-18 (2019).
- Rezaei-Ghaleh, N., Blackledge, M., Zweckstetter, M. Intrinsically disordered proteins: from sequence and conformational properties toward drug discovery. Chembiochem. 13 (7), 930-950 (2012).
- Jensen, M. R., Salmon, L., Nodet, G., Blackledge, M. Defining conformational ensembles of intrinsically disordered and partially folded proteins directly from chemical shifts. J Am Chem Soc. 132 (4), 1270-1272 (2010).
- Camacho-Zarco, A. R., et al. NMR provides unique insight into the functional dynamics and interactions of intrinsically disordered proteins. Chem Rev. 122 (10), 9331-9356 (2022).
- Mittag, T., Forman-Kay, J. D. Atomic-level characterization of disordered protein ensembles. Curr Opin Struct Biol. 17 (1), 3-14 (2007).
- Dyson, H. J., Wright, P. E. Unfolded proteins and protein folding studied by NMR. Chem Rev. 104 (8), 3607-3622 (2004).
- Dyson, H. J., Wright, P. E. NMR illuminates intrinsic disorder. Curr Opin Struct Biol. 70, 44-52 (2021).
- Sugase, K., Dyson, H. J., Wright, P. E. Mechanism of coupled folding and binding of an intrinsically disordered protein. Nature. 447 (7147), 1021-1025 (2007).
- Bessa, L. M., et al. The intrinsically disordered SARS-CoV-2 nucleoprotein in dynamic complex with its viral partner nsp3a. Sci Adv. 8 (3), eabm4034(2022).
- Milles, S., Salvi, N., Blackledge, M., Jensen, M. R. Characterization of intrinsically disordered proteins and their dynamic complexes: From in vitro to cell-like environments. Prog Nucl Magn Reson Spectrosc. 109, 79-100 (2018).
- Salvi, N., Abyzov, A., Blackledge, M. Atomic resolution conformational dynamics of intrinsically disordered proteins from NMR spin relaxation. Prog Nucl Magn Reson Spectrosc. 102 - 103, 43-60 (2017).
- Gill, M. L., Byrd, R. A., Palmer, A. G. III Dynamics of GCN4 facilitate DNA interaction: a model-free analysis of an intrinsically disordered region. Phys Chem Chem Phys. 18 (8), 5839-5849 (2016).
- Bertoncini, C. W., et al. Structural characterization of the intrinsically unfolded protein beta-synuclein, a natural negative regulator of alpha-synuclein aggregation. J Mol Biol. 372 (3), 708-722 (2007).
- Rezaei-Ghaleh, N., et al. Local and global dynamics in intrinsically disordered synuclein. Angew Chem Int Ed Engl. 57 (46), 15262-15266 (2018).
- Rezaei-Ghaleh, N., Parigi, G., Zweckstetter, M. Reorientational dynamics of amyloid-beta from NMR spin relaxation and molecular simulation. J Phys Chem Lett. 10 (12), 3369-3375 (2019).
- Palmer, A. G. NMR characterization of the dynamics of biomacromolecules. Chem Rev. 104 (8), 3623-3640 (2004).
- Palmer, A. G., Massi, F. Characterization of the dynamics of biomacromolecules using rotating-frame spin relaxation NMR spectroscopy. Chem Rev. 106 (5), 1700-1719 (2006).
- Kay, L. E., Torchia, D. A., Bax, A. Backbone dynamics of proteins as studied by 15N inverse detected heteronuclear NMR spectroscopy: application to staphylococcal nuclease. Biochemistry. 28 (23), 8972-8979 (1989).
- Mittermaier, A., Kay, L. E. Review - New tools provide new insights in NMR studies of protein dynamics. Science. 312 (5771), 224-228 (2006).
- Abragam, A. Principles of Nuclear Magnetism. , Clarendon Press. Oxford. (1983).
- Ernst, R. R., Bodenhausen, G., Wokaun, A. Principles of Nuclear Magnetic Resonance in One and Two Dimensions. , Oxford University Press. Oxford. (1990).
- Cavanagh, J., Fairbrother, W. J., Palmer, A. G. III, Skelton, N. J. Protein NMR Spectroscopy: Principles and Practice. , Elsevier, Academic Press. (1995).
- Levitt, M. H. Spin Dynamics: Basics of Nuclear Magnetic Resonance. , John Wiley and Sons. (2008).
- Keeler, J. Understanding NMR Spectroscopy. , John Wiley and Sons. (2010).
- Bloembergen, N., Purcell, E. M., Pound, R. V. Relaxation effects in nuclear magnetic resonance absorption. Phys Rev. 73 (7), 679-712 (1948).
- Wangsness, R. K., Bloch, F. The dynamical theory of nuclear induction. Phys Rev. 89 (4), 728-739 (1953).
- Redfield, A. G. On the theory of relaxation processes. IBM J Res Dev. 1 (1), 19-31 (1957).
- Peng, J. W., Wagner, G. Mapping of the spectral densities of N-H bond motions in eglin c using heteronuclear relaxation experiments. Biochemistry. 31 (36), 8571-8586 (1992).
- Farrow, N. A., et al. Backbone dynamics of a free and a phosphopeptide-complexed src homology-2 domain studied by n-15 NMR relaxation. Biochemistry. 33 (19), 5984-6003 (1994).
- Tolman, J. R., Ruan, K. NMR residual dipolar couplings as probes of biomolecular dynamics. Chem Rev. 106 (5), 1720-1736 (2006).
- Mulder, F. A. A., Skrynnikov, N. R., Hon, B., Dahlquist, F. W., Kay, L. E. Measurement of slow (mu s-ms) time scale dynamics in protein side chains by N-15 relaxation dispersion NMR spectroscopy: Application to Asn and Gln residues in a cavity mutant of T4 lysozyme. J Am Chem Soc. 123 (5), 967-975 (2001).
- Tollinger, M., Skrynnikov, N. R., Mulder, F. A. A., Forman-Kay, J. D., Kay, L. E. Slow dynamics in folded and unfolded states of an SH3 domain. J Am Chem Soc. 123 (46), 11341-11352 (2001).
- Akke, M., Palmer, A. G. Monitoring macromolecular motions on microsecond to millisecond time scales by R(1)rho-R(1) constant relaxation time NMR spectroscopy. J Am Chem Soc. 118 (4), 911-912 (1996).
- Loria, J. P., Rance, M., Palmer, A. G. A relaxation-compensated Carr-Purcell-Meiboom-Gill sequence for characterizing chemical exchange by NMR spectroscopy. J Am Chem Soc. 121 (10), 2331-2332 (1999).
- Vallurupalli, P., Bouvignies, G., Kay, L. E. Studying "Invisible" excited protein states in slow exchange with a major state conformation. J Am Chem Soc. 134 (19), 8148-8161 (2012).
- Jeener, J., Meier, B. H., Bachmann, P., Ernst, R. R. Investigation of exchange processes by 2-dimensional NMR-spectroscopy. J Chem Phys. 71 (11), 4546-4553 (1979).
- Palmer, A. G., Kroenke, C. D., Loria, J. P. Nuclear magnetic resonance methods for quantifying microsecond-to-millisecond motions in biological macromolecules. Methods Enzymol. 339, 204-238 (2001).
- Clore, G. M., Iwahara, J. Theory, practice, and applications of paramagnetic relaxation enhancement for the characterization of transient low-population states of biological macromolecules and their complexes. Chem Rev. 109 (9), 4108-4139 (2009).
- Iwahara, J., Clore, G. M. Detecting transient intermediates in macromolecular binding by paramagnetic NMR. Nature. 440 (7088), 1227-1230 (2006).
- Lakomek, N. A., et al. Residual dipolar couplings as a tool to study molecular recognition of ubiquitin. Biochem Soc Trans. 36 (Pt 6), 1433-1437 (2008).
- Johnson, C. N., Libich, D. S. Paramagnetic relaxation enhancement for detecting and characterizing self-associations of intrinsically disordered proteins. J Vis Exp. (175), e63057(2021).
- Lakomek, N. A., Ying, J., Bax, A. Measurement of (1)(5)N relaxation rates in perdeuterated proteins by TROSY-based methods. J Biomol NMR. 53 (3), 209-221 (2012).
- Stief, T., Vormann, K., Lakomek, N. A. Sensitivity-enhanced NMR (15)N R(1) and R(1rho) relaxation experiments for the investigation of intrinsically disordered proteins at high magnetic fields. Methods. 223, 1-15 (2024).
- Kay, L. E., Keifer, P., Saarinen, T. Pure absorption gradient enhanced heteronuclear single quantum correlation spectroscopy with improved sensitivity. J Am Chem Soc. 114 (26), 10663-10665 (1992).
- Geen, H., Freeman, R. Band-selective radiofrequency pulses. J Magn Reson (1969). 93 (1), 93-141 (1991).
- Desvaux, H., Berthault, P. Study of dynamic processes in liquids using off-resonance RF irradiation. Prog Nucl Magn Reson Spectrosc. 35 (4), 295-340 (1999).
- Overhauser, A. W. Polarization of nuclei in metals. Phys Rev. 91 (2), 476-476 (1953).
- Overhauser, A. W. Polarization of nuclei in metals. Phys Rev. 92 (2), 411-415 (1953).
- Kumari, P., Frey, L., Sobol, A., Lakomek, N. A., Riek, R. (15)N transverse relaxation measurements for the characterization of micros-ms dynamics are deteriorated by the deuterium isotope effect on (15)N resulting from solvent exchange. J Biomol NMR. 72 (3-4), 125-137 (2018).
- Karschin, N., Krenek, S., Heyer, D., Griesinger, C. Extension and improvement of the methanol-d(4) NMR thermometer calibration. Magn Reson Chem. 60 (4), 203-209 (2022).
- Sattler, M., Schleucher, J., Griesinger, C. Heteronuclear multidimensional NMR experiments for the structure determination of proteins in solution employing pulsed field gradients. Prog Nucl Magn Reson Spectrosc. 34 (2), 93-158 (1999).
- Ferrage, F., Cowburn, D., Ghose, R. Accurate sampling of high-frequency motions in proteins by steady-state N-{H} nuclear Overhauser effect measurements in the presence of cross-correlated relaxation. J Am Chem Soc. 131 (17), 6048-6049 (2009).
- Delaglio, F., et al. NMRPipe: a multidimensional spectral processing system based on UNIX pipes. J Biomol NMR. 6 (3), 277-293 (1995).
- Skinner, S. P., et al. CcpNmr AnalysisAssign: a flexible platform for integrated NMR analysis. J Biomol NMR. 66 (2), 111-124 (2016).
- Lee, W., Tonelli, M., Markley, J. L. NMRFAM-SPARKY: enhanced software for biomolecular NMR spectroscopy. Bioinformatics. 31 (8), 1325-1327 (2015).
- Lee, W., Rahimi, M., Lee, Y., Chiu, A. POKY: a software suite for multidimensional NMR and 3D structure calculation of biomolecules. Bioinformatics. 37 (18), 3041-3042 (2021).
- Abyzov, A., et al. Identification of dynamic modes in an intrinsically disordered protein using temperature-dependent NMR relaxation. J Am Chem Soc. 138 (19), 6240-6251 (2016).
- Singh, A., Purslow, J. A., Venditti, V. 15N CPMG relaxation dispersion for the investigation of protein conformational dynamics on the micros-ms timescale. J Vis Exp. (170), e62395(2021).
- Gopalan, A. B., Hansen, D. F., Vallurupalli, P. CPMG experiments for protein minor conformer structure determination. Methods Mol Biol. 1688, 223-242 (2018).
- Tugarinov, V., Kay, L. E. Methyl groups as probes of structure and dynamics in NMR studies of high-molecular-weight proteins. Chembiochem. 6 (9), 1567-1577 (2005).
- Lipari, G., Szabo, A. Model-free approach to the interpretation of nuclear magnetic resonance relaxation in macromolecules. 1. Theory and range of validity. J Am Chem Soc. 104 (17), 4546-4559 (1982).
- Lipari, G., Szabo, A. Model-Free Approach to the interpretation of nuclear magnetic-resonance relaxation in macromolecules .2. Analysis of experimental results. J Am Chem Soc. 104 (17), 4559-4570 (1982).
- Clore, G. M., et al. Deviations from the simple two-parameter model-free approach to the interpretation of nitrogen-15 nuclear magnetic relaxation of proteins. J Am Chem Soc. 112 (12), 4989-4991 (1990).
- Lemaster, D. M. Larmor frequency selective model free analysis of protein NMR relaxation. J Biomol NMR. 6 (4), 366-374 (1995).
- Peng, J. W., Wagner, G. Mapping of spectral density functions using heteronuclear NMR relaxation measurements. J Magn Reson (1969). 98 (2), 308-332 (1992).
- Farrow, N. A., Zhang, O. W., FormanKay, J. D., Kay, L. E. Characterization of the backbone dynamics of folded and denatured states of an SH3 domain. Biochemistry. 36 (9), 2390-2402 (1997).
- Khan, S. N., et al. Distribution of pico- and nanosecond motions in disordered proteins from nuclear spin relaxation. Biophys J. 109 (5), 988-999 (2015).
- Smith, A. A., Ernst, M., Meier, B. H., Ferrage, F. Reducing bias in the analysis of solution-state NMR data with dynamics detectors. J Chem Phys. 151 (3), 034102(2019).
- Zumpfe, K., Smith, A. A. Model-free or not. Front Mol Biosci. 8, 727553(2021).
- Robustelli, P., Trbovic, N., Friesner, R. A., Palmer, A. G. Conformational dynamics of the partially disordered yeast transcription factor GCN4. J Chem Theory Comput. 9 (11), 5190-5200 (2013).
- Salvi, N., Abyzov, A., Blackledge, M. Multi-timescale dynamics in intrinsically disordered proteins from NMR relaxation and molecular simulation. J Phys Chem Lett. 7 (13), 2483-2489 (2016).
Access restricted. Please log in or start a trial to view this content.
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved