É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Experimentos de relaxamento de RMN 15N para a investigação da dinâmica estrutural de proteínas de picossegundos a nanossegundos
Neste Artigo
Resumo
A espectroscopia de ressonância magnética nuclear (RMN) pode caracterizar a dinâmica estrutural da proteína de maneira específica do resíduo. Fornecemos um protocolo prático para registrar experimentos de relaxamento de RMN 15N R1 e R2 e {1H}-15N heteronuclear Overhauser effect (hetNOE), sensíveis à escala de tempo de picossegundos a nanossegundos.
Resumo
A espectroscopia de ressonância magnética nuclear (RMN) permite estudar proteínas em solução e sob temperaturas fisiológicas. Freqüentemente, os grupos amida da estrutura da proteína ou os grupos metil nas cadeias laterais são usados como repórteres da dinâmica estrutural em proteínas. Um estudo de dinâmica estrutural da estrutura proteica de proteínas globulares em amostras marcadas com 15N e totalmente protonadas geralmente funciona bem para proteínas com peso molecular de até 50 kDa. Quando a deuteração de cadeia lateral em combinação com espectroscopia otimizada de relaxamento transversal (TROSY) é aplicada, esse limite pode ser estendido até 200 kDa para proteínas globulares e até 1 MDa quando o foco está nas cadeias laterais. Quando proteínas intrinsecamente desordenadas (IDPs) ou proteínas com regiões intrinsecamente desordenadas (IDRs) são investigadas, essas limitações de peso não se aplicam, mas podem ir muito além. A razão é que os IDPs ou IDRs, caracterizados por alta flexibilidade interna, são frequentemente desacoplados dinamicamente. Vários métodos de RMN oferecem insights de resolução atômica sobre a dinâmica estrutural das proteínas em uma ampla gama de escalas de tempo, de picossegundos a horas. As medições de relaxamento padrão de 15N apresentam uma visão geral da flexibilidade interna de uma proteína e caracterizam a dinâmica da estrutura da proteína experimentada na escala de tempo rápida de pico a nanossegundo. Este artigo apresenta um protocolo prático para configurar e registrar experimentos de RMN 15NR1, R2 e efeito Overhauser heteronuclear (hetNOE). Mostramos dados exemplares e explicamos como interpretá-los de forma simples qualitativa antes de qualquer análise mais sofisticada.
Introdução
A função de uma proteína é determinada não apenas por sua estrutura tridimensional, mas também por sua dinâmica estrutural, englobando sua flexibilidade interna e transições estruturais entre diferentes conformações que a proteína adotará. A espectroscopia de ressonância magnética nuclear (RMN) pode investigar a dinâmica estrutural de proteínas em solução 1,2,3. Desenvolvimentos recentes na RMN de estado sólido detectada por prótons também permitem a caracterização da dinâmica de proteínas em um estado menos solúvel, como, por exemplo, uma membrana de bicamada lipídica 4,5,6. Na RMN em solução, a dinâmica estrutural da estrutura da proteína e das cadeias laterais da proteína pode ser estudada. Para uma proteína globular, um estudo de dinâmica estrutural da estrutura da proteína pode ser alcançado até 50 kDa, uma vez que a proteína é marcada isotopicamente com 15N. Quando a deuteração de cadeia lateral e a espectroscopia otimizada para relaxamento transversal (TROSY) são empregadas, esse limite pode ser estendido até 200 kDa 7,8. Quando o foco está na dinâmica da cadeia lateral, a gama de proteínas e complexos acessíveis pode ser estendida até 1 MDa 2,9.
As limitações de peso nomeadas não se aplicam a proteínas intrinsecamente desordenadas (IDPs), que frequentemente apresentam alta dinâmica intrínseca. Mais de 30% do proteoma eucariótico compreende IDPs ou regiões intrinsecamente desordenadas (IDRs)10,11,12,13. Eles desempenham papéis centrais em muitos processos celulares, como transdução e transcrição de sinal1, e estão frequentemente envolvidos na separação de fase intracelular 14,15,16,17. Os IDPs carecem de uma estrutura nativa tridimensional (3D) bem definida em condições fisiológicas e têm uma paisagem de energia fracamente canalizada ou acidentada17,18. Devido a uma baixa hidrofobicidade e forte repulsão eletrostática distribuída sobre a espinha dorsal dos IDPs ou IDRs, falta uma força motriz para dobrar em uma estrutura rígida19. Os IDPs freqüentemente adotam uma conformação dobrada quando complexos com outros parceiros de ligação 10,20,21. Além disso, as modificações pós-traducionais (PTMs) expandem as possibilidades de dobramento de IDPs ou IDRs22,23. O desdobramento de deslocados internos foi identificado como causa de várias doenças, incluindo doenças neurodegenerativas 15,24,25,26.
IDPs e IDRs mostram alta flexibilidade interna 21,27,28. Conjuntos conformacionais exibindo a variação de posições atômicas e ângulos diedros foram derivados de simulações de dinâmica molecular e restrições obtidas de dados experimentais29 , 30 , 31 , 32 . Devido à dinâmica e à desordem resultante no estado congelado, a densidade eletrônica difusa dificulta caracterizá-los estruturalmente usando métodos de última geração em biologia estrutural, como crio-EM ou cristalografia de raios-X. Além disso, as condições de cristalização ou técnicas de preparação de amostras para experimentos em temperaturas criogênicas podem afetar o espaço conformacional experimentado pelos IDPs. No entanto, a RMN da solução funciona bem para proteínas altamente dinâmicas e, portanto, é adequada para investigar IDPs 16,20,22,28,29,30,31,32,33,34,35,36,37,38.
Conforme apresentado acima, a RMN da solução oferece várias técnicas para estudar a dinâmica interna da proteína em uma ampla gama de escalas de tempo (Figura 1), principalmente com base no relaxamento de spin 31,33,38,39,40,41,42.
O relaxamento do spin dos núcleos de 15N nos grupos amida da estrutura da proteína é induzido por mudanças na orientação do ângulo de ligação 1 H-15N devido à dinâmica interna da proteína e movimentos coletivos (incluindo, quando relevante, difusão rotacional) 27 , 43 , 44 , 45 , 46 , 47 , 48 , 49 , 50 , 51. Em escalas de tempo mais curtas do que o tempo de correlação rotacional τR (o tempo que a molécula precisa para girar um radiante, também chamado de tempo de correlação geral de queda), a anisotropia de deslocamento químico (CSA) e o acoplamento dipolar (D) são ativos e não calculados pela difusão rotacional da proteína. A dinâmica interna da estrutura da proteína, compreendendo variações nos ângulos de ligação, reorientações de ligações e queda rotacional, induz flutuações estocásticas do CSA e do tensor de acoplamento dipolar, resultando em uma variação do campo magnético local, levando ao relaxamento do spin da RMN47 , 48 , 52 , 53. Essas flutuações podem ser descritas por uma função de correlação geral. A transformada de Fourier da função de correlação geral é chamada de função de densidade espectral. Na teoria semiclássica de relaxamento de Redfield, as constantes de taxa de relaxamento de RMN podem ser descritas por combinações lineares dessas funções de densidade espectral54.
Os experimentos de relaxamento de RMN de 15N da espinha dorsal desenvolvidos no início da década de 1990 compreendem experimentos de efeito Overhauser nuclear de 15N R1, R1ρ e {1H}-15N, sensíveis à escala de tempo rápida de picossegundos (ps) nanossegundos (ns), mais rápida que o tempo de correlação rotacional τR da proteína 45,55,56,57. Para caracterizar a dinâmica da espinha dorsal mais lenta do que o tempo de correlação rotacional τR, são usados os chamados experimentos de dispersão de relaxamento, R1ρ, e experimentos de Carr-Purcell-Meiboom-Gill (CPMG) sensíveis à dinâmica de microssegundos (μs) - milissegundos (ms) 44,46,58,59,60,61. A dinâmica mais lenta que microssegundos pode ser capturada por RMN de transferênciade saturação de troca química (CEST)de 15 N 62, espectroscopia de troca (EXSY, milissegundos a segundos) ou RMN em tempo real (RT) (segundos a horas) 63 , 64 . O efeito PRE (realce de relaxamento paramagnético) das sondas paramagnéticas, bem como os acoplamentos dipolares residuais (RDCs), podem ser usados para avaliar toda a faixa de dinâmica ps a ms 65,66,67,68.
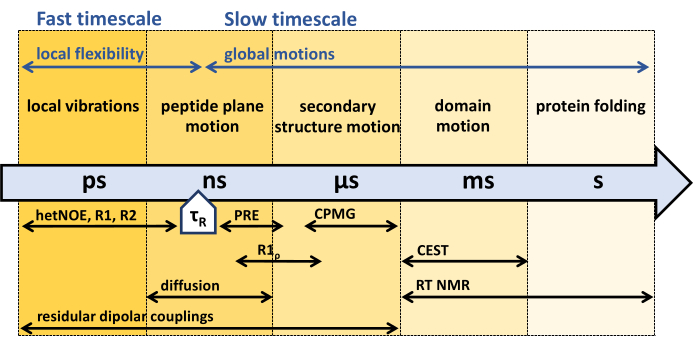
Figura 1: Escalas de tempo da dinâmica da estrutura de proteínas e janela de tempo sensível de diferentes experimentos de dinâmica de RMN. A RMN oferece uma variedade de métodos para caracterizar a dinâmica da espinha dorsal da proteína em uma ampla gama de escalas de tempo. Diferentes movimentos experimentados pela estrutura da proteína são indicados em suas respectivas escalas de tempo. O tempo de correlação rotacional da proteína, τR, é o tempo que a proteína precisa para uma rotação geral (por um radiante). Movimentos mais rápidos do que o tempo de correlação rotacional da proteína, τR, podem ser associados à flexibilidade interna da proteína. Vários experimentos de RMN e sua sensibilidade às respectivas escalas de tempo são indicados abaixo da seta. Clique aqui para ver uma versão maior desta figura.
O protocolo abaixo descreve a configuração de experimentos de relaxamento de RMN por Lakomek et al.69 e Stief et al.70, usando um esquema de detecção de coerência quântica única heteronuclear (HSQC) com sensibilidade aprimorada. Antes de prosseguir para a implementação experimental, é dada uma breve visão geral dos experimentos de relaxamento de spin de RMN e relaxamento de RMN. Devido a restrições de tamanho e para manter este protocolo compreensível, esta visão geral deve permanecer simplista (e, portanto, incompleta).
O relaxamento longitudinal ou spin-lattice, caracterizado pelo tempo T1 ou pela constante de velocidade R1 = 1 / T1 , descreve o retorno da magnetização ao equilíbrio de Boltzmann. Em equilíbrio, a magnetização é alinhada ao longo do eixo do campo magnético externo, que define o eixo z da estrutura do laboratório. Densidades espectrais em frequências Larmor altas (1H) e pequenas (15N) (as frequências de ressonância NMR, por exemplo, 600 MHz por 1H para um ímã de 14,1 Tesla) e combinações lineares dessas frequências Larmor contribuem para o relaxamento de 15N R1 , que é caracterizado pelas constantes de taxa de 15N R1 medidas em rad · s-1. Os movimentos em escalas de tempo são inversos a essas frequências de Larmor; assim, os movimentos na escala de tempo de picossegundos a nanossegundos contribuem para a constante da taxa de relaxamento R1. Para moléculas que mostram queda geral e onde um tempo de correlação rotacional pode ser definido, a curva R1 (T1) mostra um máximo (mínimo) para ωτR = 1, com a correlação rotacional τR e a frequência de Larmor ω do spin em consideração. Se várias frequências de Larmor contribuem, aquela com a frequência mais baixa é a dominante, por exemplo, ωN no caso de 15N R1. O regime de movimento rápido (ωτR muito menor que 1) se aplica a pequenas moléculas que caem muito rápido e para baixos campos magnéticos e baixa viscosidade. O regime de câmera lenta (ωτR muito maior que 1) é válido para moléculas maiores que caem mais devagar e para altos campos magnéticos e alta viscosidade.
As proteínas dobradas globulares mostram o tombamento geral em solução, e um tempo de correlação rotacional pode ser atribuído. No entanto, o conceito de queda geral não é mais válido para proteínas intrinsecamente desordenadas e freqüentemente difere da atribuição de um único tempo de correlação rotacional. Aqui, o tempo de correlação interna específico do resíduo torna-se mais crítico.
A sequência de pulso descrita medindo taxas de relaxamento de 15N R1 (Figura 2) é baseada em um experimento de leitura HSQC com sensibilidade aprimorada com detecção de eco / anti-eco para detecção de quadratura 69,70,71. Gradientes curtos com resistência e comprimento variáveis são usados para seleção de coerência e melhor supressão de água70. Durante esse tempo, a polarização longitudinal de 15N relaxará. Tempos de decaimento mais longos levam a intensidades reduzidas nos planos 2D associados a esse espectro pseudo-3D (os pontos de dados de atraso são registrados na terceira dimensão). Um elemento de loop, descrito abaixo, é executado um número crescente de vezes para tempos de relaxamento mais longos. Como o relaxamento correlacionado entre a anisotropia de deslocamento químico de 15N (CSA) e o acoplamento dipolar de 1H e 15N (D) também está ativo durante o atraso de relaxamento, um pulso central I-BURP-2 de 180 °72, seletivo nos prótons de amida, é necessário para reorientar a contribuição pelo relaxamento correlacionado cruzado (que, se não for refocado, levaria a constantes de taxa de 15N R1 distorcidas e errôneas).
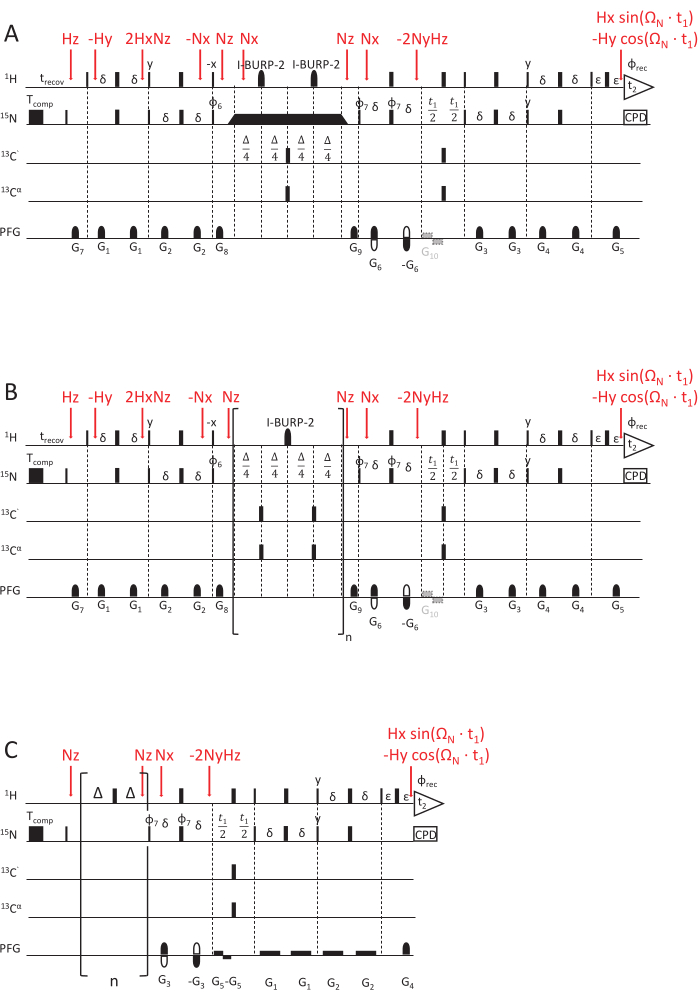
Figura 2: Esquemas de sequência de pulso de RMN para determinar as constantes de taxa de relaxamento de RMN. (A) 15N R1ρ, (B) 15N R1 e (C) experimento hetNOE, usando um esquema de leitura HSQC aprimorado por sensibilidade69,70. Os pulsos de 90 ° (x) são visualizados por retângulos estreitos e os pulsos de 180 ° (x) por retângulos largos, salvo indicação em contrário. O seguinte ciclo de fase é aplicado: φ6 = y, y, -y, -y; φ7 = y, -y, φrec = y, -y, -y, y. A detecção de quadratura é obtida invertendo a polaridade do gradiente G5 e o ciclo de fase de φ7 (detecção de eco / anti-eco). (A) 15N R1ρ experimento: O retangular preto representa o spin-lock, para o qual a duração varia para adquirir diferentes atrasos de relaxamento. Os triângulos antes e depois do bloqueio de rotação indicam os pulsos de forma adiabática que alinham a magnetização ao longo do eixo B efetivo do campo magnéticoeff. G10 é um gradiente opcional para evitar o amortecimento da radiação da magnetização da água durante a fase de evolução. (B) 15N R1 experimento: A parte entre colchetes mostra o elemento de loop da sequência, repetido um n número de vezes para corresponder ao atraso de relaxamento desejado. (C) O esquema de pulso hetNOE é semelhante à segunda metade dos esquemas de pulso R1 e R1ρ, ou seja, o tempo de evolução t1 e o elemento de detecção HSQC. No entanto, a magnetização de 15N é excitada diretamente sem qualquer INEPT. A saturação da magnetização do próton (para obter relaxamento cruzado entre 1H e 15N) é alcançada por um trem de pulsos de 180 (1H) aplicados por pelo menos 5 s. Um atraso ocioso dos mesmos comprimentos (aqui, 5 s) sem qualquer trem de pulso é aplicado para o experimento de referência. G5 é um gradiente opcional para evitar o amortecimento da radiação, e a inversão na polaridade do gradiente G4, em combinação com a fase φ7 = y, -y, -y, y, alcança a detecção de quadratura. As etapas de transferência de magnetização, representadas pelos operadores do produto, são marcadas em vermelho. Clique aqui para ver uma versão maior desta figura.
A constante de taxa de relaxamento R2 descreve o relaxamento da polarização transversal (no plano xy ortogonal ao campo magnético externo) devido à perda de coerência de fase entre os spins, levando ao decaimento da magnetização detectável53,54. A função de densidade espectral em altas e pequenas frequências contribui para R2, semelhante a R1. No entanto, a maior contribuição para R2 se origina da densidade espectral em frequência zero. Por esta razão, R2 é muito sensível à queda rotacional, descrita pelo tempo de correlação rotacional τR, que é da ordem de alguns ns para uma pequena proteína globular à temperatura ambiente. Assim, movimentos de backbone mais lentos na casa das centenas de ps para o regime de ns baixo contribuem mais. A dinâmica de troca da espinha dorsal que causa uma modulação da parte isotrópica do tensor de deslocamento químico dos núcleos de 15N, em princípio, adiciona uma contribuição de troca R2ex às constantes de taxa R2 43,44,49,60,61. Nos experimentos descritos, a contribuição do R2ex é suprimida por um bloqueio de rotação que reorienta a dinâmica mais lentamente do que a frequência circular inversa do bloqueio de rotação. O spin-lock é um pulso de radiofrequência de onda contínua longa que mantém a magnetização alinhada ao longo do eixo B do campo magnético efetivoeff (a soma vetorial do campo ω1 do spin-lock e o deslocamento químico da frequência portadora de 15N (veja abaixo)). O relaxamento da magnetização alinhada ao longo do eixo B1,eff é chamado de relaxamento R1ρ, que tem um componente R1 e um componente R2. Usando a fórmula (1), R2 pode ser calculado a partir de R1ρ e R144,73:
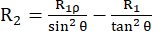 (1).
(1).
O ângulo entre o eixo do campo magnético efetivo Beff e o campo magnético externo B0 é 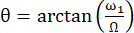 . ω1 é a amplitude de RF do spin-lock e Ω o desvio químico entre o desvio químico de 15N do resíduo correspondente e a frequência portadora de 15N 44,73.
. ω1 é a amplitude de RF do spin-lock e Ω o desvio químico entre o desvio químico de 15N do resíduo correspondente e a frequência portadora de 15N 44,73.
O esquema de pulso R1ρ (Figura 2A, 70) é muito semelhante ao esquema 15N R1 , exceto pelo atraso de relaxamento. Para medir as taxas de relaxamento de 15N R1ρ , o bloqueio de rotação deve estar ativo após a magnetização ter sido alinhada ao longo do eixo do campo efetivo Beff por um pulso adiabático com a mesma amplitude de radiofrequência (RF) que o bloqueio de rotação. O comprimento do bloqueio de rotação será variado para obter os diferentes atrasos de relaxamento.
O efeito de Overhauser nuclear {1H}-15N em estado estacionário (1 H-15N NOE), apelidado de hetNOE a seguir, é a razão entre a taxa de relaxamento cruzado e a taxa de relaxamento longitudinal de 15N. Isso leva a uma redução da polarização em estado estacionário em 15N devido ao relaxamento cruzado com o próton após a saturação da polarização do próton 45,53,54,74,75. O relaxamento cruzado depende das funções de densidade espectral da soma e diferença das frequências de Larmor de 1H e 15N. Portanto, o hetNOE é sensível tanto à dinâmica rápida de picossegundos (< 100 ps) quanto à dinâmica ps-ns (devido à sua dependência R1). A sequência69 (Figura 2C) é baseada em uma leitura HSQC aprimorada por sensibilidade com gradientes Echo / Anti-Echo para detecção de quadratura. Para a saturação da magnetização do próton e do hetNOE resultante, a magnetização do próton de equilíbrio é invertida e subsequentemente saturada por pulsos de 180 ° pulsantes rapidamente por cerca de 5 vezes o 15 N T1. Para o experimento de referência, o atraso de recuperação é igual ao atraso de saturação, mas sem o trem de pulso de 1H 180 °. Um atraso extra de D1 = 2 s é adicionado para o experimento de referência e aquele com saturação de 1H. Ambos os experimentos são registrados consecutivamente e diferem apenas na aplicação de pulsos de 1H 180 ° (saturação) ou não (referência). A razão entre as intensidades espectrais registadas na experiência com saturação de 1H dividida pelas intensidades da experiência de referência (sem o comboio de impulsos de protões de 180°) indica o valor {1H}-15N NOE (hetNOE).
O protocolo a seguir descreve a configuração dos experimentos de relaxamento por RMN por Lakomek et al.69 e Stief et al.70. Nós nos concentramos nas sequências de pulso de RMN usando um esquema de detecção HSQC aprimorado por sensibilidade. Os experimentos 15N R1 e R1ρ são implementados conforme descrito em detalhes por Stief et al.70, e o experimento hetNOE é descrito por Lakomek et al.69.
Access restricted. Please log in or start a trial to view this content.
Protocolo
1. Preparação da amostra de RMN
NOTA: A marcação isotópica das proteínas é realizada para experimentos de RMN de dimensão superior e RMN avançada. Quando a expressão de proteínas em E. coli e a purificação de proteínas foram estabelecidas usando meios ricos (por exemplo, Luria-Bertani [LB] ou meio triptona de extrato de levedura 2x [2YT]) com um rendimento de vários miligramas por litro, a preparação de uma amostra de RMN marcada isotopicamente é geralmente relativamente simples.
- Para marcação de isótopos, use meio mínimo M9 para expressão gênica, onde cloreto de amônio 15N é a única fonte de nitrogênio e glicose enriquecida com 13C é a única fonte de carbono.
- Para purificação de proteínas, siga o protocolo padrão estabelecido em laboratório para a respectiva proteína.
NOTA: A seção de resultados mostra resultados representativos para um IDP, a proteína monomérica SNARE Synaptobrevin-2 (1-96). A marcação isotópica de RMN de Synaptobrevin-2 foi realizada conforme descrito em70. Para medições de altíssima precisão, recomenda-se o uso de baixas concentrações de D2O, inferiores a 5%. Isso ocorre por causa do efeito do isótopo de deutério, que pode ser observado em Hahn-Echo 15N T2 e em experimentos de dispersão de relaxamento de baixa frequência 15N CPMG (não discutido aqui), mas tem apenas um impacto mínimo em experimentos 15N R1ρ (onde é principalmente refocado) 76 .
2. Preparativos para a execução das experiências de relaxamento de RMN no espectrómetro
NOTA: Os experimentos de relaxamento de RMN descritos são específicos para espectrômetros Bruker. Eles foram testados em sondas de ressonância tripla criogênicas e à temperatura ambiente de 1H, 15N e 13C e consoles Avance III e Avance Neo operados pelo software Bruker Topsin 3.6 ou superior.
- Siga as precauções.
- Faça um briefing de segurança antes de trabalhar no espectrômetro de RMN.
- Verifique as especificações de potência da sonda do espectrômetro usado e escolha um comprimento de pulso e potência de pulso apropriados para cada pulso, por exemplo, comparando a consistência com as entradas da tabela "prosol" Topspin.
- Importe as sequências de pulso.
- Baixe os programas de pulso de RMN do website indicado abaixo ou do repositório estendido do Banco de Ressonância Magnética Biológica (BMRB) (bmrbig102) para o diretório /opt/topspin4.1.4/exp/stan/nmr/lists/pp/user do computador que opera o espectrômetro de RMN. Por favor, ajuste a versão topspin (aqui: topspin4.1.4 para a respectiva versão instalada no espectrômetro).
- Baixe os pulsos em forma de RMN do website indicado abaixo ou do repositório estendido do Banco de Ressonância Magnética Biológica (BMRB) (bmrbig102) para o diretório /opt/topspin4.1.4/exp/stan/nmr/lists/pp/user do computador que opera o espectrômetro de RMN. Por favor, ajuste a versão topspin (aqui: topspin 4.1.4 para a respectiva versão instalada no espectrômetro).
- Calibrar o espectrómetro e a amostra.
- Defina a temperatura de interesse. Para calibração de temperatura, aplique os protocolos da instalação. Por exemplo, use uma amostra de metanol-d4 e interprete a diferença de deslocamento químico observada usando o termômetro NMR do aplicativo da web (https://chem.ch.huji.ac.il/nmr/software/thermometer.html); ver também77.
- Insira a amostra (mais 5% D2O adicionais para a trava) e aguarde aproximadamente 10 minutos para permitir que a amostra equilibre sua temperatura. Bloqueie o sinal de deutério (2H) (comando: bloquear, selecione H2O+D2O).
- Combine e sintonize os canais 1H, 15N e 13C, por exemplo, usando a rotina atma ou atmm. Se nenhuma unidade automática de correspondência e ajuste estiver instalada, combine e ajuste manualmente o teste usando o comando wobb.
- Execute o calço para um campo B0 homogêneo, por exemplo, usando a rotina topshim tg.
- Calibre o comprimento do pulso de 90° (1H).
- Use o experimento zg. Use getprosol para importar os parâmetros da sonda. Escolha uma duração curta de 5 μs para P1. Verifique e corrija o nível de potência especificado da sonda para PLW1, se necessário.
- Inicie o experimento com zg. Processe os dados com efp. Ajuste a fase com .ph para que a linha de base seja uniforme e o sinal de RMN da ressonância da água esteja em fase.
- Estime um pulso de 360° (1H) tomando um pulso comum ou aproximado de 90° (1H) (por exemplo, das especificações do espectrômetro listadas) e multiplicando-o por um fator de quatro. Execute o experimento e processe-o.
NOTA: A duração escolhida é muito longa se o sinal for positivo e muito curta se for negativo. O pulso de 360° (1H) será o cruzamento zero. - Ajuste o comprimento do pulso de acordo. Faça isso iterativamente para encontrar o cruzamento zero. Divida o valor obtido por quatro. Esse será o pulso de 90 ° (1H).
NOTA: Uma alternativa ao método 360° descrito é a rotina pulsal , que pode ser iniciada a partir da linha de comando e permite uma calibração automatizada de pulso de 1H.
- Calibre a frequência portadora O1 para a ressonância da água.
- Copie o experimento zg digitando edc na linha de comando para um novo número de experimento. Digite rpar para abrir os conjuntos de parâmetros (Figura Suplementar 1A).
- Procure o conjunto de parâmetros ZGPR. Clique no conjunto de parâmetros ZGPR (Figura Suplementar 1A), clique em Ler, escolha Executar "getprosol" e clique em OK (Figura Suplementar 1B).
- Altere P1 para o comprimento de pulso calibrado de 90° (1H). Escolha um comprimento de saturação de D1 = 2 s e uma potência de pulso de PLW9 de 50 dB. Defina o ganho do receptor (rg) para 16. Como alternativa, execute o ajuste de ganho do receptor digitando rga e definindo o ganho do receptor para o valor especificado (mas evite "estouro de ganho do receptor").
- Digite gs na linha de comando para iniciar uma aquisição repetida e contínua. Vá para a janela de aquisição e altere o O1 para reduzir a área FID dominada pelo sinal de água (Figura Suplementar 2).
- Clique abaixo do marcador para diminuir o O1 e clique acima do marcador para aumentá-lo. Use o botão de sensibilidade para alterar o tamanho do passo. Salve o O1 atual; O fechamento interrompe a aquisição repetida (Figura 2 suplementar).
- Calibre o comprimento do pulso de 90° (15N).
- Copie o experimento zg para um novo número de experimento (tipo edc). Digite rpar para ler o conjunto de parâmetros HSQCFPF3GPPHWG para alterar o programa de pulso para um experimento HSQC de 1 H-15N. Use a opção Manter os parâmetros P1, O1 e PLW1 (Figura Suplementar 1).
- Digite getprosol 1H 1H) comprimento de pulso em μs> <90° (1H) força de pulso em db> na linha de comando para importar os parâmetros da sonda. Defina o número de varreduras (NS) como 8 e as varreduras fictícias (DS) como 2.
NOTA: < > especifica aqui os valores a serem definidos. Por exemplo: getprosol 1H 8,5 -11,79. - Execute o primeiro incremento do experimento digitando zg na linha de comando. Copie o primeiro incremento para um novo número de experimento com rser 1 . Processe os dados com efp e faseie o espectro com .ph. Use o primeiro incremento (experimento 1D) do experimento para as etapas a seguir.
NOTA: O < > especificar o número escolhido para o novo experimento, por exemplo, rser 1 101. - Digite ased na linha de comando para abrir os parâmetros da sequência de pulsos do experimento 1D. Clique no E na linha PULPROG (consulte a Figura Suplementar 3A) para abrir o editor do programa de pulso.
- Use o seguinte fluxo de trabalho para salvar a sequência de pulsos com um novo nome: Clique em Arquivo (canto superior esquerdo), Salvar como, insira o novo nome e clique em OK. Em seguida, clique em Configurar PULPROG para o conjunto de dados (consulte a Figura Suplementar 3B) e clique em OK.
- Modifique a sequência HSQC original de 1 H-15N (salva com um novo nome, consulte 2.6.4.) multiplicando os 90 ° (15N) após o período de evolução t1 (antes da transferência de retorno) por um fator de 2 (o pulso de 90 ° (15N) agora é 180 ° (15N)). Pressione Ctrl S para salvar a sequência de pulsos. Ajuste o comprimento do pulso para 90° (15N) (P21) para que o sinal de RMN do primeiro incremento desapareça (no cruzamento zero) (Figura Suplementar 3B).
- Abra o experimento 1 H-15N HSQC (experimento 2D) e insira o comprimento de pulso calibrado de 90° (15N) (P21). Tipo getprosol 1H 1H) comprimento de pulso em μs> <90° (1H) força de pulso em db> 15N 15N) comprimento de pulso em μs> <90° (15N) força de pulso em db> na linha de comando.
- Determine as larguras espectrais e a apropriação dos tempos de aquisição.
- Na janela de parâmetro de aquisição para o experimento 1 H-15N HSQC (digite eda na linha de comando), ajuste a largura espectral (parâmetro de aquisição SW) e o número de incrementos de dimensão nas dimensões diretas e indiretas (parâmetro de aquisição TD).
- Escolha SW , 16 ppm na dimensão direta (1H) e 30 ppm na dimensão indireta (15N). Escolha para TD, 2048 na dimensão direta (1H) e 512 na dimensão indireta (15N).
- Adquira o experimento 1 H-15N HSQC.
- Verifique se todos os sinais de interesse foram detectados e se nenhum artefato de aliasing está presente no espectro. Altere o O3P se o centro do espectro não se encaixar no centro da distribuição do sinal da proteína ao longo do eixo 15N.
- Aumente o SW se a janela espectral for muito pequena. Aumente o TD para obter uma resolução FID mais alta (verifique as especificações da sonda para a duração máxima do desacoplamento).
NOTA: Esta é a configuração geral e o ponto de partida para a configuração dos experimentos de relaxamento de RMN. Recomenda-se registrar o experimento 1 H-15N HSQC no início e no final de todo o conjunto de experimentos e, para tempos experimentais mais longos, também entre eles para monitorar a integridade da amostra.
- Na janela de parâmetro de aquisição para o experimento 1 H-15N HSQC (digite eda na linha de comando), ajuste a largura espectral (parâmetro de aquisição SW) e o número de incrementos de dimensão nas dimensões diretas e indiretas (parâmetro de aquisição TD).
3. Implementando os experimentos de relaxamento de RMN
NOTA: As sequências de pulso de relaxamento de RMN (Figura 2) estão disponíveis em https://www.ipb.hhu.de/en/teams/team-lakomek/pulsesequences ou no repositório estendido do Banco de Ressonância Magnética Biológica (BMRB) (bmrbig102).
- Configure o 15NR1ρ experimentar.
- Copie o experimento HSQC (tipo edc) para um novo diretório. Digite ased e altere o programa de pulso para o experimento 15N R1ρ clicando nos três pontos da linha de parâmetro PULPROG nos parâmetros da sequência de pulso (ased, consulte a Figura Suplementar 3A). Clique duas vezes no programa de pulso, clique em Definir PULPROG para conjunto de dados (consulte a Figura Suplementar 3B) e clique em OK.
NOTA: Algumas mensagens de erro podem aparecer. Feche estas mensagens. - Digite gpnam na linha de comando para inserir todas as formas de gradiente ausentes (gpnam1- gpnam9) conforme descrito na sequência de pulsos (Figura Suplementar 4).
- Digite vplist na linha de comando. Clique nos três pontos (Figura Suplementar 5A), Arquivo (canto superior esquerdo) e Novo (Figura Suplementar 5B).
- Digite um nome para uma nova vplist fictícia e pressione OK. Crie a vplist fictícia (5m, 65m, 25m, 45m, 15m, 55m, 35m, 10m; uma nova linha para cada entrada da vplist). Pressione Ctrl S para salvar a lista e fechar a janela da vplist (Figura Suplementar 5C).
- Clique duas vezes no nome da vplist recém-criada (Figura Suplementar 5B).
NOTA: Isso é necessário para evitar erros de compilação na sequência de pulsos. As entradas adequadas da vplist serão determinadas mais adiante (ponto 3.1.29.). - Digite ased para abrir os parâmetros da sequência de pulsos (Figura Suplementar 6). Insira todas as intensidades e comprimentos de gradiente ausentes conforme fornecido na sequência de pulsos (Figura Suplementar 6D).
- Insira para o contador de loops L3 (número de pontos complexos na dimensão indireta) 1/2 da entrada 15N TD na janela de parâmetros de aquisição (eda). Insira para o contador de loops L6 o número de pontos de dados de relaxamento registrados usados para ajustar a curva de relaxamento.
NOTA: Este número L6 corresponde ao número de entradas vplist, aqui: 8 (Figura Suplementar 6A). - Defina o pulso adiabático TanhTan_half.nl, baixado do website especificado acima, como a forma do pulso 15N SP8. Defina o pulso adiabático TanhTan_2nd.nl do website como a forma do pulso 15N SP9 (Figura Suplementar 6C).
- Defina a entrada P8 para o comprimento do pulso adiabático TanhTan - suficientemente longo para garantir a adiabaticidade do pulso; aqui P8 = 3000 μs é escolhido (Figura Suplementar 6C).
- Defina o desacoplamento de 15N (CPDPRG 3) para um esquema de desacoplamento garp. Use para o comprimento de desacoplamento PCPD3 200 μs (Figura Suplementar 6C).
- Defina o atraso de recuperação entre varreduras para pelo menos D1 = 2 s ou mais.
NOTA: Para a escolha de um atraso de recuperação adequado, consulte a seção Discussão. - Defina as varreduras fictícias (DS) para pelo menos 64 para o experimento final (durante a configuração, DS = 4 é suficiente por razões de tempo). Como ponto de partida, use um número de varreduras (NS) de 4; use múltiplos de 4 se a relação sinal-ruído (SNR) for muito baixa (Figura Suplementar 6A).
- Defina o O1 para a frequência portadora calibrada da etapa 2.5. Defina o O2P para 176 ppm e copie o O3P do experimento 1 H-15N HSQC (Figura Suplementar 6A-C).
- Defina o comprimento de pulso P7 para o comprimento de pulso de 90° (15N) calibrado na etapa 2.6. Além disso, copie o nível de potência de pulso do pulso de 90° (15N) da etapa 2.6 para PLW3 e PLW7 (Figura Suplementar 6C).
- Defina o comprimento de pulso P1 e P19 para o comprimento de pulso de 90° (1H) calculado na etapa 2.4. (Figura 6B suplementar).
- Defina o número de incrementos na dimensão indireta TD = l3 * 2 * l6 (eda ou digite td na linha de comando).
NOTA: Todos os parâmetros da sequência de pulso descritos aqui são anotados adicionalmente na sequência de pulso. - Defina o pulso de forma SP5 para uma forma I-BURP2 (Iburp2.1000). Defina o comprimento do pulso P15 para 2000 μs. Abra a tela da ferramenta de forma clicando no E ao lado do pulso de forma I-BURP2 na janela de parâmetros da sequência de pulsos Topspin (ased) (Figura Suplementar 6B).
- Simule o pulso de forma com o comprimento correto clicando no botão Iniciar simulação de RMN. Verifique o comprimento do pulso moldado [μs] (= 2000 μs) e o ângulo de rotação [°] (= 180°) na janela de simulação. Clique em Iniciar NMR-SIM (Figura 7A suplementar).
- Verifique a faixa de excitação e escolha o comprimento de pulso I-BURP2 apropriado para cobrir a dispersão espectral de 1H da proteína, mas evite a excitação da água ao mesmo tempo. Defina P15 para o comprimento do pulso moldado da janela de simulação com o melhor pulso IBURP-2 simulado (Figura Suplementar 7B).
NOTA: O comprimento adequado é escolhido se o I-BURP2 excitar (> 95% de eficácia) toda a região da amida. Para simular um pulso I-BURP2 com um comprimento diferente, feche a janela do perfil de excitação e edite o comprimento do pulso moldado na janela de simulação. Clique em Iniciar NMR-SIM. Pulsos com comprimento menor têm uma faixa de excitação mais ampla em Hz. - Defina SPOFFS5 para alterar a frequência portadora do pulso I-BURP2 e mude a faixa de excitação para a esquerda ou direita para evitar que o pulso I-BURP2 toque e perturbe a magnetização da água (Figura Suplementar 6B).
- Para determinar o nível de potência apropriado do pulso moldado, abra a ferramenta de forma Bruker, clique no botão Iniciar simulação de RMN e defina o comprimento do pulso I-BURP2 para Comprimento do pulso moldado [μs]. Lembre-se do comprimento correspondente de um pulso retangular suave de 90° (1H), visível na janela de simulação no pulso quadrado correspondente de 90° [μs] (Figura Suplementar 7A).
- Digite calcpowlev. Use o pulso duro de 90° (1H) (P1) em microssegundos como o comprimento de referência (Figura Suplementar 8B) e o pulso retangular suave de 90° (1H) correspondente ao pulso I-BURP2 em microssegundos como o novo comprimento (Figura Suplementar 8A). Lembre-se da diferença de potência Δ em dB entre o pulso duro de 90° (1H) e o pulso retangular suave (Figura Suplementar 8C). Copie a força do pulso do pulso duro de 90 ° (1H) em dB para SPW5 e adicione a diferença memorizada Δ em dB (Figura Suplementar 6B).
- Para determinar o nível de potência do bloqueio de rotação, calcule o comprimento de pulso correspondente de 90° (15N) do bloqueio de rotação. Use a fórmula: comprimento de pulso de 90° (15N) em s = 0.25 / (potência de bloqueio de rotação em Hz).
- Use calcpowlev para calcular a diferença de potência Δ em dB entre a potência do bloqueio de rotação e o pulso forte de 90° (15N) (P7). Novo: comprimento de pulso de 90° (15N) do bloqueio de rotação em μs (Figura Suplementar 8A). Ref: comprimento de pulso duro de 90° (15N) em μs (Figura Suplementar 8B). Copie o nível de potência PLW7 do pulso forte de 90° (15N) em dB para o nível de potência de bloqueio de rotação PLW8 (Figura Suplementar 6C) e adicione a diferença de potência calculada Δ em dB (Figura Suplementar 8C).
- Copie o nível de potência do bloqueio de rotação PLW8 para os níveis de potência SPW8 e SPW9. (Figura 6C suplementar).
- Para determinar o nível de potência do desacoplamento de 15N, use calcpowlev para calcular a diferença de potência Δ entre a potência do pulso de desacoplamento de 90° (15N) (PCPD3) e o pulso duro de 90° (15N) (P7). Novo: PCPD3 em μs (200) (Figura 8A suplementar). Ref: comprimento de pulso duro de 90° (15N) em μs (Figura Suplementar 8B). Copie o nível de potência PLW7 do pulso forte de 90° (15N) em dB para o nível de desacoplamento PLW31 (Figura Suplementar 6C) e adicione Δ em dB (Figura Suplementar 8C).
- Para usar a compensação de temperatura, inclua #define TEMP_COMPENSATION (excluindo o ponto-e-vírgula na sequência de pulso). Defina P18 para a duração máxima do bloqueio de rotação usado no experimento 15N R1ρ .
NOTA: Recomenda-se desligar a compensação de temperatura para o primeiro teste do experimento. - No caso de amostras rotuladas com 15N 13C, inclua # para definir LABEL_CN no programa de pulso (excluindo o ponto e vírgula na sequência de pulso). Defina P4 para o comprimento de pulso calculado com a fórmula em78. Por exemplo, corresponde a 23,70 μs a 600 MHz e 11,85 μs a 1200 MHz. Digite calcpowlev para calcular a diferença de potência em dB entre P4 e o pulso forte (13C). Copie a potência de pulso forte (13C) em dB para PLW4 e adicione o Δ em dB. Copie PLW4 para PLW2 (Figura Suplementar 6B).
- Determine os atrasos de relaxamento para amostragem apropriada.
- Execute os primeiros oito FIDs (no caso L6 = 8) e processe-os (rser < novo número de experimento>). Escolha as entradas na vplist em que a intensidade de pico do experimento com o atraso mais longo diminui para pelo menos 1/e (aprox. 37%), mas não menos que 25% em comparação com o experimento com o atraso mais curto (que serve como referência).
- Ajuste as entradas restantes da vplist para obter uma amostragem uniforme das diminuições de intensidade entre o FID com o atraso mais longo e o FID com o atraso mais curto. Leia a NOTA e as especificações de energia da sonda.
NOTA: Os resíduos de relaxamento lento dominarão o primeiro FID. Ajuste as entradas restantes da vplist para obter uma amostragem uniforme das diminuições de intensidade entre o primeiro e o segundo FID. Não exceda a potência e o comprimento máximos permitidos da trava de rotação da sonda (consulte o manual da sonda e as especificações da sonda). Em caso de compensação de temperatura, verifique P18 (duração máxima do bloqueio de rotação), pois a duração geral do bloqueio de rotação de P18 será aplicada, dividida entre o atraso de relaxamento e a compensação de temperatura. Ele mantém o tempo (atraso de relaxamento) + tempo (compensação de temperatura) = const = P18. Para um bloqueio de rotação de 2 kHz, preferimos não exceder uma duração de bloqueio de rotação de mais de 65 ms, usando um atraso de recuperação (D1) de 2 s. Nesse caso, um compromisso entre a duração máxima do bloqueio de rotação e o atraso de relaxamento mais longo deve ser feito em favor da duração máxima permitida do bloqueio de rotação. Em caso de atrasos de recuperação D1 mais longos, poderes de bloqueio de rotação mais longos podem ser aplicáveis. O que conta é a deposição geral de energia na sonda ao longo do tempo. Se forem necessárias durações de bloqueio de rotação mais longas, aumente apenas os comprimentos de bloqueio de rotação em pequenos passos e observe o sinal de bloqueio. Como regra geral, ajuste o nível de bloqueio no terço superior da tela de bloqueio. Se o nível de bloqueio reduzir em mais de dois quadrados durante a execução da sequência de pulso, a potência de bloqueio de rotação depositada na sonda é demais.
- Digite rga na linha de comando para determinar o ganho do receptor. Para uma boa relação sinal-ruído, procure um valor de ganho do receptor (rg) entre 128 e 256, o que prova uma boa supressão de água.
NOTA: Um ganho do receptor entre 128 e 256 não pode ser alcançado indica um sinal de água muito forte. Nesse caso, a potência e as durações do gradiente são ajustadas suavemente até que o ganho do receptor seja alcançado. Veja também a seção Discussão. - Inicie uma execução de teste do experimento 15N R1ρ digitando zg na linha de comando. Verifique se o sinal de água está bem suprimido para todos os atrasos (1 a 8, no caso de 8 atrasos na vplist). Verifique também o incremento 9, que é o segundo incremento do esquema de detecção de quadratura (Eco/Anti-Eco).
- Copie o experimento HSQC (tipo edc) para um novo diretório. Digite ased e altere o programa de pulso para o experimento 15N R1ρ clicando nos três pontos da linha de parâmetro PULPROG nos parâmetros da sequência de pulso (ased, consulte a Figura Suplementar 3A). Clique duas vezes no programa de pulso, clique em Definir PULPROG para conjunto de dados (consulte a Figura Suplementar 3B) e clique em OK.
- Configure o experimento 15N R1 .
- Copie o experimento 15N R1ρ e altere o programa de pulso para o experimento 15N R1 .
- Digite vclist, gere um vclist e insira o vclist fictício descrito em70: 0, 24, 6, 20, 4, 16, 8, 12; uma nova linha para cada entrada vclist).
- Aberto ased. Verifique os comprimentos e intensidades do gradiente. Altere GPZ9 para 14%.
- Verifique os comprimentos de pulso, as formas de pulso e as intensidades de pulso (os valores corretos devem ter sido copiados do experimento 15N R1ρ ).
- No caso de amostras marcadas com 15N13C, use #define LABEL_CN no programa de pulso.
- Para o uso da compensação de temperatura, inclua #define TEMP_COMPENSATION.
- Execute os primeiros oito FIDs (no caso L6 = 8) e processe-os (rser < novo número de experimento>). Escolha as entradas na vclist em que a intensidade de pico do experimento com o atraso mais longo diminui para pelo menos 1/e (aprox. 37%), mas não menos que 25% em comparação com o experimento com o atraso mais curto. Ajuste as entradas restantes da vplist para obter uma amostragem uniforme das diminuições de intensidade entre o FID com o atraso mais longo e o FID com o atraso mais curto.
NOTA: Somente números pares são permitidos para entradas vclist.
- Configure o experimento {1H}-15N NOE.
- Copie o experimento de relaxamento 15N R1 e altere a sequência de pulsos para o esquema de pulso {1H}-15N NOE.
- Defina a duração do período de saturação com o contador de loop L8. O espaçamento entre os pulsos de 180° (1H) é de 22 ms79 (2* 1 / JNH). Para obter os resultados mais precisos, use um período de saturação de cerca de 5 vezes os 15N T1 (15N) da proteína. Aqui: 5 s. Isso corresponderia a L8 = 230.
NOTA: A seção Discussão fornece conselhos adicionais sobre como escolher o período de saturação/atraso de recuperação. - Altere o comprimento e a força dos gradientes conforme descrito no esquema de pulso. Use formas de gradiente seno.20.
- Defina o número de incrementos na dimensão indireta igual a TD = l3 * 2 * 2
- No caso de amostras marcadas com 15N13C, use #define LABEL_CN e -DLABEL_CN como ZGOPTNS.
- Execute os esquemas de pulso digitando zg na linha de comando.
4. Processamento e análise dos experimentos de RMN registrados
NOTA: Os espectros foram registrados usando um sistema Bruker. O processamento é realizado usando um sistema operacional Unix ou Linux. O processamento dos espectros e a análise dos dados foram realizados usando NMRPipe80 e python3. O software NMRPipe pode ser baixado em https://www.ibbr.umd.edu/nmrpipe/index.html. Os scripts de processamento baseados em NMRPipe são baixados do site: https://www.ipb.hhu.de/en/teams/team-lakomek/pulsesequences ou do repositório estendido do Banco de Ressonância Magnética Biológica (BMRB) (bmrbig102). Recomenda-se o uso do NMRPipe. Se o NMRPipe não estiver disponível ou desejado, alternativas como CCPN81 ou SPARKY (SPARKY 3, ou seus sucessores NMRFAM-SPARKY82 ou POKY83 ) podem ser usadas.
- Processar e analisar 15NR1 experimentar.
NOTA: Os comandos a seguir são executados em um Terminal Unix.- Copie os arquivos no diretório baixado R1_processing para o diretório que contém os dados R1 experimentais (diretório de trabalho).
- Torne os arquivos executáveis pelo comando do terminal: > chmod u+x *.com
- Execute > bruker para abrir o utilitário de conversão NMRPipe e gerar o arquivo fid.com. Clique em Ler parâmetros. Altere a Contagem de Dimensões para dois se três dimensões forem contadas e clique em Ler Parâmetros novamente. Em seguida, clique em Limpar script, Atualizar script e Salvar script (Figura suplementar 9).
NOTA: Se a frequência portadora não estiver correta, altere a posição central PPM depois de clicar em Ler parâmetros pela segunda vez. - Executar > ./fid.com
- Converta o vclist em um vplist com entradas ms: delay in ms = 40 ms * entrada do vclist. Use > cp vclist vplist e edite o vplist.
- Use > ./split.com para dividir os dados pseudo-3D em oito arquivos 2D.
- Edite os valores tauValues (valores vplist na ordem do vplist) e os valores idxExpmt (ordem dos tauValues em relação ao tempo) em nmrproc.com (Figura Suplementar 10).
- Use > ./nmrproc.com para processar os dados brutos de todos os oito espectros 2D.
- Use > nmrDraw para desenhar os espectros.
- Use o espectro de referência (primeira entrada em vplist) para essas etapas. Mude para esse espectro alterando a dimensão z para um (canto superior esquerdo). Ajuste o nível de contorno usando + e - ao lado do botão Fator . Redesenhe o espectro com o botão Desenhar.
- Use um nível de contorno onde todos os sinais de interesse possam ser reconhecidos com apenas alguns sinais provenientes do ruído (o nível de contorno está na borda do nível de ruído) (Figura 11 suplementar).
- Corrija a fase do espectro, tipo v para projeção vertical de sinal 1D e h para projeção horizontal de sinal 1D. Ajuste a fase com P0 e P1 (Figura Suplementar 11), lembre-se da diferença de fase e corrija a fase no arquivo nmrproc.com (Figura Suplementar 10).
NOTA: Se o espectro for invertido na dimensão 15N, remova o -neg na linha: | nmrPipe -fn FT -neg \ (Figura Suplementar 10). - Feche nmrDraw e processe novamente com > nmrproc.com. Para Seleção de pico (depois que o espectro é faseado corretamente e o espectro de referência é exibido com um nível de contorno onde todos os picos estão presentes), digite K (shift + k), pressione Detectar e clique em Salvar. (Figura 11 suplementar).
- Copie uma lista de picos para o diretório de trabalho e altere -assName em ass.com para o nome da lista de picos (Figura 12 suplementar).
NOTA: Um exemplo de uma lista de picos no formato correto está disponível em https://www.ipb.hhu.de/en/teams/team-lakomek/pulsesequences
e no repositório estendido do Banco de Ressonância Magnética Biológica (BMRB) (bmrbig102). - Execute > ./ass.com para atribuir os espectros. Verifique a atribuição que o script sugere para os vários picos. Use o botão esquerdo do mouse para atribuir e o botão direito do mouse para desatribuir picos. Use o botão Avançar para ir para o aminoácido seguinte e Anterior para ir para o aminoácido anterior (Figura Suplementar 13).
NOTA: O nível de contorno pode ser alterado com + e - no canto superior direito. Use o botão Draw para redesenhar o espectro com o novo nível de contorno (Figura Suplementar 13). - Depois de concluir a tarefa, clique em Salvar e em Salvar novamente. Depois, clique em Sair, Cancelar e Sair (Figura Suplementar 13).
- Execute > python3 NoneDeleter.py para criar relax.tab, que contém apenas informações sobre picos atribuídos.
- Execute > ./relax.com para gerar o script autofit.com.
- Execute > ./autoFit.com para realizar uma interpolação de Fourier e gerar pseudo proporções dos diferentes tempos de atraso para cada aminoácido (axt.tab e nlin.tab são gerados).
- Execute > ./model.com para gerar o script modelExp.com com base nos dados em nlin.tab.
- Execute > ./modelExp.com para ajustar a curva de evolução de cada resíduo com um modelo de otimização de mínimos quadrados não linear.
- Execute > bash summary.tcl -in relax.tab > t1.tab. Ignore uma possível mensagem de erro: GDB Aviso: podem ocorrer 21 valores ausentes no arquivo de tabela .
- Copie t1.tab para um diretório recém-criado R1R2.
- Processe e analise o experimento 15N R1ρ .
- Copie os arquivos no diretório baixado R1rho_processing para o diretório que contém os dados experimentais do R1ρ (diretório de trabalho).
- Execute > chmod u+x *.com
- Execute > bruker para abrir o utilitário de conversão NMRPipe e gerar o arquivo fid.com. Veja também o processamento dos espectros 15N R1 .
- Executar > ./fid.com
- Execute > ./split.com para dividir os dados pseudo-3D em oito arquivos 2D.
- Execute > ./nmrproc.com (Edite os tauValues [valores vplist] e o idxExpmt de antemão) para processar os dados brutos de todos os oito espectros 2D (Figura Suplementar 10).
- Execute > nmrDraw para desenhar os espectros e escolher os picos. Use o espectro de referência (primeira entrada em vplist) para essas etapas. Seguir o mesmo procedimento que para a experiência 15N R1 .
- Copie uma lista de picos para o diretório de trabalho e altere -assName em ass.com para o nome da lista de picos (Figura 12 suplementar).
- Execute > ./ass.com e siga o mesmo procedimento do experimento 15N R1 .
- Execute > python3 NoneDeleter.py para criar relax.tab, que contém apenas informações sobre picos atribuídos.
- Executar > ./relax.com
- Executar > ./autoFit.com
- Executar > ./model.com
- Executar > ./modelExp.com
- Execute > bash summary.tcl -in relax.tab > t1rho. tab. Ignorar possível mensagem de erro: GDB Aviso: podem ocorrer 21 valores ausentes no arquivo de tabela .
- Copie o seguinte cabeçalho no arquivo t1rho.tab (os valores podem ser obtidos no arquivo fid.com) (Figura Suplementar 14):
OBSERVAÇÃO 600MHz para T1rho
OBSERVAÇÃO -yOBS = 60.818 (coloque aqui o valor encontrado em fid.com)
OBSERVAÇÃO -yCAR = 119,06 (coloque aqui o valor encontrado em fid.com) - Execute > ./extract_PPM.com para gerar uma tabela chamada fusionOffset.tab que contém o deslocamento da frequência de pico de 15N em relação à frequência da portadora (que será necessária para calcular o ângulo θ para a extração de dados R2 dos dados R1 e R1ρ ).
- Resuma os dados de relaxamento.
- Copie o t1rho.tab e o fusionOffset.tab para o diretório R1R2.
- Copie o script crtR1p.tcl no diretório R1R2, ajuste a frequência do espectrômetro e a potência do bloqueio de rotação e execute bash crtR1p.tcl | sort -n > R1R2.tab. (Figura 15 suplementar). R1R2.tab conterá as constantes de taxa R1ρ ajustadas, o ângulo, as constantes de taxa R2 e as constantes de taxa R1.
- Processe e analise o experimento hetNOE.
- Copie os arquivos no diretório baixado NOE_processing para o diretório de dados NOE experimental (diretório de trabalho).
- Execute > chmod u+x *.com
- Execute > bruker para abrir o utilitário de conversão NMRPipe e gerar o arquivo fid.com. Clique em Ler parâmetros. Altere a Contagem de Dimensões para dois se três dimensões foram contadas e pressione Ler Parâmetros novamente. Em seguida , pressione Limpar script, Atualizar script e Salvar script.
- Executar > ./fid.com
- Execute > ./split.com (Uma vplist com duas entradas (0, 1) deve ser criada antes)
- Executar > ./ft2.com
- Execute > nmrDraw para desenhar o espectro e escolher os picos no espectro de referência (dimensão z = 1). Extraia e lembre-se do ruído (pressione T = shift + t). A correção de fase e a colheita do pico seguem o mesmo procedimento do experimento 15N R1 .
- Copie uma lista de picos para o diretório de trabalho e altere -assName em ass.com para o nome da lista de picos (Figura 12 suplementar)
- Execute > ./ass.com para atribuir os espectros. Seguir o mesmo procedimento descrito para a experiência 15N R1 .
- Execute > python3 NoneDeleter.py para criar relax.tab.
- Executar > ./script.com
- Executar > ./autoFit.com
- Altere o ruído em error_hetnoe.tcl.
- Execute > bash error_hetnoe.tcl |sort -n > hetNOE.tab
Access restricted. Please log in or start a trial to view this content.
Resultados
A seguir, são apresentados alguns dados exemplares de relaxamento de RMN registrados na proteína SNARE vesicular Synaptobrevin-2 (1-96), freqüentemente chamada de VAMP2 (vesícula-associada à proteína 2). Para registrar os dados de RMN, usamos uma amostra de 171 μM 15N Synaptobrevin-2 (1-96) (apelidada de Syb-2 a seguir) em tampão MES de 50 mM (pH 6,0) contendo 150 mM de NaCl, 0,1 mM de TCEP e 1 mM de EDTA. Todos os dados experimentais foram registrados a 278,15 K usand...
Access restricted. Please log in or start a trial to view this content.
Discussão
Este protocolo descreveu a configuração de experimentos de relaxamento de RMN 15N por Lakomek et al.69 e Stief et al.70. Nós nos concentramos nas sequências de pulso de RMN usando um esquema de detecção HSQC aprimorado por sensibilidade. Os experimentos 15N R1 e R1ρ são implementados conforme descrito em detalhes por Stief et al.70, e o experimento hetNOE é descrito por Lakomek et
Access restricted. Please log in or start a trial to view this content.
Divulgações
Os autores declaram que não têm interesses financeiros concorrentes conhecidos ou relacionamentos pessoais que possam ter influenciado o trabalho relatado neste artigo.
Agradecimentos
Agradecemos a Melinda Jaspert e Kevin Bochinsky pelas discussões úteis. N.L. agradece à Fundação Alemã de Ciência pelo financiamento através do Programa Heisenberg (número de concessão DFG 433700474). Este trabalho é ainda apoiado pelo projeto "Determinantes virológicos e imunológicos da patogênese do COVID-19 - lições para se preparar para futuras pandemias (KA1-Co-02 "COVIPA"), uma doação da Iniciativa e Fundo de Rede da Associação Helmholtz. Reconhecemos o generoso acesso ao Centro de RMN Biomolecular Jülich-Düsseldorf, administrado em conjunto pelo Forschungszentrum Jülich e pela Universidade Heinrich Heine de Düsseldorf (HHU).
Access restricted. Please log in or start a trial to view this content.
Materiais
| Name | Company | Catalog Number | Comments |
| Bruker 600 MHz AVANCE III HD spectrometer | Bruker | https://www.bruker.com/en/products-and-solutions/mr/nmr/avance-nmr-spectrometer.html | NMR experiments conducted |
Referências
- Sekhar, A., Kay, L. E. An NMR view of protein dynamics in health and disease. Annu Rev Biophys. 48, 297-319 (2019).
- Rosenzweig, R., Kay, L. E. Bringing dynamic molecular machines into focus by methyl-TROSY NMR. Annu Rev Biochem. 83, 291-315 (2014).
- Palmer, A. G. 3rd Enzyme dynamics from NMR spectroscopy. Acc Chem Res. 48 (2), 457-465 (2015).
- Reif, B. Ultra-high resolution in MAS solid-state NMR of perdeuterated proteins: implications for structure and dynamics. J Magn Reson. 216, 1-12 (2012).
- Schanda, P., Ernst, M. Studying dynamics by magic-angle spinning solid-state NMR spectroscopy: Principles and applications to biomolecules. Prog Nucl Magn Reson Spectrosc. 96, 1-46 (2016).
- Le Marchand, T., et al. (1)H-H-detected biomolecular NMR under fast magic-angle spinning. Chem Rev. 122 (10), 9943-10018 (2022).
- Pervushin, K., Riek, R., Wider, G., Wuthrich, K. Attenuated T2 relaxation by mutual cancellation of dipole-dipole coupling and chemical shift anisotropy indicates an avenue to NMR structures of very large biological macromolecules in solution. Proc Natl Acad Sci U S A. 94 (23), 12366-12371 (1997).
- Tzakos, A. G., Grace, C. R., Lukavsky, P. J., Riek, R. NMR techniques for very large proteins and RNAs in solution. Annu Rev Biophys Biomol Struct. 35, 319-342 (2006).
- Sprangers, R., Kay, L. E. Quantitative dynamics and binding studies of the 20S proteasome by NMR. Nature. 445 (7128), 618-622 (2007).
- Wright, P. E., Dyson, H. J. Intrinsically disordered proteins in cellular signalling and regulation. Nat Rev Mol Cell Biol. 16 (1), 18-29 (2015).
- van der Lee, R., et al. Classification of intrinsically disordered regions and proteins. Chem Rev. 114 (13), 6589-6631 (2014).
- Tompa, P. Intrinsically unstructured proteins. Trends Biochem Sci. 27 (10), 527-533 (2002).
- Dyson, H. J., Wright, P. E. Coupling of folding and binding for unstructured proteins. Curr Opin Struct Biol. 12 (1), 54-60 (2002).
- Uversky, V. N. Intrinsically disordered proteins in overcrowded milieu: Membrane-less organelles, phase separation, and intrinsic disorder. Current Opinion in Structural Biology. 44, 18-30 (2017).
- Patel, A., et al. A liquid-to-solid phase transition of the ALS protein FUS accelerated by disease mutation. Cell. 162 (5), 1066-1077 (2015).
- Abyzov, A., Blackledge, M., Zweckstetter, M. Conformational dynamics of intrinsically disordered proteins regulate biomolecular condensate chemistry. Chem Rev. 122 (6), 6719-6748 (2022).
- Uversky, V. N. Unusual biophysics of intrinsically disordered proteins. Biochim Biophys Acta. 1834 (5), 932-951 (2013).
- Papoian, G. A. Proteins with weakly funneled energy landscapes challenge the classical structure-function paradigm. Proc Natl Acad Sci U S A. 105 (38), 14237-14238 (2008).
- Uversky, V. N., Gillespie, J. R., Fink, A. L. Why are "natively unfolded" proteins unstructured under physiologic conditions. Proteins. 41 (3), 415-427 (2000).
- Bah, A., et al. Folding of an intrinsically disordered protein by phosphorylation as a regulatory switch. Nature. 519 (7541), 106-109 (2015).
- Tompa, P., Schad, E., Tantos, A., Kalmar, L. Intrinsically disordered proteins: emerging interaction specialists. Curr Opin Struct Biol. 35, 49-59 (2015).
- Dyson, H. J., Wright, P. E. Perspective: the essential role of NMR in the discovery and characterization of intrinsically disordered proteins. J Biomol NMR. 73 (12), 651-659 (2019).
- Adamski, W., et al. A unified description of intrinsically disordered protein dynamics under physiological conditions using NMR spectroscopy. J Am Chem Soc. 141 (44), 17817-17829 (2019).
- Dobson, C. M. Protein folding and misfolding. Nature. 426 (6968), 884-890 (2003).
- Bertoncini, C. W., et al. Release of long-range tertiary interactions potentiates aggregation of natively unstructured alpha-synuclein. Proc Natl Acad Sci U S A. 102 (5), 1430-1435 (2005).
- Buell, A. K., et al. Solution conditions determine the relative importance of nucleation and growth processes in alpha-synuclein aggregation. Proc Natl Acad Sci U S A. 111 (21), 7671-7676 (2014).
- Salvi, N., Abyzov, A., Blackledge, M. Atomic resolution conformational dynamics of intrinsically disordered proteins from NMR spin relaxation. Prog Nucl Magn Reson Spectrosc. 102, 43-60 (2017).
- Schneider, R., Blackledge, M., Jensen, M. R. Elucidating binding mechanisms and dynamics of intrinsically disordered protein complexes using NMR spectroscopy. Curr Opin Struct Biol. 54, 10-18 (2019).
- Rezaei-Ghaleh, N., Blackledge, M., Zweckstetter, M. Intrinsically disordered proteins: from sequence and conformational properties toward drug discovery. Chembiochem. 13 (7), 930-950 (2012).
- Jensen, M. R., Salmon, L., Nodet, G., Blackledge, M. Defining conformational ensembles of intrinsically disordered and partially folded proteins directly from chemical shifts. J Am Chem Soc. 132 (4), 1270-1272 (2010).
- Camacho-Zarco, A. R., et al. NMR provides unique insight into the functional dynamics and interactions of intrinsically disordered proteins. Chem Rev. 122 (10), 9331-9356 (2022).
- Mittag, T., Forman-Kay, J. D. Atomic-level characterization of disordered protein ensembles. Curr Opin Struct Biol. 17 (1), 3-14 (2007).
- Dyson, H. J., Wright, P. E. Unfolded proteins and protein folding studied by NMR. Chem Rev. 104 (8), 3607-3622 (2004).
- Dyson, H. J., Wright, P. E. NMR illuminates intrinsic disorder. Curr Opin Struct Biol. 70, 44-52 (2021).
- Sugase, K., Dyson, H. J., Wright, P. E. Mechanism of coupled folding and binding of an intrinsically disordered protein. Nature. 447 (7147), 1021-1025 (2007).
- Bessa, L. M., et al. The intrinsically disordered SARS-CoV-2 nucleoprotein in dynamic complex with its viral partner nsp3a. Sci Adv. 8 (3), eabm4034(2022).
- Milles, S., Salvi, N., Blackledge, M., Jensen, M. R. Characterization of intrinsically disordered proteins and their dynamic complexes: From in vitro to cell-like environments. Prog Nucl Magn Reson Spectrosc. 109, 79-100 (2018).
- Salvi, N., Abyzov, A., Blackledge, M. Atomic resolution conformational dynamics of intrinsically disordered proteins from NMR spin relaxation. Prog Nucl Magn Reson Spectrosc. 102 - 103, 43-60 (2017).
- Gill, M. L., Byrd, R. A., Palmer, A. G. III Dynamics of GCN4 facilitate DNA interaction: a model-free analysis of an intrinsically disordered region. Phys Chem Chem Phys. 18 (8), 5839-5849 (2016).
- Bertoncini, C. W., et al. Structural characterization of the intrinsically unfolded protein beta-synuclein, a natural negative regulator of alpha-synuclein aggregation. J Mol Biol. 372 (3), 708-722 (2007).
- Rezaei-Ghaleh, N., et al. Local and global dynamics in intrinsically disordered synuclein. Angew Chem Int Ed Engl. 57 (46), 15262-15266 (2018).
- Rezaei-Ghaleh, N., Parigi, G., Zweckstetter, M. Reorientational dynamics of amyloid-beta from NMR spin relaxation and molecular simulation. J Phys Chem Lett. 10 (12), 3369-3375 (2019).
- Palmer, A. G. NMR characterization of the dynamics of biomacromolecules. Chem Rev. 104 (8), 3623-3640 (2004).
- Palmer, A. G., Massi, F. Characterization of the dynamics of biomacromolecules using rotating-frame spin relaxation NMR spectroscopy. Chem Rev. 106 (5), 1700-1719 (2006).
- Kay, L. E., Torchia, D. A., Bax, A. Backbone dynamics of proteins as studied by 15N inverse detected heteronuclear NMR spectroscopy: application to staphylococcal nuclease. Biochemistry. 28 (23), 8972-8979 (1989).
- Mittermaier, A., Kay, L. E. Review - New tools provide new insights in NMR studies of protein dynamics. Science. 312 (5771), 224-228 (2006).
- Abragam, A. Principles of Nuclear Magnetism. , Clarendon Press. Oxford. (1983).
- Ernst, R. R., Bodenhausen, G., Wokaun, A. Principles of Nuclear Magnetic Resonance in One and Two Dimensions. , Oxford University Press. Oxford. (1990).
- Cavanagh, J., Fairbrother, W. J., Palmer, A. G. III, Skelton, N. J. Protein NMR Spectroscopy: Principles and Practice. , Elsevier, Academic Press. (1995).
- Levitt, M. H. Spin Dynamics: Basics of Nuclear Magnetic Resonance. , John Wiley and Sons. (2008).
- Keeler, J. Understanding NMR Spectroscopy. , John Wiley and Sons. (2010).
- Bloembergen, N., Purcell, E. M., Pound, R. V. Relaxation effects in nuclear magnetic resonance absorption. Phys Rev. 73 (7), 679-712 (1948).
- Wangsness, R. K., Bloch, F. The dynamical theory of nuclear induction. Phys Rev. 89 (4), 728-739 (1953).
- Redfield, A. G. On the theory of relaxation processes. IBM J Res Dev. 1 (1), 19-31 (1957).
- Peng, J. W., Wagner, G. Mapping of the spectral densities of N-H bond motions in eglin c using heteronuclear relaxation experiments. Biochemistry. 31 (36), 8571-8586 (1992).
- Farrow, N. A., et al. Backbone dynamics of a free and a phosphopeptide-complexed src homology-2 domain studied by n-15 NMR relaxation. Biochemistry. 33 (19), 5984-6003 (1994).
- Tolman, J. R., Ruan, K. NMR residual dipolar couplings as probes of biomolecular dynamics. Chem Rev. 106 (5), 1720-1736 (2006).
- Mulder, F. A. A., Skrynnikov, N. R., Hon, B., Dahlquist, F. W., Kay, L. E. Measurement of slow (mu s-ms) time scale dynamics in protein side chains by N-15 relaxation dispersion NMR spectroscopy: Application to Asn and Gln residues in a cavity mutant of T4 lysozyme. J Am Chem Soc. 123 (5), 967-975 (2001).
- Tollinger, M., Skrynnikov, N. R., Mulder, F. A. A., Forman-Kay, J. D., Kay, L. E. Slow dynamics in folded and unfolded states of an SH3 domain. J Am Chem Soc. 123 (46), 11341-11352 (2001).
- Akke, M., Palmer, A. G. Monitoring macromolecular motions on microsecond to millisecond time scales by R(1)rho-R(1) constant relaxation time NMR spectroscopy. J Am Chem Soc. 118 (4), 911-912 (1996).
- Loria, J. P., Rance, M., Palmer, A. G. A relaxation-compensated Carr-Purcell-Meiboom-Gill sequence for characterizing chemical exchange by NMR spectroscopy. J Am Chem Soc. 121 (10), 2331-2332 (1999).
- Vallurupalli, P., Bouvignies, G., Kay, L. E. Studying "Invisible" excited protein states in slow exchange with a major state conformation. J Am Chem Soc. 134 (19), 8148-8161 (2012).
- Jeener, J., Meier, B. H., Bachmann, P., Ernst, R. R. Investigation of exchange processes by 2-dimensional NMR-spectroscopy. J Chem Phys. 71 (11), 4546-4553 (1979).
- Palmer, A. G., Kroenke, C. D., Loria, J. P. Nuclear magnetic resonance methods for quantifying microsecond-to-millisecond motions in biological macromolecules. Methods Enzymol. 339, 204-238 (2001).
- Clore, G. M., Iwahara, J. Theory, practice, and applications of paramagnetic relaxation enhancement for the characterization of transient low-population states of biological macromolecules and their complexes. Chem Rev. 109 (9), 4108-4139 (2009).
- Iwahara, J., Clore, G. M. Detecting transient intermediates in macromolecular binding by paramagnetic NMR. Nature. 440 (7088), 1227-1230 (2006).
- Lakomek, N. A., et al. Residual dipolar couplings as a tool to study molecular recognition of ubiquitin. Biochem Soc Trans. 36 (Pt 6), 1433-1437 (2008).
- Johnson, C. N., Libich, D. S. Paramagnetic relaxation enhancement for detecting and characterizing self-associations of intrinsically disordered proteins. J Vis Exp. (175), e63057(2021).
- Lakomek, N. A., Ying, J., Bax, A. Measurement of (1)(5)N relaxation rates in perdeuterated proteins by TROSY-based methods. J Biomol NMR. 53 (3), 209-221 (2012).
- Stief, T., Vormann, K., Lakomek, N. A. Sensitivity-enhanced NMR (15)N R(1) and R(1rho) relaxation experiments for the investigation of intrinsically disordered proteins at high magnetic fields. Methods. 223, 1-15 (2024).
- Kay, L. E., Keifer, P., Saarinen, T. Pure absorption gradient enhanced heteronuclear single quantum correlation spectroscopy with improved sensitivity. J Am Chem Soc. 114 (26), 10663-10665 (1992).
- Geen, H., Freeman, R. Band-selective radiofrequency pulses. J Magn Reson (1969). 93 (1), 93-141 (1991).
- Desvaux, H., Berthault, P. Study of dynamic processes in liquids using off-resonance RF irradiation. Prog Nucl Magn Reson Spectrosc. 35 (4), 295-340 (1999).
- Overhauser, A. W. Polarization of nuclei in metals. Phys Rev. 91 (2), 476-476 (1953).
- Overhauser, A. W. Polarization of nuclei in metals. Phys Rev. 92 (2), 411-415 (1953).
- Kumari, P., Frey, L., Sobol, A., Lakomek, N. A., Riek, R. (15)N transverse relaxation measurements for the characterization of micros-ms dynamics are deteriorated by the deuterium isotope effect on (15)N resulting from solvent exchange. J Biomol NMR. 72 (3-4), 125-137 (2018).
- Karschin, N., Krenek, S., Heyer, D., Griesinger, C. Extension and improvement of the methanol-d(4) NMR thermometer calibration. Magn Reson Chem. 60 (4), 203-209 (2022).
- Sattler, M., Schleucher, J., Griesinger, C. Heteronuclear multidimensional NMR experiments for the structure determination of proteins in solution employing pulsed field gradients. Prog Nucl Magn Reson Spectrosc. 34 (2), 93-158 (1999).
- Ferrage, F., Cowburn, D., Ghose, R. Accurate sampling of high-frequency motions in proteins by steady-state N-{H} nuclear Overhauser effect measurements in the presence of cross-correlated relaxation. J Am Chem Soc. 131 (17), 6048-6049 (2009).
- Delaglio, F., et al. NMRPipe: a multidimensional spectral processing system based on UNIX pipes. J Biomol NMR. 6 (3), 277-293 (1995).
- Skinner, S. P., et al. CcpNmr AnalysisAssign: a flexible platform for integrated NMR analysis. J Biomol NMR. 66 (2), 111-124 (2016).
- Lee, W., Tonelli, M., Markley, J. L. NMRFAM-SPARKY: enhanced software for biomolecular NMR spectroscopy. Bioinformatics. 31 (8), 1325-1327 (2015).
- Lee, W., Rahimi, M., Lee, Y., Chiu, A. POKY: a software suite for multidimensional NMR and 3D structure calculation of biomolecules. Bioinformatics. 37 (18), 3041-3042 (2021).
- Abyzov, A., et al. Identification of dynamic modes in an intrinsically disordered protein using temperature-dependent NMR relaxation. J Am Chem Soc. 138 (19), 6240-6251 (2016).
- Singh, A., Purslow, J. A., Venditti, V. 15N CPMG relaxation dispersion for the investigation of protein conformational dynamics on the micros-ms timescale. J Vis Exp. (170), e62395(2021).
- Gopalan, A. B., Hansen, D. F., Vallurupalli, P. CPMG experiments for protein minor conformer structure determination. Methods Mol Biol. 1688, 223-242 (2018).
- Tugarinov, V., Kay, L. E. Methyl groups as probes of structure and dynamics in NMR studies of high-molecular-weight proteins. Chembiochem. 6 (9), 1567-1577 (2005).
- Lipari, G., Szabo, A. Model-free approach to the interpretation of nuclear magnetic resonance relaxation in macromolecules. 1. Theory and range of validity. J Am Chem Soc. 104 (17), 4546-4559 (1982).
- Lipari, G., Szabo, A. Model-Free Approach to the interpretation of nuclear magnetic-resonance relaxation in macromolecules .2. Analysis of experimental results. J Am Chem Soc. 104 (17), 4559-4570 (1982).
- Clore, G. M., et al. Deviations from the simple two-parameter model-free approach to the interpretation of nitrogen-15 nuclear magnetic relaxation of proteins. J Am Chem Soc. 112 (12), 4989-4991 (1990).
- Lemaster, D. M. Larmor frequency selective model free analysis of protein NMR relaxation. J Biomol NMR. 6 (4), 366-374 (1995).
- Peng, J. W., Wagner, G. Mapping of spectral density functions using heteronuclear NMR relaxation measurements. J Magn Reson (1969). 98 (2), 308-332 (1992).
- Farrow, N. A., Zhang, O. W., FormanKay, J. D., Kay, L. E. Characterization of the backbone dynamics of folded and denatured states of an SH3 domain. Biochemistry. 36 (9), 2390-2402 (1997).
- Khan, S. N., et al. Distribution of pico- and nanosecond motions in disordered proteins from nuclear spin relaxation. Biophys J. 109 (5), 988-999 (2015).
- Smith, A. A., Ernst, M., Meier, B. H., Ferrage, F. Reducing bias in the analysis of solution-state NMR data with dynamics detectors. J Chem Phys. 151 (3), 034102(2019).
- Zumpfe, K., Smith, A. A. Model-free or not. Front Mol Biosci. 8, 727553(2021).
- Robustelli, P., Trbovic, N., Friesner, R. A., Palmer, A. G. Conformational dynamics of the partially disordered yeast transcription factor GCN4. J Chem Theory Comput. 9 (11), 5190-5200 (2013).
- Salvi, N., Abyzov, A., Blackledge, M. Multi-timescale dynamics in intrinsically disordered proteins from NMR relaxation and molecular simulation. J Phys Chem Lett. 7 (13), 2483-2489 (2016).
Access restricted. Please log in or start a trial to view this content.
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados