Bu içeriği görüntülemek için JoVE aboneliği gereklidir. Oturum açın veya ücretsiz deneme sürümünü başlatın.
Method Article
NMR 15N Proteinlerin Pikosaniye ve Nanosaniye Yapısal Dinamiğinin İncelenmesi için Gevşeme Deneyleri
Bu Makalede
Özet
Nükleer manyetik rezonans (NMR) spektroskopisi, yapısal protein dinamiklerini kalıntıya özgü bir şekilde karakterize edebilir. NMR 15N R1 ve R2 gevşeme ve {1H}-15N heteronükleer Overhauser etkisi (hetNOE) deneylerini kaydetmek için pikosaniye ila nanosaniye zaman ölçeğine duyarlı uygulamalı bir protokol sunuyoruz.
Özet
Nükleer manyetik rezonans (NMR) spektroskopisi, çözelti içindeki ve fizyolojik sıcaklıklar altında proteinlerin incelenmesine izin verir. Sıklıkla, ya protein omurgasının amid grupları ya da yan zincirlerdeki metil grupları, proteinlerdeki yapısal dinamiklerin raporlayıcıları olarak kullanılır. 15N etiketli ve tamamen protonlanmış numuneler üzerinde küresel proteinlerin protein omurgasının yapısal dinamik çalışması, genellikle 50 kDa'ya kadar moleküler ağırlığa sahip proteinler için iyi sonuç verir. Enine gevşeme optimize edilmiş spektroskopi (TROSY) ile birlikte yan zincir döterasyonu uygulandığında, bu sınır küresel proteinler için 200 kDa'ya ve odak yan zincirler üzerinde olduğunda 1 MDa'ya kadar uzatılabilir. Doğası bozuk proteinler (IDP'ler) veya içsel olarak düzensiz bölgelere sahip proteinler (IDR'ler) araştırıldığında, bu ağırlık sınırlamaları geçerli değildir, ancak çok ötesine geçebilir. Bunun nedeni, yüksek iç esneklik ile karakterize edilen IDP'lerin veya IDR'lerin sıklıkla dinamik olarak ayrıştırılmasıdır. Çeşitli NMR yöntemleri, pikosaniyelerden saatlere kadar geniş bir zaman ölçeği aralığında yapısal protein dinamikleri hakkında atomik çözünürlüklü bilgiler sunar. Standart 15N gevşeme ölçümleri, bir proteinin iç esnekliğini gözden geçirir ve hızlı piko-nanosaniye zaman ölçeğinde deneyimlenen protein omurga dinamiklerini karakterize eder. Bu makale, NMR 15N R1, R2 ve heteronükleer Overhauser etkisi (hetNOE) deneylerinin kurulması ve kaydedilmesi için uygulamalı bir protokol sunmaktadır. Örnek verileri gösteriyoruz ve daha karmaşık bir analizden önce bunların nasıl nitel olarak yorumlanacağını açıklıyoruz.
Giriş
Bir proteinin işlevi sadece üç boyutlu yapısı tarafından değil, aynı zamanda iç esnekliğini ve proteinin benimseyeceği farklı konformasyonlar arasındaki yapısal geçişleri kapsayan yapısal dinamikleri tarafından da belirlenir. Nükleer manyetik rezonans (NMR) spektroskopisi, çözelti 1,2,3'teki proteinlerin yapısal dinamiklerini araştırabilir. Proton ile tespit edilen katı hal NMR'deki son gelişmeler, örneğin bir lipid çift katmanlı zar 4,5,6 gibi daha az çözünür bir durumda protein dinamiklerinin karakterizasyonuna da izin verir. NMR çözeltisinde, protein omurgasının ve protein yan zincirlerinin yapısal dinamikleri incelenebilir. Küresel bir protein için, protein 15N izotopik olarak etiketlendikten sonra protein omurgasının yapısal dinamik çalışması 50 kDa'ya kadar elde edilebilir. Yan zincir döterasyonu ve enine gevşeme optimize edilmiş spektroskopi (TROSY) kullanıldığında, bu sınır 200 kDa'ya kadar uzatılabilir 7,8. Odak noktası yan zincir dinamikleri olduğunda, erişilebilir proteinlerin ve komplekslerin aralığı 1 MDa 2,9'a kadar genişletilebilir.
Belirtilen ağırlık sınırlamaları, sıklıkla yüksek içsel dinamikler gösteren içsel olarak düzensiz proteinler (IDP'ler) için geçerli değildir. Ökaryotik proteomun %30'undan fazlası IDP'leri veya içsel olarak düzensiz bölgeleri (IDR'ler) içerir10,11,12,13. Sinyal iletimi ve transkripsiyon1 gibi birçok hücresel süreçte merkezi rol oynarlar ve sıklıkla hücre içi faz ayrımında yer alırlar 14,15,16,17. IDP'ler, fizyolojik koşullar altında iyi tanımlanmış üç boyutlu (3B) bir doğal yapıdan yoksundur ve zayıf hunili veya engebeli bir enerji manzarasına sahiptir17,18. IDP'lerin veya IDR'lerin omurgası üzerine dağıtılan düşük hidrofobiklik ve güçlü elektrostatik itme nedeniyle, sert bir yapıya katlanmak için itici bir kuvvet eksiktir19. IDP'ler, diğer bağlayıcı ortaklarla karmaşık olduklarında sıklıkla katlanmış bir uyum benimserler 10,20,21. Ayrıca, çeviri sonrası değişiklikler (PTM'ler), IDP'lerin veya IDR'lerinkatlama olanaklarını genişletir 22,23. IDP'lerin yanlış katlanması, nörodejeneratif hastalıklar da dahil olmak üzere çeşitli hastalıkların bir nedeni olarak tanımlanmıştır 15,24,25,26.
IDP'ler ve IDR'ler yüksek iç esneklik göstermektedir 21,27,28. Atomik konumların ve dihedral açıların değişimini gösteren konformasyonel topluluklar, moleküler dinamik simülasyonlarından ve deneysel verilerden elde edilen kısıtlamalardantüretilmiştir 29,30,31,32. Donmuş haldeki dinamikler ve bunun sonucunda ortaya çıkan düzensizlik nedeniyle, dağınık elektron yoğunluğu, kriyo-EM veya X-ışını kristalografisi gibi yapısal biyolojideki en son yöntemler kullanılarak yapısal olarak karakterize edilmesini zorlaştırır. Ayrıca, kriyojenik sıcaklıklarda deneyler için kristalleşme koşulları veya numune hazırlama teknikleri, IDP'lerin deneyimlediği konformasyonel alanı etkileyebilir. Bununla birlikte, NMR çözeltisi oldukça dinamik proteinler için iyi çalışır ve bu nedenle IDP'ler 16,20,22,28,29,30,31,32,33,34,35,36,37,38'i araştırmak için çok uygundur.
Yukarıda tanıtıldığı gibi, NMR çözümü, esas olarak spin gevşemesi 31,33,38,39,40,41,42'ye dayanan geniş bir zaman ölçeği aralığında iç protein dinamiklerini incelemek için çeşitli teknikler sunar.
Protein omurgasının amid gruplarındaki 15N çekirdeğin spin gevşemesi, iç protein dinamikleri ve kolektif hareketler (ilgili olduğunda rotasyonel difüzyon dahil) nedeniyle 1 H-15N bağ açısı oryantasyon değişiklikleri tarafından indüklenir27,43,44,45,46,47,48,49,50,51. Dönme korelasyon süresinden τR'den daha kısa zaman ölçeklerinde (molekülün bir radyantı döndürmesi gereken süre, aynı zamanda genel yuvarlanma korelasyon süresi olarak da adlandırılır), kimyasal kayma anizotropisi (CSA) ve dipolar eşleşme (D) aktiftir ve proteinin dönme difüzyonu ile ortalaması alınmaz. Bağ açılarındaki değişiklikleri, bağların yeniden yönlendirilmesini ve rotasyonel yuvarlanmayı içeren protein omurgasının iç dinamikleri, CSA ve dipolar eşleşme tensörünün stokastik dalgalanmalarını indükleyerek yerel manyetik alanda bir varyasyona neden olur ve sonuçta NMR spin gevşemesine yol açar 47,48,52,53. Bu dalgalanmalar genel bir korelasyon fonksiyonu ile tanımlanabilir. Genel korelasyon fonksiyonunun Fourier dönüşümüne spektral yoğunluk fonksiyonu denir. Yarı klasik Redfield gevşeme teorisinde, NMR gevşeme hızı sabitleri, bu spektral yoğunluk fonksiyonlarının54 doğrusal kombinasyonları ile tanımlanabilir.
1990'ların başında geliştirilen omurga 15N NMR gevşeme deneyleri,45,55,56,57 proteininin dönme korelasyon süresinden τR'den daha hızlı, hızlı pikosaniye (ps) nanosaniye (ns) zaman ölçeğine duyarlı, 15N R1, R 1ρ ve {1H}-15N nükleer Overhauser etkisi deneylerini içerir. Dönme korelasyon süresinden τR'den daha yavaş omurga dinamiklerini karakterize etmek için, gevşeme dağılım deneyleri, R1ρ ve mikrosaniye (μs) - milisaniye (ms) dinamiklerine duyarlı Carr-Purcell-Meiboom-Gill (CPMG) deneylerikullanılır 44,46,58,59,60,61. Mikrosaniyelerden daha yavaş dinamikler, 15N kimyasal değişim doygunluk transferi (CEST) NMR62, değişim spektroskopisi (EXSY, milisaniyelerden saniyelere) veya Gerçek zamanlı (RT) NMR (saniyelerden saatlere)63,64 ile yakalanabilir. Paramanyetik probların yanı sıra artık dipolar kuplajların (RDC'ler) PRE (paramanyetik gevşeme geliştirme) etkisi, ps ila msdinamikleri 65,66,67,68 arasındaki tüm aralığı değerlendirmek için kullanılabilir.
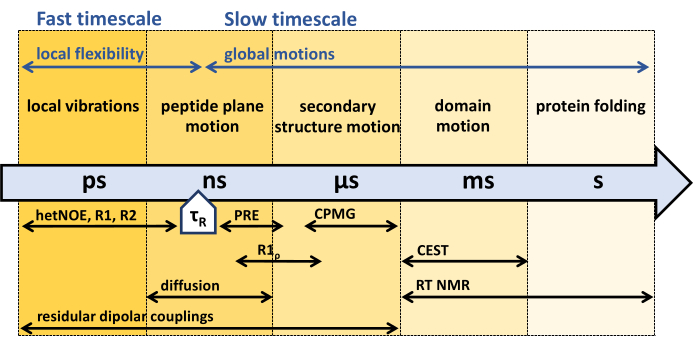
Şekil 1: Protein omurga dinamiğinin zaman ölçekleri ve farklı NMR dinamiği deneylerinin hassas zaman penceresi. NMR, çok çeşitli zaman ölçeklerinde protein omurga dinamiklerini karakterize etmek için çeşitli yöntemler sunar. Protein omurgası tarafından deneyimlenen farklı hareketler, kendi zaman ölçeklerinde gösterilir. Proteinin dönme korelasyon süresi, τR, proteinin genel bir dönüş için ihtiyaç duyduğu zamandır (bir radyant ile). Proteinin dönme korelasyon süresinden (τR) daha hızlı hareketler, proteinin iç esnekliği ile ilişkilendirilebilir. Çeşitli NMR deneyleri ve ilgili zaman ölçeklerine duyarlılıkları okun altında belirtilmiştir. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
Aşağıdaki protokol, duyarlılığı artırılmış bir heteronükleer tek kuantum tutarlılık (HSQC) algılama şeması kullanılarak Lakomek ve ark.69 ve Stief ve ark.70 tarafından NMR gevşeme deneylerinin kurulumunu açıklamaktadır. Deneysel uygulamaya geçmeden önce, NMR spin gevşemesi ve NMR gevşeme deneylerine çok kısa bir genel bakış verilmiştir. Boyut kısıtlamaları nedeniyle ve bu protokolü anlaşılır kılmak için bu genel bakış basit (ve dolayısıyla eksik) kalmalıdır.
T1 zamanı veya R1 = 1 / T1 hız sabiti ile karakterize edilen uzunlamasına veya spin-kafes gevşemesi, manyetizasyonun Boltzmann dengesine geri dönüşünü tanımlar. Dengede, manyetizasyon, laboratuvar çerçevesinin z eksenini tanımlayan dış manyetik alanın ekseni boyunca hizalanır. Yüksek (1H) ve küçük (15N) Larmor frekanslarındaki spektral yoğunluklar (NMR rezonans frekansları, örneğin, 14.1 Tesla mıknatıs için 1 H için 600MHz) ve bu Larmor frekanslarının doğrusal kombinasyonları, rad·s-1'de ölçülen 15NR 1 hız sabitleri ile karakterize edilen 15N R1 gevşemesine katkıda bulunur. Zaman ölçeklerindeki hareketler bu Larmor frekanslarının tersidir; bu nedenle, pikosaniye ila nanosaniye zaman ölçeğindeki hareketler, gevşeme hızı sabitiR1'e katkıda bulunur. Genel yuvarlanma gösteren ve bir dönme korelasyon süresinin tanımlanabildiği moleküller için, R1 (T1) eğrisi, ωτR = 1 için bir maksimum (minimum) gösterir, dönme korelasyonu τR ve Larmor frekansı ω dikkate alınan spinin. Birden fazla Larmor frekansı katkıda bulunursa, en düşük frekansa sahip olan baskın olandır, örneğin, 15NR 1 durumunda ω N. Hızlı hareket rejimi (ωτR, 1'den çok daha küçük), çok hızlı yuvarlanan küçük moleküller ve düşük manyetik alanlar ve düşük viskozite için geçerlidir. Yavaş hareket rejimi (ωτR, 1'den çok daha büyük), daha yavaş yuvarlanan daha büyük moleküller ve yüksek manyetik alanlar ve yüksek viskozite için geçerlidir.
Küresel katlanmış proteinler, çözeltide genel yuvarlanma gösterir ve bir rotasyonel korelasyon süresi atanabilir. Bununla birlikte, genel yuvarlanma kavramı artık içsel olarak düzensiz proteinler için geçerli değildir ve sıklıkla tek bir rotasyonel korelasyon süresi atamaktan farklıdır. Burada, kalıntıya özgü iç korelasyon süresi daha kritik hale gelir.
15N R1 gevşeme oranlarını ölçen tarif edilen darbe dizisi (Şekil 2), dörtlü algılama69,70,71 için bir Yankı / Anti-Yankı algılamalı duyarlılıkla geliştirilmiş bir HSQC okuma deneyine dayanmaktadır. Tutarlılık seçimi ve geliştirilmiş su bastırma70 için değişken mukavemet ve uzunluğa sahip kısa gradyanlar kullanılır. Bu süre zarfında, 15N uzunlamasına polarizasyon gevşeyecektir. Daha uzun bozunma süreleri, bu sözde 3B spektrumun ilişkili 2B düzlemlerinde azalmış yoğunluklara yol açar (gecikme veri noktaları üçüncü boyutta kaydedilir). Aşağıda açıklanan bir döngü elemanı, daha uzun gevşeme süreleri için artan sayıda yürütülür. 15N kimyasal kayma anizotropisi (CSA) ile 1H ve 15N dipolar kuplaj (D) arasındaki çapraz ilişkili gevşeme de gevşeme gecikmesi sırasında aktif olduğundan, merkezi bir I-BURP-2 180 ° darbe72, amid protonları üzerinde seçici, çapraz ilişkili gevşeme ile katkıyı yeniden odaklamak için gereklidir (yeniden odaklanmazsa, çarpık ve hatalı 15N R1 oran sabitlerine yol açacaktır).
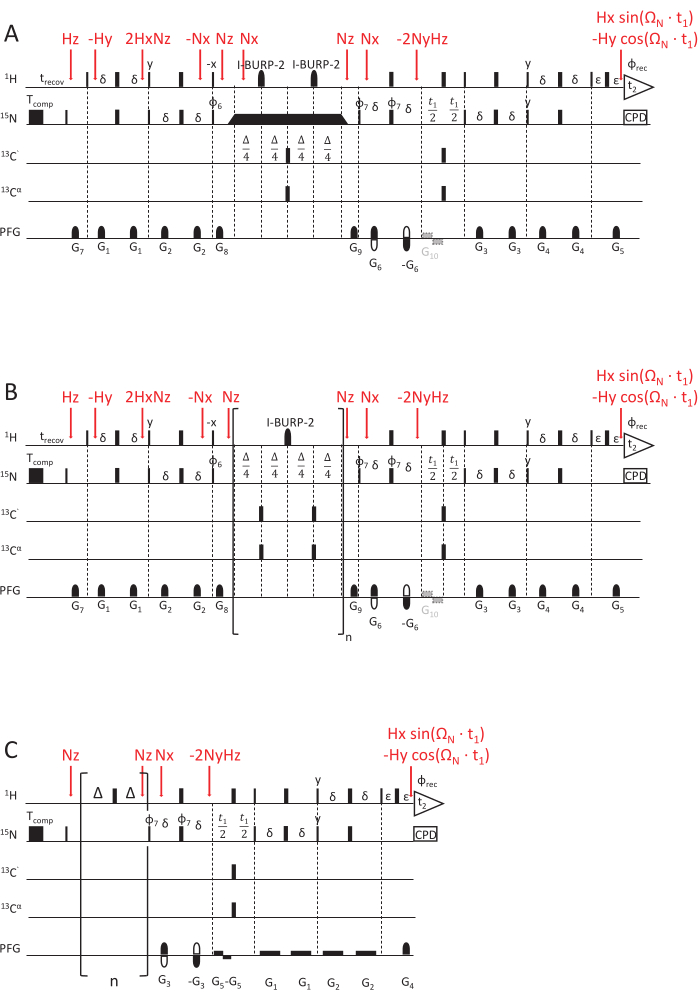
Şekil 2: NMR gevşeme hızı sabitlerini belirlemek için NMR darbe dizisi şemaları. (A) 15N R1ρ, (B) 15N R1 ve (C) hetNOE deneyi, duyarlılığı artırılmış bir HSQC okuma şeması69,70 kullanılarak. Aksi belirtilmedikçe, 90°(x) darbeler dar dikdörtgenlerle ve 180°(x) darbeler geniş dikdörtgenlerle görselleştirilir. Aşağıdaki faz döngüsü uygulanır: φ6 = y, y, -y, -y; φ7 = Y, -Y, φRec = Y, -Y, -Y, Y. Dörtlü algılama, gradyan G5'in polaritesinin ve φ7'nin faz döngüsünün (Yankı / Anti-Yankı algılama) ters çevrilmesiyle elde edilir. (A) 15N R1ρ deneyi: Siyah dikdörtgen, süresi farklı gevşeme gecikmeleri elde etmek için değişen spin-lock'u temsil eder. Döndürme kilidinden önceki ve sonraki üçgenler, manyetizasyonu etkili manyetik alan ekseni Beff boyunca hizalayan adyabatik şekil darbelerini gösterir. G10, evrim aşaması sırasında su manyetizasyonunun radyasyon sönümlemesini önlemek için isteğe bağlı bir gradyandır. (B) 15N R1 deneyi: Parantezli kısım, istenen gevşeme gecikmesine uyması için n sayıda tekrarlanan dizinin döngü elemanını gösterir. (C) hetNOE darbe şeması, R1 ve R1ρ darbe şemalarının ikinci yarısına, yani t1 evrim süresine ve HSQC algılama elemanına benzer. Bununla birlikte, 15N manyetizasyon herhangi bir BECERIKSIZ olmadan doğrudan uyarılır. Proton manyetizasyonunun doygunluğu (1H ile 15N arasında çapraz gevşeme elde etmek için), en az 5 s boyunca uygulanan 180 (1H) darbelik bir tren ile elde edilir. Referans deneyi için herhangi bir darbe dizisi olmadan aynı uzunluklarda (burada 5 s) bir rölanti gecikmesi uygulanır. G5, radyasyon sönümlemesini önlemek için isteğe bağlı bir gradyandır ve φ7 = y, -y, -y, y fazı ile birlikte G4 gradyanının polaritesindeki ters çevirme, kareleme algılaması sağlar. Ürün operatörleri tarafından temsil edilen mıknatıslanma transfer adımları kırmızı ile işaretlenmiştir. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
Gevşeme hızı sabiti R2, spin arasındaki faz tutarlılığının kaybı nedeniyle enine polarizasyonun (dış manyetik alana ortogonal xy düzleminde) gevşemesini tanımlar ve bu da tespit edilebilir manyetizasyonunbozulmasına yol açar 53,54. Yüksek ve küçük frekanslardaki spektral yoğunluk fonksiyonu, R1'e benzer şekildeR2'ye katkıdabulunur. Bununla birlikte,R2'ye en büyük katkı, sıfır frekanstaki spektral yoğunluktan kaynaklanmaktadır. Bu nedenle,R2, oda sıcaklığında küçük bir küresel protein için birkaç ns mertebesinde olan dönme korelasyon süresi τR ile tanımlanan dönme yuvarlanmasına karşı çok hassastır. Bu nedenle, yüzlerce ps'den düşük ns'ye kadar olan rejimdeki daha yavaş omurga hareketleri en fazla katkıda bulunur. 15N çekirdeğin kimyasal kayma tensörünün izotropik kısmının modülasyonuna neden olan omurganın değişim dinamikleri, prensip olarak, R2 oran sabitleri 43,44,49,60,61'e bir değişim katkısı R 2ex ekler. Açıklanan deneylerde, R2ex katkısı, spin-lock'un ters dairesel frekansından daha yavaş dinamikleri yeniden odaklayan bir spin-lock tarafından bastırılır. Döndürme kilidi, manyetizasyonu etkili manyetik alan ekseni Beff boyunca hizalanmış tutan uzun sürekli dalgalı bir radyo frekansı darbesidir (döndürme kilidi ω1 alanının vektör toplamı ve 15N taşıyıcı frekansından kimyasal kayma ofseti (aşağıya bakınız)). B1,eff ekseni boyunca hizalanan manyetizasyonun gevşemesine, bir R1 bileşeni ve bir R 2 bileşeni olan R1ρ gevşemesi denir. Formül (1) kullanılarak, R2, R1ρ ve R1 44,73'ten hesaplanabilir:
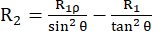 (1).
(1).
Etkin manyetik alan Beff'in ekseni ile dış manyetik alan B0 arasındaki açı . 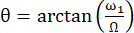 ω1, döndürme kilidinin RF genliğidir ve karşılık gelen kalıntının 15N kimyasal kayması ile 15N taşıyıcı frekansı 44,73 arasındaki kimyasal kayma ofsetini Ω.
ω1, döndürme kilidinin RF genliğidir ve karşılık gelen kalıntının 15N kimyasal kayması ile 15N taşıyıcı frekansı 44,73 arasındaki kimyasal kayma ofsetini Ω.
R1ρ darbe şeması (Şekil 2A, 70), gevşeme gecikmesi dışında 15NR 1 şemasına çok benzer. 15N R1ρ gevşeme oranlarını ölçmek için, manyetizasyon, döndürme kilidi ile aynı radyo frekansı (RF) genliğine sahip adyabatik bir darbe ile etkili alan ekseni Beff boyunca hizalandıktan sonra döndürme kilidi aktif olmalıdır. Döndürme kilidinin uzunluğu, farklı gevşeme gecikmelerini elde etmek için değiştirilecektir.
Aşağıda hetNOE olarak adlandırılan kararlı durum {1H}-15N nükleer Overhauser etkisi (1 H-15N NOE), çapraz gevşeme hızı ile 15N uzunlamasına gevşeme oranının oranıdır. Proton polarizasyonu 45,53,54,74,75'in doygunluğu üzerine proton ile çapraz gevşeme nedeniyle 15N'de kararlı durum polarizasyonunun azalmasına yol açar. Çapraz gevşeme, 1 H ve 15N Larmor frekanslarının toplamı ve farkının spektral yoğunluk fonksiyonlarına bağlıdır. Bu nedenle, hetNOE hem hızlı pikosaniye dinamiklerine (< 100 ps) hem de ps-ns dinamiklerine (R1 bağımlılığı nedeniyle) duyarlıdır. Dizi69 (Şekil 2C), karesel algılama için Yankı / Anti-Yankı gradyanları ile duyarlılığı artırılmış bir HSQC okumasına dayanmaktadır. Proton manyetizasyonunun ve ortaya çıkan hetNOE'nin doygunluğu için, denge proton manyetizasyonu ters çevrilir ve ardından 15 N T1'in yaklaşık 5 katı boyunca hızla titreşen 180° darbelerle doyurulur. Referans deneyi için, geri kazanım gecikmesi doygunluk gecikmesine eşittir, ancak 1H 180 ° darbe treni yoktur. Referans deney ve 1H doygunluğa sahip deney için D1 = 2 s'lik ekstra bir gecikme eklenir. Her iki deney de arka arkaya kaydedilir ve yalnızca 1sa 180 ° darbe (doygunluk) uygulanıp uygulanmadığında (referans) farklılık gösterir. 1H doygunluk ile deneyde kaydedilen spektral yoğunlukların oranı, referans deneyin yoğunluklarına bölünür (180° proton darbe treni olmadan) {1H}-15N NOE (hetNOE) değerini verir.
Aşağıdaki protokol, Lakomek ve ark.69 ve Stief ve ark.70 tarafından NMR gevşeme deneylerinin kurulumunu açıklamaktadır. Hassasiyeti artırılmış bir HSQC algılama şeması kullanarak NMR darbe dizilerine odaklanıyoruz. 15NR 1 ve R1ρ deneyleri, Stief ve ark.70 tarafından ayrıntılı olarak açıklandığı gibi uygulanmıştır ve hetNOE deneyi, Lakomek ve ark.69 tarafından tanımlanmıştır.
Protokol
1. NMR numune hazırlama
NOT: Proteinlerin izotop etiketlemesi, daha yüksek boyutlu NMR ve gelişmiş NMR deneyleri için yapılır. E. coli'de protein ekspresyonu ve protein saflaştırması, litre başına birkaç miligram verimle zengin ortam (örneğin, Luria-Bertani [LB] veya 2x maya özütü tripton ortamı [2YT]) kullanılarak kurulduğunda, izotopik olarak etiketlenmiş bir NMR numunesi hazırlamak genellikle nispeten basittir.
- İzotop etiketleme için, 15N amonyum klorürün tek nitrojen kaynağı ve 13C ile zenginleştirilmiş glikozun tek karbon kaynağı olduğu gen ekspresyonu için M9 minimal ortam kullanın.
- Protein saflaştırması için, ilgili protein için laboratuvarda oluşturulan standart protokolü izleyin.
NOT: Sonuçlar bölümü, bir IDP, monomerik SNARE proteini Synaptobrevin-2 (1-96) için temsili sonuçları göstermektedir. Synaptobrevin-2'nin NMR izotop etiketlemesi,70 yılında tarif edildiği gibi gerçekleştirilmiştir. Çok yüksek hassasiyetli ölçümler için, %5'ten daha düşük D2O konsantrasyonlarının kullanılması önerilir. Bunun nedeni, Hahn-Echo 15N T2'de ve düşük frekanslı 15N CPMG gevşeme dispersiyon deneylerinde (burada tartışılmamıştır) gözlemlenebilen, ancak 15N R1ρ deneylerinde (esas olarak yeniden odaklandığı) yalnızca minimum bir etkiye sahip olan döteryum izotop etkisidir76.
2. Spektrometre üzerinde NMR gevşeme deneylerini çalıştırmak için hazırlıklar
NOT: Açıklanan NMR gevşeme deneyleri Bruker spektrometreleri için özeldir. Kriyojenik ve oda sıcaklığında 1H, 15N ve 13C üçlü rezonans probları ve Bruker yazılımı Topsin 3.6 veya üstü tarafından çalıştırılan Avance III ve Avance Neo konsolları üzerinde test edilmiştir.
- Önlemleri takip edin.
- NMR spektrometresinde çalışmaya başlamadan önce bir güvenlik brifingi yapın.
- Kullanılan spektrometrenin probun güç özelliklerini kontrol edin ve her darbe için uygun bir darbe uzunluğu ve darbe gücü seçin, örneğin Topspin "prosol" tablo girişleriyle tutarlılığı karşılaştırarak.
- Darbe dizilerini içe aktarın.
- NMR darbe programlarını aşağıda belirtilen web sitesinden veya genişletilmiş Biyolojik Manyetik Rezonans Bankası (BMRB) deposundan (bmrbig102) NMR spektrometresini çalıştıran bilgisayarın /opt/topspin4.1.4/exp/stan/nmr/lists/pp/user dizinine indirin. Lütfen topspin versiyonunu ayarlayın (burada: topspin4.1.4 spektrometrede kurulu olan ilgili versiyona).
- NMR şeklindeki darbeleri aşağıda belirtilen web sitesinden veya genişletilmiş Biyolojik Manyetik Rezonans Bankası (BMRB) deposundan (bmrbig102) NMR spektrometresini çalıştıran bilgisayarın /opt/topspin4.1.4/exp/stan/nmr/lists/pp/user dizinine indirin. Lütfen topspin sürümünü ayarlayın (burada: topspin 4.1.4, spektrometrede kurulu olan ilgili sürüme).
- Spektrometreyi ve numuneyi kalibre edin.
- İlgilenilen sıcaklığı ayarlayın. Sıcaklık kalibrasyonu için tesisin protokollerini uygulayın. Örneğin, bir Metanol-d4 numunesi kullanın ve NMR termometre (https://chem.ch.huji.ac.il/nmr/software/thermometer.html) web uygulamasını kullanarak gözlemlenen kimyasal kayma farkını yorumlayın; Ayrıca bk.77.
- Numuneyi yerleştirin (artı kilit için ek bir %5 D2O) ve numunenin sıcaklığını dengelemesi için yaklaşık 10 dakika bekleyin. Döteryum (2H) sinyalini kilitleyin (komut: kilitle, H2O + D2O öğesini seçin).
- 1H, 15N ve 13C kanallarını eşleştirin ve ayarlayın, örneğin atma veya atmm rutinini kullanarak. Otomatik eşleştirme ve ayarlama ünitesi takılı değilse, wobb komutunu kullanarak probu manuel olarak eşleştirin ve ayarlayın.
- Homojen bir B0 alanı için şimleme yapın, örneğin topshim rutin tg'yi kullanarak.
- 90° (1H) darbe uzunluğunu kalibre edin.
- zg deneyini kullanın. Prob parametrelerini içe aktarmak için getprosol kullanın. P1 için 5 μs'lik kısa bir süre seçin. Gerekirse PLW1 için probun belirtilen güç seviyesini kontrol edin ve düzeltin.
- Deneyi zg ile başlatın. Verileri efp ile işleyin. ile fazı ayarlayın.ph, taban çizgisinin eşit olması ve su rezonansının NMR sinyalinin aynı fazda olması için.
- Ortak veya yaklaşık 90° (1H) darbe alarak (örneğin, listelenen spektrometre özelliklerinden) 360° (1H) darbeyi tahmin edin ve bunu dört faktörle çarpın. Denemeyi çalıştırın ve işleyin.
NOT: Seçilen süre, sinyal pozitifse çok uzun, negatifse çok kısadır. 360 ° (1H) darbe sıfır geçiş olacaktır. - Darbe uzunluğunu buna göre ayarlayın. Sıfır geçişini bulmak için bunu yinelemeli olarak yapın. Elde edilen değeri dörde bölün. Bu, 90° (1H) darbe olacaktır.
NOT: Açıklanan 360° yöntemine bir alternatif, komut satırından başlatılabilen ve otomatik 1H darbe kalibrasyonuna izin veren darbe rutinidir.
- Taşıyıcı frekansı O1'i su rezonansına göre kalibre edin.
- Komut satırına edc yazarak zg deneyini yeni bir deney numarasına kopyalayın. Parametre kümelerini açmak için rpar yazın (Ek Şekil 1A).
- ZGPR parametre setini arayın. ZGPR parametre setine tıklayın (Ek Şekil 1A), Oku'ya tıklayın, "getprosol" Çalıştır'ı seçin ve Tamam'a tıklayın (Ek Şekil 1B).
- P1'i kalibre edilmiş 90° (1H) darbe uzunluğuna değiştirin. D1 = 2 s'lik bir doygunluk uzunluğu ve 50 dB'lik bir PLW9 darbe gücü seçin. Alıcı kazancını (rg) 16'ya ayarlayın. Alternatif olarak, rga yazarak ve alıcı kazancını belirtilen değere ayarlayarak alıcı kazancı ayarını gerçekleştirin (Ancak "alıcı kazancı taşması"ndan kaçının).
- Yinelenen, sürekli bir alımı başlatmak için komut satırına gs yazın. Edinme penceresine gidin ve su sinyalinin hakim olduğu FID alanını azaltmak için O1'i değiştirin (Ek Şekil 2).
- O1'i azaltmak için işaretçinin altına tıklayın ve artırmak için işaretçinin üstüne tıklayın. Adım boyutunu değiştirmek için duyarlılık düğmesini kullanın. Mevcut O1'i kaydedin ; Close, tekrarlanan alımı durdurur (Ek Şekil 2).
- 90° (15N) darbe uzunluğunu kalibre edin.
- zg deneyini yeni bir deney numarasına kopyalayın ( edc yazın). Parametre setini okumak için rpar yazın HSQCFPF3GPPHWG darbe programını 1 H-15N HSQC deneyine değiştirmek için. P1, O1 ve PLW1 parametrelerini koru seçeneğini kullanın (Ek Şekil 1).
- Prob parametrelerini içe aktarmak için komut satırına getprosol 1H <μs cinsinden kalibre edilmiş 90° (1H) darbe uzunluğu> < db cinsinden darbe gücü> yazın. Tarama sayısını (NS) 8 ve sahte tarama sayısını (DS) 2 olarak ayarlayın.
NOT: < > burada ayarlanacak değerleri belirtir. Örneğin: getprosol 1H 8.5 -11.79. - Komut satırına zg yazarak denemenin ilk artışını çalıştırın. İlk artışı rser 1 ile yeni bir deneme numarasına kopyalayın. Verileri efp ile işleyin ve spektrumu ile aşamalandırın .ph. Aşağıdaki adımlar için denemenin ilk artışını (1B deneme) kullanın.
NOT: < >, yeni deney için seçilen numarayı belirtir, örneğin, rser 1 101. - 1D deneyin darbe dizisi parametrelerini açmak için komut satırına ased yazın. Nabız programı düzenleyicisini açmak için PULPROG satırındaki E'ye tıklayın (Ek Şekil 3A'ya bakın).
- Darbe dizisini yeni bir adla kaydetmek için aşağıdaki iş akışını kullanın: Dosya'yı tıklatın (sol üst köşe), Farklı kaydet'i tıklatın, yeni adı girin ve Tamam'ı tıklatın. Ardından PULPROG'u veri kümesine ayarla'ya tıklayın (bkz. Ek Şekil 3B) ve Tamam'a tıklayın.
- Orijinal 1 H-15N HSQC dizisini (yeni bir adla kaydedildi, bkz. 2.6.4.), t1 evrim periyodundan sonra (geri transferden önce) 90 ° (15N) 'yi 2 faktörü ile çarparak değiştirin (90 ° (15N) darbe şimdi 180 ° (15N)). Darbe sırasını kaydetmek için Ctrl S tuşuna basın. Darbe uzunluğunu 90° (15N) (P21) olarak ayarlayın, böylece ilk artışın NMR sinyali kaybolacaktır (sıfır geçişinde) (Ek Şekil 3B).
- 1 H-15N HSQC deneyini (2D deney) açın ve kalibre edilmiş 90° (15N) darbe uzunluğunu (P21) girin. Komut satırına getprosol 1H 1H) darbe uzunluğu> < db cinsinden 90° (1H) darbe gücü> 15N 15N) darbe uzunluğu> <90° (15N) darbe gücü> yazın.
- Spektral genişlikleri ve edinme sürelerinin ödeneğini belirleyin.
- 1 H-15N HSQC deneyi için edinme parametresi penceresinde (komut satırına eda yazın), spektral genişliği (edinme parametresi SW) ve doğrudan ve dolaylı boyutlardaki boyut artışlarının sayısını (edinme parametresi TD) ayarlayın.
- SW, doğrudan (1H) boyutta 16 ppm ve dolaylı (15N) boyutta 30 ppm için seçin. TD için seçin, doğrudan (1H) için 2048 ve dolaylı (15 N) boyutta 512.
- 1 H-15N HSQC deneyini edinin.
- İlgilenilen tüm sinyallerin algılandığını ve spektrumda örtüşme artefaktı bulunmadığını kontrol edin. Spektrumun merkezi, 15N ekseni boyunca protein sinyal dağılımının merkezine uymuyorsa, O3P'yi değiştirin.
- Spektral pencere çok küçükse SW'yi artırın. Daha yüksek bir FID çözünürlüğü elde etmek için TD'yi artırın (Maksimum ayırma süresi için prob özelliklerini kontrol edin).
NOT: Bu, NMR gevşeme deneylerinin kurulumu için genel kurulum ve başlangıç noktasıdır. 1 H-15N HSQC deneyinin tüm deney setinin başında ve sonunda ve daha uzun deney süreleri için numune bütünlüğünü izlemek için arada kaydedilmesi önerilir.
- 1 H-15N HSQC deneyi için edinme parametresi penceresinde (komut satırına eda yazın), spektral genişliği (edinme parametresi SW) ve doğrudan ve dolaylı boyutlardaki boyut artışlarının sayısını (edinme parametresi TD) ayarlayın.
3. NMR gevşeme deneylerinin uygulanması
NOT: NMR gevşeme darbe dizileri (Şekil 2) https://www.ipb.hhu.de/en/teams/team-lakomek/pulsesequences'da veya genişletilmiş Biyolojik Manyetik Rezonans Bankası (BMRB) deposunda (bmrbig102) mevcuttur.
- Ayarlayın 15N R1ρ deney.
- HSQC deneyini ( edc yazın) yeni bir dizine kopyalayın. ased yazın ve darbe dizisi parametrelerindeki PULPROG parametre satırının üç noktasına tıklayarak darbe programını 15N R1ρ deneyine değiştirin (ased, bkz . Ek Şekil 3A). Nabız programına çift tıklayın, PULPROG'u veri setine ayarla'ya tıklayın (Ek Şekil 3B'ye bakın) ve Tamam'a tıklayın.
NOT: Bazı hata mesajları görünebilir. Bu iletileri kapatın. - Darbe dizisinde açıklandığı gibi tüm eksik gradyan şekilleri (gpnam1- gpnam9) eklemek için komut satırına gpnam yazın (Ek Şekil 4).
- Komut satırına vplist yazın. Üç noktaya (Ek Şekil 5A), Dosya (sol üst köşe) ve Yeni'ye (Ek Şekil 5B) tıklayın.
- Yeni bir kukla vplist için bir ad yazın ve Tamam'a basın. Sahte vplist'i oluşturun (5m, 65m, 25m, 45m, 15m, 55m, 35m, 10m; her vplist girişi için yeni bir satır). Listeyi kaydetmek ve vplist penceresini kapatmak için Ctrl S tuşuna basın (Ek Şekil 5C).
- Yeni oluşturulan vplist'in adına çift tıklayın (Ek Şekil 5B).
NOT: Bu, darbe dizisindeki derleme hatalarını önlemek için gereklidir. Vplist'in uygun girişleri aşağıda daha ayrıntılı olarak belirlenecektir (madde 3.1.29.). - Darbe dizisi parametrelerini açmak için ased yazın (Ek Şekil 6). Darbe dizisinde verildiği gibi tüm eksik gradyan kuvvetlerini ve gradyan uzunluklarını girin (Ek Şekil 6D).
- Döngü sayacı L3 (dolaylı boyuttaki karmaşık noktaların sayısı) için, edinim parametresi penceresindeki (eda) 15N TD girişinin 1/2'sini girin. Döngü sayacı L6 için, gevşeme eğrisini uydurmak için kullanılan kaydedilen gevşeme veri noktalarının sayısını girin.
NOT: Bu L6 sayısı, vplist girişlerinin sayısına karşılık gelir, burada: 8 (Ek Şekil 6A). - Yukarıda belirtilen web sitesinden indirilen adyabatik TanhTan_half.nl darbesini 15N SP8 darbesinin şekli olarak ayarlayın. Web sitesinden adyabatik TanhTan_2nd.nl darbesini 15N SP9 darbesinin şekli olarak ayarlayın (Ek Şekil 6C).
- P8 girişini adyabatik TanhTan nabzının uzunluğuna ayarlayın - nabzın adyabatikliğini garanti etmek için yeterince uzun; burada P8 = 3000 μs seçilir (Ek Şekil 6C).
- 15N ayırmayı (CPDPRG 3) bir garp ayırma şemasına ayarlayın. PCPD3 200 μs dekuplaj uzunluğu için kullanın (Ek Şekil 6C).
- Taramalar arası kurtarma gecikmesini en az D1 = 2 s veya daha uzun bir süre olarak ayarlayın.
NOT: Uygun bir kurtarma gecikmesi seçimi için, Tartışma bölümüne bakın. - Son deney için sahte taramaları (DS) en az 64'e ayarlayın (kurulum sırasında, zaman nedenleriyle DS = 4 yeterlidir). Başlangıç noktası olarak, 4'lük bir dizi tarama (NS) kullanın; sinyal-gürültü (SNR) çok düşükse 4'ün katlarını kullanın (Ek Şekil 6A).
- O1'i adım 2.5'in kalibre edilmiş taşıyıcı frekansına ayarlayın. O2P'yi 176 ppm'ye ayarlayın ve O3P'yi 1 H-15N HSQC deneyinden kopyalayın (Ek Şekil 6A-C).
- Darbe uzunluğunu P7'yi adım 90'da kalibre edilen 15° (2.6 N) darbe uzunluğuna ayarlayın. Ayrıca, 90° (15N) darbenin darbe gücü seviyesini adım 2.6'dan PLW3 ve PLW7'ye kopyalayın (Ek Şekil 6C).
- P1 ve P19 darbe uzunluğunu adım 90'te hesaplanan 1° (2.4H) darbe uzunluğuna ayarlayın. (Ek Şekil 6B).
- Dolaylı boyuttaki artış sayısını ayarlayın TD = l3 * 2 * l6 (eda veya komut satırına td yazın).
NOT: Burada açıklanan tüm darbe dizisi parametreleri, darbe dizisinde ek olarak açıklanmıştır. - Şekil darbesi SP5'i bir I-BURP2 şekline (Iburp2.1000) ayarlayın. Darbe uzunluğunu P15'i 2000 μs'ye ayarlayın. Topspin darbe dizisi parametre penceresinde (ased) I-BURP2 şekil darbesinin yanındaki E'ye tıklayarak şekil aracı ekranını açın (Ek Şekil 6B).
- NMR Simülasyonunu Başlat düğmesine tıklayarak şekil darbesini doğru uzunlukta simüle edin. Simülasyon penceresinde şekillendirilmiş darbe uzunluğunu [μs] (= 2000 μs) ve dönüş açısını [°] (= 180°) kontrol edin. NMR-SIM'i Başlat'a tıklayın (Ek Şekil 7A).
- Uyarma aralığını kontrol edin ve proteinin 2H spektral dağılımını kapsayacak şekilde uygun I-BURP1 darbe uzunluğunu seçin, ancak aynı anda suyun uyarılmasını önleyin. P15'i, simüle edilmiş en iyi IBURP-2 darbesi ile simülasyon penceresinin Şekilli darbe uzunluğuna ayarlayın (Ek Şekil 7B).
NOT: I-BURP2 tüm amid bölgesini uyarıyorsa (%95 etkinliğe >) uygun uzunluk seçilir. Farklı bir uzunlukta bir I-BURP2 darbesini simüle etmek için, uyarma profili penceresini kapatın ve simülasyon penceresinde şekillendirilmiş darbe uzunluğunu düzenleyin. NMR-SIM'i Başlat'a tıklayın. Daha kısa uzunluktaki darbeler, Hz cinsinden daha geniş bir uyarma aralığına sahiptir. - I-BURP2 darbesinin taşıyıcı frekansını değiştirmek için SPOFFS5 ayarlayın ve I-BURP2 darbesinin su manyetizasyonuna dokunmasını ve bozulmasını önlemek için uyarma aralığını sola veya sağa kaydırın (Ek Şekil 6B).
- Şekillendirilmiş darbenin uygun güç seviyesini belirlemek için Bruker şekil aracını açın, NMR Simülasyonunu Başlat düğmesine tıklayın ve I-BURP2 darbe uzunluğunu Şekillendirilmiş darbe uzunluğu [μs] olarak ayarlayın. Simülasyon penceresinde karşılık gelen 90° kare darbede [μs] görülebilen yumuşak dikdörtgen 90° (1H) darbenin karşılık gelen uzunluğunu hatırlayın (Ek Şekil 7A).
- calcpowlev yazın. Referans uzunluğu olarak mikrosaniye cinsinden sert 90° (1H) darbeyi (P1) kullanın (Ek Şekil 8B) ve yeni uzunluk olarak mikrosaniye cinsinden I-BURP90 darbesine karşılık gelen 1 ° (2H) yumuşak dikdörtgen darbeyi kullanın (Ek Şekil 8A). Sert 90° (1H) darbe ile yumuşak dikdörtgen darbe arasındaki dB cinsinden Δ güç farkını unutmayın (Ek Şekil 8C). Sert 90° (1H) darbenin dB cinsinden darbe gücünü SPW5'e kopyalayın ve hafızaya alınan farkı Δ dB cinsinden ekleyin (Ek Şekil 6B).
- Döndürme kilidinin güç seviyesini belirlemek için, döndürme kilidinin karşılık gelen 90° (15N) darbe uzunluğunu hesaplayın. Şu formülü kullanın: 90° (15N) darbe uzunluğu s = 0.25 / (Hz cinsinden spin-lock gücü).
- Döndürme kilidinin gücü ile 90° (15N) sert darbe (P7) arasındaki dB cinsinden güç farkını Δ hesaplamak için calcpowlev'i kullanın. Yeni: μs cinsinden döndürme kilidinin 90° (15N) darbe uzunluğu (Ek Şekil 8A). Ref: μs cinsinden sert 90° (15N) darbe uzunluğu (Ek Şekil 8B). dB cinsinden 7° (90 N) sert darbenin PLW15güç seviyesini spin-lock güç seviyesi PLW8'e kopyalayın (Ek Şekil 6C) ve hesaplanan güç farkını Δ dB cinsinden ekleyin (Ek Şekil 8C).
- Döndürmeli kilit PLW8'in güç seviyesini SPW8 ve SPW9 güç seviyelerine kopyalayın. (Ek Şekil 6C).
- 15N'luk ayırmanın güç seviyesini belirlemek için, 90° (15N) ayırma darbesinin (PCPD3) ve 90° (15N) sert darbenin (P7) gücü arasındaki güç farkını Δ hesaplamak için calcpowlev'i kullanın. Yeni: μs (200) cinsinden PCPD3 (Ek Şekil 8A). Ref: μs cinsinden sert 90° (15N) darbe uzunluğu (Ek Şekil 8B). dB cinsinden 7° (90N) sert darbenin güç seviyesi PLW15'yi PLW31 (Ek Şekil 6C) ve dB cinsinden Δ ekleyin (Ek Şekil 8C).
- Sıcaklık telafisini kullanmak için #define TEMP_COMPENSATION ekleyin (darbe dizisindeki noktalı virgülü silerek). P18'i 15N R1ρ deneyinde kullanılan döndürme kilidinin maksimum süresine ayarlayın.
NOT: Deneyin ilk denemesi için sıcaklık telafisinin kapatılması önerilir. - 15N 13C etiketli numuneler söz konusu olduğunda, darbe programındaki LABEL_CN tanımlamak için # ekleyin (darbe dizisindeki noktalı virgülü silerek). P4'ü78'deki formülle hesaplanan darbe uzunluğuna ayarlayın. Örneğin, 600 MHz'de 23.70 μs'ye ve 1200 MHz'de 11.85 μs'ye karşılık gelir. P4 ile (13C) sert darbe arasındaki dB cinsinden güç farkını hesaplamak için calcpowlev yazın. dB cinsinden (13C) sabit darbe gücünü PLW4'e kopyalayın ve Δ'yi dB cinsinden ekleyin. PLW4'ü PLW2'ye kopyalayın (Ek Şekil 6B).
- Uygun örnekleme için gevşeme gecikmelerini belirleyin.
- İlk sekiz FID'yi çalıştırın (L6 = 8 durumunda) ve bunları işleyin (rser < yeni deney numarası>). Vplist'te, en uzun gecikmeli deneyin tepe yoğunluğunun en az 1/e'ye (yaklaşık %37) düştüğü, ancak en kısa gecikmeli deneye kıyasla (referans görevi görür) %25'ten az olmadığı girişleri seçin.
- En uzun gecikmeli FID ile en kısa gecikmeli FID arasındaki yoğunluk düşüşlerinin eşit bir örneklemesini elde etmek için vplist'in kalan girişlerini ayarlayın. NOT'u ve probun güç özelliklerini okuyun.
NOT: Yavaş yavaş gevşeyen kalıntılar ilk FID'ye hakim olacaktır. Birinci ve ikinci FID arasındaki yoğunluk düşüşlerinin eşit bir örneklemesini elde etmek için vplist'in kalan girişlerini ayarlayın. Probun izin verilen maksimum döndürme kilidi gücünü ve uzunluğunu aşmayın (prob kılavuzuna ve prob özelliklerine bakın). Sıcaklık kompanzasyonu durumunda, P18'in toplam spin-lock süresi uygulanacağından, gevşeme gecikmesi ve sıcaklık kompanzasyonu arasında bölünerek P18'i (maksimum sıkma kilidi süresi) kontrol edin. Zaman (gevşeme gecikmesi) + zaman (sıcaklık telafisi) = const = P18'i tutar. 2 kHz'lik bir döndürme kilidi için, 2 s'lik bir kurtarma gecikmesi (D1) kullanarak 65 ms'den fazla bir döndürme kilidi süresini aşmamayı tercih ediyoruz. Bu durumda, maksimum spin-lock süresi ile en uzun gevşeme gecikmesi arasında, izin verilen maksimum spin-lock süresi lehine bir uzlaşma sağlanmalıdır. Daha uzun D1 kurtarma gecikmeleri durumunda, daha uzun sıkma kilidi güçleri geçerli olabilir. Önemli olan, probun zaman içindeki genel güç birikimidir. Daha uzun sıkma kilidi süreleri gerekiyorsa, sıkma kilidi uzunluklarını yalnızca küçük adımlarla artırın ve kilit sinyalini izleyin. Genel bir kural olarak, kilit ekranınızın üst üçte birlik kısmındaki kilit seviyesini ayarlayın. Darbe dizisinin yürütülmesi sırasında kilit seviyesi iki kareden fazla azalırsa, probda biriken döndürme-kilit gücü çok fazladır.
- Alıcı kazancını belirlemek için komut satırına rga yazın. İyi bir sinyal-gürültü oranı için, 128 ile 256 arasında bir alıcı kazancı (rg) değeri hedefleyin, bu da iyi bir su bastırma olduğunu kanıtlar.
NOT: 128 ile 256 arasında bir alıcı kazancına ulaşılamıyor, çok güçlü bir su sinyali olduğunu gösterir. Bu durumda, gradyan gücü ve süreleri, alıcı kazancına ulaşılana kadar nazikçe ayarlanır. Ayrıca Tartışma bölümüne bakın. - Komut satırına zg yazarak 15N R1ρ deneyinin test çalıştırmasını başlatın. Tüm gecikmeler için su sinyalinin iyi bastırıldığını kontrol edin (vplist'te 8 gecikme olması durumunda 1 ila 8). Ayrıca, dörtlü algılama şemasının (Yankı / Anti-Yankı) ikinci artışı olan 9. artışı da kontrol edin.
- HSQC deneyini ( edc yazın) yeni bir dizine kopyalayın. ased yazın ve darbe dizisi parametrelerindeki PULPROG parametre satırının üç noktasına tıklayarak darbe programını 15N R1ρ deneyine değiştirin (ased, bkz . Ek Şekil 3A). Nabız programına çift tıklayın, PULPROG'u veri setine ayarla'ya tıklayın (Ek Şekil 3B'ye bakın) ve Tamam'a tıklayın.
- 15N R1 deneyini ayarlayın.
- 15N R1ρ deneyini kopyalayın ve darbe programını 15N R1 deneyine değiştirin.
- vclist yazın, bir vclist oluşturun ve70: 0, 24, 6, 20, 4, 16, 8, 12'de açıklanan sahte vclist'i girin; her VCLIST girdisi için yeni bir satır).
- Açık ased. Degrade uzunluklarını ve güçlerini kontrol edin. GPZ9'u %14 olarak değiştirin.
- Darbe uzunluklarını, darbe şekillerini ve darbe kuvvetlerini kontrol edin (doğru değerler 15N R1ρ deneyinden kopyalanmış olmalıdır).
- 15N13C etiketli numuneler söz konusu olduğunda, darbe programında #define LABEL_CN kullanın.
- Sıcaklık kompanzasyonunun kullanımı için #define TEMP_COMPENSATION ekleyin.
- İlk sekiz FID'yi çalıştırın (L6 = 8 durumunda) ve bunları işleyin (rser < yeni deney numarası>). Vclist'te, en uzun gecikmeli deneyin en yüksek yoğunluğunun, en kısa gecikmeli deneye kıyasla en az 1/e'ye (yaklaşık %37) düştüğü, ancak %25'ten az olmadığı girişleri seçin. En uzun gecikmeli FID ile en kısa gecikmeli FID arasındaki yoğunluk düşüşlerinin eşit bir örneklemesini elde etmek için vplist'in kalan girişlerini ayarlayın.
NOT: vclist girişleri için yalnızca çift sayılara izin verilir.
- {1H}-15N NOE deneyini ayarlayın.
- 15N R1 gevşeme deneyini kopyalayın ve darbe dizisini {1H}-15N NOE darbe şemasına değiştirin.
- Döngü sayacı L8 ile doygunluk süresinin uzunluğunu ayarlayın. 180° (1H) darbeler arasındaki boşluk 22 ms79'dur (2* 1 / JNH). En doğru sonuçları elde etmek için, proteinin 15NT 1 (15N) değerinin yaklaşık 5 katı bir doygunluk süresi kullanın. Burada: 5 sn. Bu, L8 = 230'a karşılık gelir.
NOT: Tartışma bölümü, doygunluk süresi/kurtarma gecikmesinin seçilmesi konusunda ek tavsiyeler sağlar. - Degradelerin uzunluğunu ve gücünü darbe şemasında açıklandığı gibi değiştirin. Sinüs.20 gradyan şekilleri kullanın.
- Dolaylı boyuttaki artış sayısını TD = l3 * 2 * 2'ye eşit olarak ayarlayın
- 15N13C etiketli numuneler söz konusu olduğunda, ZGOPTNS olarak #define LABEL_CN ve -DLABEL_CN kullanın.
- Komut satırına zg yazarak darbe şemalarını çalıştırın.
4. Kaydedilen NMR deneylerinin işlenmesi ve analizi
NOT: Spektrumlar bir Bruker sistemi kullanılarak kaydedilmiştir. İşlem, bir Unix veya Linux işletim sistemi kullanılarak gerçekleştirilir. Spektrum işleme ve veri analizi NMRPipe80 ve python3 kullanılarak gerçekleştirilmiştir. NMRPipe yazılımı https://www.ibbr.umd.edu/nmrpipe/index.html adresinden indirilebilir. NMRPipe tabanlı işleme komut dosyaları web sitesinden indirilir: https://www.ipb.hhu.de/en/teams/team-lakomek/pulsesequences veya genişletilmiş Biyolojik Manyetik Rezonans Bankası (BMRB) deposundan (bmrbig102). NMRPipe kullanılması tavsiye edilir. NMRPipe mevcut değilse veya istenmiyorsa, CCPN81 veya SPARKY (SPARKY 3 veya halefleri NMRFAM-SPARKY82 veya POKY83 ) gibi alternatifler kullanılabilir.
- İşleyin ve analiz edin 15N R1 deney.
NOT: Aşağıdaki komutlar bir Unix Terminalinde yürütülür.- İndirilen dizindeki dosyaları R1_processing deneysel R1 verilerini içeren dizine (çalışma dizini) kopyalayın.
- Dosyaları terminal komutuyla çalıştırılabilir hale getirin: > chmod u+x *.com
- NMRPipe Conversion Utility'yi açmak ve fid.com dosyasını oluşturmak için > bruker yürütün. Parametreleri Oku'ya tıklayın. Üç boyut sayılıyorsa Boyut Sayısı'nı iki olarak değiştirin ve Parametreleri Oku'ya tekrar tıklayın. Ardından Komut Dosyasını Temizle, Komut Dosyasını Güncelle ve Komut Dosyasını Kaydet'e tıklayın (Ek Şekil 9).
NOT: Taşıyıcı frekansı doğru değilse, Parametreleri Oku'ya ikinci kez tıkladıktan sonra Merkez Konum PPM'yi değiştirin. - > ./fid.com yürütün
- vclist'i ms girişleriyle bir vplist'e dönüştürün: ms cinsinden gecikme = 40 ms * vclist girişi. > cp vclist vplist kullanın ve vplist'i düzenleyin.
- Sahte 3B verileri sekiz adet 2B dosyaya bölmek için > ./split.com kullanın.
- nmrproc.com'da tauValues'u (vplist sırasına göre vplist değerleri) ve idxExpmt değerlerini (zamana göre tauValues sırası) düzenleyin (Ek Şekil 10).
- Sekiz 2B spektrumun tümünün ham verilerini işlemek için > ./nmrproc.com kullanın.
- Spektrumları çizmek için > nmrDraw'ı kullanın.
- Bu adımlar için referans spektrumunu (vplist'teki ilk giriş) kullanın. Z boyutunu bir (sol üst köşe) olarak değiştirerek bu spektruma geçin. Faktör düğmesinin yanındaki + ve - işaretlerini kullanarak kontur seviyesini ayarlayın. Çiz düğmesiyle spektrumu yeniden çizin.
- Gürültüden gelen yalnızca birkaç sinyalle ilgilenilen tüm sinyallerin tanınabileceği bir kontur seviyesi kullanın (kontur seviyesi, gürültü seviyesinin kenarındadır) (Ek Şekil 11).
- Spektrum fazını, dikey 1D sinyal projeksiyonu için v tipini ve yatay 1D sinyal projeksiyonu için h'yi düzeltin. Fazı P0 ve P1 ile ayarlayın (Ek Şekil 11), faz farkını hatırlayın ve nmrproc.com dosyasındaki fazı düzeltin (Ek Şekil 10).
NOT: Spektrum 15N boyutunda ters çevrilirse, satırdaki -neg'i kaldırın: | nmrPipe -fn FT -neg \ (Ek Şekil 10). - nmrDraw'ı kapatın ve > nmrproc.com ile tekrar işleyin. Tepe toplama için (spektrum doğru şekilde aşamalandırıldıktan ve referans spektrumu, tüm tepe noktalarının mevcut olduğu bir kontur seviyesiyle görüntülendikten sonra), K (shift + k) yazın, Algıla'ya basın ve Kaydet'e tıklayın. (Ek Şekil 11).
- Bir tepe listesini çalışma dizinine kopyalayın ve -assName'i tepe listesinin adına ass.com olarak değiştirin (Ek Şekil 12).
NOT: Doğru formatta bir zirve listesi örneği şu adreste mevcuttur: https://www.ipb.hhu.de/en/teams/team-lakomek/pulsesequences
ve genişletilmiş Biyolojik Manyetik Rezonans Bankası (BMRB) deposunda (bmrbig102). - Spektrumları atamak için > ./ass.com yürütün. Betiğin çeşitli tepe noktaları için önerdiği atamayı doğrulayın. Tepe noktalarını atamak için farenin sol düğmesini ve tepe noktalarını atamak için farenin sağ düğmesini kullanın. Aşağıdaki amino aside gitmek için İleri düğmesini ve önceki amino aside gitmek için Prev'i kullanın (Ek Şekil 13).
NOT: Kontur seviyesi, sağ üst köşedeki + ve - ile değiştirilebilir. Spektrumu yeni kontur seviyesiyle yeniden çizmek için Çiz düğmesini kullanın (Ek Şekil 13). - Ödevi tamamladıktan sonra Kaydet'e ve ardından tekrar Kaydet'e tıklayın. Daha sonra Çık, İptal Et ve Çık'a tıklayın (Ek Şekil 13).
- Yalnızca atanan tepe noktaları hakkında bilgi içeren relax.tab oluşturmak için python3 NoneDeleter.py > yürütün.
- autofit.com komut dosyasını oluşturmak için ./relax.com > yürütün.
- Bir Fourier Enterpolasyonu gerçekleştirmek için >/autoFit.com yürütün ve her amino asit için farklı gecikme sürelerinin sahte oranlarını oluşturun (axt.tab ve nlin.tab oluşturulur).
- nlin.tab içindeki verilere göre modelExp.com betiği oluşturmak için ./model.com > dosyasını yürütün.
- Doğrusal olmayan En Küçük Kareler Optimizasyon modeliyle her kalıntının evrim eğrisini sığdırmak için >/modelExp.com yürütün.
- Bash summary.tcl -in relax.tab > t1.tab > yürütün. Olası bir hata iletisini yoksayın: GDB Bildirimi: Tablo dosyasında 21 eksik değer oluşabilir.
- t1.tab dosyasını yeni oluşturulan bir R1R2 dizinine kopyalayın.
- 15N R1ρ deneyini işleyin ve analiz edin.
- İndirilen dizindeki dosyaları R1rho_processing deneysel R1ρ verilerini içeren dizine (çalışma dizini) kopyalayın.
- Execute > chmod u+x *.com
- NMRPipe Conversion Utility'yi açmak ve fid.com dosyasını oluşturmak için > bruker yürütün. Ayrıca 15N R1 spektrumunun işlenmesine bakın.
- > ./fid.com yürütün
- Sahte > split.com verileri sekiz adet 2B dosyaya bölmek için ./3 komutunu çalıştırın.
- Sekiz 2B spektrumun tümünün ham verilerini işlemek için ./nmrproc.com > ( tauValues [vplist değerlerini] ve idxExpmt'i önceden düzenleyin) yürütün (Ek Şekil 10).
- Spektrumları çizmek ve tepe noktalarını seçmek için > nmrDraw çalıştırın. Bu adımlar için referans spektrumunu (vplist'teki ilk giriş) kullanın. 15N R1 deneyi ile aynı prosedürü izleyin.
- Bir tepe listesini çalışma dizinine kopyalayın ve tepe listesinin adına ass.com olarak -assName değerini değiştirin (Ek Şekil 12).
- ./ass.com > yürütün ve 15N R1 deneyinde olduğu gibi aynı prosedürü izleyin.
- Yalnızca atanan tepe noktaları hakkında bilgi içeren relax.tab oluşturmak için python3 NoneDeleter.py > çalıştırın.
- > yürüt ./relax.com
- > ./autoFit.com yürütün
- > ./model.com yürütün
- > ./modelExp.com yürütün
- > bash summary.tcl -in relax.tab > t1rho'yu çalıştırın. Olası hata iletisini yoksay: GDB Uyarısı: Tablo dosyasında 21 eksik değer oluşabilir.
- Aşağıdaki üst bilgiyi t1rho.tab dosyasına kopyalayın (değerler fid.com dosyasından elde edilebilir) (Ek Şekil 14):
AÇIKLAMA T1rho için 600MHz
AÇIKLAMA -yOBS = 60.818 (buraya fid.com'da bulunan değeri koyun)
AÇIKLAMA -yCAR = 119.06 (buraya fid.com'da bulunan değeri koyun) - Taşıyıcı frekansına göre tepe 15N frekansının ofsetini içeren fusionOffset.tab adlı bir tablo oluşturmak için ./extract_PPM.com > yürütün (bu, R1 ve R1ρ verilerinden R2 verilerinin çıkarılması için θ açısını hesaplamak için gerekli olacaktır).
- Gevşeme verilerini özetleyin.
- t1rho.tab ve fusionOffset.tab dosyalarını R1R2 dizinine kopyalayın.
- R1R2 dizinindeki crtR1p.tcl betiğini kopyalayın, spektrometre frekansını ve döndürme kilidi gücünü ayarlayın ve bash crtR1p.tcl | sort -n > R1R2.tab komutunu çalıştırın. (Ek Şekil 15)'dir. R1R2.tab, takılan R1ρ hız sabitlerini, teta açısını, R2 hız sabitlerini ve R1 hız sabitlerini içerecektir.
- hetNOE deneyini işleyin ve analiz edin.
- İndirilen dizindeki dosyaları NOE_processing deneysel NOE veri dizinine (çalışma dizini) kopyalayın.
- Execute > chmod u+x *.com
- NMRPipe Conversion Utility'yi açmak ve fid.com dosyasını oluşturmak için > bruker yürütün. Parametreleri Oku'ya tıklayın. Üç boyut sayılmışsa Boyut Sayısı'nı iki olarak değiştirin ve Parametreleri Oku'ya tekrar basın. Ardından Komut Dosyasını Temizle, Komut Dosyasını Güncelle ve Komut Dosyasını Kaydet'e basın.
- > ./fid.com yürütün
- Execute > ./split.com (Daha önce iki girişli (0, 1) bir vplist oluşturulmalıdır)
- > ./ft2.com yürütün
- Spektrumu çizmek ve referans spektrumundaki tepe noktalarını seçmek için nmrDraw '> çalıştırın (z-boyutu = 1). Gürültüyü çıkarın ve hatırlayın ( T = shift + t tuşlarına basın). Faz düzeltmesi ve tepe toplama, 15N R1 deneyi ile aynı prosedürü takip eder.
- Bir tepe listesini çalışma dizinine kopyalayın ve -assName'i tepe listesinin adına ass.com olarak değiştirin (Ek Şekil 12)
- Spektrumları atamak için > ./ass.com yürütün. 15N R1 deneyi için ayrıntılı olarak açıklanan prosedürün aynısını izleyin.
- Relax.tab oluşturmak için python3 NoneDeleter.py > yürütün.
- > ./script.com yürütün
- > ./autoFit.com yürütün
- error_hetnoe.tcl'deki gürültüyü değiştirin.
- Execute > bash error_hetnoe.tcl |sort -n > hetNOE.tab
Sonuçlar
Aşağıda, sıklıkla VAMP2 (vezikül ile ilişkili protein 2) olarak adlandırılan veziküler SNARE proteini Synaptobrevin-2 (1-96) üzerinde kaydedilen bazı örnek NMR gevşeme verileri gösterilmektedir. NMR verilerini kaydetmek için, 150 mM NaCl, 0.1 mM TCEP ve 1 mM EDTA içeren 50 mM MES (pH 6.0) tamponunda 171 μM 15N Sinaptobrevin-2 (1-96) örneği (aşağıda Syb-2 olarak adlandırılmıştır) kullandık. Tüm deneysel veriler, 3 mm'lik bir NMR numune tüpüne dol...
Tartışmalar
Bu protokol, Lakomek ve ark.69 ve Stief ve ark.70 tarafından NMR 15N gevşeme deneylerinin kurulumunu tanımlamıştır. Hassasiyeti artırılmış bir HSQC algılama şeması kullanarak NMR darbe dizilerine odaklandık. 15NR 1 ve R1ρ deneyleri, Stief ve ark.70 tarafından ayrıntılı olarak açıklandığı gibi uygulanmıştır ve hetNOE deneyi, Lakomek ve
Açıklamalar
Yazarlar, bu yazıda rapor edilen çalışmayı etkilemiş gibi görünebilecek bilinen hiçbir rakip mali çıkarları veya kişisel ilişkileri olmadığını beyan ederler.
Teşekkürler
Yararlı tartışmalar için Melinda Jaspert ve Kevin Bochinsky'ye teşekkür ederiz. N.L., Alman Bilim Vakfı'na Heisenberg Programı (DFG hibe numarası 433700474) aracılığıyla fon sağladığı için teşekkür eder. Bu çalışma, Helmholtz Derneği'nin Girişim ve Ağ Oluşturma Fonu'ndan bir hibe olan "COVID-19 patogenezinin virolojik ve immünolojik belirleyicileri - gelecekteki pandemilere hazırlanmak için dersler (KA1-Co-02 "COVIPA") projesi tarafından daha da desteklenmektedir. Forschungszentrum Jülich ve Heinrich Heine Üniversitesi Düsseldorf (HHU) tarafından ortaklaşa yürütülen Jülich-Düsseldorf Biyomoleküler NMR Merkezi'ne cömert erişim sağladığımızı kabul ediyoruz.
Malzemeler
| Name | Company | Catalog Number | Comments |
| Bruker 600 MHz AVANCE III HD spectrometer | Bruker | https://www.bruker.com/en/products-and-solutions/mr/nmr/avance-nmr-spectrometer.html | NMR experiments conducted |
Referanslar
- Sekhar, A., Kay, L. E. An NMR view of protein dynamics in health and disease. Annu Rev Biophys. 48, 297-319 (2019).
- Rosenzweig, R., Kay, L. E. Bringing dynamic molecular machines into focus by methyl-TROSY NMR. Annu Rev Biochem. 83, 291-315 (2014).
- Palmer, A. G. Enzyme dynamics from NMR spectroscopy. Acc Chem Res. 48 (2), 457-465 (2015).
- Reif, B. Ultra-high resolution in MAS solid-state NMR of perdeuterated proteins: implications for structure and dynamics. J Magn Reson. 216, 1-12 (2012).
- Schanda, P., Ernst, M. Studying dynamics by magic-angle spinning solid-state NMR spectroscopy: Principles and applications to biomolecules. Prog Nucl Magn Reson Spectrosc. 96, 1-46 (2016).
- Le Marchand, T., et al. (1)H-H-detected biomolecular NMR under fast magic-angle spinning. Chem Rev. 122 (10), 9943-10018 (2022).
- Pervushin, K., Riek, R., Wider, G., Wuthrich, K. Attenuated T2 relaxation by mutual cancellation of dipole-dipole coupling and chemical shift anisotropy indicates an avenue to NMR structures of very large biological macromolecules in solution. Proc Natl Acad Sci U S A. 94 (23), 12366-12371 (1997).
- Tzakos, A. G., Grace, C. R., Lukavsky, P. J., Riek, R. NMR techniques for very large proteins and RNAs in solution. Annu Rev Biophys Biomol Struct. 35, 319-342 (2006).
- Sprangers, R., Kay, L. E. Quantitative dynamics and binding studies of the 20S proteasome by NMR. Nature. 445 (7128), 618-622 (2007).
- Wright, P. E., Dyson, H. J. Intrinsically disordered proteins in cellular signalling and regulation. Nat Rev Mol Cell Biol. 16 (1), 18-29 (2015).
- van der Lee, R., et al. Classification of intrinsically disordered regions and proteins. Chem Rev. 114 (13), 6589-6631 (2014).
- Tompa, P. Intrinsically unstructured proteins. Trends Biochem Sci. 27 (10), 527-533 (2002).
- Dyson, H. J., Wright, P. E. Coupling of folding and binding for unstructured proteins. Curr Opin Struct Biol. 12 (1), 54-60 (2002).
- Uversky, V. N. Intrinsically disordered proteins in overcrowded milieu: Membrane-less organelles, phase separation, and intrinsic disorder. Current Opinion in Structural Biology. 44, 18-30 (2017).
- Patel, A., et al. A liquid-to-solid phase transition of the ALS protein FUS accelerated by disease mutation. Cell. 162 (5), 1066-1077 (2015).
- Abyzov, A., Blackledge, M., Zweckstetter, M. Conformational dynamics of intrinsically disordered proteins regulate biomolecular condensate chemistry. Chem Rev. 122 (6), 6719-6748 (2022).
- Uversky, V. N. Unusual biophysics of intrinsically disordered proteins. Biochim Biophys Acta. 1834 (5), 932-951 (2013).
- Papoian, G. A. Proteins with weakly funneled energy landscapes challenge the classical structure-function paradigm. Proc Natl Acad Sci U S A. 105 (38), 14237-14238 (2008).
- Uversky, V. N., Gillespie, J. R., Fink, A. L. Why are "natively unfolded" proteins unstructured under physiologic conditions. Proteins. 41 (3), 415-427 (2000).
- Bah, A., et al. Folding of an intrinsically disordered protein by phosphorylation as a regulatory switch. Nature. 519 (7541), 106-109 (2015).
- Tompa, P., Schad, E., Tantos, A., Kalmar, L. Intrinsically disordered proteins: emerging interaction specialists. Curr Opin Struct Biol. 35, 49-59 (2015).
- Dyson, H. J., Wright, P. E. Perspective: the essential role of NMR in the discovery and characterization of intrinsically disordered proteins. J Biomol NMR. 73 (12), 651-659 (2019).
- Adamski, W., et al. A unified description of intrinsically disordered protein dynamics under physiological conditions using NMR spectroscopy. J Am Chem Soc. 141 (44), 17817-17829 (2019).
- Dobson, C. M. Protein folding and misfolding. Nature. 426 (6968), 884-890 (2003).
- Bertoncini, C. W., et al. Release of long-range tertiary interactions potentiates aggregation of natively unstructured alpha-synuclein. Proc Natl Acad Sci U S A. 102 (5), 1430-1435 (2005).
- Buell, A. K., et al. Solution conditions determine the relative importance of nucleation and growth processes in alpha-synuclein aggregation. Proc Natl Acad Sci U S A. 111 (21), 7671-7676 (2014).
- Salvi, N., Abyzov, A., Blackledge, M. Atomic resolution conformational dynamics of intrinsically disordered proteins from NMR spin relaxation. Prog Nucl Magn Reson Spectrosc. 102, 43-60 (2017).
- Schneider, R., Blackledge, M., Jensen, M. R. Elucidating binding mechanisms and dynamics of intrinsically disordered protein complexes using NMR spectroscopy. Curr Opin Struct Biol. 54, 10-18 (2019).
- Rezaei-Ghaleh, N., Blackledge, M., Zweckstetter, M. Intrinsically disordered proteins: from sequence and conformational properties toward drug discovery. Chembiochem. 13 (7), 930-950 (2012).
- Jensen, M. R., Salmon, L., Nodet, G., Blackledge, M. Defining conformational ensembles of intrinsically disordered and partially folded proteins directly from chemical shifts. J Am Chem Soc. 132 (4), 1270-1272 (2010).
- Camacho-Zarco, A. R., et al. NMR provides unique insight into the functional dynamics and interactions of intrinsically disordered proteins. Chem Rev. 122 (10), 9331-9356 (2022).
- Mittag, T., Forman-Kay, J. D. Atomic-level characterization of disordered protein ensembles. Curr Opin Struct Biol. 17 (1), 3-14 (2007).
- Dyson, H. J., Wright, P. E. Unfolded proteins and protein folding studied by NMR. Chem Rev. 104 (8), 3607-3622 (2004).
- Dyson, H. J., Wright, P. E. NMR illuminates intrinsic disorder. Curr Opin Struct Biol. 70, 44-52 (2021).
- Sugase, K., Dyson, H. J., Wright, P. E. Mechanism of coupled folding and binding of an intrinsically disordered protein. Nature. 447 (7147), 1021-1025 (2007).
- Bessa, L. M., et al. The intrinsically disordered SARS-CoV-2 nucleoprotein in dynamic complex with its viral partner nsp3a. Sci Adv. 8 (3), eabm4034 (2022).
- Milles, S., Salvi, N., Blackledge, M., Jensen, M. R. Characterization of intrinsically disordered proteins and their dynamic complexes: From in vitro to cell-like environments. Prog Nucl Magn Reson Spectrosc. 109, 79-100 (2018).
- Salvi, N., Abyzov, A., Blackledge, M. Atomic resolution conformational dynamics of intrinsically disordered proteins from NMR spin relaxation. Prog Nucl Magn Reson Spectrosc. 102 - 103, 43-60 (2017).
- Gill, M. L., Byrd, R. A., Palmer, A. G. Dynamics of GCN4 facilitate DNA interaction: a model-free analysis of an intrinsically disordered region. Phys Chem Chem Phys. 18 (8), 5839-5849 (2016).
- Bertoncini, C. W., et al. Structural characterization of the intrinsically unfolded protein beta-synuclein, a natural negative regulator of alpha-synuclein aggregation. J Mol Biol. 372 (3), 708-722 (2007).
- Rezaei-Ghaleh, N., et al. Local and global dynamics in intrinsically disordered synuclein. Angew Chem Int Ed Engl. 57 (46), 15262-15266 (2018).
- Rezaei-Ghaleh, N., Parigi, G., Zweckstetter, M. Reorientational dynamics of amyloid-beta from NMR spin relaxation and molecular simulation. J Phys Chem Lett. 10 (12), 3369-3375 (2019).
- Palmer, A. G. NMR characterization of the dynamics of biomacromolecules. Chem Rev. 104 (8), 3623-3640 (2004).
- Palmer, A. G., Massi, F. Characterization of the dynamics of biomacromolecules using rotating-frame spin relaxation NMR spectroscopy. Chem Rev. 106 (5), 1700-1719 (2006).
- Kay, L. E., Torchia, D. A., Bax, A. Backbone dynamics of proteins as studied by 15N inverse detected heteronuclear NMR spectroscopy: application to staphylococcal nuclease. Biochemistry. 28 (23), 8972-8979 (1989).
- Mittermaier, A., Kay, L. E. Review - New tools provide new insights in NMR studies of protein dynamics. Science. 312 (5771), 224-228 (2006).
- Abragam, A. . Principles of Nuclear Magnetism. , (1983).
- Ernst, R. R., Bodenhausen, G., Wokaun, A. . Principles of Nuclear Magnetic Resonance in One and Two Dimensions. , (1990).
- Cavanagh, J., Fairbrother, W. J., Palmer, A. G., Skelton, N. J. . Protein NMR Spectroscopy: Principles and Practice. , (1995).
- Levitt, M. H. . Spin Dynamics: Basics of Nuclear Magnetic Resonance. , (2008).
- Keeler, J. . Understanding NMR Spectroscopy. , (2010).
- Bloembergen, N., Purcell, E. M., Pound, R. V. Relaxation effects in nuclear magnetic resonance absorption. Phys Rev. 73 (7), 679-712 (1948).
- Wangsness, R. K., Bloch, F. The dynamical theory of nuclear induction. Phys Rev. 89 (4), 728-739 (1953).
- Redfield, A. G. On the theory of relaxation processes. IBM J Res Dev. 1 (1), 19-31 (1957).
- Peng, J. W., Wagner, G. Mapping of the spectral densities of N-H bond motions in eglin c using heteronuclear relaxation experiments. Biochemistry. 31 (36), 8571-8586 (1992).
- Farrow, N. A., et al. Backbone dynamics of a free and a phosphopeptide-complexed src homology-2 domain studied by n-15 NMR relaxation. Biochemistry. 33 (19), 5984-6003 (1994).
- Tolman, J. R., Ruan, K. NMR residual dipolar couplings as probes of biomolecular dynamics. Chem Rev. 106 (5), 1720-1736 (2006).
- Mulder, F. A. A., Skrynnikov, N. R., Hon, B., Dahlquist, F. W., Kay, L. E. Measurement of slow (mu s-ms) time scale dynamics in protein side chains by N-15 relaxation dispersion NMR spectroscopy: Application to Asn and Gln residues in a cavity mutant of T4 lysozyme. J Am Chem Soc. 123 (5), 967-975 (2001).
- Tollinger, M., Skrynnikov, N. R., Mulder, F. A. A., Forman-Kay, J. D., Kay, L. E. Slow dynamics in folded and unfolded states of an SH3 domain. J Am Chem Soc. 123 (46), 11341-11352 (2001).
- Akke, M., Palmer, A. G. Monitoring macromolecular motions on microsecond to millisecond time scales by R(1)rho-R(1) constant relaxation time NMR spectroscopy. J Am Chem Soc. 118 (4), 911-912 (1996).
- Loria, J. P., Rance, M., Palmer, A. G. A relaxation-compensated Carr-Purcell-Meiboom-Gill sequence for characterizing chemical exchange by NMR spectroscopy. J Am Chem Soc. 121 (10), 2331-2332 (1999).
- Vallurupalli, P., Bouvignies, G., Kay, L. E. Studying "Invisible" excited protein states in slow exchange with a major state conformation. J Am Chem Soc. 134 (19), 8148-8161 (2012).
- Jeener, J., Meier, B. H., Bachmann, P., Ernst, R. R. Investigation of exchange processes by 2-dimensional NMR-spectroscopy. J Chem Phys. 71 (11), 4546-4553 (1979).
- Palmer, A. G., Kroenke, C. D., Loria, J. P. Nuclear magnetic resonance methods for quantifying microsecond-to-millisecond motions in biological macromolecules. Methods Enzymol. 339, 204-238 (2001).
- Clore, G. M., Iwahara, J. Theory, practice, and applications of paramagnetic relaxation enhancement for the characterization of transient low-population states of biological macromolecules and their complexes. Chem Rev. 109 (9), 4108-4139 (2009).
- Iwahara, J., Clore, G. M. Detecting transient intermediates in macromolecular binding by paramagnetic NMR. Nature. 440 (7088), 1227-1230 (2006).
- Lakomek, N. A., et al. Residual dipolar couplings as a tool to study molecular recognition of ubiquitin. Biochem Soc Trans. 36 (Pt 6), 1433-1437 (2008).
- Johnson, C. N., Libich, D. S. Paramagnetic relaxation enhancement for detecting and characterizing self-associations of intrinsically disordered proteins. J Vis Exp. (175), e63057 (2021).
- Lakomek, N. A., Ying, J., Bax, A. Measurement of (1)(5)N relaxation rates in perdeuterated proteins by TROSY-based methods. J Biomol NMR. 53 (3), 209-221 (2012).
- Stief, T., Vormann, K., Lakomek, N. A. Sensitivity-enhanced NMR (15)N R(1) and R(1rho) relaxation experiments for the investigation of intrinsically disordered proteins at high magnetic fields. Methods. 223, 1-15 (2024).
- Kay, L. E., Keifer, P., Saarinen, T. Pure absorption gradient enhanced heteronuclear single quantum correlation spectroscopy with improved sensitivity. J Am Chem Soc. 114 (26), 10663-10665 (1992).
- Geen, H., Freeman, R. Band-selective radiofrequency pulses. J Magn Reson (1969). 93 (1), 93-141 (1991).
- Desvaux, H., Berthault, P. Study of dynamic processes in liquids using off-resonance RF irradiation. Prog Nucl Magn Reson Spectrosc. 35 (4), 295-340 (1999).
- Overhauser, A. W. Polarization of nuclei in metals. Phys Rev. 91 (2), 476-476 (1953).
- Overhauser, A. W. Polarization of nuclei in metals. Phys Rev. 92 (2), 411-415 (1953).
- Kumari, P., Frey, L., Sobol, A., Lakomek, N. A., Riek, R. (15)N transverse relaxation measurements for the characterization of micros-ms dynamics are deteriorated by the deuterium isotope effect on (15)N resulting from solvent exchange. J Biomol NMR. 72 (3-4), 125-137 (2018).
- Karschin, N., Krenek, S., Heyer, D., Griesinger, C. Extension and improvement of the methanol-d(4) NMR thermometer calibration. Magn Reson Chem. 60 (4), 203-209 (2022).
- Sattler, M., Schleucher, J., Griesinger, C. Heteronuclear multidimensional NMR experiments for the structure determination of proteins in solution employing pulsed field gradients. Prog Nucl Magn Reson Spectrosc. 34 (2), 93-158 (1999).
- Ferrage, F., Cowburn, D., Ghose, R. Accurate sampling of high-frequency motions in proteins by steady-state N-{H} nuclear Overhauser effect measurements in the presence of cross-correlated relaxation. J Am Chem Soc. 131 (17), 6048-6049 (2009).
- Delaglio, F., et al. NMRPipe: a multidimensional spectral processing system based on UNIX pipes. J Biomol NMR. 6 (3), 277-293 (1995).
- Skinner, S. P., et al. CcpNmr AnalysisAssign: a flexible platform for integrated NMR analysis. J Biomol NMR. 66 (2), 111-124 (2016).
- Lee, W., Tonelli, M., Markley, J. L. NMRFAM-SPARKY: enhanced software for biomolecular NMR spectroscopy. Bioinformatics. 31 (8), 1325-1327 (2015).
- Lee, W., Rahimi, M., Lee, Y., Chiu, A. POKY: a software suite for multidimensional NMR and 3D structure calculation of biomolecules. Bioinformatics. 37 (18), 3041-3042 (2021).
- Abyzov, A., et al. Identification of dynamic modes in an intrinsically disordered protein using temperature-dependent NMR relaxation. J Am Chem Soc. 138 (19), 6240-6251 (2016).
- Singh, A., Purslow, J. A., Venditti, V. 15N CPMG relaxation dispersion for the investigation of protein conformational dynamics on the micros-ms timescale. J Vis Exp. (170), e62395 (2021).
- Gopalan, A. B., Hansen, D. F., Vallurupalli, P. CPMG experiments for protein minor conformer structure determination. Methods Mol Biol. 1688, 223-242 (2018).
- Tugarinov, V., Kay, L. E. Methyl groups as probes of structure and dynamics in NMR studies of high-molecular-weight proteins. Chembiochem. 6 (9), 1567-1577 (2005).
- Lipari, G., Szabo, A. Model-free approach to the interpretation of nuclear magnetic resonance relaxation in macromolecules. 1. Theory and range of validity. J Am Chem Soc. 104 (17), 4546-4559 (1982).
- Lipari, G., Szabo, A. Model-Free Approach to the interpretation of nuclear magnetic-resonance relaxation in macromolecules .2. Analysis of experimental results. J Am Chem Soc. 104 (17), 4559-4570 (1982).
- Clore, G. M., et al. Deviations from the simple two-parameter model-free approach to the interpretation of nitrogen-15 nuclear magnetic relaxation of proteins. J Am Chem Soc. 112 (12), 4989-4991 (1990).
- Lemaster, D. M. Larmor frequency selective model free analysis of protein NMR relaxation. J Biomol NMR. 6 (4), 366-374 (1995).
- Peng, J. W., Wagner, G. Mapping of spectral density functions using heteronuclear NMR relaxation measurements. J Magn Reson (1969). 98 (2), 308-332 (1992).
- Farrow, N. A., Zhang, O. W., FormanKay, J. D., Kay, L. E. Characterization of the backbone dynamics of folded and denatured states of an SH3 domain. Biochemistry. 36 (9), 2390-2402 (1997).
- Khan, S. N., et al. Distribution of pico- and nanosecond motions in disordered proteins from nuclear spin relaxation. Biophys J. 109 (5), 988-999 (2015).
- Smith, A. A., Ernst, M., Meier, B. H., Ferrage, F. Reducing bias in the analysis of solution-state NMR data with dynamics detectors. J Chem Phys. 151 (3), 034102 (2019).
- Zumpfe, K., Smith, A. A. Model-free or not. Front Mol Biosci. 8, 727553 (2021).
- Robustelli, P., Trbovic, N., Friesner, R. A., Palmer, A. G. Conformational dynamics of the partially disordered yeast transcription factor GCN4. J Chem Theory Comput. 9 (11), 5190-5200 (2013).
- Salvi, N., Abyzov, A., Blackledge, M. Multi-timescale dynamics in intrinsically disordered proteins from NMR relaxation and molecular simulation. J Phys Chem Lett. 7 (13), 2483-2489 (2016).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiThis article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır