需要订阅 JoVE 才能查看此. 登录或开始免费试用。
Method Article
NMR 15N 弛豫实验,用于研究蛋白质皮秒至纳秒的结构动力学
摘要
核磁共振 (NMR) 波谱可以以残基特异性方式表征结构蛋白质动力学。我们提供了一个动手实验方案,用于记录 NMR 15N R1 和 R2 弛豫和 {1H}-15N 异核 Overhauser 效应 (hetNOE) 实验,对皮秒到纳秒的时间尺度敏感。
摘要
核磁共振 (NMR) 波谱允许在溶液和生理温度下研究蛋白质。通常,蛋白质骨架的酰胺基团或侧链中的甲基被用作蛋白质结构动力学的报告基因。在 15N 标记和完全质子化的样品上对球状蛋白质骨架的结构动力学研究通常适用于分子量高达 50 kDa 的蛋白质。当侧链氘与横向弛豫优化光谱 (TROSY) 相结合时,球状蛋白质的极限可以扩展到 200 kDa,当重点放在侧链上时,该极限可以扩展到 1 MDa。当研究固有无序蛋白 (IDP) 或具有固有无序区 (IDR) 的蛋白质时,这些重量限制不适用,但可能远远超出范围。原因是 IDP 或 IDR 具有高度内部灵活性的特点,经常是动态解耦的。各种 NMR 方法提供了对蛋白质结构动力学的原子分辨率见解,范围很广,从皮秒到几小时。标准的 15N 弛豫测量概述了蛋白质的内部柔韧性,并表征了在快速皮秒到纳秒时间尺度上经历的蛋白质骨架动力学。本文介绍了一种用于设置和记录 NMR 15NR1、R2 和异核 Overhauser 效应 (hetNOE) 实验的动手实验方案。我们展示了示例性数据,并解释了如何在任何更复杂的分析之前简单地定性地解释它们。
引言
蛋白质的功能不仅取决于其三维结构,还取决于其结构动力学,包括其内部灵活性和蛋白质将采用的不同构象之间的结构转变。核磁共振 (NMR) 波谱可以研究溶液 1,2,3 中蛋白质的结构动力学。质子检测固体 NMR 的最新发展还允许表征难溶状态下的蛋白质动力学,例如脂质双层膜 4,5,6。在溶液 NMR 中,可以研究蛋白质骨架和蛋白质侧链的结构动力学。对于球状蛋白质,一旦蛋白质被 15N 同位素标记,就可以实现高达 50 kDa 的蛋白质骨架的结构动力学研究。当采用侧链氘化和横向弛豫优化光谱 (TROSY) 时,该极限可以扩展到 200 kDa 7,8。当重点在于侧链动力学时,可接近的蛋白质和复合物的范围可以扩展到 1 MDa 2,9。
命名的重量限制不适用于固有无序蛋白 (IDP),它们通常表现出高内在动力学。超过 30% 的真核蛋白质组包含 IDP 或固有无序区 (IDR)10,11,12,13。它们在许多细胞过程中起着核心作用,例如信号转导和转录1,并且经常参与细胞内相分离 14,15,16,17。IDP 在生理条件下缺乏明确的三维 (3D) 天然结构,并且具有弱漏斗或崎岖的能量景观17,18。由于疏水性低且静电排斥力强,分布在 IDP 或 IDR 的主链上,因此缺少折叠成刚性结构的驱动力19。IDP 在与其他结合伴侣复合时经常采用折叠构象 10,20,21。此外,翻译后修饰 (PTM) 扩展了 IDP 或 IDR 的折叠可能性22,23。IDP 的错误折叠已被确定为各种疾病的原因,包括神经退行性疾病 15,24,25,26。
IDP 和 IDR 显示出高度的内部灵活性 21,27,28。显示原子位置和二面角变化的构象集合源自从实验数据 29,30,31,32 获得的分子动力学模拟和约束。由于动力学和由此产生的冻结状态的无序性,漫散电子密度使得很难使用结构生物学中最先进的方法(如冷冻电镜或 X 射线晶体学)对它们进行结构表征。此外,低温下实验的结晶条件或样品制备技术可能会影响 IDP 所经历的构象空间。然而,该解决方案 NMR 适用于高动态蛋白质,因此非常适合研究 IDP16、20、22、28、29、30、31、32、33、34、35、36、37、38。
如上所述,溶液 NMR 提供了各种技术来研究各种时间尺度上的蛋白质内部动力学(图 1),主要基于自旋弛豫 31,33,38,39,40,41,42。
蛋白质骨架酰胺基团中 15N 细胞核的自旋弛豫是由蛋白质内部动力学和集体运动(包括相关时的旋转扩散)引起的 1 H-15N 键角取向变化诱导的27,43,44,45,46,47,48,49,50,51 .在短于旋转相关时间 τR(分子需要旋转一个辐射点的时间,也称为整体翻滚相关时间)的时间尺度上,化学位移各向异性 (CSA) 和偶极耦合 (D) 是活跃的,而不是被蛋白质的旋转扩散平均。蛋白质骨架的内部动力学,包括键角的变化、键的重新取向和旋转翻滚,诱导 CSA 和偶极耦合张量的随机波动,导致局部磁场的变化,最终导致 NMR 自旋弛豫 47,48,52,53.这些波动可以用整体相关函数来描述。整体相关函数的傅里叶变换称为谱密度函数。在半经典的 Redfield 弛豫理论中,NMR 弛豫速率常数可以用这些光谱密度函数的线性组合来描述54。
1990 年代初期开发的主干 15N NMR 弛豫实验包括 15N R1、R1ρ 和 {1H}-15N 核奥弗豪瑟效应实验,对快速皮秒 (ps) 纳秒 (ns) 时间尺度敏感,比蛋白质的旋转相关时间 τR 快 45,55,56,57。为了表征慢于旋转相关时间 τR 的主链动力学,使用了所谓的弛豫色散实验 R1ρ 和 Carr-Purcell-Meiboom-Gill (CPMG) 实验,对微秒 (μs) - 毫秒 (ms) 动力学 44,46,58,59,60,61 敏感。慢于微秒的动力学可以通过 15N 化学交换饱和转移 (CEST) NMR62、交换光谱(EXSY,毫秒到秒)或实时 (RT) NMR(秒到小时)63,64 来捕获。顺磁探针的 PRE(顺磁弛豫增强)效应以及残余偶极耦合 (RDC) 可用于评估 ps 到 ms 动力学 65,66,67,68 的整个范围。
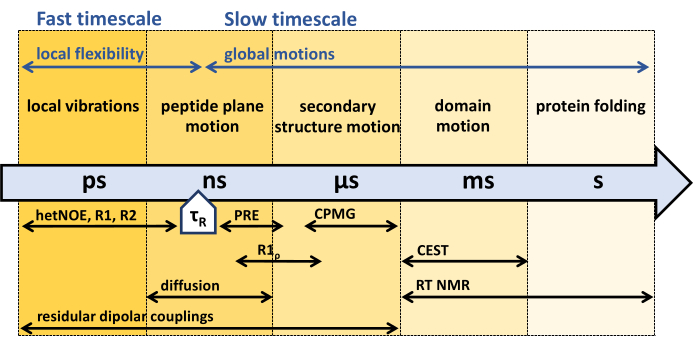
图 1:蛋白质骨架动力学的时间尺度和不同 NMR 动力学实验的敏感时间窗口。 NMR 提供了多种方法来表征各种时间尺度上的蛋白质骨架动力学。蛋白质骨架所经历的不同运动在其各自的时间尺度上表示。蛋白质的旋转相关时间 τR 是蛋白质整体旋转(1 辐射点)所需的时间。比蛋白质的旋转相关时间 τR 快的运动可能与蛋白质的内部柔韧性有关。箭头下方显示了各种 NMR 实验及其对相应时间尺度的敏感性。 请单击此处查看此图的较大版本。
以下协议描述了 Lakomek 等人 69 和 Stief 等人 70 使用灵敏度增强的异核单量子相干 (HSQC) 检测方案进行 NMR 弛豫实验的设置。在进行实验实施之前,本文对 NMR 自旋弛豫和 NMR 弛豫实验进行了非常简短的概述。由于大小限制并保持此协议的可理解性,此概述必须保持简单(因此不完整)。
纵向或自旋晶格弛豫,以 T1 时间或 R1 = 1/ T1 速率常数为特征,描述了磁化强度恢复到玻尔兹曼平衡。在平衡状态下,磁化强度沿外部磁场的轴线对齐,该磁场定义了实验室框架的 z 轴。高 (1H) 和小 (15N) Larmor 频率(NMR 共振频率,例如,14.1 Tesla 磁体在 600 MHz 内持续 1H)的光谱密度以及这些 Larmor 频率的线性组合有助于 15N R1 弛豫,其特征是以 rad·s-1 为单位测量的 15N R1 速率常数.时间尺度上的运动与这些 Larmor 频率相反;因此,皮秒到纳秒时间尺度上的运动有助于弛豫速率常数 R1。对于显示整体翻滚且可以定义旋转相关时间的分子,R1 (T1) 曲线显示了 ωτR = 1 的最大值(最小值),其中旋转相关性 τR 和自旋的 Larmor 频率 ω。如果多个 Larmor 频率有贡献,则频率最低的那个是主导频率,例如,在 15N R1 的情况下为 ωN。快运动状态 (ωτR 远小于 1) 适用于快速翻滚的小分子以及低磁场和低粘度。慢动作状态(ωτR 远大于 1)适用于翻滚速度较慢的较大分子以及高磁场和高粘度。
球状折叠蛋白质在溶液中显示整体翻滚,并且可以分配旋转相关时间。然而,整体翻滚的概念不再适用于固有无序的蛋白质,并且通常与分配单个旋转相关时间不同。在这里,残基特异性内部相关时间变得更加关键。
所描述的测量 15N R1 弛豫率的脉冲序列(图 2)基于灵敏度增强的 HSQC 读出实验,其中回声/反回声检测用于正交检测 69,70,71。具有可变强度和长度的短梯度用于相干性选择和改进的水分抑制70。在此期间,15N 纵向极化将松弛。较长的衰减时间会导致该伪 3D 光谱的相关 2D 平面的强度降低(延迟数据点记录在第三维中)。下面描述的 loop 元素的执行次数会增加,以获得更长的松弛时间。由于 15N 化学位移各向异性 (CSA) 与 1H 和 15N 偶极耦合 (D) 之间的互相关弛豫在弛豫延迟期间也很活跃,因此需要一个对酰胺质子有选择性的中央 I-BURP-2 180° 脉冲72,以重新聚焦互相关弛豫的贡献(如果不重新聚焦,将导致偏斜和错误的 15N R1 速率常数)。
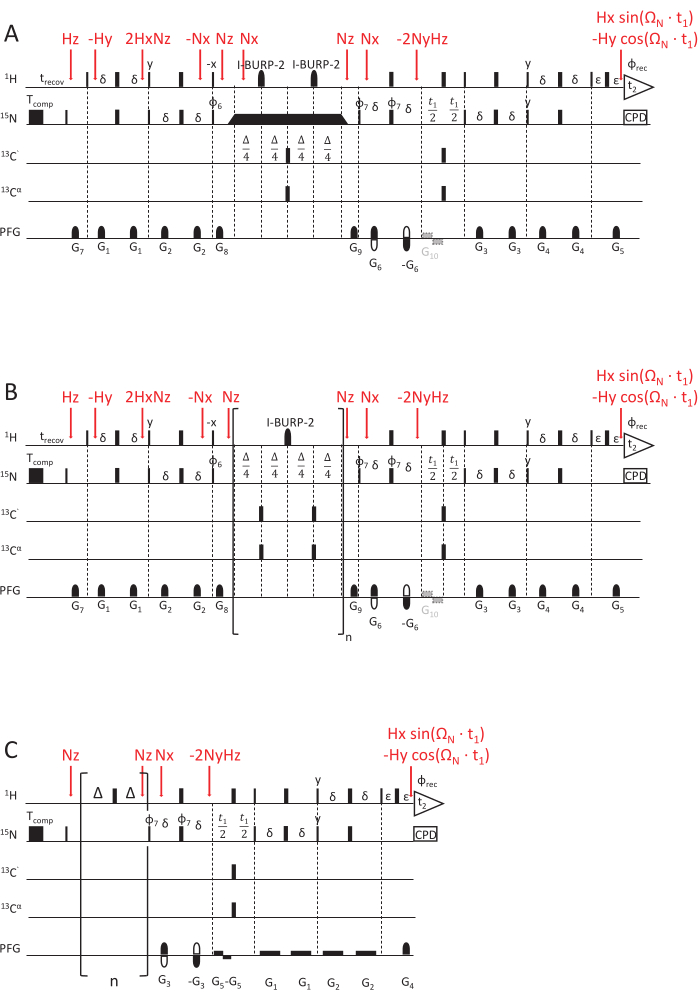
图 2:用于确定 NMR 弛豫速率常数的 NMR 脉冲序列方案。 (A) 15N R1ρ,(B) 15N R1 和 (C) hetNOE 实验,使用灵敏度增强的 HSQC 读数方案69,70。除非另有说明,否则 90°(x) 脉冲由窄矩形表示,180°(x) 脉冲由宽矩形表示。应用以下相位周期:φ6 = y、y、-y、-y;φ7 = y, -y, φrec = y, -y, -y, y.通过反转梯度 G5 的极性和 φ7 的相位周期(回波/反回波检测)来实现正交检测。(A) 15N R1ρ 实验:黑色矩形代表自旋锁,其持续时间会发生变化以获得不同的弛豫延迟。自旋锁前后的三角形表示沿有效磁场轴 Beff 对齐磁化强度的绝热形状脉冲。G10 是一个可选的梯度,用于防止演化阶段水磁化的辐射阻尼。(B) 15N R1 实验:括号部分显示序列的 loop 元素,重复 n 次以匹配所需的弛豫延迟。(C) hetNOE 脉冲方案类似于 R1 和 R1ρ 脉冲方案的后半部分,即 t1 演化时间和 HSQC 检测元件。然而,15N 磁化强度是直接激发的,没有任何 INEPT。质子磁化的饱和(以实现 1H 和 15N 之间的交叉弛豫)是通过施加至少 5 秒的 180(1) 脉冲序列来实现的。参考实验应用了相同长度(此处为 5 s)且没有任何脉冲序列的空闲延迟。G5 是防止辐射阻尼的可选梯度,梯度 G4 的极性反转与相位 φ7 = y、-y、-y、y 相结合,实现正交检测。由产品操作员表示的磁化转移步骤标记为红色。请单击此处查看此图的较大版本。
弛豫速率常数 R2 描述了由于自旋之间的相位相干性丧失而导致的横向极化弛豫(在与外部磁场正交的 xy 平面中),导致可检测磁化强度衰减53,54。高频和小频的频谱密度函数对 R2 有贡献,类似于 R1。然而,对 R2 的最大贡献来自零频率下的频谱密度。因此,R2 对旋转翻滚非常敏感,旋转相关时间 τR 表示,在室温下,小球状蛋白质的旋转翻滚大约为几纳秒。因此,数百 ps 到低 ns 范围内的较慢主干运动贡献最大。原则上,导致 15N 原子核的化学位移张量的各向同性部分调制的骨架的交换动力学,将交换贡献 R2ex 添加到 R2 速率常数 43,44,49,60,61。在所描述的实验中,R2ex 贡献被自旋锁抑制,该自旋锁重新聚焦动力学的速度比自旋锁的倒圆频率慢。自旋锁是一个长连续波射频脉冲,它使磁化强度沿有效磁场轴 Beff(自旋锁 ω1 场的矢量和与 15N 载波频率的化学位移偏移量(见下文))对齐。沿 B1,eff 轴对齐的磁化强度的弛豫称为 R1ρ 弛豫,它具有 R1 分量和 R2 分量。使用公式 (1),R2 可以从 R1ρ 和 R144,73 计算出来:
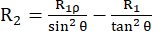 (1).
(1).
有效磁场 B的轴 eff 与外部磁场 B0 之间的夹角为 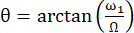 .ω1 是自旋锁的 RF 振幅,Ω相应残基的 15N 化学位移与 15N 载波频率之间的化学位移偏移 44,73。
.ω1 是自旋锁的 RF 振幅,Ω相应残基的 15N 化学位移与 15N 载波频率之间的化学位移偏移 44,73。
R1ρ脉冲方案(图2A,70)与15N R1方案非常相似,只是松弛延迟不同。 为了测量 15N R1ρ 弛豫率,在磁化强度沿有效磁场轴 Beff 通过与自旋锁具有相同射频 (RF) 幅度的绝热脉冲对准后,自旋锁必须处于活动状态。spin-lock 的长度将发生变化以获得不同的 relaxation delays。
稳态 {1H}-15N 核 Overhauser 效应 (1 H-15N NOE),在下文中称为 hetNOE,是交叉弛豫速率与 15N 纵向弛豫速率的比率。由于质子极化饱和时与质子的交叉弛豫,它导致 15N 上的稳态极化减少 45,53,54,74,75。交叉弛豫取决于 1H 和 15N Larmor 频率之和之差的频谱密度函数。因此,hetNOE 对快速皮秒动力学 (< 100 ps) 和 ps-ns 动力学(由于其 R1 依赖性)都敏感。序列69(图 2C)基于灵敏度增强的 HSQC 读数,带有用于正交检测的回声/反回声梯度。对于质子磁化强度的饱和和由此产生的 hetNOE,平衡质子磁化强度被反转,随后通过快速脉冲 180° 脉冲达到饱和,约为 15 N T1 的 5 倍。对于参考实验,恢复延迟等于饱和延迟,但没有 1H 180°脉冲序列。为参考实验和饱和度为 1H 的实验增加了 D1 = 2 s 的额外延迟。两个实验都是背靠背记录的,仅在施加 1H 180°脉冲(饱和)或不施加(参考)方面有所不同。实验中记录的 1H 饱和度的光谱强度除以参考实验(没有 180° 质子脉冲序列)的强度之比,得出 {1H}-15N NOE (hetNOE) 值。
以下协议描述了 Lakomek 等人 69 和 Stief 等人 70 的 NMR 弛豫实验的设置。我们专注于使用灵敏度增强的 HSQC 检测方案的 NMR 脉冲序列。 15N R1 和 R1ρ 实验按照 Stief 等人 70 的详细描述实现,hetNOE 实验由 Lakomek 等人 69 描述。
Access restricted. Please log in or start a trial to view this content.
研究方案
1. NMR 样品制备
注:蛋白质的同位素标记用于高维 NMR 和高级 NMR 实验。当使用富培养基(例如,Luria-Bertani [LB] 或 2x 酵母提取物胰蛋白胨培养基 [2YT])以每升几毫克的产量建立大 肠杆菌 中的蛋白质表达和蛋白质纯化时,制备同位素标记的 NMR 样品通常相对简单。
- 对于同位素标记,使用 M9 最低培养基进行基因表达,其中 15N 氯化铵是唯一的氮源, 13N 富集 C 的葡萄糖是唯一的碳源。
- 对于蛋白质纯化,请遵循实验室中为相应蛋白质制定的标准方案。
注:结果部分显示了 IDP(单体 SNARE 蛋白 Synaptobrevin-2 (1-96))的代表性结果。Synaptobrevin-2 的 NMR 同位素标记已按照70 中的描述进行。对于非常高精度的测量,建议使用低于 5% 的低 D2O 浓度。这是因为氘同位素效应,可以在 Hahn-Echo 15N T2 和低频 15N CPMG 弛豫色散实验(此处不讨论)中观察到,但在 15N R1ρ 实验(主要重新聚焦)中影响很小76。
2. 在波谱仪上运行 NMR 弛豫实验的准备工作
注意:所描述的 NMR 弛豫实验是特定于布鲁克波谱仪的。它们已在低温和室温 1H、 15N 和 13C 三共振探头以及由布鲁克软件 Topsin 3.6 或更高版本操作的 Avance III 和 Avance Neo 控制台上进行了测试。
- 请遵循预防措施。
- 在使用 NMR 波谱仪之前,请先进行安全简报。
- 检查所用光谱仪的探头功率规格,并为每个脉冲选择合适的脉冲长度和脉冲功率,例如,通过与 Topspin “prosol” 表条目的一致性进行比较。
- 导入脉冲序列。
- 从下面指示的网站或扩展的生物磁共振库 (BMRB) 存储库 (bmrbig102) 下载 NMR 脉冲程序,下载到操作 NMR 波谱仪的计算机的目录 /opt/topspin4.1.4/exp/stan/nmr/lists/pp/user。请调整 topspin 版本(此处:topspin4.1.4 为光谱仪上安装的相应版本)。
- 从下面指示的网站或扩展的生物磁共振库 (BMRB) 存储库 (bmrbig102) 下载 NMR 形状的脉冲,到操作 NMR 波谱仪的计算机的目录 /opt/topspin4.1.4/exp/stan/nmr/lists/pp/user。请调整 topspin 版本(此处:topspin 4.1.4 为光谱仪上安装的相应版本)。
- 校准光谱仪和样品。
- 设置感兴趣的温度。对于温度校准,请应用设施的协议。例如,使用甲醇-d4 样品,并使用网络应用程序 NMR 温度计 (https://chem.ch.huji.ac.il/nmr/software/thermometer.html) 解释观察到的化学位移差异;另见77.
- 插入样品(加上额外的 5% D2O 用于锁定)并等待大约 10 分钟,让样品平衡其温度。锁定氘 (2H) 信号(命令: lock,选择 H2O+D2O)。
- 匹配和调整 1H、 15N 和 13C 通道,例如,使用 atma 或 atmm 例程。 如果未安装自动匹配和调整单元,请使用 wobb 命令手动匹配和调整探头。
- 对同质 B0 字段执行匀场,例如,使用 topshim 例程 tg。
- 校准 90° (1H) 脉冲长度。
- 使用 zg 实验。使用 getprosol 导入探测参数。为 P1 选择 5 μs 的短持续时间。如果需要,检查并更正 PLW1 探头的指定功率电平。
- 使用 zg 开始实验。使用 efp 处理数据。使用 调整相位 。pH 值,使基线均匀,并且水共振的 NMR 信号同相。
- 通过取一个公共或近似 90° (1H) 脉冲(例如,根据列出的光谱仪规格)并将其乘以 4 倍来估计 360° (1H) 脉冲。运行 Experiment 并对其进行处理。
注意: 如果信号为正,则所选持续时间太长,如果信号为负,则所选持续时间太短。360° (1H) 脉冲将是过零点。 - 相应地调整脉冲长度。迭代执行此操作以找到过零点。将获得的值除以 4。这将是 90° (1H) 脉冲。
注意:所述 360° 方法的替代方案是 脉冲例程 ,它可以从命令行启动,并允许自动 1H 脉冲校准。
- 将载波频率 O1 校准到水谐振。
- 通过在命令行中键入 edc 将 zg 实验复制到新的实验编号。键入 rpar 以打开参数集(补充图 1A)。
- 搜索参数集 ZGPR。单击参数集 ZGPR (补充图 1A),单击 读取,选择执行“getprosol”,然后单击 确定 (补充图 1B)。
- 将 P1 更改为校准的 90° (1H) 脉冲长度。选择 D1 = 2 s 的饱和长度和 50 dB 的 PLW9 脉冲功率。将接收器增益 (rg) 设置为 16。或者,通过键入 rga 并将接收器增益设置为指定值来执行接收器增益调整(但要避免“接收器增益溢出”)。
- 在命令行中键入 gs 以开始重复、连续的采集。转到采集窗口并更改 O1 以减少以水信号为主的 FID 区域(补充图 2)。
- 单击标记下方可减小 O1,单击标记上方可增加 O1。使用灵敏度按钮更改步长。 保存 当前的 O1;Close 停止重复采集(补充图 2)。
- 校准 90° (15N) 脉冲长度。
- 将 zg 实验复制到新的实验编号(键入 edc)。键入 rpar 读取参数集 HSQCFPF3GPPHWG 将脉冲程序更改为 1 H-15N HSQC 实验。使用 选项保留参数 P1、O1 和 PLW1 (补充图 1)。
- 在命令行中键入 getprosol 1H<校准的 90° (1H) 脉冲长度,以 μs> <90° (1H) 脉冲强度,以 db> 为单位)以导入探头参数。将扫描次数 (NS) 设置为 8,将虚拟扫描 (DS) 设置为 2。
注意: < > 在此处指定要设置的值。例如:getprosol 1H 8.5 -11.79。 - 通过在命令行中键入 zg 来运行实验的第一个增量。将第一个增量复制到 rser 1 <新实验编号>的新 实验编号。使用 efp 处理数据,并使用 对频谱进行相位 。ph.在以下步骤中,使用实验的第一个增量 (1D 实验)。
注意:< >为新实验指定所选数字,例如 rser 1 101。 - 在命令行中键入 ased 以打开 1D 实验的脉冲序列参数。单击 PULPROG 行中的 E(参见补充图 3A)以打开脉冲程序编辑器。
- 使用以下工作流程以新名称保存脉冲序列:单击 File(文件)(左上角)、Save as(另存为),输入新名称,然后单击 OK(确定)。然后单击将 PULPROG 设置为数据集(参见补充图 3B)并单击 OK。
- 通过将 t1 演化期后(回传前)后的 90° (15N) 乘以 2 倍(90° (15N) 脉冲现在是 180° (15N)),修改原来的 1 H-15N HSQC 序列(以新名称保存,参见 2.6.4)。按 Ctrl S 保存脉冲序列。将脉冲长度设置为 90° (15N) (P21),以便第一个增量的 NMR 信号消失(在过零处)(补充图 3B)。
- 打开 1 H-15N HSQC 实验(2D 实验)并输入校准的 90° (15N) 脉冲长度 (P21)。在命令行中键入 getprosol 1H<校准的 90° (1H) 脉冲长度,以 μs 为单位> <90° (1H) 脉冲强度,以 db 为单位> 15N<校准的 90° (15N) 脉冲长度,以 μs 为单位> <90° (15N) 脉冲强度,以 db 为单位> 。
- 确定光谱宽度和采集时间的分配。
- 在 1 H-15N HSQC 实验的采集参数窗口中(在命令行中键入 eda),调整光谱宽度(采集参数 SW)以及直接和间接维度的维度增量数(采集参数 TD)。
- 对于 SW,请选择直接 (1H) 为 16 ppm,间接 (15N) 尺寸为 30 ppm。对于 TD,选择直接 (1H) 为 2048,间接 (1N) 尺寸为 512。
- 获取 1 H-15N HSQC 实验。
- 检查是否检测到所有感兴趣的信号,并且频谱中是否存在混叠伪影。如果光谱中心与沿 15 N 轴的蛋白质信号分布中心不匹配,请更改 O3P。
- 如果光谱窗口太小,请增加 SW 。增加 TD 以获得更高的 FID 分辨率(检查探头规格以了解去耦的最长持续时间)。
注意:这是 NMR 弛豫实验设置的一般设置和起点。建议在整个实验集的开始和结束时记录 1 H-15N HSQC 实验,并且对于更长的实验时间,也要在两者之间记录,以监测样品完整性。
- 在 1 H-15N HSQC 实验的采集参数窗口中(在命令行中键入 eda),调整光谱宽度(采集参数 SW)以及直接和间接维度的维度增量数(采集参数 TD)。
3. 实施 NMR 弛豫实验
注意:NMR 弛豫脉冲序列(图 2)可在 https://www.ipb.hhu.de/en/teams/team-lakomek/pulsesequences 或扩展的生物磁共振库 (BMRB) 存储库 (bmrbig102) 中获得。
- 设置 15编号1ρ 实验。
- 将 HSQC 实验(键入 edc)复制到新目录。键入 ased 并通过单击脉冲序列参数中参数行 PULPROG 的三个点将脉冲程序更改为 15N R1ρ 实验(ased,参见 补充图 3A)。双击脉冲程序,单击 将 PULPROG 设置为数据集(参见 补充图 3B),然后单击 确定。
注意:可能会出现一些错误消息。关闭这些消息。 - 在命令行中键入 gpnam 以插入所有缺失的梯度形状 (gpnam1-gpnam9),如脉冲序列中所述(补充图 4)。
- 在命令行中键入 vplist 。单击三个点(补充图 5A)、 文件 (左上角)和 新建 (补充图 5B)。
- 键入新虚拟 vplist 的名称,然后按 OK。创建虚拟 vplist(5m、65m、25m、45m、15m、55m、35m、10m;每个 vplist 条目的新行)。按 Ctrl S 保存列表并关闭 vplist 窗口(补充图 5C)。
- 双击新创建的 vplist 的名称(补充图 5B)。
注意: 这是避免 pulse sequence 中出现编译错误的必要条件。vplist 的相应条目将在下面进一步确定(第 3.1.29 点)。 - 键入 ased 以打开脉冲序列参数(补充图 6)。输入脉冲序列中给出的所有缺失的 梯度强度 和 梯度长度 (补充图 6D)。
- 在采集参数窗口 (eda) 中为循环计数器 L 输入3(间接维度中的复数点数)15N TD 条目的 1/2。为循环计数器 L6 输入用于拟合松弛曲线的记录的松弛数据点的数量。
注意:这个数字 L6 对应于 vplist 条目的数量,这里:8(补充图 6A)。 - 将从上述网站下载的绝热 TanhTan_half.nl 脉冲设置为 15N SP8 脉冲的形状。将网站上的绝热 TanhTan_2nd.nl 脉冲设置为 15N SP9 脉冲的形状(补充图 6C)。
- 将 P8 条目设置为绝热 TanhTan 脉冲的长度 - 足够长以保证脉冲的绝热性;这里选择 P8 = 3000 μs(补充图 6C)。
- 将 15N decoupling (CPDPRG 3) 设置为 garp decoupling 方案。用于去耦 PCPD3 的长度 200 μs(补充图 6C)。
- 将 interscan recovery delay 设置为至少 D1 = 2 s 或更长。
注意:有关选择合适的恢复延迟,请参阅讨论部分。 - 对于最终实验,将虚拟扫描 (DS) 设置为至少 64(在设置过程中,出于时间原因,DS = 4 就足够了)。作为起点,使用 4 次扫描次数 (NS);如果信噪比 (SNR) 太低,请使用 4 的倍数(补充图 6A)。
- 将 O1 设置为步骤 2.5 的校准载波频率。将 O 2 P 设置为 176 ppm 并从 1 H-15N HSQC 实验中复制 O 3 P(补充图 6A-C)。
- 将脉冲长度 P7 设置为在步骤 2.6 中校准的 90° (15N) 脉冲长度。此外,将步骤 2.6 中 90° (15N) 脉冲的脉冲功率电平复制到 PLW3 和 PLW7(补充图 6C)。
- 将脉冲长度 P1 和 P19 设置为步骤 2.4 中计算的 90° (1H) 脉冲长度。(补充图 6B)。
- 在间接维度 TD = l3 * 2 * l6 中设置增量数(eda 或在命令行中键入 td)。
注意: 此处描述的所有脉冲序列参数都在脉冲序列中额外注释。 - 将形状脉冲 SP5 设置为 I-BURP2 形状 (Iburp2.1000)。将脉冲长度 P15 设置为 2000 μs。通过在 Topspin 脉冲序列参数窗口 (ased) 中单击 I-BURP2 形状脉冲旁边的 E 打开形状工具显示(补充图 6B)。
- 单击 Start NMR Simulation(开始 NMR 模拟 )按钮,以正确的长度模拟形状脉冲。在仿真窗口中检查整形脉冲长度 [μs] (= 2000 μs) 和旋转角度 [°] (= 180°)。单击 启动 NMR-SIM (补充图 7A)。
- 检查激发范围并选择合适的 I-BURP2 脉冲长度,以覆盖蛋白质的 1H 光谱分散,但同时避免水的激发。将 P15 设置为模拟最佳 IBURP-2 脉冲的模拟窗口的整形脉冲长度(补充图 7B)。
注意:如果 I-BURP2 激发(> 95% 的有效性)整个酰胺区域,则选择合适的长度。要模拟不同长度的 I-BURP2 脉冲,请关闭激发曲线窗口,并在仿真窗口中编辑整形脉冲长度。单击 启动 NMR-SIM。长度较短的脉冲具有更宽的 Hz 激励范围。 - 设置 SPOFFS5 以改变 I-BURP2 脉冲的载波频率,并将激励范围向左或向右移动,以避免 I-BURP2 脉冲接触和干扰水磁化(补充图 6B)。
- 要确定整形脉冲的适当功率水平,请打开布鲁克形状工具,单击 “开始 NMR 模拟 ”按钮,然后将 I-BURP2 脉冲长度设置为“整形脉冲长度 [μs]”。请记住软矩形 90° (1H) 脉冲的相应长度,在仿真窗口中相应的 90° 方形脉冲 [μs] 处可见(补充图 7A)。
- 键入 calcpowlev。使用以微秒为单位的硬 90° (1H) 脉冲 (P1) 作为参考长度(补充图 8B),使用对应于 I-BURP2 脉冲(以微秒为单位)的 90° (1H) 软矩形脉冲作为新长度(补充图 8A)。请记住硬 90° (1H) 脉冲和软矩形脉冲之间的功率差 Δ(以 dB 为单位)(补充图 8C)。将以 dB 为单位的硬 90° (1H) 脉冲的脉冲强度复制到 SPW5 并添加以 dB 为单位的记忆差 Δ(补充图 6B)。
- 要确定自旋锁的功率水平,请计算自旋锁的相应 90° (15N) 脉冲长度。使用公式:90° (15N) 脉冲长度,单位为 s = 0.25 /(自旋锁定功率,单位为 Hz)。
- 使用 calcpowlev 计算自旋锁功率与 90° (15N) 硬脉冲 (P7) 之间的功率差 Δ,以 dB 为单位。新:自旋锁的 90° (15N) 脉冲长度,单位为 μs(补充图 8A)。参考:硬 90° (15N) 脉冲长度,单位为 μs(补充图 8B)。将 90° (15N) 硬脉冲(以 dB 为单位)的功率电平 PLW7 复制到自旋锁定功率电平 PLW8(补充图 6C),并添加计算的功率差 Δ(以 dB 为单位)(补充图 8C)。
- 将旋转锁定 PLW8 的功率级别复制到功率级别 SPW8 和 SPW9。(补充图 6C)。
- 要确定 15N 去耦的功率水平,请使用 calcpowlev 计算 90° (15N) 去耦脉冲 (PCPD3) 和 90° (15N) 硬脉冲 (P7) 的功率差 Δ。新:以 μs 为单位的 PCPD3 (200)(补充图 8A)。参考:硬 90° (15N) 脉冲长度,单位为 μs(补充图 8B)。将 90° (15N) 硬脉冲(以 dB 为单位)的功率电平 PLW7 复制到去耦电平 PLW31(补充图 6C),并添加以 dB 为单位的 Δ(补充图 8C)。
- 要使用温度补偿,请包括 #define TEMP_COMPENSATION (通过删除脉冲序列中的分号)。将 P18 设置为 15N R1ρ 实验中使用的自旋锁的最大持续时间。
注意:建议在实验的第一次试验中关闭温度补偿。 - 对于 15N 13C 标记的样品,请包括 # 以 定义脉冲程序中的LABEL_CN(通过删除脉冲序列中的分号)。将 P4 设置为使用78 中的公式计算的脉冲长度。例如,它对应于 600 MHz 时的 23.70 μs 和 1200 MHz 时的 11.85 μs。键入 calcpowlev 以计算 P4 和 (13C) 硬脉冲之间的功率差(以 dB 为单位)。将 (13C) 硬脉冲功率(以 dB 为单位)复制到 PLW4 中,并添加以 dB 为单位的 Δ。将 PLW4 复制到 PLW2(补充图 6B)。
- 确定适当采样的松弛延迟。
- 运行前 8 个 FID(如果 L6 = 8)并处理这些 FID(rser <新实验编号>)。 在 vplist 中选择延迟最长的实验的峰值强度降低到至少 1/e(约 37%)的条目,但与具有最短延迟的实验(用作参考)相比不低于 25%。
- 调整 vplist 的其余条目,以实现延迟最长的 FID 和延迟最短的 FID 之间的强度降低的均匀采样。阅读 NOTE 和探头的电源规格。
注意:缓慢松弛的残基将主导第一个 FID。调整 vplist 的其余条目,以实现第一个和第二个 FID 之间强度降低的均匀采样。不要超过探头允许的最大自旋锁定功率和长度(请参阅探头手册和探头规格)。在温度补偿的情况下,检查 P18(最大自旋锁定持续时间),因为将应用 P18 的总自旋锁定持续时间,在松弛延迟和温度补偿之间分配。它保持时间(弛豫延迟)+ 时间(温度补偿)= const = P18。对于 2 kHz 自旋锁,我们倾向于使用 2 s 的恢复延迟 (D1) ,自旋锁持续时间不超过 65 ms。在这种情况下,必须权衡最大自旋锁持续时间和最长松弛延迟,以支持允许的最大自旋锁持续时间。如果 D1 恢复延迟较长,则可能适用更长的自旋锁定功率。重要的是探针中随时间推移的总功率沉积。如果需要更长的 spin-lock 持续时间,请仅以小步长增加 spin-lock 长度并观察 lock 信号。根据经验,调整锁显示屏上三分之一的锁级别。如果在执行脉冲序列期间锁定电平降低两个以上,则探头中沉积的自旋锁定功率过大。
- 在命令行中键入 rga 以确定接收器增益。为了获得良好的信噪比,请将接收器增益 (rg) 值设在 128 到 256 之间,这证明具有良好的水抑制效果。
注意: 无法达到 128 到 256 之间的接收器增益表示水信号太强。在这种情况下,会轻轻调整梯度功率和持续时间,直到达到接收器增益。另请参阅 讨论 部分。 - 通过在命令行中键入 zg 来开始 15N R1ρ 实验的试运行。检查所有延迟(如果 vplist 中有 8 个延迟,则为 1 到 8)的水信号是否被很好地抑制。另请检查增量 9,这是正交检测方案(回声/反回声)的第二个增量。
- 将 HSQC 实验(键入 edc)复制到新目录。键入 ased 并通过单击脉冲序列参数中参数行 PULPROG 的三个点将脉冲程序更改为 15N R1ρ 实验(ased,参见 补充图 3A)。双击脉冲程序,单击 将 PULPROG 设置为数据集(参见 补充图 3B),然后单击 确定。
- 设置 15N R1 试验。
- 复制 15N R1ρ 实验,并将脉冲程序更改为 15N R1 实验。
- 键入 vclist,生成一个 vclist,然后输入70 中描述的虚拟 vclist:0、24、6、20、4、16、8、12;每个 VCList 条目的新行)。
- 打开 ASED。检查渐变长度和强度。将 GPZ9 更改为 14%。
- 检查脉冲长度、脉冲形状和脉冲强度(正确的值应从 15N R1ρ 实验中复制)。
- 对于 15N13C 标记的样品,请在脉冲程序中使用 #define LABEL_CN。
- 对于温度补偿的使用,包括 #define TEMP_COMPENSATION。
- 运行前 8 个 FID(如果 L6 = 8)并处理这些 (rser < new experiment number>)。在 vclist 中选择延迟时间最长的实验的峰值强度降低到至少 1/e(约 37%)但与延迟最短的实验相比不小于 25% 的条目。调整 vplist 的其余条目,以实现延迟最长的 FID 和延迟最短的 FID 之间的强度降低的均匀采样。
注意:vclist 条目只允许使用偶数。
- 设置 {1H}-15N NOE 实验。
- 复制 15N R1 弛豫实验,并将脉冲序列更改为 {1H}-15N NOE 脉冲方案。
- 使用 Loop 计数器 L8 设置饱和周期的长度。180° (1H) 脉冲之间的间距为 22 ms79 (2* 1 / JNH)。为了获得最准确的结果,请使用大约蛋白质 15N T1(15N) 的 5 倍的饱和期。此处:5 秒。这对应于 L8 = 230。
注:讨论部分提供了有关选择饱和期/恢复延迟的其他建议。 - 按照脉冲方案中的说明更改梯度的长度和强度。使用 sine.20 渐变形状。
- 将间接维度中的增量数设置为等于 TD = l3 * 2 * 2
- 对于 15N13C 标记的样品,使用 #define LABEL_CN 和 -DLABEL_CN 作为 ZGOPTNS。
- 通过在命令行中键入 zg 来运行 pulse 方案。
4. 处理和分析记录的 NMR 实验
注意:光谱已使用 Bruker 系统记录。使用 Unix 或 Linux 操作系统执行处理。使用 NMRPipe80 和 python3 进行光谱处理和数据分析。NMRPipe 软件可在 https://www.ibbr.umd.edu/nmrpipe/index.html 下载。基于 NMRPipe 的处理脚本可从网站下载:https://www.ipb.hhu.de/en/teams/team-lakomek/pulsesequences 或扩展的生物磁共振库 (BMRB) 存储库 (bmrbig102)。建议使用 NMRPipe。如果 NMRPipe 不可用或不需要,则可以使用 CCPN81 或 SPARKY(SPARKY 3 或其后继者 NMRFAM-SPARKY82 或 POKY83 )等替代品。
- 处理和分析 15编号1 实验。
注意: 以下命令在 Unix 终端中执行。- 将下载的目录 R1_processing 中的文件复制到包含实验性 R1 数据的目录(工作目录)。
- 通过终端命令使文件可执行: > chmod u+x *.com
- 执行 > bruker 打开 NMRPipe Conversion Utility 并生成 fid.com 文件。单击 Read Parameters (读取参数)。如果计算了三个维度,请将 Dimension Count (维度计数 ) 更改为 2,然后再次单击 Read Parameters (读取参数 )。然后单击 Clear Script、Update Script 和 Save Script (补充图 9)。
注意: 如果载波频率不正确,请在第二次单击 读取参数 后更改中心位置 PPM。 - 执行 > ./fid.com
- 使用 ms 条目转换 vplist 中的 vclist:延迟(毫秒)= 40 ms * vclist 条目。使用 cp vclist vplist 并编辑 vplist > 。
- 使用 > ./split.com 将伪 3D 数据拆分为 8 个 2D 文件。
- 在 nmrproc.com 中编辑 tauValues (vplist 值 vplist 值) 和 idxExpmt 值 (tauValues 关于时间的顺序) (补充图 10)。
- 使用 > ./nmrproc.com 处理所有 8 个 2D 光谱的原始数据。
- 使用 > nmrDraw 绘制光谱。
- 在这些步骤中使用参考光谱(vplist 中的第一个条目)。通过将 z 维度更改为 1(左上角)来切换到该频谱。使用 Factor (系数 ) 按钮旁边的 + 和 - 调整等值线级别。使用 Draw 按钮重绘频谱。
- 使用一个等值线水平,其中只需来自噪声的少数信号就可以识别所有感兴趣的信号(等值线水平位于噪声水平的边缘)(补充图 11)。
- 校正频谱相位,键入 v 用于垂直 1D 信号投影,键入 h 用于水平 1D 信号投影。用 P0 和 P1 调整相位(补充图 11),记住相位差,并在 nmrproc.com 文件中校正相位(补充图 10)。
注意:如果光谱在 15N 维度上翻转,请删除以下行中的 -neg:| nmrPipe -fn FT -neg \(补充图 10)。 - 关闭 nmrDraw 并使用 > nmrproc.com 再次处理。对于峰拾取(在光谱正确定相并且参考光谱显示所有峰的等值线水平后),键入 K (shift + k),按 Detect(检测),然后单击 Save(保存)。(补充图 11)。
- 将峰列表复制到工作目录,并将 ass.com 中的 -assName 更改为峰列表的名称(补充图 12)。
注:https://www.ipb.hhu.de/en/teams/team-lakomek/pulsesequences 上提供了正确格式的 peaklist 示例
以及扩展的生物磁共振库 (BMRB) 存储库 (bmrbig102)。 - 执行 > ./ass.com 以分配光谱。验证脚本为各种峰建议的分配。使用鼠标左键进行分配,使用鼠标右键取消分配峰。使用 Next 按钮转到下一个氨基酸,使用 Prev 转到上一个氨基酸(补充图 13)。
注意: 可以使用右上角的 + 和 - 更改等值线级别。使用按钮 Draw 以新的等值线级别重新绘制光谱(补充图 13)。 - 完成任务后,单击 Save (保存 ),然后再次单击 Save (保存 )。之后,单击 Quit、Cancel 和 Exit (补充图 13)。
- 执行 > python3 NoneDeleter.py 以创建 relax.tab,其中仅包含有关分配峰的信息。
- 执行 > ./relax.com 以生成 autofit.com 脚本。
- 执行 > ./autoFit.com 执行傅里叶插值并为每个氨基酸生成不同延迟时间的伪比率(生成 axt.tab 和 nlin.tab )。
- 执行 > ./model.com ,根据 nlin.tab 中的数据生成 modelExp.com 脚本。
- 执行 > ./modelExp.com 以使用非线性最小二乘优化模型拟合每个残基的演变曲线。
- 执行 > bash summary.tcl -in relax.tab > t1.tab。忽略潜在的错误消息: GDB Notice: 21 missing values in the table file can occur.
- 将 t1.tab 复制到新创建的目录 R1R2 中。
- 处理和分析 15N R1ρ 实验。
- 将下载目录 R1rho_processing 中的文件复制到包含实验性 R1ρ 数据的目录(工作目录)。
- 执行 > chmod u+x *.com
- 执行 > bruker 打开 NMRPipe Conversion Utility 并生成 fid.com 文件。另请参阅 15N R1 光谱的处理。
- 执行 > ./fid.com
- 执行 > ./split.com 将伪 3D 数据拆分为 8 个 2D 文件。
- 执行 > ./nmrproc.com (事先编辑 tauValues [vplist 值] 和 idxExpmt )以处理所有八个 2D 光谱的原始数据(补充图 10)。
- 执行 > nmrDraw 绘制光谱并选择峰。在这些步骤中使用参考光谱(vplist 中的第一个条目)。遵循与 15N R1 实验相同的程序。
- 将峰列表复制到工作目录,并将 ass.com 中的 -assName 更改为峰列表的名称(补充图 12)。
- 执行 > ./ass.com 并遵循与 15N R1 实验相同的程序。
- 执行 > python3 NoneDeleter.py 以创建 relax.tab,其中仅包含有关已分配峰的信息。
- 执行 > ./relax.com
- 执行 > ./autoFit.com
- 执行 > ./model.com
- 执行 > ./modelExp.com
- 执行 > bash summary.tcl -in relax.tab > t1rho. tab.忽略潜在错误消息: GDB Notice: 21 missing values in the table file may occur.
- 在 t1rho.tab 文件中复制以下标头(值可以从 fid.com 文件中获取)(补充图 14):
备注 T1rho 为 600MHz
备注 -yOBS = 60.818 (在此处输入 fid.com 中的值)
备注 -yCAR = 119.06 (在此处输入 fid.com 中的值) - 执行 > ./extract_PPM.com 生成一个名为 fusionOffset.tab 的表,其中包含峰值 15N 频率相对于载波频率的偏移量(计算从 R1 和 R1ρ 数据中提取 R2 数据的角度 θ 需要)。
- 汇总松弛数据。
- 将 t1rho.tab 和 fusionOffset.tab 复制到 R1R2 目录中。
- 复制 R1R2 目录下的脚本 crtR1p.tcl,调整波谱仪频率和自旋锁功率,执行 bash crtR1p.tcl | sort -n > R1R2.tab。(补充图 15)。 R1R2.tab 将包含拟合的 R1ρ 速率常数、θ 角、R2 速率常数和 R1 速率常数。
- 处理和分析 hetNOE 实验。
- 将下载目录 NOE_processing 中的文件复制到实验性 NOE 数据目录(工作目录)。
- 执行 > chmod u+x *.com
- 执行 > bruker 打开 NMRPipe Conversion Utility 并生成 fid.com 文件。单击 Read Parameters (读取参数)。如果计算了三个维度,请将 Dimension Count 更改为 2,然后再次按 Read Parameters。然后按 清除脚本, 更新脚本及 保存脚本.
- 执行 > ./fid.com
- 执行 > ./split.com (必须先创建包含两个条目 (0, 1) 的 vplist)
- 执行 > ./ft2.com
- 执行 > nmrDraw 绘制光谱并在参考光谱(z 维度 = 1)中选择峰。提取并记住噪声(按 T = shift + t)。相位校正和峰拾取遵循与 15N R1 实验相同的程序。
- 将峰列表复制到工作目录,并将 ass.com 中的 -assName 更改为峰列表的名称(补充图 12)
- 执行 > ./ass.com 以分配光谱。遵循与 15N R1 实验相同的程序。
- 执行 > python3 NoneDeleter.py 创建 relax.tab。
- 执行 > ./script.com
- 执行 > ./autoFit.com
- 更改 error_hetnoe.tcl 中的杂色。
- 执行 > bash error_hetnoe.tcl |sort -n > hetNOE.tab
Access restricted. Please log in or start a trial to view this content.
结果
下面显示了在囊泡 SNARE 蛋白 Synaptobrevin-2 (1-96) 上记录的一些示例 NMR 弛豫数据,通常称为 VAMP2(囊泡相关蛋白 2)。为了记录 NMR 数据,我们在含有 150 mM NaCl、0.1 mM TCEP 和 1 mM EDTA 的 50 mM MES (pH 6.0) 缓冲液中使用了 171 μM 15N Synaptobrevin-2 (1-96) 样品(以下称为 Syb-2)。所有实验数据均在 278.15 K 下使用 250 μL 体积填充在 3 mm NMR 样品管中记录。实验是在配备布鲁...
Access restricted. Please log in or start a trial to view this content.
讨论
该协议描述了 Lakomek 等人 69 和 Stief 等人 70 的 NMR 15N 弛豫实验的设置。我们专注于使用灵敏度增强的 HSQC 检测方案的 NMR 脉冲序列。15N R1 和 R1ρ 实验按照 Stief 等人 70 的详细描述实现,hetNOE 实验由 Lakomek 等人 69 描述。
在设置 NMR 弛豫实验时,
Access restricted. Please log in or start a trial to view this content.
披露声明
作者声明,他们没有已知的竞争性经济利益或个人关系,这些利益或个人关系似乎可能会影响本文报告的工作。
致谢
我们感谢 Melinda Jaspert 和 Kevin Bochinsky 的有益讨论。N.L. 感谢德国科学基金会通过海森堡计划(DFG 资助号 433700474)提供资金。这项工作得到了“COVID-19 发病机制的病毒学和免疫学决定因素 - 为未来大流行做好准备的经验教训”(KA1-Co-02 “COVIPA”)项目的进一步支持,该项目由亥姆霍兹协会的倡议和网络基金提供资助。我们感谢 Jülich-Düsseldorf 生物分子核磁共振中心的慷慨支持,该中心由 Forschungszentrum Jülich 和杜塞尔多夫大学 (HHU) 联合运营。
Access restricted. Please log in or start a trial to view this content.
材料
| Name | Company | Catalog Number | Comments |
| Bruker 600 MHz AVANCE III HD spectrometer | Bruker | https://www.bruker.com/en/products-and-solutions/mr/nmr/avance-nmr-spectrometer.html | NMR experiments conducted |
参考文献
- Sekhar, A., Kay, L. E. An NMR view of protein dynamics in health and disease. Annu Rev Biophys. 48, 297-319 (2019).
- Rosenzweig, R., Kay, L. E. Bringing dynamic molecular machines into focus by methyl-TROSY NMR. Annu Rev Biochem. 83, 291-315 (2014).
- Palmer, A. G. 3rd Enzyme dynamics from NMR spectroscopy. Acc Chem Res. 48 (2), 457-465 (2015).
- Reif, B. Ultra-high resolution in MAS solid-state NMR of perdeuterated proteins: implications for structure and dynamics. J Magn Reson. 216, 1-12 (2012).
- Schanda, P., Ernst, M. Studying dynamics by magic-angle spinning solid-state NMR spectroscopy: Principles and applications to biomolecules. Prog Nucl Magn Reson Spectrosc. 96, 1-46 (2016).
- Le Marchand, T., et al. (1)H-H-detected biomolecular NMR under fast magic-angle spinning. Chem Rev. 122 (10), 9943-10018 (2022).
- Pervushin, K., Riek, R., Wider, G., Wuthrich, K. Attenuated T2 relaxation by mutual cancellation of dipole-dipole coupling and chemical shift anisotropy indicates an avenue to NMR structures of very large biological macromolecules in solution. Proc Natl Acad Sci U S A. 94 (23), 12366-12371 (1997).
- Tzakos, A. G., Grace, C. R., Lukavsky, P. J., Riek, R. NMR techniques for very large proteins and RNAs in solution. Annu Rev Biophys Biomol Struct. 35, 319-342 (2006).
- Sprangers, R., Kay, L. E. Quantitative dynamics and binding studies of the 20S proteasome by NMR. Nature. 445 (7128), 618-622 (2007).
- Wright, P. E., Dyson, H. J. Intrinsically disordered proteins in cellular signalling and regulation. Nat Rev Mol Cell Biol. 16 (1), 18-29 (2015).
- van der Lee, R., et al. Classification of intrinsically disordered regions and proteins. Chem Rev. 114 (13), 6589-6631 (2014).
- Tompa, P. Intrinsically unstructured proteins. Trends Biochem Sci. 27 (10), 527-533 (2002).
- Dyson, H. J., Wright, P. E. Coupling of folding and binding for unstructured proteins. Curr Opin Struct Biol. 12 (1), 54-60 (2002).
- Uversky, V. N. Intrinsically disordered proteins in overcrowded milieu: Membrane-less organelles, phase separation, and intrinsic disorder. Current Opinion in Structural Biology. 44, 18-30 (2017).
- Patel, A., et al. A liquid-to-solid phase transition of the ALS protein FUS accelerated by disease mutation. Cell. 162 (5), 1066-1077 (2015).
- Abyzov, A., Blackledge, M., Zweckstetter, M. Conformational dynamics of intrinsically disordered proteins regulate biomolecular condensate chemistry. Chem Rev. 122 (6), 6719-6748 (2022).
- Uversky, V. N. Unusual biophysics of intrinsically disordered proteins. Biochim Biophys Acta. 1834 (5), 932-951 (2013).
- Papoian, G. A. Proteins with weakly funneled energy landscapes challenge the classical structure-function paradigm. Proc Natl Acad Sci U S A. 105 (38), 14237-14238 (2008).
- Uversky, V. N., Gillespie, J. R., Fink, A. L. Why are "natively unfolded" proteins unstructured under physiologic conditions. Proteins. 41 (3), 415-427 (2000).
- Bah, A., et al. Folding of an intrinsically disordered protein by phosphorylation as a regulatory switch. Nature. 519 (7541), 106-109 (2015).
- Tompa, P., Schad, E., Tantos, A., Kalmar, L. Intrinsically disordered proteins: emerging interaction specialists. Curr Opin Struct Biol. 35, 49-59 (2015).
- Dyson, H. J., Wright, P. E. Perspective: the essential role of NMR in the discovery and characterization of intrinsically disordered proteins. J Biomol NMR. 73 (12), 651-659 (2019).
- Adamski, W., et al. A unified description of intrinsically disordered protein dynamics under physiological conditions using NMR spectroscopy. J Am Chem Soc. 141 (44), 17817-17829 (2019).
- Dobson, C. M. Protein folding and misfolding. Nature. 426 (6968), 884-890 (2003).
- Bertoncini, C. W., et al. Release of long-range tertiary interactions potentiates aggregation of natively unstructured alpha-synuclein. Proc Natl Acad Sci U S A. 102 (5), 1430-1435 (2005).
- Buell, A. K., et al. Solution conditions determine the relative importance of nucleation and growth processes in alpha-synuclein aggregation. Proc Natl Acad Sci U S A. 111 (21), 7671-7676 (2014).
- Salvi, N., Abyzov, A., Blackledge, M. Atomic resolution conformational dynamics of intrinsically disordered proteins from NMR spin relaxation. Prog Nucl Magn Reson Spectrosc. 102, 43-60 (2017).
- Schneider, R., Blackledge, M., Jensen, M. R. Elucidating binding mechanisms and dynamics of intrinsically disordered protein complexes using NMR spectroscopy. Curr Opin Struct Biol. 54, 10-18 (2019).
- Rezaei-Ghaleh, N., Blackledge, M., Zweckstetter, M. Intrinsically disordered proteins: from sequence and conformational properties toward drug discovery. Chembiochem. 13 (7), 930-950 (2012).
- Jensen, M. R., Salmon, L., Nodet, G., Blackledge, M. Defining conformational ensembles of intrinsically disordered and partially folded proteins directly from chemical shifts. J Am Chem Soc. 132 (4), 1270-1272 (2010).
- Camacho-Zarco, A. R., et al. NMR provides unique insight into the functional dynamics and interactions of intrinsically disordered proteins. Chem Rev. 122 (10), 9331-9356 (2022).
- Mittag, T., Forman-Kay, J. D. Atomic-level characterization of disordered protein ensembles. Curr Opin Struct Biol. 17 (1), 3-14 (2007).
- Dyson, H. J., Wright, P. E. Unfolded proteins and protein folding studied by NMR. Chem Rev. 104 (8), 3607-3622 (2004).
- Dyson, H. J., Wright, P. E. NMR illuminates intrinsic disorder. Curr Opin Struct Biol. 70, 44-52 (2021).
- Sugase, K., Dyson, H. J., Wright, P. E. Mechanism of coupled folding and binding of an intrinsically disordered protein. Nature. 447 (7147), 1021-1025 (2007).
- Bessa, L. M., et al. The intrinsically disordered SARS-CoV-2 nucleoprotein in dynamic complex with its viral partner nsp3a. Sci Adv. 8 (3), eabm4034(2022).
- Milles, S., Salvi, N., Blackledge, M., Jensen, M. R. Characterization of intrinsically disordered proteins and their dynamic complexes: From in vitro to cell-like environments. Prog Nucl Magn Reson Spectrosc. 109, 79-100 (2018).
- Salvi, N., Abyzov, A., Blackledge, M. Atomic resolution conformational dynamics of intrinsically disordered proteins from NMR spin relaxation. Prog Nucl Magn Reson Spectrosc. 102 - 103, 43-60 (2017).
- Gill, M. L., Byrd, R. A., Palmer, A. G. III Dynamics of GCN4 facilitate DNA interaction: a model-free analysis of an intrinsically disordered region. Phys Chem Chem Phys. 18 (8), 5839-5849 (2016).
- Bertoncini, C. W., et al. Structural characterization of the intrinsically unfolded protein beta-synuclein, a natural negative regulator of alpha-synuclein aggregation. J Mol Biol. 372 (3), 708-722 (2007).
- Rezaei-Ghaleh, N., et al. Local and global dynamics in intrinsically disordered synuclein. Angew Chem Int Ed Engl. 57 (46), 15262-15266 (2018).
- Rezaei-Ghaleh, N., Parigi, G., Zweckstetter, M. Reorientational dynamics of amyloid-beta from NMR spin relaxation and molecular simulation. J Phys Chem Lett. 10 (12), 3369-3375 (2019).
- Palmer, A. G. NMR characterization of the dynamics of biomacromolecules. Chem Rev. 104 (8), 3623-3640 (2004).
- Palmer, A. G., Massi, F. Characterization of the dynamics of biomacromolecules using rotating-frame spin relaxation NMR spectroscopy. Chem Rev. 106 (5), 1700-1719 (2006).
- Kay, L. E., Torchia, D. A., Bax, A. Backbone dynamics of proteins as studied by 15N inverse detected heteronuclear NMR spectroscopy: application to staphylococcal nuclease. Biochemistry. 28 (23), 8972-8979 (1989).
- Mittermaier, A., Kay, L. E. Review - New tools provide new insights in NMR studies of protein dynamics. Science. 312 (5771), 224-228 (2006).
- Abragam, A. Principles of Nuclear Magnetism. , Clarendon Press. Oxford. (1983).
- Ernst, R. R., Bodenhausen, G., Wokaun, A. Principles of Nuclear Magnetic Resonance in One and Two Dimensions. , Oxford University Press. Oxford. (1990).
- Cavanagh, J., Fairbrother, W. J., Palmer, A. G. III, Skelton, N. J. Protein NMR Spectroscopy: Principles and Practice. , Elsevier, Academic Press. (1995).
- Levitt, M. H. Spin Dynamics: Basics of Nuclear Magnetic Resonance. , John Wiley and Sons. (2008).
- Keeler, J. Understanding NMR Spectroscopy. , John Wiley and Sons. (2010).
- Bloembergen, N., Purcell, E. M., Pound, R. V. Relaxation effects in nuclear magnetic resonance absorption. Phys Rev. 73 (7), 679-712 (1948).
- Wangsness, R. K., Bloch, F. The dynamical theory of nuclear induction. Phys Rev. 89 (4), 728-739 (1953).
- Redfield, A. G. On the theory of relaxation processes. IBM J Res Dev. 1 (1), 19-31 (1957).
- Peng, J. W., Wagner, G. Mapping of the spectral densities of N-H bond motions in eglin c using heteronuclear relaxation experiments. Biochemistry. 31 (36), 8571-8586 (1992).
- Farrow, N. A., et al. Backbone dynamics of a free and a phosphopeptide-complexed src homology-2 domain studied by n-15 NMR relaxation. Biochemistry. 33 (19), 5984-6003 (1994).
- Tolman, J. R., Ruan, K. NMR residual dipolar couplings as probes of biomolecular dynamics. Chem Rev. 106 (5), 1720-1736 (2006).
- Mulder, F. A. A., Skrynnikov, N. R., Hon, B., Dahlquist, F. W., Kay, L. E. Measurement of slow (mu s-ms) time scale dynamics in protein side chains by N-15 relaxation dispersion NMR spectroscopy: Application to Asn and Gln residues in a cavity mutant of T4 lysozyme. J Am Chem Soc. 123 (5), 967-975 (2001).
- Tollinger, M., Skrynnikov, N. R., Mulder, F. A. A., Forman-Kay, J. D., Kay, L. E. Slow dynamics in folded and unfolded states of an SH3 domain. J Am Chem Soc. 123 (46), 11341-11352 (2001).
- Akke, M., Palmer, A. G. Monitoring macromolecular motions on microsecond to millisecond time scales by R(1)rho-R(1) constant relaxation time NMR spectroscopy. J Am Chem Soc. 118 (4), 911-912 (1996).
- Loria, J. P., Rance, M., Palmer, A. G. A relaxation-compensated Carr-Purcell-Meiboom-Gill sequence for characterizing chemical exchange by NMR spectroscopy. J Am Chem Soc. 121 (10), 2331-2332 (1999).
- Vallurupalli, P., Bouvignies, G., Kay, L. E. Studying "Invisible" excited protein states in slow exchange with a major state conformation. J Am Chem Soc. 134 (19), 8148-8161 (2012).
- Jeener, J., Meier, B. H., Bachmann, P., Ernst, R. R. Investigation of exchange processes by 2-dimensional NMR-spectroscopy. J Chem Phys. 71 (11), 4546-4553 (1979).
- Palmer, A. G., Kroenke, C. D., Loria, J. P. Nuclear magnetic resonance methods for quantifying microsecond-to-millisecond motions in biological macromolecules. Methods Enzymol. 339, 204-238 (2001).
- Clore, G. M., Iwahara, J. Theory, practice, and applications of paramagnetic relaxation enhancement for the characterization of transient low-population states of biological macromolecules and their complexes. Chem Rev. 109 (9), 4108-4139 (2009).
- Iwahara, J., Clore, G. M. Detecting transient intermediates in macromolecular binding by paramagnetic NMR. Nature. 440 (7088), 1227-1230 (2006).
- Lakomek, N. A., et al. Residual dipolar couplings as a tool to study molecular recognition of ubiquitin. Biochem Soc Trans. 36 (Pt 6), 1433-1437 (2008).
- Johnson, C. N., Libich, D. S. Paramagnetic relaxation enhancement for detecting and characterizing self-associations of intrinsically disordered proteins. J Vis Exp. (175), e63057(2021).
- Lakomek, N. A., Ying, J., Bax, A. Measurement of (1)(5)N relaxation rates in perdeuterated proteins by TROSY-based methods. J Biomol NMR. 53 (3), 209-221 (2012).
- Stief, T., Vormann, K., Lakomek, N. A. Sensitivity-enhanced NMR (15)N R(1) and R(1rho) relaxation experiments for the investigation of intrinsically disordered proteins at high magnetic fields. Methods. 223, 1-15 (2024).
- Kay, L. E., Keifer, P., Saarinen, T. Pure absorption gradient enhanced heteronuclear single quantum correlation spectroscopy with improved sensitivity. J Am Chem Soc. 114 (26), 10663-10665 (1992).
- Geen, H., Freeman, R. Band-selective radiofrequency pulses. J Magn Reson (1969). 93 (1), 93-141 (1991).
- Desvaux, H., Berthault, P. Study of dynamic processes in liquids using off-resonance RF irradiation. Prog Nucl Magn Reson Spectrosc. 35 (4), 295-340 (1999).
- Overhauser, A. W. Polarization of nuclei in metals. Phys Rev. 91 (2), 476-476 (1953).
- Overhauser, A. W. Polarization of nuclei in metals. Phys Rev. 92 (2), 411-415 (1953).
- Kumari, P., Frey, L., Sobol, A., Lakomek, N. A., Riek, R. (15)N transverse relaxation measurements for the characterization of micros-ms dynamics are deteriorated by the deuterium isotope effect on (15)N resulting from solvent exchange. J Biomol NMR. 72 (3-4), 125-137 (2018).
- Karschin, N., Krenek, S., Heyer, D., Griesinger, C. Extension and improvement of the methanol-d(4) NMR thermometer calibration. Magn Reson Chem. 60 (4), 203-209 (2022).
- Sattler, M., Schleucher, J., Griesinger, C. Heteronuclear multidimensional NMR experiments for the structure determination of proteins in solution employing pulsed field gradients. Prog Nucl Magn Reson Spectrosc. 34 (2), 93-158 (1999).
- Ferrage, F., Cowburn, D., Ghose, R. Accurate sampling of high-frequency motions in proteins by steady-state N-{H} nuclear Overhauser effect measurements in the presence of cross-correlated relaxation. J Am Chem Soc. 131 (17), 6048-6049 (2009).
- Delaglio, F., et al. NMRPipe: a multidimensional spectral processing system based on UNIX pipes. J Biomol NMR. 6 (3), 277-293 (1995).
- Skinner, S. P., et al. CcpNmr AnalysisAssign: a flexible platform for integrated NMR analysis. J Biomol NMR. 66 (2), 111-124 (2016).
- Lee, W., Tonelli, M., Markley, J. L. NMRFAM-SPARKY: enhanced software for biomolecular NMR spectroscopy. Bioinformatics. 31 (8), 1325-1327 (2015).
- Lee, W., Rahimi, M., Lee, Y., Chiu, A. POKY: a software suite for multidimensional NMR and 3D structure calculation of biomolecules. Bioinformatics. 37 (18), 3041-3042 (2021).
- Abyzov, A., et al. Identification of dynamic modes in an intrinsically disordered protein using temperature-dependent NMR relaxation. J Am Chem Soc. 138 (19), 6240-6251 (2016).
- Singh, A., Purslow, J. A., Venditti, V. 15N CPMG relaxation dispersion for the investigation of protein conformational dynamics on the micros-ms timescale. J Vis Exp. (170), e62395(2021).
- Gopalan, A. B., Hansen, D. F., Vallurupalli, P. CPMG experiments for protein minor conformer structure determination. Methods Mol Biol. 1688, 223-242 (2018).
- Tugarinov, V., Kay, L. E. Methyl groups as probes of structure and dynamics in NMR studies of high-molecular-weight proteins. Chembiochem. 6 (9), 1567-1577 (2005).
- Lipari, G., Szabo, A. Model-free approach to the interpretation of nuclear magnetic resonance relaxation in macromolecules. 1. Theory and range of validity. J Am Chem Soc. 104 (17), 4546-4559 (1982).
- Lipari, G., Szabo, A. Model-Free Approach to the interpretation of nuclear magnetic-resonance relaxation in macromolecules .2. Analysis of experimental results. J Am Chem Soc. 104 (17), 4559-4570 (1982).
- Clore, G. M., et al. Deviations from the simple two-parameter model-free approach to the interpretation of nitrogen-15 nuclear magnetic relaxation of proteins. J Am Chem Soc. 112 (12), 4989-4991 (1990).
- Lemaster, D. M. Larmor frequency selective model free analysis of protein NMR relaxation. J Biomol NMR. 6 (4), 366-374 (1995).
- Peng, J. W., Wagner, G. Mapping of spectral density functions using heteronuclear NMR relaxation measurements. J Magn Reson (1969). 98 (2), 308-332 (1992).
- Farrow, N. A., Zhang, O. W., FormanKay, J. D., Kay, L. E. Characterization of the backbone dynamics of folded and denatured states of an SH3 domain. Biochemistry. 36 (9), 2390-2402 (1997).
- Khan, S. N., et al. Distribution of pico- and nanosecond motions in disordered proteins from nuclear spin relaxation. Biophys J. 109 (5), 988-999 (2015).
- Smith, A. A., Ernst, M., Meier, B. H., Ferrage, F. Reducing bias in the analysis of solution-state NMR data with dynamics detectors. J Chem Phys. 151 (3), 034102(2019).
- Zumpfe, K., Smith, A. A. Model-free or not. Front Mol Biosci. 8, 727553(2021).
- Robustelli, P., Trbovic, N., Friesner, R. A., Palmer, A. G. Conformational dynamics of the partially disordered yeast transcription factor GCN4. J Chem Theory Comput. 9 (11), 5190-5200 (2013).
- Salvi, N., Abyzov, A., Blackledge, M. Multi-timescale dynamics in intrinsically disordered proteins from NMR relaxation and molecular simulation. J Phys Chem Lett. 7 (13), 2483-2489 (2016).
Access restricted. Please log in or start a trial to view this content.
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。