このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
タンパク質のピコ秒からナノ秒の構造ダイナミクスを調べるためのNMR 15N緩和実験
要約
核磁気共鳴(NMR)分光法は、構造タンパク質のダイナミクスを残留物特異的な方法で特徴付けることができます。私たちは、ピコ秒からナノ秒の時間スケールに敏感なNMR 15N R1 およびR2 緩和、および{1H}-15Nヘテロ核オーバーハウザー効果(hetNOE)実験を記録するための実践的なプロトコルを提供します。
要約
核磁気共鳴(NMR)分光法により、溶液中および生理学的温度下でタンパク質を研究できます。多くの場合、タンパク質骨格のアミド基または側鎖のメチル基のいずれかが、タンパク質の構造ダイナミクスのレポーターとして使用されます。 15N標識および完全プロトン化サンプル上の球状タンパク質のタンパク質骨格の構造動力学研究は、通常、分子量が最大50 kDaのタンパク質に対して良好に機能します。横方向緩和最適化分光法(TROSY)と組み合わせた側鎖重水素化を適用すると、球状タンパク質では最大200 kDaまで、側鎖に焦点が当てられている場合は最大1 MDaまでこの制限を延長できます。天然変性タンパク質(IDP)または天然変性領域(IDR)を持つタンパク質を調査する場合、これらの重量制限は適用されませんが、それをはるかに超える可能性があります。その理由は、内部の柔軟性が高いという特徴を持つ IDP または IDR は、頻繁に動的に分離されるためです。さまざまなNMR法により、ピコ秒から数時間までの幅広い時間スケールで構造タンパク質のダイナミクスを原子分解能で把握することができます。標準的な 15Nリラクゼーション測定は、タンパク質の内部柔軟性を概観し、ピコ秒からナノ秒の高速タイムスケールで経験するタンパク質骨格のダイナミクスを特徴付けます。この記事では、NMR 15N R1、R2、およびヘテロ核オーバーハウザー効果(hetNOE)実験をセットアップおよび記録するための実践的なプロトコルを紹介します。私たちは、より高度な分析の前に、例示的なデータを示し、それらを単純に定性的に解釈する方法を説明します。
概要
タンパク質の機能は、その三次元構造だけでなく、タンパク質が採用する異なるコンフォメーション間の内部柔軟性と構造遷移を含む構造ダイナミクスによっても決定されます。核磁気共鳴(NMR)分光法は、溶液1,2,3中のタンパク質の構造ダイナミクスを調べることができます。プロトン検出固体NMRの最近の開発により、例えば脂質二重膜4,5,6のような難溶性状態でのタンパク質ダイナミクスの特性評価も可能になりました。溶液NMRでは、タンパク質骨格とタンパク質側鎖の構造動態を調べることができます。球状タンパク質の場合、タンパク質が15N同位体標識されると、タンパク質骨格の構造ダイナミクス研究を最大50 kDaで達成できます。側鎖重水素化および横緩和最適化分光法(TROSY)を使用する場合、この制限は最大200 kDaまで拡張できます7,8。側鎖ダイナミクスに焦点が当てられると、アクセス可能なタンパク質と複合体の範囲を最大1 MDa 2,9まで拡張できます。
名前付きの重み制限は、高い内因性ダイナミクスを頻繁に示す天然変性タンパク質(IDP)には適用されません。真核生物のプロテオームの30%以上は、IDPまたは天然変性領域(IDR)10,11,12,13で構成されています。それらは、シグナル伝達や転写1などの多くの細胞プロセスで中心的な役割を果たし、細胞内相分離14,15,16,17に頻繁に関与しています。IDPは、生理学的条件下で明確に定義された3次元(3D)ネイティブ構造を欠いており、弱く漏斗状または起伏のあるエネルギーランドスケープを持っています17,18。IDPまたはIDRのバックボーンに分布する低い疎水性と強い静電反発力のために、剛性構造に折り畳むための駆動力が欠けています19。IDPは、他の結合パートナーと複合体を形成するときに、折り畳まれたコンフォメーションを頻繁に採用する10,20,21。また、翻訳後修飾(PTM)は、IDPまたはIDRのフォールディングの可能性を広げる22,23。IDPのミスフォールディングは、神経変性疾患15,24,25,26を含むさまざまな疾患の原因として特定されています。
IDPとIDRは高い内部柔軟性を示しています21,27,28。原子位置と二面角の変化を示すコンフォメーションアンサンブルは、分子動力学シミュレーションと実験データ29,30,31,32から得られた拘束から導き出された。凍結状態ではダイナミクスとそれに伴う乱れにより、拡散電子密度により、クライオ電子顕微鏡やX線結晶構造解析などの構造生物学の最先端の方法を使用してそれらを構造的に特徴付けることが困難になります。また、極低温での実験のための結晶化条件やサンプル調製技術は、IDPが経験するコンフォメーション空間に影響を与える可能性があります。しかし、溶液NMRは高ダイナミックなタンパク質にうまく機能するため、IDP16、20、22、28、29、30、31、32、33、34、35、36、37、38の研究に適しています。
上記で紹介したように、溶液NMRは、主にスピン緩和31,33,38,39,40,41,42に基づいて、広範囲の時間スケールで内部タンパク質のダイナミクスを研究するためのさまざまな手法を提供します(図1)。
タンパク質骨格のアミド基における15N核のスピン緩和は、内部タンパク質のダイナミクスと集団運動(関連する場合は回転拡散を含む)による1 H-15N結合角の配向変化によって誘導されます27,43,44,45,46,47,48,49,50,51.回転相関時間τR(分子が1つの放射体を回転させる必要がある時間、全体のタンブリング相関時間とも呼ばれる)よりも短い時間スケールでは、化学シフト異方性(CSA)と双極子結合(D)はアクティブであり、タンパク質の回転拡散によって平均化されません。結合角の変動、結合の再配向、および回転タンブリングを含むタンパク質骨格の内部ダイナミクスは、CSAと双極子結合テンソルの確率的揺らぎを誘発し、局所磁場の変動をもたらし、最終的にNMRスピン緩和47,48,52,53をもたらす.これらの変動は、全体的な相関関数で表すことができます。全体の相関関数のフーリエ変換は、スペクトル密度関数と呼ばれます。半古典的レッドフィールド緩和理論では、NMR緩和速度定数は、これらのスペクトル密度関数の線形結合によって記述することができる54。
1990年代初頭に開発されたバックボーン15N NMR緩和実験は、15N R1、R1ρ、および{1H}-15N核オーバーハウザー効果実験を含み、タンパク質45,55,56,57の回転相関時間τRよりも速い高速ピコ秒(ps)ナノ秒(ns)の時間スケールに敏感です。回転相関時間τRよりも遅いバックボーンダイナミクスを特徴付けるために、いわゆる緩和分散実験、R1ρ、およびマイクロ秒(μs)-ミリ秒(ms)ダイナミクス44,46,58,59,60,61に敏感なCarr-Purcell-Meiboom-Gill(CPMG)実験が使用されます。マイクロ秒より遅いダイナミクスは、15N化学交換飽和移動(CEST)NMR62、交換分光法(EXSY、ミリ秒から秒)、またはリアルタイム(RT)NMR(秒から時間)63,64によって捕捉することができる。常磁性プローブのPRE(常磁性緩和強化)効果、および残留双極子カップリング(RDC)を使用して、psからmsのダイナミクス65,66,67,68の全範囲を評価できます。
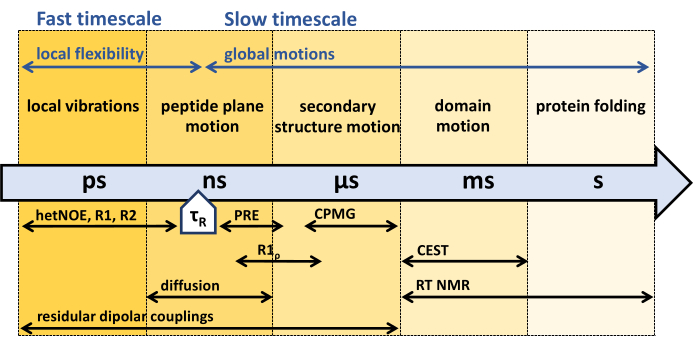
図1:タンパク質骨格ダイナミクスの時間スケールと、さまざまなNMRダイナミクス実験の感度の高い時間枠。 NMRは、幅広い時間スケールでタンパク質骨格のダイナミクスを特徴付けるためのさまざまな方法を提供します。タンパク質骨格が経験するさまざまな動きが、それぞれの時間スケールで示されます。タンパク質の回転相関時間τRは、タンパク質が全体の回転(1ラジアント分)に必要な時間です。タンパク質の回転相関時間τRよりも速い運動は、タンパク質の内部の柔軟性と関連している可能性があります。さまざまなNMR実験とそれぞれの時間スケールに対する感度は、矢印の下に示されています。 この図の拡大版を表示するには、ここをクリックしてください。
以下のプロトコルは、Lakomek et al.69 およびStief et al.70による、感度向上型異種核単一量子コヒーレンス(HSQC)検出スキームを用いたNMR緩和実験のセットアップについて説明する。実験の実施に進む前に、NMRスピン緩和とNMR緩和実験の非常に簡単な概要を説明します。サイズの制限と、このプロトコルを理解できるようにするために、この概要は単純化する必要があります (したがって不完全です)。
T1時間またはR1 = 1 / T1レート定数によって特徴付けられる縦方向またはスピン格子緩和は、磁化がボルツマン平衡に戻ることを説明しています。平衡状態では、磁化は外部磁場の軸に沿って整列し、実験室フレームのz軸を定義します。高(1H)および小(15N)のLarmor周波数(NMR共振周波数、たとえば、14.1テスラ磁石の1Hで600 MHz)でのスペクトル密度と、これらのLarmor周波数の線形の組み合わせは、rad·s-1で測定された15N R1レート定数によって特徴付けられる15N R1緩和に寄与します.タイムスケール上の運動は、これらのラーマー周波数に反比例します。したがって、ピコ秒からナノ秒の時間スケールでの動きは、緩和速度定数R1に寄与します。全体的なタンブリングを示し、回転相関時間を定義できる分子の場合、R1 (T1) 曲線は ωτR = 1 の最大 (最小) を示し、回転相関 τR とスピンのラーマー周波数 ω を考慮します。複数のLarmor周波数が寄与する場合、周波数が最も低い周波数が支配的な周波数になります(たとえば、15N R1の場合はωN)。高速運動領域(ωτRは1よりはるかに小さい)は、非常に速く転倒する小分子に適用され、低磁場と低粘度に適用されます。スローモーション領域(ωτRは1よりはるかに大きい)は、ゆっくりと転がる大きな分子や、高磁場、高粘度に有効です。
球状折り畳まれたタンパク質は、溶液中で全体的にタンブリングを示し、回転相関時間を割り当てることができます。しかし、全体的なタンブリングの概念は、天然変性タンパク質にはもはや有効ではなく、単一の回転相関時間を割り当てることとは異なることがよくあります。ここでは、残基特異的な内部相関時間がより重要になります。
15N R1緩和率を測定する記載のパルスシーケンス(図2)は、直交検出69,70,71のためのエコー/アンチエコー検出を用いた感度向上HSQC読み出し実験に基づいています。可変強度および長さを有する短いグラジエントは、コヒーレンス選択および改善された水抑制70に使用される。その間、15Nの縦偏光は緩和されます。減衰時間が長くなると、この疑似 3D スペクトルの関連する 2D 平面の強度が低下します (遅延データ ポイントは 3 次元に記録されます)。以下で説明するループ要素は、緩和時間が長くなるほど実行回数が増えます。15Nの化学シフト異方性(CSA)と1Hおよび15Nの双極子結合(D)との間の相互相関緩和も緩和遅延中にアクティブであるため、アミドプロトンに選択的な中央のI-BURP-2 180°パルス72は、相互相関緩和による寄与を再焦点化する必要があります(これは、再焦点を合わせない場合、歪んだ誤った15NR1レート定数につながります)。
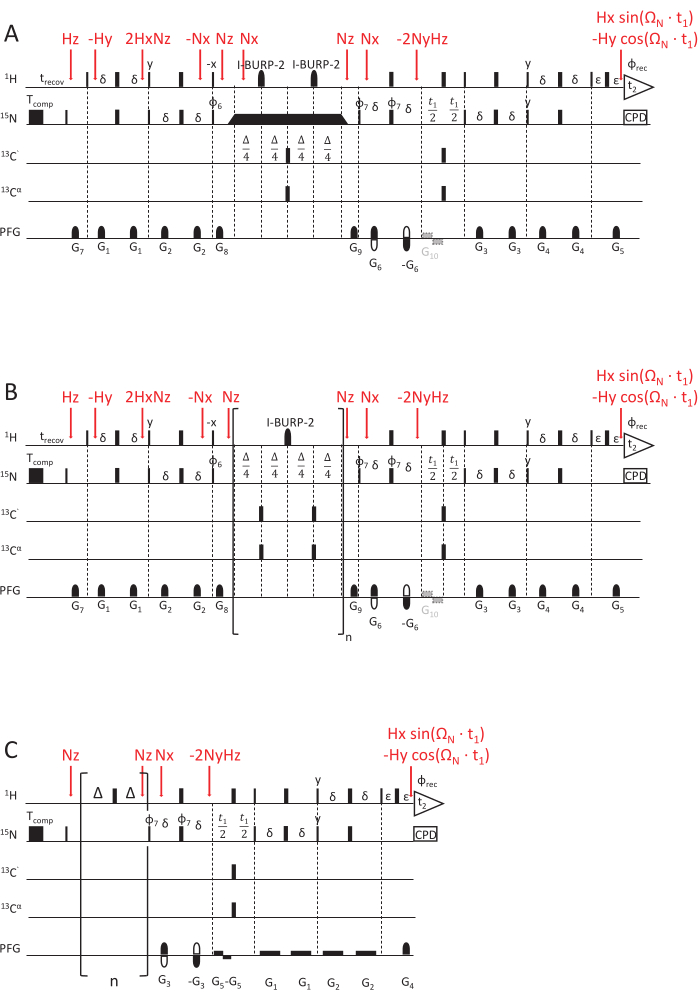
図2:NMR緩和速度定数を決定するためのNMRパルスシーケンススキーム。 (A)15N R1ρ、(B)15N R1、および(C)hetNOE実験、感度向上HSQC読み出しスキーム69,70を使用。90°(x)パルスは幅の狭い長方形で視覚化され、180°(x)パルスは幅の広い長方形で視覚化されます(特に明記されていない限り)。次の位相サイクルが適用されます:φ6 = y、y、-y、-y;φ7 = y、-y、φrec = y、-y、y、y。直交検出は、グラジエントG5の極性とφ7の位相サイクルを反転させることで実現します(エコー/アンチエコー検出)。(A)15N R1ρ実験:黒い長方形はスピンロックを表し、持続時間は異なる緩和遅延を獲得するために変化します。スピンロックの前後の三角形は、有効磁場軸Beffに沿って磁化を整列させる断熱形状パルスを示しています。G10は、進化段階での水磁化の放射減衰を防ぐためのオプションの勾配です。(B)15N R1実験:括弧で囲まれた部分は、シーケンスのループ要素を示しており、所望の緩和遅延に一致するようにn回繰り返されます。(C)hetNOEパルス方式は、R1およびR1ρパルス方式の後半、すなわちt1 evolution timeおよびHSQC検出素子と類似しています。ただし、15Nの磁化はINEPTなしで直接励起されます。陽子磁化の飽和(1Hと15Nの間の交差緩和を達成するため)は、少なくとも5秒間印加された180(1H)パルスの列によって達成されます。同じ長さ(ここでは5秒)のパルス列のないアイドル遅延が参照実験に適用されます。G5は輻射減衰を防止するためのオプションのグラジエントであり、グラジエントG4の極性の反転は、位相φ7=y、-y、-y、yとの組み合わせにより、直交検出を実現します。製品オペレーターによって表される磁化転写ステップは、赤でマークされています。この図の拡大版を表示するには、ここをクリックしてください。
緩和速度定数R2は、スピン間の位相コヒーレンスの喪失による横分極(外部磁場に直交するxy面内)の緩和を表し、検出可能な磁化53,54の減衰につながる。高周波数と低周波数でのスペクトル密度関数は、R1と同様にR2に寄与します。ただし、R2 に最も大きく寄与するのは、ゼロ周波数でのスペクトル密度です。このため、R2は回転タンブリングに非常に敏感であり、回転相関時間τRで表され、室温での小さな球状タンパク質の場合、数nsのオーダーです。したがって、数百psから低ns領域の遅いバックボーンの動きが最も寄与します。15N原子核の化学シフトテンソルの等方性部分の変調を引き起こすバックボーンの交換ダイナミクスは、原則として、R2速度定数43,44,49,60,61に交換寄与R2exを追加します。説明された実験では、R2exの寄与は、スピンロックの逆円周波数よりも遅いダイナミクスを再焦点化するスピンロックによって抑制されます。スピンロックは、磁化を有効磁場軸Beff(スピンロックω1フィールドと15Nキャリア周波数からの化学シフトオフセットのベクトル和(下記参照))に沿って整列させる長波無線周波パルスです。B1,eff軸に沿って整列した磁化の緩和をR1ρ緩和と呼び、R1成分とR2成分があります。式(1)を使用すると、R2はR1ρとR144,73から計算できます。
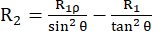 (1).
(1).
実効磁場Beffと外部磁場B0の軸との間の角度は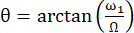 です。 ω1はスピンロックのRF振幅であり、対応する残基の15N化学シフトと15Nキャリア周波数44,73との間の化学シフトオフセットΩです。
です。 ω1はスピンロックのRF振幅であり、対応する残基の15N化学シフトと15Nキャリア周波数44,73との間の化学シフトオフセットΩです。
R1ρ パルス方式(図2A、 70)は、緩和遅延を除いて 15NのR1 方式と非常によく似ています。 15N R1ρ 緩和率を測定するには、スピンロックと同じ無線周波数(RF)振幅の断熱パルスによって磁化が有効磁場軸Beff に沿って整列した後、スピンロックがアクティブになる必要があります。スピンロックの長さは、異なる緩和遅延を得るために変化します。
定常状態{1H}-15N核オーバーハウザー効果(1 H-15N NOE)は、以下でhetNOEと呼ばれ、交差緩和率と15Nの縦緩和率の比です。これは、陽子分極45,53,54,74,75の飽和時に陽子との交差緩和により、15Nの定常分極の減少につながります。交差緩和は、1H と 15N の Larmor 周波数の合計と差のスペクトル密度関数に依存します。したがって、hetNOEは、高速ピコ秒ダイナミクス(< 100ps)とps-nsダイナミクス(R1依存性による)の両方に敏感です。シーケンス69(図2C)は、直交検出用のエコー/アンチエコー勾配を使用した感度向上HSQC読み出しに基づいています。陽子磁化とその結果生じるhetNOEの飽和のために、平衡陽子磁化は反転され、続いて15N T1の約5倍の180°パルスを急速にパルスすることによって飽和します。参照実験では、回復遅延は飽和遅延と等しくなりますが、1H 180°パルス列はありません。参照実験と飽和時間が1Hの実験には、D1 = 2秒の遅延が追加されます。どちらの実験も連続して記録され、1H180°パルス(飽和)を適用するかどうか(参照)のみが異なります。実験で記録されたスペクトル強度と 1H 飽和の比を参照実験の強度 (180° 陽子パルス列なし) で割ると、{1H}-15N NOE (hetNOE) 値が得られます。
以下のプロトコルは、Lakomekら69およびStiefら70によるNMR緩和実験の設定を説明しています。私たちは、感度を向上させたHSQC検出スキームを用いたNMRパルス配列に焦点を当てています。15N R1およびR1ρ実験は、Stief et al.70によって詳細に記述されているように実施されており、hetNOE実験はLakomek et al.69によって記述されている。
Access restricted. Please log in or start a trial to view this content.
プロトコル
1. NMRサンプル調製
注:タンパク質の同位体標識は、高次元NMRおよび高度なNMR実験のために行われます。 大腸菌 でのタンパク質発現およびタンパク質精製が、リッチ培地(Luria-Bertani [LB]や2x 酵母抽出物トリプトン培地[2YT]など)を用いて数ミリグラム/リットルの収量で確立されていた場合、同位体標識NMRサンプルの調製は通常比較的簡単です。
- 同位体標識には、 15N塩化アンモニウムが唯一の窒素源であり、 13Cが高濃度グルコースが唯一の炭素源である遺伝子発現にM9最小培地を使用します。
- タンパク質の精製には、それぞれのタンパク質について実験室で確立された標準プロトコルに従ってください。
注:結果セクションは、IDP、単量体SNAREタンパク質Synaptobrevin-2(1-96)の代表的な結果を示しています。シナプトブレビン-2のNMR同位体標識は、70に記載されているように行われてきた。非常に高精度な測定を行うには、低濃度のD2O(5%未満)を使用することをお勧めします。これは重水素同位体効果によるもので、これはHahn-Echo 15N T2 および低周波 15N CPMG緩和分散実験(ここでは議論しない)で観察できるが、 15N R1ρ 実験(主に再焦点化される)では最小限の影響しか持たない76。
2. 分光器上でのNMR緩和実験の実施準備
注:記載されているNMR緩和実験は、ブルカー分光器に特有のものです。これらの製品は、極低温および室温の 1H、 15N、 13C 三重共鳴プローブ、および Bruker のソフトウェア Topsin 3.6 以降で操作する Avance III および Avance Neo コンソールでテストされています。
- 注意事項に従ってください。
- NMR分光計で働く前に、安全ブリーフィングを行ってください。
- 使用した分光器のプローブのパワー仕様を確認し、Topspinの「prosol」テーブルエントリとの一貫性を比較するなどして、各パルスに適切なパルス長とパルスパワーを選択します。
- パルスシーケンスをインポートします。
- NMRパルスプログラムは、以下に示すウェブサイトから、または拡張されたBiological Magnetic Resonance Bank(BMRB)リポジトリ(bmrbig102)から、NMR分光計を操作しているコンピューターのディレクトリ/opt/topspin4.1.4/exp/stan/nmr/lists/pp/userにダウンロードしてください。topspinのバージョンを調整してください(ここでは、topspin4.1.4を分光器にインストールされているそれぞれのバージョンに合わせます)。
- NMR形状パルスは、以下に示すウェブサイトから、または拡張されたBiological Magnetic Resonance Bank(BMRB)リポジトリ(bmrbig102)から、NMR分光計を操作するコンピューターのディレクトリ/opt/topspin4.1.4/exp/stan/nmr/lists/pp/userにダウンロードしてください。topspinのバージョンを調整してください(ここでは、topspin 4.1.4を分光計にインストールされているそれぞれのバージョンに合わせます)。
- 分光計とサンプルを校正します。
- 対象の温度を設定します。温度校正については、施設のプロトコルを適用してください。例えば、メタノール-d4サンプルを使用し、ウェブアプリケーションの NMR温度計 (https://chem.ch.huji.ac.il/nmr/software/thermometer.html)を使用して観察された化学シフト差を解釈します。77も参照してください。
- サンプル(およびロック用に5% D2Oを追加)を挿入し、サンプルの温度が平衡化するまで約10分間待ちます。重水素(2H)信号をロックします(コマンド: ロック、H2O + D2Oを選択)。
- 1H、15N、13Cチャネルを一致させ、調整します(例:atmaまたはatmmルーチンを使用)。 自動マッチングおよびチューニングユニットが取り付けられていない場合は、wobbコマンドを使用してプローブを手動でマッチングおよびチューニングします。
- 均質なB0 フィールドに対してシミングを実行します(例:topshimルーチンtgを使用)。
- 90°(1H)のパルス長を校正します。
- zg 実験を使用します。 getprosol を使用してプローブ パラメーターをインポートします。P1には5μsの短い持続時間を選択します。必要に応じて、PLW1のプローブの指定された電力レベルを確認して修正します。
- zgで実験を開始します。efp でデータを処理します。で位相を調整します。ベースラインが均一で、水共鳴のNMR信号が同相になるようにph。
- 360°(1H)パルスを推定するには、一般的なまたは約90°(1H)のパルス(たとえば、リストされた分光器の仕様から)を取り、それに4を掛けます。実験を実行し、処理します。
注意: 信号が正の場合は選択した持続時間が長すぎ、負の場合は短すぎます。360°(1H)パルスがゼロクロッシングになります。 - それに応じてパルス長を調整します。これを繰り返して、ゼロクロッシングを見つけます。取得した値を4で割ります。これが 90° (1H) パルスになります。
注:説明されている360°メソッドの代替は、コマンドラインから開始でき、自動1Hパルスキャリブレーションを可能にするパルサルルーチンです。
- キャリア周波数O1を水の共振に校正します。
- コマンドラインに 「edc 」と入力して、zg experiment を新しい実験番号にコピーします。 「rpar 」と入力して、パラメーター・セットを開きます (補足図 1A)。
- パラメータ・セット ZGPR を検索します。パラメータセット ZGPR (補足図1A)をクリックし、[ 読み取り]をクリックし、["getprosol"の実行]を選択して [OK ]をクリックします(補足図1B)。
- P1を校正済みの90°(1H)パルス長に変更します。飽和長を D1 = 2 秒、パルス電力を PLW9 に 50 dB に選択します。レシーバーゲイン(rg)を16に設定します。または、 rga と入力し、レシーバーゲインを指定された値に設定して、レシーバーゲイン調整を実行します(ただし、「レシーバーゲインオーバーフロー」は避けてください)。
- コマンドラインに gs と入力すると、連続的な集録が繰り返されます。集録ウィンドウに移動し、O1を変更して、水信号が支配するFID領域を減らします(補足図2)。
- マーカーの下をクリックしてO1を減らし、マーカーの上をクリックして増やします。感度ボタンを使用して、ステップサイズを変更します。現在のO1 を保存します 。「閉じる」は、繰り返しの収集を停止します(補足図2)。
- 90°(15N)のパルス長を校正します。
- zg experiment を新しい実験番号 ( edc と入力) にコピーします。 rpar と入力してパラメータセットを読み取り HSQCFPF3GPPHWG パルスプログラムを 1 H-15N HSQC実験に変更します。 「パラメータ P1、O1、および PLW1 を保持する 」オプションを使用します (補足図 1)。
- コマンドラインに getprosol 1H 1H) pulse length in μs> <90° (1H) pulse strength in db> と入力して、プローブパラメータをインポートします。スキャン数 (NS) を 8 に、ダミー スキャン数 (DS) を 2 に設定します。
注: < > ここで設定する値を指定します。たとえば、getprosol 1H 8.5 -11.79 です。 - 実験の最初のインクリメントを実行するには、コマンド ラインに 「zg 」と入力します。最初の増分を rser 1 <新しい実験番号> の新しい実験番号にコピーします。 データを efp で処理し、 でスペクトルを位相化します 。ph.次の手順では、実験の最初の増分 (1D 実験) を使用します。
注:< >は、新しい実験に選択された番号を指定します(例:rser 1 101)。 - コマンドラインにasedと入力して、1D実験のパルスシーケンスパラメータを開きます。PULPROG行のE(補足図3Aを参照)をクリックして、パルスプログラムエディターを開きます。
- 次のワークフローを使用して、パルス シーケンスを新しい名前で保存します: [ファイル ] (左上隅)、[ 名前を付けて保存] の順にクリックし、新しい名前を入力して [OK] をクリックします。次に、「 PULPROG を データ・セットに設定」をクリックし ( 補足図 3B を参照)、「 OK」をクリックします。
- 元の1 H-15N HSQCシーケンス(新しい名前で保存、2.6.4.を参照)を、t1進化期間後(バックトランスファー前)の90°(15N)に2を掛けて変更します(90°(15N)パルスは180°(15N)になります)。Ctrl Sを押して、パルスシーケンスを保存します。パルス長を90°(15N)(P21)に設定して、最初の増分のNMR信号が(ゼロクロッシングで)消えるようにします(補足図3B)。
- 1 H-15N HSQC実験(2D実験)を開き、校正済みの90°(15N)パルス長(P21)を入力します。コマンドラインで「getprosol 1H 1H) pulse length in μs> <90° (1H) pulse strength in db> 15N 15N) pulse length in μs> <90° (15N) pulse strength in db>と入力します。
- スペクトル幅と取得時間の割り当てを決定します。
- 1 H-15N HSQC実験の取り込みパラメータウィンドウ(コマンドラインにedaを入力)で、スペクトル幅(取り込みパラメータSW)と直接寸法と間接寸法の次元増分数(取り込みパラメータTD)を調整します。
- SWは、直接寸法(1H)で16 ppm、間接寸法(15N)で30 ppmを選択します。TD の場合は、直接 (1H) 次元で 2048、間接 (15N) 次元で 512 を選択します。
- 1 H-15N HSQC実験を取得します。
- 対象のすべての信号が検出され、スペクトルにエイリアシング アーティファクトが存在しないことを確認します。スペクトルの中心が 15N軸に沿ったタンパク質シグナル分布の中心に適合しない場合は、O3Pを変更します。
- スペクトルウィンドウが小さすぎる場合は 、SW を大きくします。 TD を上げると、FID分解能が高くなります(デカップリングの最大持続時間については、プローブの仕様を確認してください)。
注:これは一般的なセットアップであり、NMR緩和実験のセットアップの出発点です。 1 H-15N HSQC実験は、全実験セットの開始時と終了時に記録し、さらに実験時間が長くなる場合は、サンプルの完全性を監視するために、その間に記録することをお勧めします。
- 1 H-15N HSQC実験の取り込みパラメータウィンドウ(コマンドラインにedaを入力)で、スペクトル幅(取り込みパラメータSW)と直接寸法と間接寸法の次元増分数(取り込みパラメータTD)を調整します。
3. NMR緩和実験の実施
注:NMR緩和パルスシーケンス(図2)は、https://www.ipb.hhu.de/en/teams/team-lakomek/pulsesequences または拡張Biological Magnetic Resonance Bank(BMRB)リポジトリ(bmrbig102)で入手できます。
- を設定します。 15N・R1ρ 実験。
- HSQC の実験 ( edc と入力) を新しいディレクトリにコピーします。 ased と入力し、パルスシーケンスパラメータのパラメータラインPULPROGの3つのドットをクリックして、パルスプログラムを 15N R1ρ 実験に変更します(ased、 補足図3Aを参照)。パルスプログラムをダブルクリックし、[ Set PULPROG to dataset]( 補足図3Bを参照)をクリックし、[ OK]をクリックします。
注: 一部のエラー メッセージが表示される場合があります。これらのメッセージを閉じます。 - コマンドラインに gpnam と入力して、パルスシーケンス(補足図4)で説明されているように、欠落しているすべての勾配形状(gpnam1-gpnam9)を挿入します。
- コマンドラインに vplist と入力します。3 つのドット (補足図 5A)、 ファイル (左上隅)、 および新規 (補足図 5B) をクリックします。
- 新しいダミーvplistの名前を入力し、[ OK]を押します。ダミーの vplist を作成します (5m、65m、25m、45m、15m、55m、35m、10m、vplist エントリごとに新しい行)。 Ctrl S を押してリストを保存し、vplist のウィンドウを閉じます (補足図 5C)。
- 新しく作成したvplistの名前をダブルクリックします(補足図5B)。
注:これは、パルスシーケンスのコンパイルエラーを回避するために必要です。vplist の適切なエントリは、以下 (ポイント 3.1.29.) で決定されます。 - asedと入力して、パルスシーケンスパラメータを開きます(補足図6)。パルスシーケンスで指定されているすべての欠落しているグラジエント強度とグラジエントの長さを入力します(補足図6D)。
- ループカウンタ L3 (間接次元の複素点の数) に、取得パラメータウィンドウ (eda) の 15N TD エントリの 1/2 を入力します。ループカウンターL6に、緩和曲線のフィッティングに使用される記録された緩和データポイントの数を入力します。
注: この番号 L6 は、vplist エントリの数 (ここでは 8) に対応します (補足図 6A)。 - 上記で指定したWebサイトからダウンロードした断熱TanhTan_half.nlパルスを 15NSP8パルスの形状として設定します。Webサイトから断熱TanhTan_2nd.nlパルスを 15N SP9パルスの形状として設定します(補足図6C)。
- P8エントリを断熱TanhTanパルスの長さに設定します-パルスの断熱性を保証するのに十分な長さです。ここでは、P8 = 3000 μs が選択されます (補足図 6C)。
- 15N デカップリング (CPDPRG 3) を garp デカップリング・スキームに設定します。PCPD3 200 μsのデカップリングの長さに使用します(補足図6C)。
- スキャン間回復遅延を少なくとも D1 = 2 秒以上に設定します。
注: 適切な回復遅延の選択については、「ディスカッション」セクションを参照してください。 - 最終実験では、ダミースキャン(DS)を少なくとも64に設定します(セットアップ中は、時間上の理由からDS = 4で十分です)。開始点として、4 のスキャン数 (NS) を使用します。信号対雑音比(SNR)が低すぎる場合は、4の倍数を使用します(補足図6A)。
- O1をステップ2.5の校正済みキャリア周波数に設定します。O2Pを176ppmに設定し、 1 H-15N HSQC実験(補足図6A-C)のO3Pをコピーします。
- パルス長P7を、手順90で校正した15°(2.6N)パルス長に設定します。また、ステップ 2.6 の 90°(15N)パルスのパルス電力レベルを PLW3 と PLW7 にコピーします (補足図 6C)。
- パルス長P1とP19を、手順2.4で計算した90°(1H)のパルス長に設定します。(補足図6B)。
- 間接寸法 TD = l3 * 2 * l6 の増分数を設定します (eda またはコマンド ラインに td と入力します)。
注意: ここで説明するすべてのパルスシーケンスパラメータは、パルスシーケンスに追加で注釈が付けられます。 - シェイプパルスSP5をI-BURP2形状(Iburp2.1000)にセットします。パルス長P15を2000μsに設定します。Topspin pulse sequence parameter window (ased) で I-BURP2 shape pulse の隣にある E をクリックして、シェイプツールディスプレイを開きます (補足図 6B)。
- 正しい長さで形状パルスをシミュレートするには、「NMRシミュレーションの開始」ボタンをクリックします。シミュレーションウィンドウで、成形パルス長[μs](=2000μs)と回転角[°](=180°)を確認します。 「NMR-SIMの開始」をクリックします(補足図7A)。
- 励起範囲を確認し、タンパク質の 1Hスペクトル分散をカバーする適切なI-BURP2パルス長を選択しますが、同時に水の励起は避けてください。P15 を、最適な IBURP-2 パルスがシミュレートされたシミュレーション ウィンドウの Shaped パルス長に設定します (補足図 7B)。
注:I-BURP2がアミド領域全体を励起(95%の効果>)する場合は、適切な長さが選択されます。異なる長さの I-BURP2 パルスをシミュレートするには、励起プロファイル ウィンドウを閉じ、シミュレーション ウィンドウで成形パルス長を編集します。 Start NMR-SIMをクリックします。長さが短いパルスは、Hz単位での励起範囲が広くなります。 - I-BURP2パルスのキャリア周波数を変更し、励起範囲を左または右にシフトして、I-BURP2パルスが水の磁化に触れて乱れないように SPOFFS5 を設定します(補足図6B)。
- 整形パルスの適切なパワーレベルを決定するには、Bruker Shapeツールを開き、「 Start NMR Simulation 」ボタンをクリックして、I-BURP2パルス長を「Shaped pulse length [μs]」に設定します。ソフトな長方形の90°(1H)パルスの対応する長さは、シミュレーションウィンドウに対応する90°の方形パルス[μs]で表示されることに注意してください(補足図7A)。
- 「calcpowlev」と入力します。基準長としてマイクロ秒単位のハード90°(1H)パルス(P1)を使用し(補足図8B)、新しい長さとしてI-BURP2パルスに対応する90°(1H)ソフト長方形パルス(マイクロ秒)を使用します(補足図8A)。ハード90°(1H)パルスとソフト矩形パルスの間の電力差Δ(dB)を覚えておいてください(補足図8C)。ハード90°(1H)パルスのパルス強度(dB)をSPW5にコピーし、記憶された差Δ(dB)をdBで追加します(補足図6B)。
- スピンロックのパワーレベルを決定するには、スピンロックの対応する90°(15N)パルス長を計算します。式を使用します:90°(15N)パルス長(s = 0.25 )/(スピンロック電力(Hz))。
- calcpowlev を使用して、スピン ロックのパワーと 90° (15N) ハード パルス (P7) の間のパワー差 Δ (dB) を計算します。新規:スピンロックのパルス長90°(15N)(μs)(補足図8A)。参照:硬質90°(15N)パルス長(μs)(補足図8B)。90°(15N)のハードパルス(dB)のパワーレベルPLW7をスピンロックパワーレベルPLW8(補足図6C)にコピーし、計算されたパワー差Δ(dB)を加算します(補足図8C)。
- スピンロック PLW8 の電力レベルを電力レベル SPW8 と SPW9 にコピーします。(補足図6C)。
- 15N デカップリングの電力レベルを決定するには、calcpowlev を使用して、90° (15N) のデカップリング パルス (PCPD3) と 90° (15N) ハード パルス (P7) の電力の間の電力差 Δ を計算します。新規:PCPD3(μs)(200)(補足図8A)。参照:硬質90°(15N)パルス長(μs)(補足図8B)。90°(15N)のハードパルスの電力レベルPLW7(dB)をデカップリングレベルPLW31(補足図6C)にコピーし、Δ(dB)をdBで追加します(補足図8C)。
- 温度補償を使用するには、 #define TEMP_COMPENSATION を含めます(パルスシーケンスのセミコロンを削除します)。P18 を 15N R1ρ 実験で使用したスピンロックの最大持続時間に設定します。
注:実験の最初の試行では、温度補正をオフにすることをお勧めします。 - 15N 13Cラベル付きサンプルの場合は、パルスプログラムにLABEL_CNを定義するために#を含めます(パルスシーケンスのセミコロンを削除します)。P4を78の式で計算されたパルス長に設定します。たとえば、600 MHz で 23.70 μs、1200 MHz で 11.85 μs に相当します。「calcpowlev」と入力して、P4 と (13C) ハード パルスの電力差を dB 単位で計算します。(13C)ハードパルス電力(dB)をPLW4にコピーし、Δ(dB)を加算します。PLW4 を PLW2 にコピーします (補足図 6B)。
- 適切なサンプリングのための緩和遅延を決定します。
- 最初の 8 つの FID (L6 = 8 の場合) を実行し、これらを処理します (rser <新しい実験番号>)。 遅延が最も長い実験のピーク強度が、遅延が最も短い実験(参照として)と比較して、少なくとも1/e(約37%)に減少するが、25%以上に減少するエントリをvplistで選択します。
- vplist の残りのエントリを調整して、遅延が最も長い FID と遅延が最も短い FID の間で強度が減少するサンプリングを均等にします。プローブの注と電源仕様をお読みください。
注:ゆっくりと緩和する残留物が最初のFIDを支配します。vplist の残りのエントリを調整して、1 番目と 2 番目の FID の間で強度が減少するサンプリングが均等になるようにします。プローブの最大許容スピンロック電力と長さを超えないようにしてください(プローブのマニュアルとプローブの仕様を参照してください)。温度補償の場合は、P18 (最大スピンロック持続時間) にチェックを入れます。これは、P18 の全体的なスピンロック持続時間が適用され、緩和遅延と温度補償に分割されます。時間(緩和遅延)+時間(温度補償)= const = P18を保持します。2kHzのスピンロックの場合、2秒の回復遅延(D1)を使用して、65ミリ秒を超えるスピンロック時間を超えないようにします。その場合、最大スピンロック期間と最長緩和遅延との間の妥協点を、最大許容スピンロック期間を優先する必要があります。D1回復の遅延が長い場合は、より長いスピンロックパワーが適用される場合があります。重要なのは、プローブ内の経時的な電力蓄積です。スピン ロックの持続時間を長くする必要がある場合は、スピン ロックの長さを少しずつ増やし、ロック信号を監視します。経験則として、ロックディスプレイの上3分の1でロックレベルを調整します。パルス・シーケンスの実行中にロック・レベルが2乗以上減少すると、プローブに蓄積されるスピン・ロック電力が大きすぎます。
- コマンドラインにrgaと入力して、レシーバーゲインを決定します。良好な信号対雑音比を得るには、レシーバーゲイン(rg)値が128〜256であることを目指してください。これは、良好な水抑制を証明しています。
注意: レシーバーゲインが128〜256に到達できない場合は、水信号が強すぎることを示しています。その場合、レシーバーのゲインに達するまで、勾配の電力と持続時間は緩やかに調整されます。ディスカッションのセクションも参照してください。 - 15N R1ρ 実験のテスト実行を開始するには、コマンド ラインに「zg」と入力します。すべての遅延(vplistで8つの遅延の場合、1〜8)で水信号が十分に抑制されていることを確認します。直交検出方式(エコー/アンチエコー)の2番目の増分であるインクリメント9もオンにします。
- HSQC の実験 ( edc と入力) を新しいディレクトリにコピーします。 ased と入力し、パルスシーケンスパラメータのパラメータラインPULPROGの3つのドットをクリックして、パルスプログラムを 15N R1ρ 実験に変更します(ased、 補足図3Aを参照)。パルスプログラムをダブルクリックし、[ Set PULPROG to dataset]( 補足図3Bを参照)をクリックし、[ OK]をクリックします。
- 15N R1 実験を設定します。
- 15N R1ρ 実験をコピーし、パルス プログラムを 15N R1 実験に変更します。
- 「vclist」と入力し、vclistを生成し、70で説明されているダミーのvclistを入力します:0、24、6、20、4、16、8、12。vclist エントリごとに新しい行)。
- オープン アス。グラデーションの長さと強度を確認します。GPZ9を14%に変更します。
- パルス長、パルス形状、およびパルス強度を確認します( 正しい値は15N R1ρ 実験からコピーされているはずです)。
- 15N13C標識サンプルの場合は、パルスプログラムで #define LABEL_CN を使用します。
- 温度補償を使用する場合は、 #define TEMP_COMPENSATION を含めます。
- 最初の 8 つの FID (L6 = 8 の場合) を実行し、これらを処理します (rser <新しい実験番号>)。遅延が最も長い実験のピーク強度が、遅延が最も短い実験と比較して少なくとも 1/e (約 37%) に減少し、25% 以上になる vclist のエントリを選択します。vplist の残りのエントリを調整して、遅延が最も長い FID と遅延が最も短い FID の間で強度が減少するサンプリングを均等にします。
注: vclist エントリに使用できるのは偶数のみです。
- {1H}-15N NOE 実験を設定します。
- 15N R1緩和実験をコピーし、パルスシーケンスを{1H}-15N NOEパルススキームに変更します。
- 飽和期間の長さをループカウンターL8で設定します。180°(1H)パルス間の間隔は22 ms79 (2 * 1 / JNH)です。最も正確な結果を得るには、タンパク質の 15N T1(15N)の約5倍の飽和期間を使用します。ここ:5秒。これは L8 = 230 に相当します。
注: 「ディスカッション」セクションでは、飽和期間/回復遅延の選択に関する追加のアドバイスを提供します。 - パルス方式で説明されているように、勾配の長さと強度を変更します。sine.20グラデーション形状を使用します。
- 間接寸法の増分数を TD = l3 * 2 * 2 に等しく設定します
- 15N13C標識サンプルの場合、#define LABEL_CNと-DLABEL_CNをZGOPTNSとして使用します。
- コマンドラインに「zg」と入力して、パルススキームを実行します。
4. 記録されたNMR実験の処理と分析
注:スペクトルはBrukerシステムを使用して記録されています。処理は、Unix または Linux オペレーティング システムを使用して実行されます。スペクトル処理とデータ解析は、NMRPipe80 とpython3を用いて行いました。NMRPipeソフトウェアは https://www.ibbr.umd.edu/nmrpipe/index.html からダウンロードできます。NMRPipeベースの処理スクリプトは、Webサイト(https://www.ipb.hhu.de/en/teams/team-lakomek/pulsesequences)または拡張Biological Magnetic Resonance Bank(BMRB)リポジトリ(bmrbig102)からダウンロードできます。NMRPipeの使用を推奨します。NMRPipeが利用できない場合、または望ましくない場合は、CCPN81 またはSPARKY(SPARKY 3、またはその後継のNMRFAM-SPARKY82 またはPOKY83 )などの代替手段を使用できます。
- 処理と分析 15N・R1 実験。
注: 次のコマンドは Unix ターミナルで実行されます。- ダウンロードしたディレクトリ内のファイルをR1_processing実験用R1データを含むディレクトリ(作業ディレクトリ)にコピーします。
- ターミナルコマンドでファイルを実行可能にします > chmod u+x *.com
- > brukerを実行してNMRPipe変換ユーティリティを開き、fid.com ファイルを生成します。「パラメータの読み取り」をクリックします。3 つのディメンションがカウントされる場合は [ディメンション数] を 2 に変更し、再度 [パラメーターの読み取り] をクリックします。次に、[Clear Script]、[Update Script]、および [Save Script] をクリックします (補足図 9)。
注:キャリア周波数が正しくない場合は、 2回目に[パラメータの読み取り ]をクリックした後で[中心位置]PPMを変更してください。 - > ./fid.com を実行します。
- vplist の vclist を ms エントリで変換します: 遅延 (ミリ秒 = 40 ミリ秒 * vclist のエントリ)。 > cp vclist vplist を使用して、vplist を編集します。
- > ./split.com を使用して、疑似 3D データを 8 つの 2D ファイルに分割します。
- nmrproc.com の tauValues (vplist の値を vplist の順に並べる) と idxExpmt の値 (tauValues の時間の順序) を編集します (補足図 10)。
- > ./nmrproc.com を使用して、8 つの 2D スペクトルすべての生データを処理します。
- > nmrDrawを使用してスペクトルを描画します。
- これらの手順では、参照スペクトル (vplist の最初のエントリ) を使用します。z 次元を 1 (左上隅) に変更して、そのスペクトルに切り替えます。 [係数 ]ボタンの横にある[+]と[-]を使用して、等高線レベルを調整します。 Drawボタンでスペクトルを再描画します。
- ノイズから来る信号はごくわずかで、関心のあるすべての信号を認識できるコンター・レベルを使用します(コンター・レベルはノイズ・レベルの端にあります)(補足図11)。
- スペクトル位相を補正し、垂直 1D 信号投影の場合は v 型、水平 1D 信号投影の場合は h を入力します。P0とP1で位相を調整し(補足図11)、位相差を覚えて、nmrproc.com ファイル(補足図10)で位相を修正します。
注:スペクトルが15N次元で反転している場合は、行の-negを削除します:| nmrPipe -fn FT -neg \(補足図10)。 - nmrDrawを閉じて、>nmrproc.com で再度処理します。ピークピッキング(スペクトルが正しく位相化され、参照スペクトルがすべてのピークが存在する等高線レベルで表示された後)の場合、K(シフト+ k)と入力し、検出を押して保存をクリックします。(補足図11)。
- ピークリストを作業ディレクトリにコピーし、ass.com の-assNameをピークリストの名前に変更します(補足図12)。
注:正しい形式のピークリストの例については、https://www.ipb.hhu.de/en/teams/team-lakomek/pulsesequences
拡張されたBiological Magnetic Resonance Bank(BMRB)リポジトリ(bmrbig102)にあります。 - > ./ass.com を実行してスペクトルを割り当てます。スクリプトがさまざまなピークに対して提案する割り当てを確認します。マウスの左ボタンを使用してピークを割り当て、マウスの右ボタンを使用してピークの割り当てを解除します。 [次へ ] ボタンを使用して次のアミノ酸に移動し、[ 前へ ] ボタンを使用して前のアミノ酸に移動します (補足図 13)。
注:輪郭レベルは、右上隅の+と-で変更できます。 [Draw ] ボタンを使用して、新しい等高線レベルでスペクトルを再描画します (補足図 13)。 - 割り当てが完了したら、[保存] をクリックし、もう一度 [保存] をクリックします。その後、[Quit]、[Cancel]、および[Exit]をクリックします(補足図13)。
- python3 NoneDeleter.py を実行して>割り当てられたピークに関する情報のみを含む relax.tab を作成します。
- > ./relax.com を実行して、autofit.com スクリプトを生成します。
- > ./autoFit.com を実行してフーリエ補間を実行し、各アミノ酸の異なる遅延時間の擬似比を生成します (axt.tab と nlin.tab が生成されます)。
- > ./model.com を実行して、 nlin.tab のデータに基づいて modelExp.com スクリプトを生成します。
- > ./modelExp.com を実行して、非線形最小二乗最適化モデルを使用して各残基の進化曲線を適合させます。
- bash summary.tcl -in relax.tab > t1.tab >を実行します。潜在的なエラー メッセージを無視します: GDB Notice: 21 missing values in the table file can occur.
- t1.tab を新しく作成したディレクトリ R1R2 にコピーします。
- 15N R1ρ 実験を処理および分析します。
- ダウンロードしたディレクトリR1rho_processing内のファイルを、実験的なR1ρ データを含むディレクトリ(作業ディレクトリ)にコピーします。
- > chmod u+x *.com を実行します。
- > brukerを実行してNMRPipe変換ユーティリティを開き、fid.com ファイルを生成します。15N R1 スペクトルの処理も参照してください。
- > ./fid.com を実行します。
- > ./split.com を実行して、疑似 3D データを 8 つの 2D ファイルに分割します。
- > ./nmrproc.com を実行し ( tauValues [vplist values] と idxExpmt を事前に編集)、8 つの 2D スペクトルすべての生データを処理します (補足図 10)。
- nmrDraw>を実行してスペクトルを描画し、ピークを選択します。これらの手順では、参照スペクトル (vplist の最初のエントリ) を使用します。15N R1 の実験と同じ手順に従います。
- ピークリストを作業ディレクトリにコピーし、ass.com の-assNameをピークリストの名前に変更します(補足図12)。
- > ./ass.com を実行し、15N R1 実験と同じ手順に従います。
- python3 NoneDeleter.py>を実行して、割り当てられたピークに関する情報のみを含む relax.tab を作成します。
- > ./relax.com を実行
- > ./autoFit.com を実行
- > ./model.com を実行
- > ./modelExp.com を実行
- bash summary.tcl -in relax.tab > t1rho. tab >実行します。潜在的なエラー メッセージを無視する: GDB Notice: 21 missing values in the table file may occur.
- t1rho.tab ファイルに次のヘッダーをコピーします (値は fid.com ファイルから取得できます) (補足図 14)。
備考 600MHz for T1rho
備考 -yOBS = 60.818 (ここに fid.com で見つかった値を入れてください)
備考 -yCAR = 119.06 (fid.com で求められた値をここに入れてください) - > ./extract_PPM.com を実行して、キャリア周波数に対するピーク 15N 周波数のオフセットを含む fusionOffset.tab というテーブルを生成します (これは、R1 データと R1ρ データから R2 データを抽出するための角度 θ を計算するために必要になります)。
- リラクゼーションデータを要約します。
- t1rho.tab と fusionOffset.tab をディレクトリ R1R2 にコピーします。
- R1R2ディレクトリ内のスクリプトcrtR1p.tclをコピーし、分光器の周波数とスピンロック電力を調整して、bash crtR1p.tcl | sort -n > R1R2.tabを実行します。(補足図15)で囲まれています。 R1R2.tabには、適合されたR1ρレート定数、シータ角、R2レート定数、およびR1レート定数が含まれます。
- hetNOE実験を処理および分析します。
- ダウンロードしたディレクトリ内のファイルをNOE_processing実験NOEデータディレクトリ(作業ディレクトリ)にコピーします。
- > chmod u+x *.com を実行します。
- > brukerを実行してNMRPipe Conversion Utilityを開き、fid.com ファイルを生成します。「パラメータの読み取り」をクリックします。3 つのディメンションがカウントされた場合は [ディメンション数] を 2 に変更し、もう一度 [パラメーターの読み取り] を押します。次に、[スクリプトのクリア]、[スクリプトの更新]、および [スクリプトの保存] を押します。
- > ./fid.com を実行します。
- > ./split.com を実行 (2 つのエントリ (0, 1) を持つ vplist を事前に作成する必要があります)
- > ./ft2.com を実行
- nmrDraw >を実行してスペクトルを描画し、参照スペクトル (z 次元 = 1) のピークを選択します。ノイズを抽出して記憶します(T = shift + tを押します)。位相補正とピークピッキングは、15N R1 実験と同じ手順に従います。
- ピークリストを作業ディレクトリにコピーし、ass.com の-assNameをピークリストの名前に変更します(補足図12)
- > ./ass.com を実行してスペクトルを割り当てます。 15N R1 実験で詳述したのと同じ手順に従います。
- python3 NoneDeleter.py を実行して>relax.tab を作成します。
- > ./script.com を実行
- > ./autoFit.com を実行
- error_hetnoe.tcl のノイズを変更します。
- bash error_hetnoe.tcl |sort -n > hetNOE.tab >を実行します。
Access restricted. Please log in or start a trial to view this content.
結果
以下は、水疱性SNAREタンパク質Synaptobrevin-2(1-96)、しばしばVAMP2(小胞関連タンパク質2)に記録されたいくつかの例示的なNMR緩和データを示す。NMRデータの記録には、150 mM NaCl、0.1 mM TCEP、および1 mM EDTAを含む50 mM MES(pH 6.0)緩衝液中の171 μM 15N Synaptobrevin-2 (1-96)サンプル(以下、Syb-2と呼びます)を使用しました。すべての実験データは、3 mm NMRサンプルチューブに充填?...
Access restricted. Please log in or start a trial to view this content.
ディスカッション
このプロトコルは、Lakomekら69およびStiefら70によるNMR 15N緩和実験のセットアップについて説明しました。私たちは、感度を高めたHSQC検出スキームを用いたNMRパルス配列に着目しました。15N R1およびR1ρ実験は、Stief et al.70によって詳細に記述されているように実施されており、hetNOE?...
Access restricted. Please log in or start a trial to view this content.
開示事項
著者らは、この論文で報告された研究に影響を与えたと思われる可能性のある既知の競合する金銭的利益や個人的な関係がないことを宣言します。
謝辞
有益な議論をしてくれたMelinda Jaspert氏とKevin Bochinsky氏に感謝します。N.L.は、ハイゼンベルクプログラム(DFG助成金番号433700474)を通じて資金提供してくれたドイツ科学財団に感謝します。この研究は、ヘルムホルツ協会のイニシアチブとネットワーキング基金からの助成金であるプロジェクト「COVID-19病因のウイルス学的および免疫学的決定要因-将来のパンデミックに備えるための教訓」(KA1-Co-02「COVIPA」)によってさらにサポートされています。私たちは、ユーリッヒ国立大学とデュッセルドルフ・ハインリッヒ・ハイネ大学(HHU)が共同で運営するユーリッヒ・デュッセルドルフ生体分子NMRセンターへの寛大なアクセスを認めます。
Access restricted. Please log in or start a trial to view this content.
資料
| Name | Company | Catalog Number | Comments |
| Bruker 600 MHz AVANCE III HD spectrometer | Bruker | https://www.bruker.com/en/products-and-solutions/mr/nmr/avance-nmr-spectrometer.html | NMR experiments conducted |
参考文献
- Sekhar, A., Kay, L. E. An NMR view of protein dynamics in health and disease. Annu Rev Biophys. 48, 297-319 (2019).
- Rosenzweig, R., Kay, L. E. Bringing dynamic molecular machines into focus by methyl-TROSY NMR. Annu Rev Biochem. 83, 291-315 (2014).
- Palmer, A. G. 3rd Enzyme dynamics from NMR spectroscopy. Acc Chem Res. 48 (2), 457-465 (2015).
- Reif, B. Ultra-high resolution in MAS solid-state NMR of perdeuterated proteins: implications for structure and dynamics. J Magn Reson. 216, 1-12 (2012).
- Schanda, P., Ernst, M. Studying dynamics by magic-angle spinning solid-state NMR spectroscopy: Principles and applications to biomolecules. Prog Nucl Magn Reson Spectrosc. 96, 1-46 (2016).
- Le Marchand, T., et al. (1)H-H-detected biomolecular NMR under fast magic-angle spinning. Chem Rev. 122 (10), 9943-10018 (2022).
- Pervushin, K., Riek, R., Wider, G., Wuthrich, K. Attenuated T2 relaxation by mutual cancellation of dipole-dipole coupling and chemical shift anisotropy indicates an avenue to NMR structures of very large biological macromolecules in solution. Proc Natl Acad Sci U S A. 94 (23), 12366-12371 (1997).
- Tzakos, A. G., Grace, C. R., Lukavsky, P. J., Riek, R. NMR techniques for very large proteins and RNAs in solution. Annu Rev Biophys Biomol Struct. 35, 319-342 (2006).
- Sprangers, R., Kay, L. E. Quantitative dynamics and binding studies of the 20S proteasome by NMR. Nature. 445 (7128), 618-622 (2007).
- Wright, P. E., Dyson, H. J. Intrinsically disordered proteins in cellular signalling and regulation. Nat Rev Mol Cell Biol. 16 (1), 18-29 (2015).
- van der Lee, R., et al. Classification of intrinsically disordered regions and proteins. Chem Rev. 114 (13), 6589-6631 (2014).
- Tompa, P. Intrinsically unstructured proteins. Trends Biochem Sci. 27 (10), 527-533 (2002).
- Dyson, H. J., Wright, P. E. Coupling of folding and binding for unstructured proteins. Curr Opin Struct Biol. 12 (1), 54-60 (2002).
- Uversky, V. N. Intrinsically disordered proteins in overcrowded milieu: Membrane-less organelles, phase separation, and intrinsic disorder. Current Opinion in Structural Biology. 44, 18-30 (2017).
- Patel, A., et al. A liquid-to-solid phase transition of the ALS protein FUS accelerated by disease mutation. Cell. 162 (5), 1066-1077 (2015).
- Abyzov, A., Blackledge, M., Zweckstetter, M. Conformational dynamics of intrinsically disordered proteins regulate biomolecular condensate chemistry. Chem Rev. 122 (6), 6719-6748 (2022).
- Uversky, V. N. Unusual biophysics of intrinsically disordered proteins. Biochim Biophys Acta. 1834 (5), 932-951 (2013).
- Papoian, G. A. Proteins with weakly funneled energy landscapes challenge the classical structure-function paradigm. Proc Natl Acad Sci U S A. 105 (38), 14237-14238 (2008).
- Uversky, V. N., Gillespie, J. R., Fink, A. L. Why are "natively unfolded" proteins unstructured under physiologic conditions. Proteins. 41 (3), 415-427 (2000).
- Bah, A., et al. Folding of an intrinsically disordered protein by phosphorylation as a regulatory switch. Nature. 519 (7541), 106-109 (2015).
- Tompa, P., Schad, E., Tantos, A., Kalmar, L. Intrinsically disordered proteins: emerging interaction specialists. Curr Opin Struct Biol. 35, 49-59 (2015).
- Dyson, H. J., Wright, P. E. Perspective: the essential role of NMR in the discovery and characterization of intrinsically disordered proteins. J Biomol NMR. 73 (12), 651-659 (2019).
- Adamski, W., et al. A unified description of intrinsically disordered protein dynamics under physiological conditions using NMR spectroscopy. J Am Chem Soc. 141 (44), 17817-17829 (2019).
- Dobson, C. M. Protein folding and misfolding. Nature. 426 (6968), 884-890 (2003).
- Bertoncini, C. W., et al. Release of long-range tertiary interactions potentiates aggregation of natively unstructured alpha-synuclein. Proc Natl Acad Sci U S A. 102 (5), 1430-1435 (2005).
- Buell, A. K., et al. Solution conditions determine the relative importance of nucleation and growth processes in alpha-synuclein aggregation. Proc Natl Acad Sci U S A. 111 (21), 7671-7676 (2014).
- Salvi, N., Abyzov, A., Blackledge, M. Atomic resolution conformational dynamics of intrinsically disordered proteins from NMR spin relaxation. Prog Nucl Magn Reson Spectrosc. 102, 43-60 (2017).
- Schneider, R., Blackledge, M., Jensen, M. R. Elucidating binding mechanisms and dynamics of intrinsically disordered protein complexes using NMR spectroscopy. Curr Opin Struct Biol. 54, 10-18 (2019).
- Rezaei-Ghaleh, N., Blackledge, M., Zweckstetter, M. Intrinsically disordered proteins: from sequence and conformational properties toward drug discovery. Chembiochem. 13 (7), 930-950 (2012).
- Jensen, M. R., Salmon, L., Nodet, G., Blackledge, M. Defining conformational ensembles of intrinsically disordered and partially folded proteins directly from chemical shifts. J Am Chem Soc. 132 (4), 1270-1272 (2010).
- Camacho-Zarco, A. R., et al. NMR provides unique insight into the functional dynamics and interactions of intrinsically disordered proteins. Chem Rev. 122 (10), 9331-9356 (2022).
- Mittag, T., Forman-Kay, J. D. Atomic-level characterization of disordered protein ensembles. Curr Opin Struct Biol. 17 (1), 3-14 (2007).
- Dyson, H. J., Wright, P. E. Unfolded proteins and protein folding studied by NMR. Chem Rev. 104 (8), 3607-3622 (2004).
- Dyson, H. J., Wright, P. E. NMR illuminates intrinsic disorder. Curr Opin Struct Biol. 70, 44-52 (2021).
- Sugase, K., Dyson, H. J., Wright, P. E. Mechanism of coupled folding and binding of an intrinsically disordered protein. Nature. 447 (7147), 1021-1025 (2007).
- Bessa, L. M., et al. The intrinsically disordered SARS-CoV-2 nucleoprotein in dynamic complex with its viral partner nsp3a. Sci Adv. 8 (3), eabm4034(2022).
- Milles, S., Salvi, N., Blackledge, M., Jensen, M. R. Characterization of intrinsically disordered proteins and their dynamic complexes: From in vitro to cell-like environments. Prog Nucl Magn Reson Spectrosc. 109, 79-100 (2018).
- Salvi, N., Abyzov, A., Blackledge, M. Atomic resolution conformational dynamics of intrinsically disordered proteins from NMR spin relaxation. Prog Nucl Magn Reson Spectrosc. 102 - 103, 43-60 (2017).
- Gill, M. L., Byrd, R. A., Palmer, A. G. III Dynamics of GCN4 facilitate DNA interaction: a model-free analysis of an intrinsically disordered region. Phys Chem Chem Phys. 18 (8), 5839-5849 (2016).
- Bertoncini, C. W., et al. Structural characterization of the intrinsically unfolded protein beta-synuclein, a natural negative regulator of alpha-synuclein aggregation. J Mol Biol. 372 (3), 708-722 (2007).
- Rezaei-Ghaleh, N., et al. Local and global dynamics in intrinsically disordered synuclein. Angew Chem Int Ed Engl. 57 (46), 15262-15266 (2018).
- Rezaei-Ghaleh, N., Parigi, G., Zweckstetter, M. Reorientational dynamics of amyloid-beta from NMR spin relaxation and molecular simulation. J Phys Chem Lett. 10 (12), 3369-3375 (2019).
- Palmer, A. G. NMR characterization of the dynamics of biomacromolecules. Chem Rev. 104 (8), 3623-3640 (2004).
- Palmer, A. G., Massi, F. Characterization of the dynamics of biomacromolecules using rotating-frame spin relaxation NMR spectroscopy. Chem Rev. 106 (5), 1700-1719 (2006).
- Kay, L. E., Torchia, D. A., Bax, A. Backbone dynamics of proteins as studied by 15N inverse detected heteronuclear NMR spectroscopy: application to staphylococcal nuclease. Biochemistry. 28 (23), 8972-8979 (1989).
- Mittermaier, A., Kay, L. E. Review - New tools provide new insights in NMR studies of protein dynamics. Science. 312 (5771), 224-228 (2006).
- Abragam, A. Principles of Nuclear Magnetism. , Clarendon Press. Oxford. (1983).
- Ernst, R. R., Bodenhausen, G., Wokaun, A. Principles of Nuclear Magnetic Resonance in One and Two Dimensions. , Oxford University Press. Oxford. (1990).
- Cavanagh, J., Fairbrother, W. J., Palmer, A. G. III, Skelton, N. J. Protein NMR Spectroscopy: Principles and Practice. , Elsevier, Academic Press. (1995).
- Levitt, M. H. Spin Dynamics: Basics of Nuclear Magnetic Resonance. , John Wiley and Sons. (2008).
- Keeler, J. Understanding NMR Spectroscopy. , John Wiley and Sons. (2010).
- Bloembergen, N., Purcell, E. M., Pound, R. V. Relaxation effects in nuclear magnetic resonance absorption. Phys Rev. 73 (7), 679-712 (1948).
- Wangsness, R. K., Bloch, F. The dynamical theory of nuclear induction. Phys Rev. 89 (4), 728-739 (1953).
- Redfield, A. G. On the theory of relaxation processes. IBM J Res Dev. 1 (1), 19-31 (1957).
- Peng, J. W., Wagner, G. Mapping of the spectral densities of N-H bond motions in eglin c using heteronuclear relaxation experiments. Biochemistry. 31 (36), 8571-8586 (1992).
- Farrow, N. A., et al. Backbone dynamics of a free and a phosphopeptide-complexed src homology-2 domain studied by n-15 NMR relaxation. Biochemistry. 33 (19), 5984-6003 (1994).
- Tolman, J. R., Ruan, K. NMR residual dipolar couplings as probes of biomolecular dynamics. Chem Rev. 106 (5), 1720-1736 (2006).
- Mulder, F. A. A., Skrynnikov, N. R., Hon, B., Dahlquist, F. W., Kay, L. E. Measurement of slow (mu s-ms) time scale dynamics in protein side chains by N-15 relaxation dispersion NMR spectroscopy: Application to Asn and Gln residues in a cavity mutant of T4 lysozyme. J Am Chem Soc. 123 (5), 967-975 (2001).
- Tollinger, M., Skrynnikov, N. R., Mulder, F. A. A., Forman-Kay, J. D., Kay, L. E. Slow dynamics in folded and unfolded states of an SH3 domain. J Am Chem Soc. 123 (46), 11341-11352 (2001).
- Akke, M., Palmer, A. G. Monitoring macromolecular motions on microsecond to millisecond time scales by R(1)rho-R(1) constant relaxation time NMR spectroscopy. J Am Chem Soc. 118 (4), 911-912 (1996).
- Loria, J. P., Rance, M., Palmer, A. G. A relaxation-compensated Carr-Purcell-Meiboom-Gill sequence for characterizing chemical exchange by NMR spectroscopy. J Am Chem Soc. 121 (10), 2331-2332 (1999).
- Vallurupalli, P., Bouvignies, G., Kay, L. E. Studying "Invisible" excited protein states in slow exchange with a major state conformation. J Am Chem Soc. 134 (19), 8148-8161 (2012).
- Jeener, J., Meier, B. H., Bachmann, P., Ernst, R. R. Investigation of exchange processes by 2-dimensional NMR-spectroscopy. J Chem Phys. 71 (11), 4546-4553 (1979).
- Palmer, A. G., Kroenke, C. D., Loria, J. P. Nuclear magnetic resonance methods for quantifying microsecond-to-millisecond motions in biological macromolecules. Methods Enzymol. 339, 204-238 (2001).
- Clore, G. M., Iwahara, J. Theory, practice, and applications of paramagnetic relaxation enhancement for the characterization of transient low-population states of biological macromolecules and their complexes. Chem Rev. 109 (9), 4108-4139 (2009).
- Iwahara, J., Clore, G. M. Detecting transient intermediates in macromolecular binding by paramagnetic NMR. Nature. 440 (7088), 1227-1230 (2006).
- Lakomek, N. A., et al. Residual dipolar couplings as a tool to study molecular recognition of ubiquitin. Biochem Soc Trans. 36 (Pt 6), 1433-1437 (2008).
- Johnson, C. N., Libich, D. S. Paramagnetic relaxation enhancement for detecting and characterizing self-associations of intrinsically disordered proteins. J Vis Exp. (175), e63057(2021).
- Lakomek, N. A., Ying, J., Bax, A. Measurement of (1)(5)N relaxation rates in perdeuterated proteins by TROSY-based methods. J Biomol NMR. 53 (3), 209-221 (2012).
- Stief, T., Vormann, K., Lakomek, N. A. Sensitivity-enhanced NMR (15)N R(1) and R(1rho) relaxation experiments for the investigation of intrinsically disordered proteins at high magnetic fields. Methods. 223, 1-15 (2024).
- Kay, L. E., Keifer, P., Saarinen, T. Pure absorption gradient enhanced heteronuclear single quantum correlation spectroscopy with improved sensitivity. J Am Chem Soc. 114 (26), 10663-10665 (1992).
- Geen, H., Freeman, R. Band-selective radiofrequency pulses. J Magn Reson (1969). 93 (1), 93-141 (1991).
- Desvaux, H., Berthault, P. Study of dynamic processes in liquids using off-resonance RF irradiation. Prog Nucl Magn Reson Spectrosc. 35 (4), 295-340 (1999).
- Overhauser, A. W. Polarization of nuclei in metals. Phys Rev. 91 (2), 476-476 (1953).
- Overhauser, A. W. Polarization of nuclei in metals. Phys Rev. 92 (2), 411-415 (1953).
- Kumari, P., Frey, L., Sobol, A., Lakomek, N. A., Riek, R. (15)N transverse relaxation measurements for the characterization of micros-ms dynamics are deteriorated by the deuterium isotope effect on (15)N resulting from solvent exchange. J Biomol NMR. 72 (3-4), 125-137 (2018).
- Karschin, N., Krenek, S., Heyer, D., Griesinger, C. Extension and improvement of the methanol-d(4) NMR thermometer calibration. Magn Reson Chem. 60 (4), 203-209 (2022).
- Sattler, M., Schleucher, J., Griesinger, C. Heteronuclear multidimensional NMR experiments for the structure determination of proteins in solution employing pulsed field gradients. Prog Nucl Magn Reson Spectrosc. 34 (2), 93-158 (1999).
- Ferrage, F., Cowburn, D., Ghose, R. Accurate sampling of high-frequency motions in proteins by steady-state N-{H} nuclear Overhauser effect measurements in the presence of cross-correlated relaxation. J Am Chem Soc. 131 (17), 6048-6049 (2009).
- Delaglio, F., et al. NMRPipe: a multidimensional spectral processing system based on UNIX pipes. J Biomol NMR. 6 (3), 277-293 (1995).
- Skinner, S. P., et al. CcpNmr AnalysisAssign: a flexible platform for integrated NMR analysis. J Biomol NMR. 66 (2), 111-124 (2016).
- Lee, W., Tonelli, M., Markley, J. L. NMRFAM-SPARKY: enhanced software for biomolecular NMR spectroscopy. Bioinformatics. 31 (8), 1325-1327 (2015).
- Lee, W., Rahimi, M., Lee, Y., Chiu, A. POKY: a software suite for multidimensional NMR and 3D structure calculation of biomolecules. Bioinformatics. 37 (18), 3041-3042 (2021).
- Abyzov, A., et al. Identification of dynamic modes in an intrinsically disordered protein using temperature-dependent NMR relaxation. J Am Chem Soc. 138 (19), 6240-6251 (2016).
- Singh, A., Purslow, J. A., Venditti, V. 15N CPMG relaxation dispersion for the investigation of protein conformational dynamics on the micros-ms timescale. J Vis Exp. (170), e62395(2021).
- Gopalan, A. B., Hansen, D. F., Vallurupalli, P. CPMG experiments for protein minor conformer structure determination. Methods Mol Biol. 1688, 223-242 (2018).
- Tugarinov, V., Kay, L. E. Methyl groups as probes of structure and dynamics in NMR studies of high-molecular-weight proteins. Chembiochem. 6 (9), 1567-1577 (2005).
- Lipari, G., Szabo, A. Model-free approach to the interpretation of nuclear magnetic resonance relaxation in macromolecules. 1. Theory and range of validity. J Am Chem Soc. 104 (17), 4546-4559 (1982).
- Lipari, G., Szabo, A. Model-Free Approach to the interpretation of nuclear magnetic-resonance relaxation in macromolecules .2. Analysis of experimental results. J Am Chem Soc. 104 (17), 4559-4570 (1982).
- Clore, G. M., et al. Deviations from the simple two-parameter model-free approach to the interpretation of nitrogen-15 nuclear magnetic relaxation of proteins. J Am Chem Soc. 112 (12), 4989-4991 (1990).
- Lemaster, D. M. Larmor frequency selective model free analysis of protein NMR relaxation. J Biomol NMR. 6 (4), 366-374 (1995).
- Peng, J. W., Wagner, G. Mapping of spectral density functions using heteronuclear NMR relaxation measurements. J Magn Reson (1969). 98 (2), 308-332 (1992).
- Farrow, N. A., Zhang, O. W., FormanKay, J. D., Kay, L. E. Characterization of the backbone dynamics of folded and denatured states of an SH3 domain. Biochemistry. 36 (9), 2390-2402 (1997).
- Khan, S. N., et al. Distribution of pico- and nanosecond motions in disordered proteins from nuclear spin relaxation. Biophys J. 109 (5), 988-999 (2015).
- Smith, A. A., Ernst, M., Meier, B. H., Ferrage, F. Reducing bias in the analysis of solution-state NMR data with dynamics detectors. J Chem Phys. 151 (3), 034102(2019).
- Zumpfe, K., Smith, A. A. Model-free or not. Front Mol Biosci. 8, 727553(2021).
- Robustelli, P., Trbovic, N., Friesner, R. A., Palmer, A. G. Conformational dynamics of the partially disordered yeast transcription factor GCN4. J Chem Theory Comput. 9 (11), 5190-5200 (2013).
- Salvi, N., Abyzov, A., Blackledge, M. Multi-timescale dynamics in intrinsically disordered proteins from NMR relaxation and molecular simulation. J Phys Chem Lett. 7 (13), 2483-2489 (2016).
Access restricted. Please log in or start a trial to view this content.
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved