È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Esperimenti di rilassamento NMR 15N per lo studio della dinamica strutturale delle proteine da picosecondi a nanosecondi
In questo articolo
Riepilogo
La spettroscopia di risonanza magnetica nucleare (NMR) è in grado di caratterizzare la dinamica strutturale delle proteine in modo specifico per i residui. Forniamo un protocollo pratico per la registrazione di esperimenti di rilassamento NMR 15N R1 e R2 e {1H}-15N heteronuclear Overhauser effect (hetNOE), sensibili alla scala temporale da picosecondi a nanosecondi.
Abstract
La spettroscopia di risonanza magnetica nucleare (NMR) consente di studiare le proteine in soluzione e a temperature fisiologiche. Frequentemente, i gruppi ammidici della spina dorsale proteica o i gruppi metilici nelle catene laterali sono utilizzati come reporter della dinamica strutturale nelle proteine. Uno studio di dinamica strutturale della spina dorsale proteica di proteine globulari su campioni marcati con 15N e completamente protonati di solito funziona bene per proteine con un peso molecolare fino a 50 kDa. Quando viene applicata la deuterazione a catena laterale in combinazione con la spettroscopia ottimizzata a rilassamento trasverso (TROSY), questo limite può essere esteso fino a 200 kDa per le proteine globulari e fino a 1 MDa quando l'attenzione è rivolta alle catene laterali. Quando si studiano le proteine intrinsecamente disordinate (IDP) o le proteine con regioni intrinsecamente disordinate (IDR), queste limitazioni di peso non si applicano, ma possono andare ben oltre. Il motivo è che gli IDP o IDR, caratterizzati da un'elevata flessibilità interna, sono spesso disaccoppiati dinamicamente. Vari metodi NMR offrono informazioni sulla risoluzione atomica della dinamica strutturale delle proteine in un'ampia gamma di scale temporali, dai picosecondi fino alle ore. Le misure standard di rilassamento a 15N analizzano la flessibilità interna di una proteina e caratterizzano la dinamica della spina dorsale proteica sperimentata su una scala temporale da pico a nanosecondi. Questo articolo presenta un protocollo pratico per l'impostazione e la registrazione di esperimenti NMR 15N R1, R2 e heteronuclear Overhauser effect (hetNOE). Mostriamo dati esemplari e spieghiamo come interpretarli in modo semplice qualitativo prima di qualsiasi analisi più sofisticata.
Introduzione
La funzione di una proteina è determinata non solo dalla sua struttura tridimensionale, ma anche dalla sua dinamica strutturale, che comprende la sua flessibilità interna e le transizioni strutturali tra le diverse conformazioni che la proteina adotterà. La spettroscopia di risonanza magnetica nucleare (NMR) può studiare la dinamica strutturale delle proteine in soluzione 1,2,3. I recenti sviluppi nella NMR allo stato solido rilevata con protoni consentono anche la caratterizzazione della dinamica delle proteine in uno stato meno solubile, come, ad esempio, una membrana a doppio strato lipidico 4,5,6. Nella NMR in soluzione, è possibile studiare la dinamica strutturale della spina dorsale proteica e delle catene laterali proteiche. Per una proteina globulare, uno studio della dinamica strutturale della spina dorsale proteica può essere raggiunto fino a 50 kDa una volta che la proteina è marcata isotopicamente a 15N. Quando si impiegano la deuterazione a catena laterale e la spettroscopia ottimizzata a rilassamento trasversale (TROSY), questo limite può essere esteso fino a 200 kDa 7,8. Quando l'attenzione si concentra sulla dinamica della catena laterale, la gamma di proteine e complessi accessibili può essere estesa fino a 1 MDa 2,9.
Le limitazioni di peso indicate non si applicano alle proteine intrinsecamente disordinate (IDP), che spesso mostrano un'elevata dinamica intrinseca. Oltre il 30% del proteoma eucariotico comprende IDP o regioni intrinsecamente disordinate (IDR)10,11,12,13. Svolgono un ruolo centrale in molti processi cellulari, come la trasduzione e la trascrizionedel segnale 1, e sono spesso coinvolti nella separazione di fase intracellulare 14,15,16,17. Gli IDP mancano di una struttura nativa tridimensionale (3D) ben definita in condizioni fisiologiche e hanno un paesaggio energetico debolmente incanalato o accidentato17,18. A causa di una bassa idrofobicità e di una forte repulsione elettrostatica distribuita sulla spina dorsale degli IDP o degli IDR, manca una forza motrice per il ripiegamento in una struttura rigida19. Gli IDP adottano spesso una conformazione ripiegata quando sono complessi con altri partner di legame 10,20,21. Inoltre, le modifiche post-traduzionali (PTM) ampliano le possibilità di ripiegamento degli IDP o IDR22,23. Il ripiegamento errato delle IDP è stato identificato come causa di varie malattie, comprese le malattie neurodegenerative 15,24,25,26.
Gli IDP e gli IDR mostrano un'elevata flessibilità interna 21,27,28. Gli insiemi conformazionali che mostrano la variazione delle posizioni atomiche e degli angoli diedri sono stati derivati da simulazioni di dinamica molecolare e restrizioni ottenute dai dati sperimentali 29,30,31,32. A causa della dinamica e del conseguente disordine nello stato congelato, la densità elettronica diffusa rende difficile caratterizzarli strutturalmente utilizzando metodi all'avanguardia in biologia strutturale, come la crio-EM o la cristallografia a raggi X. Inoltre, le condizioni di cristallizzazione o le tecniche di preparazione del campione per esperimenti a temperature criogeniche possono influire sullo spazio conformazionale sperimentato dagli IDP. Tuttavia, la soluzione NMR funziona bene per proteine altamente dinamiche ed è quindi adatta per studiare le IDP 16,20,22,28,29,30,31,32,33,34,35,36,37,38.
Come introdotto in precedenza, la NMR in soluzione offre varie tecniche per studiare la dinamica interna delle proteine in un'ampia gamma di scale temporali (Figura 1), principalmente basate sul rilassamento di spin 31,33,38,39,40,41,42.
Il rilassamento di spin dei nuclei 15N nei gruppi ammidici della spina dorsale proteica è indotto da cambiamenti di orientamento dell'angolo di legame 1 H-15N dovuti alla dinamica interna della proteina e ai movimenti collettivi (inclusa, quando rilevante, la diffusione rotazionale)27,43,44,45,46,47,48,49,50,51. Su scale temporali più brevi del tempo di correlazione rotazionale τR (il tempo necessario alla molecola per ruotare un radiante, chiamato anche tempo di correlazione globale di tumbling), l'anisotropia di spostamento chimico (CSA) e l'accoppiamento dipolare (D) sono attivi e non mediati dalla diffusione rotazionale della proteina. La dinamica interna della spina dorsale proteica, che comprende variazioni negli angoli di legame, riorientamento dei legami e rotolamento rotazionale, induce fluttuazioni stocastiche del CSA e del tensore di accoppiamento dipolare, con conseguente variazione del campo magnetico locale, portando infine al rilassamento dello spin NMR 47,48,52,53 . Queste fluttuazioni possono essere descritte da una funzione di correlazione complessiva. La trasformata di Fourier della funzione di correlazione complessiva è chiamata funzione di densità spettrale. Nella teoria semi-classica del rilassamento di Redfield, le costanti di velocità di rilassamento NMR possono essere descritte da combinazioni lineari di queste funzioni di densità spettrale54.
Gli esperimenti di rilassamento NMR della spina dorsale 15N sviluppati nei primi anni '90 comprendono 15N R1, R1ρ e {1H}-15N esperimenti nucleari sull'effetto Overhauser, sensibili alla scala temporale veloce del picosecondo (ps) e del nanosecondo (ns), più veloce del tempo di correlazione rotazionale τR della proteina 45,55,56,57. Per caratterizzare la dinamica della spina dorsale più lenta del tempo di correlazione rotazionale τR, vengono utilizzati i cosiddetti esperimenti di dispersione di rilassamento, R1ρ, e gli esperimenti di Carr-Purcell-Meiboom-Gill (CPMG) sensibili alla dinamica dei microsecondi (μs) - millisecondi (ms) 44,46,58,59,60,61. Le dinamiche più lente dei microsecondi possono essere catturate mediante trasferimento di saturazione a scambio chimico (CEST) a 15N NMR62, spettroscopia di scambio (EXSY, da millisecondi a secondi) o NMR in tempo reale (RT) (da secondi a ore)63,64. L'effetto PRE (paramagnetic relaxation enhancement) delle sonde paramagnetiche, così come gli accoppiamenti dipolari residui (RDC), possono essere utilizzati per valutare l'intero intervallo di dinamica da ps a ms 65,66,67,68.
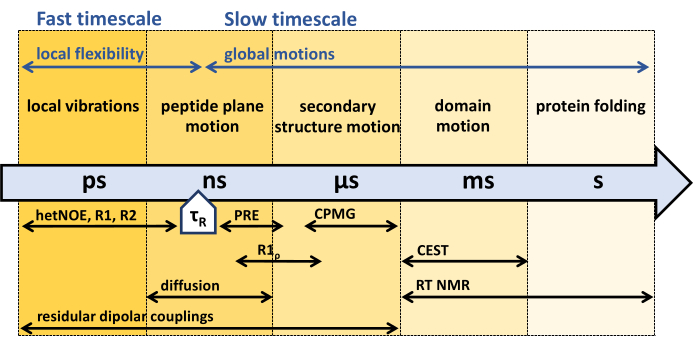
Figura 1: Scale temporali della dinamica della spina dorsale proteica e finestra temporale sensibile di diversi esperimenti di dinamica NMR. La risonanza magnetica nucleare offre una varietà di metodi per caratterizzare la dinamica della dorsale proteica in un'ampia gamma di scale temporali. I diversi movimenti sperimentati dalla spina dorsale proteica sono indicati nelle rispettive scale temporali. Il tempo di correlazione rotazionale della proteina, τR, è il tempo di cui la proteina ha bisogno per una rotazione complessiva (di un radiante). Movimenti più veloci del tempo di correlazione rotazionale della proteina, τR, possono essere associati alla flessibilità interna della proteina. Vari esperimenti NMR e la loro sensibilità alle rispettive scale temporali sono indicati sotto la freccia. Clicca qui per visualizzare una versione più grande di questa figura.
Il protocollo seguente descrive l'impostazione degli esperimenti di rilassamento NMR di Lakomek et al.69 e Stief et al.70, utilizzando uno schema di rilevamento a coerenza quantistica singola eteronucleare (HSQC) potenziato dalla sensibilità. Prima di procedere all'implementazione sperimentale, viene fornita una breve panoramica del rilassamento di spin NMR e degli esperimenti di rilassamento NMR. A causa dei limiti di dimensioni e per mantenere comprensibile questo protocollo, questa panoramica deve rimanere semplicistica (e quindi incompleta).
Il rilassamento longitudinale o reticolo di spin, caratterizzato dal tempo T1 o dalla costante di velocità R1 = 1/ T1 , descrive il ritorno della magnetizzazione all'equilibrio di Boltzmann. In equilibrio, la magnetizzazione è allineata lungo l'asse del campo magnetico esterno, che definisce l'asse z del telaio di laboratorio. Le densità spettrali alle frequenze di Larmor alte (1H) e piccole (15N) (le frequenze di risonanza NMR, ad esempio, 600 MHz per 1H per un magnete da 14,1 Tesla) e le combinazioni lineari di queste frequenze di Larmor contribuiscono al rilassamento di 15N R1 , che è caratterizzato dalle costanti di velocità di 15N R1 misurate in rad·s-1. I movimenti su scale temporali sono inversi a queste frequenze di Larmor; quindi, i movimenti sulla scala temporale da picosecondi a nanosecondi contribuiscono alla costante del tasso di rilassamento R1. Per le molecole che mostrano un tumbling complessivo e dove può essere definito un tempo di correlazione rotazionale, la curva R1 (T1) mostra un massimo (minimo) per ωτR = 1, con la correlazione rotazionale τR e la frequenza di Larmor ω dello spin in esame. Se più frequenze di Larmor contribuiscono, quella con la frequenza più bassa è quella dominante, ad esempio, ωN nel caso di 15N R1. Il regime di moto veloce (ωτR molto più piccolo di 1) si applica a piccole molecole che rotolano molto velocemente e per bassi campi magnetici e bassa viscosità. Il regime di slow motion (ωτR molto più grande di 1) è valido per molecole più grandi che rotolano più lentamente e per campi magnetici elevati e alta viscosità.
Le proteine ripiegate globulari mostrano un tumbling complessivo in soluzione e può essere assegnato un tempo di correlazione rotazionale. Tuttavia, il concetto di tumbling complessivo non è più valido per le proteine intrinsecamente disordinate e spesso differisce dall'assegnazione di un singolo tempo di correlazione rotazionale. Qui, il tempo di correlazione interna residuo specifico diventa più critico.
La sequenza di impulsi descritta che misura 15velocità di rilassamento N R1 (Figura 2) si basa su un esperimento di lettura HSQC con sensibilità migliorata con un rilevamento Echo/Anti-Echo per il rilevamento in quadratura 69,70,71. Brevi gradienti con intensità e lunghezza variabili vengono utilizzati per la selezione della coerenza e una migliore soppressione dell'acqua70. Durante questo periodo, la polarizzazione longitudinale di 15N si rilasserà. Tempi di decadimento più lunghi portano a intensità ridotte nei piani 2D associati di questo spettro pseudo-3D (i punti dati di ritardo sono registrati nella terza dimensione). Un elemento del ciclo, descritto di seguito, viene eseguito un numero crescente di volte per ottenere tempi di rilassamento più lunghi. Poiché il rilassamento cross-correlato tra l'anisotropia di spostamento chimico (CSA) di 15N e l'accoppiamento dipolare (D) di 1 H e 15N è attivo anche durante il ritardo di rilassamento, è necessarioun impulso centrale I-BURP-2 a 180°, selettivo sui protoni ammidici, per rifocalizzare il contributo mediante rilassamento cross-correlato (che se non rifocalizzato porterebbe a costanti di velocità di 15N R1 distorte ed errate).
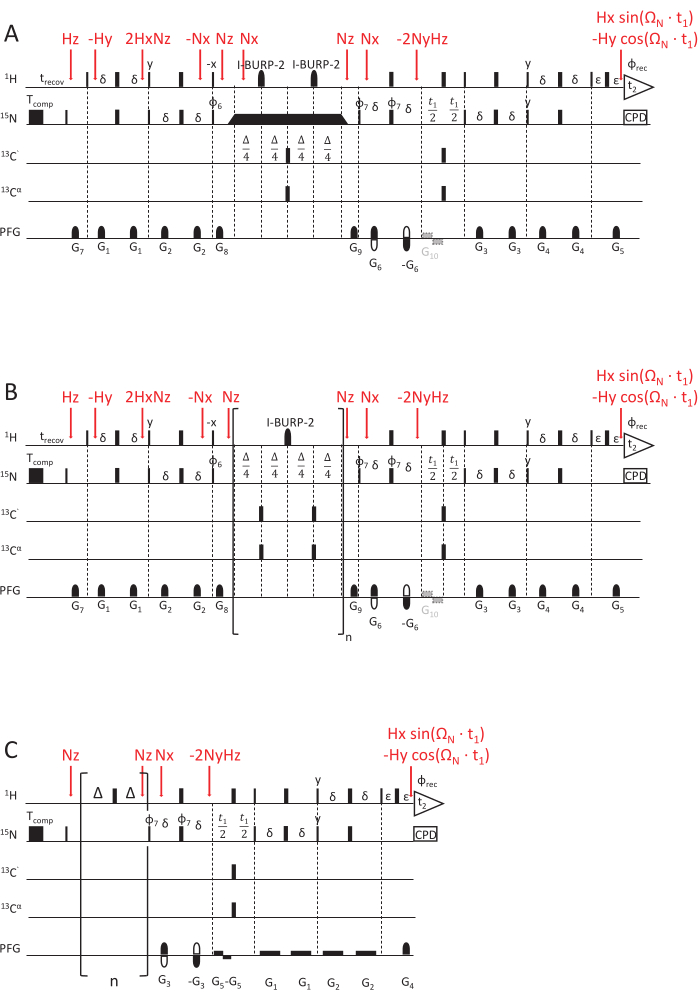
Figura 2: Schemi di sequenza di impulsi NMR per determinare le costanti del tasso di rilassamento NMR. (A) 15N R1ρ, (B) 15N R1 e (C) esperimento hetNOE, utilizzando uno schema di lettura HSQC69,70 con sensibilità aumentata. Gli impulsi a 90°(x) sono visualizzati da rettangoli stretti e gli impulsi a 180°(x) da rettangoli larghi, se non diversamente indicato. Viene applicato il seguente ciclo di fase: φ6 = y, y, -y, -y; φ7 = y, -y, φrec = y, -y, -y, y. Il rilevamento in quadratura si ottiene invertendo la polarità del gradiente G5 e il ciclo di fase di φ7 (rilevamento eco/anti-eco). (A) Esperimento 15N R1ρ: Il rettangolo nero rappresenta lo spin-lock, per il quale la durata varia per acquisire diversi ritardi di rilassamento. I triangoli prima e dopo lo spin-lock indicano gli impulsi di forma adiabatica che allineano la magnetizzazione lungo l'asse del campo magnetico effettivo Beff. G10 è un gradiente opzionale per prevenire lo smorzamento delle radiazioni della magnetizzazione dell'acqua durante la fase di evoluzione. (B) 15N R1 esperimento: la parte tra parentesi mostra l'elemento del ciclo della sequenza, ripetuto un numero n di volte per corrispondere al ritardo di rilassamento desiderato. (C) Lo schema dell'impulso hetNOE è simile alla seconda metà degli schemi dell'impulso R1 e R1ρ, vale a dire il tempo di evoluzione t1 e l'elemento di rilevamento HSQC. Tuttavia, la magnetizzazione a 15N viene eccitata direttamente senza alcun INEPT. La saturazione della magnetizzazione del protone (per ottenere un cross-relaxation tra 1H e 15N) è ottenuta da un treno di 180 (1H) impulsi applicati per almeno 5 s. Per l'esperimento di riferimento viene applicato un ritardo al minimo della stessa lunghezza (qui, 5 s) senza alcun treno di impulsi. G5 è un gradiente opzionale per prevenire lo smorzamento delle radiazioni e l'inversione di polarità del gradiente G4, in combinazione con la fase φ7 = y, -y, -y, y, raggiunge il rilevamento in quadratura. Le fasi di trasferimento della magnetizzazione, rappresentate dagli operatori del prodotto, sono contrassegnate in rosso. Clicca qui per visualizzare una versione più grande di questa figura.
La costante di velocità di rilassamento R2 descrive il rilassamento della polarizzazione trasversale (nel piano xy ortogonale al campo magnetico esterno) dovuta alla perdita di coerenza di fase tra lo spin, che porta al decadimento della magnetizzazione rilevabile53,54. La funzione di densità spettrale alle alte e piccole frequenze contribuisce a R2, simile a R1. Tuttavia, il contributo maggiore a R2 proviene dalla densità spettrale a frequenza zero. Per questo motivo, R2 è molto sensibile al tumbling rotazionale, descritto dal tempo di correlazione rotazionale τR, che è dell'ordine di pochi ns per una piccola proteina globulare a temperatura ambiente. Pertanto, i movimenti più lenti della spina dorsale nell'ordine delle centinaia di ps e del regime di ns basso contribuiscono maggiormente. Le dinamiche di scambio della spina dorsale che causano una modulazione della parte isotropa del tensore di spostamento chimico dei nuclei 15N, in linea di principio, aggiungono un contributo di scambio R2ex alle costanti di velocità R2 43,44,49,60,61. Negli esperimenti descritti, il contributo di R2ex è soppresso da un blocco di spin che rifocalizza la dinamica più lentamente della frequenza circolare inversa del blocco di rotazione. Lo spin-lock è un impulso a radiofrequenza a onda continua lunga che mantiene la magnetizzazione allineata lungo l'asse del campo magnetico effettivo Beff (la somma vettoriale del campo di spin-lock ω1 e lo spostamento chimico sfalsato dalla frequenza portante di 15N (vedi sotto)). Il rilassamento della magnetizzazione allineata lungo l'asse B1,eff è chiamato rilassamento R1ρ, che ha una componente R1 e una componente R2. Utilizzando la formula (1), R2 può essere calcolato da R1ρ e R144,73:
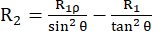 (1).
(1).
L'angolo tra l'asse del campo magnetico effettivo Beff e il campo magnetico esterno B0 è 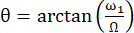 . ω1 è l'ampiezza RF dello spin-lock e Ω lo spostamento chimico tra lo spostamento chimico di 15N del residuo corrispondente e la frequenza portante di 15N 44,73.
. ω1 è l'ampiezza RF dello spin-lock e Ω lo spostamento chimico tra lo spostamento chimico di 15N del residuo corrispondente e la frequenza portante di 15N 44,73.
Lo schema degli impulsi R1ρ (Figura 2A, 70) è molto simile allo schema 15N R1 tranne che per il ritardo di rilassamento. Per misurare i tassi di rilassamento di 15N R1ρ , lo spin-lock deve essere attivo dopo che la magnetizzazione è stata allineata lungo l'asse di campo efficace Beff da un impulso adiabatico con la stessa ampiezza di radiofrequenza (RF) dello spin-lock. La lunghezza dello spin-lock verrà variata per ottenere i diversi ritardi di rilassamento.
L'effetto Overhauser nucleare allo stato stazionario {1H}-15N (1 H-15N NOE), soprannominato hetNOE in seguito, è il rapporto tra il tasso di rilassamento incrociato e il tasso di rilassamento longitudinale di 15N. Porta ad una riduzione della polarizzazione allo stato stazionario su 15N a causa del cross-rilassamento con il protone alla saturazione della polarizzazione del protone 45,53,54,74,75. Il cross-rilassamento dipende dalle funzioni di densità spettrale della somma e della differenza delle frequenze di Larmor 1H e 15N. Pertanto, l'hetNOE è sensibile sia alla dinamica dei picosecondi veloci (< 100 ps) che alla dinamica ps-ns (a causa della sua dipendenza da R1). La sequenza69 (Figura 2C) si basa su una lettura HSQC con gradienti Echo/Anti-Echo per il rilevamento in quadratura. Per la saturazione della magnetizzazione del protone e l'hetNOE risultante, la magnetizzazione del protone di equilibrio viene invertita e successivamente saturata da impulsi a impulsi di 180° per circa 5 volte il 15 N T1. Per l'esperimento di riferimento, il ritardo di recupero è uguale al ritardo di saturazione ma senza il treno di impulsi di 1ora a 180°. Viene aggiunto un ritardo extra di D1 = 2 s per l'esperimento di riferimento e quello con saturazione di 1H. Entrambi gli esperimenti sono registrati uno dopo l'altro e differiscono solo per l'applicazione di impulsi di 1ora e 180° (saturazione) o meno (riferimento). Il rapporto tra le intensità spettrali registrate nell'esperimento con una saturazione di 1H diviso per le intensità dell'esperimento di riferimento (senza il treno di impulsi di protoni a 180°) dà il valore {1H}-15N NOE (hetNOE).
Il seguente protocollo descrive l'impostazione degli esperimenti di rilassamento NMR di Lakomek et al.69 e Stief et al.70. Ci concentriamo sulle sequenze di impulsi NMR utilizzando uno schema di rilevamento HSQC con sensibilità migliorata. Gli esperimenti 15N R1 e R1ρ sono implementati come descritto in dettaglio da Stief et al.70, e l'esperimento hetNOE è descritto da Lakomek et al.69.
Access restricted. Please log in or start a trial to view this content.
Protocollo
1. Preparazione del campione NMR
NOTA: La marcatura isotopica delle proteine viene eseguita per esperimenti NMR di dimensione superiore e NMR avanzati. Quando l'espressione proteica in E. coli e la purificazione delle proteine sono state stabilite utilizzando terreni ricchi (ad esempio, Luria-Bertani [LB] o 2x estratto di lievito triptone [2YT]) con una resa di diversi milligrammi per litro, la preparazione di un campione NMR marcato isotopicamente è di solito relativamente semplice.
- Per la marcatura degli isotopi, utilizzare terreni minimi M9 per l'espressione genica, dove il cloruro di ammonio 15N è l'unica fonte di azoto e il glucosio arricchito con 13C è l'unica fonte di carbonio.
- Per la purificazione delle proteine, seguire il protocollo standard stabilito in laboratorio per la rispettiva proteina.
NOTA: La sezione dei risultati mostra risultati rappresentativi per una IDP, la proteina monomerica SNARE Sinaptobrevina-2 (1-96). La marcatura isotopica NMR di Synaptobrevin-2 è stata eseguita come descritto in70. Per misure di altissima precisione, si consiglia di utilizzare basse concentrazioni di D2O, inferiori al 5%. Ciò è dovuto all'effetto isotopico del deuterio, che può essere osservato negli esperimenti di dispersione di rilassamento CPMG 15N T2 di Hahn-Echo e 15N CPMG a bassa frequenza (non discussi qui) ma ha solo un impatto minimo negli esperimenti di 15N R1ρ (dove è principalmente rifocalizzato)76.
2. Preparativi per l'esecuzione degli esperimenti di rilassamento NMR sullo spettrometro
NOTA: Gli esperimenti di rilassamento NMR descritti sono specifici per gli spettrometri Bruker. Sono stati testati su sonde criogeniche e a temperatura ambiente a tripla risonanza da 1H, 15N e 13C e console Avance III e Avance Neo gestite dal software Bruker Topsin 3.6 o superiore.
- Seguire le precauzioni.
- Fai un briefing sulla sicurezza prima di lavorare allo spettrometro NMR.
- Controllare le specifiche di potenza della sonda dello spettrometro utilizzato e scegliere una lunghezza e una potenza dell'impulso appropriate per ogni impulso, ad esempio confrontando la coerenza con le voci della tabella "prosol" Topspin.
- Importare le sequenze di impulsi.
- Scaricare i programmi a impulsi NMR dal sito Web indicato di seguito o dal repository esteso della Banca di Risonanza Magnetica Biologica (BMRB) (bmrbig102) nella directory /opt/topspin4.1.4/exp/stan/nmr/lists/pp/user del computer che utilizza lo spettrometro NMR. Si prega di regolare la versione topspin (qui: topspin4.1.4 alla rispettiva versione installata sullo spettrometro).
- Scaricare gli impulsi a forma di NMR dal sito Web indicato di seguito o dal repository esteso della Banca di Risonanza Magnetica Biologica (BMRB) (bmrbig102) nella directory /opt/topspin4.1.4/exp/stan/nmr/lists/pp/user del computer che utilizza lo spettrometro NMR. Si prega di regolare la versione topspin (qui: topspin 4.1.4 alla rispettiva versione installata sullo spettrometro).
- Calibrare lo spettrometro e il campione.
- Impostare la temperatura di interesse. Per la calibrazione della temperatura, applicare i protocolli della struttura. Ad esempio, utilizzare un campione di metanolo-d4 e interpretare la differenza di spostamento chimico osservata utilizzando l'applicazione web NMR termometro (https://chem.ch.huji.ac.il/nmr/software/thermometer.html); Vedi anche77.
- Inserire il campione (più un ulteriore 5% D2O per il blocco) e attendere circa 10 minuti per consentire al campione di equilibrare la sua temperatura. Bloccare il segnale del deuterio (2H) (comando: blocco, selezionare H2O+D2O).
- Abbina e sintonizza i canali 1H, 15N e 13C, ad esempio utilizzando la routine atma o atmm. Se non è installata alcuna unità di abbinamento e sintonizzazione automatica, abbinare e sintonizzare manualmente la sonda utilizzando il comando wobb.
- Eseguire lo shimming per un campo B0 omogeneo, ad esempio utilizzando la routine topshim tg.
- Calibrare la lunghezza dell'impulso di 90° (1H).
- Usa l'esperimento zg. Utilizzare getprosol per importare i parametri della sonda. Scegliere una breve durata di 5 μs per P1. Se necessario, controllare e correggere il livello di potenza specificato della sonda per PLW1.
- Inizia l'esperimento con zg. Elabora i dati con efp. Regolare la fase con .ph in modo che la linea di base sia uniforme e il segnale NMR della risonanza dell'acqua sia in fase.
- Stimare un impulso a 360° (1ora) prendendo un impulso comune o approssimativo a 90° (1ora) (ad esempio, dalle specifiche dello spettrometro elencate) e moltiplicandolo per un fattore quattro. Eseguire l'esperimento ed elaborarlo.
NOTA: La durata scelta è troppo lunga se il segnale è positivo e troppo breve se negativa. L'impulso a 360° (1H) sarà il passaggio per lo zero. - Regolare la lunghezza dell'impulso di conseguenza. Fallo in modo iterativo per trovare il passaggio per lo zero. Dividi il valore ottenuto per quattro. Questo sarà l'impulso a 90° (1H).
NOTA: Un'alternativa al metodo a 360° descritto è la routine pulscal , che può essere avviata dalla riga di comando e consente una calibrazione automatica degli impulsi di 1ora.
- Calibrare la frequenza portante O1 alla risonanza dell'acqua.
- Copia l'esperimento zg digitando edc nella riga di comando con un nuovo numero di esperimento. Digitare rpar per aprire i set di parametri (Figura 1A supplementare).
- Cercare il set di parametri ZGPR. Fare clic sul set di parametri ZGPR (Figura supplementare 1A), fare clic su Leggi, scegliere Esegui "getprosol" e fare clic su OK (Figura supplementare 1B).
- Modificare P1 con la lunghezza dell'impulso calibrata di 90° (1H). Scegliere una lunghezza di saturazione di D1 = 2 s e una potenza di impulso di PLW9 di 50 dB. Impostare il guadagno del ricevitore (rg) su 16. In alternativa, eseguire la regolazione del guadagno del ricevitore digitando rga e impostando il guadagno del ricevitore sul valore specificato (ma evitare "overflow del guadagno del ricevitore").
- Digitare gs nella riga di comando per avviare un'acquisizione ripetuta e continua. Andare alla finestra di acquisizione e modificare l'O1 per ridurre l'area FID dominata dal segnale dell'acqua (Figura supplementare 2).
- Fare clic sotto l'indicatore per diminuire l'O1 e fare clic sopra l'indicatore per aumentarlo. Utilizzare il pulsante della sensibilità per modificare la dimensione del passo. Salva l'O1 corrente; Close interrompe l'acquisizione ripetuta (Figura 2 supplementare).
- Calibrare la lunghezza dell'impulso di 90° (15N).
- Copiare l'esperimento zg in un nuovo numero di esperimento (digitare edc). Digitare rpar per leggere il set di parametri HSQCFPF3GPPHWG per modificare il programma di impulsi in un esperimento HSQC 1 H-15N. Utilizzare l'opzione Mantieni parametri P1, O1 e PLW1 (Figura supplementare 1).
- Digitare getprosol 1H 1H) di lunghezza dell'impulso in μs> <90° (1H) di intensità dell'impulso in db> nella riga di comando per importare i parametri della sonda. Impostare il numero di scansioni (NS) su 8 e le scansioni fittizie (DS) su 2.
NOTA: < > specifica qui i valori da impostare. Ad esempio: getprosol 1H 8.5 -11.79. - Esegui il primo incremento dell'esperimento digitando zg nella riga di comando. Copia il primo incremento in un nuovo numero dell'esperimento con rser 1 . Elabora i dati con efp e sfasa lo spettro con .ph. Utilizzare il primo incremento (esperimento 1D) dell'esperimento per i passaggi seguenti.
NOTA: Il < > specificare il numero scelto per il nuovo esperimento, ad esempio rser 1 101. - Digitare ased nella riga di comando per aprire i parametri della sequenza di impulsi dell'esperimento 1D. Fare clic sulla E nella riga PULPROG (vedere la Figura supplementare 3A) per aprire l'editor del programma a impulsi.
- Utilizzare il seguente flusso di lavoro per salvare la sequenza di impulsi con un nuovo nome: fare clic su File (angolo in alto a sinistra), Salva con nome, immettere il nuovo nome e fare clic su OK. Quindi fare clic su Imposta PULPROG sul set di dati (vedere la Figura 3B supplementare) e fare clic su OK.
- Modificare la sequenza originale 1 H-15N HSQC (salvata con un nuovo nome, vedere 2.6.4.) moltiplicando i 90° (15N) dopo il periodo di evoluzione t1 (prima del trasferimento all'indietro) per un fattore 2 (l'impulso di 90° (15N) è ora 180° (15N)). Premere Ctrl S per salvare la sequenza di impulsi. Impostare la lunghezza dell'impulso a 90° (15N) (P21) in modo che il segnale NMR del primo incremento svanisca (al passaggio per lo zero) (Figura supplementare 3B).
- Aprire l'esperimento HSQC 1 H-15N (esperimento 2D) e inserire la lunghezza dell'impulso calibrata di 90° (15N) (P21). Digitare getprosol 1H 1H) lunghezza dell'impulso in μs> <90° (1H) intensità dell'impulso in db> 15N 15N) lunghezza dell'impulso in μs> <90° (15N) intensità dell'impulso in db> nella riga di comando.
- Determinare le larghezze spettrali e l'appropriazione dei tempi di acquisizione.
- Nella finestra dei parametri di acquisizione per l'esperimento HSQC 1 H-15N (digitare eda nella riga di comando), regolare l'ampiezza spettrale (parametro di acquisizione SW) e il numero di incrementi dimensionali in dimensioni dirette e indirette (parametro di acquisizione TD).
- Scegliere per SW, 16 ppm nella dimensione diretta (1H) e 30 ppm in quella indiretta (15N). Scegliere per TD, 2048 nella dimensione diretta (1H) e 512 in quella indiretta (15N).
- Acquisisci l'esperimento HSQC 1 H-15N.
- Verificare che tutti i segnali di interesse vengano rilevati e che non siano presenti artefatti di aliasing nello spettro. Cambiare l'O3P se il centro dello spettro non si adatta al centro della distribuzione del segnale proteico lungo l'asse 15N.
- Aumentare il SW se la finestra spettrale è troppo piccola. Aumentare il TD per ottenere una risoluzione FID più elevata (controllare le specifiche della sonda per la durata massima del disaccoppiamento).
NOTA: Questa è la configurazione generale e il punto di partenza per l'impostazione degli esperimenti di rilassamento NMR. Si raccomanda di registrare l'esperimento HSQC 1 H-15N all'inizio e alla fine dell'intera serie di esperimenti e, per tempi sperimentali più lunghi, anche tra un esperimento e l'altro per monitorare l'integrità del campione.
- Nella finestra dei parametri di acquisizione per l'esperimento HSQC 1 H-15N (digitare eda nella riga di comando), regolare l'ampiezza spettrale (parametro di acquisizione SW) e il numero di incrementi dimensionali in dimensioni dirette e indirette (parametro di acquisizione TD).
3. Implementazione degli esperimenti di rilassamento NMR
NOTA: Le sequenze di impulsi di rilassamento NMR (Figura 2) sono disponibili presso https://www.ipb.hhu.de/en/teams/team-lakomek/pulsesequences o presso il repository esteso della Banca di Risonanza Magnetica Biologica (BMRB) (bmrbig102).
- Impostare il 15N R1ρ rsperimento.
- Copiare l'esperimento HSQC (digitare edc) in una nuova directory. Digitare ased e modificare il programma di impulsi nell'esperimento 15N R1ρ facendo clic sui tre punti della linea di parametri PULPROG nei parametri della sequenza di impulsi (ased, vedere la Figura supplementare 3A). Fare doppio clic sul programma a impulsi, fare clic su Imposta PULPROG sul set di dati (vedere la Figura 3B supplementare) e fare clic su OK.
NOTA: Potrebbero essere visualizzati alcuni messaggi di errore. Chiudi questi messaggi. - Digitare gpnam nella riga di comando per inserire tutte le forme di gradiente mancanti (gpnam1- gpnam9) come descritto nella sequenza di impulsi (Figura 4 supplementare).
- Digita vplist nella riga di comando. Fare clic sui tre punti (Figura supplementare 5A), File (angolo in alto a sinistra) e Nuovo (Figura supplementare 5B).
- Digitare un nome per una nuova vplist fittizia e premere OK. Crea la vplist fittizia (5m, 65m, 25m, 45m, 15m, 55m, 35m, 10m; una nuova riga per ogni voce vplist). Premere Ctrl S per salvare l'elenco e chiudere la finestra della vplist (Figura supplementare 5C).
- Fare doppio clic sul nome della vplist appena creata (Figura supplementare 5B).
NOTA: Ciò è necessario per evitare errori di compilazione nella sequenza di impulsi. Le voci appropriate della vplist saranno determinate più avanti (punto 3.1.29.). - Digitare ased per aprire i parametri della sequenza di impulsi (Figura 6 supplementare). Inserire tutte le intensità e le lunghezze del gradiente mancanti come indicato nella sequenza di impulsi (Figura 6D supplementare).
- Inserire per il contatore di loop L3 (numero di punti complessi nella dimensione indiretta) 1/2 della voce 15N TD nella finestra dei parametri di ripresa (eda). Inserire per il contatore di loop L6 il numero di punti dati di rilassamento registrati utilizzati per l'adattamento della curva di rilassamento.
NOTA: Questo numero L6 corrisponde al numero di voci vplist, qui: 8 (Figura 6A supplementare). - Impostare l'impulso adiabatico TanhTan_half.nl, scaricato dal sito Web sopra specificato, come forma dell'impulso 15N SP8. Impostare l'impulso adiabatico TanhTan_2nd.nl dal sito Web come la forma dell'impulso 15N SP9 (Figura supplementare 6C).
- Impostare l'ingresso P8 alla lunghezza dell'impulso adiabatico TanhTan - sufficientemente lungo da garantire l'adiabaticità dell'impulso; qui si sceglie P8 = 3000 μs (Figura supplementare 6C).
- Impostare il disaccoppiamento 15N (CPDPRG 3) su uno schema di disaccoppiamento garp. Utilizzare per la lunghezza del disaccoppiamento PCPD3 200 μs (Figura supplementare 6C).
- Impostare il ritardo di recupero dell'interscansione su almeno D1 = 2 s o più.
NOTA: Per la scelta di un ritardo di recupero appropriato, vedere la sezione Discussione. - Impostare le scansioni fittizie (DS) su almeno 64 per l'esperimento finale (durante la configurazione, DS = 4 è sufficiente per motivi di tempo). Come punto di partenza, utilizzare un numero di scansioni (NS) di 4; utilizzare multipli di 4 se il rapporto segnale/rumore (SNR) è troppo basso (Figura 6A supplementare).
- Impostare l'O1 sulla frequenza portante calibrata del passaggio 2.5. Impostare l'O2P a 176 ppm e copiare l'O3P dall'esperimento HSQC 1 H-15N (Figura supplementare 6A-C).
- Impostare la lunghezza dell'impulso P7 sulla lunghezza dell'impulso di 90° (15N) calibrata al punto 2.6. Inoltre, copiare il livello di potenza dell'impulso a 90° (15N) dal passaggio 2.6 a PLW3 e PLW7 (Figura 6C supplementare).
- Impostare la lunghezza dell'impulso P1 e P19 sulla lunghezza dell'impulso di 90° (1H) calcolata al punto 2.4. (Figura 6B supplementare).
- Impostare il numero di incrementi nella dimensione indiretta TD = l3 * 2 * l6 (eda o digitare td nella riga di comando).
NOTA: Tutti i parametri della sequenza di impulsi qui descritti sono ulteriormente annotati nella sequenza di impulsi. - Impostare l'impulso di forma SP5 su una forma I-BURP2 (Iburp2.1000). Impostare la lunghezza dell'impulso P15 su 2000 μs. Aprire la visualizzazione dello strumento forma facendo clic sulla E accanto all'impulso di forma I-BURP2 nella finestra dei parametri della sequenza di impulsi Topspin (ased) (Figura 6B supplementare).
- Simulare l'impulso di forma con la lunghezza corretta facendo clic sul pulsante Avvia simulazione NMR. Controllare la lunghezza dell'impulso sagomato [μs] (= 2000 μs) e l'angolo di rotazione [°] (= 180°) nella finestra di simulazione. Fare clic su Avvia NMR-SIM (Figura supplementare 7A).
- Controllare l'intervallo di eccitazione e scegliere la lunghezza dell'impulso I-BURP2 appropriata per coprire la dispersione spettrale di 1H della proteina, ma allo stesso tempo evitare l'eccitazione dell'acqua. Impostare P15 sulla lunghezza dell'impulso sagomato della finestra di simulazione con il miglior impulso IBURP-2 simulato (Figura 7B supplementare).
NOTA: La lunghezza corretta viene scelta se l'I-BURP2 eccita (> un'efficacia del 95%) l'intera regione ammidica. Per simulare un impulso I-BURP2 con una lunghezza diversa, chiudere la finestra del profilo di eccitazione e modificare la lunghezza dell'impulso sagomato nella finestra di simulazione. Fare clic su Avvia NMR-SIM. Gli impulsi con una lunghezza inferiore hanno un intervallo di eccitazione più ampio in Hz. - Impostare SPOFFS5 per modificare la frequenza portante dell'impulso I-BURP2 e spostare l'intervallo di eccitazione a sinistra o a destra per evitare che l'impulso I-BURP2 tocchi e disturbi la magnetizzazione dell'acqua (Figura 6B supplementare).
- Per determinare il livello di potenza appropriato dell'impulso sagomato, aprire lo strumento Forma Bruker, fare clic sul pulsante Avvia simulazione NMR e impostare la lunghezza dell'impulso I-BURP2 su Lunghezza dell'impulso sagomato [μs]. Ricordare la lunghezza corrispondente di un impulso rettangolare morbido a 90° (1H), visibile nella finestra di simulazione al corrispondente impulso quadrato di 90° [μs] (Figura 7A supplementare).
- Tipo calcpowlev. Utilizzare l'impulso duro a 90° (1H) (P1) in microsecondi come lunghezza di riferimento (Figura supplementare 8B) e l'impulso rettangolare morbido a 90° (1H) corrispondente all'impulso I-BURP2 in microsecondi come nuova lunghezza (Figura supplementare 8A). Ricordare la differenza di potenza Δ in dB tra l'impulso duro a 90° (1H) e l'impulso rettangolare morbido (Figura supplementare 8C). Copiare l'intensità dell'impulso duro a 90° (1H) in dB su SPW5 e aggiungere la differenza memorizzata Δ in dB (Figura 6B supplementare).
- Per determinare il livello di potenza dello spin-lock, calcolare la corrispondente lunghezza dell'impulso di 90° (15N) dello spin-lock. Utilizzare la formula: lunghezza dell'impulso di 90° (15N) in s = 0,25 / (potenza di spin-lock in Hz).
- Utilizzare calcpowlev per calcolare la differenza di potenza Δ in dB tra la potenza dello spin-lock e l'impulso forte di 90° (15N) (P7). Novità: lunghezza dell'impulso di 90° (15N) dello spin-lock in μs (Figura supplementare 8A). Rif: lunghezza dell'impulso dura a 90° (15N) in μs (Figura supplementare 8B). Copiare il livello di potenza PLW7 dell'impulso duro a 90° (15N) in dB nel livello di potenza di spin-lock PLW8 (Figura 6C supplementare) e aggiungere la differenza di potenza calcolata Δ in dB (Figura 8C supplementare).
- Copiare il livello di potenza dello spin-lock PLW8 nei livelli di potenza SPW8 e SPW9. (Figura supplementare 6C).
- Per determinare il livello di potenza del disaccoppiamento a 15N, utilizzare calcpowlev per calcolare la differenza di potenza Δ tra la potenza dell'impulso di disaccoppiamento a 90° (15N) (PCPD3) e l'impulso duro a 90° (15N) (P7). Novità: PCPD3 in μs (200) (Figura supplementare 8A). Rif: lunghezza dell'impulso dura a 90° (15N) in μs (Figura supplementare 8B). Copiare il livello di potenza PLW7 dell'impulso duro a 90° (15N) in dB al livello di disaccoppiamento PLW31 (Figura 6C supplementare) e aggiungere Δ in dB (Figura 8C supplementare).
- Per utilizzare la compensazione della temperatura, includere #define TEMP_COMPENSATION (eliminando il punto e virgola nella sequenza di impulsi). Impostare P18 sulla durata massima dello spin-lock utilizzato nell'esperimento 15N R1ρ .
NOTA: Si consiglia di disattivare la compensazione della temperatura per la prima prova dell'esperimento. - Nel caso di campioni marcati con 15N 13C, includere # per definire LABEL_CN nel programma a impulsi (eliminando il punto e virgola nella sequenza a impulsi). Impostare P4 sulla lunghezza dell'impulso calcolata con la formula in78. Ad esempio, corrisponde a 23,70 μs a 600 MHz e 11,85 μs a 1200 MHz. Digitare calcpowlev per calcolare la differenza di potenza in dB tra P4 e l'impulso duro (13C). Copiare la potenza dell'impulso duro (13C) in dB in PLW4 e aggiungere il Δ in dB. Copiare PLW4 in PLW2 (Figura 6B supplementare).
- Determinare i ritardi di rilassamento per un campionamento appropriato.
- Esegui i primi otto FID (nel caso L6 = 8) ed elaborali (rser < numero del nuovo esperimento>). Scegli le voci nella vplist in modo che l'intensità di picco dell'esperimento con il ritardo più lungo diminuisca ad almeno 1/e (circa il 37%), ma non meno del 25% rispetto all'esperimento con il ritardo più breve (che funge da riferimento).
- Regolare le voci rimanenti della vplist per ottenere un campionamento uniforme delle diminuzioni di intensità tra il FID con il ritardo più lungo e il FID con il ritardo più breve. Leggere la NOTA e le specifiche di alimentazione della sonda.
NOTA: I residui che si rilassano lentamente domineranno il primo FID. Regolare le voci rimanenti della vplist per ottenere un campionamento uniforme delle diminuzioni di intensità tra il primo e il secondo FID. Non superare la potenza e la lunghezza massima consentita per la rotazione della sonda (consultare il manuale della sonda e le specifiche della sonda). In caso di compensazione della temperatura, controllare P18 (durata massima del blocco della rotazione), poiché verrà applicata la durata complessiva del blocco della rotazione di P18, suddivisa tra il ritardo di rilassamento e la compensazione della temperatura. Mantiene il tempo (ritardo di rilassamento) + il tempo (compensazione della temperatura) = const = P18. Per uno spin-lock a 2 kHz, preferiamo non superare una durata dello spin-lock superiore a 65 ms, utilizzando un ritardo di recupero (D1) di 2 s. In tal caso, è necessario adottare un compromesso tra la durata massima dello spin-lock e il ritardo di rilassamento più lungo a favore della durata massima consentita dello spin-lock. In caso di ritardi di recupero D1 più lunghi, potrebbero essere applicabili potenze di spin-lock più lunghe. Ciò che conta è la deposizione complessiva di potenza nella sonda nel tempo. Se sono necessarie durate di spin-lock più lunghe, aumentare le lunghezze di spin-lock solo a piccoli passi e osservare il segnale di blocco. Come regola generale, regola il livello di blocco nel terzo superiore del display del lucchetto. Se il livello di blocco si riduce di più di due quadrati durante l'esecuzione della sequenza di impulsi, la potenza di spin-lock depositata nella sonda è eccessiva.
- Digita rga nella riga di comando per determinare il guadagno del ricevitore. Per un buon rapporto segnale/rumore, puntare a un valore di guadagno del ricevitore (rg) compreso tra 128 e 256, che dimostra una buona soppressione dell'acqua.
NOTA: Un guadagno del ricevitore compreso tra 128 e 256 non può essere raggiunto indica un segnale dell'acqua troppo forte. In tal caso, la potenza del gradiente e la durata vengono regolate delicatamente fino a raggiungere il guadagno del ricevitore. Vedi anche la sezione Discussione. - Avviare un test dell'esperimento 15N R1ρ digitando zg nella riga di comando. Verificare che il segnale dell'acqua sia ben soppresso per tutti i ritardi (da 1 a 8, in caso di 8 ritardi nella vplist). Controllare anche l'incremento 9, che è il secondo incremento dello schema di rilevamento in quadratura (Eco/Anti-Eco).
- Copiare l'esperimento HSQC (digitare edc) in una nuova directory. Digitare ased e modificare il programma di impulsi nell'esperimento 15N R1ρ facendo clic sui tre punti della linea di parametri PULPROG nei parametri della sequenza di impulsi (ased, vedere la Figura supplementare 3A). Fare doppio clic sul programma a impulsi, fare clic su Imposta PULPROG sul set di dati (vedere la Figura 3B supplementare) e fare clic su OK.
- Imposta l'esperimento 15N R1 .
- Copiare l'esperimento 15N R1ρ e cambiare il programma di impulsi nell'esperimento 15N R1 .
- Digitare vclist, generare un vclist e immettere il dummy vclist descritto in70: 0, 24, 6, 20, 4, 16, 8, 12; una nuova riga per ogni voce vclist).
- Aperto ased. Controllare le lunghezze e le intensità del gradiente. Cambia GPZ9 al 14%.
- Controllare la lunghezza dell'impulso, la forma dell'impulso e l'intensità dell'impulso (i valori corretti dovrebbero essere stati copiati dall'esperimento 15N R1ρ ).
- Nel caso di campioni marcati con 15N13C, utilizzare #define LABEL_CN nel programma a impulsi.
- Per l'uso della compensazione della temperatura, includere #define TEMP_COMPENSATION.
- Esegui i primi otto FID (nel caso L6 = 8) ed elaborali (rser < numero del nuovo esperimento>). Scegliere le voci nella vclist in modo che l'intensità di picco dell'esperimento con il ritardo più lungo diminuisca ad almeno 1/e (circa il 37%) ma non meno del 25% rispetto all'esperimento con il ritardo più breve. Regolare le voci rimanenti della vplist per ottenere un campionamento uniforme delle diminuzioni di intensità tra il FID con il ritardo più lungo e il FID con il ritardo più breve.
NOTA: Sono consentiti solo numeri pari per le voci vclist.
- Impostare l'esperimento {1H}-15N NOE.
- Copiare l'esperimento di rilassamento 15N R1 e modificare la sequenza di impulsi nello schema di impulsi {1H}-15N NOE.
- Impostare la durata del periodo di saturazione con il contatore di loop L8. La spaziatura tra gli impulsi di 180° (1H) è di 22 ms79 (2* 1 / JNH). Per ottenere i risultati più accurati, utilizzare un periodo di saturazione di circa 5 volte i 15N T1(15N) della proteina. Qui: 5 s. Ciò corrisponderebbe a L8 = 230.
NOTA: La sezione Discussione fornisce ulteriori consigli sulla scelta del periodo di saturazione/ritardo di recupero. - Modificare la lunghezza e l'intensità dei gradienti come descritto nello schema degli impulsi. Usa le forme sfumate sinusoidali.20.
- Impostare il numero di incrementi nella dimensione indiretta uguale a TD = l3 * 2 * 2
- Nel caso di campioni marcati con 15N13C, utilizzare #define LABEL_CN e -DLABEL_CN come ZGOPTNS.
- Esegui gli schemi a impulsi digitando zg nella riga di comando.
4. Elaborazione e analisi degli esperimenti NMR registrati
NOTA: Gli spettri sono stati registrati utilizzando un sistema Bruker. L'elaborazione viene eseguita utilizzando un sistema operativo Unix o Linux. L'elaborazione degli spettri e l'analisi dei dati sono state eseguite utilizzando NMRPipe80 e python3. Il software NMRPipe può essere scaricato all'indirizzo https://www.ibbr.umd.edu/nmrpipe/index.html. Gli script di elaborazione basati su NMRPipe possono essere scaricati dal sito web: https://www.ipb.hhu.de/en/teams/team-lakomek/pulsesequences o dal repository esteso Biological Magnetic Resonance Bank (BMRB) (bmrbig102). Si consiglia di utilizzare NMRPipe. Se NMRPipe non è disponibile o desiderato, è possibile utilizzare alternative come CCPN81 o SPARKY (SPARKY 3, o i suoi successori NMRFAM-SPARKY82 o POKY83 ).
- Elaborazione e analisi 15N R1 rsperimento.
NOTA: I seguenti comandi vengono eseguiti in un terminale Unix.- Copiare i file nella directory scaricata R1_processing nella directory contenente i dati R1 sperimentali (directory di lavoro).
- Rendi i file eseguibili dal comando del terminale: > chmod u+x *.com
- Eseguire > bruker per aprire l'utilità di conversione NMRPipe e generare il file fid.com. Fare clic su Leggi parametri. Impostate su due il conteggio delle quote se vengono contate tre dimensioni, quindi fate nuovamente clic su Leggi parametri (Read Parameters ). Fare quindi clic su Cancella script, Aggiorna script e Salva script (Figura 9 supplementare).
NOTA: Se la frequenza portante non è corretta, modificare la posizione centrale PPM dopo aver fatto clic su Leggi parametri la seconda volta. - Esegui > ./fid.com
- Converti la vclist in una vplist con ms voci: ritardo in ms = 40 ms * voce di vclist. Utilizzare > cp vclist vplist e modificare la vplist.
- Utilizzare > ./split.com per dividere i dati pseudo 3D in otto file 2D.
- Modificare i valori tauValues (valori vplist nell'ordine della vplist) e i valori idxExpmt (ordine dei tauValues relativi all'ora) in nmrproc.com (Figura 10 supplementare).
- Utilizzare > ./nmrproc.com per elaborare i dati grezzi di tutti gli otto spettri 2D.
- Utilizzare > nmrDraw per disegnare gli spettri.
- Utilizzare lo spettro di riferimento (prima voce in vplist) per questi passaggi. Passa a quello spettro modificando la dimensione z a uno (angolo in alto a sinistra). Regolare il livello del contorno utilizzando i tasti + e - accanto al pulsante Fattore . Ridisegna lo spettro con il pulsante Disegna.
- Utilizzare un livello di contorno in cui tutti i segnali di interesse possono essere riconosciuti con solo pochi segnali provenienti dal rumore (il livello di contorno è al limite del livello di rumore) (Figura 11 supplementare).
- Correggere la fase dello spettro, digitare v per la proiezione verticale del segnale 1D e h per la proiezione orizzontale del segnale 1D. Regolare la fase con P0 e P1 (Figura supplementare 11), ricordare la differenza di fase e correggere la fase nel file nmrproc.com (Figura supplementare 10).
NOTA: Se lo spettro è invertito nella dimensione 15N, rimuovere il -neg nella riga: | nmrPipe -fn FT -neg \ (Figura supplementare 10). - Chiudi nmrDraw ed elabora di nuovo con > nmrproc.com. Per il prelievo dei picchi (dopo che lo spettro è stato sfasato correttamente e lo spettro di riferimento è stato visualizzato con un livello di contorno in cui sono presenti tutti i picchi), digitare K (Maiusc + k), premere Rileva e fare clic su Salva. (Figura supplementare 11).
- Copiare un elenco di picchi nella directory di lavoro e modificare -assName in ass.com con il nome dell'elenco di picchi (Figura 12 supplementare).
NOTA: Un esempio di lista dei picchi nel formato corretto è disponibile all'indirizzo https://www.ipb.hhu.de/en/teams/team-lakomek/pulsesequences
e presso il deposito esteso della Banca di Risonanza Magnetica Biologica (BMRB) (bmrbig102). - Eseguire > ./ass.com per assegnare gli spettri. Verificare l'assegnazione che lo script suggerisce per i vari picchi. Utilizzare il tasto sinistro del mouse per assegnare e il tasto destro del mouse per deassegnare i picchi. Utilizzare il pulsante Avanti per passare all'amminoacido successivo e Indietro per passare all'amminoacido precedente (Figura 13 supplementare).
NOTA: Il livello del contorno può essere modificato con + e - nell'angolo in alto a destra. Utilizzare il pulsante Disegna per ridisegnare lo spettro con il nuovo livello di contorno (Figura supplementare 13). - Dopo aver completato l'assegnazione, fare clic su Salva e quindi su Salva di nuovo. Successivamente, fare clic su Esci, Annulla ed Esci (Figura 13 supplementare).
- Esegui > python3 NoneDeleter.py per creare relax.tab, che contiene solo informazioni sui picchi assegnati.
- Eseguire > ./relax.com per generare lo script autofit.com.
- Esegui > ./autoFit.com per eseguire un'interpolazione di Fourier e generare pseudo rapporti dei diversi tempi di ritardo per ogni amminoacido (vengono generati axt.tab e nlin.tab ).
- Eseguire > ./model.com per generare lo script modelExp.com in base ai dati in nlin.tab.
- Esegui > ./modelExp.com per adattare la curva di evoluzione di ciascun residuo con un modello di ottimizzazione dei minimi quadrati non lineare.
- Esegui > bash summary.tcl -in relax.tab > t1.tab. Ignorare un potenziale messaggio di errore: GDB Avviso: possono verificarsi 21 valori mancanti nel file della tabella .
- Copiare t1.tab in una directory R1R2 appena creata.
- Elaborare e analizzare l'esperimento 15N R1ρ .
- Copiare i file nella directory scaricata R1rho_processing nella directory contenente i dati sperimentali R1ρ (directory di lavoro).
- Esegui > chmod u+x *.com
- Eseguire > bruker per aprire l'utilità di conversione NMRPipe e generare il file fid.com. Vedi anche l'elaborazione degli spettri 15N R1 .
- Esegui > ./fid.com
- Esegui > ./split.com per dividere i dati pseudo 3D in otto file 2D.
- Eseguire > ./nmrproc.com (modificare in anticipo i valori tauValues [vplist] e idxExpmt ) per elaborare i dati grezzi di tutti gli otto spettri 2D (Figura 10 supplementare).
- Esegui > nmrDraw per disegnare gli spettri e selezionare i picchi. Utilizzare lo spettro di riferimento (prima voce in vplist) per questi passaggi. Seguire la stessa procedura dell'esperimento 15N R1 .
- Copiare un elenco di picchi nella directory di lavoro e modificare -assName in ass.com con il nome dell'elenco di picchi (Figura 12 supplementare).
- Eseguire > ./ass.com e seguire la stessa procedura dell'esperimento 15N R1 .
- Esegui > python3 NoneDeleter.py per creare relax.tab, che contiene solo informazioni sui picchi assegnati.
- Esegui > ./relax.com
- Esegui > ./autoFit.com
- Esecuzione > ./model.com
- Esecuzione > ./modelExp.com
- Esegui > bash summary.tcl -in relax.tab > t1rho. tab. Ignora il potenziale messaggio di errore: GDB Avviso: potrebbero verificarsi 21 valori mancanti nel file della tabella .
- Copiare la seguente intestazione nel file t1rho.tab (i valori possono essere ottenuti dal file fid.com) (Figura 14 supplementare):
NOTA 600MHz per T1rho
NOTA -yOBS = 60.818 (metti qui il valore trovato in fid.com)
NOTA -yCAR = 119,06 (metti qui il valore trovato in fid.com) - Eseguire > ./extract_PPM.com per generare una tabella denominata fusionOffset.tab che contiene l'offset della frequenza di picco 15N rispetto alla frequenza portante (che sarà necessaria per calcolare l'angolo θ per l'estrazione dei dati R2 dai dati R1 e R1ρ ).
- Riassumi i dati sul rilassamento.
- Copiate il file t1rho.tab e il file fusionOffset.tab nella directory R1R2.
- Copia lo script crtR1p.tcl nella directory R1R2, regola la frequenza dello spettrometro e la potenza di spin-lock, ed esegui bash crtR1p.tcl | sort -n > R1R2.tab. (Figura supplementare 15). R1R2.tab conterrà le costanti di velocità R1ρ adattate, l'angolo theta, le costanti di velocità R2 e le costanti di velocità R1 .
- Elaborare e analizzare l'esperimento hetNOE.
- Copiare i file nella directory scaricata NOE_processing nella directory dei dati NOE sperimentale (directory di lavoro).
- Esegui > chmod u+x *.com
- Eseguire > bruker per aprire l'utilità di conversione NMRPipe e generare il file fid.com. Fare clic su Leggi parametri. Modificare il conteggio delle dimensioni su due se sono state contate tre dimensioni e premere nuovamente Leggi parametri . Quindi premere Cancella script, Aggiorna script e Salva script.
- Esegui > ./fid.com
- Esegui > ./split.com (prima deve essere creata una vplist con due voci (0, 1))
- Esecuzione > ./ft2.com
- Eseguire > nmrDraw per disegnare lo spettro e selezionare i picchi nello spettro di riferimento (dimensione z = 1). Estrai e ricorda il rumore (premi T = Maiusc + t). La correzione di fase e il prelievo dei picchi seguono la stessa procedura dell'esperimento 15N R1 .
- Copiare un elenco di picchi nella directory di lavoro e modificare -assName in ass.com con il nome dell'elenco di picchi (Figura 12 supplementare)
- Eseguire > ./ass.com per assegnare gli spettri. Seguire la stessa procedura descritta per l'esperimento 15N R1 .
- Esegui > python3 NoneDeleter.py per creare relax.tab.
- Esecuzione > ./script.com
- Esegui > ./autoFit.com
- Cambia il rumore in error_hetnoe.tcl.
- Esegui > bash error_hetnoe.tcl |sort -n > hetNOE.tab
Access restricted. Please log in or start a trial to view this content.
Risultati
Di seguito sono riportati alcuni dati esemplari di rilassamento NMR registrati sulla proteina vescicolare SNARE Sinaptobrevin-2 (1-96), frequentemente chiamata VAMP2 (proteina associata alla vescicola 2). Per registrare i dati NMR, abbiamo utilizzato un campione di 171 μM 15N Synaptobrevin-2 (1-96) (soprannominato Syb-2 di seguito) in 50 mM di tampone MES (pH 6.0) contenente 150 mM di NaCl, 0.1 mM di TCEP e 1 mM di EDTA. Tutti i dati sperimentali sono stati registrati a 278,1...
Access restricted. Please log in or start a trial to view this content.
Discussione
Questo protocollo descriveva l'impostazione degli esperimenti di rilassamento NMR 15N di Lakomek et al.69 e Stief et al.70. Ci siamo concentrati sulle sequenze di impulsi NMR utilizzando uno schema di rilevamento HSQC con sensibilità migliorata. Gli esperimenti 15N R1 e R1ρ sono implementati come descritto in dettaglio da Stief et al.70, e l'esperimento hetNOE è descritto da Lakomek et
Access restricted. Please log in or start a trial to view this content.
Divulgazioni
Gli autori dichiarano di non avere interessi finanziari concorrenti noti o relazioni personali che possano aver influenzato il lavoro riportato in questo articolo.
Riconoscimenti
Ringraziamo Melinda Jaspert e Kevin Bochinsky per le utili discussioni. N.L. ringrazia la Fondazione Tedesca per la Scienza per il finanziamento attraverso il Programma Heisenberg (DFG grant number 433700474). Questo lavoro è ulteriormente supportato dal progetto "Determinanti virologici e immunologici della patogenesi COVID-19 - lezioni per prepararsi alle future pandemie (KA1-Co-02 "COVIPA"), una sovvenzione dell'Iniziativa e del Fondo di Networking dell'Associazione Helmholtz. Ringraziamo l'accesso generoso al Centro NMR Biomolecolare Jülich-Düsseldorf, gestito congiuntamente dal Forschungszentrum Jülich e dall'Università Heinrich Heine di Düsseldorf (HHU).
Access restricted. Please log in or start a trial to view this content.
Materiali
| Name | Company | Catalog Number | Comments |
| Bruker 600 MHz AVANCE III HD spectrometer | Bruker | https://www.bruker.com/en/products-and-solutions/mr/nmr/avance-nmr-spectrometer.html | NMR experiments conducted |
Riferimenti
- Sekhar, A., Kay, L. E. An NMR view of protein dynamics in health and disease. Annu Rev Biophys. 48, 297-319 (2019).
- Rosenzweig, R., Kay, L. E. Bringing dynamic molecular machines into focus by methyl-TROSY NMR. Annu Rev Biochem. 83, 291-315 (2014).
- Palmer, A. G. 3rd Enzyme dynamics from NMR spectroscopy. Acc Chem Res. 48 (2), 457-465 (2015).
- Reif, B. Ultra-high resolution in MAS solid-state NMR of perdeuterated proteins: implications for structure and dynamics. J Magn Reson. 216, 1-12 (2012).
- Schanda, P., Ernst, M. Studying dynamics by magic-angle spinning solid-state NMR spectroscopy: Principles and applications to biomolecules. Prog Nucl Magn Reson Spectrosc. 96, 1-46 (2016).
- Le Marchand, T., et al. (1)H-H-detected biomolecular NMR under fast magic-angle spinning. Chem Rev. 122 (10), 9943-10018 (2022).
- Pervushin, K., Riek, R., Wider, G., Wuthrich, K. Attenuated T2 relaxation by mutual cancellation of dipole-dipole coupling and chemical shift anisotropy indicates an avenue to NMR structures of very large biological macromolecules in solution. Proc Natl Acad Sci U S A. 94 (23), 12366-12371 (1997).
- Tzakos, A. G., Grace, C. R., Lukavsky, P. J., Riek, R. NMR techniques for very large proteins and RNAs in solution. Annu Rev Biophys Biomol Struct. 35, 319-342 (2006).
- Sprangers, R., Kay, L. E. Quantitative dynamics and binding studies of the 20S proteasome by NMR. Nature. 445 (7128), 618-622 (2007).
- Wright, P. E., Dyson, H. J. Intrinsically disordered proteins in cellular signalling and regulation. Nat Rev Mol Cell Biol. 16 (1), 18-29 (2015).
- van der Lee, R., et al. Classification of intrinsically disordered regions and proteins. Chem Rev. 114 (13), 6589-6631 (2014).
- Tompa, P. Intrinsically unstructured proteins. Trends Biochem Sci. 27 (10), 527-533 (2002).
- Dyson, H. J., Wright, P. E. Coupling of folding and binding for unstructured proteins. Curr Opin Struct Biol. 12 (1), 54-60 (2002).
- Uversky, V. N. Intrinsically disordered proteins in overcrowded milieu: Membrane-less organelles, phase separation, and intrinsic disorder. Current Opinion in Structural Biology. 44, 18-30 (2017).
- Patel, A., et al. A liquid-to-solid phase transition of the ALS protein FUS accelerated by disease mutation. Cell. 162 (5), 1066-1077 (2015).
- Abyzov, A., Blackledge, M., Zweckstetter, M. Conformational dynamics of intrinsically disordered proteins regulate biomolecular condensate chemistry. Chem Rev. 122 (6), 6719-6748 (2022).
- Uversky, V. N. Unusual biophysics of intrinsically disordered proteins. Biochim Biophys Acta. 1834 (5), 932-951 (2013).
- Papoian, G. A. Proteins with weakly funneled energy landscapes challenge the classical structure-function paradigm. Proc Natl Acad Sci U S A. 105 (38), 14237-14238 (2008).
- Uversky, V. N., Gillespie, J. R., Fink, A. L. Why are "natively unfolded" proteins unstructured under physiologic conditions. Proteins. 41 (3), 415-427 (2000).
- Bah, A., et al. Folding of an intrinsically disordered protein by phosphorylation as a regulatory switch. Nature. 519 (7541), 106-109 (2015).
- Tompa, P., Schad, E., Tantos, A., Kalmar, L. Intrinsically disordered proteins: emerging interaction specialists. Curr Opin Struct Biol. 35, 49-59 (2015).
- Dyson, H. J., Wright, P. E. Perspective: the essential role of NMR in the discovery and characterization of intrinsically disordered proteins. J Biomol NMR. 73 (12), 651-659 (2019).
- Adamski, W., et al. A unified description of intrinsically disordered protein dynamics under physiological conditions using NMR spectroscopy. J Am Chem Soc. 141 (44), 17817-17829 (2019).
- Dobson, C. M. Protein folding and misfolding. Nature. 426 (6968), 884-890 (2003).
- Bertoncini, C. W., et al. Release of long-range tertiary interactions potentiates aggregation of natively unstructured alpha-synuclein. Proc Natl Acad Sci U S A. 102 (5), 1430-1435 (2005).
- Buell, A. K., et al. Solution conditions determine the relative importance of nucleation and growth processes in alpha-synuclein aggregation. Proc Natl Acad Sci U S A. 111 (21), 7671-7676 (2014).
- Salvi, N., Abyzov, A., Blackledge, M. Atomic resolution conformational dynamics of intrinsically disordered proteins from NMR spin relaxation. Prog Nucl Magn Reson Spectrosc. 102, 43-60 (2017).
- Schneider, R., Blackledge, M., Jensen, M. R. Elucidating binding mechanisms and dynamics of intrinsically disordered protein complexes using NMR spectroscopy. Curr Opin Struct Biol. 54, 10-18 (2019).
- Rezaei-Ghaleh, N., Blackledge, M., Zweckstetter, M. Intrinsically disordered proteins: from sequence and conformational properties toward drug discovery. Chembiochem. 13 (7), 930-950 (2012).
- Jensen, M. R., Salmon, L., Nodet, G., Blackledge, M. Defining conformational ensembles of intrinsically disordered and partially folded proteins directly from chemical shifts. J Am Chem Soc. 132 (4), 1270-1272 (2010).
- Camacho-Zarco, A. R., et al. NMR provides unique insight into the functional dynamics and interactions of intrinsically disordered proteins. Chem Rev. 122 (10), 9331-9356 (2022).
- Mittag, T., Forman-Kay, J. D. Atomic-level characterization of disordered protein ensembles. Curr Opin Struct Biol. 17 (1), 3-14 (2007).
- Dyson, H. J., Wright, P. E. Unfolded proteins and protein folding studied by NMR. Chem Rev. 104 (8), 3607-3622 (2004).
- Dyson, H. J., Wright, P. E. NMR illuminates intrinsic disorder. Curr Opin Struct Biol. 70, 44-52 (2021).
- Sugase, K., Dyson, H. J., Wright, P. E. Mechanism of coupled folding and binding of an intrinsically disordered protein. Nature. 447 (7147), 1021-1025 (2007).
- Bessa, L. M., et al. The intrinsically disordered SARS-CoV-2 nucleoprotein in dynamic complex with its viral partner nsp3a. Sci Adv. 8 (3), eabm4034(2022).
- Milles, S., Salvi, N., Blackledge, M., Jensen, M. R. Characterization of intrinsically disordered proteins and their dynamic complexes: From in vitro to cell-like environments. Prog Nucl Magn Reson Spectrosc. 109, 79-100 (2018).
- Salvi, N., Abyzov, A., Blackledge, M. Atomic resolution conformational dynamics of intrinsically disordered proteins from NMR spin relaxation. Prog Nucl Magn Reson Spectrosc. 102 - 103, 43-60 (2017).
- Gill, M. L., Byrd, R. A., Palmer, A. G. III Dynamics of GCN4 facilitate DNA interaction: a model-free analysis of an intrinsically disordered region. Phys Chem Chem Phys. 18 (8), 5839-5849 (2016).
- Bertoncini, C. W., et al. Structural characterization of the intrinsically unfolded protein beta-synuclein, a natural negative regulator of alpha-synuclein aggregation. J Mol Biol. 372 (3), 708-722 (2007).
- Rezaei-Ghaleh, N., et al. Local and global dynamics in intrinsically disordered synuclein. Angew Chem Int Ed Engl. 57 (46), 15262-15266 (2018).
- Rezaei-Ghaleh, N., Parigi, G., Zweckstetter, M. Reorientational dynamics of amyloid-beta from NMR spin relaxation and molecular simulation. J Phys Chem Lett. 10 (12), 3369-3375 (2019).
- Palmer, A. G. NMR characterization of the dynamics of biomacromolecules. Chem Rev. 104 (8), 3623-3640 (2004).
- Palmer, A. G., Massi, F. Characterization of the dynamics of biomacromolecules using rotating-frame spin relaxation NMR spectroscopy. Chem Rev. 106 (5), 1700-1719 (2006).
- Kay, L. E., Torchia, D. A., Bax, A. Backbone dynamics of proteins as studied by 15N inverse detected heteronuclear NMR spectroscopy: application to staphylococcal nuclease. Biochemistry. 28 (23), 8972-8979 (1989).
- Mittermaier, A., Kay, L. E. Review - New tools provide new insights in NMR studies of protein dynamics. Science. 312 (5771), 224-228 (2006).
- Abragam, A. Principles of Nuclear Magnetism. , Clarendon Press. Oxford. (1983).
- Ernst, R. R., Bodenhausen, G., Wokaun, A. Principles of Nuclear Magnetic Resonance in One and Two Dimensions. , Oxford University Press. Oxford. (1990).
- Cavanagh, J., Fairbrother, W. J., Palmer, A. G. III, Skelton, N. J. Protein NMR Spectroscopy: Principles and Practice. , Elsevier, Academic Press. (1995).
- Levitt, M. H. Spin Dynamics: Basics of Nuclear Magnetic Resonance. , John Wiley and Sons. (2008).
- Keeler, J. Understanding NMR Spectroscopy. , John Wiley and Sons. (2010).
- Bloembergen, N., Purcell, E. M., Pound, R. V. Relaxation effects in nuclear magnetic resonance absorption. Phys Rev. 73 (7), 679-712 (1948).
- Wangsness, R. K., Bloch, F. The dynamical theory of nuclear induction. Phys Rev. 89 (4), 728-739 (1953).
- Redfield, A. G. On the theory of relaxation processes. IBM J Res Dev. 1 (1), 19-31 (1957).
- Peng, J. W., Wagner, G. Mapping of the spectral densities of N-H bond motions in eglin c using heteronuclear relaxation experiments. Biochemistry. 31 (36), 8571-8586 (1992).
- Farrow, N. A., et al. Backbone dynamics of a free and a phosphopeptide-complexed src homology-2 domain studied by n-15 NMR relaxation. Biochemistry. 33 (19), 5984-6003 (1994).
- Tolman, J. R., Ruan, K. NMR residual dipolar couplings as probes of biomolecular dynamics. Chem Rev. 106 (5), 1720-1736 (2006).
- Mulder, F. A. A., Skrynnikov, N. R., Hon, B., Dahlquist, F. W., Kay, L. E. Measurement of slow (mu s-ms) time scale dynamics in protein side chains by N-15 relaxation dispersion NMR spectroscopy: Application to Asn and Gln residues in a cavity mutant of T4 lysozyme. J Am Chem Soc. 123 (5), 967-975 (2001).
- Tollinger, M., Skrynnikov, N. R., Mulder, F. A. A., Forman-Kay, J. D., Kay, L. E. Slow dynamics in folded and unfolded states of an SH3 domain. J Am Chem Soc. 123 (46), 11341-11352 (2001).
- Akke, M., Palmer, A. G. Monitoring macromolecular motions on microsecond to millisecond time scales by R(1)rho-R(1) constant relaxation time NMR spectroscopy. J Am Chem Soc. 118 (4), 911-912 (1996).
- Loria, J. P., Rance, M., Palmer, A. G. A relaxation-compensated Carr-Purcell-Meiboom-Gill sequence for characterizing chemical exchange by NMR spectroscopy. J Am Chem Soc. 121 (10), 2331-2332 (1999).
- Vallurupalli, P., Bouvignies, G., Kay, L. E. Studying "Invisible" excited protein states in slow exchange with a major state conformation. J Am Chem Soc. 134 (19), 8148-8161 (2012).
- Jeener, J., Meier, B. H., Bachmann, P., Ernst, R. R. Investigation of exchange processes by 2-dimensional NMR-spectroscopy. J Chem Phys. 71 (11), 4546-4553 (1979).
- Palmer, A. G., Kroenke, C. D., Loria, J. P. Nuclear magnetic resonance methods for quantifying microsecond-to-millisecond motions in biological macromolecules. Methods Enzymol. 339, 204-238 (2001).
- Clore, G. M., Iwahara, J. Theory, practice, and applications of paramagnetic relaxation enhancement for the characterization of transient low-population states of biological macromolecules and their complexes. Chem Rev. 109 (9), 4108-4139 (2009).
- Iwahara, J., Clore, G. M. Detecting transient intermediates in macromolecular binding by paramagnetic NMR. Nature. 440 (7088), 1227-1230 (2006).
- Lakomek, N. A., et al. Residual dipolar couplings as a tool to study molecular recognition of ubiquitin. Biochem Soc Trans. 36 (Pt 6), 1433-1437 (2008).
- Johnson, C. N., Libich, D. S. Paramagnetic relaxation enhancement for detecting and characterizing self-associations of intrinsically disordered proteins. J Vis Exp. (175), e63057(2021).
- Lakomek, N. A., Ying, J., Bax, A. Measurement of (1)(5)N relaxation rates in perdeuterated proteins by TROSY-based methods. J Biomol NMR. 53 (3), 209-221 (2012).
- Stief, T., Vormann, K., Lakomek, N. A. Sensitivity-enhanced NMR (15)N R(1) and R(1rho) relaxation experiments for the investigation of intrinsically disordered proteins at high magnetic fields. Methods. 223, 1-15 (2024).
- Kay, L. E., Keifer, P., Saarinen, T. Pure absorption gradient enhanced heteronuclear single quantum correlation spectroscopy with improved sensitivity. J Am Chem Soc. 114 (26), 10663-10665 (1992).
- Geen, H., Freeman, R. Band-selective radiofrequency pulses. J Magn Reson (1969). 93 (1), 93-141 (1991).
- Desvaux, H., Berthault, P. Study of dynamic processes in liquids using off-resonance RF irradiation. Prog Nucl Magn Reson Spectrosc. 35 (4), 295-340 (1999).
- Overhauser, A. W. Polarization of nuclei in metals. Phys Rev. 91 (2), 476-476 (1953).
- Overhauser, A. W. Polarization of nuclei in metals. Phys Rev. 92 (2), 411-415 (1953).
- Kumari, P., Frey, L., Sobol, A., Lakomek, N. A., Riek, R. (15)N transverse relaxation measurements for the characterization of micros-ms dynamics are deteriorated by the deuterium isotope effect on (15)N resulting from solvent exchange. J Biomol NMR. 72 (3-4), 125-137 (2018).
- Karschin, N., Krenek, S., Heyer, D., Griesinger, C. Extension and improvement of the methanol-d(4) NMR thermometer calibration. Magn Reson Chem. 60 (4), 203-209 (2022).
- Sattler, M., Schleucher, J., Griesinger, C. Heteronuclear multidimensional NMR experiments for the structure determination of proteins in solution employing pulsed field gradients. Prog Nucl Magn Reson Spectrosc. 34 (2), 93-158 (1999).
- Ferrage, F., Cowburn, D., Ghose, R. Accurate sampling of high-frequency motions in proteins by steady-state N-{H} nuclear Overhauser effect measurements in the presence of cross-correlated relaxation. J Am Chem Soc. 131 (17), 6048-6049 (2009).
- Delaglio, F., et al. NMRPipe: a multidimensional spectral processing system based on UNIX pipes. J Biomol NMR. 6 (3), 277-293 (1995).
- Skinner, S. P., et al. CcpNmr AnalysisAssign: a flexible platform for integrated NMR analysis. J Biomol NMR. 66 (2), 111-124 (2016).
- Lee, W., Tonelli, M., Markley, J. L. NMRFAM-SPARKY: enhanced software for biomolecular NMR spectroscopy. Bioinformatics. 31 (8), 1325-1327 (2015).
- Lee, W., Rahimi, M., Lee, Y., Chiu, A. POKY: a software suite for multidimensional NMR and 3D structure calculation of biomolecules. Bioinformatics. 37 (18), 3041-3042 (2021).
- Abyzov, A., et al. Identification of dynamic modes in an intrinsically disordered protein using temperature-dependent NMR relaxation. J Am Chem Soc. 138 (19), 6240-6251 (2016).
- Singh, A., Purslow, J. A., Venditti, V. 15N CPMG relaxation dispersion for the investigation of protein conformational dynamics on the micros-ms timescale. J Vis Exp. (170), e62395(2021).
- Gopalan, A. B., Hansen, D. F., Vallurupalli, P. CPMG experiments for protein minor conformer structure determination. Methods Mol Biol. 1688, 223-242 (2018).
- Tugarinov, V., Kay, L. E. Methyl groups as probes of structure and dynamics in NMR studies of high-molecular-weight proteins. Chembiochem. 6 (9), 1567-1577 (2005).
- Lipari, G., Szabo, A. Model-free approach to the interpretation of nuclear magnetic resonance relaxation in macromolecules. 1. Theory and range of validity. J Am Chem Soc. 104 (17), 4546-4559 (1982).
- Lipari, G., Szabo, A. Model-Free Approach to the interpretation of nuclear magnetic-resonance relaxation in macromolecules .2. Analysis of experimental results. J Am Chem Soc. 104 (17), 4559-4570 (1982).
- Clore, G. M., et al. Deviations from the simple two-parameter model-free approach to the interpretation of nitrogen-15 nuclear magnetic relaxation of proteins. J Am Chem Soc. 112 (12), 4989-4991 (1990).
- Lemaster, D. M. Larmor frequency selective model free analysis of protein NMR relaxation. J Biomol NMR. 6 (4), 366-374 (1995).
- Peng, J. W., Wagner, G. Mapping of spectral density functions using heteronuclear NMR relaxation measurements. J Magn Reson (1969). 98 (2), 308-332 (1992).
- Farrow, N. A., Zhang, O. W., FormanKay, J. D., Kay, L. E. Characterization of the backbone dynamics of folded and denatured states of an SH3 domain. Biochemistry. 36 (9), 2390-2402 (1997).
- Khan, S. N., et al. Distribution of pico- and nanosecond motions in disordered proteins from nuclear spin relaxation. Biophys J. 109 (5), 988-999 (2015).
- Smith, A. A., Ernst, M., Meier, B. H., Ferrage, F. Reducing bias in the analysis of solution-state NMR data with dynamics detectors. J Chem Phys. 151 (3), 034102(2019).
- Zumpfe, K., Smith, A. A. Model-free or not. Front Mol Biosci. 8, 727553(2021).
- Robustelli, P., Trbovic, N., Friesner, R. A., Palmer, A. G. Conformational dynamics of the partially disordered yeast transcription factor GCN4. J Chem Theory Comput. 9 (11), 5190-5200 (2013).
- Salvi, N., Abyzov, A., Blackledge, M. Multi-timescale dynamics in intrinsically disordered proteins from NMR relaxation and molecular simulation. J Phys Chem Lett. 7 (13), 2483-2489 (2016).
Access restricted. Please log in or start a trial to view this content.
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon