Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Expériences de relaxation RMN 15N pour l’étude de la dynamique structurelle des protéines de la picoseconde à la nanoseconde
Dans cet article
Résumé
La spectroscopie par résonance magnétique nucléaire (RMN) peut caractériser la dynamique des protéines structurelles d’une manière spécifique aux résidus. Nous fournissons un protocole pratique pour l’enregistrement d’expériences de relaxation RMN 15N R1 et R2 et d’expériences à effet Overhauser hétéronucléaire {1H}-15N, sensibles à l’échelle de temps de la picoseconde à la nanoseconde.
Résumé
La spectroscopie par résonance magnétique nucléaire (RMN) permet d’étudier les protéines en solution et à des températures physiologiques. Fréquemment, les groupes amides du squelette protéique ou les groupes méthyle dans les chaînes latérales sont utilisés comme rapporteurs de la dynamique structurelle des protéines. Une étude de la dynamique structurale du squelette protéique des protéines globulaires sur des échantillons marqués 15N et entièrement protonés fonctionne généralement bien pour les protéines d’un poids moléculaire allant jusqu’à 50 kDa. Lorsque la deutération de la chaîne latérale en combinaison avec la spectroscopie optimisée de relaxation transverse (TROSY) est appliquée, cette limite peut être étendue jusqu’à 200 kDa pour les protéines globulaires et jusqu’à 1 MDa lorsque l’accent est mis sur les chaînes latérales. Lorsque des protéines intrinsèquement désordonnées (IDP) ou des protéines avec des régions intrinsèquement désordonnées (IDR) sont étudiées, ces limitations de poids ne s’appliquent pas mais peuvent aller bien au-delà. La raison en est que les PDI ou IDR, caractérisés par une grande flexibilité interne, sont souvent découplés dynamiquement. Diverses méthodes de RMN offrent des informations à résolution atomique sur la dynamique des protéines structurelles sur une large gamme d’échelles de temps, de la picoseconde à l’heure. Les mesures de relaxation standard 15N permettent d’obtenir un aperçu de la flexibilité interne d’une protéine et de caractériser la dynamique du squelette protéique sur une échelle de temps rapide de l’ordre de la pico à la nanoseconde. Cet article présente un protocole pratique pour la mise en place et l’enregistrement d’expériences de RMN 15N R1, R2 et d’effets Overhauser hétéronucléaires (hetNOE). Nous présentons des données exemplaires et expliquons comment les interpréter simplement qualitativement avant toute analyse plus sophistiquée.
Introduction
La fonction d’une protéine est déterminée non seulement par sa structure tridimensionnelle, mais aussi par sa dynamique structurelle, englobant sa flexibilité interne et les transitions structurelles entre les différentes conformations que la protéine adoptera. La spectroscopie par résonance magnétique nucléaire (RMN) permet d’étudier la dynamique structurale des protéines dans la solution 1,2,3. Les développements récents de la RMN à l’état solide détectée par des protons permettent également de caractériser la dynamique des protéines dans un état moins soluble, comme, par exemple, une membrane bicouche lipidique 4,5,6. En RMN en solution, la dynamique structurelle du squelette protéique et des chaînes latérales protéiques peut être étudiée. Pour une protéine globulaire, une étude de dynamique structurale du squelette protéique peut être réalisée jusqu’à 50 kDa une fois que la protéine est marquée isotopiquement à 15N. Lorsque la deutération à chaîne latérale et la spectroscopie optimisée de relaxation transversale (TROSY) sont utilisées, cette limite peut être étendue jusqu’à 200 kDa 7,8. Lorsque l’accent est mis sur la dynamique de la chaîne latérale, la gamme de protéines et de complexes accessibles peut être étendue jusqu’à 1 MDa 2,9.
Les limitations de poids nommées ne s’appliquent pas aux protéines intrinsèquement désordonnées (IDP), qui présentent souvent une dynamique intrinsèque élevée. Plus de 30 % du protéome eucaryote est constitué d’IDP ou de régions intrinsèquement désordonnées (IDR)10,11,12,13. Ils jouent un rôle central dans de nombreux processus cellulaires, tels que la transduction et la transcription du signal1, et sont fréquemment impliqués dans la séparation de phase intracellulaire 14,15,16,17. Les PDI n’ont pas de structure native tridimensionnelle (3D) bien définie dans des conditions physiologiques et ont un paysage énergétique faiblement canalisé ou accidenté17,18. En raison d’une faible hydrophobie et d’une forte répulsion électrostatique répartie sur l’épine dorsale des IDP ou des IDR, il manque une force motrice pour le pliage en une structure rigide19. Les IDP adoptent fréquemment une conformation pliée lorsqu’elles sont complexes avec d’autres partenaires de liaison 10,20,21. De plus, les modifications post-traductionnelles (MTP) élargissent les possibilités de pliage des IDP ou des IDR22,23. Le mauvais repliement des PDI a été identifié comme une cause de diverses maladies, y compris les maladies neurodégénératives 15,24,25,26.
Les PDI et les IDR font preuve d’une grande flexibilité interne 21,27,28. Des ensembles conformationnels montrant la variation des positions atomiques et des angles dièdres ont été dérivés de simulations de dynamique moléculaire et de contraintes obtenues à partir de données expérimentales 29,30,31,32. En raison de la dynamique et du désordre qui en résulte à l’état gelé, la densité électronique diffuse rend difficile leur caractérisation structurelle à l’aide de méthodes de pointe en biologie structurale, telles que la cryo-EM ou la cristallographie aux rayons X. De plus, les conditions de cristallisation ou les techniques de préparation d’échantillons pour des expériences à des températures cryogéniques peuvent avoir un impact sur l’espace conformationnel expérimenté par les PDI. Cependant, la RMN en solution fonctionne bien pour les protéines hautement dynamiques et est donc bien adaptée à l’étude des IDP16, 20, 22, 28, 29, 30, 31, 32, 33, 34, 35, 36, 37, 38.
Comme nous l’avons présenté ci-dessus, la RMN en solution offre diverses techniques pour étudier la dynamique interne des protéines sur une large gamme d’échelles de temps (Figure 1), principalement basées sur la relaxation de spin 31,33,38,39,40,41,42.
La relaxation de spin des noyaux 15N dans les groupes amides du squelette protéique est induite par des changements d’orientation de l’angle de liaison 1 H-15N dus à la dynamique interne des protéines et aux mouvements collectifs (y compris, le cas échéant, la diffusion rotationnelle)27,43,44,45,46,47,48,49,50,51. À des échelles de temps plus courtes que le temps de corrélation de rotation τR (le temps dont la molécule a besoin pour tourner d’un radiant, également appelé temps de corrélation de culbutage global), l’anisotropie de décalage chimique (CSA) et le couplage dipolaire (D) sont actifs et ne sont pas moyennés par la diffusion rotationnelle de la protéine. La dynamique interne du squelette protéique, comprenant les variations des angles de liaison, les réorientations des liaisons et le culbutage rotationnel, induit des fluctuations stochastiques du CSA et du tenseur de couplage dipolaire, entraînant une variation du champ magnétique local, conduisant finalement à une relaxation de spin RMN 47,48,52,53. Ces fluctuations peuvent être décrites par une fonction de corrélation globale. La transformée de Fourier de la fonction de corrélation globale est appelée fonction de densité spectrale. Dans la théorie semi-classique de la relaxation de Redfield, les constantes de vitesse de relaxation RMN peuvent être décrites par des combinaisons linéaires de ces fonctions de densité spectrale54.
Les expériences de relaxation RMN 15N développées au début des années 1990 comprennent des expériences nucléaires à effet Overhauser 15N R1, R1ρ et {1H}-15N, sensibles à l’échelle de temps de la nanoseconde (ns) picoseconde (ps) rapide, plus rapide que le temps de corrélation de rotation τR de la protéine 45,55,56,57. Pour caractériser la dynamique du squelette plus lente que le temps de corrélation rotationnelle τR, on utilise des expériences dites de dispersion de relaxation, R1ρ et des expériences de Carr-Purcell-Meiboom-Gill (CPMG) sensibles à la dynamique microseconde (μs) - milliseconde (ms) 44,46,58,59,60,61. Des dynamiques inférieures à la microseconde peuvent être capturées par la RMN 15N par transfert de saturation par échange chimique (CEST)62, la spectroscopie d’échange (EXSY, millisecondes à secondes) ou la RMN en temps réel (RT) (secondes en heures)63,64. L’effet PRE (paramagnetic relaxation enhancement) des sondes paramagnétiques, ainsi que des couplages dipolaires résiduels (RDC), peut être utilisé pour évaluer toute la gamme de dynamiques ps à ms 65,66,67,68.
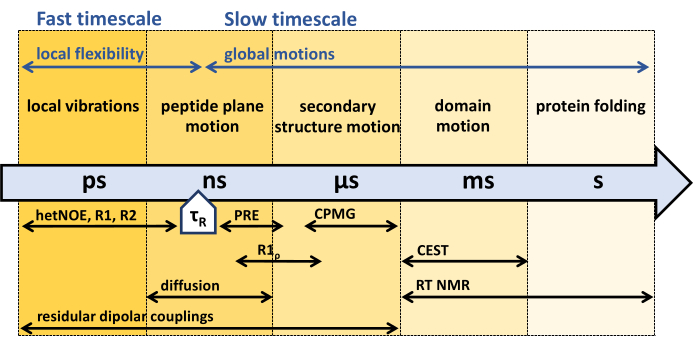
Figure 1 : Échelles de temps de la dynamique du squelette protéique et fenêtre temporelle sensible de différentes expériences de dynamique RMN. La RMN offre une variété de méthodes pour caractériser la dynamique du squelette protéique sur une large gamme d’échelles de temps. Les différents mouvements subis par le squelette protéique sont indiqués à leurs échelles de temps respectives. Le temps de corrélation rotationnelle de la protéine, τR, est le temps dont la protéine a besoin pour une rotation globale (d’un radiant). Des mouvements plus rapides que le temps de corrélation rotationnelle de la protéine, τR, peuvent être associés à la flexibilité interne de la protéine. Diverses expériences de RMN et leur sensibilité aux échelles de temps respectives sont indiquées sous la flèche. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Le protocole ci-dessous décrit la mise en place d’expériences de relaxation RMN par Lakomek et al.69 et Stief et al.70, à l’aide d’un schéma de détection de cohérence quantique unique (HSQC) hétéronucléaire à sensibilité améliorée. Avant de procéder à la mise en œuvre expérimentale, un très bref aperçu des expériences de relaxation de spin RMN et de relaxation RMN est donné. En raison de contraintes de taille et pour que ce protocole reste compréhensible, cette vue d’ensemble doit rester simpliste (et donc incomplète).
La relaxation longitudinale ou en spin-réseau, caractérisée par le temps T1 ou la constante de vitesse R1 = 1/ T1 , décrit le retour de l’aimantation à l’équilibre de Boltzmann. À l’équilibre, l’aimantation est alignée le long de l’axe du champ magnétique externe, qui définit l’axe z du cadre du laboratoire. Les densités spectrales aux fréquences de Larmor élevées (1H) et petites (15N) (les fréquences de résonance RMN, par exemple, 600 MHz pour 1H pour un aimant de 14,1 Tesla) et les combinaisons linéaires de ces fréquences de Larmor contribuent à la relaxation 15N R1 , qui est caractérisée par les constantes de vitesse 15N R1 mesurées en rad·s-1. Les mouvements sur les échelles de temps sont inverses à ces fréquences de Larmor ; ainsi, les mouvements sur l’échelle de temps de la picoseconde à la nanoseconde contribuent à la constante de vitesse de relaxation R1. Pour les molécules qui présentent un culbutage global et où un temps de corrélation de rotation peut être défini, la courbe R1 (T1) montre un maximum (minimum) pour ωτR = 1, avec la corrélation de rotation τR et la fréquence de Larmor ω du spin considérée. Si plusieurs fréquences de Larmor contribuent, celle avec la fréquence la plus basse est la dominante, par exemple, ωN dans le cas de 15N R1. Le régime de mouvement rapide (ωτR beaucoup plus petit que 1) s’applique aux petites molécules qui culbutent très rapidement et aux faibles champs magnétiques et à faible viscosité. Le régime du ralenti (ωτR beaucoup plus grand que 1) est valable pour les molécules plus grosses qui culbutent plus lentement et pour les champs magnétiques élevés et la viscosité élevée.
Les protéines globulaires repliées montrent un culbutage global dans la solution, et un temps de corrélation de rotation peut être attribué. Cependant, le concept de culbutage global n’est plus valable pour les protéines intrinsèquement désordonnées et diffère souvent de l’attribution d’un seul temps de corrélation rotationnelle. Ici, le temps de corrélation interne spécifique au résidu devient plus critique.
La séquence d’impulsions décrite mesurant des taux de relaxation de 15N R1 (figure 2) est basée sur une expérience de lecture HSQC à sensibilité améliorée avec une détection écho/anti-écho pour la détection en quadrature 69,70,71. Des gradients courts de force et de longueur variables sont utilisés pour la sélection de la cohérence et une meilleure suppression de l’eau70. Pendant ce temps, la polarisation longitudinale de 15N se détendra. Des temps de décroissance plus longs entraînent une réduction des intensités dans les plans 2D associés à ce spectre pseudo-3D (les points de données de retard sont enregistrés dans la troisième dimension). Un élément de boucle, décrit ci-dessous, est exécuté un nombre croissant de fois pour des temps de relaxation plus longs. Comme la relaxation croisée entre l’anisotropie de décalage chimique 15N (CSA) et le couplage dipolaire 1H et 15N (D) est également active pendant le retard de relaxation, une impulsion centrale I-BURP-2 180°72, sélective sur les protons amides, est nécessaire pour recentrer la contribution par relaxation corrélée croisée (qui, si elle n’était pas recentrée, conduirait à des constantes de vitesse 15N R1 asymétriques et erronées).
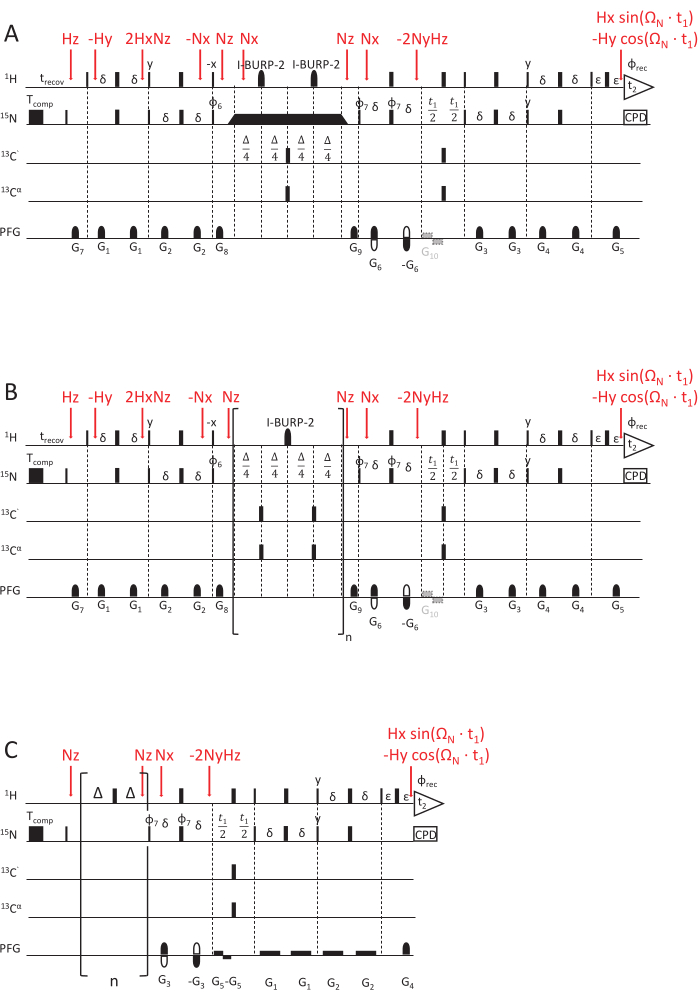
Figure 2 : Schémas de séquences d’impulsions RMN pour déterminer les constantes de vitesse de relaxation RMN. (A) 15N R1ρ, (B) 15N R1, et (C) expérience hetNOE, à l’aide d’un schéma de lecture HSQC à sensibilité améliorée69,70. Les impulsions de 90°(x) sont visualisées par des rectangles étroits et les impulsions de 180°(x) par des rectangles larges, sauf indication contraire. Le cycle de phase suivant est appliqué : φ6 = y, y, -y, -y ; φ7 = y, -y, φrec = y, -y, -y, y. La détection en quadrature est obtenue en inversant la polarité du gradient G5 et le cycle de phase de φ7 (détection Echo/Anti-Echo). (A) Expérience 15N R1ρ : Le rectangle noir représente le spin-lock, dont la durée varie pour acquérir différents retards de relaxation. Les triangles avant et après le spin-lock indiquent les impulsions de forme adiabatique qui alignent l’aimantation le long de l’axe du champ magnétique effectif Beff. G10 est un gradient optionnel pour éviter l’amortissement par rayonnement de l’aimantation de l’eau pendant la phase d’évolution. (B) Expérience 15N R1 : La partie entre crochets montre l’élément de boucle de la séquence, répété un n fois pour correspondre au retard de relaxation souhaité. (C) Le schéma d’impulsions hetNOE est similaire à la seconde moitié des schémas d’impulsions R1 et R1ρ, à savoir le temps d’évolution t1 et l’élément de détection HSQC. Cependant, l’aimantation 15N est excitée directement sans aucun INEPT. La saturation de l’aimantation du proton (pour obtenir une relaxation croisée entre 1H et 15N) est obtenue par un train de 180 impulsions (1H) appliquées pendant au moins 5 s. Un retard en repos de mêmes longueurs (ici, 5 s) sans aucun train d’impulsions est appliqué pour l’expérience de référence. G5 est un gradient facultatif pour empêcher l’amortissement des rayonnements, et l’inversion de polarité du gradient G4, en combinaison avec la phase φ7 = y, -y, -y, y, permet d’obtenir une détection en quadrature. Les étapes de transfert de magnétisation, représentées par les opérateurs de produit, sont marquées en rouge. Veuillez cliquer ici pour voir une version agrandie de cette figure.
La constante de vitesse de relaxation R2 décrit la relaxation de la polarisation transversale (dans le plan xy orthogonal au champ magnétique externe) due à la perte de cohérence de phase entre le spin, conduisant à une décroissance de l’aimantation détectable53,54. La fonction de densité spectrale aux hautes et petites fréquences contribue à R2, similaire à R1. Cependant, la plus grande contribution à R2 provient de la densité spectrale à fréquence nulle. Pour cette raison, R2 est très sensible au culbutage rotationnel, décrit par le temps de corrélation rotationnelle τR, qui est de l’ordre de quelques ns pour une petite protéine globulaire à température ambiante. Ainsi, les mouvements plus lents de l’épine dorsale dans les centaines de ps à faible régime ns contribuent le plus. La dynamique d’échange du squelette qui provoque une modulation de la partie isotrope du tenseur de décalage chimique des noyaux 15N, en principe, ajoute une contribution d’échange R2ex aux constantes de vitesse R2 43,44,49,60,61. Dans les expériences décrites, la contribution de R2ex est supprimée par un verrou de spin qui recentre la dynamique plus lentement que la fréquence circulaire inverse du verrou de spin. Le spin-lock est une longue impulsion radiofréquence à onde continue qui maintient l’aimantation alignée le long de l’axe du champ magnétique effectif Beff (la somme vectorielle du champ de spin-lock ω1 et du décalage chimique par rapport à la fréquence porteuse 15N (voir ci-dessous)). La relaxation de l’aimantation alignée le long de l’axe B1,eff est appelée relaxation R1ρ, qui a une composante R1 et une composante R2. À l’aide de la formule (1), R2 peut être calculé à partir de R1ρ et R144,73 :
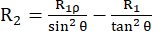 (1).
(1).
L’angle entre l’axe du champ magnétique effectif Beff et le champ magnétique externe B0 est 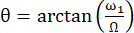 . ω1 est l’amplitude RF du spin-lock et Ω le décalage chimique entre le décalage chimique 15N du résidu correspondant et la fréquence porteuse 15N 44,73.
. ω1 est l’amplitude RF du spin-lock et Ω le décalage chimique entre le décalage chimique 15N du résidu correspondant et la fréquence porteuse 15N 44,73.
Le schéma d’impulsions R1ρ (figure 2A, 70) est très similaire au schéma 15N R1 , à l’exception du retard de relaxation. Pour mesurer les taux de relaxation de 15N R1ρ , le spin-lock doit être actif après que l’aimantation a été alignée le long de l’axe de champ effectif Beff par une impulsion adiabatique de la même amplitude de radiofréquence (RF) que le spin-lock. La longueur du spin-lock sera modifiée pour obtenir les différents délais de relaxation.
L’effet de Overhauser nucléaire {1H}-15N à l’état stationnaire (1 H-15N NOE), appelé hetNOE dans ce qui suit, est le rapport entre le taux de relaxation croisée et le taux de relaxation longitudinale de 15N. Elle conduit à une réduction de la polarisation à l’état stationnaire sur 15N due à la relaxation croisée avec le proton lors de la saturation de la polarisation du proton 45,53,54,74,75. La relaxation croisée dépend des fonctions de densité spectrale de la somme et de la différence des fréquences de Larmor 1H et 15N. Par conséquent, l’hetNOE est sensible à la fois à la dynamique picoseconde rapide (< 100 ps) et à la dynamique ps-ns (en raison de sa dépendance R1). La séquence69 (Figure 2C) est basée sur une lecture HSQC à sensibilité améliorée avec gradients Echo/Anti-Echo pour la détection en quadrature. Pour la saturation de l’aimantation des protons et l’hetNOE qui en résulte, l’aimantation des protons à l’équilibre est inversée puis saturée par des impulsions rapides de 180° pendant environ 5 fois les 15 N T1. Pour l’expérience de référence, le retard de récupération est égal au retard de saturation mais sans le train d’impulsions de 1H à 180°. Un délai supplémentaire de D1 = 2 s est ajouté pour l’expérience de référence et celle avec une saturation de 1H. Les deux expériences sont enregistrées l’une après l’autre et ne diffèrent que par l’application d’impulsions de 1H à 180° (saturation) ou non (référence). Le rapport des intensités spectrales enregistrées dans l’expérience avec une saturation de 1H divisé par les intensités de l’expérience de référence (sans le train d’impulsions de protons de 180°) donne la valeur {1H}-15N NOE (hetNOE).
Le protocole suivant décrit la mise en place d’expériences de relaxation RMN par Lakomek et al.69 et Stief et al.70. Nous nous concentrons sur les séquences d’impulsions RMN à l’aide d’un schéma de détection HSQC à sensibilité améliorée. Les expériences 15N R1 et R1ρ sont mises en œuvre comme décrit en détail par Stief et al.70, et l’expérience hetNOE est décrite par Lakomek et al.69.
Access restricted. Please log in or start a trial to view this content.
Protocole
1. Préparation des échantillons RMN
REMARQUE : Le marquage isotopique des protéines est effectué pour les expériences de RMN de dimension supérieure et de RMN avancées. Lorsque l’expression des protéines chez E. coli et la purification des protéines ont été établies à l’aide de milieux riches (par exemple, Luria-Bertani [LB] ou milieu tryptone d’extrait de levure 2x [2YT]) avec un rendement de plusieurs milligrammes par litre, la préparation d’un échantillon RMN marqué isotopiquement est généralement relativement simple.
- Pour le marquage isotopique, utilisez un milieu minimal M9 pour l’expression des gènes, où le chlorure d’ammonium 15N est la seule source d’azote et le glucose enrichi en carbone 13est la seule source de carbone.
- Pour la purification des protéines, suivez le protocole standard établi en laboratoire pour la protéine concernée.
REMARQUE : La section des résultats montre des résultats représentatifs pour une IDP, la protéine monomère SNARE Synaptobrevin-2 (1-96). Le marquage isotopique RMN de Synaptobrevin-2 a été effectué comme décrit dans70. Pour les mesures de très haute précision, il est recommandé d’utiliser de faibles concentrations de D2O, inférieures à 5 %. Cela est dû à l’effet isotopique du deutérium, qui peut être observé dans l’écho de Hahn 15N T2 et dans les expériences de dispersion de relaxation de relaxation 15N CPMG à basse fréquence (non abordées ici), mais qui n’a qu’un impact minime dans les expériences 15N R1ρ (où il est principalement recentré )76.
2. Préparatifs pour la réalisation des expériences de relaxation RMN sur le spectromètre
REMARQUE : Les expériences de relaxation RMN décrites sont spécifiques aux spectromètres Bruker. Ils ont été testés sur des sondes à triple résonance cryogéniques et à température ambiante 1H, 15N et 13C et des consoles Avance III et Avance Neo exploitées par le logiciel Bruker Topsin 3.6 ou supérieur.
- Suivez les précautions.
- Faites un briefing de sécurité avant de travailler sur le spectromètre RMN.
- Vérifiez les spécifications de puissance de la sonde du spectromètre utilisé et choisissez une longueur d’impulsion et une puissance d’impulsion appropriées pour chaque impulsion, par exemple en comparant la cohérence avec les entrées du tableau « prosol » de Topspin.
- Importez les séquences d’impulsions.
- Téléchargez les programmes d’impulsions RMN soit à partir du site Web indiqué ci-dessous, soit à partir du référentiel étendu de la Banque de Résonance Magnétique Biologique (BMRB) (bmrbig102) dans le répertoire /opt/topspin4.1.4/exp/stan/nmr/lists/pp/user de l’ordinateur utilisant le spectromètre RMN. Veuillez ajuster la version topspin (ici : topspin4.1.4 à la version respective installée sur le spectromètre).
- Téléchargez les impulsions en forme de RMN soit à partir du site Web indiqué ci-dessous, soit à partir du référentiel étendu de la Banque de Résonance Magnétique Biologique (BMRB) (bmrbig102) dans le répertoire /opt/topspin4.1.4/exp/stan/nmr/lists/pp/user de l’ordinateur utilisant le spectromètre RMN. Veuillez ajuster la version topspin (ici : topspin 4.1.4 à la version respective installée sur le spectromètre).
- Calibrez le spectromètre et l’échantillon.
- Réglez la température qui vous intéresse. Pour l’étalonnage de la température, appliquez les protocoles de l’installation. Par exemple, utilisez un échantillon de méthanol-d4 et interprétez la différence de décalage chimique observée à l’aide du thermomètre RMN de l’application Web (https://chem.ch.huji.ac.il/nmr/software/thermometer.html) ; Voir aussi77.
- Insérez l’échantillon (plus 5 % de D2O supplémentaires pour le verrou) et attendez environ 10 min pour permettre à l’échantillon d’équilibrer sa température. Verrouillez le signal de deutérium (2H) (commande : verrouiller, sélectionner H2O+D2O).
- Faites correspondre et réglez les canaux 1H, 15N et 13C, par exemple à l’aide de la routine atma ou atmm. Si aucune unité d’appariement et de réglage automatique n’est installée, faites correspondre et régler manuellement la sonde à l’aide de la commande wobb.
- Effectuez un calage pour un champ B0 homogène, par exemple en utilisant la routine topshim tg.
- Calibrez la longueur d’impulsion de 90° (1H).
- Utilisez l’expérience zg. Utilisez getprosol pour importer les paramètres de la sonde. Choisissez une courte durée de 5 μs pour P1. Vérifiez et corrigez le niveau de puissance spécifié de la sonde pour PLW1 si nécessaire.
- Démarrez l’expérience avec zg. Traitez les données avec efp. Ajustez la phase à l’aide de .ph de sorte que la ligne de base soit uniforme et que le signal RMN de la résonance de l’eau soit en phase.
- Estimez une impulsion de 360° (1H) en prenant une impulsion commune ou approximative de 90° (1H) (par exemple, à partir des spécifications du spectromètre énuméré) et en la multipliant par un facteur de quatre. Exécutez l’expérience et traitez-la.
REMARQUE : La durée choisie est trop longue si le signal est positif et trop courte si négatif. L’impulsion de 360° (1H) sera le passage à zéro. - Ajustez la durée de l’impulsion en conséquence. Faites-le de manière itérative pour trouver le passage à zéro. Divisez la valeur obtenue par quatre. Il s’agit de l’impulsion de 90° (1H).
REMARQUE : Une alternative à la méthode à 360° décrite est la routine pulscale , qui peut être démarrée à partir de la ligne de commande et permet un étalonnage automatisé des impulsions 1H.
- Calibrez la fréquence porteuse O1 en fonction de la résonance de l’eau.
- Copiez l’expérience zg en tapant edc dans la ligne de commande vers un nouveau numéro d’expérience. Tapez rpar pour ouvrir les jeux de paramètres (Figure supplémentaire 1A).
- Recherchez le jeu de paramètres ZGPR. Cliquez sur le jeu de paramètres ZGPR (figure supplémentaire 1A), cliquez sur Lire, choisissez Exécuter « getprosol » et cliquez sur OK (figure supplémentaire 1B).
- Modifiez P1 sur la longueur d’impulsion calibrée de 90° (1H). Choisissez une durée de saturation de D1 = 2 s et une puissance d’impulsion de PLW9 de 50 dB. Réglez le gain du récepteur (rg) sur 16. Vous pouvez également exécuter le réglage du gain du récepteur en tapant rga et en réglant le gain du récepteur sur la valeur spécifiée (mais évitez le « dépassement de gain du récepteur »).
- Tapez gs dans la ligne de commande pour démarrer une acquisition répétée et continue. Allez dans la fenêtre d’acquisition et modifiez l’O1 pour réduire la zone FID dominée par le signal d’eau (Figure supplémentaire 2).
- Cliquez sous le marqueur pour diminuer l’O1, puis cliquez au-dessus du marqueur pour l’augmenter. Utilisez le bouton de sensibilité pour modifier la taille du pas. Enregistrez l’O1 actuel ; Close arrête l’acquisition répétée (figure supplémentaire 2).
- Calibrez la longueur d’impulsion de 90° (15N).
- Copiez l’expérience zg sous un nouveau numéro d’expérience (type edc). Tapez rpar pour lire l’ensemble des paramètres HSQCFPF3GPPHWG pour changer le programme d’impulsions en une expérience HSQC 1 H-15N. Utilisez l’option Conserver les paramètres P1, O1 et PLW1 (Figure supplémentaire 1).
- Tapez getprosol 1H 1H) en μs> <90° (1H) intensité d’impulsion en db> dans la ligne de commande pour importer les paramètres de la sonde. Réglez le nombre de balayages (NS) sur 8 et le nombre de balayages factices (DS) sur 2.
REMARQUE : < > spécifie ici les valeurs à définir. Par exemple : getprosol 1H 8,5 -11,79. - Exécutez le premier incrément de l’expérience en tapant zg dans la ligne de commande. Copiez le premier incrément sur un nouveau numéro d’expérience avec rser 1 . Traitez les données avec efp et phasez le spectre avec .ph. Utilisez le premier incrément (expérience 1D) de l’expérience pour les étapes suivantes.
REMARQUE : Le < > spécifier le numéro choisi pour la nouvelle expérience, par exemple, rser 1 101. - Tapez ased dans la ligne de commande pour ouvrir les paramètres de séquence d’impulsions de l’expérience 1D. Cliquez sur le E dans la ligne PULPROG (voir Figure 3A supplémentaire) pour ouvrir l’éditeur de programme d’impulsions.
- Utilisez le flux de travail suivant pour enregistrer la séquence d’impulsions sous un nouveau nom : Cliquez sur Fichier (coin supérieur gauche), Enregistrer sous, entrez le nouveau nom, puis cliquez sur OK. Cliquez ensuite sur Définir PULPROG sur l’ensemble de données (voir la figure supplémentaire 3B) et cliquez sur OK.
- Modifier la séquence HSQC originale 1 H-15N (enregistrée sous un nouveau nom, voir 2.6.4.) en multipliant les 90° (15N) après la période d’évolution de t1 (avant le rétrotransfert) par un facteur 2 (l’impulsion de 90° (15N) est maintenant de 180° (15N)). Appuyez sur Ctrl S pour enregistrer la séquence d’impulsions. Réglez la longueur d’impulsion à 90° (15N) (P21) de sorte que le signal RMN du premier incrément disparaisse (au passage à zéro) (figure supplémentaire 3B).
- Ouvrez l’expérience HSQC 1 H-15N (expérience 2D) et entrez la longueur d’impulsion calibrée à 90° (15N) (P21). Type getprosol 1H 1H) en μs> <90° (1H) intensité d’impulsion en db> 15N 15N) en μs> <90° (15N) intensité d’impulsion en db> dans la ligne de commande.
- Déterminer les largeurs spectrales et l’appropriation des temps d’acquisition.
- Dans la fenêtre des paramètres d’acquisition de l’expérience HSQC 1 H-15N (tapez eda dans la ligne de commande), ajustez la largeur spectrale (paramètre d’acquisition SW) et le nombre d’incréments de dimension en dimensions directes et indirectes (paramètre d’acquisition TD).
- Choisissez pour SW , 16 ppm dans la dimension directe (1H) et 30 ppm dans la dimension indirecte (15N). Choisissez pour TD, 2048 dans la dimension directe (1H) et 512 dans la dimension indirecte (15N).
- Acquérir l’expérience HSQC 1 H-15N.
- Vérifiez que tous les signaux d’intérêt sont détectés et qu’aucun artefact de repliement n’est présent dans le spectre. Modifiez l’O3P si le centre du spectre ne correspond pas au centre de la distribution du signal protéique le long de l’axe 15N.
- Augmentez le SW si la fenêtre spectrale est trop petite. Augmentez le TD pour obtenir une résolution FID plus élevée (vérifiez les spécifications de la sonde pour la durée maximale de découplage).
REMARQUE : Il s’agit de la configuration générale et du point de départ pour la configuration des expériences de relaxation RMN. Il est recommandé d’enregistrer l’expérience HSQC 1 H-15N au début et à la fin de l’ensemble des expériences et, pour des durées d’expérience plus longues, également entre les deux pour surveiller l’intégrité de l’échantillon.
- Dans la fenêtre des paramètres d’acquisition de l’expérience HSQC 1 H-15N (tapez eda dans la ligne de commande), ajustez la largeur spectrale (paramètre d’acquisition SW) et le nombre d’incréments de dimension en dimensions directes et indirectes (paramètre d’acquisition TD).
3. Mise en œuvre des expériences de relaxation RMN
REMARQUE : Les séquences d’impulsions de relaxation RMN (Figure 2) sont disponibles à https://www.ipb.hhu.de/en/teams/team-lakomek/pulsesequences ou au dépôt étendu de la Banque de résonance magnétique biologique (BMRB) (bmrbig102).
- Configurez le 15N R1ρ expérience.
- Copiez l’expérience HSQC (type edc) dans un nouveau répertoire. Tapez ased et changez le programme d’impulsions pour l’expérience 15N R1ρ en cliquant sur les trois points de la ligne de paramètres PULPROG dans les paramètres de séquence d’impulsions (ased, voir figure supplémentaire 3A). Double-cliquez sur le programme d’impulsions, cliquez sur Définir PULPROG sur l’ensemble de données (voir la figure supplémentaire 3B), puis cliquez sur OK.
REMARQUE : Certains messages d’erreur peuvent apparaître. Fermez ces messages. - Tapez gpnam dans la ligne de commande pour insérer toutes les formes de dégradé manquantes (gpnam1- gpnam9) comme décrit dans la séquence d’impulsions (Figure supplémentaire 4).
- Tapez vplist dans la ligne de commande. Cliquez sur les trois points (Figure supplémentaire 5A), Fichier (coin supérieur gauche) et Nouveau (Figure supplémentaire 5B).
- Tapez un nom pour un nouveau vplist factice, puis appuyez sur OK. Créez le vplist factice (5m, 65m, 25m, 45m, 15m, 55m, 35m, 10m ; une nouvelle ligne pour chaque entrée vplist). Appuyez sur Ctrl S pour enregistrer la liste et fermer la fenêtre de la vplist (Figure supplémentaire 5C).
- Double-cliquez sur le nom de la vplist nouvellement créée (Figure supplémentaire 5B).
REMARQUE : Ceci est nécessaire pour éviter les erreurs de compilation dans la séquence d’impulsions. Les entrées appropriées de la vplist seront déterminées plus loin (point 3.1.29.). - Tapez ased pour ouvrir les paramètres de séquence d’impulsions (Figure supplémentaire 6). Entrez toutes les intensités de gradient et les longueurs de gradient manquantes telles qu’elles sont indiquées dans la séquence d’impulsions (figure supplémentaire 6D).
- Entrez pour le compteur de boucles L3 (nombre de points complexes dans la dimension indirecte) 1/2 de l’entrée TD 15N dans la fenêtre des paramètres d’acquisition (eda). Entrez pour le compteur de boucles L6 le nombre de points de données de relaxation enregistrés utilisés pour l’ajustement de la courbe de relaxation.
REMARQUE : Ce numéro L6 correspond au nombre d’entrées vplist, ici : 8 (Figure supplémentaire 6A). - Définissez l’impulsion adiabatique TanhTan_half.nl, téléchargée à partir du site Web spécifié ci-dessus, comme la forme de l’impulsion 15N SP8. Réglez l’impulsion adiabatique TanhTan_2nd.nl du site Web sur la forme de l’impulsion 15N SP9 (figure supplémentaire 6C).
- Réglez l’entrée P8 sur la longueur de l’impulsion adiabatique de TanhTan - suffisamment longue pour garantir l’adiabaticité de l’impulsion ; ici, on choisit P8 = 3000 μs (figure supplémentaire 6C).
- Définissez le découplage 15N (CPDPRG 3) sur un schéma de découplage garp. À utiliser pour la longueur de découplage PCPD3 200 μs (figure supplémentaire 6C).
- Réglez le délai de récupération entre les balayages sur au moins D1 = 2 s ou plus.
REMARQUE : Pour le choix d’un délai de récupération approprié, consultez la section Discussion. - Réglez les balayages factices (DS) à au moins 64 pour l’expérience finale (lors de la configuration, DS = 4 est suffisant pour des raisons de temps). Comme point de départ, utilisez un nombre de balayages (NS) de 4 ; utiliser des multiples de 4 si le rapport signal/bruit (SNR) est trop faible (Figure supplémentaire 6A).
- Réglez l’O1 sur la fréquence porteuse calibrée de l’étape 2.5. Réglez l’O2P à 176 ppm et copiez l’O3P de l’expérience HSQC 1 H-15N (figure supplémentaire 6A-C).
- Réglez la longueur d’impulsion P7 sur la longueur d’impulsion de 90° (15N) étalonnée à l’étape 2.6. De plus, copiez le niveau de puissance d’impulsion de 90° (15N) de l’étape 2.6 sur PLW3 et PLW7 (Figure supplémentaire 6C).
- Réglez la longueur d’impulsion P1 et P19 sur la longueur d’impulsion de 90° (1H) calculée à l’étape 2.4. (Figure supplémentaire 6B).
- Définissez le nombre d’incréments dans la dimension indirecte TD = l3 * 2 * l6 (eda ou tapez td dans la ligne de commande).
REMARQUE : Tous les paramètres de séquence d’impulsions décrits ici sont également annotés dans la séquence d’impulsions. - Réglez la forme pulse SP5 sur une forme I-BURP2 (Iburp2.1000). Réglez la longueur d’impulsion P15 sur 2000 μs. Ouvrez l’affichage de l’outil de forme en cliquant sur le E à côté de l’impulsion de forme I-BURP2 dans la fenêtre de paramètre de séquence d’impulsion Topspin (ased) (Figure supplémentaire 6B).
- Simulez l’impulsion de forme avec la bonne longueur en cliquant sur le bouton Démarrer la simulation RMN. Vérifiez la longueur d’impulsion profilée [μs] (= 2000 μs) et l’angle de rotation [°] (= 180°) dans la fenêtre de simulation. Cliquez sur Démarrer NMR-SIM (Figure supplémentaire 7A).
- Vérifiez la plage d’excitation et choisissez la longueur d’impulsion I-BURP2 appropriée pour couvrir la dispersion spectrale 1 H dela protéine, mais évitez d’exciter l’eau en même temps. Réglez P15 sur la longueur d’impulsion façonnée de la fenêtre de simulation avec la meilleure impulsion IBURP-2 simulée (Figure supplémentaire 7B).
REMARQUE : La longueur appropriée est choisie si l’I-BURP2 excite (> efficacité de 95%) toute la région de l’amide. Pour simuler une impulsion I-BURP2 d’une longueur différente, fermez la fenêtre du profil d’excitation et modifiez la longueur de l’impulsion façonnée dans la fenêtre de simulation. Cliquez sur Démarrer NMR-SIM. Les impulsions de longueur plus courte ont une plage d’excitation plus large en Hz. - Réglez SPOFFS5 pour changer la fréquence porteuse de l’impulsion I-BURP2 et décaler la plage d’excitation vers la gauche ou la droite pour éviter que l’impulsion I-BURP2 ne touche et ne perturbe l’aimantation de l’eau (Figure supplémentaire 6B).
- Pour déterminer le niveau de puissance approprié de l’impulsion façonnée, ouvrez l’outil Forme Bruker, cliquez sur le bouton Démarrer la simulation RMN et réglez la longueur d’impulsion I-BURP2 sur Longueur d’impulsion façonnée [μs]. Rappelez-vous la longueur correspondante d’une impulsion rectangulaire douce de 90° (1H), visible dans la fenêtre de simulation à l’impulsion carrée de 90° correspondante [μs] (Figure supplémentaire 7A).
- Type calcpowlev. Utiliser l’impulsion dure de 90° (1H) (P1) en microsecondes comme longueur de référence (figure supplémentaire 8B) et l’impulsion rectangulaire douce de 90° (1H) correspondant à l’impulsion I-BURP2 en microsecondes comme nouvelle longueur (figure supplémentaire 8A). Rappelez-vous la différence de puissance Δ en dB entre l’impulsion dure de 90° (1H) et l’impulsion rectangulaire douce (Figure supplémentaire 8C). Copiez l’intensité d’impulsion de l’impulsion dure de 90° (1H) en dB dans SPW5 et ajoutez la différence mémorisée Δ en dB (figure supplémentaire 6B).
- Pour déterminer le niveau de puissance du verrou de rotation, calculez la longueur d’impulsion correspondante de 90° (15N) du verrou de rotation. Utilisez la formule : longueur d’impulsion de 90° (15N) en s = 0,25 / (puissance de verrouillage de spin en Hz).
- Utilisez calcpowlev pour calculer la différence de puissance Δ en dB entre la puissance du spin-lock et l’impulsion dure à 90° (15N) (P7). Nouveau : longueur d’impulsion de 90° (15N) du verrou de spin en μs (figure supplémentaire 8A). Réf : longueur d’impulsion dure de 90° (15N) en μs (figure supplémentaire 8B). Copiez le niveau de puissance PLW7 de l’impulsion dure de 90° (15N) en dB sur le niveau de puissance de verrouillage de spin PLW8 (figure supplémentaire 6C) et ajoutez la différence de puissance calculée Δ en dB (figure supplémentaire 8C).
- Copiez le niveau de puissance du verrou rotatif PLW8 dans les niveaux de puissance SPW8 et SPW9. (Figure supplémentaire 6C).
- Pour déterminer le niveau de puissance de l’impulsion de découplage 15N, utilisez calcpowlev pour calculer la différence de puissance Δ entre la puissance de l’impulsion de découplage à 90° (15N) (PCPD3) et l’impulsion dure à 90° (15N) (P7). Nouveau : PCPD3 en μs (200) (figure supplémentaire 8A). Réf : longueur d’impulsion dure de 90° (15N) en μs (figure supplémentaire 8B). Copiez le niveau de puissance PLW7 de l’impulsion dure de 90° (15N) en dB sur le niveau de découplage PLW31 (figure supplémentaire 6C) et ajoutez Δ en dB (figure supplémentaire 8C).
- Pour utiliser la compensation de température, incluez #define TEMP_COMPENSATION (en supprimant le point-virgule dans la séquence d’impulsions). Régler P18 sur la durée maximale du verrou de spin utilisé dans l’expérience 15N R1ρ .
REMARQUE : Il est recommandé de désactiver la compensation de température pour le premier essai de l’expérience. - Dans le cas d’échantillons marqués 15N 13C, inclure # pour définir LABEL_CN dans le programme d’impulsions (en supprimant le point-virgule dans la séquence d’impulsions). Réglez P4 sur la longueur d’impulsion calculée avec la formule en78. Par exemple, il correspond à 23,70 μs à 600 MHz et 11,85 μs à 1200 MHz. Tapez calcpowlev pour calculer la différence de puissance en dB entre P4 et l’impulsion dure (13C). Copiez la puissance d’impulsion dure (13C) en dB dans PLW4 et ajoutez le Δ en dB. Copiez PLW4 dans PLW2 (Figure supplémentaire 6B).
- Déterminer les délais de relaxation pour un échantillonnage approprié.
- Exécutez les huit premiers FID (dans le cas L6 = 8) et traitez-les (rser < nouveau numéro d’expérience>). Choisissez les entrées de la liste des utilisateurs indiquant que l’intensité maximale de l’expérience avec le retard le plus long diminue à au moins 1/e (environ 37%), mais pas moins de 25% par rapport à l’expérience avec le retard le plus court (qui sert de référence).
- Ajustez les entrées restantes de la vplist pour obtenir un échantillonnage uniforme des diminutions d’intensité entre le FID avec le retard le plus long et le FID avec le retard le plus court. Lisez la REMARQUE et les spécifications de puissance de la sonde.
REMARQUE : Les résidus qui se relâchent lentement domineront le premier FID. Ajustez les entrées restantes de la vplist pour obtenir un échantillonnage uniforme des diminutions d’intensité entre le premier et le deuxième FID. Ne dépassez pas la puissance et la longueur maximales autorisées de la sonde (consultez le manuel de la sonde et les spécifications de la sonde). En cas de compensation de température, cochez P18 (durée maximale de spin-lock), car la durée globale de spin-lock de P18 sera appliquée, répartie entre le retard de relaxation et la compensation de température. Il contient le temps (délai de relaxation) + le temps (compensation de température) = const = P18. Pour un spin-lock de 2 kHz, on préfère ne pas dépasser une durée de spin-lock supérieure à 65 ms, en utilisant un délai de récupération (D1) de 2 s. Dans ce cas, un compromis entre la durée maximale du spin-lock et le délai de relaxation le plus long doit être pris en faveur de la durée maximale autorisée du spin-lock. En cas de délais de récupération D1 plus longs, des puissances de verrouillage de rotation plus longues peuvent être applicables. Ce qui compte, c’est le dépôt de puissance global dans la sonde au fil du temps. Si des durées de verrouillage plus longues sont nécessaires, n’augmentez les longueurs de verrouillage que par petits pas et surveillez le signal de verrouillage. En règle générale, ajustez le niveau de verrouillage dans le tiers supérieur de l’écran de votre cadenas. Si le niveau de verrouillage diminue de plus de deux cases pendant l’exécution de la séquence d’impulsions, la puissance de verrouillage de spin déposée dans la sonde est trop importante.
- Tapez rga dans la ligne de commande pour déterminer le gain du récepteur. Pour un bon rapport signal/bruit, visez une valeur de gain récepteur (rg) comprise entre 128 et 256, ce qui prouve une bonne suppression de l’eau.
REMARQUE : Un gain de récepteur compris entre 128 et 256 ne peut pas être atteint indique un signal d’eau trop fort. Dans ce cas, la puissance et les durées du gradient sont ajustées doucement jusqu’à ce que le gain du récepteur soit atteint. Voir aussi la section Discussion. - Démarrez une série d’essai de l’expérience 15N R1ρ en tapant zg dans la ligne de commande. Vérifiez que le signal d’eau est bien supprimé pour tous les retards (1 à 8, en cas de 8 retards dans la vplist). Vérifiez également l’incrément 9, qui est le deuxième incrément du schéma de détection en quadrature (Echo/ Anti-Echo).
- Copiez l’expérience HSQC (type edc) dans un nouveau répertoire. Tapez ased et changez le programme d’impulsions pour l’expérience 15N R1ρ en cliquant sur les trois points de la ligne de paramètres PULPROG dans les paramètres de séquence d’impulsions (ased, voir figure supplémentaire 3A). Double-cliquez sur le programme d’impulsions, cliquez sur Définir PULPROG sur l’ensemble de données (voir la figure supplémentaire 3B), puis cliquez sur OK.
- Configurez l’expérience 15N R1 .
- Copiez l’expérience 15N R1ρ et remplacez le programme d’impulsions par l’expérience 15N R1 .
- Tapez vclist, générez une vclist et entrez la vclist factice décrite à lasection 70 : 0, 24, 6, 20, 4, 16, 8, 12 ; une nouvelle ligne pour chaque entrée vclist).
- Ouvrez l’ased. Vérifiez les longueurs et les intensités des pentes. Changez GPZ9 à 14 %.
- Vérifiez la longueur des impulsions, la forme des impulsions et l’intensité des impulsions (les valeurs correctes doivent avoir été copiées de l’expérience 15N R1ρ ).
- Dans le cas d’échantillons marqués 15N13C, utiliser #define LABEL_CN dans le programme d’impulsions.
- Pour l’utilisation de la compensation de température, inclure #define TEMP_COMPENSATION.
- Exécutez les huit premiers FID (dans le cas L6 = 8) et traitez-les (rser < nouveau numéro d’expérience>). Choisissez les entrées de la vclist indiquant que l’intensité maximale de l’expérience avec le retard le plus long diminue à au moins 1/e (environ 37 %) mais pas moins de 25 % par rapport à l’expérience avec le retard le plus court. Ajustez les entrées restantes de la vplist pour obtenir un échantillonnage uniforme des diminutions d’intensité entre le FID avec le retard le plus long et le FID avec le retard le plus court.
REMARQUE : Seuls les nombres pairs sont autorisés pour les entrées vclist.
- Configurez l’expérience {1H}-15N NOE.
- Copiez l’expérience de relaxation 15N R1 et changez la séquence d’impulsions en schéma d’impulsions {1H}-15N NOE.
- Réglez la durée de la période de saturation à l’aide du compteur de boucles L8. L’espacement entre les impulsions de 180° (1H) est de 22 ms79 (2* 1 / JNH). Pour obtenir les résultats les plus précis, utilisez une période de saturation d’environ 5 fois les 15N T1(15N) de la protéine. Ici : 5 s. Cela correspondrait à L8 = 230.
REMARQUE : La section Discussion fournit des conseils supplémentaires sur le choix de la période de saturation/délai de récupération. - Modifiez la longueur et l’intensité des pentes comme décrit dans le schéma d’impulsions. Utilisez des formes dégradées sine.20.
- Définissez le nombre d’incréments dans la dimension indirecte égal à TD = l3 * 2 * 2
- Dans le cas d’échantillons marqués 15N13C, utiliser #define LABEL_CN et -DLABEL_CN comme ZGOPTNS.
- Exécutez les schémas d’impulsions en tapant zg dans la ligne de commande.
4. Traitement et analyse des expériences RMN enregistrées
REMARQUE : Les spectres ont été enregistrés à l’aide d’un système Bruker. Le traitement est effectué à l’aide d’un système d’exploitation Unix ou Linux. Le traitement des spectres et l’analyse des données ont été effectués à l’aide de NMRPipe80 et python3. Le logiciel NMRPipe peut être téléchargé à l’adresse https://www.ibbr.umd.edu/nmrpipe/index.html. Les scripts de traitement basés sur la NMRPipe sont téléchargés à partir du site Web : https://www.ipb.hhu.de/en/teams/team-lakomek/pulsesequences ou à partir du référentiel étendu de la Banque de Résonance Magnétique Biologique (BMRB) (bmrbig102). Il est recommandé d’utiliser NMRPipe. Si NMRPipe n’est pas disponible ou souhaité, des alternatives telles que CCPN81 ou SPARKY (SPARKY 3, ou ses successeurs NMRFAM-SPARKY82 ou POKY83 ) peuvent être utilisées.
- Traiter et analyser 15N R1 expérience.
REMARQUE : Les commandes suivantes sont exécutées dans un terminal Unix.- Copiez les fichiers du répertoire téléchargé R1_processing dans le répertoire contenant les données expérimentales R1 (répertoire de travail).
- Rendez les fichiers exécutables par la commande terminal : > chmod u+x *.com
- Exécutez > bruker pour ouvrir l’utilitaire de conversion NMRPipe et générer le fichier fid.com. Cliquez sur Lire les paramètres. Définissez le nombre de dimensions sur deux si trois dimensions sont comptées, puis cliquez à nouveau sur Lire les paramètres . Cliquez ensuite sur Effacer le script, Mettre à jour le script et Enregistrer le script (Figure 9 supplémentaire).
REMARQUE : Si la fréquence porteuse n’est pas correcte, modifiez la position centrale PPM après avoir cliqué sur Lire les paramètres une deuxième fois. - Exécuter > ./fid.com
- Convertissez le vclist en vplist avec ms entries : delay in ms = 40 ms * entrée de vclist. Utilisez > cp vclist vplist et éditez le vplist.
- Utilisez > ./split.com pour diviser les pseudo-données 3D en huit fichiers 2D.
- Modifiez les valeurs tauValues (valeurs vplist dans l’ordre de la vplist) et les valeurs idxExpmt (ordre des tauValues par rapport au temps) dans nmrproc.com (Figure supplémentaire 10).
- Utilisez > ./nmrproc.com pour traiter les données brutes des huit spectres 2D.
- Utilisez > nmrDraw pour dessiner les spectres.
- Utilisez le spectre de référence (première entrée dans vplist) pour ces étapes. Basculez vers ce spectre en changeant la dimension z en un (coin supérieur gauche). Ajustez le niveau de contour à l’aide du + et du - à côté du bouton Facteur . Redessinez le spectre avec le bouton Dessiner.
- Utilisez un niveau de contour où tous les signaux d’intérêt peuvent être reconnus, avec seulement quelques signaux provenant du bruit (le niveau de contour est à la limite du niveau de bruit) (Figure supplémentaire 11).
- Corrigez la phase spectrale, de type v pour la projection verticale du signal 1D et h pour la projection horizontale du signal 1D. Ajustez la phase avec P0 et P1 (Figure supplémentaire 11), mémorisez la différence de phase et corrigez la phase dans le fichier nmrproc.com (Figure supplémentaire 10).
REMARQUE : Si le spectre est inversé dans la dimension 15N, supprimez le -neg dans la ligne : | nmrPipe -fn FT -neg \ (Figure supplémentaire 10). - Fermez nmrDraw et traitez à nouveau avec > nmrproc.com. Pour la sélection des pics (une fois que le spectre est correctement mis en phase et que le spectre de référence est affiché avec un niveau de contour où tous les pics sont présents), tapez K (décalage + k), appuyez sur Détecter et cliquez sur Enregistrer. (Figure supplémentaire 11).
- Copiez une liste de pics dans le répertoire de travail et remplacez -assName dans ass.com par le nom de la liste de pics (Figure supplémentaire 12).
REMARQUE : Un exemple de liste de pics dans le bon format est disponible à l’adresse https://www.ipb.hhu.de/en/teams/team-lakomek/pulsesequences
et au dépôt étendu de la Banque de résonance magnétique biologique (BMRB) (bmrbig102). - Exécutez > ./ass.com pour attribuer les spectres. Vérifiez l’affectation suggérée par le script pour les différents pics. Utilisez le bouton gauche de la souris pour attribuer et le bouton droit de la souris pour désattribuer les pics. Utilisez le bouton Suivant pour accéder à l’acide aminé suivant et Prev pour accéder à l’acide aminé précédent (Figure supplémentaire 13).
REMARQUE : Le niveau de contour peut être modifié avec + et - dans le coin supérieur droit. Utilisez le bouton Draw pour redessiner le spectre avec le nouveau niveau de contour (Figure supplémentaire 13). - Une fois le devoir terminé, cliquez sur Enregistrer , puis sur Enregistrer à nouveau. Ensuite, cliquez sur Quitter, Annuler et Quitter (Figure supplémentaire 13).
- Exécutez > python3 NoneDeleter.py pour créer relax.tab, qui ne contient que des informations sur les pics attribués.
- Exécutez > ./relax.com pour générer le script autofit.com.
- Exécutez > ./autoFit.com pour effectuer une interpolation de Fourier et générer des pseudo-rapports des différents temps de retard pour chaque acide aminé (axt.tab et nlin.tab sont générés).
- Exécutez > ./model.com pour générer le script modelExp.com basé sur les données de nlin.tab.
- Exécutez > ./modelExp.com pour ajuster la courbe d’évolution de chaque résidu avec un modèle d’optimisation des moindres carrés non linéaire.
- Exécutez > bash summary.tcl -in relax.tab > t1.tab. Ignorez un message d’erreur potentiel : GDB Remarque : 21 valeurs manquantes dans le fichier de table peuvent se produire.
- Copiez t1.tab dans le répertoire R1R2 que vous venez de créer.
- Traiter et analyser l’expérience 15N R1ρ .
- Copiez les fichiers du répertoire téléchargé R1rho_processing dans le répertoire contenant les données expérimentales R1ρ (répertoire de travail).
- Exécuter > chmod u+x *.com
- Exécutez > bruker pour ouvrir l’utilitaire de conversion NMRPipe et générer le fichier fid.com. Voir aussi le traitement des spectres 15N R1 .
- Exécuter > ./fid.com
- Exécutez > ./split.com pour diviser les pseudo-données 3D en huit fichiers 2D.
- Exécutez > ./nmrproc.com (modifiez les tauValues [valeurs vplist] et idxExpmt au préalable) pour traiter les données brutes des huit spectres 2D (Figure supplémentaire 10).
- Exécutez > nmrDraw pour dessiner les spectres et choisir les pics. Utilisez le spectre de référence (première entrée dans vplist) pour ces étapes. Suivez la même procédure que pour l’expérience 15N R1 .
- Copiez une liste de pics dans le répertoire de travail et remplacez -assName dans ass.com par le nom de la liste de pics (Figure supplémentaire 12).
- Exécuter > ./ass.com et suivre la même procédure que pour l’expérience 15N R1 .
- Exécutez > python3 NoneDeleter.py pour créer relax.tab, qui ne contient que des informations sur les pics attribués.
- Exécuter > ./relax.com
- Exécuter > ./autoFit.com
- Exécuter > ./model.com
- Exécuter > ./modelExp.com
- Exécutez > bash summary.tcl -in relax.tab > t1rho. tab. Ignorer le message d’erreur potentiel : GDB Remarque : 21 valeurs manquantes dans le fichier de table peuvent se produire.
- Copiez l’en-tête suivant dans le fichier t1rho.tab (les valeurs peuvent être obtenues à partir du fichier fid.com) (Figure supplémentaire 14) :
REMARQUE 600MHz pour T1rho
REMARQUE -yOBS = 60.818 (mettre ici la valeur trouvée dans fid.com)
REMARQUE -yCAR = 119,06 (mettre ici la valeur trouvée dans fid.com) - Exécutez > ./extract_PPM.com pour générer une table appelée fusionOffset.tab qui contient le décalage de la fréquence de crête 15N par rapport à la fréquence porteuse (qui sera nécessaire pour calculer l’angle θ pour l’extraction des données R2 à partir des données R1 et R1ρ ).
- Résumez les données de relaxation.
- Copiez les fichiers t1rho.tab et fusionOffset.tab dans le répertoire R1R2.
- Copiez le script crtR1p.tcl dans le répertoire R1R2, ajustez la fréquence du spectromètre et la puissance du verrouillage de spin-lock, et exécutez bash crtR1p.tcl | sort -n > R1R2.tab. (Figure supplémentaire 15). R1R2.tab contiendra les constantes de vitesse R1ρ ajustées, l’angle thêta, les constantes de vitesse R2 et les constantes de vitesse R1 .
- Traiter et analyser l’expérience hetNOE.
- Copiez les fichiers du répertoire téléchargé NOE_processing dans le répertoire de données expérimental NOE (répertoire de travail).
- Exécuter > chmod u+x *.com
- Exécutez > bruker pour ouvrir l’utilitaire de conversion NMRPipe et générer le fichier fid.com. Cliquez sur Lire les paramètres. Remplacez le nombre de dimensions par deux si trois dimensions ont été comptées et appuyez à nouveau sur Lire les paramètres . Appuyez ensuite sur Effacer le script, Mettre à jour le script et Enregistrer le script.
- Exécuter > ./fid.com
- Exécuter > ./split.com (un vplist avec deux entrées (0, 1) doit être créé avant)
- Exécuter > ./ft2.com
- Exécutez > nmrDraw pour dessiner le spectre et choisir les pics dans le spectre de référence (dimension z = 1). Extrayez et mémorisez le bruit (appuyez sur T = Maj + t). La correction de phase et le pic de sélection suivent la même procédure que l’expérience 15N R1 .
- Copiez une liste de pics dans le répertoire de travail et remplacez -assName dans ass.com par le nom de la liste de pics (Figure supplémentaire 12)
- Exécutez > ./ass.com pour attribuer les spectres. Suivez la même procédure que celle décrite pour l’expérience 15N R1 .
- Exécutez > python3 NoneDeleter.py pour créer relax.tab.
- Exécuter > ./script.com
- Exécuter > ./autoFit.com
- Modifiez le bruit dans error_hetnoe.tcl.
- Exécuter > bash error_hetnoe.tcl |sort -n > hetNOE.tab
Access restricted. Please log in or start a trial to view this content.
Résultats
Ce qui suit présente quelques données exemplaires de relaxation RMN enregistrées sur la protéine vésiculaire SNARE Synaptobrevin-2 (1-96), fréquemment appelée VAMP2 (vésicule-associated protein 2). Pour l’enregistrement des données RMN, nous avons utilisé un échantillon de 171 μM 15N Synaptobrevin-2 (1-96) (surnommé Syb-2 ci-dessous) dans un tampon MES de 50 mM (pH 6,0) contenant 150 mM de NaCl, 0,1 mM de TCE et 1 mM d’EDTA. Toutes les données expérimentales...
Access restricted. Please log in or start a trial to view this content.
Discussion
Ce protocole décrit la mise en place d’expériences de relaxation RMN 15N par Lakomek et al.69 et Stief et al.70. Nous nous sommes concentrés sur les séquences d’impulsions RMN à l’aide d’un schéma de détection HSQC à sensibilité améliorée. Les expériences 15N R1 et R1ρ sont mises en œuvre comme décrit en détail par Stief et al.70, et l’expérience hetNOE est décrite par ...
Access restricted. Please log in or start a trial to view this content.
Déclarations de divulgation
Les auteurs déclarent qu’ils n’ont pas d’intérêts financiers concurrents connus ou de relations personnelles qui auraient pu sembler influencer les travaux rapportés dans cet article.
Remerciements
Nous remercions Melinda Jaspert et Kevin Bochinsky pour les discussions utiles. N.L. remercie la Fondation allemande pour la science pour son financement par le biais du programme Heisenberg (numéro de subvention DFG 433700474). Ce travail est également soutenu par le projet « Virological and immunological determinants of COVID-19 pathogenesis - lessons to get prepared for future pandemics » (KA1-Co-02 « COVIPA »), une subvention du Fonds d’initiative et de mise en réseau de l’Association Helmholtz. Nous reconnaissons l’accès généreux au Centre de RMN biomoléculaire de Jülich-Düsseldorf, géré conjointement par le Forschungszentrum Jülich et l’Université Heinrich Heine de Düsseldorf (HHU).
Access restricted. Please log in or start a trial to view this content.
matériels
| Name | Company | Catalog Number | Comments |
| Bruker 600 MHz AVANCE III HD spectrometer | Bruker | https://www.bruker.com/en/products-and-solutions/mr/nmr/avance-nmr-spectrometer.html | NMR experiments conducted |
Références
- Sekhar, A., Kay, L. E. An NMR view of protein dynamics in health and disease. Annu Rev Biophys. 48, 297-319 (2019).
- Rosenzweig, R., Kay, L. E. Bringing dynamic molecular machines into focus by methyl-TROSY NMR. Annu Rev Biochem. 83, 291-315 (2014).
- Palmer, A. G. 3rd Enzyme dynamics from NMR spectroscopy. Acc Chem Res. 48 (2), 457-465 (2015).
- Reif, B. Ultra-high resolution in MAS solid-state NMR of perdeuterated proteins: implications for structure and dynamics. J Magn Reson. 216, 1-12 (2012).
- Schanda, P., Ernst, M. Studying dynamics by magic-angle spinning solid-state NMR spectroscopy: Principles and applications to biomolecules. Prog Nucl Magn Reson Spectrosc. 96, 1-46 (2016).
- Le Marchand, T., et al. (1)H-H-detected biomolecular NMR under fast magic-angle spinning. Chem Rev. 122 (10), 9943-10018 (2022).
- Pervushin, K., Riek, R., Wider, G., Wuthrich, K. Attenuated T2 relaxation by mutual cancellation of dipole-dipole coupling and chemical shift anisotropy indicates an avenue to NMR structures of very large biological macromolecules in solution. Proc Natl Acad Sci U S A. 94 (23), 12366-12371 (1997).
- Tzakos, A. G., Grace, C. R., Lukavsky, P. J., Riek, R. NMR techniques for very large proteins and RNAs in solution. Annu Rev Biophys Biomol Struct. 35, 319-342 (2006).
- Sprangers, R., Kay, L. E. Quantitative dynamics and binding studies of the 20S proteasome by NMR. Nature. 445 (7128), 618-622 (2007).
- Wright, P. E., Dyson, H. J. Intrinsically disordered proteins in cellular signalling and regulation. Nat Rev Mol Cell Biol. 16 (1), 18-29 (2015).
- van der Lee, R., et al. Classification of intrinsically disordered regions and proteins. Chem Rev. 114 (13), 6589-6631 (2014).
- Tompa, P. Intrinsically unstructured proteins. Trends Biochem Sci. 27 (10), 527-533 (2002).
- Dyson, H. J., Wright, P. E. Coupling of folding and binding for unstructured proteins. Curr Opin Struct Biol. 12 (1), 54-60 (2002).
- Uversky, V. N. Intrinsically disordered proteins in overcrowded milieu: Membrane-less organelles, phase separation, and intrinsic disorder. Current Opinion in Structural Biology. 44, 18-30 (2017).
- Patel, A., et al. A liquid-to-solid phase transition of the ALS protein FUS accelerated by disease mutation. Cell. 162 (5), 1066-1077 (2015).
- Abyzov, A., Blackledge, M., Zweckstetter, M. Conformational dynamics of intrinsically disordered proteins regulate biomolecular condensate chemistry. Chem Rev. 122 (6), 6719-6748 (2022).
- Uversky, V. N. Unusual biophysics of intrinsically disordered proteins. Biochim Biophys Acta. 1834 (5), 932-951 (2013).
- Papoian, G. A. Proteins with weakly funneled energy landscapes challenge the classical structure-function paradigm. Proc Natl Acad Sci U S A. 105 (38), 14237-14238 (2008).
- Uversky, V. N., Gillespie, J. R., Fink, A. L. Why are "natively unfolded" proteins unstructured under physiologic conditions. Proteins. 41 (3), 415-427 (2000).
- Bah, A., et al. Folding of an intrinsically disordered protein by phosphorylation as a regulatory switch. Nature. 519 (7541), 106-109 (2015).
- Tompa, P., Schad, E., Tantos, A., Kalmar, L. Intrinsically disordered proteins: emerging interaction specialists. Curr Opin Struct Biol. 35, 49-59 (2015).
- Dyson, H. J., Wright, P. E. Perspective: the essential role of NMR in the discovery and characterization of intrinsically disordered proteins. J Biomol NMR. 73 (12), 651-659 (2019).
- Adamski, W., et al. A unified description of intrinsically disordered protein dynamics under physiological conditions using NMR spectroscopy. J Am Chem Soc. 141 (44), 17817-17829 (2019).
- Dobson, C. M. Protein folding and misfolding. Nature. 426 (6968), 884-890 (2003).
- Bertoncini, C. W., et al. Release of long-range tertiary interactions potentiates aggregation of natively unstructured alpha-synuclein. Proc Natl Acad Sci U S A. 102 (5), 1430-1435 (2005).
- Buell, A. K., et al. Solution conditions determine the relative importance of nucleation and growth processes in alpha-synuclein aggregation. Proc Natl Acad Sci U S A. 111 (21), 7671-7676 (2014).
- Salvi, N., Abyzov, A., Blackledge, M. Atomic resolution conformational dynamics of intrinsically disordered proteins from NMR spin relaxation. Prog Nucl Magn Reson Spectrosc. 102, 43-60 (2017).
- Schneider, R., Blackledge, M., Jensen, M. R. Elucidating binding mechanisms and dynamics of intrinsically disordered protein complexes using NMR spectroscopy. Curr Opin Struct Biol. 54, 10-18 (2019).
- Rezaei-Ghaleh, N., Blackledge, M., Zweckstetter, M. Intrinsically disordered proteins: from sequence and conformational properties toward drug discovery. Chembiochem. 13 (7), 930-950 (2012).
- Jensen, M. R., Salmon, L., Nodet, G., Blackledge, M. Defining conformational ensembles of intrinsically disordered and partially folded proteins directly from chemical shifts. J Am Chem Soc. 132 (4), 1270-1272 (2010).
- Camacho-Zarco, A. R., et al. NMR provides unique insight into the functional dynamics and interactions of intrinsically disordered proteins. Chem Rev. 122 (10), 9331-9356 (2022).
- Mittag, T., Forman-Kay, J. D. Atomic-level characterization of disordered protein ensembles. Curr Opin Struct Biol. 17 (1), 3-14 (2007).
- Dyson, H. J., Wright, P. E. Unfolded proteins and protein folding studied by NMR. Chem Rev. 104 (8), 3607-3622 (2004).
- Dyson, H. J., Wright, P. E. NMR illuminates intrinsic disorder. Curr Opin Struct Biol. 70, 44-52 (2021).
- Sugase, K., Dyson, H. J., Wright, P. E. Mechanism of coupled folding and binding of an intrinsically disordered protein. Nature. 447 (7147), 1021-1025 (2007).
- Bessa, L. M., et al. The intrinsically disordered SARS-CoV-2 nucleoprotein in dynamic complex with its viral partner nsp3a. Sci Adv. 8 (3), eabm4034(2022).
- Milles, S., Salvi, N., Blackledge, M., Jensen, M. R. Characterization of intrinsically disordered proteins and their dynamic complexes: From in vitro to cell-like environments. Prog Nucl Magn Reson Spectrosc. 109, 79-100 (2018).
- Salvi, N., Abyzov, A., Blackledge, M. Atomic resolution conformational dynamics of intrinsically disordered proteins from NMR spin relaxation. Prog Nucl Magn Reson Spectrosc. 102 - 103, 43-60 (2017).
- Gill, M. L., Byrd, R. A., Palmer, A. G. III Dynamics of GCN4 facilitate DNA interaction: a model-free analysis of an intrinsically disordered region. Phys Chem Chem Phys. 18 (8), 5839-5849 (2016).
- Bertoncini, C. W., et al. Structural characterization of the intrinsically unfolded protein beta-synuclein, a natural negative regulator of alpha-synuclein aggregation. J Mol Biol. 372 (3), 708-722 (2007).
- Rezaei-Ghaleh, N., et al. Local and global dynamics in intrinsically disordered synuclein. Angew Chem Int Ed Engl. 57 (46), 15262-15266 (2018).
- Rezaei-Ghaleh, N., Parigi, G., Zweckstetter, M. Reorientational dynamics of amyloid-beta from NMR spin relaxation and molecular simulation. J Phys Chem Lett. 10 (12), 3369-3375 (2019).
- Palmer, A. G. NMR characterization of the dynamics of biomacromolecules. Chem Rev. 104 (8), 3623-3640 (2004).
- Palmer, A. G., Massi, F. Characterization of the dynamics of biomacromolecules using rotating-frame spin relaxation NMR spectroscopy. Chem Rev. 106 (5), 1700-1719 (2006).
- Kay, L. E., Torchia, D. A., Bax, A. Backbone dynamics of proteins as studied by 15N inverse detected heteronuclear NMR spectroscopy: application to staphylococcal nuclease. Biochemistry. 28 (23), 8972-8979 (1989).
- Mittermaier, A., Kay, L. E. Review - New tools provide new insights in NMR studies of protein dynamics. Science. 312 (5771), 224-228 (2006).
- Abragam, A. Principles of Nuclear Magnetism. , Clarendon Press. Oxford. (1983).
- Ernst, R. R., Bodenhausen, G., Wokaun, A. Principles of Nuclear Magnetic Resonance in One and Two Dimensions. , Oxford University Press. Oxford. (1990).
- Cavanagh, J., Fairbrother, W. J., Palmer, A. G. III, Skelton, N. J. Protein NMR Spectroscopy: Principles and Practice. , Elsevier, Academic Press. (1995).
- Levitt, M. H. Spin Dynamics: Basics of Nuclear Magnetic Resonance. , John Wiley and Sons. (2008).
- Keeler, J. Understanding NMR Spectroscopy. , John Wiley and Sons. (2010).
- Bloembergen, N., Purcell, E. M., Pound, R. V. Relaxation effects in nuclear magnetic resonance absorption. Phys Rev. 73 (7), 679-712 (1948).
- Wangsness, R. K., Bloch, F. The dynamical theory of nuclear induction. Phys Rev. 89 (4), 728-739 (1953).
- Redfield, A. G. On the theory of relaxation processes. IBM J Res Dev. 1 (1), 19-31 (1957).
- Peng, J. W., Wagner, G. Mapping of the spectral densities of N-H bond motions in eglin c using heteronuclear relaxation experiments. Biochemistry. 31 (36), 8571-8586 (1992).
- Farrow, N. A., et al. Backbone dynamics of a free and a phosphopeptide-complexed src homology-2 domain studied by n-15 NMR relaxation. Biochemistry. 33 (19), 5984-6003 (1994).
- Tolman, J. R., Ruan, K. NMR residual dipolar couplings as probes of biomolecular dynamics. Chem Rev. 106 (5), 1720-1736 (2006).
- Mulder, F. A. A., Skrynnikov, N. R., Hon, B., Dahlquist, F. W., Kay, L. E. Measurement of slow (mu s-ms) time scale dynamics in protein side chains by N-15 relaxation dispersion NMR spectroscopy: Application to Asn and Gln residues in a cavity mutant of T4 lysozyme. J Am Chem Soc. 123 (5), 967-975 (2001).
- Tollinger, M., Skrynnikov, N. R., Mulder, F. A. A., Forman-Kay, J. D., Kay, L. E. Slow dynamics in folded and unfolded states of an SH3 domain. J Am Chem Soc. 123 (46), 11341-11352 (2001).
- Akke, M., Palmer, A. G. Monitoring macromolecular motions on microsecond to millisecond time scales by R(1)rho-R(1) constant relaxation time NMR spectroscopy. J Am Chem Soc. 118 (4), 911-912 (1996).
- Loria, J. P., Rance, M., Palmer, A. G. A relaxation-compensated Carr-Purcell-Meiboom-Gill sequence for characterizing chemical exchange by NMR spectroscopy. J Am Chem Soc. 121 (10), 2331-2332 (1999).
- Vallurupalli, P., Bouvignies, G., Kay, L. E. Studying "Invisible" excited protein states in slow exchange with a major state conformation. J Am Chem Soc. 134 (19), 8148-8161 (2012).
- Jeener, J., Meier, B. H., Bachmann, P., Ernst, R. R. Investigation of exchange processes by 2-dimensional NMR-spectroscopy. J Chem Phys. 71 (11), 4546-4553 (1979).
- Palmer, A. G., Kroenke, C. D., Loria, J. P. Nuclear magnetic resonance methods for quantifying microsecond-to-millisecond motions in biological macromolecules. Methods Enzymol. 339, 204-238 (2001).
- Clore, G. M., Iwahara, J. Theory, practice, and applications of paramagnetic relaxation enhancement for the characterization of transient low-population states of biological macromolecules and their complexes. Chem Rev. 109 (9), 4108-4139 (2009).
- Iwahara, J., Clore, G. M. Detecting transient intermediates in macromolecular binding by paramagnetic NMR. Nature. 440 (7088), 1227-1230 (2006).
- Lakomek, N. A., et al. Residual dipolar couplings as a tool to study molecular recognition of ubiquitin. Biochem Soc Trans. 36 (Pt 6), 1433-1437 (2008).
- Johnson, C. N., Libich, D. S. Paramagnetic relaxation enhancement for detecting and characterizing self-associations of intrinsically disordered proteins. J Vis Exp. (175), e63057(2021).
- Lakomek, N. A., Ying, J., Bax, A. Measurement of (1)(5)N relaxation rates in perdeuterated proteins by TROSY-based methods. J Biomol NMR. 53 (3), 209-221 (2012).
- Stief, T., Vormann, K., Lakomek, N. A. Sensitivity-enhanced NMR (15)N R(1) and R(1rho) relaxation experiments for the investigation of intrinsically disordered proteins at high magnetic fields. Methods. 223, 1-15 (2024).
- Kay, L. E., Keifer, P., Saarinen, T. Pure absorption gradient enhanced heteronuclear single quantum correlation spectroscopy with improved sensitivity. J Am Chem Soc. 114 (26), 10663-10665 (1992).
- Geen, H., Freeman, R. Band-selective radiofrequency pulses. J Magn Reson (1969). 93 (1), 93-141 (1991).
- Desvaux, H., Berthault, P. Study of dynamic processes in liquids using off-resonance RF irradiation. Prog Nucl Magn Reson Spectrosc. 35 (4), 295-340 (1999).
- Overhauser, A. W. Polarization of nuclei in metals. Phys Rev. 91 (2), 476-476 (1953).
- Overhauser, A. W. Polarization of nuclei in metals. Phys Rev. 92 (2), 411-415 (1953).
- Kumari, P., Frey, L., Sobol, A., Lakomek, N. A., Riek, R. (15)N transverse relaxation measurements for the characterization of micros-ms dynamics are deteriorated by the deuterium isotope effect on (15)N resulting from solvent exchange. J Biomol NMR. 72 (3-4), 125-137 (2018).
- Karschin, N., Krenek, S., Heyer, D., Griesinger, C. Extension and improvement of the methanol-d(4) NMR thermometer calibration. Magn Reson Chem. 60 (4), 203-209 (2022).
- Sattler, M., Schleucher, J., Griesinger, C. Heteronuclear multidimensional NMR experiments for the structure determination of proteins in solution employing pulsed field gradients. Prog Nucl Magn Reson Spectrosc. 34 (2), 93-158 (1999).
- Ferrage, F., Cowburn, D., Ghose, R. Accurate sampling of high-frequency motions in proteins by steady-state N-{H} nuclear Overhauser effect measurements in the presence of cross-correlated relaxation. J Am Chem Soc. 131 (17), 6048-6049 (2009).
- Delaglio, F., et al. NMRPipe: a multidimensional spectral processing system based on UNIX pipes. J Biomol NMR. 6 (3), 277-293 (1995).
- Skinner, S. P., et al. CcpNmr AnalysisAssign: a flexible platform for integrated NMR analysis. J Biomol NMR. 66 (2), 111-124 (2016).
- Lee, W., Tonelli, M., Markley, J. L. NMRFAM-SPARKY: enhanced software for biomolecular NMR spectroscopy. Bioinformatics. 31 (8), 1325-1327 (2015).
- Lee, W., Rahimi, M., Lee, Y., Chiu, A. POKY: a software suite for multidimensional NMR and 3D structure calculation of biomolecules. Bioinformatics. 37 (18), 3041-3042 (2021).
- Abyzov, A., et al. Identification of dynamic modes in an intrinsically disordered protein using temperature-dependent NMR relaxation. J Am Chem Soc. 138 (19), 6240-6251 (2016).
- Singh, A., Purslow, J. A., Venditti, V. 15N CPMG relaxation dispersion for the investigation of protein conformational dynamics on the micros-ms timescale. J Vis Exp. (170), e62395(2021).
- Gopalan, A. B., Hansen, D. F., Vallurupalli, P. CPMG experiments for protein minor conformer structure determination. Methods Mol Biol. 1688, 223-242 (2018).
- Tugarinov, V., Kay, L. E. Methyl groups as probes of structure and dynamics in NMR studies of high-molecular-weight proteins. Chembiochem. 6 (9), 1567-1577 (2005).
- Lipari, G., Szabo, A. Model-free approach to the interpretation of nuclear magnetic resonance relaxation in macromolecules. 1. Theory and range of validity. J Am Chem Soc. 104 (17), 4546-4559 (1982).
- Lipari, G., Szabo, A. Model-Free Approach to the interpretation of nuclear magnetic-resonance relaxation in macromolecules .2. Analysis of experimental results. J Am Chem Soc. 104 (17), 4559-4570 (1982).
- Clore, G. M., et al. Deviations from the simple two-parameter model-free approach to the interpretation of nitrogen-15 nuclear magnetic relaxation of proteins. J Am Chem Soc. 112 (12), 4989-4991 (1990).
- Lemaster, D. M. Larmor frequency selective model free analysis of protein NMR relaxation. J Biomol NMR. 6 (4), 366-374 (1995).
- Peng, J. W., Wagner, G. Mapping of spectral density functions using heteronuclear NMR relaxation measurements. J Magn Reson (1969). 98 (2), 308-332 (1992).
- Farrow, N. A., Zhang, O. W., FormanKay, J. D., Kay, L. E. Characterization of the backbone dynamics of folded and denatured states of an SH3 domain. Biochemistry. 36 (9), 2390-2402 (1997).
- Khan, S. N., et al. Distribution of pico- and nanosecond motions in disordered proteins from nuclear spin relaxation. Biophys J. 109 (5), 988-999 (2015).
- Smith, A. A., Ernst, M., Meier, B. H., Ferrage, F. Reducing bias in the analysis of solution-state NMR data with dynamics detectors. J Chem Phys. 151 (3), 034102(2019).
- Zumpfe, K., Smith, A. A. Model-free or not. Front Mol Biosci. 8, 727553(2021).
- Robustelli, P., Trbovic, N., Friesner, R. A., Palmer, A. G. Conformational dynamics of the partially disordered yeast transcription factor GCN4. J Chem Theory Comput. 9 (11), 5190-5200 (2013).
- Salvi, N., Abyzov, A., Blackledge, M. Multi-timescale dynamics in intrinsically disordered proteins from NMR relaxation and molecular simulation. J Phys Chem Lett. 7 (13), 2483-2489 (2016).
Access restricted. Please log in or start a trial to view this content.
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon