JoVE 비디오를 활용하시려면 도서관을 통한 기관 구독이 필요합니다. 전체 비디오를 보시려면 로그인하거나 무료 트라이얼을 시작하세요.
Method Article
단백질의 피코초에서 나노초까지의 구조 역학 조사를 위한 NMR 15N 이완 실험
요약
핵 자기 공명(NMR) 분광법은 잔류물 특이적 방식으로 구조적 단백질 역학을 특성화할 수 있습니다. 당사는 피코초에서 나노초의 시간 척도에 민감한 NMR 15NR1 및 R2 이완 및 {1H}-15N 이종 핵 오버하우저 효과(hetNOE) 실험을 기록하기 위한 실습 프로토콜을 제공합니다.
초록
핵 자기 공명(NMR) 분광법을 사용하면 용액 내 단백질과 생리학적 온도에서 단백질을 연구할 수 있습니다. 종종, 단백질 골격의 아미드 그룹 또는 곁사슬의 메틸 그룹은 단백질의 구조 역학을 보고하는 데 사용됩니다. 15N표지 및 완전 양성자 화된 샘플에서 구상 단백질의 단백질 골격에 대한 구조 역학 연구는 일반적으로 분자량이 최대 50kDa인 단백질에 대해 잘 작동합니다. TROSY(Transverse Relaxation Optimized Spectroscopy)와 함께 곁사슬 중수소화가 적용되면 이 한계는 구상 단백질의 경우 최대 200kDa, 곁사슬에 초점이 맞춰진 경우 최대 1MDa까지 확장될 수 있습니다. 내재적 장애 단백질(IDP) 또는 내재적 무질서 영역(IDR)을 가진 단백질을 조사할 때 이러한 중량 제한은 적용되지 않지만 그 이상일 수 있습니다. 그 이유는 높은 내부 유연성을 특징으로 하는 IDP 또는 IDR이 동적으로 분리되는 경우가 많기 때문입니다. 다양한 NMR 분석법은 피코초에서 최대 시간에 이르는 광범위한 시간 척도에 걸쳐 구조 단백질 역학에 대한 원자 분해능 통찰력을 제공합니다. 표준 15N 이완 측정은 단백질의 내부 유연성을 개관하고 빠른 피코에서 나노초까지의 시간 척도에서 경험하는 단백질 골격 역학을 특성화합니다. 이 기사에서는 NMR 15NR1, R2 및 hetNOE(Heteronuclear Overhauser effect) 실험을 설정하고 기록하기 위한 실습 프로토콜을 제시합니다. 우리는 예시적인 데이터를 보여주고 보다 정교한 분석을 하기 전에 이를 간단하게 정성적으로 해석하는 방법을 설명합니다.
서문
단백질의 기능은 3차원 구조뿐만 아니라 내부 유연성과 단백질이 채택할 다양한 형태 간의 구조적 전이를 포함하는 구조적 역학에 의해서도 결정됩니다. 핵 자기 공명(NMR) 분광법은 용액 1,2,3에서 단백질의 구조 역학을 조사할 수 있습니다. 양성자 검출 고체 상태 NMR의 최근 개발은 예를 들어 지질 이중층 멤브레인 4,5,6과 같은 덜 용해성 상태의 단백질 역학의 특성 분석을 허용합니다. 용액 NMR에서는 단백질 골격 및 단백질 곁사슬의 구조 역학을 연구할 수 있습니다. 구상 단백질의 경우, 단백질이 15N동위원소로 표지되면 단백질 골격의 구조 역학 연구를 최대 50kDa까지 달성할 수 있습니다. 측쇄 중수소 및 횡방향 이완 최적화 분광법(TROSY)을 사용하는 경우 이 한계는 최대 200kDa 7,8까지 확장될 수 있습니다. 측쇄 역학에 초점을 맞추면 접근 가능한 단백질 및 복합체의 범위를 최대 1 MDa 2,9까지 확장할 수 있습니다.
명명된 중량 제한은 종종 높은 내재적 역동성을 보이는 내재적 무질서 단백질(IDP)에는 적용되지 않습니다. 진핵생물 단백질체의 30% 이상은 IDP 또는 본질적으로 무질서한 영역(IDR)으로 구성됩니다10,11,12,13. 이들은 신호 전달(signal transduction) 및 전사(transcription)1와 같은 많은 세포 과정에서 중심적인 역할을 하며, 세포 내 위상 분리(phase separation)에 자주 관여한다 14,15,16,17. 국내실향민은 생리학적 조건 하에서 잘 정의된 3차원(3D) 본래 구조가 부족하며, 유입경로가 약하거나 울퉁불퉁한 에너지 환경을 가지고 있다17,18. IDP 또는 IDR의 백본에 분포된 낮은 소수성과 강한 정전기 반발력으로 인해 단단한 구조로 접히기 위한 추진력이 누락되었습니다19. IDP는 다른 결합 파트너(10,20,21)와 복잡할 때 접힌 형태를 자주 채택합니다. 또한, PTM(Post-translational modifications)은 IDP 또는 IDR의 폴딩 가능성을 확장한다22,23. 국내 실향민의 잘못 접힘은 신경 퇴행성 질환 15,24,25,26을 포함한 다양한 질병의 원인으로 확인되었습니다.
IDP 및 IDR은 높은 내부 유연성을 보여줍니다 21,27,28. 원자 위치와 이면각의 변화를 표시하는 구조적 앙상블은 실험 데이터 29,30,31,32에서 얻은 분자 역학 시뮬레이션 및 구속에서 파생되었습니다. 동결 상태에서의 역학 및 그로 인한 무질서로 인해 확산 전자 밀도는 Cryo-EM 또는 X선 결정학과 같은 구조 생물학의 최첨단 방법을 사용하여 구조적으로 특성화하기 어렵게 만듭니다. 또한 극저온 실험을 위한 결정화 조건 또는 시료 전처리 기술은 IDP가 경험하는 구조적 공간에 영향을 미칠 수 있습니다. 그러나 용액 NMR은 매우 동적인 단백질에 대해 잘 작동하므로 IDP 16,20,22,28,29,30,31,32,33,34,35,36,37,38을 조사하는 데 적합합니다.
위에서 소개한 바와 같이, 용액 NMR은 주로 스핀 이완 31,33,38,39,40,41,42를 기반으로 광범위한 시간 척도(그림 1)에 걸쳐 내부 단백질 역학을 연구하기 위한 다양한 기술을 제공합니다.
단백질 골격의 아미드 그룹에서 15N핵의 스핀 이완은 내부 단백질 역학 및 집단 운동(해당되는 경우 회전 확산 포함)으로 인한 1 H-15N 결합 각도 방향 변화에 의해 유도됩니다.27,43,44,45,46,47,48,49,50,51. 회전 상관 시간 τR (분자가 하나의 복사체를 회전시키는 데 필요한 시간, 전체 텀블링 상관 시간이라고도 함)보다 짧은 시간 척도에서 화학적 이동 이방성 (CSA) 및 쌍극자 결합 (D)은 활성화되며 단백질의 회전 확산에 의해 평균화되지 않습니다. 결합 각도의 변화, 결합의 방향 전환 및 회전 텀블링을 포함하는 단백질 골격의 내부 역학은 CSA 및 쌍극자 커플링 텐서의 확률적 변동을 유도하여 국소 자기장의 변동을 초래하고 궁극적으로 NMR 스핀 이완 47,48,52,53을 유발합니다. 이러한 변동은 전체 상관 함수로 설명할 수 있습니다. 전체 상관 함수의 푸리에 변환을 스펙트럼 밀도 함수라고 합니다. 반(半)고전적 레드필드 이완 이론에서, NMR 이완 속도 상수는 이들 스펙트럼 밀도 함수(54)의 선형 조합으로 설명될 수 있다.
1990년대 초에 개발된 백본 15N NMR 이완 실험은 15N R1, R 1ρ 및 {1H}-15N 핵 오버하우저 효과 실험으로 구성되며, 단백질 45,55,56,57의 회전 상관 시간 τR보다 빠른 피코초(ps) 나노초(ns) 시간 척도에 민감합니다. 회전 상관 시간 τR보다 느린 백본 동역학을 특성화하기 위해 소위 이완 분산 실험, R1ρ 및 마이크로초(μs) - 밀리초(ms) 동역학 44,46,58,59,60,61에 민감한 CPMG(Carr-Purcell-Meiboom-Gill) 실험이 사용됩니다. 마이크로초보다 느린 역학은 15N화학 교환 포화 전달(CEST) NMR62, 교환 분광법(EXSY, 밀리초에서 초로) 또는 실시간(RT) NMR(초에서 시간)63,64로 캡처할 수 있습니다. 잔류 쌍극자 커플링(RDC)뿐만 아니라 상자성 프로브의 PRE(상자성 이완 향상) 효과는 ps에서 ms 역학 65,66,67,68까지의 전체 범위를 평가하는 데 사용할 수 있습니다.
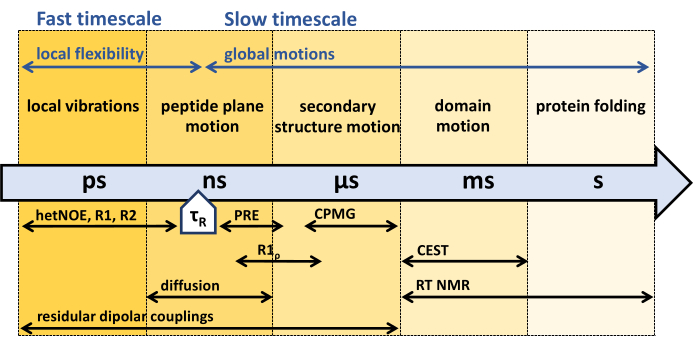
그림 1: 다양한 NMR 역학 실험의 단백질 백본 역학 및 민감한 시간 창의 시간 척도. NMR은 광범위한 시간 척도에 걸쳐 단백질 골격 역학을 특성화하는 다양한 방법을 제공합니다. 단백질 골격에 의해 경험되는 다른 운동은 그들의 각각의 시간 척도에 나타난다. 단백질의 회전 상관 시간 τR은 단백질이 전체 회전(1 복사점)에 필요한 시간입니다. 단백질의 회전 상관 시간 τR보다 빠른 움직임은 단백질의 내부 유연성과 관련될 수 있습니다. 다양한 NMR 실험과 각 시간 척도에 대한 민감도가 화살표 아래에 표시됩니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
아래 프로토콜은 감도 향상 HSQC(heteronuclear single quantum coherence) 검출 체계를 사용하여 Lakomek et al.69 및 Stief et al.70에 의한 NMR 이완 실험의 설정을 설명합니다. 실험적 구현을 진행하기 전에 NMR 스핀 이완 및 NMR 이완 실험에 대한 매우 간략한 개요가 제공됩니다. 크기 제한으로 인해 이 프로토콜을 이해할 수 있도록 하기 위해 이 개요는 단순해야 합니다(따라서 불완전해야 함).
T1 회 또는 R1 = 1 / T1 속도 상수를 특징으로하는 종방향 또는 스핀 격자 이완은 자화가 볼츠만 평형으로 돌아가는 것을 설명합니다. 평형 상태에서 자화는 실험실 프레임의 z축을 정의하는 외부 자기장의 축을 따라 정렬됩니다. 높은(1H) 및 작은(15N) Larmor 주파수(NMR 공진 주파수, 예: 14.1 Tesla 자석의 경우 1H에 대해 600MHz)의 스펙트럼 밀도와 이러한 Larmor 주파수의 선형 조합은 rad·s-1로 측정된 15NR1 속도 상수를 특징으로 하는 15N R1 완화에 기여합니다. 시간 척도에서의 움직임은 이 Larmor 주파수에 반비례합니다. 따라서 피코초에서 나노초까지의 시간 척도에서의 움직임은 완화 속도 상수 R1에 기여합니다. 전체 텀블링을 나타내고 회전 상관 시간을 정의할 수 있는 분자의 경우, R1 (T1) 곡선은 회전 상관 관계 τR 및 스핀의 Larmor 주파수 ω와 함께 ωτR = 1에 대한 최대(최소)를 보여줍니다. 여러 Larmor 주파수가 기여하는 경우 주파수가 가장 낮은 주파수가 지배적인 주파수입니다(예: 15NR1의 경우 ωN). 빠른 운동 영역(ωτR이 1보다 훨씬 작음)은 매우 빠르게 회전하는 작은 분자와 낮은 자기장과 낮은 점도에 적용됩니다. 슬로우 모션 영역(ωτR이 1보다 훨씬 큼)은 더 느리게 떨어지는 더 큰 분자와 높은 자기장 및 높은 점도에 유효합니다.
구형 접힌 단백질은 용액에서 전체적으로 텀블링(tumbling)을 보여주며, 회전 상관 시간(rotational correlation time)을 할당할 수 있습니다. 그러나 전체 텀블링의 개념은 본질적으로 무질서한 단백질에 대해 더 이상 유효하지 않으며 단일 회전 상관 시간을 할당하는 것과 종종 다릅니다. 여기서 잔류물 특이적 내부 상관 시간이 더욱 중요해집니다.
15NR1 이완 속도를 측정하는 설명 된 펄스 시퀀스 (그림 2)는 직교 검출 69,70,71을 위한 에코/안티 에코 검출을 사용한 감도 향상 HSQC 판독 실험을 기반으로 합니다. 다양한 강도 및 길이를 갖는 짧은 그래디언트는 일관성 선택 및 향상된 수분 억제70을 위해 사용됩니다. 그 시간 동안 15N세로 편광이 완화됩니다. 감쇠 시간이 길수록 이 의사 3D 스펙트럼의 관련 2D 평면의 강도가 감소합니다(지연 데이터 포인트는 3차원에 기록됨). 후술하는 루프 엘레멘트는 더 긴 이완 시간을 위해 점점 더 많은 횟수로 실행된다. 15N 화학적 이동 이방성(CSA)과 1H 및 15N 쌍극자 커플링(D) 사이의 상호 상관 이완이 이완 지연 동안에도 활성화되므로, 아미드 양성자에 선택적인 중앙 I-BURP-2 180° 펄스(72)는 상호 상관 이완에 의한 기여를 재초점을 맞추기 위해 필요하다(재초점이 맞지 않으면 편향되고 잘못된 15N R1 속도 상수로 이어질 수 있음).
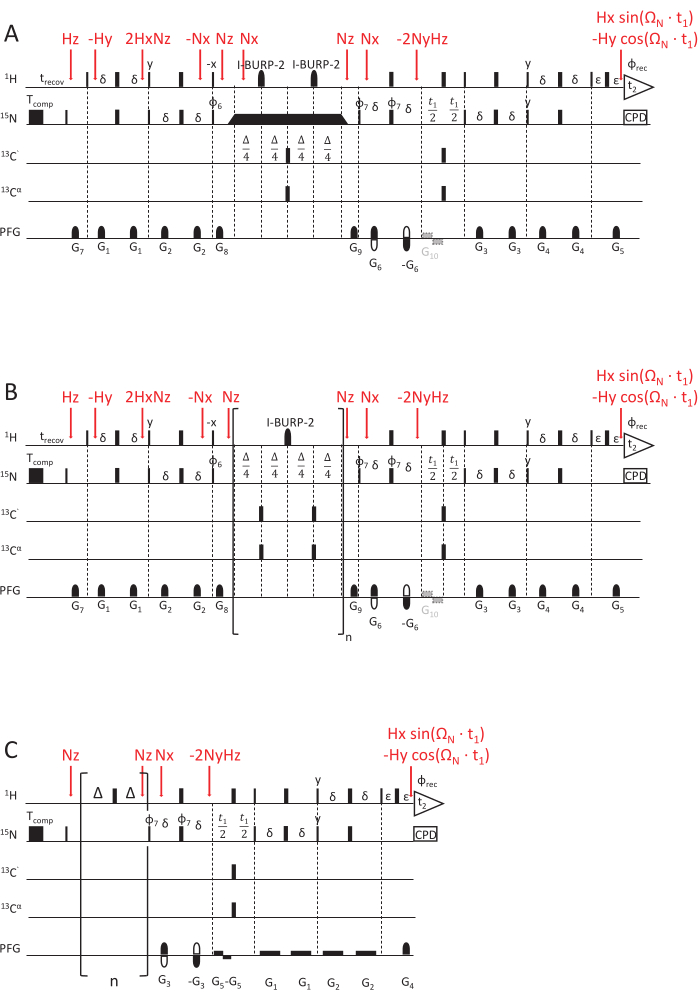
그림 2: NMR 완화율 상수를 결정하기 위한 NMR 펄스 시퀀스 체계. (A) 15NR1ρ, (B) 15NR1 및 (C) 감도 강화 HSQC 판독 체계69,70을 사용한 hetNOE 실험. 90°(x) 펄스는 달리 명시되지 않는 한 좁은 직사각형으로 시각화되고 180°(x) 펄스는 넓은 직사각형으로 시각화됩니다. 다음 위상 주기가 적용됩니다: φ6 = y, y, -y, -y; φ7 = y, -y, φrec = y, -y, -y, y. 직교 검출은 그래디언트 G5의 극성과 φ7의 위상 사이클을 반전시킴으로써 이루어집니다(에코/안티 에코 감지). (A) 15NR1ρ 실험: 검은색 직사각형은 스핀 잠금을 나타내며, 지속 시간은 다른 이완 지연을 획득하기 위해 달라집니다. spin-lock 전후의 삼각형은 유효 자기장 축 Beff를 따라 자화를 정렬하는 단열 형상 펄스를 나타냅니다. G10은 진화 단계에서 물 자화의 방사선 감쇠를 방지하기 위한 선택적 그래디언트입니다. (B) 15N R1 실험 : 브라켓 부분은 원하는 이완 지연과 일치시키기 위해 n 번 반복 된 시퀀스의 루프 요소를 보여줍니다. (C) hetNOE 펄스 방식은R1 및R1ρ 펄스 방식의 후반부, 즉t1 전개 시간 및 HSQC 검출 요소와 유사합니다. 그러나 15N자화는 INEPT 없이 직접 여기됩니다. 양성자 자화의 포화(1H와 15N사이의 교차 이완을 달성하기 위해)는 최소 5초 동안 적용된 180(1H) 펄스의 트레인에 의해 달성됩니다. 펄스 트레인 없이 동일한 길이(여기서는 5초)의 유휴 지연이 참조 실험에 적용됩니다. G5는 방사선 감쇠를 방지하기 위한 선택적 그래디언트이며, 그래디언트 G4의 극성 반전은 위상 φ7 = y, -y, -y, y와 결합하여 직교 검출을 달성합니다. 제품 작업자로 표시되는 자화 전달 단계는 빨간색으로 표시됩니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
이완 속도 상수 R2 는 스핀 사이의 위상 일관성 손실로 인한 횡 편파 (외부 자기장에 직교하는 xy 평면에서)의 이완을 설명하며, 이는 검출 가능한 자화(53,54)의 붕괴를 초래합니다. 고주파 및 소주파에서의 스펙트럼 밀도 함수는 R1과 유사하게 R2 에 기여합니다. 그러나 R2에 대한 가장 큰 기여는 제로 주파수에서의 스펙트럼 밀도에서 비롯됩니다. 이러한 이유로,R2 는 회전 상관 시간 τ R로 설명되는 회전 텀블링에 매우 민감하며, 이는 실온에서 작은 구상 단백질에 대해 몇 ns 정도입니다. 따라서 수백 ps에서 낮은 ns 영역에서 느린 백본 동작이 가장 많이 기여합니다. 15N 핵의 화학적 이동 텐서의 등방성 부분의 조절을 유발하는 백본의 교환 역학은 원칙적으로 R2 속도 상수 43,44,49,60,61 에 교환 기여도 R2ex를 추가합니다. 설명된 실험에서, R2ex 기여도는 스핀 잠금의 역 원형 주파수보다 느린 역학을 재초점을 맞추는 스핀 잠금에 의해 억제됩니다. 스핀 잠금은 유효 자기장 축 Beff(스핀 잠금 ω1 필드의 벡터 합과 15N캐리어 주파수의 화학적 이동 오프셋(아래 참조))를 따라 자화를 정렬하는 긴 연속파 무선 주파수 펄스입니다. B1,eff 축을 따라 정렬된 자화의 이완을 R1ρ 이완이라고 하며, 이는 R1 성분과 R2 성분을 갖습니다. 식 (1)을 사용하여,R2는R1ρ 및 R144,73으로부터 계산할 수 있다.
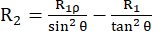 (1).
(1).
유효 자기장Beff의 축과 외부 자기장B0 사이의 각도는 이다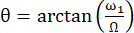 .ω1은 스핀-록의 RF 진폭이며, 해당 잔류물의 15N화학적 이동과 15N캐리어 주파수 44,73 사이의 화학적 이동 오프셋을 Ω한다.
.ω1은 스핀-록의 RF 진폭이며, 해당 잔류물의 15N화학적 이동과 15N캐리어 주파수 44,73 사이의 화학적 이동 오프셋을 Ω한다.
R1ρ 펄스 방식(그림 2A, 70)은 이완 지연을 제외하고는 15NR1 방식과 매우 유사합니다. 15NR1ρ 이완 속도를 측정하려면 스핀 잠금과 동일한 무선 주파수(RF) 진폭을 가진 단열 펄스에 의해 유효 필드 축 Beff 를 따라 자화가 정렬된 후 스핀 잠금이 활성화되어야 합니다. 스핀 잠금의 길이는 다른 이완 지연을 얻기 위해 다양해집니다.
정상 상태 {1H}-15N 핵 오버하우저 효과(1 H-15N NOE)는 다음에서 hetNOE라고 하며, 교차 이완 속도와 15N 종방향 이완 속도의 비율입니다. 이는 양성자 편광 45,53,54,74,75의 포화 시 양성자와의 교차 이완으로 인해 15N에서 정상 상태 편광의 감소로 이어집니다. 교차 이완은 1H 및 15N Larmor 주파수의 합과 차이의 스펙트럼 밀도 함수에 따라 달라집니다. 따라서 hetNOE는 빠른 피코초 동역학(< 100ps)과 ps-ns 동역학(R1 종속성으로 인해) 모두에 민감합니다. 시퀀스69(그림 2C)는 직교 검출을 위한 에코/반향 방지 그래디언트를 사용한 감도 강화 HSQC 판독을 기반으로 합니다. 양성자 자화 및 그에 따른 hetNOE의 포화를 위해, 평형 양성자 자화는 반전되고 이어서 15 N T1의 약 5 배 동안 180 ° 펄스에 의해 포화됩니다. 참조 실험의 경우 회복 지연은 포화 지연과 동일하지만 1H180°펄스 트레인은 없습니다. 참조 실험과 1H포화도가 있는 실험에 대해 D1 = 2초의 추가 지연이 추가됩니다. 두 실험 모두 연속적으로 기록되며 1H180° 펄스(포화) 적용 여부(참조)만 다릅니다. 1H포화도의 실험에서 기록된 스펙트럼 강도의 비율을 참조 실험의 강도(180° 프로톤 펄스 트레인 제외)로 나눈 값은 {1H}-15N NOE(hetNOE) 값을 제공합니다.
다음 프로토콜은 Lakomek et al.69 및 Stief et al.70에 의한 NMR 이완 실험의 설정을 설명합니다. 우리는 감도가 향상된 HSQC 검출 체계를 사용하여 NMR 펄스 시퀀스에 중점을 둡니다. 15NR1 및R1ρ 실험은 Stief et al.70에 의해 상세히 기술된 바와 같이 구현되고, hetNOE 실험은 Lakomek et al.69에 의해 설명된다.
프로토콜
1. NMR 시료 전처리
참고: 단백질의 동위원소 라벨링은 고차원 NMR 및 고급 NMR 실험을 위해 수행됩니다. 대장균 의 단백질 발현 및 단백질 정제가 리치 배지(예: Luria-Bertani[LB] 또는 2x 효모 추출물 트립톤 배지[2YT])를 사용하여 리터당 수 밀리그램의 수율로 확립된 경우, 동위원소 표지된 NMR 샘플을 준비하는 것은 일반적으로 비교적 간단합니다.
- 동위원소 라벨링의 경우 유전자 발현을 위해 M9 최소 배지를 사용하며, 여기서 15N염화암모늄이 유일한 질소 공급원이고 13C농축 포도당이 유일한 탄소원입니다.
- 단백질 정제의 경우 각 단백질에 대해 실험실에서 수립된 표준 프로토콜을 따르십시오.
참고: 결과 섹션에는 IDP, 단량체 SNARE 단백질 Synaptobrevin-2(1-96)에 대한 대표적인 결과가 나와 있습니다. Synaptobrevin-2의 NMR 동위원소 표지는70에 기재된 바와 같이 수행되었다. 매우 고정밀 측정의 경우 5% 미만의 낮은 D2O 농도를 사용하는 것이 좋습니다. 이는 중수소 동위원소 효과 때문이며, 이는 Hahn-Echo 15NT2 및 저주파 15N CPMG 이완 분산 실험에서 관찰 될 수 있지만 (여기서는 논의되지 않음) 15NR1ρ 실험 (주로 재초점이 맞춰지는 경우)에서는 최소한의 영향만 미칩니다 (76).
2. 분광계에서 NMR 이완 실험을 실행하기 위한 준비
참고: 설명된 NMR 완화 실험은 Bruker 분광계에만 해당됩니다. 극저온 및 실온 1H, 15N및 13C삼중 공명 프로브와 Bruker 소프트웨어 Topsin 3.6 이상에서 작동하는 Avance III 및 Avance Neo 콘솔에서 테스트되었습니다.
- 주의사항을 따르십시오.
- NMR 분광계에서 작업하기 전에 안전 브리핑을 하십시오.
- 사용된 분광계의 프로브 전력 사양을 확인하고 모든 펄스에 대해 적절한 펄스 길이와 펄스 전력을 선택합니다(예: Topspin "prosol" 테이블 항목과 일관성 비교).
- 펄스 시퀀스를 가져옵니다.
- 아래 표시된 웹 사이트 또는 확장된 BMRB(Biological Magnetic Resonance Bank) 저장소(bmrbig102)에서 NMR 분광계를 작동하는 컴퓨터의 /opt/topspin4.1.4/exp/stan/nmr/lists/pp/user 디렉토리로 NMR 펄스 프로그램을 다운로드합니다. topspin 버전을 조정하십시오(여기: topspin4.1.4를 분광계에 설치된 해당 버전으로 변경).
- 아래 표시된 웹 사이트 또는 확장된 BMRB(Biological Magnetic Resonance Bank) 저장소(bmrbig102)에서 NMR 분광계를 작동하는 컴퓨터의 /opt/topspin4.1.4/exp/stan/nmr/lists/pp/user 디렉토리로 NMR 모양의 펄스를 다운로드합니다. topspin 버전을 조정하십시오 (여기 : topspin 4.1.4를 분광계에 설치된 해당 버전으로 조정하십시오).
- 분광계와 샘플을 보정합니다.
- 원하는 온도를 설정합니다. 온도 보정을 위해 시설의 프로토콜을 적용하십시오. 예를 들어, 메탄올-d4 샘플을 사용하고 웹 애플리케이션 NMR 온도계 (https://chem.ch.huji.ac.il/nmr/software/thermometer.html)를 사용하여 관찰된 화학적 이동 차이를 해석합니다. 또한77 참조.
- 샘플을 삽입하고(잠금 장치에 추가 5% D2O 추가) 샘플이 온도를 평형화할 때까지 약 10분 동안 기다립니다. 중수소(2H) 신호를 잠급니다(명령: 잠금, H2O+D2O 선택).
- 예를 들어 atma 또는 atmm 루틴을 사용하여 1H, 15N및 13C채널을 일치시키고 조정합니다. 자동 매칭 및 튜닝 장치가 설치되지 않은 경우 wobb 명령을 사용하여 프로브를 수동으로 매칭하고 튜닝합니다.
- 예를 들어 topshim 루틴 tg를 사용하여 균질한 B0 필드에 대해 shimming을 수행합니다.
- 90°(1H) 펄스 길이를 보정합니다.
- zg 실험을 사용합니다. getprosol 을 사용하여 프로브 파라미터를 가져옵니다. P1에 대해 5μs의 짧은 지속 시간을 선택합니다. 필요한 경우 PLW1에 대한 프로브의 지정된 전력 수준을 확인하고 수정하십시오.
- zg로 실험을 시작합니다. efp를 사용하여 데이터를 처리합니다. 를 사용하여 위상을 조정합니다.ph 기준선이 균일하고 물 공명의 NMR 신호가 위상이 되도록 합니다.
- 360°(1H) 펄스를 추정하려면 공통 또는 대략적인 90°(1H) 펄스(예: 나열된 분광계 사양에서)를 취하고 여기에 4배를 곱합니다. 실험을 실행하고 처리합니다.
알림: 신호가 양수이면 선택한 지속 시간이 너무 길고 음수이면 너무 짧습니다. 360°(1H) 펄스는 제로 크로싱이 됩니다. - 그에 따라 펄스 길이를 조정하십시오. 이 작업을 반복적으로 수행하여 영점교차를 찾습니다. 얻은 값을 4로 나눕니다. 그것은 90°(1H) 펄스가 될 것입니다.
알림: 설명된 360° 방법의 대안은 명령줄에서 시작할 수 있고 자동화된 1H펄스 보정을 허용하는 펄스칼 루틴입니다.
- 캐리어 주파수 O1을 물 공명에 맞게 보정합니다.
- 명령줄에 edc 를 입력하여 zg 실험을 새 실험 번호에 복사합니다. rpar 을 입력하여 매개변수 세트를 엽니다(보충 그림 1A).
- 매개 변수 집합 ZGPR을 검색합니다. 매개변수 세트 ZGPR (보충 그림 1A)을 클릭하고, 읽기를 클릭하고, "getprosol" 실행을 선택하고 확인을 클릭합니다(보충 그림 1B).
- P1을 보정된 90°(1H) 펄스 길이로 변경합니다. D1 = 2s의 포화 길이와 50dB의 PLW9 펄스 전력을 선택합니다. 수신기 게인(rg)을 16으로 설정합니다. 또는 rga 를 입력하고 수신기 게인을 지정된 값으로 설정하여 수신기 게인 조정을 실행합니다(그러나 "수신기 게인 오버플로"는 피하십시오).
- 명령줄에 gs를 입력하여 반복적이고 연속적인 수집을 시작합니다. 획득 창으로 이동하여 O1을 변경하여 물 신호가 지배하는 FID 영역을 줄입니다(보충 그림 2).
- 마커 아래를 클릭하여 O1을 낮추고 마커 위를 클릭하여 늘립니다. 감도 버튼을 사용하여 단계 크기를 변경합니다. 현재 O1을 저장 하십시오. Close는 반복 수집을 중지합니다(보충 그림 2).
- 90°(15N) 펄스 길이를 보정합니다.
- zg 실험을 새 실험 번호( edc 유형)에 복사합니다. rpar 을 입력하여 파라미터 세트를 읽 HSQCFPF3GPPHWG 펄스 프로그램을 1 H-15N HSQC 실험으로 변경합니다. 매개 변수 P1, O1 및 PLW1 유지 옵션을 사용합니다(보충 그림 1).
- 명령줄에 getprosol 1H 1H) 펄스 길이 μs> <90° (1H) 펄스 강도 db> 를 입력하여 프로브 파라미터를 가져옵니다. 스캔 횟수(NS)를 8로 설정하고 더미 스캔(DS)을 2로 설정합니다.
참고 : < >는 여기에 설정할 값을 지정합니다. 예: getprosol 1H 8.5 -11.79. - 명령줄에 zg 를 입력하여 실험의 첫 번째 증분을 실행합니다. 첫 번째 증분을 rser 1<새 실험 번호> 를 사용하여 새 실험 번호로 복사합니다. efp 를 사용하여 데이터를 처리하고 를 사용하여 스펙트럼을 위상 지정합니다 .박사 다음 단계에 대해 실험의 첫 번째 증분(1D 실험)을 사용합니다.
참고: < >는 새 실험에 대해 선택한 번호(예: rser 1 101)를 지정합니다. - 명령줄에 ased를 입력하여 1D 실험의 펄스 시퀀스 파라미터를 엽니다. PULPROG 라인에서 E를 클릭하여(보충 그림 3A 참조) 펄스 프로그램 편집기를 엽니다.
- 다음 워크플로를 사용하여 펄스 시퀀스를 새 이름으로 저장합니다. 파일 (왼쪽 상단 모서리)을 클릭하고, 다른 이름으로 저장하고, 새 이름을 입력하고, 확인을 클릭합니다. 그런 다음 Set PULPROG to the dataset( 추가 그림 3B 참조)를 클릭하고 OK(확인)를 클릭합니다.
- t1 진화 기간(역전달 전) 후 90°(15N)에 2배(90°(15N) 펄스는 이제 180°(15N))를 곱하여 원래 1 H-15N HSQC 시퀀스(새 이름으로 저장됨, 2.6.4 참조)를 수정합니다. Ctrl S를 눌러 펄스 시퀀스를 저장합니다. 펄스 길이를 90°(15N)(P21)로 설정하여 첫 번째 증분의 NMR 신호가 (제로 크로싱에서) 사라지도록 합니다(보충 그림 3B).
- 1 H-15N HSQC 실험(2D 실험)을 열고 보정된 90°(15N) 펄스 길이(P21)를 입력합니다. 명령줄에 getprosol 1H<교정된 90°(1H) 펄스 길이(μs> <90°(1H) 펄스 강도(db)> 15N< 보정된 90°(15N) 펄스 길이(μs> <90°(15N) 펄스 강도(db>)를 입력합니다.
- 스펙트럼 폭과 획득 시간의 할당을 결정합니다.
- 1 H-15N HSQC 실험에 대한 획득 파라미터 창(명령줄에 eda 입력)에서 스펙트럼 폭(획득 파라미터 SW)과 직접 및 간접 치수의 치수 증분 수(획득 파라미터 TD)를 조정합니다.
- SW의 경우 직접(1H) 치수에서 16ppm, 간접(15N) 치수에서 30ppm을 선택합니다. TD, 직접(1H) 치수에서 2048, 간접(15N) 치수에서 512를 선택합니다.
- 1 H-15N HSQC 실험을 획득합니다.
- 관심 있는 모든 신호가 감지되고 스펙트럼에 앨리어싱 아티팩트가 없는지 확인합니다. 스펙트럼의 중심이 15N축을 따라 단백질 신호 분포의 중심과 맞지 않는 경우 O3P를 변경합니다.
- 스펙트럼 윈도우가 너무 작으면 SW 를 늘립니다. TD 를 높이면 더 높은 FID 분해능을 얻을 수 있습니다(최대 디커플링 기간은 프로브 사양을 확인하십시오).
알림: 이것은 NMR 이완 실험 설정의 일반적인 설정이자 시작점입니다. 전체 실험 세트의 시작과 끝에 1 H-15N HSQC 실험을 기록하는 것이 좋으며, 더 연장된 실험 시간의 경우 시료 무결성을 모니터링하기 위해 그 사이에도 기록하는 것이 좋습니다.
- 1 H-15N HSQC 실험에 대한 획득 파라미터 창(명령줄에 eda 입력)에서 스펙트럼 폭(획득 파라미터 SW)과 직접 및 간접 치수의 치수 증분 수(획득 파라미터 TD)를 조정합니다.
3. NMR 완화 실험 구현
참고: NMR 이완 펄스 시퀀스(그림 2)는 https://www.ipb.hhu.de/en/teams/team-lakomek/pulsesequences 또는 확장된 BMRB(Biological Magnetic Resonance Bank) 저장소(bmrbig102)에서 사용할 수 있습니다.
- 를 설정합니다. 15N R1ρ 실험.
- HSQC 실험( edc 입력)을 새 디렉토리에 복사합니다. ased 를 입력하고 펄스 시퀀스 파라미터에서 파라미터 라인 PULPROG의 세 점을 클릭하여 펄스 프로그램을 15N R1ρ 실험으로 변경합니다(ased, 보충 그림 3A 참조). 펄스 프로그램을 두 번 클릭하고 PULPROG 를 데이터 세트로 설정( 보충 그림 3B 참조)을 클릭한 다음 확인을 클릭합니다.
참고: 일부 오류 메시지가 나타날 수 있습니다. 이 메시지를 닫습니다. - 명령줄에 gpnam 을 입력하여 펄스 시퀀스(보충 그림 4)에 설명된 대로 누락된 모든 그래디언트 모양(gpnam1-gpnam9)을 삽입합니다.
- 명령줄에 vplist 를 입력합니다. 세 개의 점(보충 그림 5A), 파일 (왼쪽 상단 모서리) 및 새로 만들기 (보충 그림 5B)를 클릭합니다.
- 새 더미 vplist의 이름을 입력하고 OK를 누릅니다. 더미 vplist(5m, 65m, 25m, 45m, 15m, 55m, 35m, 10m, 각 vplist 항목에 대한 새 행)를 만듭니다. Ctrl S 를 눌러 목록을 저장하고 vplist의 창을 닫습니다(보충 그림 5C).
- 새로 생성된 vplist의 이름을 두 번 클릭합니다(보충 그림 5B).
참고: 이는 펄스 시퀀스에서 컴파일 오류를 방지하기 위해 필요합니다. vplist 의 적절한 항목은 아래에서 더 결정됩니다 (포인트 3.1.29). - ased를 입력하여 펄스 시퀀스 매개변수를 엽니다(보충 그림 6). 펄스 시퀀스에 제공된 대로 누락된 모든 기울기 강도와 기울기 길이를 입력합니다(보충 그림 6D).
- 루프 카운터 L3(간접 차원의 복소 점 수)에 대해 획득 매개변수 창(eda)에 15NTD 항목의 1/2을 입력합니다. 루프 카운터 L6에 대해 이완 곡선을 맞추는 데 사용되는 기록된 이완 데이터 포인트의 수를 입력합니다.
참고: 이 숫자 L6 은 vplist 항목의 수에 해당하며, 여기서는 8(보충 그림 6A)에 해당합니다. - 위에 지정된 웹 사이트에서 다운로드한 단열 TanhTan_half.nl 펄스를 15NSP8 펄스의 모양으로 설정합니다. 웹 사이트에서 단열 TanhTan_2nd.nl 펄스를 15NSP9 펄스의 모양으로 설정합니다(보충 그림 6C).
- P8 항목을 단열 TanhTan 펄스의 길이로 설정하십시오 - 펄스의 단열성을 보장하기에 충분히 길다. 여기서 P8 = 3000 μs가 선택됩니다(보충 그림 6C).
- 15N디커플링(CPDPRG 3)을 가프 디커플링 방식으로 설정합니다. PCPD3 200 μs의 디커플링 길이에 사용합니다(보충 그림 6C).
- 스캔 간 복구 지연을 최소 D1 = 2초 이상으로 설정합니다.
참고: 적절한 복구 지연을 선택하려면 토론 섹션을 참조하십시오. - 최종 실험을 위해 더미 스캔(DS)을 64 이상으로 설정합니다(설정 중에는 시간상의 이유로 DS = 4로 충분함). 시작점으로 4의 스캔 수(NS)를 사용합니다. SNR(Signal-to-Noise)이 너무 낮으면 4의 배수를 사용합니다(보충 그림 6A).
- O1을 2.5단계의 보정된 캐리어 주파수로 설정합니다. O2P를 176ppm으로 설정하고 1 H-15N HSQC 실험에서 O3P를 복사합니다(보충 그림 6A-C).
- 펄스 길이 P7을 90단계에서 보정된 15°(2.6N) 펄스 길이로 설정합니다. 또한 90단계에서 15°(2.6N) 펄스의 펄스 전력 레벨을 PLW3 및 PLW7로 복사합니다(보충 그림 6C).
- 펄스 길이 P1 및 P19를 90단계에서 계산된 1°(2.4H) 펄스 길이로 설정합니다. (보충 그림 6B).
- 간접 치수 TD = l3 * 2 * l6 (eda 또는 명령줄에 td 입력)의 증분 수를 설정합니다.
참고: 여기에 설명된 모든 펄스 시퀀스 매개변수는 펄스 시퀀스에 추가로 주석이 추가됩니다. - 모양 펄스 SP5를 I-BURP2 모양(Iburp2.1000)으로 설정합니다. 펄스 길이 P15를 2000μs로 설정합니다. Topspin 펄스 시퀀스 매개변수 창(ased)에서 I-BURP2 모양 펄스 옆에 있는 E를 클릭하여 모양 도구 표시를 엽니다(보충 그림 6B).
- Start NMR Simulation 버튼을 클릭하여 올바른 길이로 형상 펄스를 시뮬레이션합니다. 시뮬레이션 창에서 성형된 펄스 길이[μs](= 2000μs)와 회전 각도[°](= 180°)를 확인합니다. NMR-SIM 시작을 클릭합니다(보충 그림 7A).
- 여기 범위를 확인하고 단백질의 1H스펙트럼 분산을 커버하는 적절한 I-BURP2 펄스 길이를 선택하되 동시에 물의 여기(excitation)를 피하십시오. P15를 시뮬레이션된 최상의 IBURP-2 펄스가 있는 시뮬레이션 창의 성형 펄스 길이로 설정합니다(보충 그림 7B).
알림: I-BURP2가 전체 아미드 영역을 자극(> 95% 효과)하는 경우 적절한 길이가 선택됩니다. 다른 길이의 I-BURP2 펄스를 시뮬레이션하려면 여기 프로파일 창을 닫고 시뮬레이션 창에서 형성된 펄스 길이를 편집하십시오. NMR-SIM 시작을 클릭합니다. 길이가 더 짧은 펄스는 Hz 단위의 여기 범위가 더 넓습니다. - I-BURP2 펄스의 반송 주파수를 변경하고 여기 범위를 왼쪽이나 오른쪽으로 이동하여 I-BURP2 펄스가 물 자화를 만지거나 방해하지 않도록 하려면 SPOFFS5 설정합니다(보충 그림 6B).
- 형상 펄스의 적절한 전력 수준을 결정하려면 Bruker 형상 도구를 열고 NMR 시뮬레이션 시작 버튼을 클릭한 다음 I-BURP2 펄스 길이를 형상 펄스 길이[μs]로 설정합니다. 해당 90° 사각 펄스[μs]에서 시뮬레이션 창에서 볼 수 있는 부드러운 직사각형 90°(1H) 펄스의 해당 길이를 기억하십시오(보충 그림 7A).
- calcpowlev를 입력합니다. 마이크로초 단위의 하드 90°(1H) 펄스(P1)를 기준 길이(보충 그림 8B)로 사용하고 I-BURP2 펄스(마이크로초)에 해당하는 90°(1H) 소프트 직사각형 펄스를 새 길이(보충 그림 8A)로 사용합니다. 하드 90°(1H) 펄스와 부드러운 직사각형 펄스 사이의 전력 차이 Δ(dB)를 기억하십시오(보충 그림 8C). 하드 90°(1H) 펄스의 펄스 강도(dB)를 SPW5에 복사하고 기억된 차이 Δ(dB)를 추가합니다(보충 그림 6B).
- 스핀 잠금의 전력 수준을 결정하려면 스핀 잠금의 해당 90°(15N) 펄스 길이를 계산하십시오. 공식 사용: 90°(15N) 펄스 길이(s = 0.25/(스핀 잠금 전력(Hz)).
- calcpowlev를 사용하여 스핀 잠금 전력과 90°(15N) 하드 펄스(P7) 사이의 전력 차이 Δ(dB)를 계산합니다. 신규: 스핀 록의 90°(15N) 펄스 길이(μs)(보충 그림 8A). 참조: 하드 90°(15N) 펄스 길이(μs)(보충 그림 8B). dB 단위의 90°(15N) 하드 펄스의 전력 레벨 PLW7을 스핀 잠금 전력 레벨 PLW8(보충 그림 6C)에 복사하고 계산된 전력 차이 Δ(dB)를 추가합니다(보충 그림 8C).
- 스핀 잠금 PLW8의 전원 수준을 SPW8 및 SPW9의 전원 수준으로 복사합니다. (보충 그림 6C).
- 15N디커플링의 전력 수준을 확인하려면 calcpowlev를 사용하여 90°(15N) 디커플링 펄스(PCPD3)와 90°(15N) 하드 펄스(P7)의 전력 차이 Δ를 계산합니다. 신규: μs 단위의 PCPD3 (200) (보충 그림 8A). 참조: 하드 90°(15N) 펄스 길이(μs)(보충 그림 8B). dB 단위의 90°(15N) 하드 펄스의 전력 수준 PLW7을 디커플링 수준 PLW31(보충 그림 6C)에 복사하고 dB에 Δ를 추가합니다(보충 그림 8C).
- 온도 보정을 사용하려면 #define TEMP_COMPENSATION 포함합니다(펄스 시퀀스에서 세미콜론을 삭제). P18을 15NR1ρ 실험에 사용된 스핀 잠금의 최대 기간으로 설정합니다.
알림: 실험의 첫 번째 시도에서는 온도 보상을 끄는 것이 좋습니다. - 15N 13C라벨링 샘플의 경우 #을 포함하여 펄스 프로그램에서 LABEL_CN 정의합니다(펄스 시퀀스에서 세미콜론을 삭제하여). P4를78의 공식으로 계산된 펄스 길이로 설정합니다. 예를 들어, 600MHz에서 23.70μs, 1200MHz에서 11.85μs에 해당합니다. calcpowlev를 입력하여 P4와 (13C) 하드 펄스 사이의 전력 차이(dB)를 계산합니다. (13C) 하드 펄스 전력(dB)을 PLW4에 복사하고 Δ(dB)를 추가합니다. PLW4를 PLW2에 복사합니다(보충 그림 6B).
- 적절한 표본추출을 위해 완화 지연을 결정합니다.
- 처음 8개의 FID(L6 = 8의 경우)를 실행하고 이를 처리합니다(rser < 새 실험 번호>). 지연이 가장 긴 실험의 피크 강도가 지연이 가장 짧은 실험(참조 역할을 함)에 비해 최소 1/e(약 37%)로 감소하지만 25% 이상으로 감소하는 vplist 항목을 선택합니다.
- 지연이 가장 긴 FID와 지연이 가장 짧은 FID 사이의 강도 감소를 균일하게 샘플링할 수 있도록 vplist의 나머지 항목을 조정합니다. 프로브의 참고 사항과 전원 사양을 읽으십시오.
알림: 천천히 이완되는 잔류물이 첫 번째 FID를 지배합니다. 첫 번째 FID와 두 번째 FID 사이의 강도 감소를 균일하게 샘플링할 수 있도록 vplist의 나머지 항목을 조정합니다. 프로브의 최대 허용 스핀 잠금 전력 및 길이를 초과하지 마십시오(프로브 설명서 및 프로브 사양 참조). 온도 보상의 경우 P18(최대 스핀 잠금 기간)을 확인하고 P18의 전체 스핀 잠금 기간이 적용되며 이완 지연과 온도 보상으로 나뉩니다. 시간(이완 지연) + 시간(온도 보상) = const = P18을 유지합니다. 2kHz 스핀 잠금의 경우 2초의 복구 지연(D1)을 사용하여 65ms 이상의 스핀 잠금 기간을 초과하지 않는 것이 좋습니다. 이 경우 허용되는 최대 스핀 잠금 기간을 위해 최대 스핀 잠금 기간과 가장 긴 완화 지연 간의 절충안을 사용해야 합니다. D1 복구 지연이 더 긴 경우 더 긴 스핀 잠금 전원이 적용될 수 있습니다. 중요한 것은 시간 경과에 따른 프로브의 전체 전력 증착입니다. 더 긴 스핀 잠금 기간이 필요한 경우 스핀 잠금 길이를 작은 단계로만 늘리고 잠금 신호를 확인합니다. 일반적으로 잠금 디스플레이의 상단 1/3에서 잠금 수준을 조정하십시오. 펄스 시퀀스를 실행하는 동안 잠금 레벨이 두 칸 이상 감소하면 프로브에 증착된 스핀 잠금 전력이 너무 큽니다.
- 명령줄에 rga를 입력하여 수신기 게인을 확인합니다. 좋은 신호 대 잡음비를 위해 128에서 256 사이의 수신기 게인(rg) 값을 목표로 하며, 이는 우수한 수분 억제를 입증합니다.
알림: 128에서 256 사이의 수신기 이득에 도달할 수 없음은 물 신호가 너무 강함을 나타냅니다. 이 경우 그래디언트 파워와 지속 시간은 수신기 게인에 도달할 때까지 부드럽게 조정됩니다. 토론 섹션도 참조하십시오. - 명령줄에 zg를 입력하여 15N R1ρ 실험의 테스트 실행을 시작합니다. 모든 지연에 대해 물 신호가 잘 억제되어 있는지 확인합니다(vplist에서 8 지연의 경우 1 - 8). 또한 직교 감지 체계(Echo/Anti-Echo)의 두 번째 증분인 증분 9를 선택합니다.
- HSQC 실험( edc 입력)을 새 디렉토리에 복사합니다. ased 를 입력하고 펄스 시퀀스 파라미터에서 파라미터 라인 PULPROG의 세 점을 클릭하여 펄스 프로그램을 15N R1ρ 실험으로 변경합니다(ased, 보충 그림 3A 참조). 펄스 프로그램을 두 번 클릭하고 PULPROG 를 데이터 세트로 설정( 보충 그림 3B 참조)을 클릭한 다음 확인을 클릭합니다.
- 15N R1 실험을 설정합니다.
- 15NR1ρ 실험을 복사하고 펄스 프로그램을 15NR1 실험으로 변경합니다.
- vclist를 입력하고, vclist를 생성하고,70에 설명된 더미 vclist를 입력합니다: 0, 24, 6, 20, 4, 16, 8, 12; 각 vclist 항목에 대한 새 행).
- ased를 엽니다. 그래디언트 길이와 강도를 확인합니다. GPZ9를 14%로 변경합니다.
- 펄스 길이, 펄스 모양 및 펄스 강도를 확인합니다(올바른 값은 15NR1ρ 실험에서 복사해야 함).
- 15N13C 라벨이 부착된 샘플의 경우 펄스 프로그램에서 #define LABEL_CN를 사용하십시오.
- 온도 보상의 사용을 위해, 포함하십시오 #define TEMP_COMPENSATION.
- 처음 8개의 FID(L6 = 8의 경우)를 실행하고 이를 처리합니다(rser < 새 실험 번호>). 지연이 가장 긴 실험의 피크 강도가 지연이 가장 짧은 실험에 비해 최소 1/e(약 37%)로 감소하지만 25% 이상으로 감소하는 vclist의 항목을 선택합니다. 지연이 가장 긴 FID와 지연이 가장 짧은 FID 사이의 강도 감소를 균일하게 샘플링할 수 있도록 vplist의 나머지 항목을 조정합니다.
참고: vclist 항목에는 짝수만 허용됩니다.
- {1H}-15N NOE 실험을 설정합니다.
- 15NR1 이완 실험을 복사하고 펄스 시퀀스를 {1H}-15N NOE 펄스 스키마로 변경합니다.
- 루프 카운터 L8로 포화 기간의 길이를 설정합니다. 180°(1H) 펄스 사이의 간격은 22ms79 (2* 1/JNH)입니다. 가장 정확한 결과를 얻으려면 단백질의 15N T1 (15N)의 약 5 배의 포화 기간을 사용하십시오. 여기: 5초. 이는 L8 = 230에 해당합니다.
참고: 토론 섹션에서는 포화 기간/복구 지연 선택에 대한 추가 조언을 제공합니다. - 펄스 구성표에 설명된 대로 기울기의 길이와 강도를 변경합니다. 사인.20 그라디언트 모양을 사용합니다.
- 간접 차원의 증분 수를 TD = l3 * 2 * 2로 설정합니다.
- 15N13C 라벨링된 샘플의 경우 #define LABEL_CN 및 -DLABEL_CN를 ZGOPTNS로 사용합니다.
- 명령줄에 zg를 입력하여 펄스 계산법을 실행합니다.
4. 기록된 NMR 실험의 처리 및 분석
참고: 스펙트럼은 Bruker 시스템을 사용하여 기록되었습니다. 처리는 Unix 또는 Linux 운영 체제를 사용하여 수행됩니다. 스펙트럼 처리 및 데이터 분석은 NMRPipe80 및 python3을 사용하여 수행되었습니다. NMRPipe 소프트웨어는 https://www.ibbr.umd.edu/nmrpipe/index.html 에서 다운로드할 수 있습니다. NMRPipe 기반 처리 스크립트는 웹 사이트 https://www.ipb.hhu.de/en/teams/team-lakomek/pulsesequences 또는 확장된 BMRB(Biological Magnetic Resonance Bank) 저장소(bmrbig102)에서 다운로드할 수 있습니다. NMRPipe를 사용하는 것이 좋습니다. NMRPipe가 이용 가능하지 않거나 바람직하지 않은 경우, CCPN81 또는 SPARKY(SPARKY 3, 또는 그 후속 제품인 NMRFAM-SPARKY82 또는 POKY83 )와 같은 대안을 사용할 수 있다.
- 처리 및 분석 15N R1 실험.
참고: 다음 명령은 Unix 터미널에서 실행됩니다.- 다운로드한 디렉토리 R1_processing의 파일을 실험적 R1 데이터가 포함된 디렉토리(작업 디렉토리)에 복사합니다.
- 터미널 명령으로 파일을 실행 가능하게 만듭니다: > chmod u+x *.com
- bruker> 실행하여 NMRPipe 변환 유틸리티를 열고 fid.com 파일을 생성합니다. 매개 변수 읽기를 클릭합니다. 3개의 차원이 계산되는 경우 Dimension Count(차원 수)를 2로 변경하고 Read Parameters(매개 변수 읽기)를 다시 클릭합니다. 그런 다음 Clear Script(스크립트 지우기), Update Script(스크립트 업데이트) 및 Save Script(스크립트 저장)를 클릭합니다(보충 그림 9).
알림: 캐리어 주파수가 올바르지 않으면 Read Parameters 를 두 번째로 클릭한 후 Center Position PPM을 변경하십시오. - > ./fid.com 실행
- ms 항목이 있는 vplist의 vclist를 변환합니다: delay in ms = 40 ms * entry of vclist. cp vclist vplist > 사용하고 vplist를 편집합니다.
- > ./split.com 를 사용하여 의사 3D 데이터를 8개의 2D 파일로 분할합니다.
- nmrproc.com 에서 tauValues (vplist 순서의 vplist 값) 및 idxExpmt 값(시간에 대한 tauValues의 순서)을 편집합니다(보충 그림 10).
- > ./nmrproc.com 를 사용하여 8개의 모든 2D 스펙트럼의 원시 데이터를 처리할 수 있습니다.
- > nmrDraw를 사용하여 스펙트럼을 그립니다.
- 다음 단계에서는 기준 스펙트럼(vplist의 첫 번째 항목)을 사용합니다. z 차원을 1(왼쪽 위 모서리)로 변경하여 해당 스펙트럼으로 전환합니다. Factor 버튼 옆에 있는 +와 -를 사용하여 윤곽선 레벨을 조정합니다. Draw 버튼으로 스펙트럼을 다시 그립니다.
- 노이즈에서 나오는 몇 개의 신호만으로 관심 있는 모든 신호를 인식할 수 있는 윤곽 수준을 사용합니다(윤곽 수준은 소음 수준의 가장자리에 있음)(보충 그림 11).
- 스펙트럼 위상을 수정하고, 수직 1D 신호 투영의 경우 v 를 입력하고 수평 1D 신호 투영의 경우 h 를 입력합니다. P0 및 P1로 위상을 조정하고(보충 그림 11), 위상 차이를 기억하고, nmrproc.com 파일에서 위상을 수정합니다(보충 그림 10).
참고: 스펙트럼이 15N차원에서 대칭 이동된 경우 | nmrPipe -fn FT -neg \ (보충 그림 10) 줄 에서 -neg를 제거합니다. - nmrDraw를 닫고 > nmrproc.com 로 다시 처리하십시오. 피크 선택의 경우(스펙트럼이 올바르게 위상화되고 참조 스펙트럼이 모든 피크가 있는 윤곽 수준으로 표시된 후) K (shift + k)를 입력하고 Detect를 누른 다음 Save를 클릭합니다. (보충 그림 11).
- 피크 목록을 작업 디렉토리에 복사하고 -assName 을 ass.com 피크 목록의 이름으로 변경합니다(보충 그림 12).
참고: 올바른 형식의 피크리스트의 예는 다음에서 확인할 수 https://www.ipb.hhu.de/en/teams/team-lakomek/pulsesequences
확장된 BMRB(Biological Magnetic Resonance Bank) 저장소(bmrbig102)에서 사용할 수 있습니다. - > ./ass.com 를 실행하여 스펙트럼을 할당합니다. 스크립트가 다양한 피크에 대해 제안하는 할당을 확인합니다. 마우스 왼쪽 버튼을 사용하여 할당하고 마우스 오른쪽 버튼을 사용하여 피크를 할당 해제합니다. Next 버튼을 사용하여 다음 아미노산으로 이동하고 Prev를 사용하여 이전 아미노산으로 이동합니다(보충 그림 13).
참고: 윤곽선 수준은 오른쪽 상단 모서리에 있는 + 및 -로 변경할 수 있습니다. Draw 버튼을 사용하여 새 윤곽 레벨로 스펙트럼을 다시 그립니다(보충 그림 13). - 과제를 완료한 후 Save(저장 )를 클릭한 다음 Save(저장 )를 다시 클릭합니다. 그런 다음 Quit(종료), Cancel(취소) 및 Exit(종료 )를 클릭합니다(보충 그림 13).
- python3 NoneDeleter.py> 실행하여 할당된 피크에 대한 정보만 포함하는 relax.tab을 만듭니다.
- ./relax.com> 실행하여 autofit.com 스크립트를 생성합니다.
- ./autoFit.com> 실행하여 푸리에 보간을 수행하고 각 아미노산에 대해 서로 다른 지연 시간의 의사 비율을 생성합니다(axt.tab 및 nlin.tab이 생성됨).
- ./model.com> 실행하여 nlin.tab의 데이터를 기반으로 modelExp.com 스크립트를 생성합니다.
- > ./modelExp.com 를 실행하여 비선형 최소제곱 최적화 모델을 사용하여 각 잔차의 진화 곡선을 맞춥니다.
- > bash summary.tcl -in relax.tab > t1.tab을 실행합니다. 잠재적 오류 메시지 무시: GDB 알림: 테이블 파일에서 21개의 누락된 값이 발생할 수 있습니다.
- t1.tab을 새로 만든 디렉토리 R1R2에 복사 합니다.
- 15N R1ρ 실험을 처리하고 분석합니다.
- 다운로드한 디렉토리 R1rho_processing의 파일을 실험적 R1ρ 데이터가 포함된 디렉토리(작업 디렉토리)에 복사합니다.
- chmod u+x *> 실행합니다.com
- bruker> 실행하여 NMRPipe Conversion Utility를 열고 fid.com 파일을 생성합니다. 15N R1 스펙트럼 처리도 참조하십시오.
- > ./fid.com 실행
- > ./split.com 를 실행하여 의사 3D 데이터를 8개의 2D 파일로 분할합니다.
- > ./nmrproc.com 를 실행하여(tauValues [vplist values] 및 idxExpmt 미리 편집) 8개의 2D 스펙트럼 모두의 원시 데이터를 처리합니다(보충 그림 10).
- nmrDraw> 실행하여 스펙트럼을 그리고 피크를 선택합니다. 다음 단계에서는 기준 스펙트럼(vplist의 첫 번째 항목)을 사용합니다. 15NR1 실험과 동일한 절차를 따르십시오.
- 피크 목록을 작업 디렉토리에 복사하고 -assName을 피크 목록의 이름으로 ass.com 변경합니다(보충 그림 12).
- ./ass.com> 실행하고 15NR1 실험과 동일한 절차를 따릅니다.
- python3 NoneDeleter.py> 실행하여 할당된 피크에 대한 정보만 포함하는 relax.tab을 만듭니다.
- > ./relax.com 실행
- > ./autoFit.com 실행
- ./model.com> 실행
- ./modelExp.com> 실행
- bash summary.tcl -in relax.tab > t1rho> 실행합니다. 탭을 클릭합니다. 잠재적 오류 메시지 무시: GDB 알림: 테이블 파일에서 21개의 누락된 값이 발생할 수 있습니다.
- t1rho.tab 파일에 다음 헤더를 복사합니다(값은 fid.com 파일에서 얻을 수 있음)(보충 그림 14).
주목 T600rho의 경우 1MHz
REMARK -yOBS = 60.818 (여기에 fid.com 에 있는 값을 넣음)
REMARK -yCAR = 119.06 (여기에 fid.com 있는 값을 입력) - > ./extract_PPM.com를 실행하여 반송파 주파수에 상대적인 피크 15N주파수의 오프셋을 포함하는 fusionOffset.tab이라는 테이블을 생성합니다(R1 및 R1ρ 데이터에서 R2 데이터를 추출하기 위한 각도 θ를 계산하는 데 필요함).
- 완화 데이터를 요약합니다.
- t1rho.tab 및 fusionOffset.tab을 R1R2 디렉토리에 복사합니다.
- R1R2 디렉토리에서 crtR1p.tcl 스크립트를 복사하고, 분광계 주파수와 스핀 잠금 전력을 조정하고, bash crtR1p.tcl | sort -n > R1R2.tab을 실행합니다. (보충 그림 15)을 클릭합니다. R1R2.tab에는 피팅된 R1ρ 속도 상수, 세타 각도, R2 속도 상수 및 R1 속도 상수가 포함됩니다.
- hetNOE 실험을 처리하고 분석합니다.
- 다운로드한 디렉토리의 파일을 NOE_processing 실험용 NOE 데이터 디렉토리(작업 디렉토리)에 복사합니다.
- chmod u+x *> 실행합니다.com
- bruker> 실행하여 NMRPipe 변환 유틸리티를 열고 fid.com 파일을 생성합니다. 매개 변수 읽기를 클릭합니다. 3개의 차원이 계산된 경우 Dimension Count(차원 수)를 2로 변경하고 Read Parameters(매개변수 읽기)를 다시 누릅니다. 그런 다음 스크립트 지우기, 스크립트 업데이트 및 스크립트 저장을 누릅니다.
- > ./fid.com 실행
- > ./split.com 실행합니다(두 개의 항목(0, 1)이 있는 vplist를 먼저 만들어야 함).
- > ./ft2.com 실행
- nmrDraw> 실행하여 스펙트럼을 그리고 기준 스펙트럼에서 피크를 선택합니다(z 차원 = 1). 노이즈를 추출하고 기억합니다(T = shift + t 누름). 위상 보정 및 피크 피킹은 15N R1 실험과 동일한 절차를 따릅니다.
- 피크 목록을 작업 디렉토리에 복사하고 -assName in ass.com peak list의 이름으로 변경합니다(보충 그림 12)
- > ./ass.com 를 실행하여 스펙트럼을 할당합니다. 15NR1 실험에 대해 자세히 설명된 것과 동일한 절차를 따릅니다.
- python3 NoneDeleter.py> 실행하여 relax.tab을 만듭니다.
- > ./script.com 실행
- > ./autoFit.com 실행
- error_hetnoe.tcl에서 노이즈를 변경합니다.
- bash error_hetnoe.tcl |sort -n > hetNOE.tab> 실행합니다.
결과
다음은 종종 VAMP2(소포 관련 단백질 2)라고 불리는 소포성 SNARE 단백질 Synaptobrevin-2(1-96)에 기록된 몇 가지 예시적인 NMR 이완 데이터를 보여줍니다. NMR 데이터를 기록하기 위해 150mM NaCl, 0.1mM TCEP 및 1mM EDTA를 포함하는 50mM MES(pH 6.0) 완충액에 171μM 15NSynaptobrevin-2(1-96) 샘플(다음에서는 Syb-2라고 함)을 사용했습니다. 모든 실험 데이터는 3mm NMR 샘플 튜브에 채워진 250μL 부피...
토론
이 프로토콜은 Lakomek et al.69 및 Stief et al.70에 의한 NMR 15N 이완 실험의 설정을 설명했습니다. 우리는 감도가 향상된 HSQC 검출 체계를 사용하여 NMR 펄스 시퀀스에 중점을 두었습니다. 15NR1 및R1ρ 실험은 Stief et al.70에 의해 상세히 기술된 바와 같이 구현되고, hetNOE 실험은 Lakomek et
공개
저자들은 이 논문에 보고된 연구에 영향을 미칠 수 있는 경쟁적인 재정적 이해관계나 개인적 관계를 알지 못한다고 선언한다.
감사의 말
유익한 토론에 참여해 주신 Melinda Jaspert와 Kevin Bochinsky에게 감사드립니다. N.L.은 하이젠베르크 프로그램(DFG 보조금 번호 433700474)을 통해 자금을 지원해 준 독일 과학 재단에 감사를 표합니다. 이 연구는 헬름홀츠 협회의 이니셔티브 및 네트워킹 기금(Helmholtz Association's Initiative and Networking Fund)의 보조금인 "COVID-19 발병기전의 바이러스 및 면역학적 결정 요인 - 미래 팬데믹에 대비하기 위한 교훈(KA1-Co-02 "COVIPA") 프로젝트의 지원을 받고 있습니다. 우리는 Forschungszentrum Jülich와 Heinrich Heine University Düsseldorf (HHU)가 공동으로 운영하는 Jülich-Düsseldorf Biomolecular NMR Center에 대한 관대한 접근을 인정합니다.
자료
| Name | Company | Catalog Number | Comments |
| Bruker 600 MHz AVANCE III HD spectrometer | Bruker | https://www.bruker.com/en/products-and-solutions/mr/nmr/avance-nmr-spectrometer.html | NMR experiments conducted |
참고문헌
- Sekhar, A., Kay, L. E. An NMR view of protein dynamics in health and disease. Annu Rev Biophys. 48, 297-319 (2019).
- Rosenzweig, R., Kay, L. E. Bringing dynamic molecular machines into focus by methyl-TROSY NMR. Annu Rev Biochem. 83, 291-315 (2014).
- Palmer, A. G. Enzyme dynamics from NMR spectroscopy. Acc Chem Res. 48 (2), 457-465 (2015).
- Reif, B. Ultra-high resolution in MAS solid-state NMR of perdeuterated proteins: implications for structure and dynamics. J Magn Reson. 216, 1-12 (2012).
- Schanda, P., Ernst, M. Studying dynamics by magic-angle spinning solid-state NMR spectroscopy: Principles and applications to biomolecules. Prog Nucl Magn Reson Spectrosc. 96, 1-46 (2016).
- Le Marchand, T., et al. (1)H-H-detected biomolecular NMR under fast magic-angle spinning. Chem Rev. 122 (10), 9943-10018 (2022).
- Pervushin, K., Riek, R., Wider, G., Wuthrich, K. Attenuated T2 relaxation by mutual cancellation of dipole-dipole coupling and chemical shift anisotropy indicates an avenue to NMR structures of very large biological macromolecules in solution. Proc Natl Acad Sci U S A. 94 (23), 12366-12371 (1997).
- Tzakos, A. G., Grace, C. R., Lukavsky, P. J., Riek, R. NMR techniques for very large proteins and RNAs in solution. Annu Rev Biophys Biomol Struct. 35, 319-342 (2006).
- Sprangers, R., Kay, L. E. Quantitative dynamics and binding studies of the 20S proteasome by NMR. Nature. 445 (7128), 618-622 (2007).
- Wright, P. E., Dyson, H. J. Intrinsically disordered proteins in cellular signalling and regulation. Nat Rev Mol Cell Biol. 16 (1), 18-29 (2015).
- van der Lee, R., et al. Classification of intrinsically disordered regions and proteins. Chem Rev. 114 (13), 6589-6631 (2014).
- Tompa, P. Intrinsically unstructured proteins. Trends Biochem Sci. 27 (10), 527-533 (2002).
- Dyson, H. J., Wright, P. E. Coupling of folding and binding for unstructured proteins. Curr Opin Struct Biol. 12 (1), 54-60 (2002).
- Uversky, V. N. Intrinsically disordered proteins in overcrowded milieu: Membrane-less organelles, phase separation, and intrinsic disorder. Current Opinion in Structural Biology. 44, 18-30 (2017).
- Patel, A., et al. A liquid-to-solid phase transition of the ALS protein FUS accelerated by disease mutation. Cell. 162 (5), 1066-1077 (2015).
- Abyzov, A., Blackledge, M., Zweckstetter, M. Conformational dynamics of intrinsically disordered proteins regulate biomolecular condensate chemistry. Chem Rev. 122 (6), 6719-6748 (2022).
- Uversky, V. N. Unusual biophysics of intrinsically disordered proteins. Biochim Biophys Acta. 1834 (5), 932-951 (2013).
- Papoian, G. A. Proteins with weakly funneled energy landscapes challenge the classical structure-function paradigm. Proc Natl Acad Sci U S A. 105 (38), 14237-14238 (2008).
- Uversky, V. N., Gillespie, J. R., Fink, A. L. Why are "natively unfolded" proteins unstructured under physiologic conditions. Proteins. 41 (3), 415-427 (2000).
- Bah, A., et al. Folding of an intrinsically disordered protein by phosphorylation as a regulatory switch. Nature. 519 (7541), 106-109 (2015).
- Tompa, P., Schad, E., Tantos, A., Kalmar, L. Intrinsically disordered proteins: emerging interaction specialists. Curr Opin Struct Biol. 35, 49-59 (2015).
- Dyson, H. J., Wright, P. E. Perspective: the essential role of NMR in the discovery and characterization of intrinsically disordered proteins. J Biomol NMR. 73 (12), 651-659 (2019).
- Adamski, W., et al. A unified description of intrinsically disordered protein dynamics under physiological conditions using NMR spectroscopy. J Am Chem Soc. 141 (44), 17817-17829 (2019).
- Dobson, C. M. Protein folding and misfolding. Nature. 426 (6968), 884-890 (2003).
- Bertoncini, C. W., et al. Release of long-range tertiary interactions potentiates aggregation of natively unstructured alpha-synuclein. Proc Natl Acad Sci U S A. 102 (5), 1430-1435 (2005).
- Buell, A. K., et al. Solution conditions determine the relative importance of nucleation and growth processes in alpha-synuclein aggregation. Proc Natl Acad Sci U S A. 111 (21), 7671-7676 (2014).
- Salvi, N., Abyzov, A., Blackledge, M. Atomic resolution conformational dynamics of intrinsically disordered proteins from NMR spin relaxation. Prog Nucl Magn Reson Spectrosc. 102, 43-60 (2017).
- Schneider, R., Blackledge, M., Jensen, M. R. Elucidating binding mechanisms and dynamics of intrinsically disordered protein complexes using NMR spectroscopy. Curr Opin Struct Biol. 54, 10-18 (2019).
- Rezaei-Ghaleh, N., Blackledge, M., Zweckstetter, M. Intrinsically disordered proteins: from sequence and conformational properties toward drug discovery. Chembiochem. 13 (7), 930-950 (2012).
- Jensen, M. R., Salmon, L., Nodet, G., Blackledge, M. Defining conformational ensembles of intrinsically disordered and partially folded proteins directly from chemical shifts. J Am Chem Soc. 132 (4), 1270-1272 (2010).
- Camacho-Zarco, A. R., et al. NMR provides unique insight into the functional dynamics and interactions of intrinsically disordered proteins. Chem Rev. 122 (10), 9331-9356 (2022).
- Mittag, T., Forman-Kay, J. D. Atomic-level characterization of disordered protein ensembles. Curr Opin Struct Biol. 17 (1), 3-14 (2007).
- Dyson, H. J., Wright, P. E. Unfolded proteins and protein folding studied by NMR. Chem Rev. 104 (8), 3607-3622 (2004).
- Dyson, H. J., Wright, P. E. NMR illuminates intrinsic disorder. Curr Opin Struct Biol. 70, 44-52 (2021).
- Sugase, K., Dyson, H. J., Wright, P. E. Mechanism of coupled folding and binding of an intrinsically disordered protein. Nature. 447 (7147), 1021-1025 (2007).
- Bessa, L. M., et al. The intrinsically disordered SARS-CoV-2 nucleoprotein in dynamic complex with its viral partner nsp3a. Sci Adv. 8 (3), eabm4034 (2022).
- Milles, S., Salvi, N., Blackledge, M., Jensen, M. R. Characterization of intrinsically disordered proteins and their dynamic complexes: From in vitro to cell-like environments. Prog Nucl Magn Reson Spectrosc. 109, 79-100 (2018).
- Salvi, N., Abyzov, A., Blackledge, M. Atomic resolution conformational dynamics of intrinsically disordered proteins from NMR spin relaxation. Prog Nucl Magn Reson Spectrosc. 102 - 103, 43-60 (2017).
- Gill, M. L., Byrd, R. A., Palmer, A. G. Dynamics of GCN4 facilitate DNA interaction: a model-free analysis of an intrinsically disordered region. Phys Chem Chem Phys. 18 (8), 5839-5849 (2016).
- Bertoncini, C. W., et al. Structural characterization of the intrinsically unfolded protein beta-synuclein, a natural negative regulator of alpha-synuclein aggregation. J Mol Biol. 372 (3), 708-722 (2007).
- Rezaei-Ghaleh, N., et al. Local and global dynamics in intrinsically disordered synuclein. Angew Chem Int Ed Engl. 57 (46), 15262-15266 (2018).
- Rezaei-Ghaleh, N., Parigi, G., Zweckstetter, M. Reorientational dynamics of amyloid-beta from NMR spin relaxation and molecular simulation. J Phys Chem Lett. 10 (12), 3369-3375 (2019).
- Palmer, A. G. NMR characterization of the dynamics of biomacromolecules. Chem Rev. 104 (8), 3623-3640 (2004).
- Palmer, A. G., Massi, F. Characterization of the dynamics of biomacromolecules using rotating-frame spin relaxation NMR spectroscopy. Chem Rev. 106 (5), 1700-1719 (2006).
- Kay, L. E., Torchia, D. A., Bax, A. Backbone dynamics of proteins as studied by 15N inverse detected heteronuclear NMR spectroscopy: application to staphylococcal nuclease. Biochemistry. 28 (23), 8972-8979 (1989).
- Mittermaier, A., Kay, L. E. Review - New tools provide new insights in NMR studies of protein dynamics. Science. 312 (5771), 224-228 (2006).
- Abragam, A. . Principles of Nuclear Magnetism. , (1983).
- Ernst, R. R., Bodenhausen, G., Wokaun, A. . Principles of Nuclear Magnetic Resonance in One and Two Dimensions. , (1990).
- Cavanagh, J., Fairbrother, W. J., Palmer, A. G., Skelton, N. J. . Protein NMR Spectroscopy: Principles and Practice. , (1995).
- Levitt, M. H. . Spin Dynamics: Basics of Nuclear Magnetic Resonance. , (2008).
- Keeler, J. . Understanding NMR Spectroscopy. , (2010).
- Bloembergen, N., Purcell, E. M., Pound, R. V. Relaxation effects in nuclear magnetic resonance absorption. Phys Rev. 73 (7), 679-712 (1948).
- Wangsness, R. K., Bloch, F. The dynamical theory of nuclear induction. Phys Rev. 89 (4), 728-739 (1953).
- Redfield, A. G. On the theory of relaxation processes. IBM J Res Dev. 1 (1), 19-31 (1957).
- Peng, J. W., Wagner, G. Mapping of the spectral densities of N-H bond motions in eglin c using heteronuclear relaxation experiments. Biochemistry. 31 (36), 8571-8586 (1992).
- Farrow, N. A., et al. Backbone dynamics of a free and a phosphopeptide-complexed src homology-2 domain studied by n-15 NMR relaxation. Biochemistry. 33 (19), 5984-6003 (1994).
- Tolman, J. R., Ruan, K. NMR residual dipolar couplings as probes of biomolecular dynamics. Chem Rev. 106 (5), 1720-1736 (2006).
- Mulder, F. A. A., Skrynnikov, N. R., Hon, B., Dahlquist, F. W., Kay, L. E. Measurement of slow (mu s-ms) time scale dynamics in protein side chains by N-15 relaxation dispersion NMR spectroscopy: Application to Asn and Gln residues in a cavity mutant of T4 lysozyme. J Am Chem Soc. 123 (5), 967-975 (2001).
- Tollinger, M., Skrynnikov, N. R., Mulder, F. A. A., Forman-Kay, J. D., Kay, L. E. Slow dynamics in folded and unfolded states of an SH3 domain. J Am Chem Soc. 123 (46), 11341-11352 (2001).
- Akke, M., Palmer, A. G. Monitoring macromolecular motions on microsecond to millisecond time scales by R(1)rho-R(1) constant relaxation time NMR spectroscopy. J Am Chem Soc. 118 (4), 911-912 (1996).
- Loria, J. P., Rance, M., Palmer, A. G. A relaxation-compensated Carr-Purcell-Meiboom-Gill sequence for characterizing chemical exchange by NMR spectroscopy. J Am Chem Soc. 121 (10), 2331-2332 (1999).
- Vallurupalli, P., Bouvignies, G., Kay, L. E. Studying "Invisible" excited protein states in slow exchange with a major state conformation. J Am Chem Soc. 134 (19), 8148-8161 (2012).
- Jeener, J., Meier, B. H., Bachmann, P., Ernst, R. R. Investigation of exchange processes by 2-dimensional NMR-spectroscopy. J Chem Phys. 71 (11), 4546-4553 (1979).
- Palmer, A. G., Kroenke, C. D., Loria, J. P. Nuclear magnetic resonance methods for quantifying microsecond-to-millisecond motions in biological macromolecules. Methods Enzymol. 339, 204-238 (2001).
- Clore, G. M., Iwahara, J. Theory, practice, and applications of paramagnetic relaxation enhancement for the characterization of transient low-population states of biological macromolecules and their complexes. Chem Rev. 109 (9), 4108-4139 (2009).
- Iwahara, J., Clore, G. M. Detecting transient intermediates in macromolecular binding by paramagnetic NMR. Nature. 440 (7088), 1227-1230 (2006).
- Lakomek, N. A., et al. Residual dipolar couplings as a tool to study molecular recognition of ubiquitin. Biochem Soc Trans. 36 (Pt 6), 1433-1437 (2008).
- Johnson, C. N., Libich, D. S. Paramagnetic relaxation enhancement for detecting and characterizing self-associations of intrinsically disordered proteins. J Vis Exp. (175), e63057 (2021).
- Lakomek, N. A., Ying, J., Bax, A. Measurement of (1)(5)N relaxation rates in perdeuterated proteins by TROSY-based methods. J Biomol NMR. 53 (3), 209-221 (2012).
- Stief, T., Vormann, K., Lakomek, N. A. Sensitivity-enhanced NMR (15)N R(1) and R(1rho) relaxation experiments for the investigation of intrinsically disordered proteins at high magnetic fields. Methods. 223, 1-15 (2024).
- Kay, L. E., Keifer, P., Saarinen, T. Pure absorption gradient enhanced heteronuclear single quantum correlation spectroscopy with improved sensitivity. J Am Chem Soc. 114 (26), 10663-10665 (1992).
- Geen, H., Freeman, R. Band-selective radiofrequency pulses. J Magn Reson (1969). 93 (1), 93-141 (1991).
- Desvaux, H., Berthault, P. Study of dynamic processes in liquids using off-resonance RF irradiation. Prog Nucl Magn Reson Spectrosc. 35 (4), 295-340 (1999).
- Overhauser, A. W. Polarization of nuclei in metals. Phys Rev. 91 (2), 476-476 (1953).
- Overhauser, A. W. Polarization of nuclei in metals. Phys Rev. 92 (2), 411-415 (1953).
- Kumari, P., Frey, L., Sobol, A., Lakomek, N. A., Riek, R. (15)N transverse relaxation measurements for the characterization of micros-ms dynamics are deteriorated by the deuterium isotope effect on (15)N resulting from solvent exchange. J Biomol NMR. 72 (3-4), 125-137 (2018).
- Karschin, N., Krenek, S., Heyer, D., Griesinger, C. Extension and improvement of the methanol-d(4) NMR thermometer calibration. Magn Reson Chem. 60 (4), 203-209 (2022).
- Sattler, M., Schleucher, J., Griesinger, C. Heteronuclear multidimensional NMR experiments for the structure determination of proteins in solution employing pulsed field gradients. Prog Nucl Magn Reson Spectrosc. 34 (2), 93-158 (1999).
- Ferrage, F., Cowburn, D., Ghose, R. Accurate sampling of high-frequency motions in proteins by steady-state N-{H} nuclear Overhauser effect measurements in the presence of cross-correlated relaxation. J Am Chem Soc. 131 (17), 6048-6049 (2009).
- Delaglio, F., et al. NMRPipe: a multidimensional spectral processing system based on UNIX pipes. J Biomol NMR. 6 (3), 277-293 (1995).
- Skinner, S. P., et al. CcpNmr AnalysisAssign: a flexible platform for integrated NMR analysis. J Biomol NMR. 66 (2), 111-124 (2016).
- Lee, W., Tonelli, M., Markley, J. L. NMRFAM-SPARKY: enhanced software for biomolecular NMR spectroscopy. Bioinformatics. 31 (8), 1325-1327 (2015).
- Lee, W., Rahimi, M., Lee, Y., Chiu, A. POKY: a software suite for multidimensional NMR and 3D structure calculation of biomolecules. Bioinformatics. 37 (18), 3041-3042 (2021).
- Abyzov, A., et al. Identification of dynamic modes in an intrinsically disordered protein using temperature-dependent NMR relaxation. J Am Chem Soc. 138 (19), 6240-6251 (2016).
- Singh, A., Purslow, J. A., Venditti, V. 15N CPMG relaxation dispersion for the investigation of protein conformational dynamics on the micros-ms timescale. J Vis Exp. (170), e62395 (2021).
- Gopalan, A. B., Hansen, D. F., Vallurupalli, P. CPMG experiments for protein minor conformer structure determination. Methods Mol Biol. 1688, 223-242 (2018).
- Tugarinov, V., Kay, L. E. Methyl groups as probes of structure and dynamics in NMR studies of high-molecular-weight proteins. Chembiochem. 6 (9), 1567-1577 (2005).
- Lipari, G., Szabo, A. Model-free approach to the interpretation of nuclear magnetic resonance relaxation in macromolecules. 1. Theory and range of validity. J Am Chem Soc. 104 (17), 4546-4559 (1982).
- Lipari, G., Szabo, A. Model-Free Approach to the interpretation of nuclear magnetic-resonance relaxation in macromolecules .2. Analysis of experimental results. J Am Chem Soc. 104 (17), 4559-4570 (1982).
- Clore, G. M., et al. Deviations from the simple two-parameter model-free approach to the interpretation of nitrogen-15 nuclear magnetic relaxation of proteins. J Am Chem Soc. 112 (12), 4989-4991 (1990).
- Lemaster, D. M. Larmor frequency selective model free analysis of protein NMR relaxation. J Biomol NMR. 6 (4), 366-374 (1995).
- Peng, J. W., Wagner, G. Mapping of spectral density functions using heteronuclear NMR relaxation measurements. J Magn Reson (1969). 98 (2), 308-332 (1992).
- Farrow, N. A., Zhang, O. W., FormanKay, J. D., Kay, L. E. Characterization of the backbone dynamics of folded and denatured states of an SH3 domain. Biochemistry. 36 (9), 2390-2402 (1997).
- Khan, S. N., et al. Distribution of pico- and nanosecond motions in disordered proteins from nuclear spin relaxation. Biophys J. 109 (5), 988-999 (2015).
- Smith, A. A., Ernst, M., Meier, B. H., Ferrage, F. Reducing bias in the analysis of solution-state NMR data with dynamics detectors. J Chem Phys. 151 (3), 034102 (2019).
- Zumpfe, K., Smith, A. A. Model-free or not. Front Mol Biosci. 8, 727553 (2021).
- Robustelli, P., Trbovic, N., Friesner, R. A., Palmer, A. G. Conformational dynamics of the partially disordered yeast transcription factor GCN4. J Chem Theory Comput. 9 (11), 5190-5200 (2013).
- Salvi, N., Abyzov, A., Blackledge, M. Multi-timescale dynamics in intrinsically disordered proteins from NMR relaxation and molecular simulation. J Phys Chem Lett. 7 (13), 2483-2489 (2016).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유