A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
NMR 15N ניסויי הרפיה לחקר דינמיקה מבנית של פיקו-שנייה עד ננו-שניות של חלבונים
In This Article
Summary
ספקטרוסקופיית תהודה מגנטית גרעינית (NMR) יכולה לאפיין דינמיקה מבנית של חלבונים באופן ספציפי לשאריות. אנו מספקים פרוטוקול מעשי להקלטת NMR 15N R1 ו- R2 הרפיה ו {1H}-15N אפקט Overhauser הטרוגרעיני (hetNOE), רגיש לפיקושניות לציר הזמן של ננו-שניות.
Abstract
ספקטרוסקופיית תהודה מגנטית גרעינית (NMR) מאפשרת לחקור חלבונים בתמיסה ובטמפרטורות פיזיולוגיות. לעתים קרובות, קבוצות האמיד של עמוד השדרה של החלבונים או קבוצות המתיל בשרשראות צדדיות משמשות כמדווחות על דינמיקה מבנית בחלבונים. מחקר דינמי מבני של עמוד השדרה החלבוני של חלבונים כדוריים על דגימות 15N מסומנות ופרוטונים מלאות בדרך כלל עובד טוב עבור חלבונים עם משקל מולקולרי של עד 50 kDa. כאשר מיושמת דאוטרציה של שרשרת צדדית בשילוב עם ספקטרוסקופיה מותאמת להרפיה רוחבית (TROSY), ניתן להרחיב מגבלה זו עד 200 kDa עבור חלבונים כדוריים ועד 1 MDa כאשר המיקוד הוא בשרשראות הצד. כאשר חלבונים בעלי הפרעה פנימית (IDPs) או חלבונים עם אזורים בעלי הפרעה פנימית (IDR) נחקרים, מגבלות משקל אלה אינן חלות אך יכולות ללכת הרבה מעבר לכך. הסיבה לכך היא שעקורים או IDR, המאופיינים בגמישות פנימית גבוהה, מנותקים לעתים קרובות באופן דינמי. שיטות NMR שונות מציעות תובנות ברזולוציה אטומית על דינמיקה מבנית של חלבונים בטווח רחב של סקאלות זמן, מפיקושניות ועד שעות. מדידות הרפיה סטנדרטיות של 15N סוקרות את הגמישות הפנימית של חלבון ומאפיינות את דינמיקת עמוד השדרה של החלבון הנחווית בסקאלת הזמן המהירה פיקו עד ננו-שנייה. מאמר זה מציג פרוטוקול מעשי להקמה והקלטה של ניסויי NMR 15N R1, R2 ואפקט אוברהאוזר הטרו-גרעיני (hetNOE). אנו מציגים נתונים לדוגמה ומסבירים כיצד לפרש אותם בפשטות איכותית לפני כל ניתוח מתוחכם יותר.
Introduction
תפקידו של חלבון נקבע לא רק על ידי המבנה התלת-ממדי שלו אלא גם על ידי הדינמיקה המבנית שלו, הכוללת את גמישותו הפנימית ואת המעברים המבניים בין קונפורמציות שונות שהחלבון יאמץ. ספקטרוסקופיית תהודה מגנטית גרעינית (NMR) יכולה לחקור את הדינמיקה המבנית של חלבונים בתמיסה 1,2,3. התפתחויות אחרונות ב-NMR של מצב מוצק שזוהה על-ידי פרוטונים מאפשרות גם אפיון דינמיקה של חלבונים במצב פחות מסיס, כגון, למשל, קרום דו-שכבתי שומני 4,5,6. בתמיסה NMR, ניתן לחקור את הדינמיקה המבנית של עמוד השדרה של החלבון ושרשראות הצד של החלבון. עבור חלבון כדורי, מחקר דינמי מבני של עמוד השדרה של החלבון יכול להיות מושג עד 50 kDa ברגע שהחלבון מסומן איזוטופית 15N. כאשר משתמשים בדאוטרציה של שרשרת צידית ובספקטרוסקופיה מותאמת להרפיה רוחבית (TROSY), ניתן להרחיב מגבלה זו עד 200 kDa 7,8. כאשר ההתמקדות היא בדינמיקה של שרשרת צד, ניתן להרחיב את טווח החלבונים והקומפלקסים הנגישים עד 1 MDa 2,9.
מגבלות המשקל הנקובות אינן חלות על חלבונים בעלי הפרעה פנימית (IDPs), אשר לעתים קרובות מראים דינמיקה פנימית גבוהה. יותר מ-30% מהפרוטאום האיקריוטי מורכב מעקורים פנימיים או אזורים בעלי אי-סדר מהותי (IDRs)10,11,12,13. הם ממלאים תפקידים מרכזיים בתהליכים תאיים רבים, כגון העברת אותות ושעתוק1, ומעורבים לעתים קרובות בהפרדת פאזה תוך-תאית 14,15,16,17. עקורים חסרים מבנה ילידי תלת-ממדי (תלת-ממדי) מוגדר היטב בתנאים פיזיולוגיים, ויש להם נוף אנרגיה חלש או מחוספס17,18. בשל הידרופוביות נמוכה ודחייה אלקטרוסטטית חזקה המופצת על עמוד השדרה של עקורים או IDR, חסר כוח מניע לקיפול למבנה קשיח19. עקורים מאמצים לעתים קרובות קונפורמציה מקופלת כאשר הם מורכבים עם שותפים מחייבים אחרים 10,20,21. כמו כן, שינויים לאחר תרגום (PTM) מרחיבים את אפשרויות הקיפול של עקורים או IDR22,23. קיפול שגוי של עקורים זוהה כגורם למחלות שונות, כולל מחלות נוירודגנרטיביות 15,24,25,26.
עקורים ו- IDR מראים גמישות פנימית גבוהה 21,27,28. הרכבים קונפורמטיביים המציגים את השונות של מיקומים אטומיים וזוויות דיהדרליות נגזרו מהדמיות דינמיקה מולקולרית וממגבלות שהתקבלו מנתוני ניסוי 29,30,31,32. בשל הדינמיקה ואי הסדר הנובע מכך במצב הקפוא, צפיפות האלקטרונים המפוזרת מקשה על אפיונם מבחינה מבנית באמצעות שיטות חדישות בביולוגיה מבנית, כגון קריו-EM או קריסטלוגרפיה של קרני רנטגן. כמו כן, תנאי התגבשות או טכניקות הכנת דגימות לניסויים בטמפרטורות קריוגניות עשויים להשפיע על מרחב הקונפורמציה שחווים עקורים. עם זאת, תמיסה NMR עובד היטב עבור חלבונים דינמיים מאוד ולכן הוא מתאים היטב לחקירת עקורים 16,20,22,28,29,30,31,32,33,34,35,36,37,38.
כפי שהוצג לעיל, תמיסה NMR מציעה טכניקות שונות לחקר דינמיקה פנימית של חלבונים בטווח רחב של סקאלות זמן (איור 1), המבוססות בעיקר על הרפיית ספין 31,33,38,39,40,41,42.
הרפיית הספין של גרעיני 15N בקבוצות האמידים של עמוד השדרה של החלבון נגרמת על ידי שינויי כיוון זווית הקשר 1 H-15N עקב דינמיקה פנימית של חלבונים ותנועות קולקטיביות (כולל, כאשר רלוונטי, דיפוזיה סיבובית)27,43,44,45,46,47,48,49,50,51. בסקאלות זמן קצרות יותר מזמן המתאם הסיבובי τR (הזמן שהמולקולה צריכה לסובב קורן אחד, המכונה גם זמן מתאם כולל של טמבל), האנאיזוטרופיה של השינוי הכימי (CSA) והצמידות הדיפולרית (D) פעילות ואינן ממוצעות על ידי הדיפוזיה הסיבובית של החלבון. דינמיקה פנימית של עמוד השדרה של החלבון, הכוללת שינויים בזוויות הקשרים, כיוונים מחדש של קשרים וטלטלה סיבובית, גורמת לתנודות סטוכסטיות של CSA וטנזור צימוד דיפולרי, וכתוצאה מכך וריאציה של השדה המגנטי המקומי, מה שמוביל בסופו של דבר להרפיית ספין NMR 47,48,52,53. תנודות אלה יכולות להיות מתוארות על ידי פונקציית מתאם כוללת. התמרת פורייה של פונקציית המתאם הכוללת נקראת פונקציית הצפיפות הספקטרלית. בתאוריית הרפיית רדפילד הקלאסית למחצה, ניתן לתאר את קבועי קצב ההרפיה של NMR על ידי שילובים ליניאריים של פונקציות צפיפות ספקטרליות אלה54.
ניסויי הרפיה של עמוד השדרה 15N NMR שפותחו בתחילת שנות התשעים כוללים 15N R1, R1ρ ו- {1H}-15N ניסויי אפקט אוברהאוזר גרעיניים, רגישים לציר הזמן המהיר של פיקו-שנייה (ps) ננו-שנייה (ns), מהר יותר מזמן המתאם הסיבובי τR של החלבון 45,55,56,57. כדי לאפיין דינמיקה של עמוד השדרה איטית יותר מזמן המתאם הסיבובי τR, נעשה שימוש במה שמכונה ניסויי פיזור הרפיה, R1ρ, וניסויי קאר-פרסל-מייבום-גיל (CPMG) הרגישים לדינמיקה של מיקרו-שנייה (μs) - אלפית שנייה (ms) 44,46,58,59,60,61. דינמיקה איטית יותר ממיקרו-שניות יכולה להילכד על-ידי 15N העברת רוויה של חילופי כימיקלים (CEST) NMR62, ספקטרוסקופיית חליפין (EXSY, אלפיות שנייה לשניות) או NMR בזמן אמת (RT) (שניות לשעות)63,64. ניתן להשתמש באפקט PRE (שיפור הרפיה פאראמגנטי) של בדיקות פאראמגנטיות, כמו גם צימודים דיפולריים שיוריים (RDC), כדי להעריך את כל הטווח של דינמיקת ps עד ms 65,66,67,68.
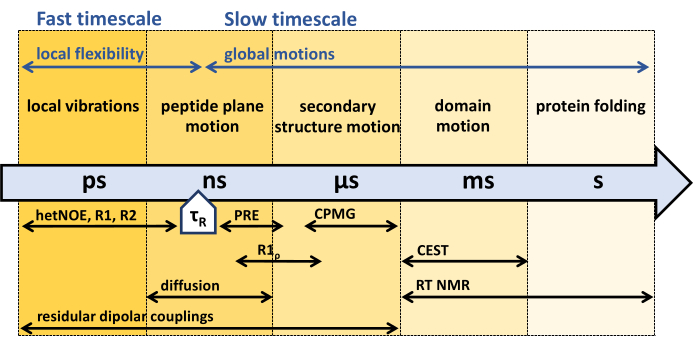
איור 1: סקאלות זמן של דינמיקה של עמוד השדרה של חלבונים וחלון זמן רגיש של ניסויים שונים בדינמיקת NMR. NMR מציע מגוון שיטות לאפיון דינמיקה של עמוד השדרה של חלבונים על פני מגוון רחב של טווחי זמן. תנועות שונות הנחוות על ידי עמוד השדרה של החלבון מסומנות בטווחי הזמן שלהן. זמן המתאם הסיבובי של החלבון, τR, הוא הזמן הדרוש לחלבון לסיבוב כולל (על ידי קורן אחד). תנועות מהירות יותר מזמן המתאם הסיבובי של החלבון, τR, יכולות להיות קשורות לגמישות הפנימית של החלבון. ניסויי תמ"ג שונים ורגישותם לטווחי הזמן המתאימים מסומנים מתחת לחץ. אנא לחץ כאן כדי להציג גרסה גדולה יותר של איור זה.
הפרוטוקול שלהלן מתאר את ההתקנה של ניסויי הרפיה NMR על ידי Lakomek et al.69 ו- Stief et al.70, תוך שימוש בסכימת זיהוי קוהרנטיות קוונטית יחידה (HSQC) משופרת רגישות. לפני שנמשיך ביישום הניסוי, ניתנת סקירה קצרה מאוד של ניסויי הרפיית ספין NMR וניסויי הרפיה של NMR. בשל מגבלות גודל וכדי לשמור על פרוטוקול זה מובן, סקירה זו חייבת להישאר פשטנית (ולכן חלקית).
הרפיית הסריג האורכית או הסחרור, המאופיינת בזמן T1 או קבוע קצב R1 = 1/ T1 , מתארת את חזרת המגנטיזציה לשיווי משקל בולצמן. בשיווי משקל, המגנטיזציה מיושרת לאורך ציר השדה המגנטי החיצוני, המגדיר את ציר z של מסגרת המעבדה. צפיפויות ספקטרליות בתדרי לרמור גבוהים (1H) וקטנים (15N) (תדרי תהודה NMR, למשל, 600 MHz עבור 1H עבור מגנט 14.1 טסלה) וצירופים ליניאריים של תדרי Larmor אלה תורמים להרפיה של 15N R1 , המאופיינת בקבועי קצב 15N R1 הנמדדים ב- rad·s-1. תנועות בסקאלות זמן הפוכות לתדרי לרמור אלה; לפיכך, תנועות בסקאלת הזמן פיקו-שנייה לננו-שנייה תורמות לקבוע קצב ההרפיה R1. עבור מולקולות המראות גלגול כולל ושבהן ניתן להגדיר זמן מתאם סיבובי, עקומת R1 (T1) מראה מקסימום (מינימום) עבור ωτR = 1, עם מתאם סיבוב τR ותדירות לרמור ω של הספין הנדון. אם מספר תדרי לרמור תורמים, התדר עם התדר הנמוך ביותר הוא התדר הדומיננטי, למשל ωN במקרה של 15N R1. משטר התנועה המהירה (ωτR קטן בהרבה מ-1) חל על מולקולות קטנות שנופלות מהר מאוד ועל שדות מגנטיים נמוכים וצמיגות נמוכה. משטר ההילוך האיטי (ωτR גדול בהרבה מ-1) תקף למולקולות גדולות יותר שנופלות לאט יותר ולשדות מגנטיים גבוהים וצמיגות גבוהה.
חלבונים מקופלים כדוריים מראים התהפכות כללית בתמיסה, וניתן להקצות זמן מתאם סיבובי. עם זאת, הרעיון של טמבל כללי אינו תקף עוד עבור חלבונים בעלי אי-סדר מהותי ולעתים קרובות שונה מהקצאת זמן מתאם סיבובי יחיד. כאן, זמן המתאם הפנימי הספציפי לשאריות הופך קריטי יותר.
רצף הדופק המתואר המודד 15N R1 קצבי הרפיה (איור 2) מבוסס על ניסוי קריאה HSQC משופר רגישות עם זיהוי Echo/ Anti-Echo לזיהוי ריבועי 69,70,71. שיפועים קצרים עם חוזק ואורך משתנים משמשים לבחירת קוהרנטיות ודיכוי מים משופר70. במהלך תקופה זו, קיטוב אורכי 15N יירגע. זמני דעיכה ארוכים יותר מובילים לעוצמות מופחתות במישורים הדו-ממדיים הקשורים לספקטרום פסאודו-תלת-ממדי זה (נקודות נתוני עיכוב נרשמות בממד השלישי). אלמנט לולאה, המתואר להלן, מבוצע מספר הולך וגדל של פעמים לזמני הרפיה ארוכים יותר. מכיוון שההרפיה הצולבת בין האנאיזוטרופיה של השינוי הכימי 15N (CSA) לבין הצימוד הדיפולרי של 1H ו-15 N (D) פעילה גם במהלך עיכוב ההרפיה, יש צורך בפעימת I-BURP-2 180°72 מרכזית, סלקטיבית על פרוטוני האמיד, כדי למקד מחדש את התרומה על ידי הרפיה צולבת (שאם לא תתמקד מחדש תוביל לקבועי קצב מוטים ושגויים של 15N R1).
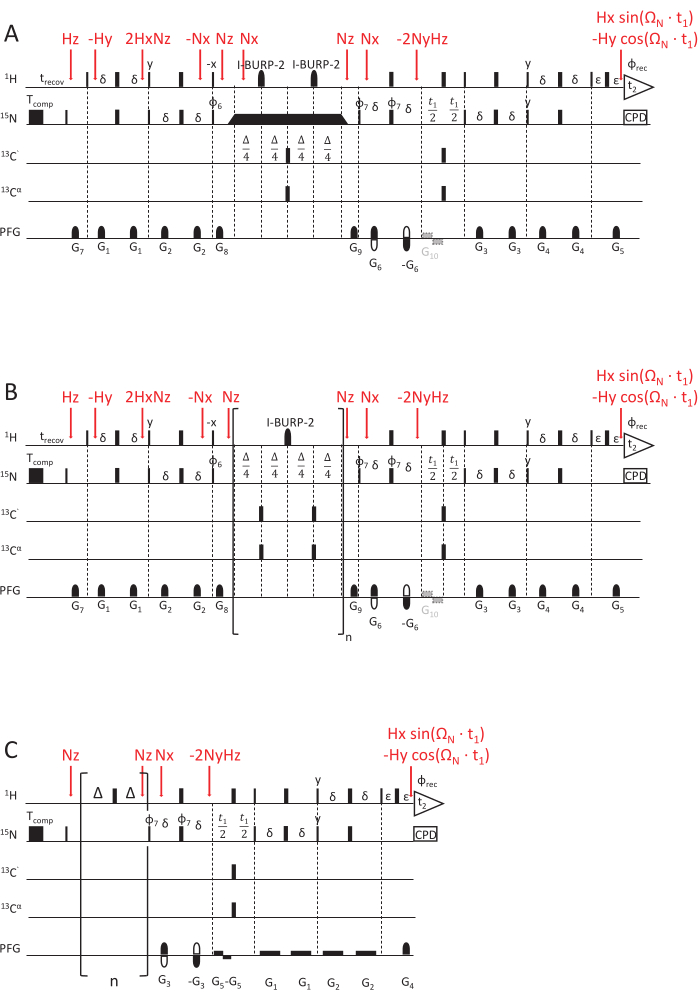
איור 2: סכמות רצף דופק NMR לקביעת קבועי קצב ההרפיה של NMR. (A) 15N R1ρ, (B) 15N R1 ו-(C) ניסוי hetNOE, באמצעות סכימת קריאה HSQC משופרת רגישות69,70. פולסים של 90°(x) מוצגים באופן חזותי על-ידי מלבנים צרים ופולסים של 180°(x) על-ידי מלבנים רחבים, אלא אם צוין אחרת. מחזור השלב הבא מוחל: φ6 = y, y, -y, -y; φ7 = y, -y, φrec = y, -y, -y, y. זיהוי ריבועי מושג על ידי היפוך הקוטביות של שיפוע G5 ומחזור הפאזה של φ7 (זיהוי הד/אנטי-אקו). (A) ניסוי 15N R1ρ: המלבן השחור מייצג את נעילת הספין, שעבורו משך הזמן משתנה כדי להשיג עיכובי הרפיה שונים. המשולשים לפני ואחרי נעילת הספין מציינים את פעימות הצורה האדיאבטיות המיישרות את המגנטיזציה לאורך ציר השדה המגנטי האפקטיבי Beff. G10 הוא שיפוע אופציונלי למניעת דעיכת קרינה של מגנטיזציה של מים במהלך שלב האבולוציה. (B) ניסוי 15N R1: החלק הסוגריים מציג את אלמנט הלולאה של הרצף, החוזר על עצמו n פעמים כדי להתאים לעיכוב ההרפיה הרצוי. (C) סכמת הדופק של hetNOE דומה למחצית השנייה של סכמות הדופק R1 ו-R1ρ, כלומר זמן האבולוציה t1 ואלמנט זיהוי HSQC. עם זאת, מגנטיזציה של 15N מעוררת ישירות ללא כל INEPT. הרוויה של מגנטיזציה של פרוטון (כדי להשיג הרפיה צולבת בין 1H ו 15N) מושגת על ידי רכבת של 180 (1H) פולסים המופעלים לפחות 5 שניות. עיכוב סרק של אותם אורכים (כאן, 5 שניות) ללא כל רכבת דופק מוחל על ניסוי הייחוס. G5 הוא שיפוע אופציונלי למניעת דעיכת קרינה, וההיפוך בקוטביות של שיפוע G4, בשילוב עם פאזה φ7 = y, -y, -y, y, משיג זיהוי ריבועי. שלבי העברת המגנטיזציה, המיוצגים על ידי מפעילי המוצר, מסומנים באדום. אנא לחץ כאן כדי להציג גרסה גדולה יותר של איור זה.
קבוע קצב ההרפיה R2 מתאר הרפיה של קיטוב רוחבי (באורתוגונלי מישור xy לשדה המגנטי החיצוני) עקב אובדן קוהרנטיות הפאזה בין הספין, מה שמוביל לדעיכה של מגנטיזציה ניתנת לגילוי53,54. פונקציית הצפיפות הספקטרלית בתדרים גבוהים וקטנים תורמת ל-R2, בדומה ל-R1. עם זאת, התרומה הגדולה ביותר ל-R2 נובעת מהצפיפות הספקטרלית בתדר אפס. מסיבה זו, R2 רגיש מאוד לטמבל הסיבובי, המתואר על ידי זמן המתאם הסיבובי τR, שהוא בסדר גודל של כמה ns עבור חלבון כדורי קטן בטמפרטורת החדר. לפיכך, תנועות איטיות יותר בעמוד השדרה במאות PS למשטר ns נמוך תורמות הכי הרבה. דינמיקת חליפין של עמוד השדרה הגורמת למודולציה של החלק האיזוטרופי של טנזור ההזזה הכימי של גרעיני 15N, באופן עקרוני, מוסיפה תרומת חליפין R2ex לקבועי קצב R2 43,44,49,60,61. בניסויים המתוארים, תרומת R2ex מדוכאת על ידי נעילת ספין הממקדת מחדש את הדינמיקה לאט יותר מהתדירות המעגלית ההופכית של נעילת הספין. ספין-לוק הוא פולס גלי רדיו ארוך ורציף השומר על המגנטיזציה מיושרת לאורך ציר השדה המגנטי האפקטיבי Beff (הסכום הווקטורי של שדה הספין-לוק ω1 והיסט ההסטה הכימית מתדר נשא 15N (ראה להלן)). הרפיה של המגנטיזציה המיושרת לאורך ציר B1,eff נקראת הרפיה R1ρ, שיש לה רכיב R1 ורכיב R2. באמצעות נוסחה (1), R2 ניתן לחשב מ R1ρ ו R144,73:
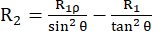 (1).
(1).
הזווית בין ציר השדה המגנטי האפקטיבי Beff לבין השדה המגנטי החיצוני B0 היא 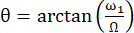 . ω1 היא משרעת RF של נעילת הספין ו- Ω היסט ההסטה הכימית בין ההסטה הכימית 15N של השארית המתאימה לבין תדר נשא 15N 44,73.
. ω1 היא משרעת RF של נעילת הספין ו- Ω היסט ההסטה הכימית בין ההסטה הכימית 15N של השארית המתאימה לבין תדר נשא 15N 44,73.
סכמת הדופק R1ρ (איור 2A, 70) דומה מאוד לסכמת 15N R1 למעט עיכוב ההרפיה. כדי למדוד את קצבי ההרפיה של 15N R1ρ , נעילת הספין חייבת להיות פעילה לאחר שהמגנטיזציה מיושרת לאורך ציר השדה האפקטיבי Beff על ידי פולס אדיאבטי עם אותה משרעת תדר רדיו (RF) כמו נעילת ספין. אורך הסחרור ישתנה כדי להשיג את עיכובי ההרפיה השונים.
אפקט אוברהאוזר גרעיני במצב יציב {1H}-15N (1 H-15N NOE), המכונה hetNOE להלן, הוא היחס בין קצב ההרפיה הצולבת לבין קצב ההרפיה האורכי 15N. זה מוביל להפחתה של הקיטוב במצב יציב על 15N עקב הרפיה צולבת עם הפרוטון עם הרוויה של קיטוב הפרוטון 45,53,54,74,75. הרפיה צולבת תלויה בפונקציות צפיפות ספקטרליות של הסכום וההפרש של תדרי לרמור 1H ו- 15N. לכן, ה-hetNOE רגיש הן לדינמיקת פיקו-שניות מהירה (<-100 ps) והן לדינמיקה של ps-ns (בשל התלות שלו ב-R1). רצף69 (איור 2C) מבוסס על קריאת HSQC משופרת רגישות עם שיפועי Echo/Anti-Echo לזיהוי ריבוע. עבור רוויה של מגנטיזציה של פרוטון וכתוצאה מכך hetNOE, מגנטיזציה של פרוטון שיווי משקל הפוכה ולאחר מכן רוויה על ידי פעימות מהירות של 180° במשך בערך פי 5 מ- 15 N T1. עבור ניסוי הייחוס, עיכוב ההתאוששות שווה לעיכוב הרוויה אך ללא רכבת הדופק1 H 180°. עיכוב נוסף של D1 = 2 שניות נוסף עבור ניסוי הייחוס וזה עם רוויה של 1H. שני הניסויים מוקלטים גב אל גב ונבדלים זה מזה רק ביישום פעימות של 1H 180 מעלות (רוויה) או לא (סימוכין). היחס בין העוצמות הספקטרליות שנרשמו בניסוי עם רוויה של 1H חלקי העוצמות של ניסוי הייחוס (ללא רכבת דופק פרוטונים של 180°) נותן את הערך {1H}-15N NOE (hetNOE).
הפרוטוקול הבא מתאר את ההתקנה של ניסויי הרפיה NMR על ידי Lakomek et al.69 ו- Stief et al.70. אנו מתמקדים ברצפי פולסי NMR באמצעות סכימת זיהוי HSQC משופרת רגישות. ניסויי 15N R1 ו- R1ρ מיושמים כמתואר בפירוט על ידי Stief et al.70, וניסוי hetNOE מתואר על ידי Lakomek et al.69.
Protocol
1. הכנת דגימת תמ"ג
הערה: סימון איזוטופים של החלבונים מבוצע עבור NMR ממדי גבוה יותר וניסויי NMR מתקדמים. כאשר ביטוי חלבונים באי-קולי וטיהור חלבונים נקבעו באמצעות מדיה עשירה (למשל, לוריא-ברטאני [LB] או 2x תמצית שמרים טריפטון בינוני [2YT]) עם תפוקה של כמה מיליגרם לליטר, הכנת דגימת NMR עם תווית איזוטופית היא בדרך כלל פשוטה יחסית.
- לסימון איזוטופים, השתמשו במדיה מינימלית M9 לביטוי גנים, כאשר 15N אמוניום כלורי הוא מקור החנקן היחיד וגלוקוז מועשר ב-13C הוא מקור הפחמן היחיד.
- לטיהור חלבונים, פעל לפי הפרוטוקול הסטנדרטי שנקבע במעבדה עבור החלבון המתאים.
הערה: סעיף התוצאות מציג תוצאות מייצגות עבור IDP, חלבון SNARE מונומרי Synaptobrevin-2 (1-96). תיוג איזוטופ NMR של Synaptobrevin-2 בוצע כמתוארב-70. למדידות ברמת דיוק גבוהה מאוד, מומלץ להשתמש בריכוזי D2O נמוכים, פחות מ-5%. הסיבה לכך היא אפקט איזוטופ הדאוטריום, שניתן לצפות בו בניסויי פיזור הרפיה של Hahn-Echo 15N T2 ובתדירות נמוכה של 15N CPMG (שלא נדונו כאן), אך יש לו השפעה מינימלית בלבד בניסויים של 15N R1ρ (שבהם הוא מתמקד בעיקר מחדש)76.
2. הכנות להפעלת ניסויי הרפיה NMR על הספקטרומטר
הערה: ניסויי ההרפיה המתוארים של NMR הם ספציפיים עבור ספקטרומטרים של Bruker. הם נבדקו על בדיקות תהודה משולשת קריוגניות ובטמפרטורת החדר 1H, 15N ו-13C וקונסולות Avance III ו-Avance Neo המופעלות על ידי תוכנת Bruker Topsin 3.6 ומעלה.
- בצע את אמצעי הזהירות.
- בצע תדריך בטיחות לפני העבודה בספקטרומטר NMR.
- בדוק את מפרט ההספק של הגשושית של הספקטרומטר המשומש ובחר אורך פולס ועוצמת פולס מתאימים לכל פולס, למשל, על ידי השוואת עקביות עם ערכי טבלת "פרוסול" Topspin.
- ייבא את רצפי הפולסים.
- הורד את תוכניות דופק NMR מהאתר המצוין להלן או מהמאגר המורחב של בנק התהודה המגנטית הביולוגית (BMRB) (bmrbig102) לספרייה /opt/topspin4.1.4/exp/stan/nmr/lists/pp/user של המחשב המפעיל את ספקטרומטר NMR. התאם את גרסת topspin (כאן: topspin4.1.4 לגרסה המתאימה המותקנת על הספקטרומטר).
- הורד את הפולסים בצורת NMR מהאתר המצוין להלן או מהמאגר המורחב של בנק התהודה המגנטית הביולוגית (BMRB) (bmrbig102) לספרייה /opt/topspin4.1.4/exp/stan/nmr/lists/pp/user של המחשב המפעיל את ספקטרומטר ה- NMR. אנא התאם את גרסת topspin (כאן: topspin 4.1.4 לגרסה המתאימה המותקנת על הספקטרומטר).
- כיול הספקטרומטר והדגימה.
- הגדר את טמפרטורת העניין. לכיול טמפרטורה, החל את הפרוטוקולים של המתקן. לדוגמה, השתמש בדגימת מתנול-d4 ולפרש את הפרש ההזזה הכימי שנצפה באמצעות מדחום NMR (https://chem.ch.huji.ac.il/nmr/software/thermometer.html); ראו גם77.
- הכנס את הדגימה (בתוספת 5% D2O נוספים עבור המנעול) והמתן כ- 10 דקות כדי לאפשר לדגימה לאזן את הטמפרטורה שלה. נעל את אות הדאוטריום (2H) (פקודה: נעל, בחר H2O+D2O).
- התאם וכוונן את ערוצי 1H, 15N ו- 13C, לדוגמה, באמצעות שגרת atma או atmm. אם לא מותקנת יחידת התאמה וכוונון אוטומטיים, התאימו וכווננו ידנית את הבדיקה באמצעות הפקודה wobb.
- בצעו שיימינג עבור שדה B0 הומוגני, למשל, באמצעות ה-tg השגרתי topshim.
- כייל את אורך הפולס של 90° (1H).
- השתמש בניסוי zg. השתמש getprosol לייבא את הפרמטרים בדיקה. בחר משך זמן קצר של 5 μs עבור P1. בדוק ותקן את רמת ההספק שצוינה של הבדיקה עבור PLW1 במידת הצורך.
- התחל את הניסוי עם zg. עבד את הנתונים עם efp. התאם את הפאזה עם .ph כך שקו הבסיס אחיד ואות ה- NMR של תהודת המים נמצא בפאזה.
- הערך פולס של 360° (1H) על-ידי לקיחת פולס נפוץ או משוער של 90° (1H) (למשל, ממפרטי הספקטרומטר המפורטים) והכפלתו פי ארבע. הפעל את הניסוי ועבד אותו.
הערה: משך הזמן שנבחר ארוך מדי אם האות חיובי וקצר מדי אם שלילי. פעימת 360° (1H) תהיה חציית האפס. - התאימו את אורך הדופק בהתאם. עשו זאת באופן איטרטיבי כדי למצוא את מעבר האפס. חלק את הערך המתקבל בארבע. זה יהיה הדופק של 90° (1H).
הערה: חלופה לשיטת 360° המתוארת היא שגרת פולסקל , אשר ניתן להתחיל משורת הפקודה ומאפשרת כיול אוטומטי של פעימת 1H.
- כיול תדר המוביל O1 לתהודה של המים.
- העתק את ניסוי zg על-ידי הקלדת edc בשורת הפקודה למספר ניסוי חדש. הקלד rpar כדי לפתוח את ערכות הפרמטרים (איור משלים 1A).
- חפש את ערכת הפרמטרים ZGPR. לחץ על קבוצת הפרמטרים ZGPR (איור משלים 1A), לחץ על קריאה, בחר ביצוע "getprosol" ולחץ על אישור (איור משלים 1B).
- שנה את P1 לאורך הפולס המכויל של 90° (1H). בחר אורך רוויה של D1 = 2 שניות ועוצמת פולס של PLW9 של 50 dB. הגדר את רווח המקלט (rg) ל- 16. לחלופין, בצע את התאמת רווח המקלט על ידי הקלדת rga והגדרת רווח המקלט לערך שצוין (אך הימנע מ"גלישת רווח מקלט").
- הקלד gs בשורת הפקודה כדי להתחיל ברכישה רציפה חוזרת. עבור אל חלון הרכישה ושנה את O1 כדי להקטין את אזור ה-FID הנשלט על-ידי אות המים (איור משלים 2).
- לחץ מתחת לסמן כדי להקטין את O1, ולחץ מעל הסמן כדי להגדיל אותו. השתמש בלחצן הרגישות כדי לשנות את גודל השלב. שמור את O1 הנוכחי; הסגירה עוצרת את הרכישה החוזרת (תרשים משלים 2).
- כייל את אורך הפולס של 90° (15N).
- העתק את ניסוי zg למספר ניסוי חדש (הקלד edc). הקלד rpar כדי לקרוא את ערכת הפרמטרים HSQCFPF3GPPHWG כדי לשנות את תוכנית הדופק לניסוי H-15N HSQC 1. השתמש באפשרות שמור פרמטרים P1, O1 ו- PLW1 (איור משלים 1).
- הקלד getprosol 1H <מכויל 90° (1H) אורך פולס ב μs> <90° (1H) עוצמת פולס ב db> בשורת הפקודה כדי לייבא את הפרמטרים בדיקה. הגדר את מספר הסריקות (NS) ל- 8 ואת סריקות הדמה (DS) ל- 2.
הערה: < > מציין כאן את הערכים שיש להגדיר. לדוגמה: getprosol 1H 8.5 -11.79. - הפעל את ההפרש הראשון של הניסוי על-ידי הקלדת zg בשורת הפקודה. העתק את ההפרש הראשון למספר ניסוי חדש עם rser 1 <מספר ניסוי חדש>. עבד את הנתונים עם efp ושלב את הספקטרום עם .יח. השתמש בהפרש הקבוע הראשון (ניסוי 1D) של הניסוי עבור השלבים הבאים.
הערה: < > לציין את המספר שנבחר עבור הניסוי החדש, לדוגמה, rser 1 101. - הקלד ased בשורת הפקודה כדי לפתוח את פרמטרי רצף הדופק של ניסוי 1D. לחץ על E בשורה PULPROG (ראה איור משלים 3A) כדי לפתוח את עורך תוכנית הדופק.
- השתמשו בתהליך העבודה הבא כדי לשמור את רצף הדופק בשם חדש: לחצו על 'קובץ' (בפינה השמאלית העליונה), על 'שמירה בשם', הזינו את השם החדש ולחצו על 'אשר'. לאחר מכן לחצו על Set PULPROG לערכת הנתונים (ראו איור משלים 3B) ולחצו על OK.
- שנה את רצף H-15N HSQC המקורי (נשמר תחת שם חדש, ראה 2.6.4.) על-ידי הכפלת 90° (15N) לאחר תקופת האבולוציה t1 (לפני ההעברה האחורית) בפקטור של 2 (פעימת 90° (15N) היא כעת 180° (15N)). הקש Ctrl S כדי לשמור את רצף הדופק. הגדר את אורך הפולס ל- 90° (15N) (P21) כך שאות ה- NMR של ההפרש הראשון ייעלם (במעבר האפס) (איור משלים 3B).
- פתח את ניסוי H-15N HSQC (ניסוי דו-ממדי) והזן את אורך הפולס המכויל של 90° (15N) (P21). הקלד getprosol 1H <מכויל 90° (1 H) אורך פולס ב μs> <90° (1 H) עוצמת פולס ב db> 15N <מכויל 90° (15N) אורך פולס ב μs> <90° (15N) עוצמת פולס ב db> בשורת הפקודה.
- לקבוע את רוחב ספקטרלי וניכוס זמני רכישה.
- בחלון פרמטר הרכישה עבור ניסוי H-15N HSQC 1(הקלד eda בשורת הפקודה), התאם את הרוחב הספקטרלי (פרמטר הרכישה SW) ואת מספר הפרשי הממדים בממדים ישירים ועקיפים (פרמטר רכישה TD).
- בחר עבור SW, 16 עמודים לדקה בממד הישיר (1H) ו- 30 עמודים לדקה בממד העקיף (15N). בחר עבור TD, 2048 בממד הישיר (1H) ו- 512 בממד העקיף (15N).
- לרכוש את 1 H-15N HSQC ניסוי.
- בדוק שכל האותות המעניינים מזוהים ושאין ממצאים כינוייים בספקטרום. שנה את O3P אם מרכז הספקטרום אינו מתאים למרכז התפלגות אות החלבון לאורך ציר 15N.
- הגדל את SW אם החלון הספקטרלי קטן מדי. הגדל את ה - TD כדי לקבל רזולוציית FID גבוהה יותר (בדוק את מפרט הבדיקה למשך הזמן המרבי של ניתוק).
הערה: זוהי ההגדרה הכללית ונקודת המוצא להגדרת ניסויי הרפיה NMR. מומלץ להקליט את ניסוי 1 H-15N HSQC בתחילת ובסוף כל סדרת הניסויים, ולמשך זמני ניסוי ארוכים יותר, גם בין לבין כדי לפקח על שלמות הדגימה.
- בחלון פרמטר הרכישה עבור ניסוי H-15N HSQC 1(הקלד eda בשורת הפקודה), התאם את הרוחב הספקטרלי (פרמטר הרכישה SW) ואת מספר הפרשי הממדים בממדים ישירים ועקיפים (פרמטר רכישה TD).
3. יישום ניסויי ההרפיה של תמ"ג
הערה: רצפי פעימות ההרפיה של NMR (איור 2) זמינים במאגר https://www.ipb.hhu.de/en/teams/team-lakomek/pulsesequences או במאגר המורחב של בנק התהודה המגנטית הביולוגית (BMRB) (bmrbig102).
- הגדר את 15נ ר1ρ ניסוי.
- העתק את ניסוי HSQC (הקלד edc) לספרייה חדשה. הקלד ased ושנה את תוכנית הדופק לניסוי 15N R1ρ על-ידי לחיצה על שלוש הנקודות של שורת הפרמטרים PULPROG בפרמטרים של רצף הדופק (ased, ראה איור משלים 3A). לחץ פעמיים על תוכנית הדופק, לחץ על הגדר PULPROG לערכת נתונים (ראה איור משלים 3B), ולחץ על אישור.
הערה: הודעות שגיאה מסוימות עשויות להופיע. סגור הודעות אלה. - הקלד gpnam בשורת הפקודה כדי להוסיף את כל צורות מעבר הצבע החסרות (gpnam1- gpnam9) כמתואר ברצף הפולסים (איור משלים 4).
- הקלד vplist בשורת הפקודה. לחצו על שלוש הנקודות (איור משלים 5A), קובץ (בפינה השמאלית העליונה) וחדש (איור משלים 5B).
- הקלד שם עבור vplist דמה חדש ולחץ על אישור. צור את vplist דמה (5m, 65m, 25m, 45m, 15m, 55m, 35m, 10m; שורה חדשה עבור כל ערך vplist). הקש Ctrl S כדי לשמור את הרשימה ולסגור את החלון של ה- vplist (איור משלים 5C).
- לחץ פעמיים על שם ה- vplist החדש שנוצר (איור משלים 5B).
הערה: זה הכרחי כדי למנוע שגיאות הידור ברצף הדופק. הערכים המתאימים של הסמנכ"ל ייקבעו בהמשך (נקודה 3.1.29). - הקלד ased כדי לפתוח את הפרמטרים של רצף הדופק (איור משלים 6). הזן את כל עוצמות השיפוע ואורכי השיפוע החסרים כפי שניתן ברצף הפולסים (איור משלים 6D).
- הזן עבור מונה הלולאה L3 (מספר נקודות מורכבות בממד העקיף) 1/2 מהערך 15N TD בחלון פרמטר הרכישה (eda). הזן עבור מונה הלולאה L6 את מספר נקודות נתוני ההרפיה המתועדות המשמשות להתאמת עקומת ההרפיה.
הערה: מספר זה L6 מתאים למספר ערכי vplist, כאן: 8 (איור משלים 6A). - הגדר את פעימת TanhTan_half.nl האדיאבטית, שהורדה מאתר האינטרנט שצוין לעיל, כצורה של פולס 15N SP8. הגדר את פולס TanhTan_2nd.nl האדיאבטי מהאתר כצורה של פולס 15N SP9 (איור משלים 6C).
- הגדר את ערך P8 לאורך דופק TanhTan האדיאבטי - מספיק זמן כדי להבטיח את האדיאבטיות של הדופק; כאן נבחר P8 = 3000 μs (איור משלים 6C).
- הגדר את ניתוק 15N (CPDPRG 3) לסכימת ניתוק garp. יש להשתמש לאורך ניתוק PCPD3 200 μs (איור משלים 6C).
- הגדר את השהיית השחזור הבין-סריקה ל- D1 = 2 שניות או יותר.
הערה: לבחירת עיכוב שחזור מתאים, עיין בסעיף דיון. - הגדר את סריקות הדמה (DS) ל- 64 לפחות עבור הניסוי הסופי (במהלך ההתקנה, DS = 4 מספיק מטעמי זמן). כנקודת התחלה, השתמש במספר סריקות (NS) של 4; השתמש בכפולות של 4 אם האות לרעש (SNR) נמוך מדי (איור משלים 6A).
- הגדר את O1 לתדר המוביל המכויל של שלב 2.5. הגדר את ה-O2P ל-176 עמודים לדקה והעתק את ה-O3P מניסוי H-15N HSQC (איור משלים 6A-C).
- הגדר את אורך הפולס P7 לאורך הפולס של 90° (15N) המכויל בשלב 2.6. כמו כן, העתק את עוצמת הפולס של פעימת 90° (15N) משלב 2.6 ל- PLW3 ו- PLW7 (איור משלים 6C).
- הגדר את אורך הפולס P1 ו- P19 לאורך הפולס של 90° (1H) המחושב בשלב 2.4. (תרשים משלים 6ב).
- הגדר את מספר ההפרשים הקבועים בממד העקיף TD = l3 * 2 * l6 (eda או הקלד td בשורת הפקודה).
הערה: כל הפרמטרים של רצף הדופק המתוארים כאן מבוארים בנוסף ברצף הדופק. - הגדר את פולס הצורה SP5 לצורת I-BURP2 (Iburp2.1000). הגדר את אורך הפולס P15 עד 2000 μs. פתח את תצוגת כלי הצורה על-ידי לחיצה על E לצד פעימת הצורה I-BURP2 בחלון פרמטר רצף הדופק Topspin (ased) (איור משלים 6B).
- הדמיית פולס הצורה באורך הנכון על-ידי לחיצה על הלחצן Start NMR Simulation . בדוק את אורך הפולס המעוצב [μs] (= 2000 μs) ואת זווית הסיבוב [°] (= 180°) בחלון הסימולציה. לחץ על Start NMR-SIM (איור משלים 7A).
- בדוק את טווח העירור ובחר את אורך הפולס המתאים של I-BURP2 כדי לכסות את הפיזור הספקטרלי של 1H של החלבון, אך הימנע מעירור של המים בו זמנית. הגדר את P15 לאורך הפולס המעוצב של חלון הסימולציה עם ההדמיה הטובה ביותר של פולס IBURP-2 (איור משלים 7B).
הערה: האורך המתאים נבחר אם I-BURP2 מעורר (> 95% יעילות) את כל אזור האמיד. כדי לדמות פולס I-BURP2 באורך שונה, סגור את חלון פרופיל העירור וערוך את אורך הפולס המעוצב בחלון הסימולציה. לחץ על התחל NMR-SIM. פולסים באורך קצר יותר הם בעלי טווח עירור רחב יותר בהרץ. - הגדר SPOFFS5 לשנות את תדר הנשא של פולס I-BURP2 ולהסיט את טווח העירור שמאלה או ימינה כדי למנוע מפולס I-BURP2 לגעת ולהפריע למגנטיזציה של המים (איור משלים 6B).
- כדי לקבוע את רמת העוצמה המתאימה של הפולס המעוצב, פתחו את הכלי צורת Bruker, לחצו על הלחצן Start NMR Simulation והגדירו את אורך הפולס I-BURP2 לאורך פולס מעוצב [μs]. זכרו את האורך המתאים של פולס מלבני רך של 90° (1H), הנראה בחלון הסימולציה בפולס המרובע המתאים של 90° [μs] (איור משלים 7A).
- סוג calcpowlev. השתמשו בפעימה הקשה של 90° (1H) (P1) במיקרו-שניות כאורך הייחוס (איור משלים 8B) והפולס המלבני הרך של 90° (1H) המתאים לפעימת I-BURP2 במיקרו-שניות כאורך החדש (איור משלים 8A). זכרו את הפרש ההספק Δ ב-dB בין הפולס הקשה של 90° (1H) לבין הפולס המלבני הרך (איור משלים 8C). העתק את עוצמת הדופק של הדופק הקשיח 90° (1H) ב- dB ל- SPW5 והוסף את ההבדל המשונן Δ ב- dB (איור משלים 6B).
- כדי לקבוע את רמת הכוח של נעילת הסחרור, חשב את אורך הפולס המתאים של 90° (15N) של נעילת הסחרור. השתמש בנוסחה: אורך פולס 90° (15N) ב- s = 0.25 / (כוח נעילת סיבוב בהרץ).
- השתמש ב- calcpowlev כדי לחשב את הפרש ההספק Δ ב- dB בין עוצמת נעילת הספין לבין הדופק הקשיח של 90° (15N) (P7). חדש: אורך פולס של 90° (15N) של נעילת הספין ב-μs (איור משלים 8A). Ref: אורך פולס קשה של 90° (15N) ב-μs (איור משלים 8B). העתק את רמת ההספק PLW7 של הפולס הקשיח 90° (15N) ב- dB לרמת הספק נעילת הספין PLW8 (איור משלים 6C), והוסף את הפרש ההספק המחושב Δ ב- dB (איור משלים 8C).
- העתק את רמת ההספק של נעילת הספין PLW8 לרמות צריכת החשמל SPW8 ו- SPW9. (איור משלים 6ג).
- כדי לקבוע את רמת ההספק של ניתוק 15N, השתמש ב- calcpowlev כדי לחשב את הפרש ההספק Δ בין העוצמה של פולס ניתוק 90° (15N) (PCPD3) לבין הדופק הקשיח של 90° (15N) (P7). חדש: PCPD3 ב-μs (200) (איור משלים 8A). Ref: אורך פולס קשה של 90° (15N) ב-μs (איור משלים 8B). העתק את רמת ההספק PLW7 של הפולס הקשיח 90° (15N) ב- dB לרמת הניתוק PLW31 (איור משלים 6C) והוסף Δ ב- dB (איור משלים 8C).
- כדי להשתמש בפיצוי טמפרטורה, כלול #define TEMP_COMPENSATION (על-ידי מחיקת הנקודה-פסיק ברצף הדופק). הגדר את P18 למשך הזמן המרבי של נעילת הספין ששימשה בניסוי 15N R1ρ .
הערה: מומלץ לכבות את פיצוי הטמפרטורה עבור הניסוי הראשון של הניסוי. - במקרה של 15דגימות מסומנות N 13C, כלול # כדי להגדיר LABEL_CN בתוכנית הדופק (על ידי מחיקת הנקודה-פסיק ברצף הדופק). הגדר את P4 לאורך הדופק המחושב באמצעות הנוסחה ב-78. לדוגמה, זה מתאים 23.70 μs ב 600 MHz ו 11.85 μs ב 1200 MHz. הקלד calcpowlev כדי לחשב את הפרש הכוח dB בין P4 לבין הדופק הקשה (13C). העתק את עוצמת הדופק הקשיח (13C) ב- dB ל- PLW4 והוסף את Δ ב- dB. העתק את PLW4 ל-PLW2 (איור משלים 6B).
- לקבוע את עיכובי ההרפיה עבור דגימה מתאימה.
- הפעל את שמונת ה- FIDs הראשונים (במקרה L6 = 8) ועבד אותם (rser <מספר FID> < מספר ניסוי חדש>). בחר את הערכים ב- vplist שעוצמת השיא של הניסוי עם ההשהיה הארוכה ביותר יורדת לפחות ל- 1/e (כ- 37%), אך לא פחות מ -25% בהשוואה לניסוי עם ההשהיה הקצרה ביותר (המשמשת כנקודת ייחוס).
- התאם את הערכים הנותרים של ה- vplist כדי להשיג דגימה אחידה של העוצמה פוחתת בין FID עם ההשהיה הארוכה ביותר לבין FID עם ההשהיה הקצרה ביותר. קרא את ההערה ואת מפרטי ההספק של הבדיקה.
הערה: השאריות המרגיעות לאט ישלטו ב- FID הראשון. התאם את הערכים הנותרים של ה- vplist כדי להשיג דגימה שווה של העוצמה פוחתת בין ה- FID הראשון והשני. אין לחרוג מעוצמת נעילת הספין והאורך המרביים המותרים של הגשושית (עיין במדריך הבדיקה ובמפרטי הבדיקה). במקרה של פיצוי טמפרטורה, בדוק את P18 (משך נעילת הספין המרבי), מכיוון שיוחל משך נעילת הספין הכולל של P18, מחולק בין עיכוב ההרפיה לפיצוי הטמפרטורה. הוא מחזיק זמן (עיכוב הרפיה) + זמן (פיצוי טמפרטורה) = const = P18. עבור נעילת ספין של 2 קילו-הרץ, אנו מעדיפים לא לחרוג ממשך נעילת סיבוב של יותר מ-65 אלפיות השנייה, באמצעות השהיית שחזור (D1) של 2 שניות. במקרה כזה, יש להתפשר בין משך נעילת הסחרור המרבי לבין עיכוב ההרפיה הארוך ביותר לטובת משך נעילת הסחרור המרבי המותר. במקרה של עיכובים ארוכים יותר בשחזור D1, ייתכן שיחולו כוחות נעילה ארוכים יותר. מה שקובע הוא שקיעת הכוח הכוללת בבדיקה לאורך זמן. אם נדרשים משכי זמן ארוכים יותר של נעילת סיבוב, הגדל את אורכי נעילת הספין רק בצעדים קטנים וצפה באות הנעילה. ככלל אצבע, התאימו את רמת הנעילה בשליש העליון של תצוגת המנעול. אם רמת הנעילה פוחתת ביותר משני ריבועים במהלך ביצוע רצף הדופק, כוח נעילת הספין המופקד בבדיקה הוא יותר מדי.
- הקלד rga בשורת הפקודה כדי לקבוע את רווח המקלט. לקבלת יחס אות לרעש טוב, כוון לערך רווח מקלט (rg) בין 128 ל- 256, מה שמוכיח דיכוי מים טוב.
הערה: לא ניתן להגיע לרווח מקלט בין 128 ל- 256 מציין אות מים חזק מדי. במקרה זה, עוצמת השיפוע ומשכי הזמן מכווננים בעדינות עד שמגיעים לרווח המקלט. ראה גם את פרק הדיון. - התחל ריצת בדיקה של הניסוי 15N R1ρ על-ידי הקלדת zg בשורת הפקודה. בדוק כי אות המים מדוכא היטב עבור כל העיכובים (1 עד 8, במקרה של 8 עיכובים vplist). בדוק גם את התוספת 9, שהיא התוספת השנייה של סכימת זיהוי הריבוע (Echo/ Anti-Echo).
- העתק את ניסוי HSQC (הקלד edc) לספרייה חדשה. הקלד ased ושנה את תוכנית הדופק לניסוי 15N R1ρ על-ידי לחיצה על שלוש הנקודות של שורת הפרמטרים PULPROG בפרמטרים של רצף הדופק (ased, ראה איור משלים 3A). לחץ פעמיים על תוכנית הדופק, לחץ על הגדר PULPROG לערכת נתונים (ראה איור משלים 3B), ולחץ על אישור.
- הגדר את ניסוי 15N R1 .
- העתק את ניסוי 15N R1ρ ושנה את תוכנית הדופק לניסוי 15N R1 .
- הקלד vclist, צור vclist והזן את vclist הדמה המתואר ב- 70: 0, 24, 6, 20, 4, 16, 8, 12; שורה חדשה עבור כל ערך VCLIST).
- פתח ased. בדוק את אורכי השיפוע ועוצמתם. שנה את GPZ9 ל- 14%.
- בדוק את אורכי הדופק, צורות הדופק ועוצמות הדופק (הערכים הנכונים היו צריכים להיות מועתקים מהניסוי 15N R1ρ ).
- במקרה של 15דגימות מסומנות N13C, השתמש #define LABEL_CN בתוכנית הדופק.
- לשימוש בפיצוי טמפרטורה, כלול #define TEMP_COMPENSATION.
- הפעל את שמונת ה- FIDs הראשונים (במקרה L6 = 8) ועבד אותם (rser <מספר FID> < מספר ניסוי חדש>). בחר את הערכים ב- vclist שעוצמת השיא של הניסוי עם ההשהיה הארוכה ביותר יורדת לפחות ל- 1/e (כ- 37%) אך לא פחות מ- 25% בהשוואה לניסוי עם ההשהיה הקצרה ביותר. התאם את הערכים הנותרים של ה- vplist כדי להשיג דגימה אחידה של העוצמה פוחתת בין FID עם ההשהיה הארוכה ביותר לבין FID עם ההשהיה הקצרה ביותר.
הערה: רק מספרים זוגיים מותרים עבור ערכי vclist.
- הגדר את ניסוי {1H}-15N NOE.
- העתק את ניסוי ההרפיה 15N R1 ושנה את רצף הדופק לסכמת הדופק {1H}-15N NOE.
- הגדר את אורך תקופת הרוויה עם מונה הלולאה L8. המרווח בין פולסים של 180° (1H) הוא 22 ms79 (2* 1 / JNH). כדי להשיג את התוצאות המדויקות ביותר, השתמש בתקופת רוויה של בערך פי 5 מהחלבון 15N T1(15N). הנה: 5 שניות. זה יתאים L8 = 230.
הערה: המקטע 'דיון' מספק עצות נוספות לבחירת תקופת הרוויה/עיכוב ההתאוששות. - שנה את אורך וחוזק שיפועי הצבע כמתואר בערכת הדופק. השתמש בצורות מעבר צבע sine.20.
- הגדר את מספר ההפרשים בממד העקיף השווה ל - TD = l3 * 2 * 2
- במקרה של 15דגימות מסומנות N13C, השתמש #define LABEL_CN ו -DLABEL_CN כמו ZGOPTNS.
- הפעל את ערכות הדופק על ידי הקלדת zg בשורת הפקודה.
4. עיבוד וניתוח ניסויי תמ"ג מוקלטים
הערה: Spectra הוקלטו באמצעות מערכת Bruker. העיבוד מתבצע באמצעות מערכת הפעלה יוניקס או לינוקס. עיבוד ספקטרום וניתוח נתונים בוצעו באמצעות NMRPipe80 ו- python3. ניתן להוריד את תוכנת NMRPipe בכתובת https://www.ibbr.umd.edu/nmrpipe/index.html. סקריפטים מבוססי NMRPipe מורדים מהאתר: https://www.ipb.hhu.de/en/teams/team-lakomek/pulsesequences או מהמאגר המורחב של בנק התהודה המגנטית הביולוגית (BMRB) (bmrbig102). מומלץ להשתמש ב- NMRPipe. אם NMRPipe אינו זמין או רצוי, ניתן להשתמש בחלופות כגון CCPN81 או SPARKY (SPARKY 3, או יורשיו NMRFAM-SPARKY82 או POKY83 ).
- עיבוד וניתוח 15נ ר1 ניסוי.
הערה: הפקודות הבאות מבוצעות במסוף יוניקס.- העתק את הקבצים בספריה שהורדת R1_processing לספרייה המכילה את נתוני R1 הניסיוניים (ספריית עבודה).
- הפוך את הקבצים להפעלה לפי פקודת המסוף: > chmod u+x *.com
- הפעל > bruker כדי לפתוח את כלי ההמרה NMRPipe וליצור את קובץ fid.com. לחץ על Read Parameters. שנה את ספירת הממדים לשתיים אם סופרים שלושה ממדים, ולחץ שוב על קריאת פרמטרים . לאחר מכן לחצו על Clear Script, Update Script ו-Save Script (איור משלים 9).
הערה: אם תדר המוביל אינו נכון, שנה את PPM מיקום המרכז לאחר לחיצה על קריאת פרמטרים בפעם השנייה. - ביצוע > ./fid.com
- המר את ה- vclist ב- vplist עם ערכי ms: עיכוב ב- ms = 40 ms * ערך של vclist. השתמש > cp vclist vplist וערוך את vplist.
- השתמש ב- ./split.com > כדי לפצל את נתוני התלת-ממד המדומים לשמונה קבצים דו-ממדיים.
- ערוך את ערכי tauValues (ערכי vplist לפי סדר ה- vplist) ואת ערכי idxExpmt (סדר ערכי tauValues ביחס לזמן) ב- nmrproc.com (איור משלים 10).
- השתמש ב- ./nmrproc.com > כדי לעבד את הנתונים הגולמיים של כל שמונת הספקטרום הדו-ממדי.
- השתמש ב- nmrDraw > כדי לצייר את הספקטרה.
- השתמש בספקטרום ההפניות (הערך הראשון ב- vplist) עבור שלבים אלה. עבור לספקטרום זה על-ידי שינוי ממד z לאחד (בפינה השמאלית העליונה). התאם את רמת קווי המתאר באמצעות + ו- לצד לחצן גורם . צייר מחדש את הספקטרום באמצעות לחצן צייר.
- השתמשו ברמת מתאר שבה ניתן לזהות את כל האותות המעניינים באמצעות אותות מעטים בלבד שמגיעים מהרעש (רמת קווי המתאר נמצאת בקצה רמת הרעש) (איור משלים 11).
- תקן את שלב הספקטרום, הקלד v עבור הקרנת אות 1D אנכית ו - h עבור הקרנת אות 1D אופקית. התאימו את הפאזה עם P0 ו-P1 (איור משלים 11), זכרו את הפרש הפאזה ותקנו את הפאזה בקובץ nmrproc.com (איור משלים 10).
הערה: אם הספקטרום הפוך בממד 15N, הסר את -neg בשורה: | nmrPipe -fn FT -neg \ (איור משלים 10). - סגור nmrצייר ועבד שוב עם > nmrproc.com. לבחירת מרב (לאחר שהספקטרום נמצא בשלבים נכונים וספקטרום הייחוס מוצג ברמת מיתאר שבה כל הפסגות קיימות), הקלד K (shift + k), הקש Detect ולחץ על Save. (תרשים משלים 11).
- העתק רשימת שיא לספריית העבודה ושנה את -assName ב- ass.com לשם רשימת השיא (איור משלים 12).
הערה: דוגמה לרשימת שיא בתבנית הנכונה זמינה בכתובת https://www.ipb.hhu.de/en/teams/team-lakomek/pulsesequences
ובמאגר המורחב של בנק התהודה המגנטית הביולוגית (BMRB) (bmrbig102). - בצע > ./ass.com כדי להקצות את הספקטרה. אמת את המשימה שהסקריפט מציע עבור הפסגות השונות. השתמש בלחצן העכבר השמאלי כדי להקצות ובלחצן העכבר הימני כדי לבטל את הקצאת הפסגות. השתמשו בלחצן הבא כדי לעבור לחומצת האמינו הבאה וב-Prev כדי לעבור לחומצת האמינו הקודמת (איור משלים 13).
הערה: ניתן לשנות את רמת קווי המתאר באמצעות + ו- בפינה השמאלית העליונה. השתמש בלחצן צייר כדי לצייר מחדש את הספקטרום עם רמת קווי המתאר החדשה (איור משלים 13). - לאחר השלמת המשימה, לחץ על שמור ולאחר מכן על שמור שוב. לאחר מכן, לחץ על יציאה, ביטול ויציאה (איור משלים 13).
- הפעל > python3 NoneDeleter.py כדי ליצור relax.tab, המכיל רק מידע על פסגות שהוקצו.
- הפעל > ./relax.com כדי ליצור את קובץ ה- Script של autofit.com.
- בצע > ./autoFit.com כדי לבצע אינטרפולציה של פורייה וליצור יחסי פסאודו של זמני ההשהיה השונים עבור כל חומצת אמינו (axt.tab ו- nlin.tab נוצרים).
- הפעל > ./model.com כדי ליצור את קובץ ה- Script של modelExp.com בהתבסס על הנתונים ב - nlin.tab.
- בצע > ./modelExp.com כדי להתאים את עקומת האבולוציה של כל שארית עם מודל אופטימיזציה לא ליניארי של הכי פחות ריבועים.
- בצע > bash summary.tcl -in relax.tab > t1.tab. התעלם מהודעת שגיאה אפשרית: הודעת GDB: 21 ערכים חסרים בקובץ הטבלה עלולים להתרחש.
- העתק t1.tab לספריה חדשה שנוצרה R1R2.
- עבד ונתח את ניסוי 15N R1ρ .
- העתק את הקבצים בספריה שהורדת R1rho_processing לספרייה המכילה את נתוני R1ρ הניסיוניים (ספריית עבודה).
- ביצוע > chmod u+x *.com
- הפעל > bruker כדי לפתוח את כלי ההמרה NMRPipe וליצור את קובץ fid.com. ראה גם עיבוד של ספקטרום 15N R1 .
- ביצוע > ./fid.com
- הפעל > ./split.com כדי לפצל את נתוני התלת-ממד המדומים לשמונה קבצים דו-ממדיים.
- בצע > ./nmrproc.com (ערוך מראש את ערכי tauValues [vplist] ואת idxExpmt ) כדי לעבד את הנתונים הגולמיים של כל שמונת הספקטרום הדו-ממדי (איור משלים 10).
- בצע > nmrDraw כדי לצייר את הספקטרום ולבחור את הפסגות. השתמש בספקטרום ההפניות (הערך הראשון ב- vplist) עבור שלבים אלה. בצע את אותו הליך כמו בניסוי 15N R1 .
- העתק רשימת שיא לספריית העבודה ושנה את -assName ב- ass.com לשם רשימת השיא (איור משלים 12).
- בצע > ./ass.com ופעל לפי אותו הליך כמו בניסוי 15N R1 .
- הפעל > python3 NoneDeleter.py כדי ליצור relax.tab, המכיל רק מידע על פסגות שהוקצו.
- ביצוע > ./relax.com
- ביצוע > ./autoFit.com
- ביצוע > ./model.com
- ביצוע > ./modelExp.com
- ביצוע > bash summary.tcl -in relax.tab > t1rho. tab. התעלם מהודעת שגיאה אפשרית: הודעת GDB: 21 ערכים חסרים בקובץ הטבלה עלולים להתרחש.
- העתק את הכותרת הבאה בקובץ t1rho.tab (ניתן לקבל את הערכים מקובץ fid.com) (איור משלים 14):
הערה 600MHz עבור T1rho
הערה -yOBS = 60.818 (שים כאן את הערך שנמצא ב- fid.com)
הערה -yCAR = 119.06 (שים כאן את הערך שנמצא ב- fid.com) - בצע > ./extract_PPM.com כדי ליצור טבלה בשם fusionOffset.tab המכילה את ההיסט של תדר השיא 15N ביחס לתדר המוביל (שיהיה צורך לחשב את הזווית θ לחילוץ נתוני R2 מנתוני R1 ו- R1ρ ).
- סכם את נתוני ההרפיה.
- העתק את t1rho.tab ואת fusionOffset.tab לספריה R1R2.
- העתק את הסקריפט crtR1p.tcl בספריה R1R2, התאם את תדר הספקטרומטר ואת עוצמת נעילת הספין והפעל את bash crtR1p.tcl | מיין -n > R1R2.tab. (תרשים משלים 15). R1R2.tab יכיל את קבועי קצב R1ρ מותאמים, זווית תטא, קבועי קצב R2 וקבועי קצב R1 .
- לעבד ולנתח את ניסוי hetNOE.
- העתק את הקבצים בספריה שהורדת NOE_processing לספריית הנתונים הניסיונית NOE (ספריית עבודה).
- ביצוע > chmod u+x *.com
- הפעל > bruker כדי לפתוח את כלי ההמרה NMRPipe וליצור את קובץ fid.com. לחץ על Read Parameters. שנה את ספירת הממדים לשתיים אם נספרו שלושה ממדים והקש שוב על קריאת פרמטרים . לאחר מכן הקש Clear Script, Update Script ו - Save Script.
- ביצוע > ./fid.com
- ביצוע > ./split.com (יש ליצור vplist עם שני ערכים (0, 1) לפני כן)
- ביצוע > ./ft2.com
- בצע > nmrDraw כדי לצייר את הספקטרום ולבחור את הפסגות בספקטרום הייחוס (z-dimension = 1). חלץ וזכור את הרעש (הקש T = shift + t). תיקון פאזה ובחירת שיא עוקבים אחר אותו הליך כמו ניסוי 15N R1 .
- העתק רשימת שיא לספריית העבודה ושנה את -assName ב- ass.com לשם רשימת השיא (איור משלים 12)
- בצע > ./ass.com כדי להקצות את הספקטרה. בצע את אותו הליך כמפורט עבור ניסוי 15N R1 .
- הפעל > python3 NoneDeleter.py כדי ליצור relax.tab.
- ביצוע > ./script.com
- ביצוע > ./autoFit.com
- שנה את הרעש ב- error_hetnoe.tcl.
- Execute > bash error_hetnoe.tcl |sort -n > hetNOE.tab
תוצאות
להלן כמה נתוני הרפיה לדוגמה של NMR שנרשמו על חלבון SNARE שלפוחית Synaptobrevin-2 (1-96), המכונה לעתים קרובות VAMP2 (חלבון הקשור לשלפוחית 2). לצורך רישום נתוני NMR, השתמשנו בדגימה של 171 μM 15N Synaptobrevin-2 (1-96) (המכונה Syb-2 להלן) במאגר של 50 mM MES (pH 6.0) המכיל 150 mM NaCl, 0.1 mM TCEP ו-1 mM EDTA. כל נתוני הניסוי תועדו...
Discussion
פרוטוקול זה תיאר את ההתקנה של ניסויי הרפיה NMR 15N על ידי Lakomek et al.69 ו- Stief et al.70. התמקדנו ברצפי פולסי NMR באמצעות סכימת זיהוי HSQC משופרת רגישות. ניסויי 15N R1 ו- R1ρ מיושמים כמתואר בפירוט על ידי Stief et al.70, וניסוי hetNOE מתואר ע...
Disclosures
המחברים מצהירים כי אין להם אינטרסים כלכליים מתחרים ידועים או קשרים אישיים שיכלו להשפיע לכאורה על העבודה המדווחת במאמר זה.
Acknowledgements
אנו מודים למלינדה ג'ספר ולקווין בוצ'ינסקי על הדיונים המועילים. נ.ל. מודה לקרן המדע הגרמנית על המימון באמצעות תוכנית הייזנברג (מענק DFG מספר 433700474). עבודה זו נתמכת גם על ידי הפרויקט "גורמים וירולוגיים ואימונולוגיים של פתוגנזה COVID-19 - לקחים כדי להתכונן למגיפות עתידיות (KA1-Co-02 "COVIPA"), מענק מקרן היוזמה והרשתות של אגודת הלמהולץ. אנו מכירים בגישה נדיבה למרכז NMR ביומולקולרי יוליך-דיסלדורף, המנוהל במשותף על ידי Forschungszentrum Jülich ואוניברסיטת היינריך היינה דיסלדורף (HHU).
Materials
| Name | Company | Catalog Number | Comments |
| Bruker 600 MHz AVANCE III HD spectrometer | Bruker | https://www.bruker.com/en/products-and-solutions/mr/nmr/avance-nmr-spectrometer.html | NMR experiments conducted |
References
- Sekhar, A., Kay, L. E. An NMR view of protein dynamics in health and disease. Annu Rev Biophys. 48, 297-319 (2019).
- Rosenzweig, R., Kay, L. E. Bringing dynamic molecular machines into focus by methyl-TROSY NMR. Annu Rev Biochem. 83, 291-315 (2014).
- Palmer, A. G. Enzyme dynamics from NMR spectroscopy. Acc Chem Res. 48 (2), 457-465 (2015).
- Reif, B. Ultra-high resolution in MAS solid-state NMR of perdeuterated proteins: implications for structure and dynamics. J Magn Reson. 216, 1-12 (2012).
- Schanda, P., Ernst, M. Studying dynamics by magic-angle spinning solid-state NMR spectroscopy: Principles and applications to biomolecules. Prog Nucl Magn Reson Spectrosc. 96, 1-46 (2016).
- Le Marchand, T., et al. (1)H-H-detected biomolecular NMR under fast magic-angle spinning. Chem Rev. 122 (10), 9943-10018 (2022).
- Pervushin, K., Riek, R., Wider, G., Wuthrich, K. Attenuated T2 relaxation by mutual cancellation of dipole-dipole coupling and chemical shift anisotropy indicates an avenue to NMR structures of very large biological macromolecules in solution. Proc Natl Acad Sci U S A. 94 (23), 12366-12371 (1997).
- Tzakos, A. G., Grace, C. R., Lukavsky, P. J., Riek, R. NMR techniques for very large proteins and RNAs in solution. Annu Rev Biophys Biomol Struct. 35, 319-342 (2006).
- Sprangers, R., Kay, L. E. Quantitative dynamics and binding studies of the 20S proteasome by NMR. Nature. 445 (7128), 618-622 (2007).
- Wright, P. E., Dyson, H. J. Intrinsically disordered proteins in cellular signalling and regulation. Nat Rev Mol Cell Biol. 16 (1), 18-29 (2015).
- van der Lee, R., et al. Classification of intrinsically disordered regions and proteins. Chem Rev. 114 (13), 6589-6631 (2014).
- Tompa, P. Intrinsically unstructured proteins. Trends Biochem Sci. 27 (10), 527-533 (2002).
- Dyson, H. J., Wright, P. E. Coupling of folding and binding for unstructured proteins. Curr Opin Struct Biol. 12 (1), 54-60 (2002).
- Uversky, V. N. Intrinsically disordered proteins in overcrowded milieu: Membrane-less organelles, phase separation, and intrinsic disorder. Current Opinion in Structural Biology. 44, 18-30 (2017).
- Patel, A., et al. A liquid-to-solid phase transition of the ALS protein FUS accelerated by disease mutation. Cell. 162 (5), 1066-1077 (2015).
- Abyzov, A., Blackledge, M., Zweckstetter, M. Conformational dynamics of intrinsically disordered proteins regulate biomolecular condensate chemistry. Chem Rev. 122 (6), 6719-6748 (2022).
- Uversky, V. N. Unusual biophysics of intrinsically disordered proteins. Biochim Biophys Acta. 1834 (5), 932-951 (2013).
- Papoian, G. A. Proteins with weakly funneled energy landscapes challenge the classical structure-function paradigm. Proc Natl Acad Sci U S A. 105 (38), 14237-14238 (2008).
- Uversky, V. N., Gillespie, J. R., Fink, A. L. Why are "natively unfolded" proteins unstructured under physiologic conditions. Proteins. 41 (3), 415-427 (2000).
- Bah, A., et al. Folding of an intrinsically disordered protein by phosphorylation as a regulatory switch. Nature. 519 (7541), 106-109 (2015).
- Tompa, P., Schad, E., Tantos, A., Kalmar, L. Intrinsically disordered proteins: emerging interaction specialists. Curr Opin Struct Biol. 35, 49-59 (2015).
- Dyson, H. J., Wright, P. E. Perspective: the essential role of NMR in the discovery and characterization of intrinsically disordered proteins. J Biomol NMR. 73 (12), 651-659 (2019).
- Adamski, W., et al. A unified description of intrinsically disordered protein dynamics under physiological conditions using NMR spectroscopy. J Am Chem Soc. 141 (44), 17817-17829 (2019).
- Dobson, C. M. Protein folding and misfolding. Nature. 426 (6968), 884-890 (2003).
- Bertoncini, C. W., et al. Release of long-range tertiary interactions potentiates aggregation of natively unstructured alpha-synuclein. Proc Natl Acad Sci U S A. 102 (5), 1430-1435 (2005).
- Buell, A. K., et al. Solution conditions determine the relative importance of nucleation and growth processes in alpha-synuclein aggregation. Proc Natl Acad Sci U S A. 111 (21), 7671-7676 (2014).
- Salvi, N., Abyzov, A., Blackledge, M. Atomic resolution conformational dynamics of intrinsically disordered proteins from NMR spin relaxation. Prog Nucl Magn Reson Spectrosc. 102, 43-60 (2017).
- Schneider, R., Blackledge, M., Jensen, M. R. Elucidating binding mechanisms and dynamics of intrinsically disordered protein complexes using NMR spectroscopy. Curr Opin Struct Biol. 54, 10-18 (2019).
- Rezaei-Ghaleh, N., Blackledge, M., Zweckstetter, M. Intrinsically disordered proteins: from sequence and conformational properties toward drug discovery. Chembiochem. 13 (7), 930-950 (2012).
- Jensen, M. R., Salmon, L., Nodet, G., Blackledge, M. Defining conformational ensembles of intrinsically disordered and partially folded proteins directly from chemical shifts. J Am Chem Soc. 132 (4), 1270-1272 (2010).
- Camacho-Zarco, A. R., et al. NMR provides unique insight into the functional dynamics and interactions of intrinsically disordered proteins. Chem Rev. 122 (10), 9331-9356 (2022).
- Mittag, T., Forman-Kay, J. D. Atomic-level characterization of disordered protein ensembles. Curr Opin Struct Biol. 17 (1), 3-14 (2007).
- Dyson, H. J., Wright, P. E. Unfolded proteins and protein folding studied by NMR. Chem Rev. 104 (8), 3607-3622 (2004).
- Dyson, H. J., Wright, P. E. NMR illuminates intrinsic disorder. Curr Opin Struct Biol. 70, 44-52 (2021).
- Sugase, K., Dyson, H. J., Wright, P. E. Mechanism of coupled folding and binding of an intrinsically disordered protein. Nature. 447 (7147), 1021-1025 (2007).
- Bessa, L. M., et al. The intrinsically disordered SARS-CoV-2 nucleoprotein in dynamic complex with its viral partner nsp3a. Sci Adv. 8 (3), eabm4034 (2022).
- Milles, S., Salvi, N., Blackledge, M., Jensen, M. R. Characterization of intrinsically disordered proteins and their dynamic complexes: From in vitro to cell-like environments. Prog Nucl Magn Reson Spectrosc. 109, 79-100 (2018).
- Salvi, N., Abyzov, A., Blackledge, M. Atomic resolution conformational dynamics of intrinsically disordered proteins from NMR spin relaxation. Prog Nucl Magn Reson Spectrosc. 102 - 103, 43-60 (2017).
- Gill, M. L., Byrd, R. A., Palmer, A. G. Dynamics of GCN4 facilitate DNA interaction: a model-free analysis of an intrinsically disordered region. Phys Chem Chem Phys. 18 (8), 5839-5849 (2016).
- Bertoncini, C. W., et al. Structural characterization of the intrinsically unfolded protein beta-synuclein, a natural negative regulator of alpha-synuclein aggregation. J Mol Biol. 372 (3), 708-722 (2007).
- Rezaei-Ghaleh, N., et al. Local and global dynamics in intrinsically disordered synuclein. Angew Chem Int Ed Engl. 57 (46), 15262-15266 (2018).
- Rezaei-Ghaleh, N., Parigi, G., Zweckstetter, M. Reorientational dynamics of amyloid-beta from NMR spin relaxation and molecular simulation. J Phys Chem Lett. 10 (12), 3369-3375 (2019).
- Palmer, A. G. NMR characterization of the dynamics of biomacromolecules. Chem Rev. 104 (8), 3623-3640 (2004).
- Palmer, A. G., Massi, F. Characterization of the dynamics of biomacromolecules using rotating-frame spin relaxation NMR spectroscopy. Chem Rev. 106 (5), 1700-1719 (2006).
- Kay, L. E., Torchia, D. A., Bax, A. Backbone dynamics of proteins as studied by 15N inverse detected heteronuclear NMR spectroscopy: application to staphylococcal nuclease. Biochemistry. 28 (23), 8972-8979 (1989).
- Mittermaier, A., Kay, L. E. Review - New tools provide new insights in NMR studies of protein dynamics. Science. 312 (5771), 224-228 (2006).
- Abragam, A. . Principles of Nuclear Magnetism. , (1983).
- Ernst, R. R., Bodenhausen, G., Wokaun, A. . Principles of Nuclear Magnetic Resonance in One and Two Dimensions. , (1990).
- Cavanagh, J., Fairbrother, W. J., Palmer, A. G., Skelton, N. J. . Protein NMR Spectroscopy: Principles and Practice. , (1995).
- Levitt, M. H. . Spin Dynamics: Basics of Nuclear Magnetic Resonance. , (2008).
- Keeler, J. . Understanding NMR Spectroscopy. , (2010).
- Bloembergen, N., Purcell, E. M., Pound, R. V. Relaxation effects in nuclear magnetic resonance absorption. Phys Rev. 73 (7), 679-712 (1948).
- Wangsness, R. K., Bloch, F. The dynamical theory of nuclear induction. Phys Rev. 89 (4), 728-739 (1953).
- Redfield, A. G. On the theory of relaxation processes. IBM J Res Dev. 1 (1), 19-31 (1957).
- Peng, J. W., Wagner, G. Mapping of the spectral densities of N-H bond motions in eglin c using heteronuclear relaxation experiments. Biochemistry. 31 (36), 8571-8586 (1992).
- Farrow, N. A., et al. Backbone dynamics of a free and a phosphopeptide-complexed src homology-2 domain studied by n-15 NMR relaxation. Biochemistry. 33 (19), 5984-6003 (1994).
- Tolman, J. R., Ruan, K. NMR residual dipolar couplings as probes of biomolecular dynamics. Chem Rev. 106 (5), 1720-1736 (2006).
- Mulder, F. A. A., Skrynnikov, N. R., Hon, B., Dahlquist, F. W., Kay, L. E. Measurement of slow (mu s-ms) time scale dynamics in protein side chains by N-15 relaxation dispersion NMR spectroscopy: Application to Asn and Gln residues in a cavity mutant of T4 lysozyme. J Am Chem Soc. 123 (5), 967-975 (2001).
- Tollinger, M., Skrynnikov, N. R., Mulder, F. A. A., Forman-Kay, J. D., Kay, L. E. Slow dynamics in folded and unfolded states of an SH3 domain. J Am Chem Soc. 123 (46), 11341-11352 (2001).
- Akke, M., Palmer, A. G. Monitoring macromolecular motions on microsecond to millisecond time scales by R(1)rho-R(1) constant relaxation time NMR spectroscopy. J Am Chem Soc. 118 (4), 911-912 (1996).
- Loria, J. P., Rance, M., Palmer, A. G. A relaxation-compensated Carr-Purcell-Meiboom-Gill sequence for characterizing chemical exchange by NMR spectroscopy. J Am Chem Soc. 121 (10), 2331-2332 (1999).
- Vallurupalli, P., Bouvignies, G., Kay, L. E. Studying "Invisible" excited protein states in slow exchange with a major state conformation. J Am Chem Soc. 134 (19), 8148-8161 (2012).
- Jeener, J., Meier, B. H., Bachmann, P., Ernst, R. R. Investigation of exchange processes by 2-dimensional NMR-spectroscopy. J Chem Phys. 71 (11), 4546-4553 (1979).
- Palmer, A. G., Kroenke, C. D., Loria, J. P. Nuclear magnetic resonance methods for quantifying microsecond-to-millisecond motions in biological macromolecules. Methods Enzymol. 339, 204-238 (2001).
- Clore, G. M., Iwahara, J. Theory, practice, and applications of paramagnetic relaxation enhancement for the characterization of transient low-population states of biological macromolecules and their complexes. Chem Rev. 109 (9), 4108-4139 (2009).
- Iwahara, J., Clore, G. M. Detecting transient intermediates in macromolecular binding by paramagnetic NMR. Nature. 440 (7088), 1227-1230 (2006).
- Lakomek, N. A., et al. Residual dipolar couplings as a tool to study molecular recognition of ubiquitin. Biochem Soc Trans. 36 (Pt 6), 1433-1437 (2008).
- Johnson, C. N., Libich, D. S. Paramagnetic relaxation enhancement for detecting and characterizing self-associations of intrinsically disordered proteins. J Vis Exp. (175), e63057 (2021).
- Lakomek, N. A., Ying, J., Bax, A. Measurement of (1)(5)N relaxation rates in perdeuterated proteins by TROSY-based methods. J Biomol NMR. 53 (3), 209-221 (2012).
- Stief, T., Vormann, K., Lakomek, N. A. Sensitivity-enhanced NMR (15)N R(1) and R(1rho) relaxation experiments for the investigation of intrinsically disordered proteins at high magnetic fields. Methods. 223, 1-15 (2024).
- Kay, L. E., Keifer, P., Saarinen, T. Pure absorption gradient enhanced heteronuclear single quantum correlation spectroscopy with improved sensitivity. J Am Chem Soc. 114 (26), 10663-10665 (1992).
- Geen, H., Freeman, R. Band-selective radiofrequency pulses. J Magn Reson (1969). 93 (1), 93-141 (1991).
- Desvaux, H., Berthault, P. Study of dynamic processes in liquids using off-resonance RF irradiation. Prog Nucl Magn Reson Spectrosc. 35 (4), 295-340 (1999).
- Overhauser, A. W. Polarization of nuclei in metals. Phys Rev. 91 (2), 476-476 (1953).
- Overhauser, A. W. Polarization of nuclei in metals. Phys Rev. 92 (2), 411-415 (1953).
- Kumari, P., Frey, L., Sobol, A., Lakomek, N. A., Riek, R. (15)N transverse relaxation measurements for the characterization of micros-ms dynamics are deteriorated by the deuterium isotope effect on (15)N resulting from solvent exchange. J Biomol NMR. 72 (3-4), 125-137 (2018).
- Karschin, N., Krenek, S., Heyer, D., Griesinger, C. Extension and improvement of the methanol-d(4) NMR thermometer calibration. Magn Reson Chem. 60 (4), 203-209 (2022).
- Sattler, M., Schleucher, J., Griesinger, C. Heteronuclear multidimensional NMR experiments for the structure determination of proteins in solution employing pulsed field gradients. Prog Nucl Magn Reson Spectrosc. 34 (2), 93-158 (1999).
- Ferrage, F., Cowburn, D., Ghose, R. Accurate sampling of high-frequency motions in proteins by steady-state N-{H} nuclear Overhauser effect measurements in the presence of cross-correlated relaxation. J Am Chem Soc. 131 (17), 6048-6049 (2009).
- Delaglio, F., et al. NMRPipe: a multidimensional spectral processing system based on UNIX pipes. J Biomol NMR. 6 (3), 277-293 (1995).
- Skinner, S. P., et al. CcpNmr AnalysisAssign: a flexible platform for integrated NMR analysis. J Biomol NMR. 66 (2), 111-124 (2016).
- Lee, W., Tonelli, M., Markley, J. L. NMRFAM-SPARKY: enhanced software for biomolecular NMR spectroscopy. Bioinformatics. 31 (8), 1325-1327 (2015).
- Lee, W., Rahimi, M., Lee, Y., Chiu, A. POKY: a software suite for multidimensional NMR and 3D structure calculation of biomolecules. Bioinformatics. 37 (18), 3041-3042 (2021).
- Abyzov, A., et al. Identification of dynamic modes in an intrinsically disordered protein using temperature-dependent NMR relaxation. J Am Chem Soc. 138 (19), 6240-6251 (2016).
- Singh, A., Purslow, J. A., Venditti, V. 15N CPMG relaxation dispersion for the investigation of protein conformational dynamics on the micros-ms timescale. J Vis Exp. (170), e62395 (2021).
- Gopalan, A. B., Hansen, D. F., Vallurupalli, P. CPMG experiments for protein minor conformer structure determination. Methods Mol Biol. 1688, 223-242 (2018).
- Tugarinov, V., Kay, L. E. Methyl groups as probes of structure and dynamics in NMR studies of high-molecular-weight proteins. Chembiochem. 6 (9), 1567-1577 (2005).
- Lipari, G., Szabo, A. Model-free approach to the interpretation of nuclear magnetic resonance relaxation in macromolecules. 1. Theory and range of validity. J Am Chem Soc. 104 (17), 4546-4559 (1982).
- Lipari, G., Szabo, A. Model-Free Approach to the interpretation of nuclear magnetic-resonance relaxation in macromolecules .2. Analysis of experimental results. J Am Chem Soc. 104 (17), 4559-4570 (1982).
- Clore, G. M., et al. Deviations from the simple two-parameter model-free approach to the interpretation of nitrogen-15 nuclear magnetic relaxation of proteins. J Am Chem Soc. 112 (12), 4989-4991 (1990).
- Lemaster, D. M. Larmor frequency selective model free analysis of protein NMR relaxation. J Biomol NMR. 6 (4), 366-374 (1995).
- Peng, J. W., Wagner, G. Mapping of spectral density functions using heteronuclear NMR relaxation measurements. J Magn Reson (1969). 98 (2), 308-332 (1992).
- Farrow, N. A., Zhang, O. W., FormanKay, J. D., Kay, L. E. Characterization of the backbone dynamics of folded and denatured states of an SH3 domain. Biochemistry. 36 (9), 2390-2402 (1997).
- Khan, S. N., et al. Distribution of pico- and nanosecond motions in disordered proteins from nuclear spin relaxation. Biophys J. 109 (5), 988-999 (2015).
- Smith, A. A., Ernst, M., Meier, B. H., Ferrage, F. Reducing bias in the analysis of solution-state NMR data with dynamics detectors. J Chem Phys. 151 (3), 034102 (2019).
- Zumpfe, K., Smith, A. A. Model-free or not. Front Mol Biosci. 8, 727553 (2021).
- Robustelli, P., Trbovic, N., Friesner, R. A., Palmer, A. G. Conformational dynamics of the partially disordered yeast transcription factor GCN4. J Chem Theory Comput. 9 (11), 5190-5200 (2013).
- Salvi, N., Abyzov, A., Blackledge, M. Multi-timescale dynamics in intrinsically disordered proteins from NMR relaxation and molecular simulation. J Phys Chem Lett. 7 (13), 2483-2489 (2016).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved