Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Experimentos de relajación de RMN 15N para la investigación de la dinámica estructural de proteínas de picosegundos a nanosegundos
En este artículo
Resumen
La espectroscopia de resonancia magnética nuclear (RMN) puede caracterizar la dinámica estructural de proteínas de una manera específica para cada residuo. Proporcionamos un protocolo práctico para registrar la relajación de RMN 15N R1 y R2 y los experimentos de efecto Overhauser heteronuclear (hetNOE) de {1H}-15N, sensible a la escala de tiempo de picosegundos a nanosegundos.
Resumen
La espectroscopia de resonancia magnética nuclear (RMN) permite estudiar proteínas en solución y a temperaturas fisiológicas. Con frecuencia, los grupos amida de la columna vertebral de la proteína o los grupos metilo en las cadenas laterales se utilizan como indicadores de la dinámica estructural de las proteínas. Un estudio de dinámica estructural de la columna vertebral de proteínas globulares en muestras marcadas con 15N y completamente protonadas generalmente funciona bien para proteínas con un peso molecular de hasta 50 kDa. Cuando se aplica la deuteración de cadena lateral en combinación con espectroscopia optimizada de relajación transversal (TROSY), este límite puede extenderse hasta 200 kDa para proteínas globulares y hasta 1 MDa cuando el foco está en las cadenas laterales. Cuando se investigan proteínas intrínsecamente desordenadas (IDP) o proteínas con regiones intrínsecamente desordenadas (IDR), estas limitaciones de peso no se aplican, pero pueden ir mucho más allá. La razón es que los IDP o IDR, caracterizados por una alta flexibilidad interna, suelen estar desacoplados dinámicamente. Varios métodos de RMN ofrecen información de resolución atómica sobre la dinámica estructural de las proteínas en una amplia gama de escalas de tiempo, desde picosegundos hasta horas. Las mediciones estándar de relajación de 15N describen la flexibilidad interna de una proteína y caracterizan la dinámica de la columna vertebral de la proteína experimentada en la escala de tiempo rápida de picos a nanosegundos. Este artículo presenta un protocolo práctico para configurar y registrar experimentos de RMN 15N R1, R2 y de efecto Overhauser heteronuclear (hetNOE). Mostramos datos ejemplares y explicamos cómo interpretarlos de forma sencilla y cualitativa antes de cualquier análisis más sofisticado.
Introducción
La función de una proteína está determinada no solo por su estructura tridimensional, sino también por su dinámica estructural, que abarca su flexibilidad interna y las transiciones estructurales entre las diferentes conformaciones que adoptará la proteína. La espectroscopia de resonancia magnética nuclear (RMN) puede investigar la dinámica estructural de las proteínas en la solución 1,2,3. Los desarrollos recientes en RMN de estado sólido detectada por protones también permiten la caracterización de la dinámica de proteínas en un estado menos soluble, como, por ejemplo, una membrana de bicapa lipídica 4,5,6. En la RMN en solución, se puede estudiar la dinámica estructural de la columna vertebral de la proteína y las cadenas laterales de la proteína. Para una proteína globular, se puede lograr un estudio de dinámica estructural de la columna vertebral de la proteína hasta 50 kDa una vez que la proteína está marcada isotópicamente con 15N. Cuando se emplea la deuteración de cadena lateral y la espectroscopia optimizada de relajación transversal (TROSY), este límite puede extenderse hasta 200 kDa 7,8. Cuando la atención se centra en la dinámica de la cadena lateral, la gama de proteínas y complejos accesibles puede ampliarse hasta 1 MDa 2,9.
Las limitaciones de peso mencionadas no se aplican a las proteínas intrínsecamente desordenadas (IDP), que con frecuencia muestran una alta dinámica intrínseca. Más del 30% del proteoma eucariota está compuesto por IDPs o regiones intrínsecamente alteradas (IDRs)10,11,12,13. Desempeñan un papel central en muchos procesos celulares, como la transducción de señales y la transcripción1, y frecuentemente están implicados en la separación de fases intracelulares 14,15,16,17. Los desplazados internos carecen de una estructura nativa tridimensional (3D) bien definida en condiciones fisiológicas y tienen un paisaje energético débilmente canalizado o accidentado17,18. Debido a una baja hidrofobicidad y una fuerte repulsión electrostática distribuida sobre la columna vertebral de los IDP o IDR, falta una fuerza motriz para plegarse a una estructura rígida19. Con frecuencia, los PIC adoptan una conformación plegada cuando se componen con otros socios de unión 10,20,21. Además, las modificaciones postraduccionales (PTM) amplían las posibilidades de plegamiento de los IDP o IDR22,23. El mal plegamiento de los desplazados internos ha sido identificado como causa de diversas enfermedades, incluidas las neurodegenerativas 15,24,25,26.
Los IDP y los IDR muestran una gran flexibilidad interna 21,27,28. Los conjuntos conformacionales que muestran la variación de las posiciones atómicas y los ángulos diedros se derivaron de simulaciones de dinámica molecular y restricciones obtenidas a partir de datos experimentales 29,30,31,32. Debido a la dinámica y al desorden resultante en el estado congelado, la densidad difusa de electrones dificulta su caracterización estructural utilizando métodos de última generación en biología estructural, como la crio-EM o la cristalografía de rayos X. Además, las condiciones de cristalización o las técnicas de preparación de muestras para experimentos a temperaturas criogénicas pueden afectar el espacio conformacional experimentado por los desplazados internos. Sin embargo, la solución de RMN funciona bien para proteínas altamente dinámicas y, por lo tanto, es adecuada para investigar las IDP 16,20,22,28,29,30,31,32,33,34,35,36,37,38.
Como se presentó anteriormente, la RMN en solución ofrece varias técnicas para estudiar la dinámica interna de las proteínas en una amplia gama de escalas de tiempo (Figura 1), basadas principalmente en la relajación del espín 31,33,38,39,40,41,42.
La relajación del espín de los núcleos 15N en los grupos amida de la columna vertebral de la proteína es inducida por cambios en la orientación del ángulo de enlace 1H-15 N debido a la dinámica interna de la proteína y a los movimientos colectivos (incluida, cuando corresponda, la difusión rotacional)27,43,44,45,46,47,48,49,50,51 . A escalas de tiempo más cortas que el tiempo de correlación rotacional τR (el tiempo que la molécula necesita para rotar un radiante, también denominado tiempo de correlación de caída general), la anisotropía por desplazamiento químico (CSA) y el acoplamiento dipolar (D) están activos y no promediados por la difusión rotacional de la proteína. La dinámica interna de la columna vertebral de la proteína, que comprende variaciones en los ángulos de enlace, reorientaciones de enlaces y volteretas rotacionales, induce fluctuaciones estocásticas del CSA y el tensor de acoplamiento dipolar, lo que resulta en una variación del campo magnético local, lo que finalmente conduce a la relajación del espín de la RMN 47,48,52,53. Estas fluctuaciones pueden describirse mediante una función de correlación global. La transformada de Fourier de la función de correlación general se denomina función de densidad espectral. En la teoría semiclásica de la relajación de Redfield, las constantes de la tasa de relajación de la RMN se pueden describir mediante combinaciones lineales de estas funciones de densidad espectral54.
Los experimentos de relajación de RMN de 15N de la columna vertebral desarrollados a principios de la década de 1990 comprenden experimentos de efecto Overhauser nuclear de 15N R1, R1ρ y {1H}-15N, sensibles a la escala de tiempo de picosegundos (ps) nanosegundos (ns) rápidos, más rápido que el tiempo de correlación rotacional τR de la proteína 45,55,56,57. Para caracterizar la dinámica de la columna vertebral más lenta que el tiempo de correlación rotacional τR, se utilizan los llamados experimentos de dispersión de relajación, R1ρ, y los experimentos de Carr-Purcell-Meiboom-Gill (CPMG) sensibles a la dinámica de microsegundos (μs) - milisegundos (ms) 44,46,58,59,60,61. La dinámica más lenta que microsegundos puede ser capturada por RMN de transferencia de saturación de intercambio químico (CEST) de 15N62, espectroscopía de intercambio (EXSY, milisegundos a segundos) o RMN en tiempo real (RT) (segundos a horas)63,64. El efecto PRE (mejora de la relajación paramagnética) de las sondas paramagnéticas, así como de los acoplamientos dipolares residuales (RDC), se puede utilizar para evaluar todo el rango de dinámicas de ps a ms 65,66,67,68.
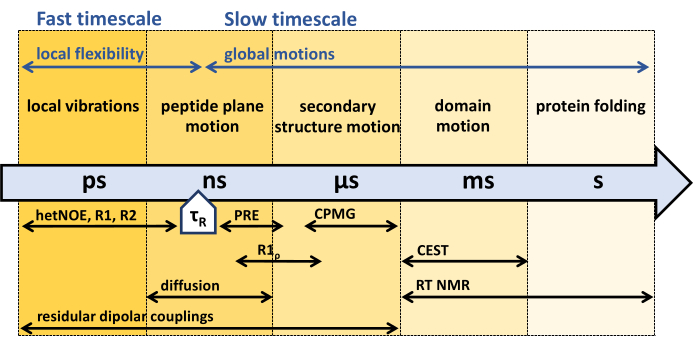
Figura 1: Escalas de tiempo de la dinámica de la columna vertebral de proteínas y ventana de tiempo sensible de diferentes experimentos de dinámica de RMN. La RMN ofrece una variedad de métodos para caracterizar la dinámica de la columna vertebral de proteínas en una amplia gama de escalas de tiempo. Los diferentes movimientos experimentados por la columna vertebral de la proteína se indican en sus respectivas escalas de tiempo. El tiempo de correlación rotacional de la proteína, τR, es el tiempo que la proteína necesita para una rotación general (en un radiante). Los movimientos más rápidos que el tiempo de correlación rotacional de la proteína, τR, pueden asociarse con la flexibilidad interna de la proteína. Debajo de la flecha se indican varios experimentos de RMN y su sensibilidad a las escalas de tiempo respectivas. Haga clic aquí para ver una versión más grande de esta figura.
El siguiente protocolo describe la configuración de los experimentos de relajación de RMN de Lakomek et al.69 y Stief et al.70, utilizando un esquema de detección de coherencia cuántica única (HSQC) heteronuclear con sensibilidad mejorada. Antes de proceder a la implementación experimental, se ofrece una breve descripción general de la relajación del espín de RMN y los experimentos de relajación de RMN. Debido a las restricciones de tamaño y para mantener la comprensión de este protocolo, esta descripción general debe seguir siendo simplista (y, por lo tanto, incompleta).
La relajación longitudinal o de espín-red, caracterizada por el tiempo T1 o la constante de velocidad R1 = 1/ T1 , describe el retorno de la magnetización al equilibrio de Boltzmann. En equilibrio, la magnetización se alinea a lo largo del eje del campo magnético exterior, que define el eje z del marco del laboratorio. Las densidades espectrales a frecuencias de Larmor altas (1H) y pequeñas (15N) (las frecuencias de resonancia de RMN, por ejemplo, 600 MHz para 1H para un imán de 14,1 Tesla) y las combinaciones lineales de estas frecuencias de Larmor contribuyen a la relajación de 15N R1 , que se caracteriza por las constantes de velocidad de 15N R1 medidas en rad·s-1. Los movimientos en escalas de tiempo son inversos a estas frecuencias de Larmor; por lo tanto, los movimientos en la escala de tiempo de picosegundos a nanosegundos contribuyen a la constante de tasa de relajación R1. Para las moléculas que muestran un volteo general y donde se puede definir un tiempo de correlación rotacional, la curva R1 (T1) muestra un máximo (mínimo) para ωτR = 1, con la correlación rotacional τR y la frecuencia de Larmor ω del espín bajo consideración. Si contribuyen varias frecuencias de Larmor, la que tiene la frecuencia más baja es la dominante, por ejemplo, ωN en el caso de 15N R1. El régimen de movimiento rápido (ωτR, mucho menor que 1) se aplica a moléculas pequeñas que giran muy rápido y para campos magnéticos bajos y baja viscosidad. El régimen de cámara lenta (ωτR mucho mayor que 1) es válido para moléculas más grandes que giran más lentamente y para campos magnéticos altos y alta viscosidad.
Las proteínas globulares plegadas muestran un volteo general en solución, y se puede asignar un tiempo de correlación rotacional. Sin embargo, el concepto de volteo general ya no es válido para proteínas intrínsecamente desordenadas y con frecuencia difiere de la asignación de un solo tiempo de correlación rotacional. Aquí, el tiempo de correlación interna específico del residuo se vuelve más crítico.
La secuencia de pulsos descrita que mide frecuencias de relajación de 15N R1 (Figura 2) se basa en un experimento de lectura HSQC con sensibilidad mejorada con una detección de eco/antieco para la detección en cuadratura69,70,71. Se utilizan gradientes cortos con fuerza y longitud variables para la selección de la coherencia y la mejora de la supresión del agua70. Durante ese tiempo, la polarización longitudinal de 15N se relajará. Los tiempos de decaimiento más largos conducen a intensidades reducidas en los planos 2D asociados de este espectro pseudo-3D (los puntos de datos de retardo se registran en la tercera dimensión). Un elemento de bucle, que se describe a continuación, se ejecuta un número creciente de veces para tiempos de relajación más largos. Dado que la relajación correlacionada entre la anisotropía de desplazamiento químico (CSA) de 15N y el acoplamiento dipolar de 1H y 15N (D) también está activa durante el retardo de relajación, es necesario un pulso central de 180° I-BURP-272, selectivo en los protones amida, para reenfocar la contribución mediante la relajación correlacionada cruzada (que si no se reenfoca conduciría a constantes de velocidad 15N R1 sesgadas y erróneas).
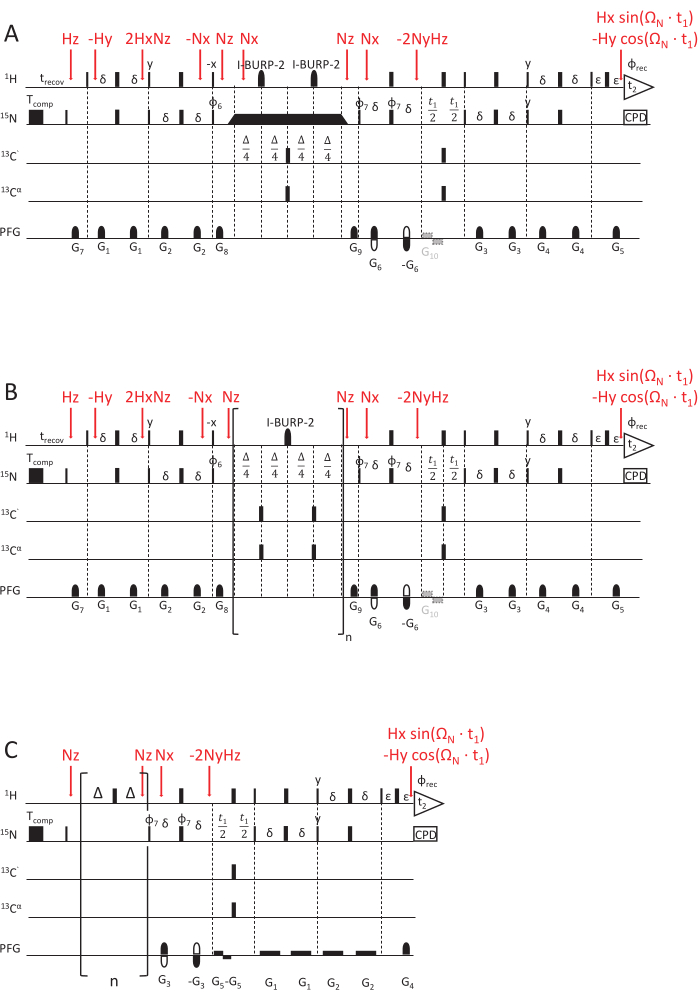
Figura 2: Esquemas de secuencia de pulsos de RMN para determinar las constantes de la tasa de relajación de RMN. (A) 15N R1ρ, (B) 15N R1 y (C) experimento hetNOE, utilizando un esquema de lectura HSQC de sensibilidad mejorada69,70. Los pulsos de 90°(x) se visualizan mediante rectángulos estrechos y los pulsos de 180°(x) mediante rectángulos anchos, a menos que se indique lo contrario. Se aplica el siguiente ciclo de fase: φ6 = y, y, -y, -y; φ7 = y, -y, φrec = y, -y, -y, y. La detección de cuadratura se logra invirtiendo la polaridad del gradiente G5 y el ciclo de fase de φ7 (detección de eco / anti-eco). (A) Experimento de 15N R1ρ: El rectángulo negro representa el espín-bloqueo, para el cual la duración varía hasta adquirir diferentes retrasos de relajación. Los triángulos antes y después del bloqueo de espín indican los pulsos de forma adiabática que alinean la magnetización a lo largo del eje del campo magnético efectivo Beff. G10 es un gradiente opcional para evitar la amortiguación de la radiación de la magnetización del agua durante la fase de evolución. (B) Experimento de 15N R1: La parte entre paréntesis muestra el elemento de bucle de la secuencia, repetido un número n de veces para que coincida con el retardo de relajación deseado. (C) El esquema de impulsos hetNOE es similar a la segunda mitad de los esquemas de impulsos R1 y R1ρ, es decir, el tiempo de evolución t1 y el elemento de detección HSQC. Sin embargo, la magnetización de 15N se excita directamente sin ningún INEPT. La saturación de la magnetización del protón (para lograr una relajación cruzada entre 1H y 15N) se logra mediante un tren de pulsos de 180 (1H) aplicados durante al menos 5 s. Para el experimento de referencia se aplica un retardo en reposo de las mismas longitudes (en este caso, 5 s) sin ningún tren de impulsos. G5 es un gradiente opcional para evitar la amortiguación de la radiación, y la inversión de la polaridad del gradiente G4, en combinación con la fase φ7 = y, -y, -y, y, logra la detección de cuadratura. Los pasos de transferencia de magnetización, representados por operadores de producto, están marcados en rojo. Haga clic aquí para ver una versión más grande de esta figura.
La constante de velocidad de relajación R2 describe la relajación de la polarización transversal (en el plano xy ortogonal al campo magnético externo) debido a la pérdida de coherencia de fase entre el espín, lo que conduce a la desintegración de la magnetización detectable53,54. La función de densidad espectral a frecuencias altas y pequeñas contribuye a R2, similar a R1. Sin embargo, la mayor contribución a R2 se origina en la densidad espectral a frecuencia cero. Por esta razón, R2 es muy sensible al volteo rotacional, descrito por el tiempo de correlación rotacional τR, que es del orden de unos pocos ns para una pequeña proteína globular a temperatura ambiente. Por lo tanto, los movimientos más lentos de la columna vertebral en el régimen de cientos de ps a ns bajos son los que más contribuyen. La dinámica de intercambio de la columna vertebral que provoca una modulación de la parte isótropa del tensor de desplazamiento químico de los núcleos 15N, en principio, añade una contribución de intercambio R2ex a las constantes de velocidad R2 43,44,49,60,61. En los experimentos descritos, la contribución de R2ex es suprimida por un bloqueo de espín que reenfoca la dinámica más lentamente que la frecuencia circular inversa del bloqueo de espín. El bloqueo de espín es un pulso de radiofrecuencia de onda continua larga que mantiene la magnetización alineada a lo largo del eje del campo magnético efectivo Beff (la suma vectorial del campo ω1 de bloqueo de espín y el desplazamiento químico de la frecuencia portadora de 15N (ver más abajo)). La relajación de la magnetización alineada a lo largo del eje B1,eff se denomina relajación R1ρ, que tiene un componente R1 y un componente R2. Usando la fórmula (1), R2 se puede calcular a partir de R1ρ y R144,73:
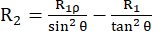 (1).
(1).
El ángulo entre el eje del campo magnético efectivo Beff y el campo magnético externo B0 es 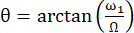 . ω1 es la amplitud de RF del bloqueo de espín y Ω el desplazamiento químico entre el desplazamiento químico de 15N del residuo correspondiente y la frecuencia portadora de 15N 44,73.
. ω1 es la amplitud de RF del bloqueo de espín y Ω el desplazamiento químico entre el desplazamiento químico de 15N del residuo correspondiente y la frecuencia portadora de 15N 44,73.
El esquema de impulsos R1ρ (Figura 2A, 70) es muy similar al esquema 15N R1 , excepto por el retardo de relajación. Para medir las tasas de relajación de 15N R1ρ , el bloqueo de espín debe estar activo después de que la magnetización se haya alineado a lo largo del eje de campo efectivo Beff mediante un pulso adiabático con la misma amplitud de radiofrecuencia (RF) que el bloqueo de espín. La duración del spin-lock se variará para obtener los diferentes retrasos de relajación.
El efecto Overhauser nuclear en estado estacionario {1H}-15N (1 H-15N NOE), denominado hetNOE en lo sucesivo, es la relación entre la tasa de relajación cruzada y la tasa de relajación longitudinal de 15N. Conduce a una reducción de la polarización de estado estacionario en 15N debido a la relajación cruzada con el protón tras la saturación de la polarización del protón 45,53,54,74,75. La relajación cruzada depende de las funciones de densidad espectral de la suma y diferencia de lasfrecuencias de Larmor 1 H y 15N. Por lo tanto, el hetNOE es sensible tanto a la dinámica de picosegundos rápidos (< 100 ps) como a la dinámica ps-ns (debido a su dependencia de R1). La secuencia69 (Figura 2C) se basa en una lectura HSQC de sensibilidad mejorada con gradientes Echo/Anti-Echo para la detección en cuadratura. Para la saturación de la magnetización de protones y el hetNOE resultante, la magnetización de protones de equilibrio se invierte y posteriormente se satura mediante pulsos de 180° que pulsan rápidamente durante aproximadamente 5 veces los 15 N T1. Para el experimento de referencia, el retardo de recuperación es igual al retardo de saturación, pero sin el tren de pulsos de 1H 180°. Se añade un retardo adicional de D1 = 2 s para el experimento de referencia y el que tiene una saturación de 1H. Ambos experimentos se registran de forma consecutiva y difieren solo en la aplicación de pulsos de 1H 180° (saturación) o no (referencia). La relación de las intensidades espectrales registradas en el experimento con una saturación de 1H dividida por las intensidades del experimento de referencia (sin el tren de impulsos de protones de 180°) da el valor de {1H}-15N NOE (hetNOE).
El siguiente protocolo describe la configuración de los experimentos de relajación por RMN de Lakomek et al.69 y Stief et al.70. Nos centramos en las secuencias de pulsos de RMN utilizando un esquema de detección HSQC de sensibilidad mejorada. Los experimentos 15N R1 y R1ρ se implementan como se describe en detalle por Stief et al.70, y el experimento hetNOE es descrito por Lakomek et al.69.
Access restricted. Please log in or start a trial to view this content.
Protocolo
1. Preparación de la muestra de RMN
NOTA: El marcaje de isótopos de las proteínas se realiza para experimentos de RMN de dimensiones superiores y RMN avanzada. Cuando la expresión de proteínas en E. coli y la purificación de proteínas se han establecido utilizando medios ricos (por ejemplo, Luria-Bertani [LB] o medio de triptona de extracto de levadura 2x [2YT]) con un rendimiento de varios miligramos por litro, la preparación de una muestra de RMN marcada isotópicamente suele ser relativamente sencilla.
- Para el marcaje de isótopos, utilice medios mínimos M9 para la expresión génica, donde el cloruro de amonio 15N es la única fuente de nitrógeno y la glucosa enriquecida con 13C es la única fuente de carbono.
- Para la purificación de proteínas, siga el protocolo estándar establecido en el laboratorio para la proteína respectiva.
NOTA: La sección de resultados muestra resultados representativos para un IDP, la proteína SNARE monomérica Sinaptobrevin-2 (1-96). El marcaje de isótopos de RMN de sinaptobrevina-2 se ha realizado como se describe en70. Para mediciones de muy alta precisión, se recomienda utilizar concentraciones bajas deD2O, inferiores al 5%. Esto se debe al efecto del isótopo de deuterio, que se puede observar en los experimentos de dispersión de relajación de Hahn-Echo 15N T2 y en los experimentos de dispersión de relajación de 15N CPMG de baja frecuencia (no discutidos aquí), pero tiene solo un impacto mínimo en los experimentos de 15N R1ρ (donde se reenfoca principalmente)76.
2. Preparativos para la ejecución de los experimentos de relajación de RMN en el espectrómetro
NOTA: Los experimentos de relajación de RMN descritos son específicos para los espectrómetros Bruker. Han sido probados en sondas de triple resonancia criogénicas y a temperatura ambiente de 1H, 15N y 13C y consolas Avance III y Avance Neo operadas por el software Bruker Topsin 3.6 o superior.
- Siga las precauciones.
- Haga una sesión informativa de seguridad antes de trabajar en el espectrómetro de RMN.
- Compruebe las especificaciones de potencia de la sonda del espectrómetro utilizado y elija una longitud de pulso y una potencia de pulso adecuadas para cada pulso, por ejemplo, comparando la consistencia con las entradas de la tabla "prosol" de Topspin.
- Importe las secuencias de pulsos.
- Descargue los programas de pulso de RMN desde el sitio web que se indica a continuación o desde el repositorio extendido del Banco de Resonancia Magnética Biológica (BMRB) (bmrbig102) en el directorio /opt/topspin4.1.4/exp/stan/nmr/lists/pp/user de la computadora que opera el espectrómetro de RMN. Ajuste la versión topspin (aquí: topspin4.1.4 a la versión respectiva instalada en el espectrómetro).
- Descargue los pulsos en forma de RMN desde el sitio web que se indica a continuación o desde el repositorio extendido del Banco de Resonancia Magnética Biológica (BMRB) (bmrbig102) en el directorio /opt/topspin4.1.4/exp/stan/nmr/lists/pp/user de la computadora que opera el espectrómetro de RMN. Ajuste la versión topspin (aquí: topspin 4.1.4 a la versión respectiva instalada en el espectrómetro).
- Calibrar el espectrómetro y la muestra.
- Establezca la temperatura de interés. Para la calibración de la temperatura, aplique los protocolos de la instalación. Por ejemplo, utilice una muestra de metanol-d4 e interprete la diferencia de desplazamiento químico observada utilizando el termómetro de RMN de la aplicación web (https://chem.ch.huji.ac.il/nmr/software/thermometer.html); Véase también77.
- Inserte la muestra (más un 5% adicional D2O para el bloqueo) y espere aproximadamente 10 minutos para permitir que la muestra equilibre su temperatura. Bloquee la señal de deuterio (2H) (comando: bloquear, seleccione H2O+D2O).
- Haga coincidir y sintonice los canales 1H, 15N y 13C, por ejemplo, utilizando la rutina atma o atmm. Si no hay instalada una unidad de ajuste y ajuste automático, haga coincidir y ajuste manualmente la sonda con el comando wobb.
- Realice el calzado para un campo B0 homogéneo, por ejemplo, utilizando la rutina topshim tg.
- Calibra la longitud del pulso de 90° (1H).
- Usa el experimento zg. Utilice getprosol para importar los parámetros de la sonda. Elija una duración corta de 5 μs para P1. Compruebe y corrija el nivel de potencia especificado de la sonda para PLW1 si es necesario.
- Comienza el experimento con zg. Procese los datos con efp. Ajuste la fase con .ph de modo que la línea de base es uniforme y la señal de RMN de la resonancia del agua está en fase.
- Estime un pulso de 360° (1H) tomando un pulso común o aproximado de 90° (1H) (por ejemplo, de las especificaciones del espectrómetro enumeradas) y multiplicándolo por un factor de cuatro. Ejecute el experimento y procéselo.
NOTA: La duración elegida es demasiado larga si la señal es positiva y demasiado corta si es negativa. El pulso de 360° (1H) será el paso por cero. - Ajuste la longitud del pulso en consecuencia. Hazlo de forma iterativa para encontrar el cruce por cero. Divide el valor obtenido por cuatro. Ese será el pulso de 90° (1H).
NOTA: Una alternativa al método 360° descrito es la rutina pulscal , que se puede iniciar desde la línea de comandos y permite una calibración automatizada de pulsos de 1H.
- Calibrar la frecuencia portadora O1 a la resonancia del agua.
- Copie el experimento zg escribiendo edc en la línea de comandos a un nuevo número de experimento. Escriba rpar para abrir los conjuntos de parámetros (Figura complementaria 1A).
- Busque el conjunto de parámetros ZGPR. Haga clic en el conjunto de parámetros ZGPR (Figura complementaria 1A), haga clic en Leer, elija Ejecutar "getprosol" y haga clic en Aceptar (Figura complementaria 1B).
- Cambie P1 a la longitud de pulso calibrada de 90° (1H). Elija una longitud de saturación de D1 = 2 s y una potencia de pulso de PLW9 de 50 dB. Ajuste la ganancia del receptor (rg) a 16. Alternativamente, ejecute el ajuste de ganancia del receptor escribiendo rga y ajustando la ganancia del receptor al valor especificado (pero evite el "desbordamiento de ganancia del receptor").
- Escriba gs en la línea de comandos para iniciar una adquisición repetida y continua. Vaya a la ventana de adquisición y cambie el O1 para reducir el área FID dominada por la señal de agua (Figura complementaria 2).
- Haga clic debajo del marcador para disminuir el O1 y haga clic encima del marcador para aumentarlo. Utilice el botón de sensibilidad para cambiar el tamaño del paso. Guarde el O1 actual; Close detiene la adquisición repetida (Figura complementaria 2).
- Calibra la longitud de pulso de 90° (15N).
- Copie el experimento zg en un nuevo número de experimento (escriba edc). Escriba rpar para leer el conjunto de parámetros HSQCFPF3GPPHWG para cambiar el programa de pulsos a un experimento HSQC de 1 H-15N. Utilice la opción Mantener los parámetros P1, O1 y PLW1 (figura complementaria 1).
- Escriba getprosol 1H 1H) en μs> <90° (1H) fuerza de pulso en db> en la línea de comandos para importar los parámetros de la sonda. Establezca el número de exploraciones (NS) en 8 y las exploraciones ficticias (DS) en 2.
NOTA: < > especifica aquí los valores que se van a establecer. Por ejemplo: getprosol 1H 8.5 -11.79. - Ejecute el primer incremento del experimento escribiendo zg en la línea de comandos. Copie el primer incremento en un nuevo número de experimento con rser 1 . Procese los datos con efp y phase el espectro con .ph. Utilice el primer incremento (experimento 1D) del experimento para los siguientes pasos.
NOTA: El < > especificar el número elegido para el nuevo experimento, por ejemplo, rser 1 101. - Escriba ased en la línea de comandos para abrir los parámetros de secuencia de pulsos del experimento 1D. Haga clic en la E en la línea PULPROG (ver Figura complementaria 3A) para abrir el editor de programas de pulsos.
- Utilice el siguiente flujo de trabajo para guardar la secuencia de pulsos con un nuevo nombre: haga clic en Archivo (esquina superior izquierda), Guardar como, introduzca el nuevo nombre y haga clic en Aceptar. A continuación, haga clic en Establecer PULPROG en el conjunto de datos (consulte la Figura complementaria 3B) y haga clic en Aceptar.
- Modifique la secuencia HSQC original de 1 H-15N (guardada con un nuevo nombre, véase 2.6.4.) multiplicando los 90° (15N) después del período de evolución t1 (antes de la transferencia inversa) por un factor de 2 (el pulso de 90° (15N) es ahora 180° (15N)). Presione Ctrl S para guardar la secuencia de pulsos. Ajuste la longitud del pulso a 90° (15N) (P21) para que la señal de RMN del primer incremento se desvanezca (en el cruce por cero) (Figura suplementaria 3B).
- Abra el experimento HSQC 1 H-15N (experimento 2D) e introduzca la longitud de pulso calibrada de 90° (15N) (P21). Tipo getprosol 1H 1H) en μs> <90° (1H) fuerza de pulso en db> 15N 15N) longitud de pulso en μs> <90° (15N) fuerza de pulso en db> en la línea de comandos.
- Determinar los anchos espectrales y la apropiación de los tiempos de adquisición.
- En la ventana de parámetros de adquisición para el experimento HSQC 1 H-15N (escriba eda en la línea de comandos), ajuste la anchura espectral (parámetro de adquisición SW) y el número de incrementos de dimensión en dimensiones directas e indirectas (parámetro de adquisición TD).
- Elija para SW, 16 ppm en la dimensión directa (1H) y 30 ppm en la indirecta (15N). Elija TD, 2048 en la dimensión directa (1H) y 512 en la dimensión indirecta (15N).
- Adquiere el experimento HSQC 1 H-15N.
- Compruebe que se detectan todas las señales de interés y que no hay artefactos de aliasing en el espectro. Cambie el O3P si el centro del espectro no encaja con el centro de la distribución de la señal de proteína a lo largo del eje 15N.
- Aumente el SW si la ventana espectral es demasiado pequeña. Aumente el TD para obtener una resolución FID más alta (consulte las especificaciones de la sonda para conocer la duración máxima del desacoplamiento).
NOTA: Esta es la configuración general y el punto de partida para la configuración de los experimentos de relajación de RMN. Se recomienda registrar el experimento HSQC 1 H-15N al principio y al final de todo el conjunto de experimentos y, para tiempos experimentales más prolongados, también en el medio para monitorear la integridad de la muestra.
- En la ventana de parámetros de adquisición para el experimento HSQC 1 H-15N (escriba eda en la línea de comandos), ajuste la anchura espectral (parámetro de adquisición SW) y el número de incrementos de dimensión en dimensiones directas e indirectas (parámetro de adquisición TD).
3. Implementación de los experimentos de relajación por RMN
NOTA: Las secuencias de pulsos de relajación de RMN (Figura 2) están disponibles en https://www.ipb.hhu.de/en/teams/team-lakomek/pulsesequences o en el repositorio ampliado del Banco de Resonancia Magnética Biológica (BMRB) (bmrbig102).
- Configura el archivo 15N R1ρ experimento.
- Copie el experimento HSQC (tipo edc) en un nuevo directorio. Escriba ased y cambie el programa de impulsos al experimento 15N R1ρ haciendo clic en los tres puntos de la línea de parámetros PULPROG en los parámetros de la secuencia de impulsos (véase la figura complementaria 3A). Haga doble clic en el programa de pulsos, haga clic en Establecer PULPROG en el conjunto de datos (consulte la Figura complementaria 3B) y haga clic en Aceptar.
NOTA: Pueden aparecer algunos mensajes de error. Cierre estos mensajes. - Escriba gpnam en la línea de comandos para insertar todas las formas de degradado que faltan (gpnam1- gpnam9) como se describe en la secuencia de pulsos (Figura complementaria 4).
- Escriba vplist en la línea de comandos. Haga clic en los tres puntos (Figura complementaria 5A), Archivo (esquina superior izquierda) y Nuevo (Figura complementaria 5B).
- Escriba un nombre para una nueva vplist ficticia y presione OK. Cree la vplist ficticia (5 m, 65 m, 25 m, 45 m, 15 m, 55 m, 35 m, 10 m; una nueva fila para cada entrada de vplist). Presione Ctrl S para guardar la lista y cerrar la ventana de la vplist (Figura complementaria 5C).
- Haga doble clic en el nombre de la vplist recién creada (Figura complementaria 5B).
NOTA: Esto es necesario para evitar errores de compilación en la secuencia de pulsos. Las entradas apropiadas de la vplist se determinarán más adelante (punto 3.1.29.). - Escriba ased para abrir los parámetros de la secuencia de pulsos (Figura complementaria 6). Introduzca todas las intensidades de gradiente y las longitudes de gradiente que faltan, tal como se indican en la secuencia de pulsos (Figura complementaria 6D).
- Introduzca para el contador de bucle L3 (número de puntos complejos en la dimensión indirecta) 1/2 de la entrada TD de 15N en la ventana de parámetros de adquisición (eda). Introduzca para el contador de bucles L6 el número de puntos de datos de relajación registrados utilizados para ajustar la curva de relajación.
NOTA: Este número L6 corresponde al número de entradas vplist, aquí: 8 (Figura complementaria 6A). - Establezca el pulso adiabático TanhTan_half.nl, descargado del sitio web especificado anteriormente, como la forma del pulso 15N SP8. Ajuste el pulso adiabático TanhTan_2nd.nl del sitio web como la forma del pulso 15N SP9 (Figura complementaria 6C).
- Ajuste la entrada P8 a la longitud del pulso TanhTan adiabático, lo suficientemente largo como para garantizar la adiabaticidad del pulso; aquí se elige P8 = 3000 μs (Figura suplementaria 6C).
- Establezca el desacoplamiento de 15N (CPDPRG 3) en un esquema de desacoplamiento garp. Utilícelo para la longitud de desacoplamiento PCPD3 200 μs (Figura complementaria 6C).
- Establezca el retardo de recuperación entre exploraciones en al menos D1 = 2 s o más.
NOTA: Para la elección de un retraso de recuperación adecuado, consulte la sección Discusión. - Establezca los escaneos ficticios (DS) en al menos 64 para el experimento final (durante la configuración, DS = 4 es suficiente por razones de tiempo). Como punto de partida, utilice un número de exploraciones (NS) de 4; utilice múltiplos de 4 si la relación señal-ruido (SNR) es demasiado baja (Figura suplementaria 6A).
- Ajuste el O1 a la frecuencia portadora calibrada del paso 2.5. Ajuste el O2P a 176 ppm y copie el O3P del experimento HSQC 1 H-15N (Figura suplementaria 6A-C).
- Ajuste la longitud de pulso P7 a la longitud de pulso de 90° (15N) calibrada en el paso 2.6. Además, copie el nivel de potencia del pulso de 90° (15N) del paso 2.6 a PLW3 y PLW7 (Figura complementaria 6C).
- Ajuste la longitud de pulso P1 y P19 a la longitud de pulso de 90° (1H) calculada en el paso 2.4. (Figura complementaria 6B).
- Establezca el número de incrementos en la dimensión indirecta TD = l3 * 2 * l6 (eda o escriba td en la línea de comandos).
NOTA: Todos los parámetros de la secuencia de pulsos descritos aquí se anotan adicionalmente en la secuencia de pulsos. - Establezca el pulso de forma SP5 en una forma I-BURP2 (Iburp2.1000). Ajuste la longitud del pulso P15 a 2000 μs. Abra la pantalla de la herramienta de forma haciendo clic en la E junto al pulso de forma I-BURP2 en la ventana de parámetros de secuencia de pulso Topspin (ased) (Figura complementaria 6B).
- Simule el pulso de forma con la longitud correcta haciendo clic en el botón Iniciar simulación de RMN. Compruebe la longitud del pulso con forma [μs] (= 2000 μs) y el ángulo de rotación [°] (= 180°) en la ventana de simulación. Haga clic en Iniciar RMN-SIM (Figura complementaria 7A).
- Verifique el rango de excitación y elija la longitud de pulso I-BURP2 adecuada para cubrir la dispersión espectral de 1H de la proteína, pero evite la excitación del agua al mismo tiempo. Establezca P15 en la longitud de pulso con forma de la ventana de simulación con el mejor pulso IBURP-2 simulado (Figura complementaria 7B).
NOTA: Se elige la longitud adecuada si el I-BURP2 excita (> un 95% de efectividad) toda la región amida. Para simular un pulso I-BURP2 con una longitud diferente, cierre la ventana del perfil de excitación y edite la longitud del pulso con forma en la ventana de simulación. Haga clic en Iniciar RMN-SIM. Los pulsos con una longitud más corta tienen un rango de excitación más amplio en Hz. - Ajuste SPOFFS5 para cambiar la frecuencia portadora del pulso I-BURP2 y desplazar el rango de excitación hacia la izquierda o la derecha para evitar que el pulso I-BURP2 toque y perturbe la magnetización del agua (Figura complementaria 6B).
- Para determinar el nivel de potencia adecuado del pulso con forma, abra la herramienta de forma Bruker, haga clic en el botón Iniciar simulación de RMN y establezca la longitud del pulso I-BURP2 en Longitud de pulso con forma [μs]. Recuerde la longitud correspondiente de un pulso rectangular suave de 90° (1H), visible en la ventana de simulación en el pulso cuadrado de 90° correspondiente [μs] (Figura suplementaria 7A).
- Escriba calcpowlev. Utilice el pulso duro de 90° (1H) (P1) en microsegundos como longitud de referencia (Figura complementaria 8B) y el pulso rectangular suave de 90° (1H) correspondiente al pulso I-BURP2 en microsegundos como la nueva longitud (Figura complementaria 8A). Recuerde la diferencia de potencia Δ en dB entre el pulso duro de 90° (1H) y el pulso rectangular suave (Figura 8C suplementaria). Copie la intensidad del pulso duro de 90° (1H) en dB a SPW5 y agregue la diferencia memorizada Δ en dB (Figura suplementaria 6B).
- Para determinar el nivel de potencia del bloqueo de giro, calcule la longitud de pulso de 90° (15N) correspondiente del bloqueo de giro. Utilice la fórmula: longitud de pulso de 90° (15N) en s = 0,25 / (potencia de bloqueo de espín en Hz).
- Utilice calcpowlev para calcular la diferencia de potencia Δ en dB entre la potencia del bloqueo de giro y el pulso duro de 90° (15N) (P7). Nuevo: Longitud de pulso de 90° (15N) del bloqueo de espín en μs (Figura suplementaria 8A). Ref: longitud de pulso dura de 90° (15N) en μs (Figura suplementaria 8B). Copie el nivel de potencia PLW7 del impulso duro de 90° (15N) en dB al nivel de potencia de bloqueo de espín PLW8 (Figura complementaria 6C) y agregue la diferencia de potencia calculada Δ en dB (Figura complementaria 8C).
- Copie el nivel de potencia del spin-lock PLW8 a los niveles de potencia SPW8 y SPW9. (Figura complementaria 6C).
- Para determinar el nivel de potencia del desacoplamiento de 15N, utilice calcpowlev para calcular la diferencia de potencia Δ entre la potencia del pulso de desacoplamiento de 90° (15N) (PCPD3) y el pulso duro de 90° (15N) (P7). Nuevo: PCPD3 en μs (200) (Figura suplementaria 8A). Ref: longitud de pulso dura de 90° (15N) en μs (Figura suplementaria 8B). Copie el nivel de potencia PLW7 del impulso duro de 90° (15N) en dB al nivel de desacoplamiento PLW31 (Figura complementaria 6C) y añada Δ en dB (Figura suplementaria 8C).
- Para utilizar la compensación de temperatura, incluya #define TEMP_COMPENSATION (eliminando el punto y coma en la secuencia de pulsos). Establezca P18 en la duración máxima del bloqueo de espín utilizado en el experimento 15N R1ρ .
NOTA: Se recomienda desactivar la compensación de temperatura para la primera prueba del experimento. - En el caso de muestras marcadas con 15N 13C, incluya # para definir LABEL_CN en el programa de pulsos (eliminando el punto y coma en la secuencia de pulsos). Ajuste P4 a la longitud de pulso calculada con la fórmula de78. Por ejemplo, corresponde a 23,70 μs a 600 MHz y 11,85 μs a 1200 MHz. Escriba calcpowlev para calcular la diferencia de potencia en dB entre P4 y el pulso duro (13C). Copie la potencia de pulso duro (13C) en dB a PLW4 y agregue la Δ en dB. Copie PLW4 a PLW2 (Figura complementaria 6B).
- Determine los retrasos de relajación para un muestreo adecuado.
- Ejecute los primeros ocho FID (en caso L6 = 8) y procéselos (rser < nuevo número de experimento>). Elija las entradas en la vplist en las que la intensidad máxima del experimento con el retraso más largo disminuya a al menos 1/e (aprox. 37%), pero no menos del 25% en comparación con el experimento con el retraso más corto (que sirve como referencia).
- Ajuste las entradas restantes de la vplist para lograr un muestreo uniforme de las disminuciones de intensidad entre el FID con el retraso más largo y el FID con el retraso más corto. Lea la NOTA y las especificaciones de potencia de la sonda.
NOTA: Los residuos que se relajan lentamente dominarán el primer FID. Ajuste las entradas restantes de la vplist para lograr un muestreo uniforme de las disminuciones de intensidad entre el primer y el segundo FID. No exceda la potencia y longitud máximas permitidas de bloqueo de giro de la sonda (consulte el manual de la sonda y las especificaciones de la sonda). En caso de compensación de temperatura, verifique P18 (duración máxima de bloqueo de giro), ya que se aplicará la duración total de bloqueo de giro de P18, dividida entre el retardo de relajación y la compensación de temperatura. Contiene tiempo (retraso de relajación) + tiempo (compensación de temperatura) = const = P18. Para un bloqueo de espín de 2 kHz, preferimos no exceder una duración de espín de más de 65 ms, utilizando un retardo de recuperación (D1) de 2 s. En ese caso, se debe tomar un compromiso entre la duración máxima del bloqueo de giro y el retraso de relajación más largo a favor de la duración máxima permitida del bloqueo de giro. En caso de retrasos de recuperación D1 más largos, pueden ser aplicables potencias de bloqueo de giro más largas. Lo que cuenta es la deposición general de potencia en la sonda a lo largo del tiempo. Si se requieren duraciones de bloqueo de giro más largas, solo aumente las longitudes de bloqueo de giro en pequeños pasos y observe la señal de bloqueo. Como regla general, ajuste el nivel de bloqueo en el tercio superior de la pantalla de bloqueo. Si el nivel de bloqueo se reduce en más de dos cuadrados durante la ejecución de la secuencia de pulsos, la potencia de bloqueo de espín depositada en la sonda es demasiado.
- Escriba rga en la línea de comandos para determinar la ganancia del receptor. Para una buena relación señal-ruido, apunte a un valor de ganancia del receptor (rg) entre 128 y 256, lo que demuestra una buena supresión de agua.
NOTA: No se puede alcanzar una ganancia del receptor entre 128 y 256 indica una señal de agua demasiado fuerte. En ese caso, la potencia y las duraciones del gradiente se ajustan suavemente hasta que se alcanza la ganancia del receptor. Véase también la sección de Discusión. - Inicie una ejecución de prueba del experimento 15N R1ρ escribiendo zg en la línea de comandos. Compruebe que la señal de agua esté bien suprimida para todos los retrasos (de 1 a 8, en caso de 8 retrasos en la vplist). Marque también el incremento 9, que es el segundo incremento del esquema de detección de cuadratura (Echo/Anti-Echo).
- Copie el experimento HSQC (tipo edc) en un nuevo directorio. Escriba ased y cambie el programa de impulsos al experimento 15N R1ρ haciendo clic en los tres puntos de la línea de parámetros PULPROG en los parámetros de la secuencia de impulsos (véase la figura complementaria 3A). Haga doble clic en el programa de pulsos, haga clic en Establecer PULPROG en el conjunto de datos (consulte la Figura complementaria 3B) y haga clic en Aceptar.
- Configure el experimento 15N R1 .
- Copie el experimento de 15N R1ρ y cambie el programa de impulsos al experimento de 15N R1 .
- Escriba vclist, genere una vclist y escriba la vclist ficticia descrita en70: 0, 24, 6, 20, 4, 16, 8, 12; una nueva fila para cada entrada vclist).
- Ased abierto. Compruebe las longitudes y las intensidades de los gradientes. Cambie GPZ9 a 14%.
- Compruebe las longitudes de los pulsos, las formas de los pulsos y las intensidades de los pulsos (los valores correctos deberían haberse copiado del experimento 15NR 1ρ ).
- En el caso de muestras marcadas con 15N13C, utilice #define LABEL_CN en el programa de pulsos.
- Para el uso de la compensación de temperatura, incluya #define TEMP_COMPENSATION.
- Ejecute los primeros ocho FID (en caso de L6 = 8) y procéselos (rser < nuevo número de experimento>). Elija las entradas en el vclist en las que la intensidad máxima del experimento con el retardo más largo disminuya a al menos 1/e (aprox. 37%) pero no menos del 25% en comparación con el experimento con el retardo más corto. Ajuste las entradas restantes de la vplist para lograr un muestreo uniforme de las disminuciones de intensidad entre el FID con el retraso más largo y el FID con el retraso más corto.
NOTA: Solo se permiten números pares para las entradas vclist.
- Configure el experimento {1H}-15N NOE.
- Copie el experimento de relajación 15N R1 y cambie la secuencia de pulsos al esquema de pulsos {1H}-15N NOE.
- Ajuste la duración del período de saturación con el contador de bucles L8. El espaciado entre pulsos de 180° (1H) es de 22 ms79 (2* 1 / JNH). Para lograr los resultados más precisos, utilice un período de saturación de aproximadamente 5 veces el 15N T1 (15N) de la proteína. Aquí: 5 s. Eso correspondería a L8 = 230.
NOTA: La sección de discusión proporciona consejos adicionales sobre cómo elegir el período de saturación/retraso de recuperación. - Cambie la longitud y la intensidad de los gradientes como se describe en el esquema de pulsos. Utilice formas de degradado sinusoidal.20.
- Establezca el número de incrementos en la dimensión indirecta igual a TD = l3 * 2 * 2
- En el caso de muestras marcadas con 15N13C, utilice #define LABEL_CN y -DLABEL_CN como ZGOPTNS.
- Ejecute los esquemas de pulso escribiendo zg en la línea de comandos.
4. Procesamiento y análisis de los experimentos de RMN registrados
NOTA: Los espectros se han registrado utilizando un sistema Bruker. El procesamiento se realiza mediante un sistema operativo Unix o Linux. El procesamiento de los espectros y el análisis de los datos se realizaron utilizando NMRPipe80 y python3. El software NMRPipe se puede descargar en https://www.ibbr.umd.edu/nmrpipe/index.html. Los scripts de procesamiento basados en NMRPipe se descargan del sitio web: https://www.ipb.hhu.de/en/teams/team-lakomek/pulsesequences o del repositorio ampliado del Banco de Resonancia Magnética Biológica (BMRB) (bmrbig102). Se recomienda el uso de NMRPipe. Si NMRPipe no está disponible o no se desea, se pueden utilizar alternativas como CCPN81 o SPARKY (SPARKY 3, o sus sucesores NMRFAM-SPARKY82 o POKY83 ).
- Procesar y analizar 15N R1 experimento.
NOTA: Los siguientes comandos se ejecutan en un terminal Unix.- Copie los archivos del directorio descargado R1_processing en el directorio que contiene los datos experimentales R1 (directorio de trabajo).
- Haga que los archivos sean ejecutables mediante el comando de terminal: > chmod u+x *.com
- Ejecute > bruker para abrir la utilidad de conversión NMRPipe y generar el archivo fid.com. Haga clic en Leer parámetros. Cambie el Recuento de dimensiones a dos si se cuentan tres dimensiones y vuelva a hacer clic en Leer parámetros . A continuación, haga clic en Borrar secuencia de comandos, Actualizar secuencia de comandos y Guardar secuencia de comandos (Figura complementaria 9).
NOTA: Si la frecuencia portadora no es correcta, cambie las PPM de la posición central después de hacer clic en Leer parámetros por segunda vez. - Ejecutar > ./fid.com
- Convierta la vclist en una vplist con entradas de ms: retraso en ms = 40 ms * entrada de vclist. Utilice > cp vclist vplist y edite la vplist.
- Utilice > ./split.com para dividir los datos pseudo 3D en ocho archivos 2D.
- Edite los valores tauValues (valores vplist en el orden de la vplist) y los valores idxExpmt (orden de los valores tauValues con respecto a la hora) en nmrproc.com (Figura complementaria 10).
- Utilice > ./nmrproc.com para procesar los datos sin procesar de los ocho espectros 2D.
- Utilice > nmrDraw para dibujar los espectros.
- Utilice el espectro de referencia (primera entrada en vplist) para estos pasos. Cambie a ese espectro cambiando la dimensión z a uno (esquina superior izquierda). Ajuste el nivel de contorno usando los botones + y - junto al botón Factor . Vuelva a dibujar el espectro con el botón Dibujar.
- Utilice un nivel de contorno en el que se puedan reconocer todas las señales de interés con solo unas pocas señales provenientes del ruido (el nivel de contorno está en el borde del nivel de ruido) (Figura complementaria 11).
- Corrija la fase del espectro, tipo v para la proyección vertical de la señal 1D y h para la proyección horizontal de la señal 1D. Ajuste la fase con P0 y P1 (Figura complementaria 11), recuerde la diferencia de fase y corrija la fase en el archivo nmrproc.com (Figura complementaria 10).
NOTA: Si el espectro se invierte en la dimensión 15N, elimine el -neg en la línea: | nmrPipe -fn FT -neg \ (Figura suplementaria 10). - Cierre nmrDraw y vuelva a procesar con > nmrproc.com. Para la selección de picos (después de que el espectro se haya faseado correctamente y el espectro de referencia se muestre con un nivel de contorno en el que todos los picos estén presentes), escriba K (shift + k), presione Detectar y haga clic en Guardar. (Figura complementaria 11).
- Copie una lista de picos en el directorio de trabajo y cambie -assName en ass.com por el nombre de la lista de picos (Figura complementaria 12).
NOTA: Un ejemplo de una lista de picos en el formato correcto está disponible en https://www.ipb.hhu.de/en/teams/team-lakomek/pulsesequences
y en el repositorio ampliado del Banco de Resonancia Magnética Biológica (BMRB) (bmrbig102). - Ejecute > ./ass.com para asignar los espectros. Compruebe la asignación que sugiere el script para los distintos picos. Utilice el botón izquierdo del ratón para asignar y el botón derecho del ratón para desasignar picos. Utilice el botón Siguiente para ir al siguiente aminoácido y Anterior para ir al aminoácido anterior (Figura complementaria 13).
NOTA: El nivel de contorno se puede cambiar con + y - en la esquina superior derecha. Utilice el botón Dibujar para volver a dibujar el espectro con el nuevo nivel de contorno (Figura complementaria 13). - Después de completar la tarea, haga clic en Guardar y, a continuación, en Guardar de nuevo. A continuación, haga clic en Salir, Cancelar y Salir (Figura complementaria 13).
- Ejecute > python3 NoneDeleter.py para crear relax.tab, que contiene solo información sobre los picos asignados.
- Ejecute > ./relax.com para generar el autofit.com script.
- Ejecute > ./autoFit.com para realizar una interpolación de Fourier y generar pseudo proporciones de los diferentes tiempos de retardo para cada aminoácido (se generan axt.tab y nlin.tab ).
- Ejecute > ./model.com para generar el script de modelExp.com basado en los datos de nlin.tab.
- Ejecute > ./modelExp.com para ajustar la curva de evolución de cada residuo con un modelo de optimización de mínimos cuadrados no lineal.
- Ejecute > bash summary.tcl -in relax.tab > t1.tab. Ignore un posible mensaje de error: GDB Aviso: Pueden aparecer 21 valores faltantes en el archivo de tabla .
- Copie t1.tab en un directorio R1R2 recién creado.
- Procese y analice el experimento 15N R1ρ .
- Copie los archivos del directorio descargado R1rho_processing en el directorio que contiene los datos experimentales de R1ρ (directorio de trabajo).
- Ejecute > chmod u+x *.com
- Ejecute > bruker para abrir la utilidad de conversión NMRPipe y generar el archivo fid.com. Véase también el procesamiento de los espectros 15N R1 .
- Ejecutar > ./fid.com
- Ejecute > ./split.com para dividir los datos pseudo 3D en ocho archivos 2D.
- Ejecute > ./nmrproc.com (edite los tauValues [valores vplist] y el idxExpmt de antemano) para procesar los datos sin procesar de los ocho espectros 2D (Figura complementaria 10).
- Ejecute > nmrDraw para dibujar los espectros y elegir los picos. Utilice el espectro de referencia (primera entrada en vplist) para estos pasos. Siga el mismo procedimiento que para el experimento 15N R1 .
- Copie una lista de picos en el directorio de trabajo y cambie -assName en ass.com por el nombre de la lista de picos (Figura complementaria 12).
- Ejecute > ./ass.com y siga el mismo procedimiento que para el experimento 15N R1 .
- Ejecute > python3 NoneDeleter.py para crear relax.tab, que contiene solo información sobre los picos asignados.
- Ejecute > ./relax.com
- Ejecutar > ./autoFit.com
- Ejecute > ./model.com
- Ejecute > ./modelExp.com
- Ejecute > bash summary.tcl -in relax.tab > t1rho. tab. Ignore el posible mensaje de error: GDB Aviso: Es posible que falten 21 valores en el archivo de tabla .
- Copie el siguiente encabezado en el archivo t1rho.tab (los valores se pueden obtener del archivo fid.com) (Figura complementaria 14):
OBSERVACIÓN: 600MHz para T1rho
OBSERVACIÓN -yOBS = 60.818 (ponga aquí el valor encontrado en fid.com)
OBSERVACIÓN -yCAR = 119.06 (ponga aquí el valor que se encuentra en fid.com) - Ejecute > ./extract_PPM.com para generar una tabla denominada fusionOffset.tab que contenga el desplazamiento de la frecuencia máxima de 15N en relación con la frecuencia portadora (que será necesaria para calcular el ángulo θ para la extracción de datos R2 a partir de los datos R1 y R1ρ ).
- Resumir los datos de relajación.
- Copie el t1rho.tab y el fusionOffset.tab en el directorio R1R2.
- Copie el script crtR1p.tcl en el directorio R1R2, ajuste la frecuencia del espectrómetro y la potencia de bloqueo de espín, y ejecute bash crtR1p.tcl | sort -n > R1R2.tab. (Figura complementaria 15). R1R2.tab contendrá las constantes de velocidad R1ρ ajustadas, el ángulo theta, las constantes de velocidad R2 y las constantes de velocidad R1 .
- Procese y analice el experimento hetNOE.
- Copie los archivos del directorio descargado NOE_processing en el directorio de datos experimental NOE (directorio de trabajo).
- Execute > chmod u+x *.com
- Ejecute > bruker para abrir la utilidad de conversión NMRPipe y generar el archivo fid.com. Haga clic en Leer parámetros. Cambie el Recuento de dimensiones a dos si se contaron tres dimensiones y vuelva a pulsar Leer parámetros . A continuación , pulse Borrar guión, Actualizar guión y Guardar guión.
- Ejecutar > ./fid.com
- Ejecute > ./split.com (Una vplist con dos entradas (0, 1) debe crearse antes)
- Ejecute > ./ft2.com
- Ejecute > nmrDraw para dibujar el espectro y seleccionar los picos en el espectro de referencia (dimensión z = 1). Extraiga y recuerde el ruido (presione T = shift + t). La corrección de fase y la selección de picos siguen el mismo procedimiento que el experimento 15N R1 .
- Copie una lista de picos en el directorio de trabajo y cambie -assName en ass.com por el nombre de la lista de picos (Figura complementaria 12)
- Ejecute > ./ass.com para asignar los espectros. Siga el mismo procedimiento que se detalla para el experimento 15N R1 .
- Ejecute > python3 NoneDeleter.py para crear relax.tab.
- Ejecutar > ./script.com
- Ejecute > ./autoFit.com
- Cambia el ruido en error_hetnoe.tcl.
- Execute > bash error_hetnoe.tcl |sort -n > hetNOE.tab
Access restricted. Please log in or start a trial to view this content.
Resultados
A continuación se muestran algunos datos ejemplares de relajación de RMN registrados en la proteína SNARE vesicular Sinaptobrevin-2 (1-96), frecuentemente llamada VAMP2 (proteína 2 asociada a vesículas). Para registrar los datos de RMN, utilizamos una muestra de 171 μM 15N Synaptobrevin-2 (1-96) (denominada Syb-2 a continuación) en un tampón MES (pH 6,0) de 50 mM que contenía 150 mM de NaCl, 0,1 mM de TCEP y 1 mM de EDTA. Todos los datos experimentales se registraron ...
Access restricted. Please log in or start a trial to view this content.
Discusión
Este protocolo describió la configuración de los experimentos de relajación de RMN 15N por Lakomek et al.69 y Stief et al.70. Nos centramos en las secuencias de pulsos de RMN utilizando un esquema de detección HSQC de sensibilidad mejorada. Los experimentos 15N R1 y R1ρ se implementan como se describe en detalle por Stief et al.70, y el experimento hetNOE es descrito por Lakomek et
Access restricted. Please log in or start a trial to view this content.
Divulgaciones
Los autores declaran que no tienen intereses financieros o relaciones personales contrapuestas que puedan haber influido en el trabajo reportado en este artículo.
Agradecimientos
Agradecemos a Melinda Jaspert y Kevin Bochinsky por las útiles discusiones. N.L. agradece a la Fundación Alemana de la Ciencia por la financiación a través del Programa Heisenberg (subvención DFG número 433700474). Este trabajo está respaldado por el proyecto "Determinantes virológicos e inmunológicos de la patogénesis de la COVID-19: lecciones para prepararse para futuras pandemias (KA1-Co-02 "COVIPA"), una subvención del Fondo de Iniciativas y Redes de la Asociación Helmholtz. Agradecemos el generoso acceso al Centro de RMN Biomolecular de Jülich-Düsseldorf, dirigido conjuntamente por Forschungszentrum Jülich y la Universidad Heinrich Heine de Düsseldorf (HHU).
Access restricted. Please log in or start a trial to view this content.
Materiales
| Name | Company | Catalog Number | Comments |
| Bruker 600 MHz AVANCE III HD spectrometer | Bruker | https://www.bruker.com/en/products-and-solutions/mr/nmr/avance-nmr-spectrometer.html | NMR experiments conducted |
Referencias
- Sekhar, A., Kay, L. E. An NMR view of protein dynamics in health and disease. Annu Rev Biophys. 48, 297-319 (2019).
- Rosenzweig, R., Kay, L. E. Bringing dynamic molecular machines into focus by methyl-TROSY NMR. Annu Rev Biochem. 83, 291-315 (2014).
- Palmer, A. G. 3rd Enzyme dynamics from NMR spectroscopy. Acc Chem Res. 48 (2), 457-465 (2015).
- Reif, B. Ultra-high resolution in MAS solid-state NMR of perdeuterated proteins: implications for structure and dynamics. J Magn Reson. 216, 1-12 (2012).
- Schanda, P., Ernst, M. Studying dynamics by magic-angle spinning solid-state NMR spectroscopy: Principles and applications to biomolecules. Prog Nucl Magn Reson Spectrosc. 96, 1-46 (2016).
- Le Marchand, T., et al. (1)H-H-detected biomolecular NMR under fast magic-angle spinning. Chem Rev. 122 (10), 9943-10018 (2022).
- Pervushin, K., Riek, R., Wider, G., Wuthrich, K. Attenuated T2 relaxation by mutual cancellation of dipole-dipole coupling and chemical shift anisotropy indicates an avenue to NMR structures of very large biological macromolecules in solution. Proc Natl Acad Sci U S A. 94 (23), 12366-12371 (1997).
- Tzakos, A. G., Grace, C. R., Lukavsky, P. J., Riek, R. NMR techniques for very large proteins and RNAs in solution. Annu Rev Biophys Biomol Struct. 35, 319-342 (2006).
- Sprangers, R., Kay, L. E. Quantitative dynamics and binding studies of the 20S proteasome by NMR. Nature. 445 (7128), 618-622 (2007).
- Wright, P. E., Dyson, H. J. Intrinsically disordered proteins in cellular signalling and regulation. Nat Rev Mol Cell Biol. 16 (1), 18-29 (2015).
- van der Lee, R., et al. Classification of intrinsically disordered regions and proteins. Chem Rev. 114 (13), 6589-6631 (2014).
- Tompa, P. Intrinsically unstructured proteins. Trends Biochem Sci. 27 (10), 527-533 (2002).
- Dyson, H. J., Wright, P. E. Coupling of folding and binding for unstructured proteins. Curr Opin Struct Biol. 12 (1), 54-60 (2002).
- Uversky, V. N. Intrinsically disordered proteins in overcrowded milieu: Membrane-less organelles, phase separation, and intrinsic disorder. Current Opinion in Structural Biology. 44, 18-30 (2017).
- Patel, A., et al. A liquid-to-solid phase transition of the ALS protein FUS accelerated by disease mutation. Cell. 162 (5), 1066-1077 (2015).
- Abyzov, A., Blackledge, M., Zweckstetter, M. Conformational dynamics of intrinsically disordered proteins regulate biomolecular condensate chemistry. Chem Rev. 122 (6), 6719-6748 (2022).
- Uversky, V. N. Unusual biophysics of intrinsically disordered proteins. Biochim Biophys Acta. 1834 (5), 932-951 (2013).
- Papoian, G. A. Proteins with weakly funneled energy landscapes challenge the classical structure-function paradigm. Proc Natl Acad Sci U S A. 105 (38), 14237-14238 (2008).
- Uversky, V. N., Gillespie, J. R., Fink, A. L. Why are "natively unfolded" proteins unstructured under physiologic conditions. Proteins. 41 (3), 415-427 (2000).
- Bah, A., et al. Folding of an intrinsically disordered protein by phosphorylation as a regulatory switch. Nature. 519 (7541), 106-109 (2015).
- Tompa, P., Schad, E., Tantos, A., Kalmar, L. Intrinsically disordered proteins: emerging interaction specialists. Curr Opin Struct Biol. 35, 49-59 (2015).
- Dyson, H. J., Wright, P. E. Perspective: the essential role of NMR in the discovery and characterization of intrinsically disordered proteins. J Biomol NMR. 73 (12), 651-659 (2019).
- Adamski, W., et al. A unified description of intrinsically disordered protein dynamics under physiological conditions using NMR spectroscopy. J Am Chem Soc. 141 (44), 17817-17829 (2019).
- Dobson, C. M. Protein folding and misfolding. Nature. 426 (6968), 884-890 (2003).
- Bertoncini, C. W., et al. Release of long-range tertiary interactions potentiates aggregation of natively unstructured alpha-synuclein. Proc Natl Acad Sci U S A. 102 (5), 1430-1435 (2005).
- Buell, A. K., et al. Solution conditions determine the relative importance of nucleation and growth processes in alpha-synuclein aggregation. Proc Natl Acad Sci U S A. 111 (21), 7671-7676 (2014).
- Salvi, N., Abyzov, A., Blackledge, M. Atomic resolution conformational dynamics of intrinsically disordered proteins from NMR spin relaxation. Prog Nucl Magn Reson Spectrosc. 102, 43-60 (2017).
- Schneider, R., Blackledge, M., Jensen, M. R. Elucidating binding mechanisms and dynamics of intrinsically disordered protein complexes using NMR spectroscopy. Curr Opin Struct Biol. 54, 10-18 (2019).
- Rezaei-Ghaleh, N., Blackledge, M., Zweckstetter, M. Intrinsically disordered proteins: from sequence and conformational properties toward drug discovery. Chembiochem. 13 (7), 930-950 (2012).
- Jensen, M. R., Salmon, L., Nodet, G., Blackledge, M. Defining conformational ensembles of intrinsically disordered and partially folded proteins directly from chemical shifts. J Am Chem Soc. 132 (4), 1270-1272 (2010).
- Camacho-Zarco, A. R., et al. NMR provides unique insight into the functional dynamics and interactions of intrinsically disordered proteins. Chem Rev. 122 (10), 9331-9356 (2022).
- Mittag, T., Forman-Kay, J. D. Atomic-level characterization of disordered protein ensembles. Curr Opin Struct Biol. 17 (1), 3-14 (2007).
- Dyson, H. J., Wright, P. E. Unfolded proteins and protein folding studied by NMR. Chem Rev. 104 (8), 3607-3622 (2004).
- Dyson, H. J., Wright, P. E. NMR illuminates intrinsic disorder. Curr Opin Struct Biol. 70, 44-52 (2021).
- Sugase, K., Dyson, H. J., Wright, P. E. Mechanism of coupled folding and binding of an intrinsically disordered protein. Nature. 447 (7147), 1021-1025 (2007).
- Bessa, L. M., et al. The intrinsically disordered SARS-CoV-2 nucleoprotein in dynamic complex with its viral partner nsp3a. Sci Adv. 8 (3), eabm4034(2022).
- Milles, S., Salvi, N., Blackledge, M., Jensen, M. R. Characterization of intrinsically disordered proteins and their dynamic complexes: From in vitro to cell-like environments. Prog Nucl Magn Reson Spectrosc. 109, 79-100 (2018).
- Salvi, N., Abyzov, A., Blackledge, M. Atomic resolution conformational dynamics of intrinsically disordered proteins from NMR spin relaxation. Prog Nucl Magn Reson Spectrosc. 102 - 103, 43-60 (2017).
- Gill, M. L., Byrd, R. A., Palmer, A. G. III Dynamics of GCN4 facilitate DNA interaction: a model-free analysis of an intrinsically disordered region. Phys Chem Chem Phys. 18 (8), 5839-5849 (2016).
- Bertoncini, C. W., et al. Structural characterization of the intrinsically unfolded protein beta-synuclein, a natural negative regulator of alpha-synuclein aggregation. J Mol Biol. 372 (3), 708-722 (2007).
- Rezaei-Ghaleh, N., et al. Local and global dynamics in intrinsically disordered synuclein. Angew Chem Int Ed Engl. 57 (46), 15262-15266 (2018).
- Rezaei-Ghaleh, N., Parigi, G., Zweckstetter, M. Reorientational dynamics of amyloid-beta from NMR spin relaxation and molecular simulation. J Phys Chem Lett. 10 (12), 3369-3375 (2019).
- Palmer, A. G. NMR characterization of the dynamics of biomacromolecules. Chem Rev. 104 (8), 3623-3640 (2004).
- Palmer, A. G., Massi, F. Characterization of the dynamics of biomacromolecules using rotating-frame spin relaxation NMR spectroscopy. Chem Rev. 106 (5), 1700-1719 (2006).
- Kay, L. E., Torchia, D. A., Bax, A. Backbone dynamics of proteins as studied by 15N inverse detected heteronuclear NMR spectroscopy: application to staphylococcal nuclease. Biochemistry. 28 (23), 8972-8979 (1989).
- Mittermaier, A., Kay, L. E. Review - New tools provide new insights in NMR studies of protein dynamics. Science. 312 (5771), 224-228 (2006).
- Abragam, A. Principles of Nuclear Magnetism. , Clarendon Press. Oxford. (1983).
- Ernst, R. R., Bodenhausen, G., Wokaun, A. Principles of Nuclear Magnetic Resonance in One and Two Dimensions. , Oxford University Press. Oxford. (1990).
- Cavanagh, J., Fairbrother, W. J., Palmer, A. G. III, Skelton, N. J. Protein NMR Spectroscopy: Principles and Practice. , Elsevier, Academic Press. (1995).
- Levitt, M. H. Spin Dynamics: Basics of Nuclear Magnetic Resonance. , John Wiley and Sons. (2008).
- Keeler, J. Understanding NMR Spectroscopy. , John Wiley and Sons. (2010).
- Bloembergen, N., Purcell, E. M., Pound, R. V. Relaxation effects in nuclear magnetic resonance absorption. Phys Rev. 73 (7), 679-712 (1948).
- Wangsness, R. K., Bloch, F. The dynamical theory of nuclear induction. Phys Rev. 89 (4), 728-739 (1953).
- Redfield, A. G. On the theory of relaxation processes. IBM J Res Dev. 1 (1), 19-31 (1957).
- Peng, J. W., Wagner, G. Mapping of the spectral densities of N-H bond motions in eglin c using heteronuclear relaxation experiments. Biochemistry. 31 (36), 8571-8586 (1992).
- Farrow, N. A., et al. Backbone dynamics of a free and a phosphopeptide-complexed src homology-2 domain studied by n-15 NMR relaxation. Biochemistry. 33 (19), 5984-6003 (1994).
- Tolman, J. R., Ruan, K. NMR residual dipolar couplings as probes of biomolecular dynamics. Chem Rev. 106 (5), 1720-1736 (2006).
- Mulder, F. A. A., Skrynnikov, N. R., Hon, B., Dahlquist, F. W., Kay, L. E. Measurement of slow (mu s-ms) time scale dynamics in protein side chains by N-15 relaxation dispersion NMR spectroscopy: Application to Asn and Gln residues in a cavity mutant of T4 lysozyme. J Am Chem Soc. 123 (5), 967-975 (2001).
- Tollinger, M., Skrynnikov, N. R., Mulder, F. A. A., Forman-Kay, J. D., Kay, L. E. Slow dynamics in folded and unfolded states of an SH3 domain. J Am Chem Soc. 123 (46), 11341-11352 (2001).
- Akke, M., Palmer, A. G. Monitoring macromolecular motions on microsecond to millisecond time scales by R(1)rho-R(1) constant relaxation time NMR spectroscopy. J Am Chem Soc. 118 (4), 911-912 (1996).
- Loria, J. P., Rance, M., Palmer, A. G. A relaxation-compensated Carr-Purcell-Meiboom-Gill sequence for characterizing chemical exchange by NMR spectroscopy. J Am Chem Soc. 121 (10), 2331-2332 (1999).
- Vallurupalli, P., Bouvignies, G., Kay, L. E. Studying "Invisible" excited protein states in slow exchange with a major state conformation. J Am Chem Soc. 134 (19), 8148-8161 (2012).
- Jeener, J., Meier, B. H., Bachmann, P., Ernst, R. R. Investigation of exchange processes by 2-dimensional NMR-spectroscopy. J Chem Phys. 71 (11), 4546-4553 (1979).
- Palmer, A. G., Kroenke, C. D., Loria, J. P. Nuclear magnetic resonance methods for quantifying microsecond-to-millisecond motions in biological macromolecules. Methods Enzymol. 339, 204-238 (2001).
- Clore, G. M., Iwahara, J. Theory, practice, and applications of paramagnetic relaxation enhancement for the characterization of transient low-population states of biological macromolecules and their complexes. Chem Rev. 109 (9), 4108-4139 (2009).
- Iwahara, J., Clore, G. M. Detecting transient intermediates in macromolecular binding by paramagnetic NMR. Nature. 440 (7088), 1227-1230 (2006).
- Lakomek, N. A., et al. Residual dipolar couplings as a tool to study molecular recognition of ubiquitin. Biochem Soc Trans. 36 (Pt 6), 1433-1437 (2008).
- Johnson, C. N., Libich, D. S. Paramagnetic relaxation enhancement for detecting and characterizing self-associations of intrinsically disordered proteins. J Vis Exp. (175), e63057(2021).
- Lakomek, N. A., Ying, J., Bax, A. Measurement of (1)(5)N relaxation rates in perdeuterated proteins by TROSY-based methods. J Biomol NMR. 53 (3), 209-221 (2012).
- Stief, T., Vormann, K., Lakomek, N. A. Sensitivity-enhanced NMR (15)N R(1) and R(1rho) relaxation experiments for the investigation of intrinsically disordered proteins at high magnetic fields. Methods. 223, 1-15 (2024).
- Kay, L. E., Keifer, P., Saarinen, T. Pure absorption gradient enhanced heteronuclear single quantum correlation spectroscopy with improved sensitivity. J Am Chem Soc. 114 (26), 10663-10665 (1992).
- Geen, H., Freeman, R. Band-selective radiofrequency pulses. J Magn Reson (1969). 93 (1), 93-141 (1991).
- Desvaux, H., Berthault, P. Study of dynamic processes in liquids using off-resonance RF irradiation. Prog Nucl Magn Reson Spectrosc. 35 (4), 295-340 (1999).
- Overhauser, A. W. Polarization of nuclei in metals. Phys Rev. 91 (2), 476-476 (1953).
- Overhauser, A. W. Polarization of nuclei in metals. Phys Rev. 92 (2), 411-415 (1953).
- Kumari, P., Frey, L., Sobol, A., Lakomek, N. A., Riek, R. (15)N transverse relaxation measurements for the characterization of micros-ms dynamics are deteriorated by the deuterium isotope effect on (15)N resulting from solvent exchange. J Biomol NMR. 72 (3-4), 125-137 (2018).
- Karschin, N., Krenek, S., Heyer, D., Griesinger, C. Extension and improvement of the methanol-d(4) NMR thermometer calibration. Magn Reson Chem. 60 (4), 203-209 (2022).
- Sattler, M., Schleucher, J., Griesinger, C. Heteronuclear multidimensional NMR experiments for the structure determination of proteins in solution employing pulsed field gradients. Prog Nucl Magn Reson Spectrosc. 34 (2), 93-158 (1999).
- Ferrage, F., Cowburn, D., Ghose, R. Accurate sampling of high-frequency motions in proteins by steady-state N-{H} nuclear Overhauser effect measurements in the presence of cross-correlated relaxation. J Am Chem Soc. 131 (17), 6048-6049 (2009).
- Delaglio, F., et al. NMRPipe: a multidimensional spectral processing system based on UNIX pipes. J Biomol NMR. 6 (3), 277-293 (1995).
- Skinner, S. P., et al. CcpNmr AnalysisAssign: a flexible platform for integrated NMR analysis. J Biomol NMR. 66 (2), 111-124 (2016).
- Lee, W., Tonelli, M., Markley, J. L. NMRFAM-SPARKY: enhanced software for biomolecular NMR spectroscopy. Bioinformatics. 31 (8), 1325-1327 (2015).
- Lee, W., Rahimi, M., Lee, Y., Chiu, A. POKY: a software suite for multidimensional NMR and 3D structure calculation of biomolecules. Bioinformatics. 37 (18), 3041-3042 (2021).
- Abyzov, A., et al. Identification of dynamic modes in an intrinsically disordered protein using temperature-dependent NMR relaxation. J Am Chem Soc. 138 (19), 6240-6251 (2016).
- Singh, A., Purslow, J. A., Venditti, V. 15N CPMG relaxation dispersion for the investigation of protein conformational dynamics on the micros-ms timescale. J Vis Exp. (170), e62395(2021).
- Gopalan, A. B., Hansen, D. F., Vallurupalli, P. CPMG experiments for protein minor conformer structure determination. Methods Mol Biol. 1688, 223-242 (2018).
- Tugarinov, V., Kay, L. E. Methyl groups as probes of structure and dynamics in NMR studies of high-molecular-weight proteins. Chembiochem. 6 (9), 1567-1577 (2005).
- Lipari, G., Szabo, A. Model-free approach to the interpretation of nuclear magnetic resonance relaxation in macromolecules. 1. Theory and range of validity. J Am Chem Soc. 104 (17), 4546-4559 (1982).
- Lipari, G., Szabo, A. Model-Free Approach to the interpretation of nuclear magnetic-resonance relaxation in macromolecules .2. Analysis of experimental results. J Am Chem Soc. 104 (17), 4559-4570 (1982).
- Clore, G. M., et al. Deviations from the simple two-parameter model-free approach to the interpretation of nitrogen-15 nuclear magnetic relaxation of proteins. J Am Chem Soc. 112 (12), 4989-4991 (1990).
- Lemaster, D. M. Larmor frequency selective model free analysis of protein NMR relaxation. J Biomol NMR. 6 (4), 366-374 (1995).
- Peng, J. W., Wagner, G. Mapping of spectral density functions using heteronuclear NMR relaxation measurements. J Magn Reson (1969). 98 (2), 308-332 (1992).
- Farrow, N. A., Zhang, O. W., FormanKay, J. D., Kay, L. E. Characterization of the backbone dynamics of folded and denatured states of an SH3 domain. Biochemistry. 36 (9), 2390-2402 (1997).
- Khan, S. N., et al. Distribution of pico- and nanosecond motions in disordered proteins from nuclear spin relaxation. Biophys J. 109 (5), 988-999 (2015).
- Smith, A. A., Ernst, M., Meier, B. H., Ferrage, F. Reducing bias in the analysis of solution-state NMR data with dynamics detectors. J Chem Phys. 151 (3), 034102(2019).
- Zumpfe, K., Smith, A. A. Model-free or not. Front Mol Biosci. 8, 727553(2021).
- Robustelli, P., Trbovic, N., Friesner, R. A., Palmer, A. G. Conformational dynamics of the partially disordered yeast transcription factor GCN4. J Chem Theory Comput. 9 (11), 5190-5200 (2013).
- Salvi, N., Abyzov, A., Blackledge, M. Multi-timescale dynamics in intrinsically disordered proteins from NMR relaxation and molecular simulation. J Phys Chem Lett. 7 (13), 2483-2489 (2016).
Access restricted. Please log in or start a trial to view this content.
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados