Для просмотра этого контента требуется подписка на Jove Войдите в систему или начните бесплатную пробную версию.
Method Article
NMR 15N Релаксационные эксперименты для исследования пикосекундной и наносекундной структурной динамики белков
В этой статье
Резюме
Спектроскопия ядерного магнитного резонанса (ЯМР) может характеризовать структурную динамику белков специфичным для остатков образом. Мы предоставляем практический протокол для записи экспериментов с релаксацией NMR 15NR 1 и R2 и гетероядерным эффектом Оверхаузера {1H}-15N (hetNOE), чувствительным к масштабу от пикосекунд до наносекунд.
Аннотация
Спектроскопия ядерного магнитного резонанса (ЯМР) позволяет изучать белки в растворе и при физиологических температурах. Часто либо амидные группы белкового скелета, либо метильные группы в боковых цепях используются в качестве репортеров структурной динамики белков. Исследование структурной динамики белковой основы глобулярных белков на меченых 15N и полностью протонированных образцах обычно хорошо работает для белков с молекулярной массой до 50 кДа. При применении дейтерации боковых цепей в сочетании с спектроскопией, оптимизированной с поперечной релаксацией (TROSY), этот предел может быть увеличен до 200 кДа для глобулярных белков и до 1 МДа, когда основное внимание уделяется боковым цепям. Когда исследуются внутренне неупорядоченные белки (IDP) или белки с внутренне неупорядоченными областями (IDR), эти ограничения по весу не применяются, но могут выходить далеко за их пределы. Причина в том, что МВУ или РДЭ, характеризующиеся высокой внутренней гибкостью, часто динамически разъединяются. Различные методы ЯМР позволяют получить представление о структурной динамике белка с атомным разрешением в широком диапазоне временных масштабов, от пикосекунд до часов. Стандартные измерения релаксации 15Н дают обзор внутренней гибкости белка и характеризуют динамику белковой основы на быстром пико- и наносекундном временном масштабе. В данной статье представлен практический протокол для постановки и записи экспериментов NMR 15N R1, R2 и гетеронуклеарного эффекта Оверхаузера (hetNOE). Мы показываем примерные данные и объясняем, как их просто качественно интерпретировать перед любым более сложным анализом.
Введение
Функция белка определяется не только его трехмерной структурой, но и его структурной динамикой, охватывающей его внутреннюю гибкость и структурные переходы между различными конформациями, которые будет принимать белок. Спектроскопия ядерного магнитного резонанса (ЯМР) позволяет исследовать структурную динамику белков в растворе 1,2,3. Последние разработки в области твердотельного ЯМР, детектируемого протонами, также позволяют охарактеризовать динамику белка в менее растворимом состоянии, например, липидной бислойной мембраны 4,5,6. В растворе ЯМР можно изучать структурную динамику белкового остова и боковых цепей белка. Для глобулярного белка исследование структурной динамики белкового остова может быть достигнуто до 50 кДа, если белок изотопно мечен 15N. При использовании дейтерации боковой цепи и спектроскопии с оптимизированной поперечной релаксацией (TROSY) этот предел может быть увеличен до 200 кДа 7,8. Когда основное внимание уделяется динамике боковых цепей, диапазон доступных белков и комплексов может быть расширен до 1 MDa 2,9.
Указанные ограничения по весу не распространяются на внутренне неупорядоченные белки (IDP), которые часто демонстрируют высокую внутреннюю динамику. Более 30% эукариотического протеома составляют МПР или внутренне неупорядоченные области (РЗ)10,11,12,13. Они играют центральную роль во многих клеточных процессах, таких как передача сигнала и транскрипция1, и часто участвуют во внутриклеточном разделении фаз 14,15,16,17. У ВПЛ отсутствует четко определенная трехмерная (3D) нативная структура в физиологических условиях и они имеют слабо направленный или пересеченный энергетический ландшафт17,18. Из-за низкой гидрофобности и сильного электростатического отталкивания, распределенного по позвоночнику МДУ или МПР, отсутствует движущая сила для складывания в жесткуюконструкцию19. ВПЛ часто принимают складчатую конформацию при сложном взаимодействии с другими партнерами по связыванию 10,20,21. Кроме того, посттрансляционные модификации (ПТМ) расширяют возможности сворачивания МВУ или РДЭ 22,23. Неправильное сворачивание ВПЛ было определено как причина различных заболеваний, включая нейродегенеративные заболевания 15,24,25,26.
ВПЛ и РДЭ демонстрируют высокую внутреннюю гибкость 21,27,28. Конформационные ансамбли, показывающие изменение атомных положений и двугранных углов, были получены на основе моделирования молекулярной динамики и ограничений, полученных на основе экспериментальных данных 29,30,31,32. Из-за динамики и возникающего в результате этого беспорядка в замороженном состоянии диффузная электронная плотность затрудняет их структурную характеристику с помощью современных методов структурной биологии, таких как крио-ЭМ или рентгеновская кристаллография. Кроме того, условия кристаллизации или методы подготовки образцов для экспериментов при криогенных температурах могут влиять на конформационное пространство, которое испытывают ИПЛ. Тем не менее, раствор ЯМР хорошо работает с высокодинамичными белками и, следовательно, хорошо подходит для исследования IDP 16,20,22,28,29,30,31,32,33,34,35,36,37,38.
Как было сказано выше, раствор ЯМР предлагает различные методы для изучения внутренней динамики белка в широком диапазоне временных масштабов (рис. 1), в первую очередь на основе спиновой релаксации 31,33,38,39,40,41,42.
Спиновая релаксация ядер 15N в амидных группах белкового остова индуцируется изменениямиориентации 1 H-15 N угла связи из-за внутренней динамики белка и коллективных движений (включая, когда это уместно, вращательную диффузию)27,43,44,45,46,47,48,49,50,51. На временных масштабах, меньших, чем время корреляции вращения τR (время, необходимое молекуле для вращения одного радианта, также называемое общим временем корреляции при переворачивании), анизотропия химического сдвига (CSA) и дипольная связь (D) активны и не усредняются при ротационной диффузии белка. Внутренняя динамика белкового каркаса, включающая изменения углов связи, переориентацию связей и вращательное вращение, индуцирует стохастические флуктуации CSA и тензора дипольной связи, что приводит к изменению локального магнитного поля, что в конечном итоге приводит к спиновой релаксации ЯМР 47,48,52,53. Эти колебания могут быть описаны общей корреляционной функцией. Преобразование Фурье общей корреляционной функции называется функцией спектральной плотности. В полуклассической теории релаксации Редфилда константы скорости релаксации ЯМР могут быть описаны линейными комбинациями этих функций спектральной плотности54.
Основные эксперименты по релаксации 15Н ЯМР, разработанные в начале 1990-х годов, включают 15N R1, R 1ρ и {1H}-15N ядерные эксперименты с эффектом Оверхаузера, чувствительные к быстрому пикосекундному (ps) наносекундному (нс) масштабу времени, более быстрому, чем время вращательной корреляции τR белка 45,55,56,57. Для характеристики динамики магистрали медленнее, чем время корреляции вращения τR, используются так называемые эксперименты с релаксационной дисперсией, R1ρ, и эксперименты Карра-Перселла-Мейбума-Гилла (CPMG), чувствительные к микросекундной (μс) - миллисекундной (мс) динамике 44,46,58,59,60,61. Динамика медленнее микросекунд может быть зафиксирована с помощью 15Н химического обменного переноса насыщения (CEST) NMR62, обменной спектроскопии (EXSY, от миллисекунд до секунд) или ЯМР в реальном времени (RT) (от секунд до часов)63,64. Эффект PRE (парамагнитная релаксация) парамагнитных зондов, а также остаточных дипольных связей (RDC) может быть использован для оценки всего диапазона динамики пс и мс 65,66,67,68.
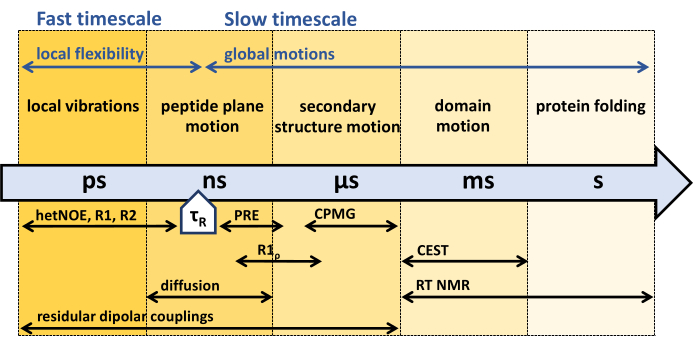
Рисунок 1: Временные шкалы динамики белковой магистрали и чувствительное временное окно различных экспериментов по динамике ЯМР. ЯМР предлагает множество методов для характеристики динамики белковой магистрали в широком диапазоне временных масштабов. Различные движения, испытываемые белковым каркасом, указываются в соответствующих временных масштабах. Время корреляции вращения белка, τR, — это время, необходимое белку для общего вращения (на одно излучение). Движения, превышающие время корреляции вращения белка, τR, могут быть связаны с внутренней гибкостью белка. Различные эксперименты с ЯМР и их чувствительность к соответствующим временным масштабам указаны ниже стрелкой. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этой цифры.
В приведенном ниже протоколе описывается постановка экспериментов по релаксации ЯМР Lakomek et al.69 и Stief et al.70 с использованием схемы детектирования гетероядерной одиночной квантовой когерентности (HSQC) с повышенной чувствительностью. Прежде чем приступить к экспериментальной реализации, дается очень краткий обзор экспериментов по спиновой релаксации ЯМР и экспериментам по релаксации ЯМР. Из-за ограничений по размеру и для того, чтобы этот протокол был понятен, этот обзор должен оставаться упрощенным (и, следовательно, неполным).
Продольная или спин-решеточная релаксация, характеризующаяся временем T1 или постоянной скорости R1 = 1/ T1 , описывает возврат намагниченности к равновесию Больцмана. В равновесии намагниченность выравнивается вдоль оси внешнего магнитного поля, которая определяет ось z лабораторной рамы. Спектральные плотности на высоких (1Н) и малых (15Н) частотах Лармора (резонансные частоты ЯМР, например, 600 МГц в течение 1Ч для магнита 14,1 Тесла) и линейные комбинации этих частот Лармора способствуют релаксации 15Н R1 , которая характеризуется константами скорости 15Н R1 , измеряемыми в рад·с-1. Движения на временных шкалах обратны этим частотам Лармора; таким образом, движения на пикосекундном и наносекундном временном масштабе вносят свой вклад в константу скорости релаксации R1. Для молекул, которые демонстрируют общую кувыркающуюся способность и для которых можно определить время корреляции вращения, кривая R1 (T1) показывает максимум (минимум) для ωτR = 1, при этом рассматривается вращательная корреляция τR и частота Лармора ω спина. Если вносят свой вклад несколько частот Лармора, то доминирующей является частота с наименьшей частотой, например, ωN в случае 15N R1. Режим быстрого движения (ωτR намного меньше 1) применяется к малым молекулам, которые кувыркаются очень быстро, а также к низким магнитным полям и низкой вязкости. Режим замедленного движения (ωτR намного больше 1) справедлив для более крупных молекул, которые кувыркаются медленнее, а также для сильных магнитных полей и высокой вязкости.
Шаровидные свернутые белки демонстрируют общее переворачивание в растворе, и может быть назначено время корреляции вращения. Тем не менее, концепция общего кувыркания больше не применима к внутренне неупорядоченным белкам и часто отличается от определения одного времени корреляции вращения. В этом случае время внутренней корреляции, специфичное для остатков, становится более критичным.
Описанная последовательность импульсов, измеряющая скорость релаксации 15NR1 (рис. 2), основана на эксперименте по считыванию HSQC с повышенной чувствительностью и детектированием эха/антиэха для квадратурного детектирования 69,70,71. Короткие градиенты с переменной прочностью и длиной используются для выбора когерентности и улучшенного подавления воды70. За это время продольная поляризация на 15Н ослабнет. Более длительное время затухания приводит к снижению интенсивности в связанных 2D-плоскостях этого псевдо-3D-спектра (точки данных задержки записываются в третьем измерении). Элемент цикла, описанный ниже, выполняется все большее количество раз для увеличения времени релаксации. Поскольку взаимно коррелированная релаксация между анизотропией химического сдвига 15N (CSA) и дипольной связью 1H и 15N (D) также активна во время задержки релаксации, необходим центральный импульс I-BURP-2 180°72, селективный по амидным протонам, для перефокусировки вклада путем перекрестной релаксации (которая, если ее не перефокусировать, приведет к искажению и ошибочным константам скорости 15NR1).
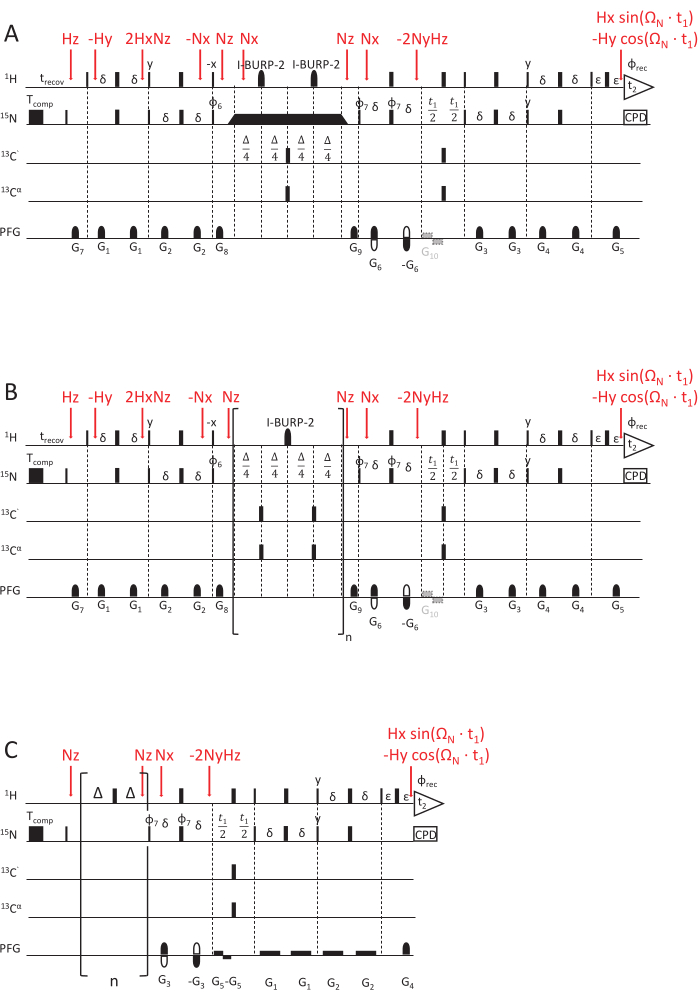
Рисунок 2: Схемы последовательности импульсов ЯМР для определения констант скорости релаксации ЯМР. (A) эксперимент 15N R1ρ, (B) 15N R1 и (C) hetNOE с использованием схемы считывания HSQC с повышенной чувствительностью69,70. Импульсы 90°(x) визуализируются узкими прямоугольниками, а импульсы 180°(x) — широкими прямоугольниками, если не указано иное. Применяется следующий фазовый цикл: φ6 = y, y, -y, -y; φ7 = y, -y, φrec = y, -y, -y, y. Квадратурное детектирование достигается путем инвертирования полярности градиента G5 и фазового цикла φ7 (Echo/Anti-Echo detection). (A) Эксперимент 15N R1ρ: Черный прямоугольник представляет спин-замок, продолжительность которого изменяется для получения различных задержек релаксации. Треугольники до и после спин-замка обозначают импульсы адиабатической формы, которые выравнивают намагниченность вдоль эффективной магнитной оси Beff. G10 является дополнительным градиентом, предотвращающим затухание излучения намагниченности воды во время фазы эволюции. (B) 15N R1 эксперимент: В квадратных скобках показан элемент петли последовательности, повторенный n раз для соответствия желаемой задержке релаксации. (C) Импульсная схема hetNOE аналогична второй половине импульсных схем R1 и R1ρ, а именно времени эволюции t1 и HSQC-детектирующего элемента. Однако намагниченность 15Н возбуждается напрямую, без какого-либо INEPT. Насыщение намагниченности протона (для достижения перекрестной релаксации между 1Ч и 15Н) достигается последовательностью из 180 (1Н) импульсов, приложенных в течение не менее 5 с. Для эталонного эксперимента применяется задержка холостого хода той же длины (в данном случае 5 с) без какой-либо последовательности импульсов. G5 является дополнительным градиентом для предотвращения затухания излучения, а инверсия полярности градиента G4 в сочетании с фазой φ7 = y, -y, -y, y обеспечивает квадратурное детектирование. Этапы переноса намагниченности, представленные операторами продукта, отмечены красным цветом. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этой цифры.
Константа скорости релаксации R2 описывает релаксацию поперечной поляризации (в плоскости xy, ортогонально внешнему магнитному полю) из-за потери фазовой когерентности между спинами, что приводит к затуханию обнаруживаемой намагниченности53,54. Функция спектральной плотности на высоких и малых частотах вносит свой вклад в R2, подобно R1. Тем не менее, наибольший вклад в R2 вносит спектральная плотность на нулевой частоте. По этой причине R2 очень чувствителен к вращательному переворачиванию, описываемому временем вращательной корреляции τR, которое составляет порядка нескольких нс для небольшого глобулярного белка при комнатной температуре. Таким образом, наибольший вклад вносят более медленные движения основной магистрали в режиме от сотен пс до низких нс. Обменная динамика остов, вызывающая модуляцию изотропной части тензора химического сдвига ядер 15N, в принципе прибавляют обменный вклад R 2ex к константам скоростей R 23,44,49,60,61. В описанных экспериментах вклад R2ex подавляется спиновой блокировкой, которая перефокусирует динамику медленнее, чем обратная круговая частота спиновой блокировки. Спиновая блокировка представляет собой длинный непрерывный радиочастотный импульс, при котором намагниченность выравнивается вдоль эффективной магнитной оси Beff (векторная сумма поля спиновой блокировки ω1 и смещения химического сдвига от несущей частоты 15Н (см. ниже)). Релаксация намагниченности, выровненной по оси B1,eff, называется релаксацией R1ρ, которая имеет компонент R1 и компонент R2. По формуле (1) R2 можно вычислить из R1ρ и R144,73:
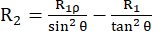 (1).
(1).
Угол между осью эффективного магнитного поля Beff и внешним магнитным полем B0 равен 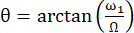 . ω1 представляет собой радиочастотную амплитуду спин-замка, а Ω смещение химического сдвига между химическим сдвигом соответствующего остатка на 15Н и несущей частотой 15Н 44,73.
. ω1 представляет собой радиочастотную амплитуду спин-замка, а Ω смещение химического сдвига между химическим сдвигом соответствующего остатка на 15Н и несущей частотой 15Н 44,73.
Схема импульса R1ρ (рис. 2A, 70) очень похожа на схему 15N R1 , за исключением задержки релаксации. Для измерения скоростей релаксации 15N R1ρ спин-блокировка должна быть активной после того, как намагниченность была выровнена вдоль эффективной оси поля Beff адиабатическим импульсом с той же амплитудой радиочастоты (РЧ), что и спин-блокировка. Длина спин-фиксатора будет варьироваться для получения различных задержек релаксации.
Стационарный ядерный эффект Оверхаузера {1H}-15N N (1 H-15N NOE), называемый далее hetNOE, представляет собой отношение скорости перекрестной релаксации и скорости продольной релаксации 15Н. Это приводит к уменьшению стационарной поляризации на 15Н за счет перекрестной релаксации с протоном при насыщении поляризацией протона 45,53,54,74,75. Перекрестная релаксация зависит от функций спектральной плотности суммы и разности частот 1Н и 15Н Лармора. Таким образом, hetNOE чувствителен как к быстрой пикосекундной динамике (< 100 пс), так и к динамике ps-ns (из-за его зависимости от R1). Последовательность69 (рисунок 2C) основана на считывании HSQC с повышенной чувствительностью и градиентами эхо/антиэхо для квадратурного детектирования. Для насыщения протонной намагниченности и результирующей hetNOE равновесную намагниченность протона инвертируют и затем насыщают быстрыми импульсами на 180° примерно в 5 раз больше 15 N T1. Для эталонного эксперимента задержка восстановления равна задержке насыщения, но без последовательностиимпульсов 1 ч 180°. Дополнительная задержка D1 = 2 с добавляется для контрольного эксперимента и эксперимента с насыщением 1Н. Оба эксперимента записываются один за другим и отличаются только применениемимпульсов 1 ч 180° (насыщение) или отсутствием (опорное). Отношение спектральных интенсивностей, зарегистрированных в эксперименте с насыщением 1H, деленное на интенсивности эталонного эксперимента (без последовательности импульсов протона 180°), дает значение {1H}-15N NOE (hetNOE).
Следующий протокол описывает постановку экспериментов по релаксации ЯМР, выполненную Lakomek et al.69 и Stief et al.70. Мы сосредоточились на последовательностях импульсов ЯМР с использованием схемы детектирования HSQC с повышенной чувствительностью. Эксперименты с 15N R1 и R1ρ реализованы так, как подробно описаны Stief et al.70, а эксперимент с hetNOE описан Lakomek et al.69.
Access restricted. Please log in or start a trial to view this content.
протокол
1. Подготовка образцов ЯМР
Примечание: Изотопное мечение белков выполняется для ЯМР более высокой размерности и расширенных экспериментов ЯМР. После того как экспрессия белка в E. coli и очистка белка были установлены с использованием насыщенных сред (например, Лурия-Бертани [LB] или 2-кратного триптонного экстракта дрожжей [2YT]) с выходом в несколько миллиграммов на литр, получение изотопно меченного образца ЯМР обычно является относительно простым.
- Для мечения изотопов используйте минимальную среду M9 для экспрессии генов, где хлорид аммония 15N является единственным источником азота, а обогащенная 13C глюкоза является единственным источником углерода.
- Для очистки белка следуйте стандартному протоколу, установленному в лаборатории для соответствующего белка.
ПРИМЕЧАНИЕ: В разделе «Результаты» представлены репрезентативные результаты для IDP, мономерного белка SNARE Synaptobrevin-2 (1-96). ЯМР-мечение изотопом синаптобревина-2 проводили, как описано в70. Для очень высокоточных измерений рекомендуется использовать низкие концентрации D2O, менее 5%. Это связано с эффектом изотопа дейтерия, который можно наблюдать в экспериментах Hahn-Echo 15N T2 и в низкочастотных экспериментах с релаксационной дисперсией 15N CPMG (здесь не обсуждается), но оказывает лишь минимальное влияние в экспериментах с 15N R1ρ (где он в основном перефокусирован)76.
2. Подготовка к проведению экспериментов по релаксации ЯМР на спектрометре
ПРИМЕЧАНИЕ: Описанные эксперименты по релаксации ЯМР специфичны для спектрометров Bruker. Они были протестированы на криогенных и трехрезонансных зондах при температуре 1ч, 15Н и 13С, а также на консолях Avance III и Avance Neo, работающих с программным обеспечением Bruker Topsin 3.6 или выше.
- Соблюдайте меры предосторожности.
- Перед работой на ЯМР-спектрометре проведите инструктаж по технике безопасности.
- Проверьте характеристики мощности щупа используемого спектрометра и выберите подходящую длину импульса и мощность импульса для каждого импульса, например, сравнив согласованность с записями таблицы Topspin «prosol».
- Импортируйте последовательности импульсов.
- Загрузите программы импульсов ЯМР либо с веб-сайта, указанного ниже, либо из расширенного репозитория Банка биологического магнитного резонанса (BMRB) (bmrbig102) в каталог /opt/topspin4.1.4/exp/stan/nmr/lists/pp/пользователь компьютера, на котором работает спектрометр ЯМР. Пожалуйста, настройте версию topspin (здесь: topspin4.1.4 на соответствующую версию, установленную на спектрометре).
- Загрузите импульсы в форме ЯМР либо с веб-сайта, указанного ниже, либо из расширенного репозитория Банка биологического магнитного резонанса (BMRB) (bmrbig102) в каталог /opt/topspin4.1.4/exp/stan/nmr/lists/pp/пользователь компьютера, на котором работает спектрометр ЯМР. Пожалуйста, настройте версию topspin (здесь: topspin 4.1.4 на соответствующую версию, установленную на спектрометре).
- Откалибруйте спектрометр и образец.
- Установите интересующую температуру. Для калибровки температуры применяйте протоколы объекта. Например, используйте образец метанола-d4 и интерпретируйте наблюдаемую разность химических сдвигов с помощью веб-приложения NMR thermometer (https://chem.ch.huji.ac.il/nmr/software/thermometer.html); см. также77.
- Вставьте образец (плюс дополнительные 5% D2O для замка) и подождите примерно 10 минут, чтобы образец сбалансировал свою температуру. Заблокируйте дейтериевый сигнал (2H) (команда: lock, выберите H2O+D2O).
- Сопоставляйте и настраивайте каналы 1H, 15N и 13C, например, с помощью процедуры atma или atmm. Если блок автоматического согласования и настройки не установлен, вручную выполните сопоставление и настройку щупа с помощью команды wobb.
- Выполните шимминг для однородного поля B0 , например, с помощью подпрограммы topshim tg.
- Откалибруйте длину импульса 90° (1ч).
- Используйте эксперимент zg. Используйте getprosol для импорта параметров зонда. Выберите короткую длительность 5 μс для P1. Проверьте и при необходимости откорректируйте указанный уровень мощности щупа для PLW1.
- Начните эксперимент с zg. Обрабатывайте данные с помощью efp. Отрегулируйте фазу с помощью .p, чтобы базовая линия была ровной, а сигнал ЯМР водного резонанса находился в фазе.
- Оцените импульс на 360° (1Н) путем взятия обычного или приблизительного импульса 90° (1Н) (например, из перечисленных спецификаций спектрометра) и умножения его на четыре раза. Запустите эксперимент и обработайте его.
ПРИМЕЧАНИЕ: Выбранная продолжительность слишком длинная, если сигнал положительный, и слишком короткая, если отрицательный. Импульс на 360° (1ч) будет пересечением нуля. - Отрегулируйте длину импульса соответствующим образом. Делайте это итеративно, чтобы найти пересечение нуля. Разделите полученное значение на четыре. Это будет импульс 90° (1ч).
ПРИМЕЧАНИЕ: Альтернативой описанному методу 360° является импульсная процедура, которая может быть запущена из командной строки и позволяет выполнять автоматическую калибровку импульса за 1ч.
- Откалибруйте несущую частоту O1 в соответствии с резонансом воды.
- Скопируйте эксперимент zg, введя edc в командной строке на новый номер эксперимента. Введите rpar для открытия наборов параметров (дополнительный рисунок 1A).
- Поиск по набору параметров ZGPR. Нажмите на набор параметров ZGPR (дополнительный рисунок 1A), нажмите на Read, выберите Execute "getprosol" и нажмите OK (дополнительный рисунок 1B).
- Измените P1 на калиброванную длину импульса 90° (1H). Выберите длину насыщения D1 = 2 с и мощность импульса PLW9 50 дБ. Установите коэффициент усиления приемника (rg) на 16. В качестве альтернативы выполните регулировку усиления ресивера, введя rga и установив усиление ресивера на указанное значение (но избегайте "переполнения ресивера").
- Введите gs в командной строке, чтобы начать повторный непрерывный сбор данных. Перейдите в окно сбора данных и измените O1, чтобы уменьшить зону FID, в которой доминирует сигнал воды (дополнительный рисунок 2).
- Щелкните ниже маркера, чтобы уменьшить O1, и щелкните над маркером, чтобы увеличить его. Используйте кнопку чувствительности для изменения размера шага. Сохранить текущий O1; Закрытие останавливает повторное получение данных (дополнительный рисунок 2).
- Откалибруйте длину импульса 90° (15Н).
- Скопируйте эксперимент zg на новый номер эксперимента (тип edc). Введите rpar для чтения набора параметров HSQCFPF3GPPHWG для изменения программы импульсов на эксперимент 1 H-15N HSQC. Используйте опцию Сохранить параметры P1, O1 и PLW1 (дополнительный рисунок 1).
- Введите getprosol 1H <откалиброван 90° (1H) длина импульса в μс> <90° (1H) сила импульса в дБ> в командной строке для импорта параметров зонда. Установите количество сканирований (NS) равным 8, а количество фиктивных сканирований (DS) — 2.
ПРИМЕЧАНИЕ: < > указывает здесь значения, которые должны быть установлены. Например: гетпросол 1Н 8.5 -11.79. - Запустите первое приращение эксперимента, введя zg в командной строке. Скопируйте первый приращение в новый номер эксперимента с помощью rser 1 <новый номер эксперимента>. Обработайте данные с помощью efp и фазируйте спектр с помощью .тел. Используйте первый инкремент (1D-эксперимент) эксперимента для выполнения следующих шагов.
ПРИМЕЧАНИЕ: В < > указать выбранный номер для нового эксперимента, например, rser 1 101. - Введите ased в командной строке, чтобы открыть параметры последовательности импульсов 1D-эксперимента. Нажмите на букву E в строке PULPROG (см. дополнительный рисунок 3A), чтобы открыть редактор импульсных программ.
- Используйте следующий рабочий процесс для сохранения последовательности импульсов под новым именем: нажмите «Файл» (левый верхний угол), «Сохранить как», введите новое имя и нажмите «ОК». Затем нажмите кнопку Установить PULPROG для набора данных (см. дополнительный рисунок 3B) и нажмите кнопку OK.
- Измените исходную последовательность 1 H-15N HSQC (сохраненную под новым именем, см. 2.6.4.), умножив 90° (15N) после периода эволюции t1 (до обратного переноса) в 2 раза (импульс 90° (15N) теперь равен 180° (15N)). Нажмите Ctrl S для сохранения последовательности импульсов. Установите длину импульса на 90° (15Н) (P21) так, чтобы сигнал ЯМР первого приращения исчезал (при пересечении нуля) (дополнительный рисунок 3B).
- Откройте эксперимент 1 H-15N HSQC (эксперимент 2D) и введите калиброванную длину импульса 90° (15N) (P21). Введите getprosol 1H <откалиброванный 90° (1H) длина импульса в мкс> <90° (1H) мощность импульса в дБ> 15 Н <откалиброванный 90° (15Н) длина импульса в мкс> <90° (15Н) в дБ> в командной строке.
- Определение спектральной ширины и распределения времени захвата.
- В окне параметра сбора данных для эксперимента 1 H-15N HSQC (введите eda в командной строке) отрегулируйте спектральную ширину (параметр сбора данных SW) и количество приращений размерности в прямых и косвенных измерениях (параметр сбора данных TD).
- Выберите для SW, 16 ppm в прямом (1H) и 30 ppm в косвенном (15N) размере. Выберите TD, 2048 в прямом (1H) и 512 в непрямом (15N) размере.
- Приобретите эксперимент 1 H-15N HSQC.
- Убедитесь, что все интересующие сигналы обнаружены и что в спектре отсутствуют артефакты наложения. Измените O3P, если центр спектра не совпадает с центром распределения сигнала белка по оси 15N.
- Увеличьте SW , если спектральное окно слишком мало. Увеличьте TD для получения более высокого разрешения FID (проверьте характеристики пробника для максимальной продолжительности развязки).
ПРИМЕЧАНИЕ: Это общая установка и отправная точка для постановки экспериментов по релаксации ЯМР. Рекомендуется записывать эксперимент 1 H-15N HSQC в начале и в конце всей серии экспериментов, а для более длительного времени эксперимента также между ними для контроля целостности образца.
- В окне параметра сбора данных для эксперимента 1 H-15N HSQC (введите eda в командной строке) отрегулируйте спектральную ширину (параметр сбора данных SW) и количество приращений размерности в прямых и косвенных измерениях (параметр сбора данных TD).
3. Проведение экспериментов по релаксации ЯМР
ПРИМЕЧАНИЕ: Последовательности релаксационных импульсов ЯМР (рис. 2) доступны в https://www.ipb.hhu.de/en/teams/team-lakomek/pulsesequences или в расширенном репозитории банка биологического магнитного резонанса (BMRB) (bmrbig102).
- Настройте параметр 15Н Р1ρ эксперимент.
- Скопируйте эксперимент HSQC (тип edc) в новый каталог. Введите ased и измените программу импульсов на эксперимент 15N R1ρ, щелкнув по трем точкам на линии параметров PULPROG в параметрах последовательности импульсов (ased, см. дополнительный рисунок 3A). Дважды щелкните по программе пульса, нажмите Set PULPROG to dataset (см. дополнительный рисунок 3B) и нажмите OK.
ПРИМЕЧАНИЕ: Могут появиться некоторые сообщения об ошибках. Закройте эти сообщения. - Введите gpnam в командной строке, чтобы вставить все отсутствующие формы градиента (gpnam1-gpnam9), как описано в последовательности импульсов (дополнительный рисунок 4).
- Введите vplist в командной строке. Нажмите на три точки (Дополнительный рисунок 5A), Файл (верхний левый угол) и Новый (Дополнительный рисунок 5B).
- Введите имя нового фиктивного vplist и нажмите OK. Создайте фиктивный vplist (5m, 65m, 25m, 45m, 15m, 55m, 35m, 10m; новая строка для каждой записи vplist). Нажмите Ctrl S, чтобы сохранить список и закрыть окно vplist (Дополнительный рисунок 5C).
- Дважды щелкните по имени только что созданного vplist (дополнительный рисунок 5B).
ПРИМЕЧАНИЕ: Это необходимо для того, чтобы избежать ошибок компиляции в последовательности импульсов. Соответствующие записи vplist будут определены далее (. 3.1.29.). - Введите ased для открытия параметров последовательности импульсов (дополнительный рисунок 6). Введите все недостающие силы градиента и длины градиента , как указано в последовательности импульсов (дополнительный рисунок 6D).
- Введите для счетчика петли L3 (количество комплексных точек в косвенной размерности) 1/2 от 15N TD записи в окне параметров сбора данных (eda). Введите для счетчика петли L6 количество записанных точек данных релаксации, используемых для аппроксимации кривой релаксации.
ПРИМЕЧАНИЕ: Это число L6 соответствует количеству записей vplist, здесь: 8 (Дополнительный рисунок 6A). - Установите адиабатический импульс TanhTan_half.nl, загруженный с указанного выше сайта, в качестве формы импульса 15Н SP8. Установите адиабатический импульс TanhTan_2nd.nl с веб-сайта в качестве формы импульса 15Н SP9 (дополнительный рисунок 6C).
- Установите вход P8 на длину адиабатического импульса Тантана - достаточную длину для гарантии адиабатичности пульса; здесь выбрано значение P8 = 3000 μs (дополнительный рисунок 6C).
- Установите развязку 15Н (CPDPRG 3) на схему развязки GARP. Используется для длины развязки PCPD3 200 мкс (дополнительный рисунок 6C).
- Установите задержку восстановления между сканированиями не менее D1 = 2 с или дольше.
ПРИМЕЧАНИЕ: Для выбора подходящей задержки восстановления см. раздел Обсуждение. - Установите фиктивное сканирование (DS) как минимум 64 для окончательного эксперимента (во время настройки DS = 4 достаточно по временным причинам). В качестве отправной точки используйте количество сканирований (NS), равное 4; используйте число, кратное 4, если отношение сигнал/шум (SNR) слишком низкое (дополнительный рисунок 6A).
- Установите O1 на калиброванную несущую частоту на шаге 2.5. Установите O2P на 176 ppm и скопируйте O3P из эксперимента 1 H-15N HSQC (дополнительный рисунок 6A-C).
- Установите длину импульса P7 равной 90° (15Н), откалиброванную на шаге 2.6. Кроме того, скопируйте уровень импульсной мощности импульса 90° (15Н) с шага 2,6 на PLW3 и PLW7 (дополнительный рисунок 6C).
- Установите длительность импульса P1 и P19 равной длине импульса 90° (1H), рассчитанной на шаге 2.4. (Дополнительный рисунок 6B).
- Задайте количество приращений в косвенном размере: TD =l 3 * 2 *l 6 (eda или введите td в командной строке).
ПРИМЕЧАНИЕ: Все описанные здесь параметры последовательности импульсов дополнительно аннотируются в последовательности импульсов. - Установите форму импульса SP5 в форму I-BURP2 (Iburp2.1000). Установите длительность импульса P15 равной 2000 μс. Откройте дисплей инструмента «Фигура», нажав на букву E рядом с импульсом формы I-BURP2 в окне параметров последовательности импульсов Topspin (ased) (дополнительный рисунок 6B).
- Смоделируйте импульс формы с нужной длиной, нажав кнопку «Начать моделирование ЯМР ». Проверьте сформированную длину импульса [μс] (= 2000 μс) и угол поворота [°] (= 180°) в окне моделирования. Нажмите « Запустить NMR-SIM » (дополнительный рисунок 7A).
- Проверьте диапазон возбуждения и выберите подходящую длину импульса I-BURP2, чтобы покрыть спектральную дисперсию белка 1H, но в то же время избежать возбуждения воды. Установите P15 в значение Shaped pulse length окна моделирования с лучшим моделируемым импульсом IBURP-2 (Дополнительный рисунок 7B).
ПРИМЕЧАНИЕ: Правильная длина выбирается в том случае, если I-BURP2 возбуждает (> 95% эффективности) всю амидную область. Чтобы смоделировать импульс I-BURP2 с другой длиной, закройте окно профиля возбуждения и отредактируйте сформированную длину импульса в окне моделирования. Нажмите « Запустить NMR-SIM». Импульсы с меньшей длиной имеют более широкий диапазон возбуждения в Гц. - Установите SPOFFS5 для изменения несущей частоты импульса I-BURP2 и смещения диапазона возбуждения влево или вправо, чтобы импульс I-BURP2 не касался и не нарушал намагниченность воды (дополнительный рисунок 6B).
- Чтобы определить подходящий уровень мощности сформированного импульса, откройте инструмент Bruker shape, нажмите кнопку «Начать моделирование ЯМР » и установите длину импульса I-BURP2 на Длина сформированного импульса [μс]. Запомните соответствующую длину мягкого прямоугольного импульса 90° (1H), видимого в окне моделирования при соответствующем квадратном импульсе 90° [μс] (дополнительный рисунок 7A).
- Тип calcpowlev. Используйте жесткий импульс 90° (1H) (P1) в микросекундах в качестве опорной длины (дополнительный рисунок 8B) и мягкий прямоугольный импульс 90° (1H), соответствующий импульсу I-BURP2 в микросекундах, в качестве новой длины (дополнительный рисунок 8A). Помните о разнице мощности Δ в дБ между жестким импульсом 90° (1H) и мягким прямоугольным импульсом (дополнительный рисунок 8C). Скопируйте силу импульса жесткого импульса 90° (1ч) в дБ в SPW5 и добавьте заученную разность Δ в дБ (дополнительный рисунок 6B).
- Чтобы определить уровень мощности спин-блокировки, рассчитайте соответствующую длину импульса спин-блокировки под углом 90° (15Н). Используйте формулу: длина импульса 90° (15Н) в s = 0,25 / (мощность блокировки спина в Гц).
- Используйте calcpowlev для вычисления разницы мощностей Δ в дБ в дБ между мощностью спин-замка и жестким импульсом (P7) под углом 90° (15Н). Новинка: длина импульса спин-фиксатора 90° (15Н) в мкс (дополнительный рисунок 8A). Ссылка: длительность импульса в жестком диапазоне 90° (15Н) в мкс (Дополнительный рисунок 8B). Скопируйте уровень мощности PLW7 жесткого импульса 90° (15Н) в дБ на уровень мощности PLW8 с блокировкой вращения (дополнительный рисунок 6C) и добавьте рассчитанную разницу мощности Δ в дБ (дополнительный рисунок 8C).
- Скопируйте уровень мощности спин-локатора PLW8 на уровни мощности SPW8 и SPW9. (Дополнительный рисунок 6C).
- Чтобы определить уровень мощности развязки 15Н, используйте calcpowlev для расчета разницы мощностей Δ между мощностью импульса развязки 90° (15Н) (PCPD3) и жесткого импульса 90° (15Н) (P7). Новинка: PCPD3 в μs (200) (дополнительный рисунок 8A). Ссылка: длительность импульса в жестком диапазоне 90° (15Н) в мкс (Дополнительный рисунок 8B). Скопируйте уровень мощности PLW7 жесткого импульса 90° (15Н) в дБ в дБ на уровень развязки PLW31 (дополнительный рисунок 6C) и добавьте Δ в дБ (дополнительный рисунок 8C).
- Чтобы использовать температурную компенсацию, включите #define TEMP_COMPENSATION (удалив точку с запятой в последовательности импульсов). Установите P18 на максимальную длительность спин-блокировки, используемой в эксперименте 15N R1ρ .
ПРИМЕЧАНИЕ: Рекомендуется отключить температурную компенсацию для первого испытания эксперимента. - В случае образцов, помеченных 15N 13C, включите # для определения LABEL_CN в программе импульсов (путем удаления точки с запятой в последовательности импульсов). Установите P4 на длину импульса, рассчитанную по формуле78. Например, она соответствует 23,70 мкс на частоте 600 МГц и 11,85 мкс на частоте 1200 МГц. Введите calcpowlev, чтобы рассчитать разницу мощности в дБ между P4 и жестким импульсом (13C). Скопируйте мощность жесткого импульса (13C) в дБ в PLW4 и добавьте Δ в дБ. Скопируйте PLW4 в PLW2 (дополнительный рисунок 6B).
- Определите задержки релаксации для соответствующего отбора проб.
- Запустите первые восемь FID (в случае, если L6 = 8) и обработайте их (rser <номер FID> < номер нового эксперимента>). Выберите записи в vplist, чтобы пиковая интенсивность эксперимента с самой большой задержкой снизилась как минимум до 1/e (около 37%), но не менее чем на 25% по сравнению с экспериментом с самой короткой задержкой (которая служит эталоном).
- Отрегулируйте оставшиеся записи vplist для достижения равномерной выборки снижения интенсивности между FID с самой большой задержкой и FID с самой короткой задержкой. Прочтите ПРИМЕЧАНИЕ и технические характеристики мощности пробника.
ПРИМЕЧАНИЕ: Медленно расслабляющие остатки будут доминировать в первом FID. Отрегулируйте оставшиеся записи vplist для достижения равномерной выборки снижения интенсивности между первым и вторым FID. Не превышайте максимально допустимую мощность и длину блокировки спин-фиксатора щупа (см. руководство по эксплуатации щупа и технические характеристики щупа). В случае температурной компенсации проверьте P18 (максимальная продолжительность блокировки спина), так как будет применяться общая продолжительность блокировки спина P18, разделенная между задержкой релаксации и температурной компенсацией. Он содержит время (задержка релаксации) + время (температурная компенсация) = const = P18. Для спин-блокировки на частоте 2 кГц мы предпочитаем не превышать длительность спин-синхронизации более 65 мс, используя задержку восстановления (D1) в 2 с. В этом случае необходимо найти компромисс между максимальной продолжительностью блокировки спина и самой большой задержкой релаксации в пользу максимально допустимой длительности блокировки спина. В случае более длительных задержек восстановления D1 могут применяться более длительные мощности блокировки вращения. Что имеет значение, так это общее отложение мощности в зонде с течением времени. Если требуется более длительная продолжительность блокировки вращения, увеличивайте длину блокировки вращения только небольшими шагами и следите за сигналом блокировки. Как правило, отрегулируйте уровень блокировки в верхней трети дисплея блокировки. Если уровень блокировки уменьшается более чем на два квадрата во время выполнения последовательности импульсов, мощность блокировки спина, передаваемая в пробник, становится слишком большой.
- Введите rga в командной строке, чтобы определить коэффициент усиления ресивера. Для получения хорошего соотношения сигнал/шум стремитесь к значению усиления приемника (rg) в диапазоне от 128 до 256, что доказывает хорошее подавление воды.
ПРИМЕЧАНИЕ: Недостижимое усиление приемника в диапазоне от 128 до 256 указывает на слишком сильный сигнал воды. В этом случае мощность градиента и длительность плавно регулируются до тех пор, пока не будет достигнуто усиление приемника. Смотрите также раздел Обсуждение. - Запустите тестовый запуск эксперимента 15N R1ρ , введя zg в командной строке. Убедитесь, что сигнал воды хорошо подавлен при всех задержках (от 1 до 8, в случае 8 задержек в vplist). Проверьте также инкремент 9, который является вторым инкрементом квадратурной схемы детектирования (Эхо/Анти-Эхо).
- Скопируйте эксперимент HSQC (тип edc) в новый каталог. Введите ased и измените программу импульсов на эксперимент 15N R1ρ, щелкнув по трем точкам на линии параметров PULPROG в параметрах последовательности импульсов (ased, см. дополнительный рисунок 3A). Дважды щелкните по программе пульса, нажмите Set PULPROG to dataset (см. дополнительный рисунок 3B) и нажмите OK.
- Поставьте задачу эксперимента 15N R1 .
- Скопируйте эксперимент с 15N R1ρ и измените программу импульсов на эксперимент 15N R1 .
- Введите vclist, сгенерируйте vclist и введите фиктивный vclist, описанный в70: 0, 24, 6, 20, 4, 16, 8, 12; новая строка для каждой записи vclist).
- Откройте ассед. Проверьте длину и силу градиента. Измените GPZ9 на 14%.
- Проверьте длину импульсов, их форму и силу (правильные значения должны были быть скопированы из эксперимента 15N R1ρ ).
- В случае образцов, меченных 15N13C, используйте #define LABEL_CN в программе импульсов.
- Для использования температурной компенсации включите #define TEMP_COMPENSATION.
- Запустите первые восемь FID (в случае, если L6 = 8) и обработайте их (rser <номер FID> < новый номер эксперимента>). Выберите записи в vclist, чтобы пиковая интенсивность эксперимента с наибольшей задержкой уменьшалась как минимум на 1/e (около 37%), но не менее чем на 25% по сравнению с экспериментом с наименьшей задержкой. Отрегулируйте оставшиеся записи vplist для достижения равномерной выборки снижения интенсивности между FID с самой большой задержкой и FID с самой короткой задержкой.
ПРИМЕЧАНИЕ: Для записей vclist допускаются только четные числа.
- Постановка эксперимента {1H}-15N NOE.
- Скопируйте эксперимент с релаксацией 15Н R1 и измените последовательность импульсов на схему импульсов {1H}-15N NOE.
- Установите длину периода насыщения с помощью счетчика петель L8. Интервал между импульсами 180° (1H) составляет 22 мс79 (2*1 / JNH). Для достижения наиболее точных результатов используйте период насыщения, примерно в 5 раз превышающий 15N T1 (15N) белка. Здесь: 5 с. Это соответствует L8 = 230.
ПРИМЕЧАНИЕ: В разделе "Обсуждение" содержатся дополнительные советы по выбору периода насыщения/задержки восстановления. - Изменяйте длину и силу градиентов, как описано в схеме импульсов. Используйте формы градиента sine.20.
- Установите количество приращений в косвенном размере, равное TD = l3 * 2 * 2
- В случае образцов, меченных 15N13C, используйте #define LABEL_CN и -DLABEL_CN в качестве ZGOPTNS.
- Запустите импульсные схемы, набрав zg в командной строке.
4. Обработка и анализ записанных ЯМР-экспериментов
ПРИМЕЧАНИЕ: Спектры были зарегистрированы с помощью системы Bruker. Обработка выполняется с помощью операционной системы Unix или Linux. Обработку спектров и анализ данных проводили с помощью NMRPipe80 и python3. Программное обеспечение NMRPipe можно загрузить по адресу https://www.ibbr.umd.edu/nmrpipe/index.html. Скрипты обработки на основе NMRPipe загружаются с веб-сайта: https://www.ipb.hhu.de/en/teams/team-lakomek/pulsesequences или из расширенного репозитория Банка биологического магнитного резонанса (BMRB) (bmrbig102). Рекомендуется использовать NMRPipe. Если NMRPipe недоступен или нежелателен, можно использовать альтернативы, такие как CCPN81 или SPARKY (SPARKY 3 или его преемники NMRFAM-SPARKY82 или POKY83 ).
- Обработка и анализ 15Н Р1 эксперимент.
ПРИМЕЧАНИЕ: Следующие команды выполняются в терминале Unix.- Скопируйте файлы из загруженного каталога R1_processing в каталог, содержащий экспериментальные данные R1 (рабочий каталог).
- Сделайте файлы исполняемыми с помощью команды терминала: > chmod u+x *.com
- Выполните > bruker , чтобы открыть утилиту преобразования NMRPipe и сгенерировать файл fid.com. Нажмите кнопку Прочитать параметры. Измените значение параметра Количество измерений на два, если подсчитываются три измерения, и снова нажмите кнопку Прочитать параметры . Затем нажмите Clear Script, Update Script и Save Script (дополнительный рисунок 9).
ПРИМЕЧАНИЕ: Если несущая частота неверна, измените положение центра PPM, нажав кнопку "Прочитать параметры " во второй раз. - Выполнить > ./fid.com
- Преобразуем vclist в vplist с ms записями: задержка в ms = 40 ms * запись vclist. Используйте > cp vclist vplist и отредактируйте vplist.
- Используйте > ./split.com для разделения псевдо 3D данных на восемь 2D файлов.
- Отредактируйте значения tauValues (значения vplist в порядке vplist) и idxExpmt (порядок значений tauValues относительно времени) в nmrproc.com (дополнительный рисунок 10).
- Используйте > ./nmrproc.com для обработки исходных данных всех восьми 2D-спектров.
- Используйте > nmrDraw для рисования спектров.
- Для этих шагов используйте эталонный спектр (первая запись в vplist). Переключитесь на этот спектр, изменив z-размерность на единицу (верхний левый угол). Отрегулируйте уровень контура с помощью кнопок + и - рядом с кнопкой Фактор . Перерисуйте спектр с помощью кнопки Draw.
- Используйте уровень контура, при котором все интересующие сигналы могут быть распознаны, и только несколько сигналов поступают от шума (уровень контура находится на краю уровня шума) (дополнительный рисунок 11).
- Корректируйте фазу спектра, тип v для вертикальной проекции 1D-сигнала и h для горизонтальной проекции 1D-сигнала. Отрегулируйте фазу с помощью P0 и P1 (дополнительный рисунок 11), запомните разность фаз и скорректируйте фазу в файле nmrproc.com (дополнительный рисунок 10).
ПРИМЕЧАНИЕ: Если спектр перевернут в размерности 15N, удалите -neg в строке: | nmrPipe -fn FT -neg \ (Дополнительный рисунок 10). - Закройте nmrDraw и снова обработайте > nmrproc.com. Для выбора пиков (после того, как спектр будет правильно разделен на фазы и эталонный спектр отобразится с уровнем контура, где присутствуют все пики), введите K (shift + k), нажмите Detect и нажмите Сохранить. (Дополнительный рисунок 11).
- Скопируйте список пиков в рабочий каталог и измените -assName в ass.com на имя списка пиков (дополнительный рисунок 12).
ПРИМЕЧАНИЕ: Пример пик-листа в правильном формате доступен по адресу https://www.ipb.hhu.de/en/teams/team-lakomek/pulsesequences
и в расширенном репозитории Банка биологического магнитного резонанса (BMRB) (bmrbig102). - Выполните > ./ass.com для назначения спектров. Проверьте назначение, которое сценарий предлагает для различных пиков. Используйте левую кнопку мыши для назначения, а правую кнопку мыши для отмены назначения пиков. Используйте кнопку «Далее », чтобы перейти к следующей аминокислоте, и «Предыдущая », чтобы перейти к предыдущей аминокислоте (дополнительный рисунок 13).
ПРИМЕЧАНИЕ: Уровень контура можно изменить с помощью + и - в правом верхнем углу. С помощью кнопки «Рисовать » перерисуйте спектр с новым уровнем контура (дополнительный рисунок 13). - После выполнения задания нажмите «Сохранить », а затем снова «Сохранить ». После этого нажмите « Выйти», «Отмена» и «Выйти » (дополнительный рисунок 13).
- Выполните > NoneDeleter.py python3 , чтобы создать relax.tab, который содержит только информацию о назначенных вершинах.
- Выполните > ./relax.com для создания сценария autofit.com.
- Выполните > ./autoFit.com , чтобы выполнить интерполяцию Фурье и сгенерировать псевдоотношения различных времен задержки для каждой аминокислоты (генерируются axt.tab и nlin.tab ).
- Выполните > ./model.com , чтобы сгенерировать modelExp.com скрипт на основе данных в nlin.tab.
- Выполните > ./modelExp.com для подгонки кривой эволюции каждого остатка с помощью нелинейной модели оптимизации по методу наименьших квадратов.
- Выполните > bash summary.tcl -in relax.tab > t1.tab. Игнорируйте потенциальное сообщение об ошибке: GDB Notice: может произойти 21 пропущенное значение в файле таблицы .
- Скопируйте t1.tab в только что созданную директорию R1R2.
- Обработайте и проанализируйте эксперимент с 15N R1ρ .
- Скопируйте файлы из загруженного каталога R1rho_processing в каталог, содержащий экспериментальные данные R1ρ (рабочий каталог).
- Выполнить > chmod u+x *.com
- Выполните команду > bruker , чтобы открыть утилиту преобразования NMRPipe и сгенерировать файл fid.com. Смотрите также обработку спектров 15N R1 .
- Выполнить > ./fid.com
- Выполните > ./split.com , чтобы разделить псевдо 3D данные на восемь 2D файлов.
- Выполните > ./nmrproc.com (предварительно отредактируйте tauValues [vplist] и idxExpmt ) для обработки необработанных данных всех восьми 2D-спектров (дополнительный рисунок 10).
- Выполните > nmrDraw, чтобы нарисовать спектры и выбрать вершины. Для этих шагов используйте эталонный спектр (первая запись в vplist). Следуйте той же процедуре, что и в эксперименте 15N R1 .
- Скопируйте список пиков в рабочий каталог и измените -assName в ass.com на имя списка пиков (дополнительный рисунок 12).
- Выполните > ./ass.com и выполните ту же процедуру, что и для эксперимента 15N R1 .
- Выполните > NoneDeleter.py python3 , чтобы создать relax.tab, который содержит только информацию о назначенных вершинах.
- Выполнить > ./relax.com
- Выполнить > ./autoFit.com
- Выполнить > ./model.com
- Выполнить > ./modelExp.com
- Выполните > bash summary.tcl -in relax.tab > t1rho. tab. Игнорировать потенциальное сообщение об ошибке: GDB Notice: в файле таблицы может отсутствовать 21 значение .
- Скопируйте следующий заголовок в файл t1rho.tab (значения можно получить из файла fid.com) (Дополнительный рисунок 14):
ПРИМЕЧАНИЕ 600 МГц для T1rho
ПРИМЕЧАНИЕ -yOBS = 60.818 (запишите здесь значение, найденное в fid.com)
ПРИМЕЧАНИЕ -yCAR = 119.06 (поместите сюда значение, найденное в fid.com) - Выполните > ./extract_PPM.com чтобы создать таблицу с именем fusionOffset.tab, содержащую смещение пиковой частоты 15N относительно несущей частоты (что потребуется для вычисления угла θ для извлечения данных R2 из данных R1 и R1ρ).
- Обобщите данные о релаксации.
- Скопируйте t1rho.tab и fusionOffset.tab в каталог R1R2.
- Скопируйте скрипт crtR1p.tcl в директорию R1R2, отрегулируйте частоту спектрометра и мощность блокировки спина, а затем выполните bash crtR1p.tcl | sort -n > R1R2.tab. (Дополнительный рисунок 15). R1R2.tab будет содержать аппроксимированные константы скорости R1ρ , тета-угол, константы скорости R2 и константы скорости R1 .
- Обработайте и проанализируйте эксперимент hetNOE.
- Скопируйте файлы из загруженного каталога NOE_processing в экспериментальный каталог данных NOE (рабочий каталог).
- Выполнить > chmod u+x *.com
- Выполните команду > bruker , чтобы открыть утилиту NMRPipe Conversion Utility и сгенерировать файл fid.com. Нажмите кнопку Прочитать параметры. Измените значение параметра «Количество измерений » на два, если было подсчитано три измерения, и нажмите «Прочитать параметры » еще раз. Затем нажмите «Очистить сценарий», «Обновить сценарий» и «Сохранить сценарий».
- Выполнить > ./fid.com
- Выполнить > ./split.com (Перед этим должен быть создан vplist с двумя записями (0, 1))
- Выполнить > ./ft2.com
- Выполните > nmrDraw, чтобы нарисовать спектр и указать пики в эталонном спектре (z-dimension = 1). Извлеките и запомните шум (нажмите T = shift + t). Фазовая коррекция и выбор пиков выполняются по той же процедуре, что и в эксперименте с 15N R1 .
- Скопируйте список пиков в рабочий каталог и измените -assName в ass.com на имя списка пиков (дополнительный рисунок 12)
- Выполните > ./ass.com для назначения спектров. Следуйте той же процедуре, что и в эксперименте 15N R1 .
- Выполните > python3 NoneDeleter.py , чтобы создать relax.tab.
- Выполнить > ./script.com
- Выполнить > ./autoFit.com
- Изменение шума в error_hetnoe.tcl.
- Execute > bash error_hetnoe.tcl |sort -n > hetNOE.tab
Access restricted. Please log in or start a trial to view this content.
Результаты
Ниже приведены некоторые примеры данных релаксации ЯМР, зарегистрированных на везикулярном белке SNARE Synaptobrevin-2 (1-96), часто называемом VAMP2 (везикулоассоциированный белок 2). Для регистрации данных ЯМР мы использовали образец 171 мкМ 15N Synaptobrevin-2 (1-96) (далее названный Syb-...
Access restricted. Please log in or start a trial to view this content.
Обсуждение
В этом протоколе описывалась постановка экспериментов по релаксации ЯМР 15N, проведенных Lakomek et al.69 и Stief et al.70. Мы сосредоточились на последовательностях импульсов ЯМР с использованием схемы детектирования HSQC с повышенной чувствительно?...
Access restricted. Please log in or start a trial to view this content.
Раскрытие информации
Авторы заявляют, что у них нет известных конкурирующих финансовых интересов или личных отношений, которые могли бы повлиять на работу, описанную в этой статье.
Благодарности
Мы благодарим Мелинду Джасперт и Кевина Бочински за полезные дискуссии. Н.Л. благодарит Немецкий научный фонд за финансирование в рамках программы Гейзенберга (грант DFG No 433700474). Эта работа также поддерживается проектом «Вирусологические и иммунологические детерминанты патогенеза COVID-19 – уроки подготовки к будущим пандемиям (KA1-Co-02 «COVIPA»), грантом Фонда инициатив и сетевого взаимодействия Ассоциации им. Гельмгольца. Мы признательны за щедрый доступ к Юлих-Дюссельдорфскому биомолекулярному ЯМР-центру, совместно управляемому Исследовательским центром Юлиха и Дюссельдорфским университетом имени Генриха Гейне (HHU).
Access restricted. Please log in or start a trial to view this content.
Материалы
| Name | Company | Catalog Number | Comments |
| Bruker 600 MHz AVANCE III HD spectrometer | Bruker | https://www.bruker.com/en/products-and-solutions/mr/nmr/avance-nmr-spectrometer.html | NMR experiments conducted |
Ссылки
- Sekhar, A., Kay, L. E. An NMR view of protein dynamics in health and disease. Annu Rev Biophys. 48, 297-319 (2019).
- Rosenzweig, R., Kay, L. E. Bringing dynamic molecular machines into focus by methyl-TROSY NMR. Annu Rev Biochem. 83, 291-315 (2014).
- Palmer, A. G. 3rd Enzyme dynamics from NMR spectroscopy. Acc Chem Res. 48 (2), 457-465 (2015).
- Reif, B. Ultra-high resolution in MAS solid-state NMR of perdeuterated proteins: implications for structure and dynamics. J Magn Reson. 216, 1-12 (2012).
- Schanda, P., Ernst, M. Studying dynamics by magic-angle spinning solid-state NMR spectroscopy: Principles and applications to biomolecules. Prog Nucl Magn Reson Spectrosc. 96, 1-46 (2016).
- Le Marchand, T., et al. (1)H-H-detected biomolecular NMR under fast magic-angle spinning. Chem Rev. 122 (10), 9943-10018 (2022).
- Pervushin, K., Riek, R., Wider, G., Wuthrich, K. Attenuated T2 relaxation by mutual cancellation of dipole-dipole coupling and chemical shift anisotropy indicates an avenue to NMR structures of very large biological macromolecules in solution. Proc Natl Acad Sci U S A. 94 (23), 12366-12371 (1997).
- Tzakos, A. G., Grace, C. R., Lukavsky, P. J., Riek, R. NMR techniques for very large proteins and RNAs in solution. Annu Rev Biophys Biomol Struct. 35, 319-342 (2006).
- Sprangers, R., Kay, L. E. Quantitative dynamics and binding studies of the 20S proteasome by NMR. Nature. 445 (7128), 618-622 (2007).
- Wright, P. E., Dyson, H. J. Intrinsically disordered proteins in cellular signalling and regulation. Nat Rev Mol Cell Biol. 16 (1), 18-29 (2015).
- van der Lee, R., et al. Classification of intrinsically disordered regions and proteins. Chem Rev. 114 (13), 6589-6631 (2014).
- Tompa, P. Intrinsically unstructured proteins. Trends Biochem Sci. 27 (10), 527-533 (2002).
- Dyson, H. J., Wright, P. E. Coupling of folding and binding for unstructured proteins. Curr Opin Struct Biol. 12 (1), 54-60 (2002).
- Uversky, V. N. Intrinsically disordered proteins in overcrowded milieu: Membrane-less organelles, phase separation, and intrinsic disorder. Current Opinion in Structural Biology. 44, 18-30 (2017).
- Patel, A., et al. A liquid-to-solid phase transition of the ALS protein FUS accelerated by disease mutation. Cell. 162 (5), 1066-1077 (2015).
- Abyzov, A., Blackledge, M., Zweckstetter, M. Conformational dynamics of intrinsically disordered proteins regulate biomolecular condensate chemistry. Chem Rev. 122 (6), 6719-6748 (2022).
- Uversky, V. N. Unusual biophysics of intrinsically disordered proteins. Biochim Biophys Acta. 1834 (5), 932-951 (2013).
- Papoian, G. A. Proteins with weakly funneled energy landscapes challenge the classical structure-function paradigm. Proc Natl Acad Sci U S A. 105 (38), 14237-14238 (2008).
- Uversky, V. N., Gillespie, J. R., Fink, A. L. Why are "natively unfolded" proteins unstructured under physiologic conditions. Proteins. 41 (3), 415-427 (2000).
- Bah, A., et al. Folding of an intrinsically disordered protein by phosphorylation as a regulatory switch. Nature. 519 (7541), 106-109 (2015).
- Tompa, P., Schad, E., Tantos, A., Kalmar, L. Intrinsically disordered proteins: emerging interaction specialists. Curr Opin Struct Biol. 35, 49-59 (2015).
- Dyson, H. J., Wright, P. E. Perspective: the essential role of NMR in the discovery and characterization of intrinsically disordered proteins. J Biomol NMR. 73 (12), 651-659 (2019).
- Adamski, W., et al. A unified description of intrinsically disordered protein dynamics under physiological conditions using NMR spectroscopy. J Am Chem Soc. 141 (44), 17817-17829 (2019).
- Dobson, C. M. Protein folding and misfolding. Nature. 426 (6968), 884-890 (2003).
- Bertoncini, C. W., et al. Release of long-range tertiary interactions potentiates aggregation of natively unstructured alpha-synuclein. Proc Natl Acad Sci U S A. 102 (5), 1430-1435 (2005).
- Buell, A. K., et al. Solution conditions determine the relative importance of nucleation and growth processes in alpha-synuclein aggregation. Proc Natl Acad Sci U S A. 111 (21), 7671-7676 (2014).
- Salvi, N., Abyzov, A., Blackledge, M. Atomic resolution conformational dynamics of intrinsically disordered proteins from NMR spin relaxation. Prog Nucl Magn Reson Spectrosc. 102, 43-60 (2017).
- Schneider, R., Blackledge, M., Jensen, M. R. Elucidating binding mechanisms and dynamics of intrinsically disordered protein complexes using NMR spectroscopy. Curr Opin Struct Biol. 54, 10-18 (2019).
- Rezaei-Ghaleh, N., Blackledge, M., Zweckstetter, M. Intrinsically disordered proteins: from sequence and conformational properties toward drug discovery. Chembiochem. 13 (7), 930-950 (2012).
- Jensen, M. R., Salmon, L., Nodet, G., Blackledge, M. Defining conformational ensembles of intrinsically disordered and partially folded proteins directly from chemical shifts. J Am Chem Soc. 132 (4), 1270-1272 (2010).
- Camacho-Zarco, A. R., et al. NMR provides unique insight into the functional dynamics and interactions of intrinsically disordered proteins. Chem Rev. 122 (10), 9331-9356 (2022).
- Mittag, T., Forman-Kay, J. D. Atomic-level characterization of disordered protein ensembles. Curr Opin Struct Biol. 17 (1), 3-14 (2007).
- Dyson, H. J., Wright, P. E. Unfolded proteins and protein folding studied by NMR. Chem Rev. 104 (8), 3607-3622 (2004).
- Dyson, H. J., Wright, P. E. NMR illuminates intrinsic disorder. Curr Opin Struct Biol. 70, 44-52 (2021).
- Sugase, K., Dyson, H. J., Wright, P. E. Mechanism of coupled folding and binding of an intrinsically disordered protein. Nature. 447 (7147), 1021-1025 (2007).
- Bessa, L. M., et al. The intrinsically disordered SARS-CoV-2 nucleoprotein in dynamic complex with its viral partner nsp3a. Sci Adv. 8 (3), eabm4034(2022).
- Milles, S., Salvi, N., Blackledge, M., Jensen, M. R. Characterization of intrinsically disordered proteins and their dynamic complexes: From in vitro to cell-like environments. Prog Nucl Magn Reson Spectrosc. 109, 79-100 (2018).
- Salvi, N., Abyzov, A., Blackledge, M. Atomic resolution conformational dynamics of intrinsically disordered proteins from NMR spin relaxation. Prog Nucl Magn Reson Spectrosc. 102 - 103, 43-60 (2017).
- Gill, M. L., Byrd, R. A., Palmer, A. G. III Dynamics of GCN4 facilitate DNA interaction: a model-free analysis of an intrinsically disordered region. Phys Chem Chem Phys. 18 (8), 5839-5849 (2016).
- Bertoncini, C. W., et al. Structural characterization of the intrinsically unfolded protein beta-synuclein, a natural negative regulator of alpha-synuclein aggregation. J Mol Biol. 372 (3), 708-722 (2007).
- Rezaei-Ghaleh, N., et al. Local and global dynamics in intrinsically disordered synuclein. Angew Chem Int Ed Engl. 57 (46), 15262-15266 (2018).
- Rezaei-Ghaleh, N., Parigi, G., Zweckstetter, M. Reorientational dynamics of amyloid-beta from NMR spin relaxation and molecular simulation. J Phys Chem Lett. 10 (12), 3369-3375 (2019).
- Palmer, A. G. NMR characterization of the dynamics of biomacromolecules. Chem Rev. 104 (8), 3623-3640 (2004).
- Palmer, A. G., Massi, F. Characterization of the dynamics of biomacromolecules using rotating-frame spin relaxation NMR spectroscopy. Chem Rev. 106 (5), 1700-1719 (2006).
- Kay, L. E., Torchia, D. A., Bax, A. Backbone dynamics of proteins as studied by 15N inverse detected heteronuclear NMR spectroscopy: application to staphylococcal nuclease. Biochemistry. 28 (23), 8972-8979 (1989).
- Mittermaier, A., Kay, L. E. Review - New tools provide new insights in NMR studies of protein dynamics. Science. 312 (5771), 224-228 (2006).
- Abragam, A. Principles of Nuclear Magnetism. , Clarendon Press. Oxford. (1983).
- Ernst, R. R., Bodenhausen, G., Wokaun, A. Principles of Nuclear Magnetic Resonance in One and Two Dimensions. , Oxford University Press. Oxford. (1990).
- Cavanagh, J., Fairbrother, W. J., Palmer, A. G. III, Skelton, N. J. Protein NMR Spectroscopy: Principles and Practice. , Elsevier, Academic Press. (1995).
- Levitt, M. H. Spin Dynamics: Basics of Nuclear Magnetic Resonance. , John Wiley and Sons. (2008).
- Keeler, J. Understanding NMR Spectroscopy. , John Wiley and Sons. (2010).
- Bloembergen, N., Purcell, E. M., Pound, R. V. Relaxation effects in nuclear magnetic resonance absorption. Phys Rev. 73 (7), 679-712 (1948).
- Wangsness, R. K., Bloch, F. The dynamical theory of nuclear induction. Phys Rev. 89 (4), 728-739 (1953).
- Redfield, A. G. On the theory of relaxation processes. IBM J Res Dev. 1 (1), 19-31 (1957).
- Peng, J. W., Wagner, G. Mapping of the spectral densities of N-H bond motions in eglin c using heteronuclear relaxation experiments. Biochemistry. 31 (36), 8571-8586 (1992).
- Farrow, N. A., et al. Backbone dynamics of a free and a phosphopeptide-complexed src homology-2 domain studied by n-15 NMR relaxation. Biochemistry. 33 (19), 5984-6003 (1994).
- Tolman, J. R., Ruan, K. NMR residual dipolar couplings as probes of biomolecular dynamics. Chem Rev. 106 (5), 1720-1736 (2006).
- Mulder, F. A. A., Skrynnikov, N. R., Hon, B., Dahlquist, F. W., Kay, L. E. Measurement of slow (mu s-ms) time scale dynamics in protein side chains by N-15 relaxation dispersion NMR spectroscopy: Application to Asn and Gln residues in a cavity mutant of T4 lysozyme. J Am Chem Soc. 123 (5), 967-975 (2001).
- Tollinger, M., Skrynnikov, N. R., Mulder, F. A. A., Forman-Kay, J. D., Kay, L. E. Slow dynamics in folded and unfolded states of an SH3 domain. J Am Chem Soc. 123 (46), 11341-11352 (2001).
- Akke, M., Palmer, A. G. Monitoring macromolecular motions on microsecond to millisecond time scales by R(1)rho-R(1) constant relaxation time NMR spectroscopy. J Am Chem Soc. 118 (4), 911-912 (1996).
- Loria, J. P., Rance, M., Palmer, A. G. A relaxation-compensated Carr-Purcell-Meiboom-Gill sequence for characterizing chemical exchange by NMR spectroscopy. J Am Chem Soc. 121 (10), 2331-2332 (1999).
- Vallurupalli, P., Bouvignies, G., Kay, L. E. Studying "Invisible" excited protein states in slow exchange with a major state conformation. J Am Chem Soc. 134 (19), 8148-8161 (2012).
- Jeener, J., Meier, B. H., Bachmann, P., Ernst, R. R. Investigation of exchange processes by 2-dimensional NMR-spectroscopy. J Chem Phys. 71 (11), 4546-4553 (1979).
- Palmer, A. G., Kroenke, C. D., Loria, J. P. Nuclear magnetic resonance methods for quantifying microsecond-to-millisecond motions in biological macromolecules. Methods Enzymol. 339, 204-238 (2001).
- Clore, G. M., Iwahara, J. Theory, practice, and applications of paramagnetic relaxation enhancement for the characterization of transient low-population states of biological macromolecules and their complexes. Chem Rev. 109 (9), 4108-4139 (2009).
- Iwahara, J., Clore, G. M. Detecting transient intermediates in macromolecular binding by paramagnetic NMR. Nature. 440 (7088), 1227-1230 (2006).
- Lakomek, N. A., et al. Residual dipolar couplings as a tool to study molecular recognition of ubiquitin. Biochem Soc Trans. 36 (Pt 6), 1433-1437 (2008).
- Johnson, C. N., Libich, D. S. Paramagnetic relaxation enhancement for detecting and characterizing self-associations of intrinsically disordered proteins. J Vis Exp. (175), e63057(2021).
- Lakomek, N. A., Ying, J., Bax, A. Measurement of (1)(5)N relaxation rates in perdeuterated proteins by TROSY-based methods. J Biomol NMR. 53 (3), 209-221 (2012).
- Stief, T., Vormann, K., Lakomek, N. A. Sensitivity-enhanced NMR (15)N R(1) and R(1rho) relaxation experiments for the investigation of intrinsically disordered proteins at high magnetic fields. Methods. 223, 1-15 (2024).
- Kay, L. E., Keifer, P., Saarinen, T. Pure absorption gradient enhanced heteronuclear single quantum correlation spectroscopy with improved sensitivity. J Am Chem Soc. 114 (26), 10663-10665 (1992).
- Geen, H., Freeman, R. Band-selective radiofrequency pulses. J Magn Reson (1969). 93 (1), 93-141 (1991).
- Desvaux, H., Berthault, P. Study of dynamic processes in liquids using off-resonance RF irradiation. Prog Nucl Magn Reson Spectrosc. 35 (4), 295-340 (1999).
- Overhauser, A. W. Polarization of nuclei in metals. Phys Rev. 91 (2), 476-476 (1953).
- Overhauser, A. W. Polarization of nuclei in metals. Phys Rev. 92 (2), 411-415 (1953).
- Kumari, P., Frey, L., Sobol, A., Lakomek, N. A., Riek, R. (15)N transverse relaxation measurements for the characterization of micros-ms dynamics are deteriorated by the deuterium isotope effect on (15)N resulting from solvent exchange. J Biomol NMR. 72 (3-4), 125-137 (2018).
- Karschin, N., Krenek, S., Heyer, D., Griesinger, C. Extension and improvement of the methanol-d(4) NMR thermometer calibration. Magn Reson Chem. 60 (4), 203-209 (2022).
- Sattler, M., Schleucher, J., Griesinger, C. Heteronuclear multidimensional NMR experiments for the structure determination of proteins in solution employing pulsed field gradients. Prog Nucl Magn Reson Spectrosc. 34 (2), 93-158 (1999).
- Ferrage, F., Cowburn, D., Ghose, R. Accurate sampling of high-frequency motions in proteins by steady-state N-{H} nuclear Overhauser effect measurements in the presence of cross-correlated relaxation. J Am Chem Soc. 131 (17), 6048-6049 (2009).
- Delaglio, F., et al. NMRPipe: a multidimensional spectral processing system based on UNIX pipes. J Biomol NMR. 6 (3), 277-293 (1995).
- Skinner, S. P., et al. CcpNmr AnalysisAssign: a flexible platform for integrated NMR analysis. J Biomol NMR. 66 (2), 111-124 (2016).
- Lee, W., Tonelli, M., Markley, J. L. NMRFAM-SPARKY: enhanced software for biomolecular NMR spectroscopy. Bioinformatics. 31 (8), 1325-1327 (2015).
- Lee, W., Rahimi, M., Lee, Y., Chiu, A. POKY: a software suite for multidimensional NMR and 3D structure calculation of biomolecules. Bioinformatics. 37 (18), 3041-3042 (2021).
- Abyzov, A., et al. Identification of dynamic modes in an intrinsically disordered protein using temperature-dependent NMR relaxation. J Am Chem Soc. 138 (19), 6240-6251 (2016).
- Singh, A., Purslow, J. A., Venditti, V. 15N CPMG relaxation dispersion for the investigation of protein conformational dynamics on the micros-ms timescale. J Vis Exp. (170), e62395(2021).
- Gopalan, A. B., Hansen, D. F., Vallurupalli, P. CPMG experiments for protein minor conformer structure determination. Methods Mol Biol. 1688, 223-242 (2018).
- Tugarinov, V., Kay, L. E. Methyl groups as probes of structure and dynamics in NMR studies of high-molecular-weight proteins. Chembiochem. 6 (9), 1567-1577 (2005).
- Lipari, G., Szabo, A. Model-free approach to the interpretation of nuclear magnetic resonance relaxation in macromolecules. 1. Theory and range of validity. J Am Chem Soc. 104 (17), 4546-4559 (1982).
- Lipari, G., Szabo, A. Model-Free Approach to the interpretation of nuclear magnetic-resonance relaxation in macromolecules .2. Analysis of experimental results. J Am Chem Soc. 104 (17), 4559-4570 (1982).
- Clore, G. M., et al. Deviations from the simple two-parameter model-free approach to the interpretation of nitrogen-15 nuclear magnetic relaxation of proteins. J Am Chem Soc. 112 (12), 4989-4991 (1990).
- Lemaster, D. M. Larmor frequency selective model free analysis of protein NMR relaxation. J Biomol NMR. 6 (4), 366-374 (1995).
- Peng, J. W., Wagner, G. Mapping of spectral density functions using heteronuclear NMR relaxation measurements. J Magn Reson (1969). 98 (2), 308-332 (1992).
- Farrow, N. A., Zhang, O. W., FormanKay, J. D., Kay, L. E. Characterization of the backbone dynamics of folded and denatured states of an SH3 domain. Biochemistry. 36 (9), 2390-2402 (1997).
- Khan, S. N., et al. Distribution of pico- and nanosecond motions in disordered proteins from nuclear spin relaxation. Biophys J. 109 (5), 988-999 (2015).
- Smith, A. A., Ernst, M., Meier, B. H., Ferrage, F. Reducing bias in the analysis of solution-state NMR data with dynamics detectors. J Chem Phys. 151 (3), 034102(2019).
- Zumpfe, K., Smith, A. A. Model-free or not. Front Mol Biosci. 8, 727553(2021).
- Robustelli, P., Trbovic, N., Friesner, R. A., Palmer, A. G. Conformational dynamics of the partially disordered yeast transcription factor GCN4. J Chem Theory Comput. 9 (11), 5190-5200 (2013).
- Salvi, N., Abyzov, A., Blackledge, M. Multi-timescale dynamics in intrinsically disordered proteins from NMR relaxation and molecular simulation. J Phys Chem Lett. 7 (13), 2483-2489 (2016).
Access restricted. Please log in or start a trial to view this content.
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеThis article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены