需要订阅 JoVE 才能查看此. 登录或开始免费试用。
Method Article
监测细胞自主的生物钟节律基因表达,利用荧光素酶生物发光记者
摘要
生物钟单个细胞内发挥作用,也就是说,它们是细胞的自我。在这里,我们描述了使用非侵入性,实时荧光素酶为基础的生物发光技术产生的细胞自主的时钟模型的方法。记者细胞提供听话的,功能性的昼夜节律生物学研究的模型系统。
摘要
在哺乳动物中,许多方面的行为和生理的,如睡眠-觉醒周期和肝脏代谢的调节内源性生物钟(审阅1,2)。时间的昼夜节律系统是一个分层的多振荡器网络,与中央的时钟位于视交叉上核(SCN)同步和协调其他额外的SCN和外设时钟1,2。单个细胞的生成和维护的昼夜节律3,4,这些振荡器在有机体份额显着相似的生化负反馈机制的不同组织类型的功能单元。然而,由于在神经元网络级在SCN和有机体一级通过节奏,系统性的线索的相互作用,在有机体一级的昼夜节律不一定细胞自主5-7。相对于传统的研究在体内的自发活动和SCN外植体离体 ,CELL-总部设在体外实验中可以发现的细胞自主的昼夜缺陷5,8。在战略上,基于细胞的实验模型更易于处理的表型特征和基本时钟机制的快速发现5,8-13。
因为昼夜节律是动态的,纵向的高时间分辨率的测量来评估时钟功能。近年来,实时使用萤火虫荧光素酶的生物发光记录,记者已成为一种常见的技术研究在哺乳动物中的昼夜节律14,15,因为它允许分子节奏的持久性和动态检查。细胞的自我监控的昼夜节律基因表达,荧光素酶记者可以被引入到细胞通过瞬时转染13,16,17或稳定转导5,10,18,19。在这里,我们描述了一个稳定传导协议的慢病毒介导的基因传递。 Ţ他慢病毒载体系统是优于传统的方法,如瞬时转染和生殖传输,因为它的效率和多功能性:它可以有效地提供稳定整合到宿主基因组的分裂和非分裂细胞20。记者细胞系建立之后,时钟功能的动态可以检查通过生物发光记录。我们首先介绍了新一代的P(Per2基因 )-D 吕克记者的线条,然后和其他昼夜记者从目前的数据。在这些实验中,3T3小鼠成纤维细胞和U2OS人骨肉瘤细胞被用作细胞模型。我们还讨论了不同的方式使用这些时钟模型在昼夜节律的研究。这里描述的方法可以应用到多种类型的细胞,研究细胞和分子基础的生物钟,并在处理其他生物系统的问题可能会被证明是有用的。
研究方案
1。构建慢病毒荧光素酶记者
哺乳动物的昼夜记者的结构通常包含一个表达盒,其中一昼夜的荧光素酶基因启动子的融合。双方结扎和重组基于策略通常用于DNA克隆。作为一个例子,在这里,我们描述了一个重组的Gateway克隆方法产生一个P(Per2基因 )-D 吕克慢记者,在不稳定的荧光素酶 (D 吕克 )的小鼠Per2基因启动子的控制下。
- Per2基因启动子的克隆。使用PCR扩增的Per2基因的启动子的526 bp的DNA片段,从小鼠Per2基因的BAC克隆9-13的转录起始位点的上游侧,用正向引物(的5'-CTCGAGCGGATTACCGAGGCTGGTCACG TC-3')和反向引物(5' CTCGAGTCCCTTGCTCGGCCCGTCAC TTGG-3'),并克隆入pENTR5'-TOPO载体(Invitrogen)中,以产生pENTR5'-P(Per2基因 )。
- 克隆的D 吕克 。的d 吕克含有萤火虫荧光素酶基因的C-末端的病虫害如先前描述的21序列的快速蛋白质降解。使用PCR扩增的d 吕克的DNA片段,并克隆到pENTR / D-TOPO载体(Invitrogen)的生成pENTR / Dd的吕克 。
- 报告载体的构建。混合使用这两种pENTR的质粒,pENTR5'-P(Per2基因 )和pENTR / Dd的吕克,与慢病毒的目标向量pLV7-BSD的(BSD的 ,杀稻瘟菌素抗性基因),并执行使用Clonase生成pLV7-BSD的 -P的重组反应(Per2基因 )的-d 吕克记者( 图1)。 pLV7-BSD是一个修改后的版本(在我们的实验室)pLenti6/R4R2/V5-DEST(Invitrogen公司)的土拨鼠肝炎病毒转录后调节元件(WPRE)序列22个直接插入下游的表达CA ssette以增强基因的表达。
2。慢病毒颗粒的生产
1。种子293T细胞(1天)
- 90-100%汇合在定期的DMEM补充有10%FBS和1x青霉素 - 链霉素-谷氨酰胺(PSG)的10厘米培养皿上培养人类胚胎肾(HEK)293T细胞。 (快速生长的细胞与低通道数的高效转染的关键。)
- 之前,通过添加0.001%聚-L-赖氨酸在PBS中的1毫升到各孔中,并在室温下孵育20分钟,转染,涂层6孔培养板中接种细胞。吸取溶液,并在使用前用1×PBS冲洗一次。
- 游离293T细胞,用胰蛋白酶和种子0.75×10 6个细胞的各孔上的预涂层钢板用2ml的DMEM定期。旋流板彻底的各孔中的细胞,以获得均匀的分布。在孵化器中于37℃培养过夜的细胞成长。
- 观察从第1天的种子细胞。细胞应达到80%至90%的汇合点。
- 准备质粒转染混合物在1.5 ml离心管中,加入2微克记者质粒DNA的慢病毒( 例如,pLV7-P(Per2基因 )-D 吕克·刘实验室)和3个包装载体(1.3微克的加格/波尔,0.5微克牧师和0.7微克VSVG Invitrogen公司)。另外,作为对照转染和随后的感染,我们通常包括一个额外的井慢病毒GFP表达载体的转染,pLV156-CMV-EGFP( 图1A),窝藏增强型绿色荧光蛋白(EGFP)的CMV启动子的控制下如前所述20。
- 加入100μl0.25 M 氯化钙 (稀释后用DNase / RNase-free的DDH 2 O从2.5 M股票)在第2步中的质粒混合调匀。然后加入100μl的2倍BBS溶液(50 mM的BES,280 mM氯化钠,1.5毫米的Na 2 HPO 4,PH值为6.95),轻轻地混合,但彻底。的DNA混合,在室温下孵育15分钟。
- 在等待过程中,从293T细胞吸出培养基,并更改为2毫升新鲜培养基。板返回到至少10分钟,平衡介质的pH值在转染前的孵化器。
- 染293T细胞一滴一滴从第3步组合。轻轻旋转板,并在显微镜下观察颗粒的形成。孵育,5%CO 2,37℃培养过夜。 (投诉警察课4 / DNA沉淀物细粒子形成的高效转染的关键。)
3。收获病毒颗粒(天3-4)
- 关于转染后16小时(3天),细胞应达到100%汇合,从细胞中吸出培养基,,更换2毫升新鲜定期DMEM。孵育,37℃培养过夜。
- 第4天,通过观察EGFP表达的转录评估转染效率sfection对照组(转染效率90-100%高EGFP表达的是一个病毒准备好一个可靠的预测。)
- 收集培养基中分泌,感染性病毒颗粒。离心> 2,000 xg离心5分钟以除去残留的293T细胞,并收集含病毒上清液。可替代地,介质也可以用0.45μm的膜过滤器清零。准备用于在感染的病毒颗粒。
3。感染的3T3细胞
1。种子3T3细胞(第3天)
第二天分割和种子适当的数字(12000)的3T3细胞在12孔板中获得20%至30%汇合。孵育,37℃培养过夜。
2。感染3T3细胞(第4天)
- 观察种子细胞。合流的20-30%(小于50%)所需的感染。
- 加入聚凝胺(polybrene)至终浓度为5微克/毫升到所收集的培养基中病毒颗粒。移液拌匀。
- 吸液介质从3T3细胞,上述病毒的混合物,每孔加入1毫升。孵育,37℃培养过夜。 (聚凝胺的使用,以提高感染效率,但不是绝对必需的,正如它可能是有毒的一些细胞,建议事先测试)。
3。选择受感染的细胞(第5天及以后)
- 二十四小时,感染后,吸出含有从受感染的细胞的病毒和聚凝胺(polybrene)的培养基中,用1×PBS洗一次,并改变到新鲜培养基。孵育在37°C过夜复苏和增长。
- 当汇合(一般1-2天),分裂的细胞,在37°C过夜孵育。
- 翌日,吸出培养基,细胞(<50%汇合需要的话)和含10μg/ ml的杀稻瘟菌素选择稳定地用新鲜培养基更换转导的细胞。 (杀稻瘟菌素杀曲线需要凭经验确定为特定的细胞系。)
4。记者细胞的生物发光的记录
1。种子记者细胞
传播稻瘟素抗性记者细胞分裂到35毫米的培养皿。在37°C孵育,直至汇合。我们通常准备≥3个菜,每一个记者的细胞在各种条件下昼夜表型。
2。同步和变更记录介质
- 吸出培养基从汇合记者细胞,用PBS洗一次,更换含10μMforskolin的(或200 nm地塞米松)。在37℃下孵育1小时,以同步的细胞。 (或者,细胞可以通过温度循环23或血清休克24同步)。
- 在等待时,作好记录3T3细胞如下:1倍DMEM(HyClone)的含有10%FBS,1×笔/链霉素/谷氨酰胺,1μM福斯克林,1mM的荧光素,25mM的HEPES,pH值7.4的介质。血清和毛喉素浓度可以凭经验确定。对于非常暗淡的细胞,无酚红培养基也可使用。
- 毛喉素治疗结束时,吸出培养基并更换新鲜配制的记录介质。
3。记者细胞的生物发光记录
- 介质的变化,包括培养皿,用40毫米的无菌盖玻片和密封的地方,真空润滑脂,以防止水分蒸发。
- 加载到的LumiCycle光度计,这是保持在36°C孵化器内无H 2 O或CO 2的菜。
- 启动实时生物发光的记录。我们通常记录的节奏每周1次,其次是介质的变化和第二周的连续记录(见,萨韦利耶夫等人 。详细信息)25。 (96我们的记录会板,协同SL2被用作记录装置,请参阅详细讨论1.1。)
5。数据分析和演示
记者细胞促进高清晰度的定量发光记录,确定表型的影响生物钟功能的关键。为了获得昼夜参数包括相位,周期长,节奏振幅,和阻尼率,我们使用的LumiCycle分析的程序(Actimetrics)分析生物发光数据5,14。简言之,原始数据是基线拟合的第一,和基线减去的数据被装配到一个正弦波,从该参数被确定。表明持久性的节奏,善良的拟合>通常达到90%的样品。由于高瞬态生物发光换液时,我们通常不包括数据分析的第一个周期。
有关数据显示,我们通常绘制原始数据(生物发光,计数/秒)对TI我(天)。在必要时,可以绘制基线减去数据用来比较的振幅和相位。
6。代表性的成果
1。特定相位的昼夜记者
生物钟是根据上的生化负反馈机制1。核心的反馈环路的转录激活因子BMAL1,CLOCK,和阻遏市盈率及晶体,产生有节奏的基因表达(早晨阶段, 例如 , 牧师雇员再培训局α)作用于昼夜E / E'箱的增强子元件。的核心循环调节和集成所述至少两个其它的昼夜顺式元素,DBP/E4BP4结合元件(D-盒;天的阶段, 例如,Per3的 )和ROR / REV-ERB结合元件(RRE;晚相例如,BMAL1)17。规管的多个昼夜元素的组合可以产生新的中间阶段。例如,转录因子CRY1tion是介导的所有三个昼夜的元素( 即 E / E'框和D-BOX元素中的第一个内含子的CRY1基因的启动子和RRES),从而引发不同的CRY1晚上的时间第13期。
这些基因表达调控的机制的基础上,我们产生了四种不同的报告结构:P( 肺癌 )-D 吕克·P(CRY1)-D吕克记者的E / E'框和D-box元件在监管区17, 26,27 P(CRY1) - 内含子 -D 吕克所有三个元素的组合调控( 即 E / E'箱,D-BOX,RRE)13,17和P(BMAL1)-D吕克·稳压完全由:RRE 9,17,19,21。我们这些记者介绍3T3细胞产生预期的不同阶段的记者表达( 图2)。
2。通过RNA干扰基因敲除和药理学激活已经化合物
当转染效率是高的,合成的siRNA可以瞬时转染到细胞中基因表达击倒。当转染在技术上是困难的,可以是一个shRNA表达载体稳定转导通过慢病毒感染的细胞,这样的shRNA处理由细胞产生的基因敲除(KD)的siRNA。在这里,我们介绍KD CRY1和CRY2基因的siRNA在U2OS细胞( 图3A)和shRNA使用pLL3.7网关表达载体( 图3B)在3T3细胞的影响。除了3T3细胞,,U2OS模式已成为另一个卓越的细胞时钟模型,主要是因为它符合市售的人siRNA库( 例如 ,人类的起源,能够产生强大的昼夜节律,验证功能的高通量筛选的关键要求所有已知的时钟基因,可以高效转染和定量的发光录制)。在这两种类型的细胞中,RNA干扰介导KD导致的时钟表型符合先前的小鼠基因敲除(KO)和细胞KD研究5,10,11,28。例如,的CRY1 KD缩短周期长度和降低节奏持久性,而CRY2 KD延长期。此外,选定的小分子可以使用药理学目标和干扰的蛋白质的功能( 图3C)。
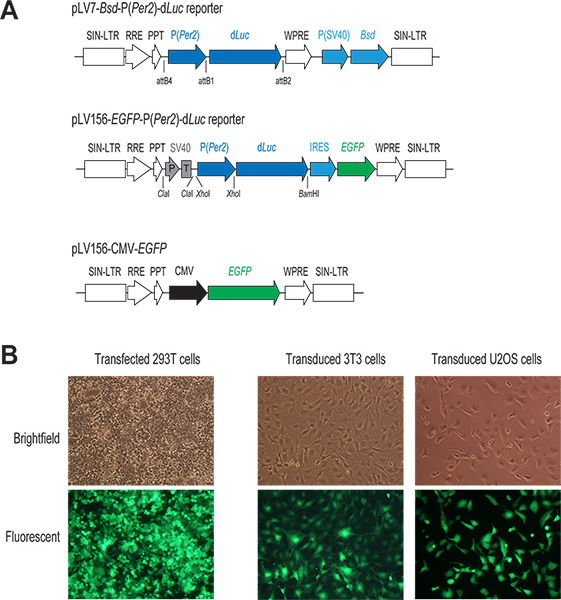
图1。慢病毒载体介导的基因传递系统。 (A)示意图2慢P( 肺癌 )-D 吕克记者载体和CMV-EGFP。只有整合到宿主细胞的基因组的区域被示出。在这两个记者的结构,直接控制下的Per2基因启动子转录的D 吕克 。在pLV7-BSD-P(Per2基因 )-D 吕克载体(重新的组合为基础的克隆),共表达的杀稻瘟菌素抗性基因(bsd)促进感染的细胞的选择。 -D: 吕克向量(基于结扎的克隆)在pLV156-P(Per2基因 ),绿色荧光蛋白翻译是由一个内部核糖体进入位点(IRES)的d 吕克下游介导的,允许的视觉观察和感染细胞的FACS分类。此外,使用SV40启动子/终止子(P / T)作为绝缘体(见讨论1.3)。在CMV-EGFP的对照载体,一个强大的CMV启动子控制下的EGFP表达(B)感染与转染表达GFP的细胞的荧光图像。通常情况下,我们实现了高效率,无论是在瞬时转染293T细胞,在慢病毒感染的细胞株,我们的利益,在这些细胞中的GFP表达。 点击此处查看大图 。
图2。第一阶段特异性表达的3T3细胞中的生物发光记者。记者的慢病毒载体在这个实验中pLV7-BSD-P(Per2基因 ),P-D 吕克(CRY1)-D,P(CRY1) - 吕克内含子 -D 吕克 ,和P(BMAL1)-D吕克 。每个记者表现出明显的振荡相位,如箭头所指示的。虽然肺癌和 cry1发起人驱动器峰值生物发光早上天相和BMAL1子在夜间阶段,组合调整的P(CRY1) - 内含子窝藏的E-box,D-BOX,和RRE元素的赋予晚上阶段的峰值生物发光点击这里查看大图 。
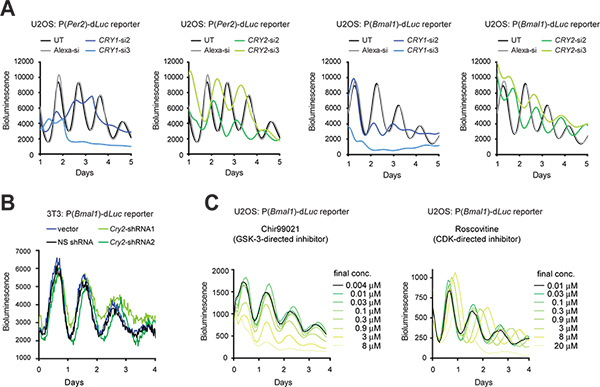
图3。记者的昼夜生物发光的节奏在细胞遗传学和药理学的扰动。 (A)CRY1和CRY2击倒siRNA对细胞节律的U2OS记者细胞的影响。 LumiCycle光度计用于在35 mm培养皿中的细胞生物发光的记录。图适用 从参考第10,爱思唯尔(2009年)的许可。(B)CRY2击倒的shRNA对细胞节律的3T3记者细胞的影响。 CRY2基因敲除一个pLL3.7 Gateway载体包含一个U6-shRNA组磁带的使用。 shRNA2有一个更好的Western blot分析(数据未示出)所确定的效率比shRNA1击倒。和合光度计用于生物发光记录细胞在96孔板中。记录的设置如下:孵化温度33°C;积分时间,15秒,间隔时间30分钟。(C)的小分子抑制剂的影响CEL的U2OS记者细胞的lular的节奏。 Chir99021和roscovitine处理,分别针对GSK-3和CDK抑制剂。用于生物发光的细胞录音ViewLux的系统(Chir99021测定)和Tecan公司光度计(roscovitine处理测定)在384孔板中。图改编自“参考编号#19(版权2008年美国国家科学院,美国), 点击这里查看大图 。
讨论
1。目前的协议的修改
1.1记录装置和吞吐量的考虑
由于其商用中,LumiCycle(Actimetrics)已成为最常用的自动光度计设备实时记录4,5,9,19,29-31的 。 LumiCycle采用光电倍增管(PMT)作为光探测器,它提供了极高的灵敏度和低噪声14,因此特别适合于数据采集极其暗淡的基于荧光素酶发光。其他相似的PMT的设备( 例如 ,克罗诺斯,阿托公司; POL...
披露声明
没有利益冲突的声明。
致谢
这项工作是支持的,部分由美国国家科学基金会(IOS-0920417)(ACL)。
材料
| 试剂名称 | 公司 | 目录编号 | 评论 |
| DMEM | 胎牛血清 | SH30243FS | 对于正常细胞生长 |
| DMEM | Invitrogen公司 | 12100-046 | 对于luminometry |
| FBS | 胎牛血清 | SH3091003 | |
| 青/链霉素/谷氨酰胺(100X) | 胎牛血清 | SV3008201 | |
| B-27 | Invitrogen公司 | 17504-044 | |
| D-荧光素 | Biosynth | L-8220 | |
| 聚-L-赖氨酸 | 西格玛 | P4707 | |
| 聚凝胺 | 密理博 | TR-1003-G | |
| 佛司可林 | 西格玛 | F6886 | |
| 所有其他化学品 | 西格玛 | ||
| 设备 | |||
| 组织培养箱 | 在37°C,5%CO2 | ||
| 组织培养罩 | BSL-2认证 | ||
| 光与荧光显微镜 | 相衬可选 | ||
| LumiCycle | Actimetrics |
参考文献
- Reppert, S. M., Weaver, D. R. Coordination of circadian timing in mammals. Nature. 418, 935-941 (2002).
- Hastings, M. H., Reddy, A. B., Maywood, E. S. A clockwork web: circadian timing in brain and periphery, in health and disease. Nat. Rev. Neurosci. 4, 649-661 (2003).
- Nagoshi, E. Circadian gene expression in individual fibroblasts: cell-autonomous and self-sustained oscillators pass time to daughter cells. Cell. 119, 693-705 (2004).
- Welsh, D. K. Bioluminescence imaging of individual fibroblasts reveals persistent, independently phased circadian rhythms of clock gene expression. Curr. Biol. 14, 2289-2295 (2004).
- Liu, A. C. Intercellular coupling confers robustness against mutations in the SCN circadian clock network. Cell. 129, 605-616 (2007).
- Kornmann, B. System-driven and oscillator-dependent circadian transcription in mice with a conditionally active liver clock. PLoS Biol. 5, e34 (2007).
- Hogenesch, J. B., Herzog, E. D. Intracellular and intercellular processes determine robustness of the circadian clock. FEBS Lett. 585, 1427-1434 (2011).
- DeBruyne, J. P., Weaver, D. R., Reppert, S. M. Peripheral circadian oscillators require CLOCK. Curr. Biol. 17, 538-539 (2007).
- Liu, A. C. Redundant function of REV-ERBalpha and beta and non-essential role for Bmal1 cycling in transcriptional regulation of intracellular circadian rhythms. PLoS Genet. 4, e1000023 (2008).
- Zhang, E. E. A genome-wide RNAi screen for modifiers of the circadian clock in human cells. Cell. 139, 199-210 (2009).
- Baggs, J. E. Network features of the mammalian circadian clock. PLoS Biol. 7, e52 (2009).
- Hirota, T. High-throughput chemical screen identifies a novel potent modulator of cellular circadian rhythms and reveals CKIalpha as a clock regulatory kinase. PLoS Biol. 8, e1000559 (2010).
- Ukai-Tadenuma, M. Delay in feedback repression by cryptochrome 1 is required for circadian clock function. Cell. 144, 268-281 (2011).
- Yamazaki, S., Takahashi, J. S. Real-time luminescence reporting of circadian gene expression in mammals. Methods Enzymol. 393, 288-301 (2005).
- Welsh, D. K., Imaizumi, T., Kay, S. A. Real-time reporting of circadian-regulated gene expression by luciferase imaging in plants and mammalian cells. Methods Enzymol. 393, 269-288 (2005).
- Sato, T. K. Feedback repression is required for mammalian circadian clock function. Nat. Genet. 38, 312-319 (2006).
- Ueda, H. R. System-level identification of transcriptional circuits underlying mammalian circadian clocks. Nat. Genet. 37, 187-192 (2005).
- Brown, S. A. The period length of fibroblast circadian gene expression varies widely among human individuals. PLoS Biol. 3, e338 (2005).
- Hirota, T. A chemical biology approach reveals period shortening of the mammalian circadian clock by specific inhibition of GSK-3beta. Proc. Natl. Acad. Sci. U.S.A. 105, 20746-20751 (2008).
- Tiscornia, G., Singer, O., Verma, I. M. Production and purification of lentiviral vectors. Nat. Protoc. 1, 241-245 (2006).
- Ueda, H. R. A transcription factor response element for gene expression during circadian night. Nature. 418, 534-539 (2002).
- Zufferey, R., Donello, J. E., Trono, D., Hope, T. J. Woodchuck hepatitis virus posttranscriptional regulatory element enhances expression of transgenes delivered by retroviral vectors. J. Virol. 73, 2886-2892 (1999).
- Buhr, E. D., Yoo, S. H., Takahashi, J. S. Temperature as a universal resetting cue for mammalian circadian oscillators. Science. 330, 379-385 (2010).
- Balsalobre, A., Damiola, F., Schibler, . U.A serum shock induces circadian gene expression in mammalian tissue culture cells. Cell. 93, 929-937 (1998).
- Savelyev, S. A., Larsson, K. C., Johansson, A., Lundkvist, G. B. S. Slice Preparation, Organotypic Tissue Culturing and Luciferase Recording of Clock Gene Activity in the Suprachiasmatic Nucleus. J. Vis. Exp. (48), e2439 (2011).
- Akashi, M., Ichise, T., Mamine, T., Takumi, T. Molecular mechanism of cell-autonomous circadian gene expression of Period2, a crucial regulator of the mammalian circadian clock. Mol. Biol. Cell. 17, 555-565 (2006).
- Ohno, T., Onishi, Y., Ishida, N. A novel E4BP4 element drives circadian expression of mPeriod2. Nucleic Acids Res. 35, 648-655 (2007).
- Maier, B. A large-scale functional RNAi screen reveals a role for CK2 in the mammalian circadian clock. Genes Dev. 23, 708-718 (2009).
- Yoo, S. H. PERIOD2::LUCIFERASE real-time reporting of circadian dynamics reveals persistent circadian oscillations in mouse peripheral tissues. Proc. Natl. Acad. Sci. U.S.A. 101, 5339-5346 (2004).
- Liu, A. C., Lewis, W. G., Kay, S. A. Mammalian circadian signaling networks and therapeutic targets. Nat. Chem. Biol. 3, 630-639 (2007).
- Ko, C. H. Emergence of noise-induced oscillations in the central circadian pacemaker. PLoS Biol. 8, e1000513 (2010).
- Izumo, M., Johnson, C. H., Yamazaki, S. Circadian gene expression in mammalian fibroblasts revealed by real-time luminescence reporting: temperature compensation and damping. Proc. Natl. Acad. Sci. U.S.A. 100, 16089-16094 (2003).
- Izumo, M., Sato, T. R., Straume, M., Johnson, C. H. Quantitative analyses of circadian gene expression in mammalian cell cultures. PLoS Comput. Biol. 2, e136 (2006).
- Chen, Z. Identification of diverse modulators of central and peripheral circadian clocks by high-throughput chemical screening. Proc. Natl. Acad. Sci. U.S.A. 109, 101-106 (2011).
- Yamaguchi, S. Synchronization of cellular clocks in the suprachiasmatic nucleus. Science. 302, 1408-1412 (2003).
- Akashi, M., Hayasaka, N., Yamazaki, S., Node, K. Mitogen-activated protein kinase is a functional component of the autonomous circadian system in the suprachiasmatic nucleus. J. Neurosci. 28, 4619-4623 (2008).
- Hoshino, H., Nakajima, Y., Ohmiya, Y. Luciferase-YFP fusion tag with enhanced emission for single-cell luminescence imaging. Nat. Methods. 4, 637-639 (2007).
- Asai, M. Visualization of mPer1 transcription in vitro: NMDA induces a rapid phase shift of mPer1 gene in cultured SCN. Curr. Biol. 11, 1524-1527 (2001).
- Wilsbacher, L. D. Photic and circadian expression of luciferase in mPeriod1-luc transgenic mice in vivo. Proc. Natl. Acad. Sci. U.S.A. 99, 489-494 (2002).
- Yamazaki, S. Resetting central and peripheral circadian oscillators in transgenic rats. Science. 288, 682-685 (2000).
- Welsh, D. K., Noguchi, T., Yuste, R. Cellular bioluminescence imaging. Imaging: A Laboratory Manual. , 369-385 (2011).
- Nakajima, Y. Enhanced beetle luciferase for high-resolution bioluminescence imaging. PLoS One. 5, e10011 (2010).
- Guilding, C. A riot of rhythms: neuronal and glial circadian oscillators in the mediobasal hypothalamus. Mol. Brain. 2, 28 (2009).
- O'Neill, J. S. cAMP-dependent signaling as a core component of the mammalian circadian pacemaker. Science. 320, 949-953 (2008).
- Fuller, P. M., Lu, J., Saper, C. B. Differential rescue of light- and food-entrainable circadian rhythms. Science. 320, 1074-1077 (2008).
- Mukherjee, S. Knockdown of Clock in the ventral tegmental area through RNA interference results in a mixed state of mania and depression-like behavior. Biol. Psychiatry. 68, 503-511 (2010).
- Saijo, K. A Nurr1/CoREST pathway in microglia and astrocytes protects dopaminergic neurons from inflammation-induced death. Cell. 137, 47-59 (2009).
- Elias, G. M. Synapse-specific and developmentally regulated targeting of AMPA receptors by a family of MAGUK scaffolding proteins. Neuron. 52, 307-320 (2006).
- Isojima, Y. CKIepsilon/delta-dependent phosphorylation is a temperature-insensitive, period-determining process in the mammalian circadian clock. Proc. Natl. Acad. Sci. U.S.A. 106, 15744-15749 (2009).
- Bucan, M., Abel, T. The mouse: genetics meets behaviour. Nat. Rev. Genet. 3, 114-123 (2002).
- Hughes, M. E. Harmonics of circadian gene transcription in mammals. PLoS Genet. 5, e1000442 (2009).
- Atwood, A. Cell-autonomous circadian clock of hepatocytes drives rhythms in transcription and polyamine synthesis. Proc. Natl. Acad. Sci. U.S.A. 108, 18560-18565 (2011).
- Panda, S. Coordinated transcription of key pathways in the mouse by the circadian clock. Cell. 109, 307-320 (2002).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。