このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
ルシフェラーゼ生物発光レポーターを用いた遺伝子発現のモニタリング細胞自律的な概日時計のリズム
要約
概日時計は、個々の細胞内で機能する、すなわち、それらは細胞自律的である。ここでは、非侵襲的、ルシフェラーゼベースのリアルタイム生物発光技術を用いた細胞自律的なクロックモデルを生成するための方法を説明します。レポーター細胞は、概日生物学を研究するのに扱いやすい、機能的なモデル系を提供する。
要約
哺乳動物では、行動や睡眠覚醒サイクルと肝臓代謝などの生理機能の多くの側面は、内因性の概日時計(1,2日 )によって規制されている。サーカディアン計時システムは、同期や他の場所で1,2エクストラSCNおよび周辺クロックを調整する視交叉上核(SCN)に位置し、中央のクロックで、階層的なマルチ発振器ネットワークです。個々の細胞は、概日リズム3,4、および生物シェアの異なる組織型著しく類似した生化学的な負のフィードバック機構のこれらの発振器の生成と維持のための機能部である。しかし、SCNの神経ネットワークレベルおよび生物体レベルでリズミカルな、全身の手がかりを通じて相互作用のために、生物体レベルで概日リズムが細胞自律的な5-7というわけではありません。 生体とSCNの外植片のex vivo、cの自発運動の伝統的な研究に比べエルベースインビトロアッセイは、細胞自律的な概日5,8欠陥の発見を可能にします。戦略的には、細胞ベースのモデルは、表現型の特徴と基本的な時計機構5,8-13の迅速な発見のために、より実験的に扱いやすいです。
概日リズムは動的であるため、高い時間分解能で縦測定は、時計機能を評価する必要があります。近年では、レポーターとしてホタルルシフェラーゼを用いたリアルタイム生物発光の記録は、それが分子リズムの永続性とダイナミクスの検討を可能にするように、哺乳類14,15の概日リズムを研究するための一般的な手法となっています。遺伝子発現の細胞自律的な概日リズムを監視するには、ルシフェラーゼレポーターは、一過性のトランスフェクション13,16,17または安定した伝達5,10,18,19を介して細胞内に導入することができる。ここでは、レンチウイルス媒介遺伝子送達を用いて安定した伝達プロトコルを記述します。 T彼は、レンチウイルスベクターシステムは、その効率性と汎用性の一過性トランスフェクション及び生殖系列伝達など、従来の方法よりも優れている:それは分割し、非分裂細胞20の両方の宿主ゲノムへの効率的な配信と安定した統合を可能にします。レポーター細胞株が確立されると、時計機能のダイナミクスは、生物発光の記録を使って調べることができます。我々は最初のP(PER2)-D Lucレポーターライン、その後これと他のサーカディアン記者から存在するデータの生成を説明します。これらのアッセイにおいて、3T3マウス線維芽細胞およびU2OSヒト骨肉腫細胞は細胞モデルとして使用されています。我々はまた、概日の研究では、これらのクロックモデルのさまざまな使用方法について説明します。ここで説明する方法は、概日時計の細胞·分子レベルで研究する細胞種の多種多様に適用することができ、かつ他の生物系で問題に取り組む上で役立つことがあります。
プロトコル
1。レンチウイルスルシフェラーゼレポーターの構築
哺乳類の概日レポーターコンストラクトは、通常の概日プロモーターがルシフェラーゼ遺伝子と融合された発現カセットを含んでいます。両方のライゲーションと組み換えベースの戦略は、一般的にDNAクローニングのために使用されます。例として、ここでは、不安定化ルシフェラーゼ (D リュック ) はマウスPER2プロモーターの制御下にあるP(PER2)-Dリュックレンチウイルスレポーターを生成するための組換えベースのGatewayクローニング法を説明します。
- PER2プロモーターのクローニング。フォワードプライマー(5'-CTCGAGCGGATTACCGAGGCTGGTCACG TC-3 ')およびリバースプライマー(5'を使用して、上流のマウスPER2 BACクローン9月13日からの転写開始部位の、526 bpのPER2プロモーターDNA断片を増幅するPCRを使用して、 -CTCGAGTCCCTTGCTCGGCCCGTCAC TTGG-3 ')、および生成するpENTR5'-TOPOベクター(Invitrogen)にクローンpENTR5'-P(PER2)。
- D リュックのクローニング。 D リュックは、前述の 21として急速なタンパク質分解のためにホタルルシフェラーゼ遺伝子とC末端PEST配列を含む。 pENTR / DD リュックを生成するためにpENTR / D-TOPOベクター(Invitrogen)にD リュックの DNA断片、およびクローンを増幅するPCRを使用しています。
- レポーターベクターの構築。レンチウイルスデスティネーションベクタpLV7-BSD(BSD、ブラストサイジン耐性遺伝子)を持つ2つのpENTRプラスミド、pENTR5'-P(PER2)とpENTR / DD リュックを混ぜ、pLV7-BSD-Pを生成するクロナーゼを使用して組換え反応を行う(PER2)-D Lucレポーター( 図1)。 pLV7-BSDは、式Caのすぐ下流に挿入された中でウッドチャック肝炎ウイルスpLenti6/R4R2/V5-DESTの修正版(我々の研究室で行われた)(Invitrogen)を用い、転写後調節エレメント(WPRE)配列22です遺伝子の発現を増強するssette。
2。レンチウイルス粒子の産生
1。シード293T細胞(1日目)
- 10cmの培養皿に10%FBSおよび1×ペニシリン - ストレプトマイシン - グルタミン(PSG)を補充したDMEM中でレギュラー90〜100%コンフルエントになるヒト胚性腎(HEK)293T細胞を成長させる。 (低継代数とともに急速に増殖している細胞は、効率的なトランスフェクションのために重要です。)
- 前にPBS中で各ウェルにポリ-L-リジン0.001%の1mlを添加し、20分間室温でインキュベートすることにより、トランスフェクションのために細胞を播種コート6ウェル培養プレートに。液を除き、使用前に1×PBSで一回すすいでください。
- 2ミリリットルレギュラーDMEMでプレコートしたプレートの各ウェルにトリプシンとシードと293T細胞0.75×10 6個の細胞を取り除きます。徹底的に渦巻プレート各ウェル内の細胞の均一な分布を得るために。 37℃で一晩インキュベーター内で細胞を成長させる。
- 1日目から、播種された細胞を観察します。細胞は80〜90%のコンフルエンスに到達する必要があります。
- と3パッケージングベクター(1.3μgのギャグ/ POL、0.5μgの、レンチウイルスレポータープラスミドDNA(劉ラボなどpLV7-P(PER2)-Dリュック )の2μgを追加することで、1.5 mlのマイクロチューブにプラスミドをトランスフェクション混合物を準備REV、および0.7μgのVSVG、Invitrogen社)。トランスフェクションとその後の感染の両方のコントロールとして、我々は通常、CMVプロモーターの制御下で増強された緑色蛍光タンパク質(EGFP)を保有するレンチウイルスGFP発現ベクターのトランスフェクションの追加だけでなく、pLV156-CMV-EGFP( 図1A)を含み、 20前述したように。
- ステップ2のプラスミドミックスに、0.25MのCaCl 2(2.5MストックからのDNase / RNaseフリーddH 2 Oで希釈したもの)を100μlを加えてよく混合します。その後、2倍のBBS溶液(50mM BE100μlを加えるS、280 mMのNaCl、1.5mMののNa 2 HPO 4、pHは6.95)と、優しく、しかし、完全に混合する。 15分間室温でDNAミックスをインキュベートする。
- 待っている間に、293T細胞から培地を吸引し、2 mlの新鮮な培地に変更します。トランスフェクションの前に培地のpHを平衡化するために、少なくとも10分間インキュベーターにプレートを返す。
- ドロップによる293T細胞降下に手順3からトランスフェクション混合物を追加します。渦巻優しくプレート、顕微鏡下で粒子形成を観察します。 CO 2、37℃で一晩5%でインキュベートする。 (カポ4 / DNA沈殿物の微粒子形成が効率的なトランスフェクションのために重要です。)
3。収穫ウイルス粒子(日3-4)
- 細胞は、100%コンフルエントに達した細胞から培地を吸引し、2 mlの新鮮なレギュラーDMEMで置き換える必要があり、その時点で約16時間トランスフェクション後(3日目)。 37℃で一晩インキュベートする。
- 4日目に、TRANでEGFP発現を観察することにより、トランスフェクション効率を評価sfectionコントロール細胞(高EGFP発現と90〜100%のトランスフェクション効率が良いウイルス予備校の信頼性の高い予測因子である。)
- 分泌され、感染性ウイルス粒子を含む培地を収集します。残留293T細胞を除去し、ウイルスを含む上清を回収し、5分間> 2,000 xgで遠心分離します。あるいは、媒体は、0.45μmのメンブランフィルターでクリアすることができます。ウイルス粒子は感染症で使用するための準備が整いました。
3。 3T3細胞への感染
1。シード3T3細胞(3日目)
翌日20〜30%コンフルエントを取得するために分割し、12ウェルプレート上の3T3細胞のシード適切な数(〜12000)。 37℃で一晩インキュベートする。
2。 3T3細胞(4日目)に感染
- 播種した細胞を観察します。 20〜30%の合流点(50%未満)は、感染症のために望まれている。
- ウイルスを含む回収した培地に5μg/ mlの最終濃度にポリブレンを追加粒子。ピペッティングでよく混ぜる。
- 3T3細胞から培地を吸引除去し、ウェルあたり上記のウイルス混合物1mlを加える。 37℃で一晩インキュベートする。 (ポリブレン感染効率を向上させるために使用されていますが、絶対に必要ではありません。それはいくつかの細胞毒性がありますので、事前の検査が推奨されています。)
3。感染した細胞を(5日目以降)を選択します
- 二十四時間感染後、感染細胞からウイルスとポリブレンを含む培地を吸引除去し、1×PBSで1回洗浄し、新鮮な培地に変更します。 37回復と成長のための℃で一晩インキュベートする。
- ときに合流します(通常1〜2日後)、37℃で一晩インキュベートした細胞を分割します。
- 翌日、細胞から吸引培地(<50%のコンフルエンスが望まれている)と安定形質導入細胞を選択するために、10μg/ mlのブラストサイジンを含む新鮮な培地と交換してください。 (ブラストサイはkill-曲線経験的に特定の細胞株のために決定する必要があります。)
- はクロック記者(一般的に4-6日合計)を発現している抗生物質耐性細胞の連続的な選択のために2〜3日毎にブラスチシジンを含む新鮮な培地に変更します。
4。レポーター細胞の生物発光の記録
1。シードレポーター細胞
ブラストサイジン耐性レポーター細胞を伝播し、35mm培養ディッシュに分割します。コンフルエントになるまで37℃でインキュベートする。私たちは通常、サーカディアン表現型の各条件の下に各レポーター細胞株に対する≥3料理を準備。
2。同期と記録媒体への変更
- コンフルエントレポーター細胞から培地を吸引除去し、PBSで1回洗浄し、DMEMに、10μMフォルスコリン(または200nMのデキサメタゾン)を含むと交換してください。細胞を同期させるために、1時間37℃でインキュベートする。 (あるいは、細胞を温度サイクル23または血清ショック24で同期させることができます。)
- 待っている間、記録を作成し次のように、3T3細胞用る培地:10%FBS、1×ペニシリン/ストレプトマイシン/ GLN、1μMフォルスコリン、1mMのルシフェリン、25mMのHEPES、pH7.4を含む1×DMEM(Hyclone社)。血清およびフォルスコリン濃度は経験的に決定することができる。薄暗い細胞については、フェノールレッドを含まない培地を用いることができる。
- フォルスコリン治療の終了時に、培地を吸引し、作りたての記録媒体と交換してください。
3。レポーター細胞の生物発光の記録
- 培地交換後、蒸発を防ぐために真空グリースで所定の位置に40ミリメートル滅菌カバーガラスとシールで培養皿をカバーしています。
- H 2 OやCO 2をせずに36℃のインキュベーターセットに内蔵されていLumiCycleルミノメーター、上に皿をロードします。
- リアルタイム生物発光の記録を開始します。私たちは通常、二週目のための媒体の変化と連続記録(Savelyev らを参照してください。詳細については)25に続いて、1週間のリズムを記録します。 (96我々の記録のためのLLプレート、シナジーSL2は、記録装置として使用されていました。細部のためのディスカッション1.1を参照)。
5。データ解析とプレゼンテーション
レポーター細胞は、概日時計機能に対する表現型への影響を決定するための重要な高分解能の定量的な発光の記録が容易になります。位相、周期の長さ、リズムの振幅、減衰率などの概日のパラメータを得るために、我々は生物発光5,14データを分析するLumiCycle解析プログラム(Actimetrics)を使用します。簡単に言えば、生データは、ベースラインは最初フィットしており、ベースラインを差し引いたデータはパラメータが決定されるから、正弦波、に取り付けられている。永続的なリズム、> 90%は通常達成されるの適合度を示すためのサンプル。培地交換時に高レベルの過渡生物発光のために、我々は通常解析からのデータの最初のサイクルを除外します。
データプレゼンテーションのために、私たちは通常、TIに対する生データ(生物発光、カウント/秒)をプロット私(日)。必要な場合には、ベースラインを差し引いたデータは、振幅と位相を比較するためにプロットすることができます。
6。代表的な結果
1。フェーズ固有の概日記者
概日時計は、生化学的な負のフィードバック機構1に基づいています。コアフィードバックループは、リズミカルな遺伝子発現を(朝の段階では、 例えば、Rev非ERBαで)を生成するために概E / E'-ボックスエンハンサーエレメントに作用転写活性化因子BMAL1やCLOCK、およびリプレッサーPERSとCRYS、から構成されています。およびROR / REV-ERBバインディング要素(RRE、夜フェーズのため、、コア·ループは、少なくとも2つの他の概日のシスエレメント、DBP/E4BP4バインディング要素(日段階のため、 例えば、Per3 D-ボックス)を調整し、統合例えば、BMAL1)17。複数概要素による組み合わせ調節は、小説の中間相を生成することができます。たとえば、Cry1転写酵素る、別個のCry1夕方時フェーズ13を生じさせる3つのすべての概要素( すなわち 、E / E'-ボックスおよびD-ボックス要素Cry1遺伝子の第1イントロン中のプロモーターとのRREで)によって媒介される。
遺伝子調節のこれらのメカニズムに基づいて、我々は、4つの異なるレポーター構築物を生成した:調節領域17に、両方のE / E'-ボックスおよびD-ボックス要素を含むP(PER2)-DリュックとP(Cry1)-Dリュックの記者、 26,27、P(Cry1) -すべての3つの要素( すなわち 、E / E'-ボックス、D-ボックス、およびRRE)13,17によって組み合わせ調節を表すイントロン -D リュック 、およびP(BMAL1)-DリュックレギュもっぱらRRE 9,17,19,21によって。我々は、レポーター発現の予想の明確なフェーズ( 図2)を生成する3T3細胞にこれらのレポーターを導入しました。
2。 RNAiおよび薬理学的にACTIによる遺伝子ノックダウンveの化合物
トランスフェクション効率が高い場合には、合成siRNAは一過性遺伝子発現をノックダウン細胞にトランスフェクトすることができます。トランスフェクションは、技術的に困難な場合には、shRNA発現ベクターを安定細胞によって産生されるshRNAを遺伝子ノックダウン(KD)のsiRNAに処理されるように、レンチウイルス感染を介して細胞内に導入することができます。ここでは、pLL3.7ゲートウェイ発現ベクター9( 図3B)を使用して、3T3細胞でU2OS細胞におけるsiRNA( 図3A)およびshRNAを用いたCry1 Cry2と遺伝子のKDの効果を提示します。 3T3細胞に加えて、U2OSモデルは、市販のヒトsiRNAライブラリーのハイスループットスクリーニング( 例えば 、ヒト起源、堅牢な概日リズムの検証関数を生成することができるための重要な要件を満たしている主な理由は別の抜群の携帯クロックモデルとなっているすべての既知の時計遺伝子、高効率なトランスフェクション及び影響を受けやすい定量的な発光記録)。両方の種類の細胞では、RNAi媒介KDは以前マウスノックアウト(KO)や携帯KDスタディ5,10,11,28と一貫クロック表現型が得られた。たとえば、Cry1 KD短縮期間の長さとリズムの永続性を低減し、Cry2 KDは期間を長くする一方。さらに、選択された小分子は、薬理学的標的と混乱させるタンパク質の機能( 図3C)に使用することができます。
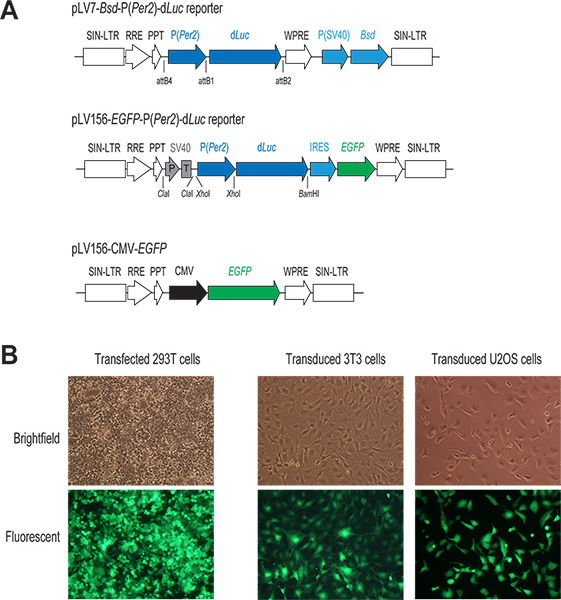
図1。レンチウイルス媒介遺伝子送達システム。 2レンチP(PER2)-D LucレポーターベクターおよびCMV-EGFPコンストラクトの(A)の模式図。宿主細胞ゲノムに統合するための唯一の領域が示されています。両方のレポーターコンストラクトでは、d リュックの転写がPER2プロモーターの直接制御下にある。 pLV7-BSD-P(PER2)-D Lucベクター(再で組合せベースのクローニング)、共発現ブラストサイジン耐性遺伝子(BSD)が感染した細胞の選択を容易にする。 pLV156-P(PER2)-D Lucベクター(ライゲーションベースのクローン作成)では、EGFPの翻訳は目視や感染した細胞のFACS選別を可能にする、Dリュックの内部リボソーム侵入部位(IRES)下流によって媒介される。また、SV40プロモーター/ターミネーター(P / T)は絶縁体(議論1.3を参照)として使用されます。 CMV-EGFP制御ベクトル、EGFP発現に強力なCMVプロモーターの制御下にある。トランスフェクションし、感染したGFP発現細胞の(B)の蛍光画像が。これらの細胞におけるGFPの発現によって示されるように、典型的には、我々は、293T細胞の一過性トランスフェクションでは、私たちの興味のある細胞株のレンチウイルス感染の両方で高い効率を達成しています。 拡大図を表示するには、ここをクリックしてください 。
図2。 3T3細胞での生物発光レポーターの位相特異的発現 、この実験で使用したレンチウイルスレポーターベクターは、P(Cry1)-Dリュック、P(Cry1)、pLV7-BSD-P(PER2)-Dリュックです- 。 イントロン -D リュック 、とP(BMAL1)-Dリュック 。矢印で示すように、各記者が、振動の異なる相を示す。 PER2とCry1プロモーターは、朝·日の各フェーズと夜の段階では、Pによる組み合わせ調節(Cry1)でBMAL1プロモーターでピーク生物発光を駆動しながら- イントロン宿すのE-box、D-ボックス、およびRRE要素がピーク生物発光の夕方の位相を与える。 拡大図を表示するにはここをクリック 。
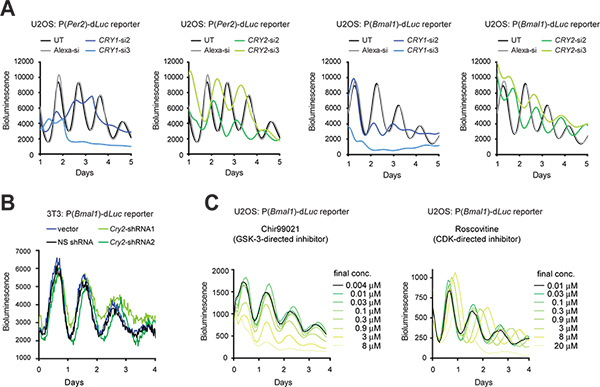
図3。レポーター細胞の概日生物発光リズムの遺伝学的および薬理学的摂動。 (A)は、Cry1の影響およびU2OSレポーター細胞の細胞リズムにsiRNAによるノックダウンCry2。 LumiCycleルミノメーターは、35 mmの培養ディッシュ中の細胞の生物発光の記録のために使用された。図は、適合されている リファレンスエルゼビア(2009年)からの許可を得て、第10位、。3T3レポーター細胞の細胞リズムにshRNAのノックダウンによってCry2の(B)の効果。からU6-shRNAのカセットを含むpLL3.7ゲートウェイベクトルはCry2遺伝子ノックダウンのために使用された。ウェスタンブロット解析(データは示していない)によって決定されるようshRNA2はshRNA1より良いノックダウン効率を有する。シナジールミノメーターは、96ウェルプレートでの細胞の生物発光の記録のために使用された。録音のための設定は次のとおりです。インキュベーター温度33℃、積分時間、15秒; CELでの小分子阻害剤のインターバル時間は、30分(C)の効果U2OSレポーター細胞のlularリズム。 CHIR99021とロスコビチンは、それぞれ、GSK-3およびCDKに対して向けられた阻害剤である。 ViewLuxシステム(CHIR99021アッセイ)とテカンルミノメーター(ロスコビチンアッセイ)384ウェルプレートでの細胞の生物発光のレコーディングのために使用された。図を参照19位(学術著作権2008国立アカデミー、米国)から適合させられます。 拡大図を表示するには、ここをクリックしてください 。
ディスカッション
1。現在のプロトコルへの変更
1.1記録デバイスとスループットの考慮事項
なぜなら、その商業的利用可能性、LumiCycle(Actimetrics)は、リアルタイムレコーディング4,5,9,19,29-31ための最も一般的に使用される自動化されたルミノデバイスとなっています。 LumiCycleは非常に高感度、低ノイズ14を提供する光検出器として、光電子増倍管...
開示事項
特別な利害関係は宣言されません。
謝辞
この作品は、国立科学財団(IOS-0920417)(ACL)によって部分的にサポートされていました。
資料
| 試薬の名称 | 会社 | カタログ番号 | 注釈 |
| DMEM | Hyclone社 | SH30243FS | 定期的な細胞増殖のための |
| DMEM | インビトロジェン | 12100-046 | luminometry用 |
| FBS | Hyclone社 | SH3091003 | |
| ペニシリン/ストレプトマイシン/ GLN(100倍) | Hyclone社 | SV3008201 | |
| B-27 | インビトロジェン | 17504-044 | |
| D-ルシフェリン | Biosynth | L-8220 | |
| ポリエルリジン | シグマ | P4707 | |
| ポリブレン | ミリポア | TR-1003-G | |
| フォルス | シグマ | F6886 | |
| 他のすべての化学物質 | シグマ | ||
| 機器 | |||
| 組織培養インキュベーター | 37℃で5%CO2の | ||
| 組織培養フード | BSL-2の認定 | ||
| ライト&蛍光顕微鏡 | オプションの位相コントラスト | ||
| LumiCycle | Actimetrics |
参考文献
- Reppert, S. M., Weaver, D. R. Coordination of circadian timing in mammals. Nature. 418, 935-941 (2002).
- Hastings, M. H., Reddy, A. B., Maywood, E. S. A clockwork web: circadian timing in brain and periphery, in health and disease. Nat. Rev. Neurosci. 4, 649-661 (2003).
- Nagoshi, E. Circadian gene expression in individual fibroblasts: cell-autonomous and self-sustained oscillators pass time to daughter cells. Cell. 119, 693-705 (2004).
- Welsh, D. K. Bioluminescence imaging of individual fibroblasts reveals persistent, independently phased circadian rhythms of clock gene expression. Curr. Biol. 14, 2289-2295 (2004).
- Liu, A. C. Intercellular coupling confers robustness against mutations in the SCN circadian clock network. Cell. 129, 605-616 (2007).
- Kornmann, B. System-driven and oscillator-dependent circadian transcription in mice with a conditionally active liver clock. PLoS Biol. 5, e34 (2007).
- Hogenesch, J. B., Herzog, E. D. Intracellular and intercellular processes determine robustness of the circadian clock. FEBS Lett. 585, 1427-1434 (2011).
- DeBruyne, J. P., Weaver, D. R., Reppert, S. M. Peripheral circadian oscillators require CLOCK. Curr. Biol. 17, 538-539 (2007).
- Liu, A. C. Redundant function of REV-ERBalpha and beta and non-essential role for Bmal1 cycling in transcriptional regulation of intracellular circadian rhythms. PLoS Genet. 4, e1000023 (2008).
- Zhang, E. E. A genome-wide RNAi screen for modifiers of the circadian clock in human cells. Cell. 139, 199-210 (2009).
- Baggs, J. E. Network features of the mammalian circadian clock. PLoS Biol. 7, e52 (2009).
- Hirota, T. High-throughput chemical screen identifies a novel potent modulator of cellular circadian rhythms and reveals CKIalpha as a clock regulatory kinase. PLoS Biol. 8, e1000559 (2010).
- Ukai-Tadenuma, M. Delay in feedback repression by cryptochrome 1 is required for circadian clock function. Cell. 144, 268-281 (2011).
- Yamazaki, S., Takahashi, J. S. Real-time luminescence reporting of circadian gene expression in mammals. Methods Enzymol. 393, 288-301 (2005).
- Welsh, D. K., Imaizumi, T., Kay, S. A. Real-time reporting of circadian-regulated gene expression by luciferase imaging in plants and mammalian cells. Methods Enzymol. 393, 269-288 (2005).
- Sato, T. K. Feedback repression is required for mammalian circadian clock function. Nat. Genet. 38, 312-319 (2006).
- Ueda, H. R. System-level identification of transcriptional circuits underlying mammalian circadian clocks. Nat. Genet. 37, 187-192 (2005).
- Brown, S. A. The period length of fibroblast circadian gene expression varies widely among human individuals. PLoS Biol. 3, e338 (2005).
- Hirota, T. A chemical biology approach reveals period shortening of the mammalian circadian clock by specific inhibition of GSK-3beta. Proc. Natl. Acad. Sci. U.S.A. 105, 20746-20751 (2008).
- Tiscornia, G., Singer, O., Verma, I. M. Production and purification of lentiviral vectors. Nat. Protoc. 1, 241-245 (2006).
- Ueda, H. R. A transcription factor response element for gene expression during circadian night. Nature. 418, 534-539 (2002).
- Zufferey, R., Donello, J. E., Trono, D., Hope, T. J. Woodchuck hepatitis virus posttranscriptional regulatory element enhances expression of transgenes delivered by retroviral vectors. J. Virol. 73, 2886-2892 (1999).
- Buhr, E. D., Yoo, S. H., Takahashi, J. S. Temperature as a universal resetting cue for mammalian circadian oscillators. Science. 330, 379-385 (2010).
- Balsalobre, A., Damiola, F., Schibler, . U.A serum shock induces circadian gene expression in mammalian tissue culture cells. Cell. 93, 929-937 (1998).
- Savelyev, S. A., Larsson, K. C., Johansson, A., Lundkvist, G. B. S. Slice Preparation, Organotypic Tissue Culturing and Luciferase Recording of Clock Gene Activity in the Suprachiasmatic Nucleus. J. Vis. Exp. (48), e2439 (2011).
- Akashi, M., Ichise, T., Mamine, T., Takumi, T. Molecular mechanism of cell-autonomous circadian gene expression of Period2, a crucial regulator of the mammalian circadian clock. Mol. Biol. Cell. 17, 555-565 (2006).
- Ohno, T., Onishi, Y., Ishida, N. A novel E4BP4 element drives circadian expression of mPeriod2. Nucleic Acids Res. 35, 648-655 (2007).
- Maier, B. A large-scale functional RNAi screen reveals a role for CK2 in the mammalian circadian clock. Genes Dev. 23, 708-718 (2009).
- Yoo, S. H. PERIOD2::LUCIFERASE real-time reporting of circadian dynamics reveals persistent circadian oscillations in mouse peripheral tissues. Proc. Natl. Acad. Sci. U.S.A. 101, 5339-5346 (2004).
- Liu, A. C., Lewis, W. G., Kay, S. A. Mammalian circadian signaling networks and therapeutic targets. Nat. Chem. Biol. 3, 630-639 (2007).
- Ko, C. H. Emergence of noise-induced oscillations in the central circadian pacemaker. PLoS Biol. 8, e1000513 (2010).
- Izumo, M., Johnson, C. H., Yamazaki, S. Circadian gene expression in mammalian fibroblasts revealed by real-time luminescence reporting: temperature compensation and damping. Proc. Natl. Acad. Sci. U.S.A. 100, 16089-16094 (2003).
- Izumo, M., Sato, T. R., Straume, M., Johnson, C. H. Quantitative analyses of circadian gene expression in mammalian cell cultures. PLoS Comput. Biol. 2, e136 (2006).
- Chen, Z. Identification of diverse modulators of central and peripheral circadian clocks by high-throughput chemical screening. Proc. Natl. Acad. Sci. U.S.A. 109, 101-106 (2011).
- Yamaguchi, S. Synchronization of cellular clocks in the suprachiasmatic nucleus. Science. 302, 1408-1412 (2003).
- Akashi, M., Hayasaka, N., Yamazaki, S., Node, K. Mitogen-activated protein kinase is a functional component of the autonomous circadian system in the suprachiasmatic nucleus. J. Neurosci. 28, 4619-4623 (2008).
- Hoshino, H., Nakajima, Y., Ohmiya, Y. Luciferase-YFP fusion tag with enhanced emission for single-cell luminescence imaging. Nat. Methods. 4, 637-639 (2007).
- Asai, M. Visualization of mPer1 transcription in vitro: NMDA induces a rapid phase shift of mPer1 gene in cultured SCN. Curr. Biol. 11, 1524-1527 (2001).
- Wilsbacher, L. D. Photic and circadian expression of luciferase in mPeriod1-luc transgenic mice in vivo. Proc. Natl. Acad. Sci. U.S.A. 99, 489-494 (2002).
- Yamazaki, S. Resetting central and peripheral circadian oscillators in transgenic rats. Science. 288, 682-685 (2000).
- Welsh, D. K., Noguchi, T., Yuste, R. Cellular bioluminescence imaging. Imaging: A Laboratory Manual. , 369-385 (2011).
- Nakajima, Y. Enhanced beetle luciferase for high-resolution bioluminescence imaging. PLoS One. 5, e10011 (2010).
- Guilding, C. A riot of rhythms: neuronal and glial circadian oscillators in the mediobasal hypothalamus. Mol. Brain. 2, 28 (2009).
- O'Neill, J. S. cAMP-dependent signaling as a core component of the mammalian circadian pacemaker. Science. 320, 949-953 (2008).
- Fuller, P. M., Lu, J., Saper, C. B. Differential rescue of light- and food-entrainable circadian rhythms. Science. 320, 1074-1077 (2008).
- Mukherjee, S. Knockdown of Clock in the ventral tegmental area through RNA interference results in a mixed state of mania and depression-like behavior. Biol. Psychiatry. 68, 503-511 (2010).
- Saijo, K. A Nurr1/CoREST pathway in microglia and astrocytes protects dopaminergic neurons from inflammation-induced death. Cell. 137, 47-59 (2009).
- Elias, G. M. Synapse-specific and developmentally regulated targeting of AMPA receptors by a family of MAGUK scaffolding proteins. Neuron. 52, 307-320 (2006).
- Isojima, Y. CKIepsilon/delta-dependent phosphorylation is a temperature-insensitive, period-determining process in the mammalian circadian clock. Proc. Natl. Acad. Sci. U.S.A. 106, 15744-15749 (2009).
- Bucan, M., Abel, T. The mouse: genetics meets behaviour. Nat. Rev. Genet. 3, 114-123 (2002).
- Hughes, M. E. Harmonics of circadian gene transcription in mammals. PLoS Genet. 5, e1000442 (2009).
- Atwood, A. Cell-autonomous circadian clock of hepatocytes drives rhythms in transcription and polyamine synthesis. Proc. Natl. Acad. Sci. U.S.A. 108, 18560-18565 (2011).
- Panda, S. Coordinated transcription of key pathways in the mouse by the circadian clock. Cell. 109, 307-320 (2002).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved