Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Überwachung Zell-autonome circadiane Uhr Rhythms der Genexpression mittels Luciferase Biolumineszenz Reporter
In diesem Artikel
Zusammenfassung
Circadiane Uhren funktionieren innerhalb einzelner Zellen, dh sie sind zellautonome. Hier beschreiben wir Verfahren zur Erzeugung von Zell-autonome Uhrenmodelle mit nicht-invasiven, Luciferase-basierte Echtzeit-Biolumineszenz-Technologie. Reporter Zellen bereitzustellen gefügig, funktionales Modell für das Studium zirkadianen Biologie.
Zusammenfassung
Bei Säugetieren sind viele Aspekte des Verhaltens und der Physiologie wie Schlaf-Wach-Zyklen und Leberstoffwechsel durch endogene circadiane Uhren (bewertet 1,2) geregelt. Die circadiane zeithaltenden System ist eine hierarchische Multi-Oszillator-Netzwerk, mit dem zentralen Takt im suprachiasmatischen Nukleus (SCN) zu synchronisieren und koordinieren extra-SCN und peripheren Uhren anderswo 1,2 liegt. Zellen sind die einzelnen Funktionseinheiten für die Erzeugung und Aufrechterhaltung der zirkadianen Rhythmen 3,4 und dieser Oszillatoren verschiedener Gewebetypen im Organismus teilen eine bemerkenswert ähnliche biochemische negativen Rückkopplungsmechanismus. Jedoch aufgrund von Wechselwirkungen an der neuronalen Netzwerk-Ebene im SCN und durch rhythmische, systemischer Cues am organismal Ebene werden Tagesrhythmus am organismal Ebene nicht notwendigerweise zellautonome 5-7. Im Vergleich zu herkömmlichen Studien der lokomotorischen Aktivität in vivo und ex vivo SCN Explantate, cell-basierte in vitro-Assays ermöglichen Entdeckung der Zell-autonome circadiane Mängeln 5,8. Strategisch sind zell-basierte Modelle mehr experimentell handhabbar zur phänotypischen Charakterisierung und schnelle Entdeckung der Grundtakt Mechanismen 5,8-13.
Da zirkadianen Rhythmen dynamisch sind, sind Längs-Messungen mit hoher zeitlicher Auflösung benötigt, um Uhrfunktion beurteilen. In den letzten Jahren hat Echtzeit Biolumineszenz Aufnahme mit Leuchtkäfer-Luciferase als Reporter zu einer gebräuchlichen Methode zur Untersuchung Tagesrhythmus in Säugetieren 14,15, wie es für die Prüfung des Persistenz und Dynamik molekularer Rhythmen ermöglicht. Um Zell-autonome circadiane Rhythmen der Genexpression überwachen, können Luciferase Reporter in Zellen durch transiente Transfektion 13,16,17 oder stabile Transduktion 5,10,18,19 eingeführt werden. Hier beschreiben wir eine stabile Transduktion Protokoll unter Verwendung Lentivirus-vermittelte Gentransfer. Ter Lentivirales Vektorsystem ist den herkömmlichen Methoden wie transiente Transfektion und Keimbahntransmission wegen ihrer Effizienz und Vielseitigkeit: er ermöglicht die effiziente Bereitstellung und stabile Integration in das Wirtsgenom von beiden Teilen und nicht teilende Zellen 20. Sobald ein Reporter-Zelllinie hergestellt ist, kann die Dynamik der Uhr-Funktion durch Biolumineszenz-Aufnahme untersucht werden. Wir beschreiben zunächst die Erzeugung von P (Per2)-d Luc Reporter Linien, und dann vorliegenden Daten aus diesem und anderen circadianen Reportern. In diesen Assays werden 3T3 Mausfibroblasten und U2OS humanen Osteosarkomzellen als zelluläre Modelle verwendet. Wir diskutieren auch verschiedene Möglichkeiten der Verwendung dieser Uhrenmodelle in zirkadianen Studien. Hier beschriebenen Methoden können zu einer Vielzahl von Zelltypen, die zellulären und molekularen Grundlagen von circadianen Uhren studieren angewendet werden, und es kann nützlich sein bei der Bewältigung von Problemen in anderen biologischen Systemen.
Protokoll
Ein. Bau Lentivirale Luciferase Reporter
Ein Säuger zirkadianen Reporterkonstrukt enthält üblicherweise eine Expressionskassette in dem eine circadiane Promotor mit dem Luciferase-Gen fusioniert ist. Sowohl Ligations-und Rekombinations-basierte Strategien werden üblicherweise für DNA-Klonierung verwendet. Als ein Beispiel beschreiben wir hier eine Rekombination basierende Gateway-Klonen Verfahren zur Erzeugung eines P (Per2)-d Luc-Reporter lentiviralen, in der die destabilisierte Luciferase (Luc d) unter Kontrolle des Maus Per2 Promotor.
- Klonierung Per2 Promotor. Verwenden, um die PCR Per2 Promotor DNA-Fragment von 526 bp amplifizieren, stromaufwärts von der Transkriptionsstartstelle aus einer Maus Per2 BAC-Klon 9-13, unter Verwendung eines Vorwärtsprimers (5'-CTCGAGCGGATTACCGAGGCTGGTCACG TC-3 ') und einen Rückwärts-Primer (5' -CTCGAGTCCCTTGCTCGGCCCGTCAC TTGG-3 ') und Klon in pENTR5'-TOPO-Vektor (Invitrogen) zu erzeugenpENTR5'-P (Per2).
- Klonierung von d Luc. Der d Luc enthält das Leuchtkäfer-Luciferase-Gen und eine C-terminale Sequenz für die schnelle PEST Proteinabbau, wie zuvor beschrieben 21. Verwenden, um die PCR-DNA-Fragment d Luc und Klon in pENTR / D-TOPO-Vektor (Invitrogen) zu amplifizieren pENTR / Dd Luc erzeugen.
- Bau-Reporter-Vektor. Mischen Sie die beiden pENTR Plasmide, pENTR5'-P (Per2) und pENTR / Dd Luc, mit dem lentiviralen Zielvektor pLV7-BSD (BSD, Blasticidin Resistenzgen), und führen Sie die Rekombination mit Clonase eine pLV7-BSD-P erzeugen (Per2)-d Luc Reporter (Abbildung 1). pLV7-BSD ist eine modifizierte Version (made in unserem Labor) der pLenti6/R4R2/V5-DEST (Invitrogen), in dem die woodchuck Hepatitis-Virus post-Transkriptionsregulatorelements (WPRE) Sequenzen 22 wurden sofort nach dem Ausdruck ca eingesetzt ssette die Genexpression zu erhöhen.
2. Produktion von Lentivirale Partikel
Ein. Seed 293T-Zellen (Tag 1)
- Wachsen menschliche embryonale Nieren (HEK) 293T-Zellen zu 90-100% Konfluenz in regelmäßigen DMEM mit 10% FBS und 1x Penicillin-Streptomycin-Glutamin (PSG) auf 10 cm Zellkulturschalen ergänzt. (Schnell wachsende Zellen mit niedrigen Durchgang Nummer sind entscheidend für die effiziente Transfektion.)
- Vor dem Aussäen der Zellen für Transfektion Mantel 6-well Kulturplatten durch Zugabe von 1 ml 0,001% Poly-L-Lysin in PBS in jede Vertiefung und Inkubieren bei Raumtemperatur für 20 min. Absaugen und spülen einmal mit 1x PBS vor der Verwendung.
- Dissoziieren 293T-Zellen mit Trypsin und Saatgut 0,75 x 10 6 Zellen auf jede Vertiefung der vorbeschichteten Platten mit 2 ml DMEM regelmäßigen. Swirl die Platten gründlich, um eine gleichmäßige Verteilung der Zellen in jeder Vertiefung erhalten. Wachsen die Zellen in den Inkubator bei 37 ° C über Nacht.
- Beachten Sie die ausgesäten Zellen von Tag 1. Zelle erreichen sollte Zusammenfluss von 80-90%.
- Bereiten Plasmidtransfektion Mischung in einem 1,5 ml Mikrozentrifugenröhrchen durch Zugabe von 2 ug eines lentiviralen Reporter Plasmid-DNA (zB pLV7-P (Per2)-d Luc; Liu lab) und die 3 Verpackungsvektoren (1,3 pg Gag / Pol, 0,5 ug Rev und 0,7 ug VSVG; Invitrogen). Als Kontrolle für sowohl Transfektion und nachfolgende Infektion, wir normalerweise einen zusätzlichen gut in Transfektion für einen lentiviralen GFP-Expressionsvektor, pLV156-CMV-EGFP (Abbildung 1A), beherbergt, Enhanced Green Fluorescent Protein (EGFP) unter der Kontrolle des CMV-Promotors wie zuvor beschrieben 20.
- Fügen Sie 100 ul von 0,25 M CaCl 2 (verdünnt mit DNase / RNase-free ddH 2 O von 2,5 M Lager) mit dem Plasmid Mischung in Schritt 2 und gründlich mischen. Dann fügen Sie 100 ul 2x BBS-Lösung (50 mM BES, 280 mM NaCl, 1,5 mM Na 2 HPO 4, pH 6,95) und mischen Sie vorsichtig, aber gründlich. Inkubieren des DNA-Mischung bei Raumtemperatur für 15 min.
- Während des Wartens, absaugen Medium aus 293T-Zellen und wechseln Sie in 2 ml frisches Medium. Bringen Sie die Platte in den Inkubator für mindestens 10 min bis mittlere pH vor der Transfektion äquilibrieren.
- Fügen Transfektion Mischung aus Schritt 3 bis 293T-Zellen Tropfen für Tropfen. Schwenken Sie die Platte vorsichtig und beobachten Partikelbildung unter dem Mikroskop. Inkubation bei 5% CO 2, 37 ° C über Nacht. (Fine Partikelbildung von CaPO 4 / DNA-Präzipitat ist entscheidend für eine effiziente Transfektion.)
3. Ernte Viruspartikel (Tage 3-4)
- Etwa 16 Stunden nach der Transfektion (Tag 3) Zu diesem Zeitpunkt Zellen sollten bis zu 100% Konfluenz absaugen Medium von Zellen und ersetzen mit 2 ml frischem regelmäßigen DMEM. Bei 37 ° C über Nacht.
- Am Tag 4 zu beurteilen Transfektionseffizienz durch Beobachtung EGFP-Expression in transfection Kontrollzellen (Transfektionseffizienz von 90-100% mit hoher EGFP-Expression ist ein zuverlässiger Indikator für eine gute virale prep.)
- Sammeln des Mediums enthaltend sekretiert, infektiöse Viruspartikel. Zentrifuge bei> 2.000 xg für 5 min, um das restliche 293T Zellen zu entfernen und sammeln das Virus enthaltende Überstand. Alternativ kann das Medium mit einer 0,45 um Membranfilter gelöscht. Die viralen Partikel sind bereit für den Einsatz in Infektion.
3. Infektion von Zellen 3T3
Ein. Seed 3T3-Zellen (Tag 3)
Split und Saatgut entsprechende Anzahl (~ 12.000) von 3T3-Zellen auf einer 12-Well-Platte auf 20-30% Konfluenz am nächsten Tag erhalten. Bei 37 ° C über Nacht.
2. Infect 3T3-Zellen (Tag 4)
- Beachten Sie die ausgesäten Zellen. Konfluenz von 20-30% (unter 50%) ist für die Infektion gewünscht.
- Hinzufügen Polybren in einer Endkonzentration von 5 pg / ml zu dem gesammelten Medium mit viralenTeilchen. Gut mischen durch Pipettieren.
- Absaugen Medium von 3T3-Zellen und 1 ml der oben viralen Mischung pro Vertiefung. Bei 37 ° C über Nacht. (Polybrene verwendet wird, um Infektionen Effizienz zu verbessern, ist aber nicht unbedingt erforderlich. Wie es zu toxisch sein einigen Zellen wird vorheriger Prüfung empfohlen.)
3. Wählen infizierten Zellen (Tag 5 und höher)
- Vierundzwanzig Stunden nach der Infektion, absaugen Medium mit Virus-und Polybren aus infizierten Zellen, einmal zu waschen mit 1x PBS, und wechseln Sie in frischem Medium. Bei 37 ° C über Nacht zur Verwertung und Wachstum.
- Wenn konfluent (in der Regel 1-2 Tage später), teilen die Zellen und bei 37 ° C über Nacht.
- Am folgenden Tag absaugen Medium von Zellen (<50% Konfluenz gewünscht) und ersetzen Sie sie durch frisches Medium mit 10 ug / ml Blasticidin für stabil wählen transduzierten Zellen. (Blasticidin kill-Kurve muss empirisch für eine bestimmte Zelllinie bestimmt werden.)
- , um frisches Medium, welches Blasticidin alle 2-3 Tage für die kontinuierliche Auswahl der Antibiotika-resistenten Zellen Uhr Reporter (in der Regel 4-6 Tage insgesamt) ändern.
4. Biolumineszenz Aufzeichnung von Reporter Cells
Ein. Seed-Reporter-Zellen
Propagieren Blasticidin-resistente Reporter Zellen und aufgeteilt auf 35-mm-Kulturschalen. Bei 37 ° C bis zur Konfluenz. Wir in der Regel bereiten ≥ 3 Gerichte für jeden Reporter-Zelllinie unter jeder Bedingung für die circadiane Phänotypisierung.
2. Synchronisation und Änderung Aufzeichnungsträger
- Absaugen Medium von konfluenten Reporter Zellen, einmal zu waschen mit PBS, und ersetzen Sie sie durch DMEM mit 10 uM Forskolin (oder 200 nM Dexamethason). Bei 37 ° C für 1 Stunde, um die Zellen zu synchronisieren. (Alternativ können Zellen durch Temperaturzyklen 23 oder Serum Schock 24 synchronisiert.)
- Während des Wartens, bereiten Rekordten Weg für 3T3-Zellen wie folgt: 1x DMEM (HyClone), das 10% FBS, 1x Pen / Strep / Gln, 1 pM Forskolin, 1 mM Luciferin, 25 mM HEPES, pH 7,4. Serum und Forskolin-Konzentration kann empirisch ermittelt werden. Für sehr dunklen Zellen kann Phenolrot-freies Medium verwendet werden.
- Am Ende der Forskolin Behandlung absaugen Medium und ersetzen mit frisch zubereiteten Speichermedium.
3. Aufzeichnung der Biolumineszenz Reporterzellen
- Nach Mediumwechsel decken Kulturschalen mit 40 mm sterile Deckgläser und Dichtung an Ort und Stelle mit Vakuumfett um Verdunstung zu verhindern.
- Legen Sie die Speisen auf den LumiCycle Luminometer, die innerhalb eines eingestellten Brutschrank bei 36 ° C ohne H 2 O oder CO 2 gehalten.
- Starten Sie real-time Biolumineszenz Aufnahme. Wir in der Regel erfassen Rhythmen für 1 Woche, gefolgt von mittleren Wandel und kontinuierliche Aufzeichnung für eine zweite Woche (siehe Savelyev et al. Für Details) 25. (Für die Aufnahme von 96-wirll Platten, Synergy SL2 wurde als Aufnahmegerät verwendet, siehe Diskussion 1.1 für Details).
5. Datenanalyse und Präsentation
Reporter Zellen zu erleichtern hochauflösende quantitative Lumineszenz Aufnahme von entscheidender Bedeutung für die Bestimmung phänotypischen Auswirkungen auf circadiane Uhr-Funktion. Um circadianen Parameter einschließlich Phase Periodenlänge, Rhythmus Amplitude und Dämpfungsrate zu erhalten, verwenden wir die LumiCycle Analyse-Programm (Actimetrics) zu analysieren Biolumineszenz Daten 5,14. Kurz gesagt sind Rohdaten Basislinie taillierter ersten und Baseline-subtrahierten Daten werden an eine Sinuswelle, aus denen die Parameter bestimmt werden angebracht. Für Proben, die persistent Rhythmen, die Güte der Passung von> 90% wird in der Regel erreicht zu zeigen. Aufgrund der hohen transienten Biolumineszenz nach Medium ändern, wir in der Regel auszuschließen den ersten Zyklus von Daten aus der Analyse.
Für Präsentationen, wir in der Regel zeichnen Rohdaten (Biolumineszenz, counts / sec) gegen time (Tage). Wenn nötig, kann basisliniengetrennten subtrahierten Daten aufgetragen, um Amplitude und Phase zu vergleichen.
6. Repräsentative Ergebnisse
Ein. Phase-spezifische circadiane Reporter
Die circadiane Uhr auf einer biochemischen negativen Feedback-Mechanismus 1 basiert. Der Kern besteht aus Rückkopplungsschleife Transkriptionsaktivatoren BMAL1 und CLOCK, und Repressoren PERs und CRYS, die auf den zirkadianen E / E'-Box Enhancerelemente wirken, um rhythmische Genexpression (mit Morgen Phase, z. B. Rev-erb α) zu erzeugen. Der Kern Schleife regelt und integriert mindestens zwei anderen zirkadianen cis-Elemente, die DBP/E4BP4 Bindeelements (D-Box, für Tage Phase, z. B. Per3) und dem ROR / REV-ERB Bindeelements (RRE; für Nachtphase, zB BMAL1) 17. Kombinatorische Regulierung von mehreren circadianen Elementen erzeugen können neuartige Zwischenphasen. Beispielsweise Cry1 Transkriptiontion wird von allen drei zirkadianen Elemente (dh E / E'-Box und D-Kastenelemente im Promotor und RRES im ersten Intron des Gens Cry1), was zu dem unterscheidbaren Cry1 Abend-Zeitphase 13 vermittelt.
Basierend auf diesen Mechanismen der Genregulation, erzeugten wir vier verschiedenen Reporter-Konstrukte: P (Per2)-d Luc und P (Cry1)-d Luc-Reporter, die sowohl E / E'-Box und D-Boxenelemente in der regulatorischen Region 17, 26,27; P (Cry1) - Intron-d Luc darstellt kombinatorischen Regulierung durch alle drei Elemente (dh E / E'-Box, D-Box, und RRE) 13,17; und P (BMAL1)-d Luc geregelten ausschließlich durch RRE 9,17,19,21. Wir führten diese Reportern in 3T3-Zellen, die zu erwartenden unterschiedlichen Phasen der Reporterexpression (Abbildung 2) zu erzeugen.
2. Gene knockdown mittels RNAi und pharmakologisch aktiviertve Verbindungen
Wenn Transfektionseffizienz hoch ist, können synthetische siRNA transient in Zellen transfiziert werden, um knock down Genexpression. Wenn Transfektion technisch schwierig ist, kann ein Expressionsvektor sein shRNA stabil transduzierten in Zellen über lentiviralen Infektion, so daß shRNA von der Zelle produziert wird, um siRNA zur Gen Knockdown (KD) verarbeitet. Hier findet KD Wirkungen Cry1 und Cry2 Gene mittels siRNA in U2OS Zellen (3A) und shRNA in 3T3 Zellen unter Verwendung eines Gateway-pLL3.7 Expressionsvektor 9 (Abbildung 3B). Neben 3T3-Zellen wurde die U2OS Modell zu einem weiteren Takt herausragende zellulären Modell zuletzt deswegen, weil die wesentlichen Anforderungen für das Hochdurchsatz-Screening von kommerziell erhältlichen humanen siRNA-Bibliotheken (zB menschlichen Ursprungs, geeignet zur Erzeugung robuste zirkadiane Rhythmen, validiert Funktion erfüllt alle bekannten Uhren-Gene und zugänglich hocheffiziente Transfektion undquantitative Lumineszenz-Aufnahme). In beiden Zelltypen führte RNAi-vermittelte KD in clock Phänotypen konsistent mit früheren Maus-Knockout (KO) und zellulären KD Studien 5,10,11,28. Zum Beispiel, Cry1 KD verkürzt Periodenlänge und reduziert Rhythmus Persistenz, während Cry2 KD verlängert Zeitraum. Zusätzlich können ausgewählte kleine Moleküle zu pharmakologisch Target und perturb Proteinfunktion (3C) eingesetzt werden.
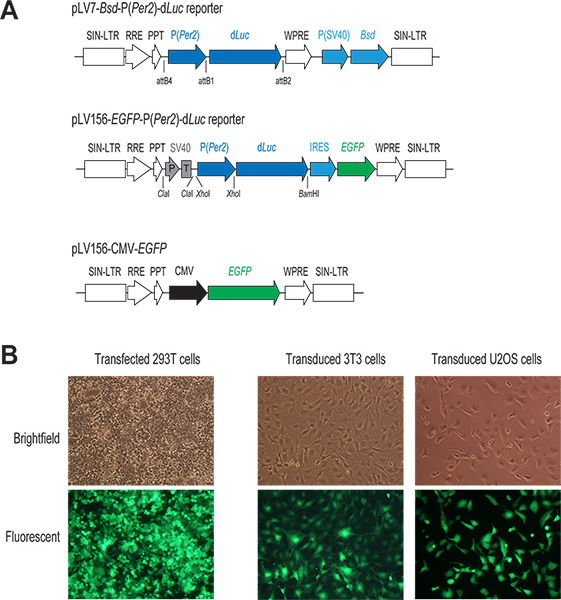
Abbildung 1. Das Lentivirus-vermittelte Genliefersystem. (A) Schematische Darstellung von zwei lentiviralen P (Per2)-d Luc Reporter Vektoren und einer CMV-EGFP konstruieren. Nur der Bereich für die Integration in das Genom der Wirtszelle gezeigt ist. In beiden Reporter-Konstrukte, die Transkription von d Luc unter direkter Kontrolle des Per2 Promotor. In der pLV7-BSD-P (Per2)-d Luc Vektor (reKombination-Klonen), erleichtert ein coexprimierten Blasticidin-Resistenzgen (BSD) Auswahl der infizierten Zellen. Im pLV156-P (Per2)-d Luc-Vektors (ligation-Klonen), wird EGFP Übersetzung durch eine interne Ribosomeneintrittsstelle (IRES) stromabwärts d Luc vermittelt, so dass für eine visuelle Beobachtung und FACS-Sortierung von infizierten Zellen. Zusätzlich ist ein SV40-Promotor / Terminator (P / T) als ein Isolator (siehe Diskussion 1.3) verwendet. In der CMV-EGFP Kontrollvektor ist EGFP-Expression unter der Kontrolle eines starken CMV-Promotors. (B) Fluoreszierende Bilder der transfizierten und infizierten GFP-exprimierenden Zellen. Normalerweise erreichen wir eine hohe Effizienz sowohl im transienten Transfektion von 293T-Zellen und in lentivirale Infektion von Zelllinien unseres Interesses, wie GFP-Expression in diesen Zellen angezeigt. Klicken Sie hier für eine größere Abbildung zu sehen .

Abbildung 2. Phase-spezifische Expression von Biolumineszenz Reportern in 3T3-Zellen Die lentiviralen Reportervektoren in diesem Experiment verwendet werden pLV7-BSD-P (Per2)-d Luc, P (Cry1)-d Luc, P (Cry1) -. Intron-d Luc, und P (BMAL1)-d Luc. Jedes Reporterkügelchen durch einen deutlichen Phase der Oszillation, wie durch die Pfeile angedeutet. Während die Per2 und Cry1 Promotoren Laufwerk peak Biolumineszenz am Morgen-Tag Phasen und der BMAL1 Promotors in der Nacht Phase kombinatorischen Regulierung durch die P (Cry1) - Intron beherbergen E-Box, verleiht D-Box und RRE Elementen Abend Phase peak Biolumineszenz . Klicken Sie hier für eine größere Abbildung zu sehen .
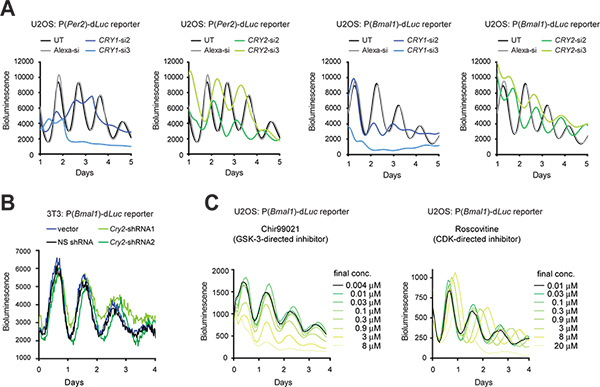
Abbildung 3. Genetische und pharmakologische Störung des zirkadianen Biolumineszenz Rhythmen Reporter Zellen. (A) Auswirkungen von Cry1 und Cry2 knockdown von siRNAs auf zelluläre Rhythmen U2OS Reporter Zellen. Die LumiCycle Luminometer wurde für Biolumineszenz Aufnahme von Zellen in 35 mm-Schalen verwendet. Abbildung angepasst ist von Referenz Nr. 10, mit Genehmigung von Elsevier (2009). (B) Auswirkungen von Cry2 knockdown von shRNAs auf zelluläre Rhythmen 3T3 Reporter Zellen. A pLL3.7 Gateway-Vektor, der ein U6-shRNA-Kassette wurde für Cry2 Gen knockdown verwendet. shRNA2 hat einen besseren Wirkungsgrad als Knockdown shRNA1 wie durch Western-Blot-Analyse (Daten nicht gezeigt) bestimmt. Eine Synergie Luminometer wurde für Biolumineszenz Aufnahme von Zellen in einer 96-Well-Platte verwendet. Die Einstellungen für die Aufnahme sind wie folgt:. Inkubatortemperatur, 33 ° C; Integrationszeit, 15 sec; Intervallzeit, 30 min (C) Wirkung von Kleinmolekül-Inhibitoren auf celzellulären Rhythmen U2OS Reporter Zellen. Chir99021 und Roscovitin sind Inhibitoren gegen GSK-3 und CDK gerichtet sind. Die ViewLux System (Chir99021 Assay) und eine Tecan Luminometer (Roscovitine Assay) wurden für Biolumineszenz-Aufnahmen von Zellen in 384-Well-Platten verwendet. Abbildung von Referenz # 19 (Copyright 2008 National Academy of Sciences, USA) angepasst. Klicken Sie hier für eine größere Abbildung zu sehen .
Diskussion
Ein. Änderungen Aktuelle Protocol
1,1 Aufnahmegeräte und Durchsatz Überlegungen
Wegen seiner kommerziellen Verfügbarkeit hat die LumiCycle (Actimetrics) werden die am häufigsten verwendeten automatisierten Luminometer Vorrichtung zur Echtzeit-Aufzeichnung 4,5,9,19,29-31. Die LumiCycle beschäftigt Photomultiplier (PMT) als Licht-Detektoren, die extrem hohe Empfindlichkeit und geringe Geräuschentwicklung 14 bereitzustellen, und deshalb ist b...
Offenlegungen
Keine Interessenskonflikte erklärt.
Danksagungen
Diese Arbeit wurde zum Teil von der National Science Foundation (IOS-0.920.417) (ACL) unterstützt.
Materialien
| Namen Reagenz | Firma | Katalog-Nummer | Kommentare |
| DMEM | HyClone | SH30243FS | Für regelmäßige Zellwachstum |
| DMEM | Invitrogen | 12100-046 | Für Luminometrie |
| FBS | HyClone | SH3091003 | |
| Pen / Strep / Gln (100x) | HyClone | SV3008201 | |
| B-27 | Invitrogen | 17504-044 | |
| D-Luciferin | Biosynth | L-8220 | |
| Poly-L-Lysin | Sigma | P4707 | |
| Polybrene | Millipore | TR-1003-G | |
| Forskolin | Sigma | F6886 | |
| Alle anderen Chemikalien | Sigma | ||
| Ausrüstung | |||
| Gewebekultur-Inkubator | 5% CO2 bei 37 ° C | ||
| Gewebekultur Haube | BSL-2 zertifiziert | ||
| Light & Fluoreszenzmikroskop | Phasenkontrast optional | ||
| LumiCycle | Actimetrics |
Referenzen
- Reppert, S. M., Weaver, D. R. Coordination of circadian timing in mammals. Nature. 418, 935-941 (2002).
- Hastings, M. H., Reddy, A. B., Maywood, E. S. A clockwork web: circadian timing in brain and periphery, in health and disease. Nat. Rev. Neurosci. 4, 649-661 (2003).
- Nagoshi, E. Circadian gene expression in individual fibroblasts: cell-autonomous and self-sustained oscillators pass time to daughter cells. Cell. 119, 693-705 (2004).
- Welsh, D. K. Bioluminescence imaging of individual fibroblasts reveals persistent, independently phased circadian rhythms of clock gene expression. Curr. Biol. 14, 2289-2295 (2004).
- Liu, A. C. Intercellular coupling confers robustness against mutations in the SCN circadian clock network. Cell. 129, 605-616 (2007).
- Kornmann, B. System-driven and oscillator-dependent circadian transcription in mice with a conditionally active liver clock. PLoS Biol. 5, e34 (2007).
- Hogenesch, J. B., Herzog, E. D. Intracellular and intercellular processes determine robustness of the circadian clock. FEBS Lett. 585, 1427-1434 (2011).
- DeBruyne, J. P., Weaver, D. R., Reppert, S. M. Peripheral circadian oscillators require CLOCK. Curr. Biol. 17, 538-539 (2007).
- Liu, A. C. Redundant function of REV-ERBalpha and beta and non-essential role for Bmal1 cycling in transcriptional regulation of intracellular circadian rhythms. PLoS Genet. 4, e1000023 (2008).
- Zhang, E. E. A genome-wide RNAi screen for modifiers of the circadian clock in human cells. Cell. 139, 199-210 (2009).
- Baggs, J. E. Network features of the mammalian circadian clock. PLoS Biol. 7, e52 (2009).
- Hirota, T. High-throughput chemical screen identifies a novel potent modulator of cellular circadian rhythms and reveals CKIalpha as a clock regulatory kinase. PLoS Biol. 8, e1000559 (2010).
- Ukai-Tadenuma, M. Delay in feedback repression by cryptochrome 1 is required for circadian clock function. Cell. 144, 268-281 (2011).
- Yamazaki, S., Takahashi, J. S. Real-time luminescence reporting of circadian gene expression in mammals. Methods Enzymol. 393, 288-301 (2005).
- Welsh, D. K., Imaizumi, T., Kay, S. A. Real-time reporting of circadian-regulated gene expression by luciferase imaging in plants and mammalian cells. Methods Enzymol. 393, 269-288 (2005).
- Sato, T. K. Feedback repression is required for mammalian circadian clock function. Nat. Genet. 38, 312-319 (2006).
- Ueda, H. R. System-level identification of transcriptional circuits underlying mammalian circadian clocks. Nat. Genet. 37, 187-192 (2005).
- Brown, S. A. The period length of fibroblast circadian gene expression varies widely among human individuals. PLoS Biol. 3, e338 (2005).
- Hirota, T. A chemical biology approach reveals period shortening of the mammalian circadian clock by specific inhibition of GSK-3beta. Proc. Natl. Acad. Sci. U.S.A. 105, 20746-20751 (2008).
- Tiscornia, G., Singer, O., Verma, I. M. Production and purification of lentiviral vectors. Nat. Protoc. 1, 241-245 (2006).
- Ueda, H. R. A transcription factor response element for gene expression during circadian night. Nature. 418, 534-539 (2002).
- Zufferey, R., Donello, J. E., Trono, D., Hope, T. J. Woodchuck hepatitis virus posttranscriptional regulatory element enhances expression of transgenes delivered by retroviral vectors. J. Virol. 73, 2886-2892 (1999).
- Buhr, E. D., Yoo, S. H., Takahashi, J. S. Temperature as a universal resetting cue for mammalian circadian oscillators. Science. 330, 379-385 (2010).
- Balsalobre, A., Damiola, F., Schibler, . U.A serum shock induces circadian gene expression in mammalian tissue culture cells. Cell. 93, 929-937 (1998).
- Savelyev, S. A., Larsson, K. C., Johansson, A., Lundkvist, G. B. S. Slice Preparation, Organotypic Tissue Culturing and Luciferase Recording of Clock Gene Activity in the Suprachiasmatic Nucleus. J. Vis. Exp. (48), e2439 (2011).
- Akashi, M., Ichise, T., Mamine, T., Takumi, T. Molecular mechanism of cell-autonomous circadian gene expression of Period2, a crucial regulator of the mammalian circadian clock. Mol. Biol. Cell. 17, 555-565 (2006).
- Ohno, T., Onishi, Y., Ishida, N. A novel E4BP4 element drives circadian expression of mPeriod2. Nucleic Acids Res. 35, 648-655 (2007).
- Maier, B. A large-scale functional RNAi screen reveals a role for CK2 in the mammalian circadian clock. Genes Dev. 23, 708-718 (2009).
- Yoo, S. H. PERIOD2::LUCIFERASE real-time reporting of circadian dynamics reveals persistent circadian oscillations in mouse peripheral tissues. Proc. Natl. Acad. Sci. U.S.A. 101, 5339-5346 (2004).
- Liu, A. C., Lewis, W. G., Kay, S. A. Mammalian circadian signaling networks and therapeutic targets. Nat. Chem. Biol. 3, 630-639 (2007).
- Ko, C. H. Emergence of noise-induced oscillations in the central circadian pacemaker. PLoS Biol. 8, e1000513 (2010).
- Izumo, M., Johnson, C. H., Yamazaki, S. Circadian gene expression in mammalian fibroblasts revealed by real-time luminescence reporting: temperature compensation and damping. Proc. Natl. Acad. Sci. U.S.A. 100, 16089-16094 (2003).
- Izumo, M., Sato, T. R., Straume, M., Johnson, C. H. Quantitative analyses of circadian gene expression in mammalian cell cultures. PLoS Comput. Biol. 2, e136 (2006).
- Chen, Z. Identification of diverse modulators of central and peripheral circadian clocks by high-throughput chemical screening. Proc. Natl. Acad. Sci. U.S.A. 109, 101-106 (2011).
- Yamaguchi, S. Synchronization of cellular clocks in the suprachiasmatic nucleus. Science. 302, 1408-1412 (2003).
- Akashi, M., Hayasaka, N., Yamazaki, S., Node, K. Mitogen-activated protein kinase is a functional component of the autonomous circadian system in the suprachiasmatic nucleus. J. Neurosci. 28, 4619-4623 (2008).
- Hoshino, H., Nakajima, Y., Ohmiya, Y. Luciferase-YFP fusion tag with enhanced emission for single-cell luminescence imaging. Nat. Methods. 4, 637-639 (2007).
- Asai, M. Visualization of mPer1 transcription in vitro: NMDA induces a rapid phase shift of mPer1 gene in cultured SCN. Curr. Biol. 11, 1524-1527 (2001).
- Wilsbacher, L. D. Photic and circadian expression of luciferase in mPeriod1-luc transgenic mice in vivo. Proc. Natl. Acad. Sci. U.S.A. 99, 489-494 (2002).
- Yamazaki, S. Resetting central and peripheral circadian oscillators in transgenic rats. Science. 288, 682-685 (2000).
- Welsh, D. K., Noguchi, T., Yuste, R. Cellular bioluminescence imaging. Imaging: A Laboratory Manual. , 369-385 (2011).
- Nakajima, Y. Enhanced beetle luciferase for high-resolution bioluminescence imaging. PLoS One. 5, e10011 (2010).
- Guilding, C. A riot of rhythms: neuronal and glial circadian oscillators in the mediobasal hypothalamus. Mol. Brain. 2, 28 (2009).
- O'Neill, J. S. cAMP-dependent signaling as a core component of the mammalian circadian pacemaker. Science. 320, 949-953 (2008).
- Fuller, P. M., Lu, J., Saper, C. B. Differential rescue of light- and food-entrainable circadian rhythms. Science. 320, 1074-1077 (2008).
- Mukherjee, S. Knockdown of Clock in the ventral tegmental area through RNA interference results in a mixed state of mania and depression-like behavior. Biol. Psychiatry. 68, 503-511 (2010).
- Saijo, K. A Nurr1/CoREST pathway in microglia and astrocytes protects dopaminergic neurons from inflammation-induced death. Cell. 137, 47-59 (2009).
- Elias, G. M. Synapse-specific and developmentally regulated targeting of AMPA receptors by a family of MAGUK scaffolding proteins. Neuron. 52, 307-320 (2006).
- Isojima, Y. CKIepsilon/delta-dependent phosphorylation is a temperature-insensitive, period-determining process in the mammalian circadian clock. Proc. Natl. Acad. Sci. U.S.A. 106, 15744-15749 (2009).
- Bucan, M., Abel, T. The mouse: genetics meets behaviour. Nat. Rev. Genet. 3, 114-123 (2002).
- Hughes, M. E. Harmonics of circadian gene transcription in mammals. PLoS Genet. 5, e1000442 (2009).
- Atwood, A. Cell-autonomous circadian clock of hepatocytes drives rhythms in transcription and polyamine synthesis. Proc. Natl. Acad. Sci. U.S.A. 108, 18560-18565 (2011).
- Panda, S. Coordinated transcription of key pathways in the mouse by the circadian clock. Cell. 109, 307-320 (2002).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten