È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Monitoraggio Cell-autonome Ritmi orologio circadiano di espressione genica mediante bioluminescenza Reporter luciferasi
In questo articolo
Riepilogo
Orologi circadiani funzionare all'interno di singole celle, cioè, sono cellule autonome. Qui, descriviamo i metodi per la generazione di cellule autonome modelli di orologio con tecniche non invasive, luciferasi a base di tecnologia in tempo reale bioluminescenza. Cellule Reporter fornire trattabili, sistemi modello per lo studio della biologia funzionale circadiano.
Abstract
Nei mammiferi, molti aspetti del comportamento e della fisiologia, come cicli sonno-veglia e il metabolismo del fegato sono regolati da orologi circadiani endogeni (recensione 1,2). Il circadiano cronometraggio è un sistema gerarchico multi-oscillatore di rete, con l'orologio centrale situato nel nucleo soprachiasmatico (SCN), la sincronizzazione e il coordinamento extra-SCN e periferiche orologi altrove 1,2. Le singole cellule sono le unità funzionali per la generazione e il mantenimento dei ritmi circadiani 3,4, e questi oscillatori di diversi tipi di tessuto della quota di un organismo molto simile meccanismo biochimico feedback negativo. Tuttavia, a causa di interazioni a livello della rete neuronale nel SCN e attraverso ritmiche, segnali sistemici a livello organismal, ritmi circadiani a livello organismal non sono necessariamente cellula-autonoma 5-7. Rispetto agli studi tradizionali di attività locomotoria in vivo e ex vivo SCN espianti, cell-base in vitro permettono la scoperta di difetti circadiani cellule autonome 5,8. Strategica, a base di cellule modelli sono più trattabili sperimentalmente per la caratterizzazione fenotipica e rapida scoperta di meccanismi di clock di base 5,8-13.
Dato che i ritmi circadiani sono dinamiche, misure longitudinali con alta risoluzione temporale sono necessari per valutare la funzione orologio. Negli ultimi anni, registrazione in tempo reale utilizzando bioluminescenza luciferasi di lucciola come reporter è diventata una tecnica comune per studiare ritmi circadiani nei mammiferi 14,15, in quanto permette di esaminare la persistenza e dinamiche di ritmi molecolare. Per monitorare ritmi circadiani cellule autonome di espressione genica, reporter luciferasi può essere introdotto nelle cellule tramite trasfezione transiente 13,16,17 o trasduzione stabile 5,10,18,19. Qui si descrive un protocollo stabile trasduzione con lentivirus-mediata consegna del gene. Tl sistema vettore lentivirale è superiore ai metodi tradizionali come trasfezione transiente e trasmissione germinale causa della sua efficienza e versatilità: consente consegna efficiente e stabile integrazione nel genoma dell'ospite sia divisione e non divisione delle cellule 20. Una volta che una linea cellulare reporter viene stabilita, la dinamica della funzione orologio può essere monitorata tramite registrazione bioluminescenza. Per prima cosa descrivere la generazione di P (Per2)-d linee giornalista Luc, e quindi i dati presenti di questo e di altri giornalisti circadiani. In questi saggi, fibroblasti di topo 3T3 e osteosarcoma U2OS cellule umani sono usati come modelli cellulari. Discutiamo anche diversi modi di utilizzo di questi modelli di clock in studi circadiani. Metodi descritti qui può essere applicato ad una grande varietà di tipi cellulari per studiare le basi cellulari e molecolari di orologi circadiani, e possono risultare utili per affrontare problemi in altri sistemi biologici.
Protocollo
1. Costruzione di lentivirali Reporter luciferasi
Un costrutto reporter di mammifero circadiano solitamente contiene una cassetta di espressione in cui è fuso con un promotore circadiano il gene della luciferasi. Sia legatura e ricombinazione basata strategie sono comunemente usati per la clonazione del DNA. Come esempio, qui si descrive un metodo basato ricombinazione clonazione per generare una Gateway (Per2) P-d giornalista lentivirale Luc, in cui la luciferasi destabilizzato (d Luc) è sotto il controllo del promotore di topo Per2.
- Clonazione di Per2 promotore. Utilizzare PCR per amplificare il frammento di DNA Per2 promotore di 526 bp, a monte del sito di inizio trascrizione da un Per2 topo clone BAC 9-13, utilizzando un primer forward (5'-CTCGAGCGGATTACCGAGGCTGGTCACG TC-3 ') e un primer reverse (5' -CTCGAGTCCCTTGCTCGGCCCGTCAC TTGG-3 '), e clone in pENTR5'-vettore TOPO (Invitrogen) per generarepENTR5'-P (Per2).
- Clonazione di d Luc. La Luc d contiene il gene della luciferasi di lucciola e un terminale C sequenza PEST per una rapida degradazione proteica come precedentemente descritto 21. Utilizzare PCR per amplificare il frammento di DNA d Luc, e clone in pENTR / D-TOPO vettore (Invitrogen) per generare pENTR / Dd Luc.
- Costruzione del vettore reporter. Mescolare i due plasmidi pENTR, pENTR5'-P (Per2) e pENTR / Dd Luc, con il vettore di destinazione lentivirale pLV7-Bsd (Bsd, gene di resistenza blasticidina), ed eseguire la reazione di ricombinazione con Clonase per generare un pLV7-Bsd-P (Per2) d-Luc reporter (Figura 1). pLV7-BSD è una versione modificata (realizzato nel nostro laboratorio) di pLenti6/R4R2/V5-DEST (Invitrogen) in cui sono stati inseriti il virus dell'epatite woodchuck post-trascrizionale di regolamentazione elemento (WPRE) sequenze 22 immediatamente a valle dell'espressione ca ssette per migliorare l'espressione genica.
2. Produzione di particelle lentivirali
1. Cellule semi 293T (giorno 1)
- Crescere renali embrionali umane (HEK) 293T al 90-100% di confluenza in regolare DMEM supplementato con 10% FBS e 1x Penicillina-Streptomicina-Glutammina (PSG) su piatti di coltura 10 centimetri. (Cellule in rapida crescita con il numero di passaggio basso sono fondamentali per la trasfezione efficiente.)
- Prima della semina delle cellule per la trasfezione, coat piastre da 6 pozzetti di coltura con l'aggiunta di 1 ml di 0,001% di poli-L-lisina in PBS ad ogni pozzetto ed incubare a temperatura ambiente per 20 min. Aspirare la soluzione e lavare una volta con PBS 1x prima dell'uso.
- Separa celle 293T con tripsina e di semi di 0.75 x 10 6 cellule su ciascun pozzetto di pre-piastre rivestite con 2 ml di DMEM regolare. Swirl le piastre accuratamente per ottenere una distribuzione uniforme delle cellule in ciascun pozzetto. Crescere le cellule in incubatore a 37 ° C per una notte.
- Osservare le cellule seminate dal giorno 1. Cellulare dovrebbe raggiungere la confluenza del 80-90%.
- Preparare mix plasmide trasfezione in una provetta da microcentrifuga 1,5 ml aggiungendo 2 pg di DNA di un plasmide reporter di lentivirale (ad esempio, pLV7-P (Per2) d-Luc; Liu lab) ed i tre vettori di imballaggio (1,3 mcg Gag / Pol, 0,5 mg Rev, e 0,7 mg VSVG, Invitrogen). Come controllo sia per trasfezione e successiva infezione, di solito comprendono un pozzo supplementare per trasfezione in un vettore lentivirale espressione di GFP, pLV156-CMV-EGFP (Figura 1A), harboring maggiore proteina fluorescente verde (EGFP) sotto il controllo del promotore CMV come descritto in precedenza 20.
- Aggiungere 100 ml di 0,25 M CaCl 2 (diluito con DNasi / RNasi-free DDH 2 O da 2,5 magazzino M) al mix plasmide al punto 2 e mescolare accuratamente. Aggiungere quindi 100 ml di soluzione di 2x BBS (50 mM BES, 280 mM NaCl, 1,5 mM Na 2 HPO 4, pH 6,95) e mescolare delicatamente ma accuratamente. Incubare la miscela DNA a temperatura ambiente per 15 min.
- In attesa, aspirare media da cellule 293T e cambiare a 2 ml di terreno fresco. Riportare la piastra alla incubatore per almeno 10 minuti per equilibrare pH medio prima della transfezione.
- Aggiungi mix di trasfezione dal punto 3 al 293T cellule goccia a goccia. Agitare delicatamente la piastra e osservare la formazione di particelle al microscopio. Incubare al 5% di CO 2, 37 ° C per una notte. (Formazione di particelle di precisione del Capo 4 precipitato / DNA è fondamentale per la trasfezione efficiente.)
3. Particelle virali Harvest (giorni 3-4)
- Circa 16 ore dopo la trasfezione (giorno 3) con cui cellule tempo dovrebbe raggiungere il 100% di confluenza, aspirare media da cellule e sostituire con 2 ml di DMEM fresco regolare. Incubare a 37 ° C per una notte.
- Il giorno 4, valutare l'efficienza di trasfezione osservando espressione EGFP nel transfection cellule di controllo (efficienza di trasfezione di 90-100% con l'espressione di EGFP elevata è un fattore predittivo affidabile di una buona preparazione virale.)
- Raccogliere il mezzo contenente secrete, particelle virali infettive. Centrifugare a> 2000 xg per 5 minuti per rimuovere i residui di cellule 293T e raccogliere il surnatante contenente virus. In alternativa, il supporto può essere eliminato con un filtro da 0,45 micron membrana. Le particelle virali sono pronti per l'uso in infezioni.
3. Infezione di cellule 3T3
1. Cellule 3T3 Seed (giorno 3)
Spalato e numero di sementi adeguate (~ 12.000) di cellule 3T3 su un 12-pozzetti per ottenere 20-30% confluenza di giorno successivo. Incubare a 37 ° C per una notte.
2. Infettare le cellule 3T3 (giorno 4)
- Osservare le cellule seminate. Confluenza del 20-30% (meno del 50%) è desiderato per l'infezione.
- Aggiungere polibrene ad una concentrazione finale di 5 pg / ml per il mezzo contenente raccolti viraleparticelle. Mescolare bene pipettando.
- Aspirare il terreno da cellule 3T3, e aggiungere 1 ml della suddetta miscela virale per pozzetto. Incubare a 37 ° C per una notte. (Polybrene viene utilizzato per migliorare l'efficienza dell'infezione, ma non è assolutamente necessaria. Come può essere tossico per alcune cellule, previo controllo è raccomandato.)
3. Selezionare cellule infette (giorno 5 e successivi)
- Ventiquattro ore dopo l'infezione, aspirare terreno contenente virus e polibrene dalle cellule infette, lavare una volta con PBS 1x, e cambiare con terreno fresco. Incubare a 37 ° C per una notte per il recupero e la crescita.
- Quando confluenti (di solito 1-2 giorni dopo), dividere le celle e incubare a 37 ° C per una notte.
- Il giorno seguente, medie aspirato dalle cellule confluenza (<50% è desiderato) e sostituirlo con mezzo fresco contenente 10 ug / ml di blasticidina per selezionare stabilmente trasdotte. (Blasticidina kill-curva deve essere empiricamente determinato per una linea cellulare di particolare.)
- Cambia in terreno fresco contenente blasticidina ogni 2-3 giorni per la selezione continua di antibiotico-resistenti cellule che esprimono i giornalisti di clock (in genere 4-6 giorni in totale).
4. Registrazione bioluminescenza di celle Reporter
1. Seed giornalista cellule
Propagare blasticidina cellule resistenti giornalista e dividere su 35 mm piastre di coltura. Incubare a 37 ° C fino confluenti. Noi di solito preparare ≥ 3 piatti per ogni linea cellulare giornalista sotto ogni condizione di fenotipizzazione circadiano.
2. Sincronizzazione e modifica di supporto di registrazione
- Aspirare il terreno dalle cellule giornalista confluenti, lavare una volta con PBS, e sostituirlo con DMEM contenente 10 pM forskolina (o 200 nM desametasone). Incubare a 37 ° C per 1 ora per sincronizzare le cellule. (In alternativa, le cellule possono essere sincronizzati da 23 cicli di temperatura o scosse siero 24.)
- Durante l'attesa, la preparazione di registrazioneMedio ING per 3T3 come segue: 1x DMEM (HyClone) contenente 10% FBS, 1x Pen / Strep / Gln, 1 pM forskolin, 1 mM luciferina, HEPES 25 mM, pH 7,4. E la concentrazione sierica forskolin possono essere determinate empiricamente. Per le cellule molto debole, rosso fenolo terreno privo possono essere utilizzati.
- Alla fine del trattamento forskolina, aspirare medio e sostituirlo con supporto di registrazione appena fatta.
3. Registrazione bioluminescenza di cellule del reporter
- Dopo il cambiamento medio, coprire piastre di coltura con 40 millimetri coprioggetto sterili e tenuta in posizione con grasso per vuoto per evitare l'evaporazione.
- Caricare le stoviglie sul luminometro LumiCycle, che è mantenuta all'interno di un incubatore a 36 ° C senza H 2 O e CO 2.
- Inizio registrazione in tempo reale bioluminescenza. Di solito registrare ritmi per 1 settimana, seguito dal cambiamento medio e registrazione continua per la seconda settimana (vedi Savelyev et al. Per i dettagli) 25. (Per la registrazione di 96-noill piatti, Synergy SL2 è stato usato come dispositivo di registrazione, vedere discussione 1.1 per i dettagli).
5. Analisi dei dati e presentazione
Cellule Reporter facilitare registrazione ad alta risoluzione quantitativa luminescenza, fondamentale per determinare effetti fenotipici sulla funzione orologio circadiano. Per ottenere i parametri circadiani, compresi l'introduzione, durata del periodo, l'ampiezza del ritmo, e il tasso di smorzamento, si utilizza il programma di analisi LumiCycle (Actimetrics) per analizzare i dati bioluminescenza 5,14. Brevemente, i dati grezzi sono basale installare prima, e basale sottratto dati sono montati una sinusoide, da cui sono determinati i parametri. Per i campioni che mostrano ritmi persistenti, la bontà di adattamento del> 90% si ottiene in genere. A causa di alta bioluminescenza transitoria upon cambiamento medio, di solito esclude il primo ciclo di analisi dati.
Per la presentazione dei dati, di solito tracciare i dati grezzi (bioluminescenza, conteggi / sec) contro time (giorni). Se necessario, i dati di base-sottratti possono essere tracciati per confrontare ampiezza e fase.
6. Risultati rappresentativi
1. Giornalisti circadiani fase-specifici
L'orologio circadiano è basato su un meccanismo biochimico feedback negativo 1. L'anello di retroazione nucleo è costituito da attivatori trascrizionali Bmal1 e orologio, e repressori PERS e cristallina, che agiscono sulle circadiani E / E'-box per la produzione di elementi enhancer del gene espressione ritmica (con fase mattina, ad esempio, Rev-erb α). Il ciclo di base regola e integra almeno altri due circadiani cis-elementi, l'elemento di legame DBP/E4BP4 (D-box, per la fase giorno, ad esempio, PER3) e il ROR / REV-ERB elemento di rilegatura (RRE, per la fase notte, ad esempio, Bmal1) 17. Regolazione combinatoria da più elementi circadiani può generare nuove fasi intermedie. Per esempio, Cry1 trascrizionezione è mediata da tre elementi circadiani (cioè E / E'-box e D-box elementi del promotore e RREs nel primo introne del gene Cry1), dando luogo alla distinta Cry1 sera-tempo di fase 13.
Sulla base di questi meccanismi di regolazione genica, abbiamo generato quattro costrutti diversi giornalista: P (Per2)-d Luc e P (Cry1)-d reporter Luc contenenti sia E / E'-box e D-box elementi nella regione regolatrice 17, 26,27, P (Cry1) - Intron-d Luc rappresenta regolazione combinatoria da tutti e tre gli elementi (ad esempio, E / E'-box, D-box, e RRE), 13,17, e P (Bmal1)-d Luc regolamentato esclusivamente da RRE 9,17,19,21. Abbiamo introdotto questi giornalisti in cellule 3T3 per produrre le fasi previste distinte di espressione giornalista (Figura 2).
2. Knockdown Gene tramite RNAi e farmacologicamente Active composti
Quando è alta efficienza di trasfezione, siRNA sintetico può essere trasfettate in cellule di abbattere espressione genica. Quando trasfezione è tecnicamente difficile, un vettore di espressione shRNA può essere stabilmente trasdotti nelle cellule tramite infezione lentivirale, così che shRNA prodotta dalla cella viene elaborato per siRNA per knockdown gene (KD). Presentiamo qui di effetti KD Cry1 e Cry2 geni usando siRNA in cellule U2OS (Figura 3A) e shRNA in cellule 3T3 usando un vettore di espressione pLL3.7 Gateway 9 (Figura 3B). Oltre alle cellule 3T3, il modello U2OS è diventato un altro modello preminente orologio cellulare soprattutto perché soddisfa i requisiti chiave per high-throughput screening di librerie disponibili in commercio siRNA umani (ad esempio, l'origine umana, in grado di generare robusti ritmi circadiani, funzione convalidato di tutti i geni orologio noti e suscettibili di transfezione altamente efficiente eregistrazione luminescenza quantitativa). In entrambi i tipi di cellule, mediato da RNAi KD portato a fenotipi orologio coerenti con knockout mouse precedente (KO) e cellulari studi KD 5,10,11,28. Ad esempio, Cry1 KD periodo accorcia lunghezza e riduce la persistenza del ritmo, mentre Cry2 KD allunga periodo. Inoltre, selezionate piccole molecole possono essere utilizzate per farmacologicamente bersaglio e funzione proteica perturbano (Figura 3C).
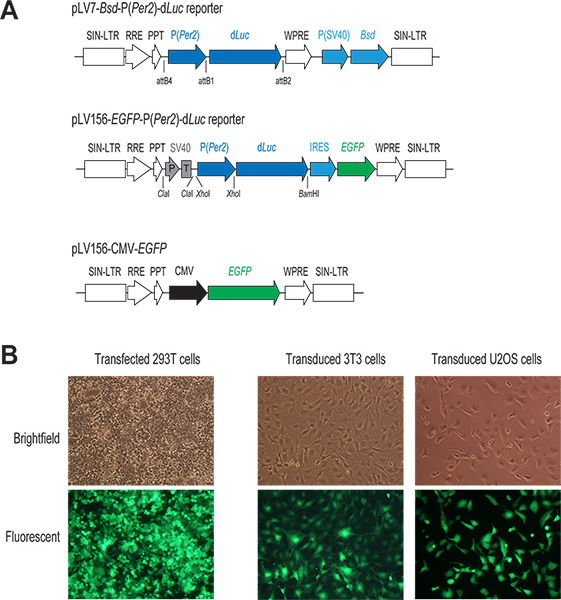
Figura 1. Il lentivirus-mediata gene delivery system. (A) Rappresentazione schematica di due lentivirale P (Per2)-d vettori giornalista Luc e CMV-EGFP costruire. Solo la regione di integrazione nel genoma della cellula ospite viene mostrato. In entrambi i costrutti reporter, la trascrizione di d Luc è sotto il controllo diretto del promotore Per2. Nel pLV7-Bsd-P (Per2)-d Luc vettore (ricombinazione basata clonazione), un gene di resistenza coexpressed blasticidina (Bsd) facilita la selezione di cellule infette. Nel pLV156-P (Per2) d-Luc vettoriale (legatura basata clonazione), traduzione EGFP è mediata da un sito interno voce ribosoma (IRES) a valle di Luc d, consentendo l'osservazione visiva e smistamento FACS di cellule infette. Inoltre, un promotore SV40 / terminatore (P / T) viene utilizzato come isolante (vedere discussione 1.3). Nel CMV-EGFP controllo vettoriale, espressione EGFP è sotto il controllo di un forte promotore CMV. (B) di immagini fluorescenti GFP-transfettate esprimenti e cellule infettate. Tipicamente, ottenere elevata efficienza sia in trasfezione transiente di cellule 293T e nelle infezioni lentivirali di linee cellulari di nostro interesse, come indicato dall'espressione GFP in queste cellule. Clicca qui per ingrandire la figura .

Figura 2. Fase-specifico espressione del reporter bioluminescenza in cellule 3T3 I vettori lentivirali del reporter usati in questo esperimento sono pLV7-BSD-P (Per2) d-Luc, P (Cry1) d-Luc, P (Cry1) -. Intron d-Luc, e P (Bmal1)-d Luc. Ogni giornalista presenta una distinta fase di oscillazione, come indicato dalle frecce. Mentre il Per2 e Cry1 unità promotori bioluminescenza picco a mattina-day fasi e il promotore Bmal1 nella fase notte, regolazione combinatoria dalla P (Cry1) - Intron ospitare E-box, D-box, e gli elementi RRE conferisce fase serata di bioluminescenza picco . Clicca qui per ingrandire la figura .
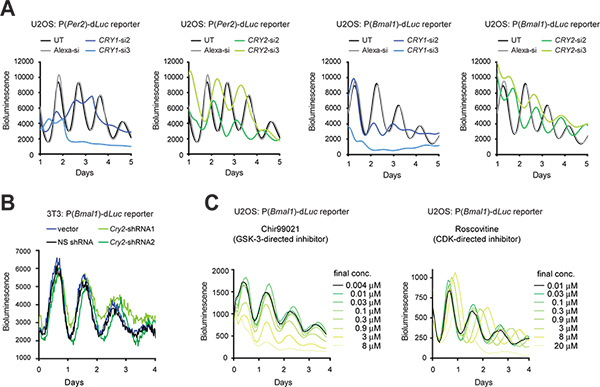
Figura 3. Perturbazione genetica e farmacologica dei ritmi circadiani bioluminescenza nelle cellule reporter. (A) Effetti di Cry1 e Cry2 atterramento da siRNA sui ritmi cellulari delle cellule del reporter U2OS. Luminometro LumiCycle utilizzata per la registrazione bioluminescenza di cellule in piastre da 35 mm. Figura è adattato da Reference # 10, con il permesso di Elsevier (2009). (B) Effetti di Cry2 knockdown da shRNAs sui ritmi cellulari delle cellule 3T3 giornalista. Un pLL3.7 vettore contenente un Gateway U6-shRNA cassetta è stata utilizzata per Cry2 knockdown gene. shRNA2 ha un'efficienza migliore di knockdown shRNA1 come determinato mediante analisi Western blot (dati non mostrati). Un luminometro sinergia è stata utilizzata per la registrazione bioluminescenza di cellule in una piastra a 96 pozzetti. Le impostazioni di registrazione sono le seguenti: temperatura dell'incubatore., 33 ° C, tempo di integrazione, 15 sec; intervallo di tempo, 30 min (C) Effetti di piccole molecole inibitrici di celritmi lular di cellule giornalista U2OS. Chir99021 e Roscovitine sono inibitori diretti contro GSK-3 e CDK, rispettivamente. Il sistema ViewLux (Chir99021 assay) e un luminometro Tecan (Roscovitine saggio) sono stati utilizzati per le registrazioni bioluminescenza di cellule in piastre a 384 pozzetti. Figura di riferimento è tratto da # 19 (Copyright 2008 National Academy of Sciences, USA). Clicca qui per ingrandire la figura .
Discussione
1. Modifiche al protocollo corrente
1,1 i dispositivi di registrazione e le considerazioni di throughput
A causa della sua disponibilità commerciale, il LumiCycle (Actimetrics) è diventato lo strumento più comunemente usato luminometro automatizzati per registrazione in tempo reale 4,5,9,19,29-31. Il LumiCycle impiega tubi fotomoltiplicatori (PMT) come rivelatori di luce, che forniscono elevata sensibilità e basso rumore 14, ed è quindi part...
Divulgazioni
Nessun conflitto di interessi dichiarati.
Riconoscimenti
Questo lavoro è stato sostenuto in parte dalla National Science Foundation (IOS-0920417) (ACL).
Materiali
| Nome del reagente | Azienda | Numero di catalogo | Commenti |
| DMEM | HyClone | SH30243FS | Per regolare la crescita cellulare |
| DMEM | Invitrogen | 12100-046 | Per luminometria |
| FBS | HyClone | SH3091003 | |
| Pen / Strep / Gln (100x) | HyClone | SV3008201 | |
| B-27 | Invitrogen | 17504-044 | |
| D-Luciferin | Biosynth | L-8220 | |
| Poli-L-lisina | Sigma | P4707 | |
| Polibrene | Millipore | TR-1003-G | |
| Forskolin | Sigma | F6886 | |
| Tutti gli altri prodotti chimici | Sigma | ||
| Attrezzatura | |||
| Coltura tissutale incubatore | 5% di CO2 a 37 ° C | ||
| Tissue cultura cappuccio | BSL-2 certificata | ||
| Light & microscopio a fluorescenza | Contrasto di fase opzionale | ||
| LumiCycle | Actimetrics |
Riferimenti
- Reppert, S. M., Weaver, D. R. Coordination of circadian timing in mammals. Nature. 418, 935-941 (2002).
- Hastings, M. H., Reddy, A. B., Maywood, E. S. A clockwork web: circadian timing in brain and periphery, in health and disease. Nat. Rev. Neurosci. 4, 649-661 (2003).
- Nagoshi, E. Circadian gene expression in individual fibroblasts: cell-autonomous and self-sustained oscillators pass time to daughter cells. Cell. 119, 693-705 (2004).
- Welsh, D. K. Bioluminescence imaging of individual fibroblasts reveals persistent, independently phased circadian rhythms of clock gene expression. Curr. Biol. 14, 2289-2295 (2004).
- Liu, A. C. Intercellular coupling confers robustness against mutations in the SCN circadian clock network. Cell. 129, 605-616 (2007).
- Kornmann, B. System-driven and oscillator-dependent circadian transcription in mice with a conditionally active liver clock. PLoS Biol. 5, e34 (2007).
- Hogenesch, J. B., Herzog, E. D. Intracellular and intercellular processes determine robustness of the circadian clock. FEBS Lett. 585, 1427-1434 (2011).
- DeBruyne, J. P., Weaver, D. R., Reppert, S. M. Peripheral circadian oscillators require CLOCK. Curr. Biol. 17, 538-539 (2007).
- Liu, A. C. Redundant function of REV-ERBalpha and beta and non-essential role for Bmal1 cycling in transcriptional regulation of intracellular circadian rhythms. PLoS Genet. 4, e1000023 (2008).
- Zhang, E. E. A genome-wide RNAi screen for modifiers of the circadian clock in human cells. Cell. 139, 199-210 (2009).
- Baggs, J. E. Network features of the mammalian circadian clock. PLoS Biol. 7, e52 (2009).
- Hirota, T. High-throughput chemical screen identifies a novel potent modulator of cellular circadian rhythms and reveals CKIalpha as a clock regulatory kinase. PLoS Biol. 8, e1000559 (2010).
- Ukai-Tadenuma, M. Delay in feedback repression by cryptochrome 1 is required for circadian clock function. Cell. 144, 268-281 (2011).
- Yamazaki, S., Takahashi, J. S. Real-time luminescence reporting of circadian gene expression in mammals. Methods Enzymol. 393, 288-301 (2005).
- Welsh, D. K., Imaizumi, T., Kay, S. A. Real-time reporting of circadian-regulated gene expression by luciferase imaging in plants and mammalian cells. Methods Enzymol. 393, 269-288 (2005).
- Sato, T. K. Feedback repression is required for mammalian circadian clock function. Nat. Genet. 38, 312-319 (2006).
- Ueda, H. R. System-level identification of transcriptional circuits underlying mammalian circadian clocks. Nat. Genet. 37, 187-192 (2005).
- Brown, S. A. The period length of fibroblast circadian gene expression varies widely among human individuals. PLoS Biol. 3, e338 (2005).
- Hirota, T. A chemical biology approach reveals period shortening of the mammalian circadian clock by specific inhibition of GSK-3beta. Proc. Natl. Acad. Sci. U.S.A. 105, 20746-20751 (2008).
- Tiscornia, G., Singer, O., Verma, I. M. Production and purification of lentiviral vectors. Nat. Protoc. 1, 241-245 (2006).
- Ueda, H. R. A transcription factor response element for gene expression during circadian night. Nature. 418, 534-539 (2002).
- Zufferey, R., Donello, J. E., Trono, D., Hope, T. J. Woodchuck hepatitis virus posttranscriptional regulatory element enhances expression of transgenes delivered by retroviral vectors. J. Virol. 73, 2886-2892 (1999).
- Buhr, E. D., Yoo, S. H., Takahashi, J. S. Temperature as a universal resetting cue for mammalian circadian oscillators. Science. 330, 379-385 (2010).
- Balsalobre, A., Damiola, F., Schibler, . U.A serum shock induces circadian gene expression in mammalian tissue culture cells. Cell. 93, 929-937 (1998).
- Savelyev, S. A., Larsson, K. C., Johansson, A., Lundkvist, G. B. S. Slice Preparation, Organotypic Tissue Culturing and Luciferase Recording of Clock Gene Activity in the Suprachiasmatic Nucleus. J. Vis. Exp. (48), e2439 (2011).
- Akashi, M., Ichise, T., Mamine, T., Takumi, T. Molecular mechanism of cell-autonomous circadian gene expression of Period2, a crucial regulator of the mammalian circadian clock. Mol. Biol. Cell. 17, 555-565 (2006).
- Ohno, T., Onishi, Y., Ishida, N. A novel E4BP4 element drives circadian expression of mPeriod2. Nucleic Acids Res. 35, 648-655 (2007).
- Maier, B. A large-scale functional RNAi screen reveals a role for CK2 in the mammalian circadian clock. Genes Dev. 23, 708-718 (2009).
- Yoo, S. H. PERIOD2::LUCIFERASE real-time reporting of circadian dynamics reveals persistent circadian oscillations in mouse peripheral tissues. Proc. Natl. Acad. Sci. U.S.A. 101, 5339-5346 (2004).
- Liu, A. C., Lewis, W. G., Kay, S. A. Mammalian circadian signaling networks and therapeutic targets. Nat. Chem. Biol. 3, 630-639 (2007).
- Ko, C. H. Emergence of noise-induced oscillations in the central circadian pacemaker. PLoS Biol. 8, e1000513 (2010).
- Izumo, M., Johnson, C. H., Yamazaki, S. Circadian gene expression in mammalian fibroblasts revealed by real-time luminescence reporting: temperature compensation and damping. Proc. Natl. Acad. Sci. U.S.A. 100, 16089-16094 (2003).
- Izumo, M., Sato, T. R., Straume, M., Johnson, C. H. Quantitative analyses of circadian gene expression in mammalian cell cultures. PLoS Comput. Biol. 2, e136 (2006).
- Chen, Z. Identification of diverse modulators of central and peripheral circadian clocks by high-throughput chemical screening. Proc. Natl. Acad. Sci. U.S.A. 109, 101-106 (2011).
- Yamaguchi, S. Synchronization of cellular clocks in the suprachiasmatic nucleus. Science. 302, 1408-1412 (2003).
- Akashi, M., Hayasaka, N., Yamazaki, S., Node, K. Mitogen-activated protein kinase is a functional component of the autonomous circadian system in the suprachiasmatic nucleus. J. Neurosci. 28, 4619-4623 (2008).
- Hoshino, H., Nakajima, Y., Ohmiya, Y. Luciferase-YFP fusion tag with enhanced emission for single-cell luminescence imaging. Nat. Methods. 4, 637-639 (2007).
- Asai, M. Visualization of mPer1 transcription in vitro: NMDA induces a rapid phase shift of mPer1 gene in cultured SCN. Curr. Biol. 11, 1524-1527 (2001).
- Wilsbacher, L. D. Photic and circadian expression of luciferase in mPeriod1-luc transgenic mice in vivo. Proc. Natl. Acad. Sci. U.S.A. 99, 489-494 (2002).
- Yamazaki, S. Resetting central and peripheral circadian oscillators in transgenic rats. Science. 288, 682-685 (2000).
- Welsh, D. K., Noguchi, T., Yuste, R. Cellular bioluminescence imaging. Imaging: A Laboratory Manual. , 369-385 (2011).
- Nakajima, Y. Enhanced beetle luciferase for high-resolution bioluminescence imaging. PLoS One. 5, e10011 (2010).
- Guilding, C. A riot of rhythms: neuronal and glial circadian oscillators in the mediobasal hypothalamus. Mol. Brain. 2, 28 (2009).
- O'Neill, J. S. cAMP-dependent signaling as a core component of the mammalian circadian pacemaker. Science. 320, 949-953 (2008).
- Fuller, P. M., Lu, J., Saper, C. B. Differential rescue of light- and food-entrainable circadian rhythms. Science. 320, 1074-1077 (2008).
- Mukherjee, S. Knockdown of Clock in the ventral tegmental area through RNA interference results in a mixed state of mania and depression-like behavior. Biol. Psychiatry. 68, 503-511 (2010).
- Saijo, K. A Nurr1/CoREST pathway in microglia and astrocytes protects dopaminergic neurons from inflammation-induced death. Cell. 137, 47-59 (2009).
- Elias, G. M. Synapse-specific and developmentally regulated targeting of AMPA receptors by a family of MAGUK scaffolding proteins. Neuron. 52, 307-320 (2006).
- Isojima, Y. CKIepsilon/delta-dependent phosphorylation is a temperature-insensitive, period-determining process in the mammalian circadian clock. Proc. Natl. Acad. Sci. U.S.A. 106, 15744-15749 (2009).
- Bucan, M., Abel, T. The mouse: genetics meets behaviour. Nat. Rev. Genet. 3, 114-123 (2002).
- Hughes, M. E. Harmonics of circadian gene transcription in mammals. PLoS Genet. 5, e1000442 (2009).
- Atwood, A. Cell-autonomous circadian clock of hepatocytes drives rhythms in transcription and polyamine synthesis. Proc. Natl. Acad. Sci. U.S.A. 108, 18560-18565 (2011).
- Panda, S. Coordinated transcription of key pathways in the mouse by the circadian clock. Cell. 109, 307-320 (2002).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon