Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Surveillance de cellules autonomes Rythmes horloge circadienne de l'expression génique par Reporters Bioluminescence luciférase
Dans cet article
Résumé
Horloges circadiennes fonctionner dans des cellules individuelles, c'est à dire qu'ils sont des cellules autonomes. Ici, nous décrivons les méthodes pour générer des modèles d'horloge cellules autonomes utilisant des méthodes non-invasives, la luciférase technologie basée sur la bioluminescence en temps réel. Cellules Reporter fournir traitables, des systèmes de modèles fonctionnels pour l'étude de la biologie circadienne.
Résumé
Chez les mammifères, de nombreux aspects du comportement et de la physiologie comme cycles veille-sommeil et le métabolisme du foie sont régis par horloges circadiennes endogènes (revu 1,2). Le rythme circadien garde-temps est un système hiérarchique multi-oscillateur réseau, avec l'horloge centrale située dans le noyau suprachiasmatique (NSC) la synchronisation et la coordination des horloges extra-SCN et périphérique ailleurs 1,2. Les cellules individuelles sont des unités fonctionnelles pour la production et l'entretien des rythmes circadiens 3,4, et ces oscillateurs de différents types de tissus de la part d'un organisme remarquablement semblable mécanisme biochimique rétroaction négative. Toutefois, en raison des interactions au niveau du réseau neuronal dans le SCN et à travers rythmiques, les indices systémiques au niveau organismique, les rythmes circadiens au niveau organismique ne sont pas nécessairement cell-autonomous 5-7. Par rapport aux études traditionnelles de l'activité locomotrice in vivo et ex vivo d'explants SCN, cell-fondé des tests in vitro permettent la découverte de cellules autonomes défauts circadiens 5,8. Stratégiquement, à base de cellules modèles sont plus dociles expérimentalement pour la caractérisation phénotypique et la découverte rapide des mécanismes d'horloge de base 5,8-13.
Parce que les rythmes circadiens sont dynamiques, les mesures longitudinales avec une haute résolution temporelle sont nécessaires pour évaluer la fonction horloge. Ces dernières années, l'enregistrement bioluminescence en temps réel en utilisant la luciférase de luciole en tant que journaliste est devenu une technique courante pour étudier les rythmes circadiens chez les mammifères 14,15, car elle permet l'examen de la persistance et de la dynamique moléculaire des rythmes. Pour surveiller des cellules autonomes rythmes circadiens de l'expression des gènes, les journalistes luciférase peut être introduit dans les cellules par transfection transitoire 13,16,17 ou transduction stable 5,10,18,19. Nous décrivons ici un protocole de transduction stable à l'aide de gènes médié par lentivirus. Te système de vecteur lentiviral est supérieur aux méthodes traditionnelles telles que la transfection transitoire et la transmission germinale en raison de son efficacité et la polyvalence: il permet l'exécution efficace et une intégration stable dans le génome de l'hôte à la fois la division et non les cellules en division 20. Une fois une lignée cellulaire journaliste est établie, la dynamique de la fonction d'horloge peut être examiné à travers l'enregistrement de bioluminescence. Nous décrivons d'abord la génération de P (Per2)-d lignes journaliste Luc, puis présentent les données de cette étude et d'autres journalistes circadiens. Dans ces essais, les fibroblastes de souris 3T3 et U2OS cellules d'ostéosarcome humains sont utilisés comme modèles cellulaires. Nous discutons également de multiples façons d'utiliser ces modèles dans les études d'horloge circadienne. Les méthodes décrites ici peuvent être appliqués à une grande variété de types de cellules pour étudier les bases cellulaires et moléculaires des horloges circadiennes, et peut s'avérer utile dans la résolution des problèmes dans d'autres systèmes biologiques.
Protocole
1. Construction de Lentiviral Reporters luciférase
Une construction reporter mammifère circadien contient généralement une cassette d'expression dans lequel un promoteur circadien est fusionné avec le gène de la luciférase. Les deux stratégies de ligature et la recombinaison basée sont couramment utilisés pour le clonage d'ADN. A titre d'exemple, nous décrivons ici une méthode basée sur la passerelle de recombinaison pour produire un clonage P (Per2)-Luc rapporteur d lentiviral, dans lequel la luciférase déstabilisé (d Luc) est sous le contrôle du promoteur de la souris Per2.
- Clonage de Per2 promoteur. Utiliser la PCR pour amplifier le fragment d'ADN de promoteur Per2 526 pb en amont du site d'initiation de la transcription à partir d'un clone BAC 9-13 Per2 souris, en utilisant une amorce sens (5'-CTCGAGCGGATTACCGAGGCTGGTCACG TC-3 ') et une amorce anti-sens (5' -CTCGAGTCCCTTGCTCGGCCCGTCAC TTGG-3 '), et clone dans pENTR5'-TOPO vecteur (Invitrogen) pour générerpENTR5'-P (PER2).
- Clonage de Luc d. Le Luc d contient le gène de la luciférase de luciole et une séquence PEST C-terminal de la dégradation rapide des protéines comme décrit précédemment 21. Utilisez PCR pour amplifier le fragment d'ADN d Luc, et clone dans pENTR / D-TOPO vecteur (Invitrogen) pour générer pENTR / Dd Luc.
- Construction du vecteur rapporteur. Mélanger les deux plasmides pENTR, pENTR5'-P (Per2) et pENTR / Dd Luc, avec le vecteur lentiviral destination pLV7-Bsd (Bsd, gène de résistance blasticidine), et d'effectuer la réaction de recombinaison utilisant clonase pour générer un pLV7-Bsd-P (Per2)-d journaliste Luc (Figure 1). pLV7-BSD est une version modifiée (faite dans notre laboratoire) de pLenti6/R4R2/V5-DEST (Invitrogen) dans lequel le virus hépatite de la marmotte post-régulation de la transcription des éléments (WPRE) séquences 22 ont été insérés immédiatement en aval de l'expression ca ssette pour améliorer l'expression des gènes.
2. Production de Particules Lentivirales
1. Cellules 293T semences (jour 1)
- Cultivez rein embryonnaires humaines (HEK) 293T jusqu'à la confluence 90-100% pour l'appui régulier DMEM supplémenté avec 10% de FBS et 1x pénicilline-streptomycine-Glutamine (PSG) sur 10 cm boîtes de culture. (Rapidement les cellules en croissance avec un nombre faible passage sont essentiels pour la transfection efficace.)
- Avant de semer les cellules pour les plaques de transfection, manteau culture à 6 puits en ajoutant 1 ml de 0,001% de poly-L-lysine dans du PBS dans chaque puits et incuber à température ambiante pendant 20 min. Aspirer la solution et rincer une fois avec du PBS 1x avant l'utilisation.
- Dissocier les cellules 293T avec de la trypsine et de graines 0,75 x 10 6 cellules sur chaque puits des plaques pré-enduites avec 2 ml de DMEM régulière. Les plaques de fond de turbulence afin d'obtenir une répartition uniforme des cellules dans chaque puits. Croître les cellules dans l'incubateur à 37 ° C pendant la nuit.
- Respecter les cellules ensemencées du jour 1. Cellule doit atteindre la confluence de 80-90%.
- Préparer le mélange de transfection plasmidique dans un tube de 1,5 ml par addition de 2 ug d'un ADN plasmidique lentiviral journaliste (par exemple, pLV7-P (Per2)-d Luc, Liu laboratoire) et les 3 vecteurs d'encapsidation (1,3 mg Gag / Pol, 0,5 pg VSVG mg Rev, et 0,7; Invitrogen). En tant que commande à la fois pour la transfection et infection subséquente, on comprend généralement un puits supplémentaire dans la transfection d'un vecteur d'expression de la GFP lentiviral, pLV156-CMV-EGFP (figure 1A), hébergeant la protéine fluorescente verte améliorée (EGFP) sous le contrôle du promoteur CMV 20 comme décrit précédemment.
- Ajouter 100 ul de 0,25 M CaCl 2 (dilué avec de la DNase / RNase-free ddH 2 O de 2,5 stock M) au mélange plasmide à l'étape 2 et bien mélanger. Puis ajouter 100 ul de 2x BBS solution (50 mM BES, 280 mM NaCl, 1,5 mM Na 2 HPO 4, pH 6,95) et mélanger doucement mais à fond. Incuber le mélange d'ADN à température ambiante pendant 15 min.
- En attendant, aspirer moyen de cellules 293T et passer à 2 ml de milieu frais. Retourner la plaque à l'étuve pendant au moins 10 min pour équilibrer le pH du milieu avant la transfection.
- Ajouter le mélange de transfection de l'étape 3 à 293T goutte à goutte cellules. Swirl doucement la plaque et d'observer la formation de particules sous un microscope. Incuber à 5% de CO 2, 37 ° C pendant la nuit. (Formation de particules fines de Capo 4 précipité / ADN est essentiel pour la transfection efficace.)
3. Particules virales récolte (jours 3-4)
- Environ 16 heures après la transfection (jour 3) par lequel les cellules de temps devrait atteindre 100% de confluence, aspirer moyen de cellules et la remplacer par 2 ml de DMEM frais réguliers. Incuber à 37 ° C pendant la nuit.
- Au jour 4, d'évaluer l'efficacité de transfection en observant expression de l'EGFP dans les trancellules témoins (sfection efficacité de la transfection de 90-100% avec haute expression de l'EGFP est un prédicteur fiable d'une bonne préparation virale).
- Recueillir le milieu contenant sécrétées, des particules virales infectieuses. Centrifuger à> 2,000 xg pendant 5 minutes pour éliminer les résidus des cellules 293T et recueillir le surnageant contenant le virus. Alternativement, le support peut être effacé par un filtre de 0,45 um membrane. Les particules virales sont prêts à être utilisés dans l'infection.
3. Infection de cellules 3T3
1. Cellules 3T3 semences (jour 3)
Split et le nombre de graines appropriée (~ 12.000) de cellules 3T3 sur une plaque à 12 puits pour obtenir la confluence de 20-30% par jour suivant. Incuber à 37 ° C pendant la nuit.
2. Infecter les cellules 3T3 (jour 4)
- Respecter les cellules ensemencées. Confluence de 20-30% (moins de 50%) est nécessaire pour l'infection.
- Ajouter polybrène à une concentration finale de 5 pg / ml dans le milieu recueilli contenant viraleparticules. Bien mélanger par pipetage.
- Aspirer moyen de cellules 3T3, et ajouter 1 ml du mélange ci-dessus virale par puits. Incuber à 37 ° C pendant la nuit. (Polybrene est utilisé pour améliorer l'efficacité de l'infection, mais n'est pas absolument nécessaire. Comme il peut être toxique pour certaines cellules, le test préalable est recommandé.)
3. Sélectionnez les cellules infectées (jour 5 et au-delà)
- Vingt-quatre heures après l'infection, aspirer un milieu contenant des virus et des cellules infectées polybrène, laver une fois avec du PBS 1x, et changer de milieu frais. Incuber à 37 ° C pendant la nuit pour la reprise et la croissance.
- Lorsque confluente (habituellement 1-2 jours plus tard), diviser les cellules et incuber à 37 ° C pendant la nuit.
- Le lendemain, moyen aspiration des cellules (confluent <50% est souhaitée) et le remplacer par du milieu frais contenant 10 pg / ml blasticidine pour sélectionner des cellules transduites de manière stable. (Blasticidine tuer la courbe doit être déterminée empiriquement pour une lignée cellulaire donnée.)
- Changer de milieu frais contenant blasticidine tous les 2-3 jours pour la sélection continue d'résistantes aux antibiotiques des cellules exprimant journalistes d'horloge (en général 4-6 jours au total).
4. Enregistrement bioluminescence de cellules Reporter
1. Cellules reporters de semences
Propager les cellules reporters blasticidine résistant et fendu sur des boîtes de culture de 35 mm. Incuber à 37 ° C jusqu'à confluence. Nous avons l'habitude de préparer ≥ 3 plats pour chaque lignée cellulaire rapporteur sous chaque condition pour le phénotypage circadien.
2. Synchronisation et de changement de support d'enregistrement
- Aspirer moyen de cellules confluentes journaliste, laver une fois avec du PBS, et le remplacer par du DMEM contenant 10 uM (ou 200 nM de dexaméthasone) forskoline. Incuber à 37 ° C pendant 1 heure pour synchroniser les cellules. (Alternativement, les cellules peuvent être synchronisés par 23 cycles de température ou des chocs sérum 24.)
- En attendant, préparez dossiertion moyen pour les cellules 3T3 comme suit: 1x DMEM (HyClone) contenant 10% de FBS, 1x Pen / Strep / Gln, 1 uM de forskoline, 1 mM de luciférine, HEPES 25 mM, pH 7,4. Sérum et la concentration de forskoline peut être déterminée empiriquement. Pour les cellules très sombres, le rouge de phénol milieu exempt peuvent être utilisés.
- À la fin du traitement forskoline, aspirer à moyen et à le remplacer par support d'enregistrement fraîchement préparé.
3. D'enregistrement bioluminescence de cellules reporters
- Suite à changement de milieu, couvrir des boîtes de culture de 40 mm lamelles stériles et le joint en place avec de la graisse à vide pour éviter l'évaporation.
- Charger la vaisselle sur le luminomètre LumiCycle, qui est maintenu à l'intérieur d'un incubateur réglé à 36 ° C sans H 2 O ou CO 2.
- Démarrez l'enregistrement en temps réel bioluminescence. Nous avons l'habitude d'enregistrer des rythmes pendant 1 semaine, puis changement de milieu et d'enregistrement en continu pour une deuxième semaine (voir Savelyev et al. Pour plus de détails) 25. (Pour l'enregistrement de 96-nousll plaques, Synergy SL2 a été utilisé comme dispositif d'enregistrement, voir Discussion 1.1 pour les détails).
5. Analyse des données et présentation
Cellules Reporter faciliter enregistrement haute résolution de luminescence quantitative, critique pour la détermination des effets phénotypiques sur la fonction horloge circadienne. Pour obtenir les paramètres circadiens, y compris la phase, durée de la période, l'amplitude du rythme, et le taux d'amortissement, nous utilisons le programme d'analyse LumiCycle (Actimetrics) pour analyser les données de bioluminescence 5,14. En bref, les données brutes sont équipés de base d'abord, et de lignes de base de données soustraites sont équipés d'une onde sinusoïdale, dont les paramètres sont déterminés. Pour les échantillons qui montrent des rythmes persistants, qualité de l'ajustement de> 90% est habituellement réalisé. En raison de la bioluminescence transitoire élevée lors de changement de milieu, nous avons l'habitude d'exclure le premier cycle de données de l'analyse.
Pour la présentation des données, nous avons l'habitude de représenter des données brutes (bioluminescence, coups / sec) contre time (jours). Lorsque cela est nécessaire, les données de base-soustraites peuvent être tracées pour comparer l'amplitude et la phase.
6. Les résultats représentatifs
1. Phase spécifiques journalistes circadiens
L'horloge circadienne est basé sur un mécanisme de rétroaction négative biochimique 1. La boucle de rétroaction de base se compose d'activateurs transcriptionnels BMAL1 et CLOCK, et répresseurs pers et cristallisation, qui agissent sur les éléments circadiens enhancer E / E'-box pour produire l'expression du gène rythmique (matin avec la phase, par exemple, Rev-erb α). Le noyau de boucle régule et intègre au moins deux autres éléments cis circadiens, l'élément de liaison DBP/E4BP4 (D-box; jour pour la phase, par exemple, PER3) et l'élément de ROR / Rev-erb liaison (RRE; pour la phase nocturne, par exemple, BMAL1) 17. Réglementation combinatoire par plusieurs éléments circadien peuvent générer de nouveaux phases intermédiaires. Par exemple, Cry1 transcriptiontion est médiée par les trois éléments circadiens (c.-à-éléments, E / E 'et D-box-box dans le promoteur et RREs dans le premier intron du gène Cry1), donnant lieu à l'distincte Cry1 soir-temps de phase 13.
Sur la base de ces mécanismes de régulation des gènes, nous avons généré des quatre constructions rapporteurs différents: P (Per2)-d-Luc et P (Cry1)-D journalistes Luc contenant à la fois E / E'-boîte et D-box éléments dans la région de régulation 17, 26,27; P (Cry1) - Intron-Luc représentant d combinatoire réglementation par les trois éléments (c.-à-E / E'-boîte, D-box, et RRE) 13,17, et P (BMAL1)-Luc d réglementé exclusivement par RRE 9,17,19,21. Nous avons introduit ces journalistes dans les cellules 3T3 de produire des phases distinctes d'expression prévus journaliste (figure 2).
2. Inactivation génique par interférence ARN et pharmacologiquement active composés
Lorsque l'efficacité de transfection est élevé, siARN synthétiques peuvent être transfectées de façon transitoire dans les cellules d'abattre l'expression des gènes. Lorsque la transfection est techniquement difficile, un vecteur d'expression peut être shRNA manière stable dans les cellules transduites par infection lentivirale, de sorte que shRNA produite par la cellule est traitée de siRNA pour effet de choc gène (KD). Nous présentons ici les effets de KD Cry1 et Cry2 gènes en utilisant des siRNA dans des cellules U2OS (figure 3A) et shRNA dans des cellules 3T3 en utilisant un vecteur d'expression pLL3.7 passerelle 9 (figure 3B). En plus des cellules 3T3, le modèle U2OS est devenu un autre modèle d'horloge prééminent cellulaire en grande partie parce qu'il répond aux exigences essentielles de criblage à haut débit disponibles dans le commerce siRNA à l'homme (par exemple, l'origine de l'homme, capable de générer des rythmes circadiens robustes, la fonction de validation tous les gènes de l'horloge sont connus, et qui se prêtent à une transfection très efficace etd'enregistrement luminescence quantitative). Dans les deux types cellulaires, l'ARNi médiée KD entraîné phénotypes d'horloge en conformité avec la souris knock-out précédente (KO) et cellulaires études KD 5,10,11,28. Par exemple, Cry1 KD durée de la période se raccourcit et réduit la persistance rythme, tandis que Cry2 KD allonge la période. En outre, certaines petites molécules peuvent être utilisées pour pharmacologiquement cible et la fonction des protéines perturbent (figure 3C).
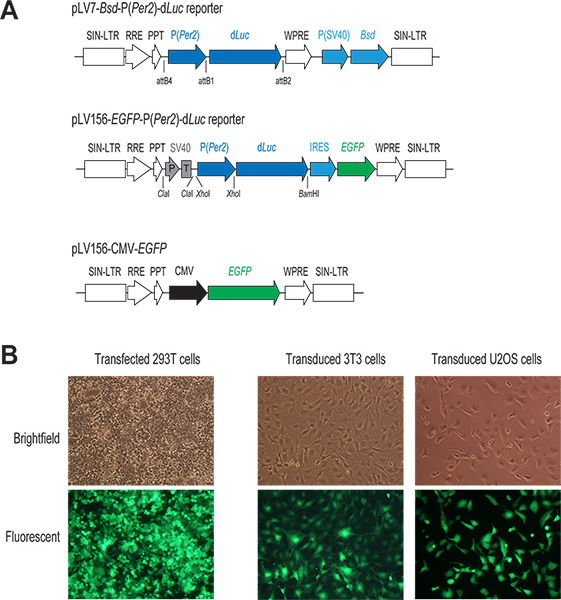
Figure 1. Le système de lentivirus gène à médiation livraison. (A) Représentation schématique de deux lentiviral P (Per2)-d vecteurs rapporteurs Luc et un CMV-EGFP construire. Seule la région à l'intégration dans le génome de la cellule hôte est représenté. Dans les deux constructions rapporteurs, la transcription de Luc d est sous le contrôle direct du promoteur Per2. Dans le pLV7-Bsd-P (Per2)-vecteur d Luc (recombinaison basée sur le clonage), un gène de résistance coexprimé blasticidine (BSD) facilite la sélection des cellules infectées. Dans le pLV156-P (Per2)-Luc vecteur d (ligature à base de clonage), traduction EGFP est médiée par un site d'entrée interne du ribosome (IRES) en aval de Luc d, permettant l'observation visuelle et tri par FACS des cellules infectées. En outre, un promoteur SV40 / terminaison (P / T) est utilisé comme isolant (voir la discussion 1.3). Dans le vecteur CMV-EGFP contrôle, expression EGFP est sous le contrôle d'un promoteur fort CMV. (B) des images fluorescentes de la GFP-exprimant les cellules transfectées et infectées. En règle générale, nous obtenons une grande efficacité à la fois dans la transfection transitoire de cellules 293T et l'infection lentiviral de lignées cellulaires de notre intérêt, comme indiqué par l'expression de GFP dans ces cellules. Cliquez ici pour agrandir la figure .

Figure 2. Phase expression spécifique des journalistes bioluminescence dans les cellules 3T3 Les vecteurs rapporteurs lentiviraux utilisés dans cette expérience sont pLV7-Bsd-P (Per2)-d-Luc, P (Cry1)-d-Luc, P (Cry1) -. Intron-d-Luc, et P (BMAL1)-d-Luc. Chaque rapporteur présente une phase distincte de l'oscillation, comme indiqué par les flèches. Alors que le Per2 et Cry1 bioluminescence disque promoteurs pic à jour phases matin et le promoteur BMAL1 à la phase nuit, la régulation par la combinatoire P (Cry1) - Intron héberger E-box, D-box, et les éléments RRE confère phase de soirée de la bioluminescence de pointe . Cliquez ici pour agrandir la figure .
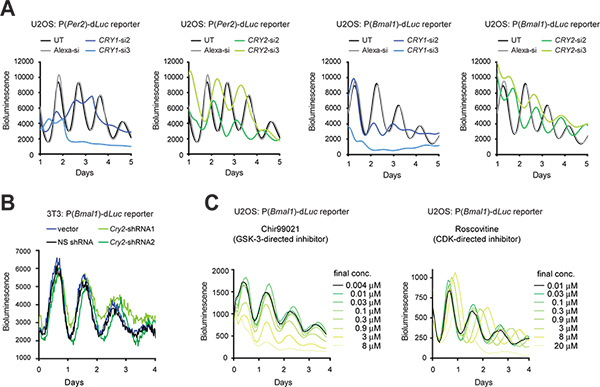
Figure 3. Perturbation génétique et pharmacologique des rythmes circadiens bioluminescence dans les cellules rapporteurs. (A) Les effets de Cry1 et Cry2 knockdown par des siRNA sur les rythmes cellulaires de cellules reporters U2OS. Le luminomètre LumiCycle a été utilisé pour l'enregistrement bioluminescence de cellules dans des boîtes de 35 mm. Figure adaptée de référence n ° 10, avec la permission d'Elsevier (2009). (B) Effets de Cry2 knockdown par shRNA sur les rythmes cellulaires de cellules 3T3 journaliste. Un vecteur pLL3.7 passerelle contenant une cassette U6-shRNA a été utilisé pour Cry2 inactivation génique. shRNA2 a une meilleure efficacité que knockdown shRNA1 tel que déterminé par analyse par Western blot (données non présentées). Un luminomètre Synergy a été utilisé pour l'enregistrement bioluminescence de cellules dans une plaque de 96 puits. Les réglages d'enregistrement sont les suivantes: température de l'incubateur., 33 ° C, le temps d'intégration, 15 sec; intervalle de temps, 30 min (C) Effets des inhibiteurs de petites molécules sur celrythmes lume de cellules reporters U2OS. Chir99021 et roscovitine sont des inhibiteurs dirigés contre GSK-3 et CDK, respectivement. Le système ViewLux (Chir99021 dosage) et un luminomètre Tecan (roscovitine dosage) ont été utilisés pour les enregistrements de bioluminescence de cellules en plaques de 384 puits. Figure adaptée de la référence # 19 (Copyright 2008 National Academy of Sciences, États-Unis). Cliquez ici pour agrandir la figure .
Discussion
1. Modifications au Protocole actuel
1,1 appareils d'enregistrement et de considérations débit
En raison de sa disponibilité commerciale, la LumiCycle (Actimetrics) est devenu l'appareil le plus couramment utilisé luminomètre automatisé pour enregistrement en temps réel 4,5,9,19,29-31. Le LumiCycle emploie des tubes photomultiplicateurs (PMT) en tant que détecteurs de lumière, qui offrent une extrême sensibilité et faible bruit 14,
Déclarations de divulgation
Aucun conflit d'intérêt déclaré.
Remerciements
Ce travail a été financé en partie par la National Science Foundation (IOS-0920417) (ACL).
matériels
| Nom du réactif | Entreprise | Numéro de catalogue | Commentaires |
| DMEM | HyClone | SH30243FS | Pour la croissance cellulaire régulier |
| DMEM | Invitrogen | 12100-046 | Pour luminométrie |
| FBS | HyClone | SH3091003 | |
| Pen / Strep / Gln (100x) | HyClone | SV3008201 | |
| B-27 | Invitrogen | 17504-044 | |
| D-luciférine | Biosynth | L-8220 | |
| La poly-L-lysine | Sigma | P4707 | |
| Polybrène | Millipore | TR-1003-G | |
| La forskoline | Sigma | F6886 | |
| Tous les autres produits chimiques | Sigma | ||
| Équipement | |||
| Incubateur de culture tissulaire | 5% de CO2 à 37 ° C | ||
| Hotte de culture de tissus | BSL-2 certifié | ||
| Lumière fluorescente et microscope | Contraste de phase optionnelle | ||
| LumiCycle | Actimetrics |
Références
- Reppert, S. M., Weaver, D. R. Coordination of circadian timing in mammals. Nature. 418, 935-941 (2002).
- Hastings, M. H., Reddy, A. B., Maywood, E. S. A clockwork web: circadian timing in brain and periphery, in health and disease. Nat. Rev. Neurosci. 4, 649-661 (2003).
- Nagoshi, E. Circadian gene expression in individual fibroblasts: cell-autonomous and self-sustained oscillators pass time to daughter cells. Cell. 119, 693-705 (2004).
- Welsh, D. K. Bioluminescence imaging of individual fibroblasts reveals persistent, independently phased circadian rhythms of clock gene expression. Curr. Biol. 14, 2289-2295 (2004).
- Liu, A. C. Intercellular coupling confers robustness against mutations in the SCN circadian clock network. Cell. 129, 605-616 (2007).
- Kornmann, B. System-driven and oscillator-dependent circadian transcription in mice with a conditionally active liver clock. PLoS Biol. 5, e34 (2007).
- Hogenesch, J. B., Herzog, E. D. Intracellular and intercellular processes determine robustness of the circadian clock. FEBS Lett. 585, 1427-1434 (2011).
- DeBruyne, J. P., Weaver, D. R., Reppert, S. M. Peripheral circadian oscillators require CLOCK. Curr. Biol. 17, 538-539 (2007).
- Liu, A. C. Redundant function of REV-ERBalpha and beta and non-essential role for Bmal1 cycling in transcriptional regulation of intracellular circadian rhythms. PLoS Genet. 4, e1000023 (2008).
- Zhang, E. E. A genome-wide RNAi screen for modifiers of the circadian clock in human cells. Cell. 139, 199-210 (2009).
- Baggs, J. E. Network features of the mammalian circadian clock. PLoS Biol. 7, e52 (2009).
- Hirota, T. High-throughput chemical screen identifies a novel potent modulator of cellular circadian rhythms and reveals CKIalpha as a clock regulatory kinase. PLoS Biol. 8, e1000559 (2010).
- Ukai-Tadenuma, M. Delay in feedback repression by cryptochrome 1 is required for circadian clock function. Cell. 144, 268-281 (2011).
- Yamazaki, S., Takahashi, J. S. Real-time luminescence reporting of circadian gene expression in mammals. Methods Enzymol. 393, 288-301 (2005).
- Welsh, D. K., Imaizumi, T., Kay, S. A. Real-time reporting of circadian-regulated gene expression by luciferase imaging in plants and mammalian cells. Methods Enzymol. 393, 269-288 (2005).
- Sato, T. K. Feedback repression is required for mammalian circadian clock function. Nat. Genet. 38, 312-319 (2006).
- Ueda, H. R. System-level identification of transcriptional circuits underlying mammalian circadian clocks. Nat. Genet. 37, 187-192 (2005).
- Brown, S. A. The period length of fibroblast circadian gene expression varies widely among human individuals. PLoS Biol. 3, e338 (2005).
- Hirota, T. A chemical biology approach reveals period shortening of the mammalian circadian clock by specific inhibition of GSK-3beta. Proc. Natl. Acad. Sci. U.S.A. 105, 20746-20751 (2008).
- Tiscornia, G., Singer, O., Verma, I. M. Production and purification of lentiviral vectors. Nat. Protoc. 1, 241-245 (2006).
- Ueda, H. R. A transcription factor response element for gene expression during circadian night. Nature. 418, 534-539 (2002).
- Zufferey, R., Donello, J. E., Trono, D., Hope, T. J. Woodchuck hepatitis virus posttranscriptional regulatory element enhances expression of transgenes delivered by retroviral vectors. J. Virol. 73, 2886-2892 (1999).
- Buhr, E. D., Yoo, S. H., Takahashi, J. S. Temperature as a universal resetting cue for mammalian circadian oscillators. Science. 330, 379-385 (2010).
- Balsalobre, A., Damiola, F., Schibler, . U.A serum shock induces circadian gene expression in mammalian tissue culture cells. Cell. 93, 929-937 (1998).
- Savelyev, S. A., Larsson, K. C., Johansson, A., Lundkvist, G. B. S. Slice Preparation, Organotypic Tissue Culturing and Luciferase Recording of Clock Gene Activity in the Suprachiasmatic Nucleus. J. Vis. Exp. (48), e2439 (2011).
- Akashi, M., Ichise, T., Mamine, T., Takumi, T. Molecular mechanism of cell-autonomous circadian gene expression of Period2, a crucial regulator of the mammalian circadian clock. Mol. Biol. Cell. 17, 555-565 (2006).
- Ohno, T., Onishi, Y., Ishida, N. A novel E4BP4 element drives circadian expression of mPeriod2. Nucleic Acids Res. 35, 648-655 (2007).
- Maier, B. A large-scale functional RNAi screen reveals a role for CK2 in the mammalian circadian clock. Genes Dev. 23, 708-718 (2009).
- Yoo, S. H. PERIOD2::LUCIFERASE real-time reporting of circadian dynamics reveals persistent circadian oscillations in mouse peripheral tissues. Proc. Natl. Acad. Sci. U.S.A. 101, 5339-5346 (2004).
- Liu, A. C., Lewis, W. G., Kay, S. A. Mammalian circadian signaling networks and therapeutic targets. Nat. Chem. Biol. 3, 630-639 (2007).
- Ko, C. H. Emergence of noise-induced oscillations in the central circadian pacemaker. PLoS Biol. 8, e1000513 (2010).
- Izumo, M., Johnson, C. H., Yamazaki, S. Circadian gene expression in mammalian fibroblasts revealed by real-time luminescence reporting: temperature compensation and damping. Proc. Natl. Acad. Sci. U.S.A. 100, 16089-16094 (2003).
- Izumo, M., Sato, T. R., Straume, M., Johnson, C. H. Quantitative analyses of circadian gene expression in mammalian cell cultures. PLoS Comput. Biol. 2, e136 (2006).
- Chen, Z. Identification of diverse modulators of central and peripheral circadian clocks by high-throughput chemical screening. Proc. Natl. Acad. Sci. U.S.A. 109, 101-106 (2011).
- Yamaguchi, S. Synchronization of cellular clocks in the suprachiasmatic nucleus. Science. 302, 1408-1412 (2003).
- Akashi, M., Hayasaka, N., Yamazaki, S., Node, K. Mitogen-activated protein kinase is a functional component of the autonomous circadian system in the suprachiasmatic nucleus. J. Neurosci. 28, 4619-4623 (2008).
- Hoshino, H., Nakajima, Y., Ohmiya, Y. Luciferase-YFP fusion tag with enhanced emission for single-cell luminescence imaging. Nat. Methods. 4, 637-639 (2007).
- Asai, M. Visualization of mPer1 transcription in vitro: NMDA induces a rapid phase shift of mPer1 gene in cultured SCN. Curr. Biol. 11, 1524-1527 (2001).
- Wilsbacher, L. D. Photic and circadian expression of luciferase in mPeriod1-luc transgenic mice in vivo. Proc. Natl. Acad. Sci. U.S.A. 99, 489-494 (2002).
- Yamazaki, S. Resetting central and peripheral circadian oscillators in transgenic rats. Science. 288, 682-685 (2000).
- Welsh, D. K., Noguchi, T., Yuste, R. Cellular bioluminescence imaging. Imaging: A Laboratory Manual. , 369-385 (2011).
- Nakajima, Y. Enhanced beetle luciferase for high-resolution bioluminescence imaging. PLoS One. 5, e10011 (2010).
- Guilding, C. A riot of rhythms: neuronal and glial circadian oscillators in the mediobasal hypothalamus. Mol. Brain. 2, 28 (2009).
- O'Neill, J. S. cAMP-dependent signaling as a core component of the mammalian circadian pacemaker. Science. 320, 949-953 (2008).
- Fuller, P. M., Lu, J., Saper, C. B. Differential rescue of light- and food-entrainable circadian rhythms. Science. 320, 1074-1077 (2008).
- Mukherjee, S. Knockdown of Clock in the ventral tegmental area through RNA interference results in a mixed state of mania and depression-like behavior. Biol. Psychiatry. 68, 503-511 (2010).
- Saijo, K. A Nurr1/CoREST pathway in microglia and astrocytes protects dopaminergic neurons from inflammation-induced death. Cell. 137, 47-59 (2009).
- Elias, G. M. Synapse-specific and developmentally regulated targeting of AMPA receptors by a family of MAGUK scaffolding proteins. Neuron. 52, 307-320 (2006).
- Isojima, Y. CKIepsilon/delta-dependent phosphorylation is a temperature-insensitive, period-determining process in the mammalian circadian clock. Proc. Natl. Acad. Sci. U.S.A. 106, 15744-15749 (2009).
- Bucan, M., Abel, T. The mouse: genetics meets behaviour. Nat. Rev. Genet. 3, 114-123 (2002).
- Hughes, M. E. Harmonics of circadian gene transcription in mammals. PLoS Genet. 5, e1000442 (2009).
- Atwood, A. Cell-autonomous circadian clock of hepatocytes drives rhythms in transcription and polyamine synthesis. Proc. Natl. Acad. Sci. U.S.A. 108, 18560-18565 (2011).
- Panda, S. Coordinated transcription of key pathways in the mouse by the circadian clock. Cell. 109, 307-320 (2002).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon