Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Monitoreo de células autónomas Ritmos reloj circadiano de expresión génica utilizando luciferase reporteros Bioluminiscencia
En este artículo
Resumen
Los relojes circadianos funcionar dentro de las células individuales, es decir, son células autónomas. A continuación, se describen los métodos para la generación de células autónomas modelos de reloj de forma no invasiva, luciferasa tiempo real basado en la tecnología de bioluminiscencia. Células Reporter proporcionar sistemas manejables, modelo funcional para el estudio de la biología circadiana.
Resumen
En los mamíferos, muchos aspectos del comportamiento y la fisiología como los ciclos de sueño-vigilia y el metabolismo del hígado son regulados por relojes circadianos endógenos (revisado 1,2). El tiempo circadiano de mantenimiento de sistema es una estructura jerárquica multi-oscilador red, con el reloj central situado en el núcleo supraquiasmático (SCN) sincronizar y coordinar los relojes extra-SCN y periférico en otra parte 1,2. Las células individuales son las unidades funcionales para la generación y el mantenimiento de los ritmos circadianos 3,4, y estos osciladores de diferentes tipos de tejidos en el organismo una proporción notablemente similar mecanismo bioquímico de retroalimentación negativa. Sin embargo, debido a las interacciones a nivel de red neuronal en el SNC y por medio de señales rítmicas, sistémicas a nivel de organismos, de los ritmos circadianos en el nivel organismal no necesariamente son células autónomas de 5-7. En comparación con los estudios tradicionales de actividad locomotora in vivo y ex vivo explantes SCN, Cell-basado en los ensayos in vitro permiten el descubrimiento de células autónomas defectos circadianos 5,8. Estratégicamente, modelos basados en células son más experimentalmente tratable para la caracterización fenotípica y rápido descubrimiento de los mecanismos de reloj básicas 5,8-13.
Debido a que los ritmos circadianos son dinámicas, las mediciones longitudinales con alta resolución temporal son necesarios para evaluar la función del reloj. En los últimos años, grabación en tiempo real utilizando bioluminiscencia luciferasa de luciérnaga como reportero se ha convertido en una técnica común para el estudio de los ritmos circadianos en mamíferos 14,15, ya que permite el examen de la persistencia y la dinámica de los ritmos moleculares. Para supervisar células autónomas ritmos circadianos de la expresión génica, luciferase reporteros se pueden introducir en las células a través de la transfección transitoria 13,16,17 o transducción estable 5,10,18,19. Aquí se describe un protocolo de transducción estable utilizando lentivirus mediada por la entrega de genes. Tsistema de vector lentiviral que es superior a los métodos tradicionales tales como la transfección transitoria y transmisión de línea germinal debido a su eficiencia y versatilidad: permite una entrega eficaz y la integración estable en el genoma huésped de tanto que se dividen y no se dividen las células 20. Una vez que una línea celular reportera se ha establecido, la dinámica de la función de reloj se pueden examinar a través de grabación de bioluminiscencia. En primer lugar, describimos la generación de P (Per2)-D líneas Luc reportero, y luego se presentan datos de este y otros reporteros circadianos. En estos ensayos, los fibroblastos 3T3 de ratón y células de osteosarcoma humano U2OS se utilizan como modelos celulares. También se discuten las diversas formas de utilizar estos modelos en estudios reloj circadiano. Los métodos descritos aquí se pueden aplicar a una gran variedad de tipos de células para estudiar la base molecular y celular de los relojes circadianos, y puede resultar útil en la lucha contra los problemas en otros sistemas biológicos.
Protocolo
1. Construcción de Lentiviral luciferase reporteros
Un constructo reportero circadiano de mamíferos por lo general contiene un casete de expresión en la que se fusiona un promotor circadiano con el gen de la luciferasa. Tanto las estrategias de ligación y basadas en la recombinación son comúnmente utilizados para la clonación del ADN. Como ejemplo, aquí se describe un método de Gateway recombinación basada en la clonación para generar una P (Per2)-d reportero Luc lentiviral, en la que la luciferasa desestabilizado (d Luc) está bajo el control del promotor de ratón Per2.
- Clonación del promotor Per2. El uso de PCR para amplificar el fragmento de promotor de ADN Per2 de 526 pb, aguas arriba del sitio de inicio de la transcripción de un ratón Per2 clon BAC 9-13, usando un cebador directo (5'-CTCGAGCGGATTACCGAGGCTGGTCACG TC-3 ') y un cebador inverso (5' -CTCGAGTCCCTTGCTCGGCCCGTCAC TTGG-3 '), y el clon en pENTR5'-TOPO vector (Invitrogen) para generarpENTR5'-P (Per2).
- Clonación de d Luc. La Luc d contiene el gen de la luciferasa de luciérnaga y una secuencia PEST C-terminal de la degradación de proteínas rápida como se ha descrito previamente 21. El uso de PCR para amplificar el fragmento de ADN d Luc, y el clon en pENTR / D-TOPO vector (Invitrogen) para generar pENTR / Dd Luc.
- Construcción del vector reportero. Mezclar los dos plásmidos pENTR, P pENTR5'-(Per2) y Luc pENTR / DD, con el vector lentiviral destino pLV7-BSD (BSD, blasticidin gen de resistencia), y llevar a cabo la reacción de recombinación utilizando Clonase para generar un pLV7-BSD-P (Per2)-d luc (Figura 1). pLV7-BSD es una versión modificada (realizado en nuestro laboratorio) de pLenti6/R4R2/V5-DEST (Invitrogen) en el que el virus de la hepatitis de la marmota post-transcripcional elemento regulador (WPRE) 22 secuencias se insertaron inmediatamente aguas abajo de la expresión ca ssette para potenciar la expresión génica.
2. Producción de partículas lentivirales
1. Semillas células 293T (día 1)
- Grow embrionarias humanas de riñón (HEK) células 293T a 90-100% de confluencia en ordinario DMEM suplementado con FBS al 10% y 1x de penicilina-estreptomicina-glutamina (PSG) en placas de 10 cm de cultivo. (Rápidamente las células que crecen con el paso número bajo son críticos para la transfección eficiente.)
- Antes de la siembra de las células de las placas de transfección, capa de cultivo de 6 pocillos mediante la adición de 1 ml de 0,001% de poli-L-lisina en PBS a cada pocillo e incubar a temperatura ambiente durante 20 min. Aspirar la solución y se lava una vez con 1x PBS antes de su uso.
- Disociar las células 293T con tripsina y semilla 0,75 x 10 6 células en cada pocillo de las placas pre-recubiertas con 2 ml de DMEM regular. Swirl las placas a fondo para obtener una distribución uniforme de células en cada pocillo. Cultivar las células en la incubadora a 37 ° C durante la noche.
- Observar las células sembradas a partir del día 1. La célula debe alcanzar confluencia de 80-90%.
- Preparar la mezcla de transfección del plásmido en un tubo de microcentrífuga de 1,5 ml mediante la adición de 2 g de un ADN plásmido reportero lentiviral (por ejemplo, pLV7-P (Per2)-d Luc; Liu laboratorio) y los vectores de empaquetamiento 3 (1,3 g Gag / Pol, 0,5 g Rev, y 0,7 g VSVG; Invitrogen). Como control de transfección tanto y la posterior infección, que por lo general incluyen un pozo adicional en la transfección de un vector de expresión GFP lentiviral, pLV156-CMV-EGFP (Figura 1A), que alberga aumento de proteína verde fluorescente (EGFP) bajo el control del promotor CMV como se ha descrito previamente 20.
- Añadir 100 l de 0,25 M de CaCl 2 (diluido con DNasa / RNasa libre ddH 2 O de 2,5 M valores) a la mezcla de plásmido en el paso 2 y mezclar bien. A continuación, añadir 100 ml de solución 2 x BBS (50 mM SERS, 280 mM NaCl, 1,5 mM Na 2 HPO 4, pH 6,95) y mezclar suavemente pero a fondo. Incubar la mezcla de ADN a temperatura ambiente durante 15 min.
- Mientras espera, aspirar medio de células 293T y cambiar a 2 ml de medio fresco. Volver a la placa de la incubadora durante al menos 10 min para equilibrar el pH del medio antes de la transfección.
- Añadir la mezcla de transfección de la etapa 3 a gota células 293T a gota. Agitar la placa suavemente y observar la formación de partículas con un microscopio. Incubar a 5% de CO 2, 37 ° C durante la noche. (Formación de partículas finas de Capo 4 precipitado / ADN es esencial para la transfección eficiente.)
3. Cosecha de partículas virales (días 3-4)
- Acerca de 16 horas después de la transfección-(día 3) por el cual las células de tiempo debe alcanzar el 100% de confluencia, aspirar el medio de las células y reemplazar con 2 ml de DMEM fresco ordinario. Incubar a 37 ° C durante la noche.
- El día 4, evaluar la eficacia de transfección mediante la observación de la expresión de EGFP en transfection células control (eficacia de la transfección de 90-100%, con expresión de EGFP alta es un predictor fiable de una preparación viral bueno.)
- Se recoge el medio que contiene secretadas, las partículas virales infecciosas. Centrifugar a> 2.000 xg durante 5 minutos para eliminar las células residuales de 293T y recoger el sobrenadante de virus que contiene. Alternativamente, el medio puede ser limpiado con un filtro de 0,45 micras membrana. Las partículas virales son listos para su uso en la infección.
3. Infección de células 3T3
1. Semillas 3T3 células (día 3)
Split y número adecuados de las semillas (~ 12.000) de las células 3T3 en una placa de 12-bien para obtener 20-30% de confluencia por día siguiente. Incubar a 37 ° C durante la noche.
2. Infectar las células 3T3 (día 4)
- Observar las células sembradas. Confluencia de 20-30% (menos del 50%) es deseable para la infección.
- Añadir polibreno a una concentración final de 5 ug / ml al medio de recogida que contiene viralpartículas. Mezclar bien pipeteando.
- Aspirar medio a partir de células 3T3, y añadir 1 ml de la mezcla anterior viral por pocillo. Incubar a 37 ° C durante la noche. (Polybrene se utiliza para mejorar la eficiencia de la infección, pero no se requiere absolutamente. Como puede ser tóxica para algunas células, de un ensayo previo se recomienda.)
3. Seleccione células infectadas (día 5 en adelante)
- Veinticuatro horas después de la infección, aspirar el medio que contiene virus y polibreno de las células infectadas, se lava una vez con PBS 1x, y cambiar a un medio fresco. Incubar a 37 ° C durante la noche para la recuperación y el crecimiento.
- Cuando confluente (normalmente 1-2 días más tarde), los trozos de las células y se incuba a 37 ° C durante la noche.
- El día siguiente, el medio aspirado de células (confluencia <50% se desea) y reemplazar con medio fresco que contiene 10 mg / ml blasticidina para seleccionar para las células transducidas de forma estable. (Blasticidina matar-curva necesita ser determinado empíricamente para una línea celular particular.)
- Cambiar a un nuevo soporte de blasticidina cada 2-3 días para la selección continua de antibióticos resistentes a las células que expresan los periodistas reloj (generalmente 4-6 días en total).
4. La bioluminiscencia Grabación de células Reporter
1. Se siembran las células indicadoras
Propagar blasticidina células resistentes a reportero y dividido en placas de cultivo de 35 mm. Incubar a 37 ° C hasta una confluencia. Por lo general, preparar ≥ 3 platos para cada línea celular reportero en cada condición para fenotipificación circadiano.
2. Sincronización y cambio de medio de grabación
- Aspirar medio de células confluentes reportero, lavar una vez con PBS, y reemplazar con DMEM que contenía 10 mM de forskolina (o 200 nM dexametasona). Incubar a 37 ° C durante 1 hora para sincronizar las células. (Alternativamente, las células pueden ser sincronizados por 23 ciclos de temperatura o choque de suero 24.)
- Mientras espera, preparar registroING medio para células 3T3 como sigue: 1x DMEM (HyClone) que contenía 10% de FBS, 1 x Pen / Strep / Gln, 1 mM de forskolina, 1 mM de luciferina, 25 mM HEPES, pH 7,4. El suero y la concentración de forscolina se puede determinar empíricamente. Para las células muy tenues, libre de rojo fenol medio puede ser utilizado.
- Al final del tratamiento forskolina, aspirar el medio y sustituirlo por medio de grabación recién hecho.
3. Bioluminiscencia de grabación de células informadoras
- Después de cambiar el medio, cubra placas de cultivo de 40 mm cubreobjetos estériles y sellar en el lugar con grasa de vacío para evitar la evaporación.
- Coloque la vajilla en el luminómetro LumiCycle, que se mantiene dentro de una incubadora a 36 ° C sin H 2 O y CO 2.
- Iniciar grabación en tiempo real bioluminiscencia. Por lo general, registrar los ritmos durante 1 semana, seguido de cambio de medio y de grabación continua de una semana segundos (ver Savelyev et al. Para más detalles) 25. (Para la grabación de 96-queplacas ll, Synergy SL2 fue utilizado como dispositivo de grabación, ver Discusión 1.1 para detalles).
5. Análisis de datos y presentación
Reportero facilitar células de alta resolución de grabación luminiscencia cuantitativa, fundamental para determinar los efectos fenotípicos sobre la función del reloj circadiano. Para obtener los parámetros circadianos incluyendo fase, la duración del período, amplitud, ritmo y grado de amortiguación, se utiliza el programa de análisis de LumiCycle (Actimetrics) para analizar los datos de bioluminiscencia 5,14. Brevemente, los datos en bruto son línea de base, montado de primera, y la sustracción de línea de base de datos se ajustan a una onda sinusoidal, de la que los parámetros son determinados. Para ver ejemplos que muestran ritmos persistentes, de bondad de ajuste de> 90% se consigue normalmente. Debido a la bioluminiscencia alta transitoria a cambio de medio, por lo general no incluyen el primer ciclo de datos de análisis.
Para la presentación de datos, por lo general graficar datos en bruto (bioluminiscencia, cuentas / seg) en contra de time (días). Cuando sea necesario, los datos de línea de base-resta se pueden trazar para comparar la amplitud y fase.
6. Los resultados representativos
1. Fase específica periodistas circadianos
El reloj circadiano se basa en un mecanismo de retroalimentación negativa bioquímica 1. El bucle de realimentación núcleo consiste de activadores de la transcripción y BMAL1 reloj, y represores pers y Crys, que actúan sobre los circadianos E / E'-box elementos potenciadores para producir la expresión de genes rítmica (con fase mañana, por ejemplo, Rev-erb α). El lazo de núcleo regula e integra al menos otros dos circadianos cis-elementos, el elemento de unión DBP/E4BP4 (D-box; para la fase de día, por ejemplo, PER3) y el elemento de ROR / REV-ERB unión (RRE; para la fase noche, por ejemplo, Bmal1) 17. Regulación combinatoria de múltiples elementos circadianos puede generar nuevas fases intermedias. Por ejemplo, Cry1 transcripciónes mediada por los tres elementos (elementos circadianos es decir, E / E'de caja y caja D-en el promotor y RRES en el primer intrón del gen Cry1), dando lugar a la distinta Cry1 noche-tiempo de fase 13.
En base a estos mecanismos de regulación génica, hemos generado cuatro construcciones indicadoras diferentes: P (Per2)-d Luc y P (Cry1)-D Luc reporteros que contienen tanto E / E'de caja y D-box elementos en la región de regulación 17, 26,27, P (Cry1) - Intron-d Luc representa regulación combinatoria de los tres elementos (es decir, E / E'-box, D-box, y RRE) 13,17, y P (Bmal1)-d Luc regulado exclusivamente por RRE 9,17,19,21. Hemos introducido estos reporteros en células 3T3 para producir las fases previstas distintas de expresión reportero (Figura 2).
2. Gene caída a través de RNAi y farmacológicamente actiVe compuestos
Cuando la eficiencia de transfección es alta, ARNsi sintético puede ser transfectados transitoriamente en las células para derribar la expresión génica. Cuando la transfección es técnicamente difícil, un vector de expresión de shRNA puede ser de forma estable en células transducidas mediante infección lentiviral, de modo que shRNA producida por la celda se procesa para siRNA para el gen desmontables (KD). Aquí se presentan los efectos de Kd de Cry1 y Cry2 genes utilizando siRNA en células U2OS (Figura 3A) y shRNA en células 3T3 usando un vector de expresión de puerta de enlace pLL3.7 9 (Figura 3B). Además de las células 3T3, el modelo U2OS ha convertido en un modelo preeminente reloj celular en gran parte porque cumple con los requisitos fundamentales para la selección de alto rendimiento de las bibliotecas siRNA humanos disponibles en el mercado (por ejemplo, el origen humano, capaz de generar fuertes ritmos circadianos, la función de validación todos los genes conocidos despertador y susceptibles de transfección altamente eficiente ygrabación luminiscencia cuantitativo). En ambos tipos de células, RNAi mediada KD dado lugar a fenotipos de reloj consistentes con ratón knockout anterior (KO) y estudios celulares KD 5,10,11,28. Por ejemplo, Cry1 KD duración del período se acorta y disminuye la persistencia ritmo, mientras que Cry2 KD alarga período. Además, pequeñas moléculas seleccionadas se pueden utilizar para farmacológicamente objetivo y función perturbar proteína (Figura 3C).
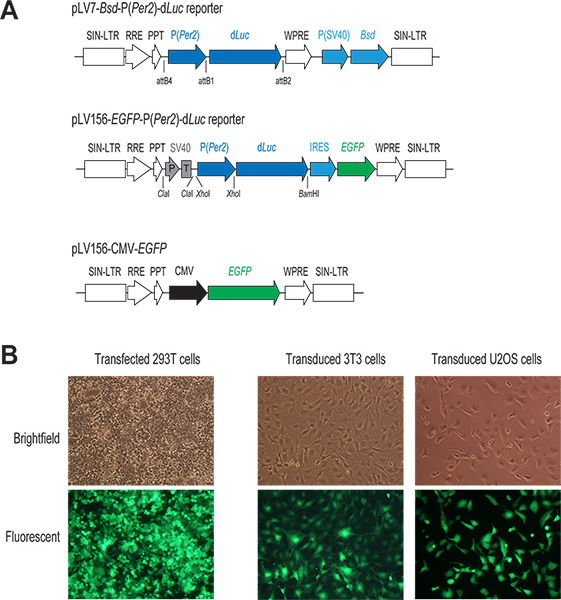
Figura 1. El lentivirus de genes mediada por sistema de suministro. (A) Diagrama esquemático de dos lentiviral P (Per2)-D vectores indicadores Luc y una EGFP-CMV construir. Sólo la región para la integración en el genoma de la célula huésped se muestra. En ambas construcciones indicadoras, la transcripción de d Luc está bajo el control directo del promotor Per2. En el pLV7-BSD-P (Per2)-d vector Luc (recombinación basada en la clonación), un gen de blasticidina coexpressed resistencia (BSD) facilita la selección de las células infectadas. En la pLV156-P (Per2)-d Luc vector (ligadura de un clonado), traducción EGFP está mediada por un sitio interno de entrada al ribosoma (IRES) aguas abajo de d Luc, lo que permite la observación visual y clasificación de FACS de las células infectadas. Además, un SV40 promotor / terminador (P / T) se utiliza como un aislador (véase la discusión 1,3). En el vector CMV-EGFP control, es la expresión de EGFP bajo el control de un fuerte promotor de CMV. (B) imágenes fluorescentes de GFP-expresando transfectadas y las células infectadas. Típicamente, se logra una alta eficiencia tanto en la transfección transitoria de células 293T y en la infección lentiviral de líneas celulares de nuestro interés, como se indica por la expresión de GFP en estas células. Haga clic aquí para ampliar la figura .

Figura 2. Fase expresión específica de los reporteros de bioluminiscencia en células 3T3 Los vectores indicadores de lentivirus utilizados en este experimento son pLV7-BSD-P (Per2)-Luc d, P (Cry1)-Luc d, P (Cry1) -. Intrón-d Luc, y P (Bmal1)-d Luc. Cada reportero exhibe una fase distinta de la oscilación, como se indica por las flechas. Mientras que el Per2 y Cry1 bioluminiscencia unidad promotores pico en fases día por la mañana y el promotor de Bmal1 en fase nocturna, la regulación combinatoria por el P (Cry1) - Intron albergar caja E, D-box, y los elementos RRE confiere fase noche de bioluminiscencia pico . Haga clic aquí para ampliar la cifra .
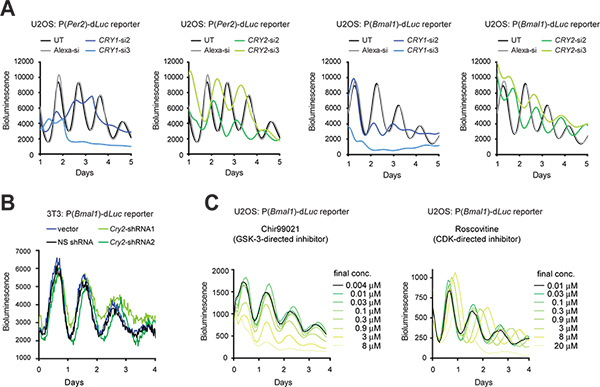
Figura 3. Perturbación genética y farmacológica de los ritmos circadianos de bioluminiscencia en las células indicadoras. (A) Efectos de Cry1 y Cry2 desmontables por siRNAs sobre los ritmos celulares de las células indicadoras U2OS. El luminómetro LumiCycle fue utilizado para la grabación de la bioluminiscencia de las células en placas de 35 mm. La figura está adaptada de la referencia n º 10, con permiso de Elsevier (2009). (B) Efectos de la caída por Cry2 shRNAs sobre los ritmos celulares de células indicadoras 3T3. Un vector de puerta de enlace que contiene un casete de pLL3.7 U6-shRNA se utilizó para gen desmontables Cry2. shRNA2 tiene una eficacia mejor que shRNA1 desmontables como se determina por análisis de transferencia Western (datos no mostrados). Un luminómetro Synergy fue utilizado para la grabación de la bioluminiscencia de las células en una placa de 96 pocillos. Los ajustes para la grabación son las siguientes: temperatura de la incubadora., 33 ° C, el tiempo de integración, 15 s; intervalo de tiempo, 30 min (C) Efectos de inhibidores de moléculas pequeñas en celritmos lular de células U2OS reportero. Chir99021 y roscovitina son inhibidores dirigidos contra GSK-3 y CDK, respectivamente. El sistema Viewlux (Chir99021 ensayo) y un luminómetro Tecan (roscovitina ensayo) se utilizaron para las grabaciones de bioluminiscencia de las células en 384-y placas. La figura es una adaptación de Referencia # 19 (Copyright 2008 National Academy of Sciences, EE.UU.). Haga clic aquí para ampliar la cifra .
Discusión
1. Modificaciones al Protocolo actual
1.1 Los dispositivos de grabación y consideraciones de rendimiento
Debido a su disponibilidad comercial, la LumiCycle (Actimetrics) se ha convertido en el dispositivo más utilizado luminómetro automatizado para grabación en tiempo real 4,5,9,19,29-31. El LumiCycle emplea tubos fotomultiplicadores (PMT) como detectores de luz, que proporcionan una sensibilidad extremadamente alta y bajo ruido 14, y por lo ...
Divulgaciones
No hay conflictos de interés declarado.
Agradecimientos
Este trabajo fue apoyado en parte por la Fundación Nacional de Ciencia (IOS-0920417) (ACL).
Materiales
| Nombre de reactivo | Empresa | Número de catálogo | Comentarios |
| DMEM | HyClone | SH30243FS | Para el crecimiento celular normal |
| DMEM | Invitrogen | 12100-046 | Para luminometría |
| FBS | HyClone | SH3091003 | |
| Pen / Strep / Gln (100x) | HyClone | SV3008201 | |
| B-27 | Invitrogen | 17504-044 | |
| D-luciferina | Biosynth | L-8220 | |
| Poli-L-lisina | Sigma | P4707 | |
| Polybrene | Millipore | TR-1003-G | |
| Forskolin | Sigma | F6886 | |
| Todos los otros productos químicos | Sigma | ||
| Equipo | |||
| Incubadora de cultivo de tejido | 5% de CO2 a 37 ° C | ||
| Tejido capucha cultura | BSL-2 certificado | ||
| Light & microscopio fluorescente | Fase opcional contraste | ||
| LumiCycle | Actimetrics |
Referencias
- Reppert, S. M., Weaver, D. R. Coordination of circadian timing in mammals. Nature. 418, 935-941 (2002).
- Hastings, M. H., Reddy, A. B., Maywood, E. S. A clockwork web: circadian timing in brain and periphery, in health and disease. Nat. Rev. Neurosci. 4, 649-661 (2003).
- Nagoshi, E. Circadian gene expression in individual fibroblasts: cell-autonomous and self-sustained oscillators pass time to daughter cells. Cell. 119, 693-705 (2004).
- Welsh, D. K. Bioluminescence imaging of individual fibroblasts reveals persistent, independently phased circadian rhythms of clock gene expression. Curr. Biol. 14, 2289-2295 (2004).
- Liu, A. C. Intercellular coupling confers robustness against mutations in the SCN circadian clock network. Cell. 129, 605-616 (2007).
- Kornmann, B. System-driven and oscillator-dependent circadian transcription in mice with a conditionally active liver clock. PLoS Biol. 5, e34 (2007).
- Hogenesch, J. B., Herzog, E. D. Intracellular and intercellular processes determine robustness of the circadian clock. FEBS Lett. 585, 1427-1434 (2011).
- DeBruyne, J. P., Weaver, D. R., Reppert, S. M. Peripheral circadian oscillators require CLOCK. Curr. Biol. 17, 538-539 (2007).
- Liu, A. C. Redundant function of REV-ERBalpha and beta and non-essential role for Bmal1 cycling in transcriptional regulation of intracellular circadian rhythms. PLoS Genet. 4, e1000023 (2008).
- Zhang, E. E. A genome-wide RNAi screen for modifiers of the circadian clock in human cells. Cell. 139, 199-210 (2009).
- Baggs, J. E. Network features of the mammalian circadian clock. PLoS Biol. 7, e52 (2009).
- Hirota, T. High-throughput chemical screen identifies a novel potent modulator of cellular circadian rhythms and reveals CKIalpha as a clock regulatory kinase. PLoS Biol. 8, e1000559 (2010).
- Ukai-Tadenuma, M. Delay in feedback repression by cryptochrome 1 is required for circadian clock function. Cell. 144, 268-281 (2011).
- Yamazaki, S., Takahashi, J. S. Real-time luminescence reporting of circadian gene expression in mammals. Methods Enzymol. 393, 288-301 (2005).
- Welsh, D. K., Imaizumi, T., Kay, S. A. Real-time reporting of circadian-regulated gene expression by luciferase imaging in plants and mammalian cells. Methods Enzymol. 393, 269-288 (2005).
- Sato, T. K. Feedback repression is required for mammalian circadian clock function. Nat. Genet. 38, 312-319 (2006).
- Ueda, H. R. System-level identification of transcriptional circuits underlying mammalian circadian clocks. Nat. Genet. 37, 187-192 (2005).
- Brown, S. A. The period length of fibroblast circadian gene expression varies widely among human individuals. PLoS Biol. 3, e338 (2005).
- Hirota, T. A chemical biology approach reveals period shortening of the mammalian circadian clock by specific inhibition of GSK-3beta. Proc. Natl. Acad. Sci. U.S.A. 105, 20746-20751 (2008).
- Tiscornia, G., Singer, O., Verma, I. M. Production and purification of lentiviral vectors. Nat. Protoc. 1, 241-245 (2006).
- Ueda, H. R. A transcription factor response element for gene expression during circadian night. Nature. 418, 534-539 (2002).
- Zufferey, R., Donello, J. E., Trono, D., Hope, T. J. Woodchuck hepatitis virus posttranscriptional regulatory element enhances expression of transgenes delivered by retroviral vectors. J. Virol. 73, 2886-2892 (1999).
- Buhr, E. D., Yoo, S. H., Takahashi, J. S. Temperature as a universal resetting cue for mammalian circadian oscillators. Science. 330, 379-385 (2010).
- Balsalobre, A., Damiola, F., Schibler, . U.A serum shock induces circadian gene expression in mammalian tissue culture cells. Cell. 93, 929-937 (1998).
- Savelyev, S. A., Larsson, K. C., Johansson, A., Lundkvist, G. B. S. Slice Preparation, Organotypic Tissue Culturing and Luciferase Recording of Clock Gene Activity in the Suprachiasmatic Nucleus. J. Vis. Exp. (48), e2439 (2011).
- Akashi, M., Ichise, T., Mamine, T., Takumi, T. Molecular mechanism of cell-autonomous circadian gene expression of Period2, a crucial regulator of the mammalian circadian clock. Mol. Biol. Cell. 17, 555-565 (2006).
- Ohno, T., Onishi, Y., Ishida, N. A novel E4BP4 element drives circadian expression of mPeriod2. Nucleic Acids Res. 35, 648-655 (2007).
- Maier, B. A large-scale functional RNAi screen reveals a role for CK2 in the mammalian circadian clock. Genes Dev. 23, 708-718 (2009).
- Yoo, S. H. PERIOD2::LUCIFERASE real-time reporting of circadian dynamics reveals persistent circadian oscillations in mouse peripheral tissues. Proc. Natl. Acad. Sci. U.S.A. 101, 5339-5346 (2004).
- Liu, A. C., Lewis, W. G., Kay, S. A. Mammalian circadian signaling networks and therapeutic targets. Nat. Chem. Biol. 3, 630-639 (2007).
- Ko, C. H. Emergence of noise-induced oscillations in the central circadian pacemaker. PLoS Biol. 8, e1000513 (2010).
- Izumo, M., Johnson, C. H., Yamazaki, S. Circadian gene expression in mammalian fibroblasts revealed by real-time luminescence reporting: temperature compensation and damping. Proc. Natl. Acad. Sci. U.S.A. 100, 16089-16094 (2003).
- Izumo, M., Sato, T. R., Straume, M., Johnson, C. H. Quantitative analyses of circadian gene expression in mammalian cell cultures. PLoS Comput. Biol. 2, e136 (2006).
- Chen, Z. Identification of diverse modulators of central and peripheral circadian clocks by high-throughput chemical screening. Proc. Natl. Acad. Sci. U.S.A. 109, 101-106 (2011).
- Yamaguchi, S. Synchronization of cellular clocks in the suprachiasmatic nucleus. Science. 302, 1408-1412 (2003).
- Akashi, M., Hayasaka, N., Yamazaki, S., Node, K. Mitogen-activated protein kinase is a functional component of the autonomous circadian system in the suprachiasmatic nucleus. J. Neurosci. 28, 4619-4623 (2008).
- Hoshino, H., Nakajima, Y., Ohmiya, Y. Luciferase-YFP fusion tag with enhanced emission for single-cell luminescence imaging. Nat. Methods. 4, 637-639 (2007).
- Asai, M. Visualization of mPer1 transcription in vitro: NMDA induces a rapid phase shift of mPer1 gene in cultured SCN. Curr. Biol. 11, 1524-1527 (2001).
- Wilsbacher, L. D. Photic and circadian expression of luciferase in mPeriod1-luc transgenic mice in vivo. Proc. Natl. Acad. Sci. U.S.A. 99, 489-494 (2002).
- Yamazaki, S. Resetting central and peripheral circadian oscillators in transgenic rats. Science. 288, 682-685 (2000).
- Welsh, D. K., Noguchi, T., Yuste, R. Cellular bioluminescence imaging. Imaging: A Laboratory Manual. , 369-385 (2011).
- Nakajima, Y. Enhanced beetle luciferase for high-resolution bioluminescence imaging. PLoS One. 5, e10011 (2010).
- Guilding, C. A riot of rhythms: neuronal and glial circadian oscillators in the mediobasal hypothalamus. Mol. Brain. 2, 28 (2009).
- O'Neill, J. S. cAMP-dependent signaling as a core component of the mammalian circadian pacemaker. Science. 320, 949-953 (2008).
- Fuller, P. M., Lu, J., Saper, C. B. Differential rescue of light- and food-entrainable circadian rhythms. Science. 320, 1074-1077 (2008).
- Mukherjee, S. Knockdown of Clock in the ventral tegmental area through RNA interference results in a mixed state of mania and depression-like behavior. Biol. Psychiatry. 68, 503-511 (2010).
- Saijo, K. A Nurr1/CoREST pathway in microglia and astrocytes protects dopaminergic neurons from inflammation-induced death. Cell. 137, 47-59 (2009).
- Elias, G. M. Synapse-specific and developmentally regulated targeting of AMPA receptors by a family of MAGUK scaffolding proteins. Neuron. 52, 307-320 (2006).
- Isojima, Y. CKIepsilon/delta-dependent phosphorylation is a temperature-insensitive, period-determining process in the mammalian circadian clock. Proc. Natl. Acad. Sci. U.S.A. 106, 15744-15749 (2009).
- Bucan, M., Abel, T. The mouse: genetics meets behaviour. Nat. Rev. Genet. 3, 114-123 (2002).
- Hughes, M. E. Harmonics of circadian gene transcription in mammals. PLoS Genet. 5, e1000442 (2009).
- Atwood, A. Cell-autonomous circadian clock of hepatocytes drives rhythms in transcription and polyamine synthesis. Proc. Natl. Acad. Sci. U.S.A. 108, 18560-18565 (2011).
- Panda, S. Coordinated transcription of key pathways in the mouse by the circadian clock. Cell. 109, 307-320 (2002).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados