A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
ניטור מקצבי Cell-אוטונומי יממת שעון של ביטוי גנים באמצעות כתבי פליטת האור לוציפראז
In This Article
Summary
שעוני יממה לתפקד בתוך תאים בודדים, כלומר, הם תאים אוטונומיים. כאן, אנו מתארים שיטות ליצירת מודלי שעון תא אוטונומי באמצעות טכנולוגיה לא פולשנית, לוציפראז מבוסס זמן אמת פליטת אור. תאי Reporter מספקים מערכות צייתניות, פונקציונליות מודל ללימוד ביולוגית יממה.
Abstract
ביונקים, היבטים רבים של התנהגות ופיזיולוגיה כגון מחזורי שינה והערות וחילוף חומרים כבדים מוסדרים בשעוני יממת אנדוגני (סקר 1,2). המערכת הביולוגית זמן שמירה, היא רשת היררכית רב מתנד, עם השעון המרכזי ממוקם בגרעין suprachiasmatic (SCN) סנכרון ותיאום שעונים-SCN נוסף והיקפיים אחר 1,2. תאים בודדים הם יחידות פונקציונליות לדור ותחזוקה של מקצבי יממה 3,4 ומתנדים אלה של סוגי רקמות שונים בנתח אורגניזם מנגנון משוב דומה להפליא ביוכימי שלילי. עם זאת, בשל אינטראקציות ברמת הרשת העצבית בSCN ובאמצעות רמזים קצביים, מערכתיים ברמת האורגניזם, מקצבי יממה ברמת האורגניזם אינם בהכרח תא אוטונומי 5-7. בהשוואה למחקרים מסורתיים של פעילות של תנועה בגוף חי וexplants SCN vivo לשעבר, גell מבוסס במבחני חוץ גופייה מאפשרת גילוי פגמי תא אוטונומי יממת 5,8. מבחינה אסטרטגית, מודלים מבוססי תאים יותר בניסוי צייתנים לאפיון פנוטיפי וגילוי מהיר של מנגנוני שעון בסיסיים 5,8-13.
בגלל מקצבי יממה הם דינמיים, מדידות אורכות עם רזולוציה גבוהה זמנית נחוצות להערכת תפקוד שעון. בשנים האחרונות, הקלטת פליטת אור בזמן אמת באמצעות בלוציפראז הגחלילית ככתב הפכה לשיטה נפוצה ללימוד מקצבי יממה ביונקים 14,15, שכן היא מאפשרת לבחינה של ההתמדה והדינמיקה של מקצבים מולקולריים. כדי לפקח על מקצבי יממת תא אוטונומי של ביטוי גנים, כתבי וציפראז ניתן הוכנסו לתאים באמצעות transfection החולף 13,16,17 או תמרה יציבה 5,10,18,19. כאן אנו מתארים פרוטוקול תמרה יציבה באמצעות משלוח הגן lentivirus בתיווך. Tמערכת וקטור lentiviral הוא עדיף על שיטות מסורתיות כגון transfection חולף ושידור germline בגלל היעילות והרבגוניות שלו: הוא מאפשר העברה יעילה ויציבה אינטגרציה לתוך הגנום של המארח הוא חלוקה ולא חלוקת תאים-20. ברגע ששורת תאי כתב הוא הקים, את הדינמיקה של פונקציית שעון יכולה להיבחן דרך הקלטת פליטת אור. אנו מתארים לראשונה את הדור (Per2) קווי P-d לוק כתב, ולאחר מכן מציגים נתונים מזה וכתבי יממה אחרים. במבחנים אלה, fibroblasts העכבר 3T3 וU2OS תאי סרטן עצמות אדם משמשים כמודל לסלולרי. אנחנו גם לדון בדרכים שונות של שימוש במודלים אלה במחקרי שעון יממה. שיטות שתוארו כאן ניתן ליישם במגוון רחב של סוגי תאים ללמוד את הבסיס התאי ומולקולרי של שעוני יממה, ועשויה להיות שימושי בהתמודדות עם בעיות במערכות ביולוגיות אחרות.
Protocol
1. בנייה של עיתונאי לוציפראז lentiviral
מבנה כתב יממת יונקים מכיל בדרך כלל ביטוי בקלטת שאמרגן יממה מעומתת עם גן לוציפראז. אסטרטגיות קשירה ושניהם רקומבינציה מבוססת משמשות בדרך כלל לשיבוט דנ"א. כדוגמה, הנה אנחנו מתארים שיטת שיבוט Gateway מבוססת רקומבינציה ליצירת כתב P (Per2)-ד לוק lentiviral, בי לוציפראז הערער (ד לוק) הוא תחת שליטה של אמרגן Per2 עכבר.
- שיבוט של Per2 אמרגן. השתמש PCR להגביר בר דנ"א אמרגן Per2 של 526 נ"ב, במעלה זרם של אתר תחילת השעתוק מעכבר Per2 BAC שיבוט 9-13, באמצעות פריימר קדימה (5'-CTCGAGCGGATTACCGAGGCTGGTCACG TC-3 ') ופריימר הפוך (5' -CTCGAGTCCCTTGCTCGGCCCGTCAC TTGG-3 "), ושיבוט לpENTR5'-topo וקטור (Invitrogen) לייצרpENTR5'-P (Per2).
- שיבוט של ד לוק. ד לוק מכיל את גן בלוציפראז גחלילית ורצף הדברה C-מסוף לפירוק חלבונים מהיר כפי שתואר קודם לכן 21. השתמש PCR להגביר בר ד לוק-DNA, והשיבוט לpENTR / D-topo וקטור (Invitrogen) כדי ליצור pENTR / DD לוק.
- בנייה של וקטור כתב. מערבב את שני פלסמידים pENTR, pENTR5'-P (Per2) וpENTR / DD לוק, עם וקטור יעד lentiviral pLV7-BSD (BSD, גן ההתנגדות blasticidin), ולבצע את תגובת רקומבינציה באמצעות Clonase לייצר pLV7-BSD-P (Per2)-D כתב לוק (איור 1). pLV7-BSD הוא גרסה שונה (שנעשה במעבדה שלנו) של pLenti6/R4R2/V5-DEST (Invitrogen) שבו וירוס צהבת מרמיטת רצפים לאחר התעתיק רגולטוריים אלמנט (WPRE) 22 הוכנסו מייד במורד זרם של הביטוי ca ssette כדי לשפר ביטוי גנים.
2. ייצור של חלקיקי lentiviral
1. תאי 293T זרעים (יום 1)
- לגדול בכליות (HEK) 293T תאים עובריים אנושיים למפגש 90-100% בDMEM הרגיל בתוספת 10% FBS ו1x פניצילין-סטרפטומיצין-גלוטמין (PSG) על 10 מנות תרבות סנטימטר. (במהירות תאים גדלים עם מספר מעבר נמוך הם קריטיים עבור transfection היעיל.)
- לפני זריעת התאים transfection ל, מעייל צלחות התרבות 6-כן על ידי הוספת 1 מ"ל של 0.001% פולי-L-ליזין בPBS לכל באר ודגירה בטמפרטורת חדר למשך 20 דקות. לשאוב את הפתרון ולשטוף פעם אחת עם 1x PBS לפני השימוש.
- לנתק את תאי 293T עם טריפסין וזרע 0.75 x 10 6 תאים על גבי כל אחד גם מהצלחות מראש המצופות עם 2 המ"ל DMEM הרגיל. מערבולת ביסודיות את הצלחות להשיג חלוקה שווה של תאים בכל אחד גם. לגדל את התאים בחממה ב 37 ° C למשך הלילה.
- הקפד על התאים נזרעו מיום 1. תא צריך להגיע מפגש של 80-90%.
- הכן תערובת transfection פלסמיד בצינור microcentrifuge מ"ל 1.5 ידי הוספת 2 מיקרוגרם של DNA פלסמיד כתב lentiviral (למשל, pLV7-P (Per2)-ד לוק; יו מעבדה) ואת 3 וקטורי האריזה (1.3 איסור פרסום מיקרוגרם / פול, 0.5 מיקרוגרם Rev, ו -0.7 מיקרוגרם VSVG; Invitrogen). כשלט עבור transfection וזיהום שלאחר מכן, אנחנו בדרך כלל כוללים גם תוספת בtransfection לוקטור ביטוי של GFP lentiviral, pLV156-CMV-EGFP (איור 1 א), חסת חלבון פלואורסצנטי ירוק משופר (EGFP) תחת השליטה של אמרגן CMV כפי שתואר לעיל 20.
- הוסף 100 μl של 0.25 מ 'CaCl 2 (מדולל עם DNase / RNase ללא DDH 2 O מהמניות 2.5 מ') לתערובת פלסמיד בשלב 2 ומערבבים היטב. ואז להוסיף 100 μl של פתרון BBS 2x (50 מ"מ BES, 280 המ"מ NaCl, 1.5 מ"מ Na 2 HPO 4, pH 6.95) ומערבב בעדינות אך ביסודיות. דגירה את תערובת ה-DNA בטמפרטורת חדר במשך 15 דקות.
- בזמן המתנה, לשאוב בינוני מתאי 293T ולשנות עד בינוני 2 מ"ל טרי. להחזיר את הצלחת לאינקובטור במשך לפחות 10 דקות כדי לאזן חומציות בינונית לפני transfection.
- הוסף תערובת transfection משלב 3 לירידת 293T תאים אחר טיפה. המערבולת בעדינות את הצלחת ולבחון היווצרות חלקיק תחת מיקרוסקופ. לדגור על 5% CO 2, 37 ° C למשך הלילה. (היווצרות חלקיקים קטנים של קאפו 4 משקע / ה-DNA הוא קריטי עבור transfection היעיל.)
3. חלקיקים נגיפיים קציר (ימים 3-4)
- כ 16 שעות לאחר transfection (יום 3) שבו תאים בזמן צריכים להגיע למפגש 100%, לשאוב בינוני מתאים ולהחליף המ"ל DMEM טרי 2 רגיל. לדגור על 37 מעלות צלזיוס למשך הלילה.
- ביום 4, להעריך את יעילות transfection על ידי התבוננות בביטוי EGFP טראןתאי בקרת sfection (יעילות Transfection של 90-100% עם ביטוי EGFP גבוה היא מנבא אמין של מכין נגיפי טוב.)
- איסוף בינוני המכילים חלקיקים מופרשים, מחלות זיהומיות ויראליות. צנטריפוגה ב> 2000 XG למשך 5 דקות להסרת תאי 293T השיורי ולאסוף את supernatant וירוס המכיל. לחלופין, ניתן הבינוני פונה עם מסנן קרום מיקרומטר 0.45. את חלקיקי הנגיף מוכנים לשימוש בזיהום.
3. זיהום של תאי 3T3
1. תאי 3T3 זרעים (יום 3)
פיצול ומספר מתאים זרע (~ 12,000) של תאי 3T3 בצלחת 12-היטב כדי להשיג מפגש 20-30% ביום שלמחרת. לדגור על 37 מעלות צלזיוס למשך הלילה.
2. להדביק תאי 3T3 (יום 4)
- הקפד על התאים נזרעו. מפגש של 20-30% (פחות מ 50%) הוא רצוי לזיהום.
- הוסף polybrene לריכוז סופי של 5 מיקרוגרם / מ"ל למדיום שנאסף המכיל נגיףחלקיקים. מערבב היטב על ידי pipetting.
- לשאוב בינוני מתאי 3T3, ולהוסיף 1 מ"ל של התערובת הנגיפית מעל לטוב. לדגור על 37 מעלות צלזיוס למשך הלילה. (Polybrene משמש כדי לשפר את יעילות זיהום, אבל אינו מחויב באופן מוחלט. כפי שזה עלול להיות רעיל לתאים מסוימים, בדיקות מוקדמות מומלצות).
3. בחר תאים נגועים (יום 5 והלאה)
- עשרים וארבע שעות לאחר פגיעה, לשאוב בינוני המכיל וירוס וpolybrene מתאים נגועים, לשטוף פעם אחת עם 1x PBS, ולשנות למדיום טרי. לדגור על 37 מעלות צלזיוס למשך הלילה להתאוששות ולצמיחה.
- כאשר מתלכדים (בדרך כלל 1-2 ימים לאחר מכן), לפצל את התאים ולדגור על 37 מעלות צלזיוס למשך הלילה.
- למחרת בינוני, aspirate מתאים (<50% מפגש הוא רצוי) ולהחליף במדיום טרי המכיל 10 מיקרוגרם / המ"ל Blasticidin כדי לבחור ליציבות transduced תאים. (Blasticidin להרוג עקום צריך להיקבע באופן אמפירי לשורת תאים מסוימות.)
- שינוי למדיום טרי המכיל Blasticidin כל 2-3 ימים לבחירה מתמשכת של תאים עמידים לאנטיביוטיקה המבטאים כתבי שעון (בדרך כלל 4-6 ימים סה"כ).
4. הקלטת פליטת אור של תאי Reporter
1. תאי כתב Seed
הפץ תאי כתב Blasticidin עמידים ולפצל על מנות התרבות 35-מ"מ. לדגור על 37 מעלות צלזיוס עד שמתלכד. אנחנו בדרך כלל להכין ≥ 3 מנות עבור כל שורת תאי כתב תחת כל תנאים לphenotyping יממה.
2. סנכרון ושינוי להקלטה בינונית
- לשאוב בינוני מתאי כתב מחוברים, לשטוף פעם עם PBS, ולהחליף עם DMEM המכיל 10 מיקרומטר forskolin (או 200 ננומטר דקסמטזון). לדגור על 37 מעלות צלזיוס במשך שעה 1 כדי לסנכרן את התאים. (לחלופין, תאים יכולים להיות מסונכרנים במחזורי הטמפרטורה 23, או הלם סרום 24.)
- בזמן המתנה, להכין שיאבינוני ing לתאי 3T3 כדלקמן: 1x DMEM (HyClone) המכיל 10% FBS, 1x עט / דלקת / Gln, 1 מיקרומטר forskolin, 1 המ"מ luciferin, 25 HEPES מ"מ, pH 7.4. יכולים להיקבע סרום וריכוז forskolin באופן אמפירי. לתאים עמומים מאוד, בינוני פנול האדום חופשי יכול להיות בשימוש.
- בסוף טיפול forskolin, לשאוב בינוני ולהחליף עם הקלטה בינונית טרי.
3. הקלטת פליטת אור של תאי כתב
- בעקבות שינוי בינוני, לכסות מנות התרבות עם 40 המ"מ coverslips סטריליים וחותם במקום עם גריז ואקום כדי למנוע אידוי.
- טען את המנות על גבי luminometer LumiCycle, שנשמר בתוך קבוצת חממה ב 36 ° C ללא H 2 O או CO 2.
- התחל הקלטת פליטת אור בזמן אמת. אנחנו בדרך כלל להקליט מקצבים ל1 שבוע, ואחרי שינוי מדיום והקלטה רציפה לשבוע שני (ראה סאבלייב et al. לפרטים) 25. (להקלטה של 96-צלחות ll, סינרגיה SL2 שמשה כמכשיר הקלטה; ראו דיון 1.1 עבור פרט).
5. ניתוח והצגת נתונים
תאי Reporter להקל על צריבה ברזולוציה גבוהה כמו הארה, קריטית לקביעת השפעות פנוטיפי על תפקוד שעון יממה. כדי לקבל פרמטרי יממה, כוללים שלב, אורך תקופה, משרעת קצב, וקצב דעיכה, אנו משתמשים בתכנית ניתוח LumiCycle (Actimetrics) לניתוח נתוני פליטת אור 5,14. בקצרה, נתונים גולמיים הם בסיס מצויד ראשונים, ונתונים בסיסיים-נגרעו מצוידים לגל סינוס, שממנו את הפרמטרים שנקבע. לדגימות שמראות מקצבים מתמשכים, טוב, של התאמה של> 90% מושגת בדרך כלל. בשל פליטת אור חולפת גבוהה על שינוי בינוני, אנחנו בדרך כלל לא לכלול את המחזור הראשון של נתונים מניתוח.
להצגת נתונים, אנחנו בדרך כלל להתוות סדרת נתוני גלם (פליטת אור, ספירות / sec) נגד ti(ימים). בעת צורך, נתונים בסיסיים-נגרעו ניתן להתוות להשוות האמפליטודה והפאזה.
6. נציג תוצאות
1. כתבי יממת שלב ספציפיים
שעון היממה המבוסס על מנגנון משוב שלילי ביוכימי 1. לולאת משוב הליבה מורכבת ממפעילי תעתיק BMAL1 ושעון, וrepressors מצו וcrys, הפועלים על אלמנטי E / E'-תיבת היממה משפר לייצר ביטוי גנים קצבי (בשלב הבוקר, למשל, Rev-Erb α). לולאת הליבה מווסתת ומשלבת לפחות שני אלמנטי CIS אחרים יממה, אלמנט כריכת DBP/E4BP4 (D-תיבה; לשלב היום, למשל, Per3) ואלמנט כריכת רע"ש / REV-Erb (RRE; ללילה שלב, למשל, Bmal1) 17. רגולצית קומבינטורית ידי גורמי יממה מרובים יכולה ליצור שלבי ביניים רומן. לדוגמה, Cry1 תמלילבו מתווך על ידי כל השלושה אלמנטי היממה (כלומר, E / E'-תיבה ותיבת D-אלמנטים באמרגן וRREs באינטרון הראשון של Cry1 הגן), והוליד Cry1 השלב המובחן הערב בזמן 13.
בהתבסס על מנגנונים אלה של ויסות גנים, שנוצרנו 4 מבני כתב שונים: P (Per2)-ד לוק ו-P (Cry1)-d כתבי לוק המכילים גם E / E'-תיבה ואלמנטי D-box באזור הרגולציה 17, 26,27, P (Cry1) - אינטרון-d לוק מייצג רגולצית קומבינטורית על ידי כל השלושה האלמנטים (כלומר, E / E'קופסה, D-תיבה, וRRE) 13,17; ו-P (Bmal1)-ד לוק המוסדר באופן בלעדי על ידי RRE 9,17,19,21. אנחנו הצגנו לכתבים אלה לתוך תאי 3T3 כדי לייצר את השלבים הברורים הצפויים של ביטוי עיתונאי (איור 2).
2. מציאת גן באמצעות RNAi ופרמקולוגית active תרכובות
כאשר יעילות transfection היא גבוהה, siRNA הסינטטי ניתן transfected זמנית לתוך תאים כדי להפיל ביטוי גנים. כאשר transfection הוא קשה מבחינה טכנית, וקטור ביטוי shRNA יכול להיות יציבות transduced לתוך תאים באמצעות זיהום lentiviral, כך שshRNA מיוצר על ידי תאי מעובד לsiRNA למציאת גן (KD). כאן אנו מציגים תופעות KD של Cry1 וCry2 גנים באמצעות siRNA בתאי U2OS (האיור 3A) וshRNA בתאי 3T3 באמצעות וקטור pLL3.7 Gateway ביטוי 9 (איור 3 ב '). בנוסף לתאי 3T3, מודל U2OS הפך אחר מודל שעון סלולרי בולט במידה רבה משום שהיא עומדת בדרישות המפתח לתפוקה גבוהה הקרנה של ספריות siRNA אדם זמינות מסחרי (למשל, מקור אנושי, מסוגל לייצר מקצב פעילות חזק, תפקוד תוקף של כל הגנים הידועים שעון ומתאימים לtransfection והיעיל ביותרהקלטת הארה כמוני). בשני סוגי התאים, KD RNAi בתיווך הביא פנוטיפים שעון עקביים עם נוקאאוט הקודם עכבר (KO) ומחקרי KD סלולריים 5,10,11,28. לדוגמה, אורך Cry1 KD מתקצר תקופה ומפחית התמדת קצב, בעוד Cry2 KD מתארך תקופה. בנוסף, מולקולות קטנות שנבחרו יכולות לשמש למטרה פרמקולוגית ותפקוד חלבון מטריד (האיור 3C).
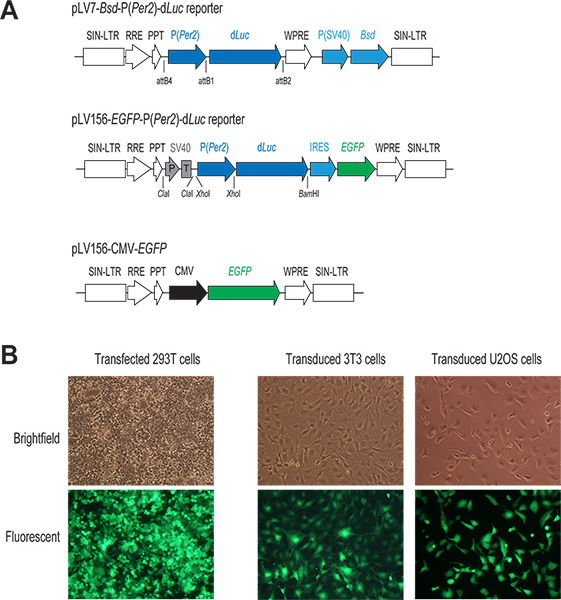
איור 1. מערכת מסירת גן lentivirus בתיווך. () תרשים סכמטי של 2 P lentiviral (Per2)-d וקטורי כתב לוק וCMV-EGFP לבנות. רק באזור להשתלבות הגנום התא המארח מוצג. בשני המבנים של הכתב, התעתיק של ד לוק נמצא תחת שליטה ישירה של אמרגן Per2. בpLV7-BSD-P (Per2)-ד לוק הווקטור (מחדששיבוט שילוב מבוסס), גן התנגדות Blasticidin coexpressed (BSD) מאפשר בחירה של תאים נגועים. בpLV156-P (Per2)-ד לוק וקטור (שיבוט קשירה מבוססת), תרגום EGFP מתווך על ידי אתר פנימי כניסה הריבוזום (ires) במורד זרם של ד לוק, המאפשר התבוננות וFACS מיון של תאים נגועים ויזואלי. בנוסף, SV40 אמרגן / שליחות קטלנית (P / T) משמש כמבודד (ראה דיון 1.3). בוקטור שליטת CMV-EGFP, ביטוי EGFP נמצא תחת שליטה של אמרגן CMV חזק. (B) תמונות של פלורסנט GFP-להביע transfected והנגוע בתאים. בדרך כלל, אנו להשיג יעילות גבוהה בשניהם transfection החולף של תאי 293T ובזיהום lentiviral של שורות תאים של האינטרס שלנו, כפי שצוינו על ידי ביטוי של GFP בתאים אלה. לחצו כאן לצפייה בדמות גדולה.

איור 2. ביטוי שלב ספציפי של כתבי פליטת אור בתאי 3T3 וקטורי כתב lentiviral משמשים בניסוי זה הם pLV7-BSD-P (Per2)-ד לוק, P (Cry1)-ד לוק, P (Cry1) -. אינטרון-d לוק, ו-P (Bmal1)-ד לוק. כל עיתונאי מציג שלב מובהק של תנודה, כפי שצוין על ידי החיצים. בעוד Per2 ופליטת אור Cry1 יזמי כונן שיא בשלבי בוקר יום ואמרגן Bmal1 בלילה השלב, רגולצית קומבינטורית ידי P (Cry1) - אינטרון מחסה תיבת דואר, D-תיבה, ואלמנטי RRE מקנים שלב הערב של פליטת אור שיא . לחץ כאן לצפייה בדמות גדולה.
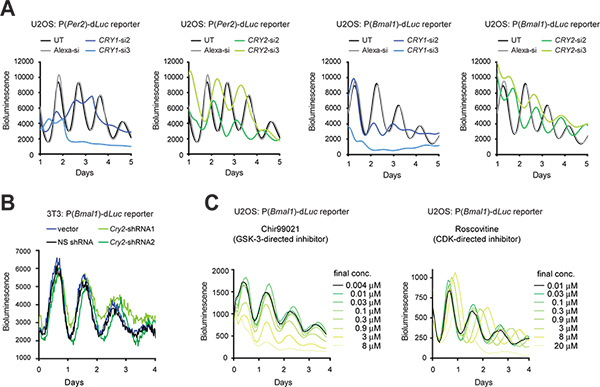
איור 3. הפרעות גנטיות ותרופתיות של מקצבי יממת פליטת אור בתאי כתב. (A) השפעות של Cry1 וCry2 מציאה ידי siRNAs על מקצבים תאיים של תאי כתב U2OS. Luminometer LumiCycle שמש להקלטת פליטת אור של תאים ב35 מנות מ"מ. איור מותאם מעיון # 10, באישור Elsevier (2009). (B) השפעות של Cry2 מציאה ידי shRNAs על מקצבים תאיים של תאי כתב 3T3. וקטור pLL3.7 Gateway המכיל קלטת U6-shRNA שמש למציאת הגן Cry2. shRNA2 יש יעילות טובה יותר מאשר מציאת shRNA1 כפי שנקבע על ידי ניתוח כתם מערבי (מידע לא מוצג). Luminometer Synergy שמש להקלטת פליטת אור של תאים בצלחת 96 היטב. ההגדרות לרישום הן כדלקמן:. טמפרטורת חממה, 33 ° C; זמן אינטגרציה, 15 שניות; זמן מרווח, 30 דקות (C) השפעות של מעכבי מולקולה קטנים על celמקצבי lular של תאי כתב U2OS. Chir99021 וRoscovitine הם מעכבים מכוונים נגד GSK-3 וCDK, בהתאמה. מערכת ViewLux (Chir99021 assay) וluminometer טקאן (Roscovitine assay) שמשו להקלטות פליטת אור של תאים בצלחות 384 כן. איור מותאם Reference # 19 (זכויות יוצרים 2008 האקדמיה הלאומית למדעים, ארה"ב). לחץ כאן לצפייה בדמות גדולה.
Discussion
1. עשיית שינויים בפרוטוקול הנוכחי
1.1 מכשירי הקלטה ושיקולי תפוקה
בגלל הזמינות המסחרית שלה, LumiCycle (Actimetrics) הפך את מכשיר luminometer האוטומטי הנפוץ ביותר להקלטה בזמן אמת 4,5,9,19,29-31. LumiCycle מעסיק צינורות ...
Disclosures
אין ניגודי האינטרסים הכריזו.
Acknowledgements
עבודה זו נתמכה בחלקו על ידי הקרן הלאומי למדע (IOS-0920417) (ACL).
Materials
| שם מגיב | חברה | מספר קטלוגים | תגובות |
| DMEM | HyClone | SH30243FS | לצמיחת תאים רגילות |
| DMEM | Invitrogen | 12100-046 | לluminometry |
| FBS | HyClone | SH3091003 | |
| עט / דלקת / Gln (100x) | HyClone | SV3008201 | |
| B-27 | Invitrogen | 17504-044 | |
| D-luciferin | ביוסינט | L-8220 | |
| פולי-L-ליזין | סיגמא | P4707 | |
| Polybrene | Millipore | TR-1003-G | |
| Forskolin | סיגמא | F6886 | |
| כל כימיקלים אחרים | סיגמא | ||
| ציוד | |||
| רקמת תרבות חממה | 5% CO2 על 37 מעלות צלזיוס | ||
| ברדס תרביות רקמה | מנהלת הליגה מוסמכת-2 | ||
| מיקרוסקופ אור וניאון | לעומת שלב אופציונלי | ||
| LumiCycle | Actimetrics |
References
- Reppert, S. M., Weaver, D. R. Coordination of circadian timing in mammals. Nature. 418, 935-941 (2002).
- Hastings, M. H., Reddy, A. B., Maywood, E. S. A clockwork web: circadian timing in brain and periphery, in health and disease. Nat. Rev. Neurosci. 4, 649-661 (2003).
- Nagoshi, E. Circadian gene expression in individual fibroblasts: cell-autonomous and self-sustained oscillators pass time to daughter cells. Cell. 119, 693-705 (2004).
- Welsh, D. K. Bioluminescence imaging of individual fibroblasts reveals persistent, independently phased circadian rhythms of clock gene expression. Curr. Biol. 14, 2289-2295 (2004).
- Liu, A. C. Intercellular coupling confers robustness against mutations in the SCN circadian clock network. Cell. 129, 605-616 (2007).
- Kornmann, B. System-driven and oscillator-dependent circadian transcription in mice with a conditionally active liver clock. PLoS Biol. 5, e34 (2007).
- Hogenesch, J. B., Herzog, E. D. Intracellular and intercellular processes determine robustness of the circadian clock. FEBS Lett. 585, 1427-1434 (2011).
- DeBruyne, J. P., Weaver, D. R., Reppert, S. M. Peripheral circadian oscillators require CLOCK. Curr. Biol. 17, 538-539 (2007).
- Liu, A. C. Redundant function of REV-ERBalpha and beta and non-essential role for Bmal1 cycling in transcriptional regulation of intracellular circadian rhythms. PLoS Genet. 4, e1000023 (2008).
- Zhang, E. E. A genome-wide RNAi screen for modifiers of the circadian clock in human cells. Cell. 139, 199-210 (2009).
- Baggs, J. E. Network features of the mammalian circadian clock. PLoS Biol. 7, e52 (2009).
- Hirota, T. High-throughput chemical screen identifies a novel potent modulator of cellular circadian rhythms and reveals CKIalpha as a clock regulatory kinase. PLoS Biol. 8, e1000559 (2010).
- Ukai-Tadenuma, M. Delay in feedback repression by cryptochrome 1 is required for circadian clock function. Cell. 144, 268-281 (2011).
- Yamazaki, S., Takahashi, J. S. Real-time luminescence reporting of circadian gene expression in mammals. Methods Enzymol. 393, 288-301 (2005).
- Welsh, D. K., Imaizumi, T., Kay, S. A. Real-time reporting of circadian-regulated gene expression by luciferase imaging in plants and mammalian cells. Methods Enzymol. 393, 269-288 (2005).
- Sato, T. K. Feedback repression is required for mammalian circadian clock function. Nat. Genet. 38, 312-319 (2006).
- Ueda, H. R. System-level identification of transcriptional circuits underlying mammalian circadian clocks. Nat. Genet. 37, 187-192 (2005).
- Brown, S. A. The period length of fibroblast circadian gene expression varies widely among human individuals. PLoS Biol. 3, e338 (2005).
- Hirota, T. A chemical biology approach reveals period shortening of the mammalian circadian clock by specific inhibition of GSK-3beta. Proc. Natl. Acad. Sci. U.S.A. 105, 20746-20751 (2008).
- Tiscornia, G., Singer, O., Verma, I. M. Production and purification of lentiviral vectors. Nat. Protoc. 1, 241-245 (2006).
- Ueda, H. R. A transcription factor response element for gene expression during circadian night. Nature. 418, 534-539 (2002).
- Zufferey, R., Donello, J. E., Trono, D., Hope, T. J. Woodchuck hepatitis virus posttranscriptional regulatory element enhances expression of transgenes delivered by retroviral vectors. J. Virol. 73, 2886-2892 (1999).
- Buhr, E. D., Yoo, S. H., Takahashi, J. S. Temperature as a universal resetting cue for mammalian circadian oscillators. Science. 330, 379-385 (2010).
- Balsalobre, A., Damiola, F., Schibler, . U.A serum shock induces circadian gene expression in mammalian tissue culture cells. Cell. 93, 929-937 (1998).
- Savelyev, S. A., Larsson, K. C., Johansson, A., Lundkvist, G. B. S. Slice Preparation, Organotypic Tissue Culturing and Luciferase Recording of Clock Gene Activity in the Suprachiasmatic Nucleus. J. Vis. Exp. (48), e2439 (2011).
- Akashi, M., Ichise, T., Mamine, T., Takumi, T. Molecular mechanism of cell-autonomous circadian gene expression of Period2, a crucial regulator of the mammalian circadian clock. Mol. Biol. Cell. 17, 555-565 (2006).
- Ohno, T., Onishi, Y., Ishida, N. A novel E4BP4 element drives circadian expression of mPeriod2. Nucleic Acids Res. 35, 648-655 (2007).
- Maier, B. A large-scale functional RNAi screen reveals a role for CK2 in the mammalian circadian clock. Genes Dev. 23, 708-718 (2009).
- Yoo, S. H. PERIOD2::LUCIFERASE real-time reporting of circadian dynamics reveals persistent circadian oscillations in mouse peripheral tissues. Proc. Natl. Acad. Sci. U.S.A. 101, 5339-5346 (2004).
- Liu, A. C., Lewis, W. G., Kay, S. A. Mammalian circadian signaling networks and therapeutic targets. Nat. Chem. Biol. 3, 630-639 (2007).
- Ko, C. H. Emergence of noise-induced oscillations in the central circadian pacemaker. PLoS Biol. 8, e1000513 (2010).
- Izumo, M., Johnson, C. H., Yamazaki, S. Circadian gene expression in mammalian fibroblasts revealed by real-time luminescence reporting: temperature compensation and damping. Proc. Natl. Acad. Sci. U.S.A. 100, 16089-16094 (2003).
- Izumo, M., Sato, T. R., Straume, M., Johnson, C. H. Quantitative analyses of circadian gene expression in mammalian cell cultures. PLoS Comput. Biol. 2, e136 (2006).
- Chen, Z. Identification of diverse modulators of central and peripheral circadian clocks by high-throughput chemical screening. Proc. Natl. Acad. Sci. U.S.A. 109, 101-106 (2011).
- Yamaguchi, S. Synchronization of cellular clocks in the suprachiasmatic nucleus. Science. 302, 1408-1412 (2003).
- Akashi, M., Hayasaka, N., Yamazaki, S., Node, K. Mitogen-activated protein kinase is a functional component of the autonomous circadian system in the suprachiasmatic nucleus. J. Neurosci. 28, 4619-4623 (2008).
- Hoshino, H., Nakajima, Y., Ohmiya, Y. Luciferase-YFP fusion tag with enhanced emission for single-cell luminescence imaging. Nat. Methods. 4, 637-639 (2007).
- Asai, M. Visualization of mPer1 transcription in vitro: NMDA induces a rapid phase shift of mPer1 gene in cultured SCN. Curr. Biol. 11, 1524-1527 (2001).
- Wilsbacher, L. D. Photic and circadian expression of luciferase in mPeriod1-luc transgenic mice in vivo. Proc. Natl. Acad. Sci. U.S.A. 99, 489-494 (2002).
- Yamazaki, S. Resetting central and peripheral circadian oscillators in transgenic rats. Science. 288, 682-685 (2000).
- Welsh, D. K., Noguchi, T., Yuste, R. Cellular bioluminescence imaging. Imaging: A Laboratory Manual. , 369-385 (2011).
- Nakajima, Y. Enhanced beetle luciferase for high-resolution bioluminescence imaging. PLoS One. 5, e10011 (2010).
- Guilding, C. A riot of rhythms: neuronal and glial circadian oscillators in the mediobasal hypothalamus. Mol. Brain. 2, 28 (2009).
- O'Neill, J. S. cAMP-dependent signaling as a core component of the mammalian circadian pacemaker. Science. 320, 949-953 (2008).
- Fuller, P. M., Lu, J., Saper, C. B. Differential rescue of light- and food-entrainable circadian rhythms. Science. 320, 1074-1077 (2008).
- Mukherjee, S. Knockdown of Clock in the ventral tegmental area through RNA interference results in a mixed state of mania and depression-like behavior. Biol. Psychiatry. 68, 503-511 (2010).
- Saijo, K. A Nurr1/CoREST pathway in microglia and astrocytes protects dopaminergic neurons from inflammation-induced death. Cell. 137, 47-59 (2009).
- Elias, G. M. Synapse-specific and developmentally regulated targeting of AMPA receptors by a family of MAGUK scaffolding proteins. Neuron. 52, 307-320 (2006).
- Isojima, Y. CKIepsilon/delta-dependent phosphorylation is a temperature-insensitive, period-determining process in the mammalian circadian clock. Proc. Natl. Acad. Sci. U.S.A. 106, 15744-15749 (2009).
- Bucan, M., Abel, T. The mouse: genetics meets behaviour. Nat. Rev. Genet. 3, 114-123 (2002).
- Hughes, M. E. Harmonics of circadian gene transcription in mammals. PLoS Genet. 5, e1000442 (2009).
- Atwood, A. Cell-autonomous circadian clock of hepatocytes drives rhythms in transcription and polyamine synthesis. Proc. Natl. Acad. Sci. U.S.A. 108, 18560-18565 (2011).
- Panda, S. Coordinated transcription of key pathways in the mouse by the circadian clock. Cell. 109, 307-320 (2002).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved