É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Monitorização das células-autônomas Ritmos relógio circadiano de expressão genética dos Repórteres Bioluminescência luciferase
Neste Artigo
Resumo
Relógios circadianos funcionam no interior das células individuais, ou seja, são células-autónomo. Aqui, descrevem métodos para a geração de modelos de células autónomas relógio usando não-invasiva, com base em tecnologia de luciferase bioluminescência em tempo real. Células repórter fornecer tratáveis, sistemas modelo funcional para estudar biologia circadiano.
Resumo
Nos mamíferos, muitos aspectos do comportamento e fisiologia, tais como ciclos de sono-vigília e metabolismo do fígado são regulados pela endógenos relógios circadianos (revisado 1,2). O sistema de cronometragem circadiano é uma rede multi-oscilador hierárquica, com o relógio central, localizado no núcleo supraquiasmático (SCN) a sincronização e coordenação de relógios SCN-extra e periférico em outro lugar 1,2. As células individuais são as unidades funcionais para a geração e manutenção de ritmos circadianos 3,4, e estes osciladores de diferentes tipos de tecidos de um organismo a percentagem notavelmente semelhante mecanismo de feedback negativo bioquímico. No entanto, devido a interacções a nível de rede neuronal no SCN e através rítmicos, pistas sistémicos ao nível dos organismos, os ritmos circadianos no nível organismal não são necessariamente células autónoma 5-7. Em comparação com estudos tradicionais de actividade locomotora in vivo e ex vivo explantes SCN, cell com base em ensaios in vitro permitir a descoberta de células autônomas defeitos circadianos 5,8. Estrategicamente, baseados em células modelos são mais tratáveis experimentalmente para a caracterização fenotípica e rápida descoberta de mecanismos de clock básico 5,8-13.
Porque os ritmos circadianos são dinâmicos, medições longitudinais com alta resolução temporal são necessários para avaliar a função do relógio. Em anos recentes, a gravação em tempo real usando a bioluminescência de luciferase do pirilampo, como repórter tornou-se uma técnica comum para o estudo de ritmos circadianos em mamíferos 14,15, uma vez que permite o exame da persistência e da dinâmica de ritmos moleculares. Para monitorar a célula-autónomos ritmos circadianos de expressão do gene, os repórteres de luciferase pode ser introduzido em células através de transfecção transiente 13,16,17 ou transdução estável 5,10,18,19. Aqui descrevemos um protocolo de transdução estável usando lentivírus mediada entrega de genes. Tele sistema vector lentiviral é superior aos métodos tradicionais, tais como a transfecção transiente e transmissão germinal devido à sua eficácia e versatilidade: ela permite a entrega eficiente e a integração estável no genoma do hospedeiro de ambos dividindo e não as células em divisão 20. Uma vez que uma linha celular repórter é estabelecida, a dinâmica da função de relógio pode ser examinado através da gravação de bioluminescência. Nós primeiro descrever a geração de P (Per2)-D linhas repórter Luc, e em seguida, apresentam dados deste e de outros repórteres circadianos. Nestes ensaios, os fibroblastos 3T3 de rato e células de osteosarcoma humano U2OS são utilizados como modelos celulares. Também discutimos várias formas de utilizar esses modelos de relógio em estudos circadianos. Métodos descritos aqui podem ser aplicados a uma grande variedade de tipos de células para o estudo da base molecular e celular do relógio circadiano, e pode ser útil na resolução de problemas em outros sistemas biológicos.
Protocolo
1. Construção de Lentiviral Repórteres luciferase
A construção repórter de mamífero circadiano geralmente contém uma cassete de expressão no qual um promotor circadiano é fundido com o gene da luciferase. Ambas as estratégias de ligação e de recombinação baseada são comumente usados para a clonagem de DNA. Como um exemplo, aqui descrevemos um método baseado em recombinação clonagem Gateway para gerar um P (Per2)-d repórter Luc lentiviral, em que a luciferase desestabilizado (d Luc) está sob o controle do promotor do rato Per2.
- A clonagem do promotor Per2. Use PCR para amplificar o fragmento de DNA Per2 promotor de 526 bp a montante do sítio de iniciação da transcrição a partir de um clone BAC Per2 rato 9-13, utilizando um iniciador directo (5'-CTCGAGCGGATTACCGAGGCTGGTCACG TC-3 ') e um iniciador reverso (5' CTCGAGTCCCTTGCTCGGCCCGTCAC-TTGG-3 '), e do clone em pENTR5'-TOPO vector (Invitrogen) para gerarpENTR5'-P (Per2).
- Clonagem de d Luc. O Luc d contém o gene da luciferase do pirilampo e de uma sequência PEST C-terminal por degradação de proteínas rápida como anteriormente descrito 21. Use PCR para amplificar o fragmento de DNA d Luc, e clone em pENTR / D-TOPO vector (Invitrogen) para gerar pENTR / Dd Luc.
- Construção do vector repórter. Misture os dois plasmídeos pENTR, pENTR5'-P (Per2) e pENTR / Dd Luc, com o vetor de destino lentiviral pLV7-BSD (BSD, blasticidina gene de resistência), e realizar a reação de recombinação usando Clonase para gerar um pLV7-BSD-P (Per2)-d repórter Luc (Figura 1). pLV7-Bsd é uma versão modificada (feito no nosso laboratório) de pLenti6/R4R2/V5-DEST (Invitrogen), em que o vírus da hepatite da marmota pós-transcricionais reguladoras de elementos (WPRE) sequências de 22 foram inseridos imediatamente a jusante da expressão de ca ssette para aumentar a expressão do gene.
2. Produção de Partículas Lentiviral
1. Células de sementes 293T (dia 1)
- Crescer de rim embrionário humano (HEK) 293T células até à confluência em DMEM 90-100% normal suplementado com FBS a 10% e penicilina-estreptomicina 1x-Glutamina (PSG) em placas de cultura de 10 cm. (Células com crescimento acelerado, com número de passagens baixo são essenciais para transfecção eficiente.)
- Antes de semear as células para transfecção, revestimento placas de cultura de 6 poços por adição de 1 ml de 0,001% de poli-L-lisina em PBS a cada poço e incubar à temperatura ambiente durante 20 min. Aspirar a solução e lavar uma vez com 1x PBS antes da utilização.
- Dissociar células 293T com tripsina e de sementes 0,75 x 10 6 células para cada poço das placas pré-revestidas com 2 ml de DMEM regular. Redemoinho as placas cuidadosamente para se obter uma distribuição uniforme de células em cada poço. Crescer as células na incubadora a 37 ° C durante a noite.
- Observe as células semeadas a partir do dia 1. Celular deve chegar a confluência de 80-90%.
- Preparar a mistura de transfecção do plasmídeo para um tubo de microcentrífuga de 1,5 ml por adição de 2 ug de ADN de um plasmídeo repórter lentiviral (por exemplo, pLV7-P (Per2) d-Luc; Liu lab) e os três vectores de empacotamento (1,3 ug Gag / Pol, 0,5 ug Rev, e 0,7 ug VSVG; Invitrogen). Como controlo, tanto para a transfecção e infecção subsequente, que geralmente incluem um poço adicional na transfecção de um vector lentiviral expressão da GFP, pLV156-CMV-EGFP (Figura 1A), abrigando proteína verde fluorescente melhorada (EGFP), sob o controlo do promotor de CMV como descrito previamente 20.
- Adicionar 100 ul de 0,25 M de CaCl2 (diluído com DNase / RNase DDH 2 O estoque de 2,5 M) à mistura de plasmídeo no passo 2 e misturar bem. Em seguida adicionar 100 ul de solução de BBS 2x (50 mM BES, 280 mM NaCl, 1,5 mM de Na 2 HPO 4, pH 6,95) e misturar suavemente mas profundamente. Incubar a mistura de ADN à temperatura ambiente durante 15 min.
- Enquanto espera, aspirar médio a partir de células 293T e altere a 2 ml de meio fresco. Regressar a placa à incubadora durante pelo menos 10 min para equilibrar o pH do meio antes da transfecção.
- Adicionar mistura de transfecção a partir do passo 3 para células 293T gota a gota. Placa Mexa suavemente e observar a formação de partículas sob um microscópio. Incubar em 5% de CO2, 37 ° C durante a noite. (Formação de partículas finas de CaPO precipitado 4 / DNA é crítica para a transfecção eficiente.)
3. Partículas virais de colheita (dias 3-4)
- Cerca de 16 horas após a transfecção (dia 3), através da qual as células de tempo deve chegar a 100% de confluência, aspirar meio de células e substituir com 2 ml de DMEM fresco regular. Incubar a 37 ° C durante a noite.
- No dia 4, avaliar a eficiência de transfecção pela observação de expressão EGFP em transfection células de controlo (A eficiência de transfecção de 90-100%, com maior expressão EGFP é uma previsão fiável de uma boa preparação viral.)
- Recolher o meio contendo segregadas, partículas virais infecciosas. Centrifugar a> 2000 xg durante 5 min para remover residuais células 293T e recolher o sobrenadante contendo vírus. Alternativamente, o meio pode ser limpo com um filtro de membrana de 0,45 um. As partículas virais estão prontos para utilização na infecção.
3. A infecção de células 3T3
1. As células 3T3 de semente (dia 3)
Dividir e número de sementes apropriada (~ 12000) de células 3T3 numa placa de 12 poços para se obter 20-30% de confluência por dia seguinte. Incubar a 37 ° C durante a noite.
2. Infectar as células 3T3 (dia 4)
- Observe as células semeadas. Confluência de 20-30% (menos do que 50%) é desejado para a infecção.
- Adicionar polibreno a uma concentração final de 5 ug / ml para o meio recolhido contendo viralpartículas. Misturar bem por pipetagem.
- Aspirar meio de células 3T3 e adicionar 1 ml da mistura acima viral por poço. Incubar a 37 ° C durante a noite. (Polybrene é utilizado para melhorar a eficiência de infecção, mas não é absolutamente necessário. Como pode ser tóxico para algumas células, o teste prévio é recomendado.)
3. Selecione as células infectadas (dia 5 em diante)
- Vinte e quatro horas após a infecção, aspirar meio contendo polibreno vírus e de células infectadas, lavar uma vez com PBS 1x e mudar para meio fresco. Incubar a 37 ° C durante a noite para a recuperação e crescimento.
- Quando confluentes (geralmente 1-2 dias mais tarde), dividir as células e incubar a 37 ° C durante a noite.
- No dia seguinte meio, aspirado a partir de células (<50% de confluência é desejada) e substitua com meio fresco contendo 10 ng / ml para seleccionar blasticidina para estavelmente transduzidas células. (Blasticidina matar-curva tem de ser determinado empiricamente para uma linha de células particular.)
- Mudar para meio fresco contendo blasticidina cada 2-3 dias para a selecção contínua de antibiótico-resistentes células que expressam os repórteres de relógio (geralmente 4-6 dias no total).
4. Gravação de bioluminescência de Células Reporter
1. Repórter células de sementes
Propagar células resistentes blasticidina repórter e dividida em placas de cultura de 35 mm. Incubar a 37 ° C até à confluência. Nós normalmente preparar ≥ 3 pratos para cada linha de células repórter em cada condição para fenotipagem circadiano.
2. Sincronização e mudança de meio de gravação
- Aspirar meio a partir de células confluentes repórter, lavar uma vez com PBS, e substituir com DMEM contendo 10 mM de forskolin (ou 200 nM de dexametasona). Incubar a 37 ° C durante 1 hora, para sincronizar as células. (Alternativamente, as células podem ser sincronizados por 23 ciclos de temperatura ou de choque de soro 24.)
- Enquanto espera, prepare recordeing meio para células 3T3 como se segue: 1x DMEM (HyClone) contendo 10% FBS, 1x pH Pen / Strep / Gln, 1 uM de forscolina, 1 mM de luciferina, 25 mM de HEPES, 7,4. Soro ea concentração de forskolin pode ser determinada empiricamente. No caso de células muito fraca, média livre de vermelho de fenol podem ser usados.
- No final do tratamento de forscolina, aspirar meio e substituir por meio de gravação feita recentemente.
3. Gravação bioluminescência de células repórter
- Após a mudança médio, cobrir placas de cultura com 40 milímetros lamínulas estéreis e selo em lugar com graxa de vácuo para evitar a evaporação.
- Carregar os pratos para o luminómetro LumiCycle, que é mantido no interior de um conjunto de incubadora a 36 ° C sem H 2 O ou CO 2.
- Iniciar gravação em tempo real de bioluminescência. Nós normalmente gravar ritmos durante 1 semana, seguido por troca de meio e de gravação contínua para uma segunda semana (ver Savelyev et al. Para mais pormenores) 25. (Para gravação de 96 nósll, placas Synergy SL2 foi usado como o dispositivo de gravação, ver Discussão 1.1 para detalhes).
5. Análise de Dados e Apresentação
Células repórter facilitar alta resolução de gravação de luminescência quantitativa, fundamental para determinar efeitos fenotípicos sobre a função do relógio circadiano. Para obter parâmetros circadianos, incluindo fase, a duração do período, amplitude, ritmo e taxa de amortecimento, usamos o programa de Análise LumiCycle (Actimetrics) para analisar os dados de bioluminescência 5,14. Resumidamente, os dados em bruto são de linha de base equipado primeiramente, e da linha de base-subtraídas dados são montados a uma onda seno, a partir da qual os parâmetros são determinados. Para amostras que mostram ritmos persistentes, bondade do ajuste de> 90% é geralmente obtida. Devido à elevada transiente bioluminescência após mudança de meio, que normalmente excluir o primeiro ciclo de análise de dados.
Para a apresentação dos dados, que geralmente traçar dados brutos (bioluminescência, contagens / s) contra time (dias). Quando necessário, os dados de linha de base, podem ser representados subtraídos para comparar a amplitude e fase.
6. Resultados representativos
1. Fase específicos repórteres circadianos
O relógio circadiano baseia-se num mecanismo de feedback negativo bioquímico 1. O ciclo de realimentação núcleo é constituído de ativadores transcricionais Bmal1 e relógio, e Pers repressores e Crys, que atuam sobre os elementos circadianos E / E'caixa-Enhancer para produzir a expressão do gene rítmica (com fase de manhã, por exemplo, Rev-erb α). O laço núcleo regula e integra pelo menos dois outros circadianos cis-elementos, o elemento de ligação DBP/E4BP4 (D-box, para fase de dia, por exemplo, Per3) eo elemento ROR / REV-ERB ligação (RRE, por fase de noite, por exemplo, Bmal1) 17. Regulação combinatória por múltiplos elementos circadianos pode gerar novas fases intermediárias. Por exemplo, a transcrição Cry1ção é mediada por todos os três elementos circadianos (isto é, os elementos E, / E'-box e D-box no promotor e RREs no primeiro intrão do gene Cry1), dando origem ao distinto Cry1 fase noite tempo 13.
Com base nestes mecanismos de regulação dos genes, foram geradas quatro construções repórter diferentes: P (Per2) d-Luc e P (Cry1)-D repórteres Luc contendo tanto E / E'-caixa e da caixa D-elementos na região reguladora 17, 26,27; P (Cry1) - Intron-d Luc representando regulação combinatória por todos os três elementos (ou seja, E / E'-box, D-box, e RRE) 13,17, e P (Bmal1)-d Luc regulada exclusivamente por RRE 9,17,19,21. Introduzimos estas repórteres em células 3T3 para produzir as fases antecipadas distintos de expressão repórter (Figura 2).
2. Knockdown do gene através de RNAi e farmacologicamente active compostos
Quando a eficiência de transfecção é elevada, siRNA sintético pode ser transientemente transfectados para células de derrubar a expressão do gene. Quando a transfecção é tecnicamente difícil, um vector de expressão podem ser estavelmente shRNA transduzida em células por meio de infecção por lentivírus, de modo a que shRNA produzido pela célula é processada para siRNA para knockdown do gene (KD). Apresentamos aqui os efeitos de KD Cry1 e cry2 genes utilizando siRNA em células U2OS (Figura 3A) e em células 3T3 shRNA, utilizando um vector de expressão de Gateway pLL3.7 9 (Figura 3B). Além de células 3T3, o modelo U2OS tornou-se outro modelo de relógio proeminente celular em grande parte porque ele atende aos requisitos fundamentais para high-throughput screening de bibliotecas disponíveis comercialmente siRNA humanos (por exemplo, origem humana, capaz de gerar fortes ritmos circadianos, função validada de todos os conhecidos genes do relógio, e passíveis de transfecção altamente eficiente egravação de luminescência quantitativa). Em ambos os tipos de células, RNAi mediada KD resultou em fenótipos de relógio consistentes com knockout rato anterior (KO) e estudos celulares KD 5,10,11,28. Por exemplo, Cry1 KD duração do período diminui e reduz a persistência do ritmo, enquanto cry2 KD prolonga período. Além disso, pequenas moléculas seleccionadas podem ser utilizadas para farmacologicamente alvo e a função da proteína perturb (Figura 3C).
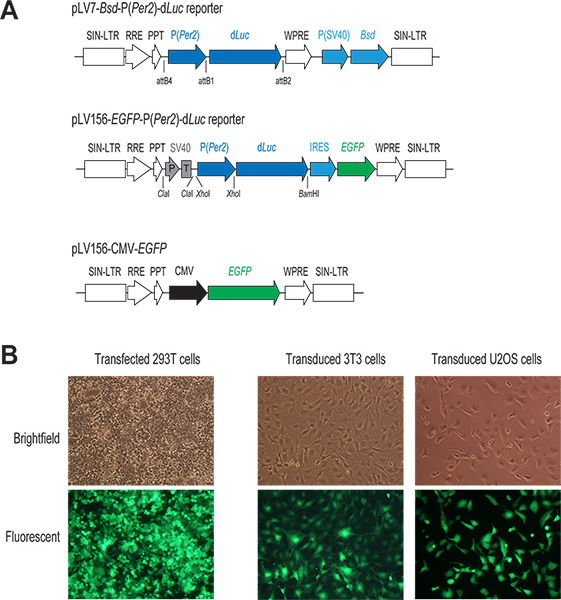
Figura 1. O lentivírus mediada por sistema de entrega de genes. (A) Diagrama esquemático de dois P lentiviral (Per2)-D vetores repórter Luc e uma CMV-EGFP construir. Apenas a região para a integração no genoma da célula hospedeira é mostrado. Em ambas as construções repórter, a transcrição de d Luc está sob o controlo directo do promotor Per2. No pLV7-BSD-P (Per2)-d Luc vetor (recombinação baseada clonagem), um gene de resistência à blasticidina coexpressas (BSD) facilita a selecção de células infectadas. No pLV156-P (Per2) d-Luc vector (ligadura baseado clonagem), tradução EGFP é mediada por um local interno de entrada do ribossoma (IRES) a jusante da d Luc, permitindo por meio de observação visual e triagem por FACS de células infectadas. Além disso, um promotor de SV40 / terminador (P / T), é usado como isolante (ver discussão 1.3). No vector de controlo CMV-EGFP, expressão EGFP está sob controle de um promotor de CMV forte. (B) as imagens fluorescentes de GFP-transfectadas e infectadas que expressam as células. Tipicamente, conseguimos alta eficiência tanto na transfecção transitória de células 293T e na infecção por lentivírus de linhas de células do nosso interesse, como indicado pela expressão de GFP nestas células. para ver figura maior .

Figura 2. Fase a expressão específica de repórteres em células 3T3 de bioluminescência Os vectores lentivirais repórter utilizados nesta experiência são pLV7-Bsd-P (Per2) d-Luc, P (Cry1) d-Luc, P (Cry1) -. Intron-d Luc, e P (Bmal1) d-Luc. Cada repórter apresenta uma fase distinta de oscilação, como indicado pelas setas. Enquanto o Per2 e Cry1 bioluminescência unidade promotores de pico na manhã de dia-fases e do promotor na fase Bmal1 noite, regulação combinatória pelo P (Cry1) - Intron abrigar E-box, D-box, e elementos RRE confere fase noite de bioluminescência de pico . Clique aqui para ver maior figura .
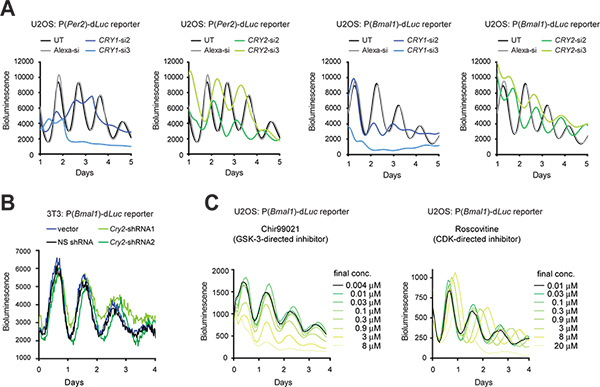
Figura 3. Perturbação genética e farmacológicas dos ritmos circadianos em células de bioluminescência repórter. (A) Efeitos de Cry1 e cry2 knockdown por siRNAs sobre ritmos celulares de células repórter U2OS. O luminómetro LumiCycle foi utilizado para a gravação de bioluminescência de células em pratos de 35 mm. A Figura é adaptado de Referência n º 10, com a permissão da Elsevier (2009). (B) Efeitos da cry2 knockdown por shRNAs sobre ritmos celulares de células repórter 3T3. Um vector de Gateway pLL3.7 contendo uma cassete de U6-shRNA foi usada para knockdown cry2 gene. shRNA2 tem uma eficiência melhor do que knockdown shRNA1 como determinado por análise de Western blot (dados não mostrados). Um luminómetro Synergy foi utilizado para a gravação de bioluminescência de células numa placa de 96 poços. As configurações de gravação são as seguintes:. Incubadora de temperatura, 33 ° C, o tempo de integração de 15 segundos, o tempo de intervalo, 30 min (C) Efeitos de inibidores de moléculas pequenas em celritmos intracelulares de células repórter U2OS. Chir99021 Roscovitina e são inibidores dirigidos contra a GSK-3 e CDK, respectivamente. O sistema ViewLux (Chir99021 ensaio) e um luminómetro Tecan (Roscovitina ensaio) foram utilizadas para as gravações de bioluminescência de células em 384 poços. Figura é adaptado de Referência # 19 (Copyright 2008 Academia Nacional de Ciências dos EUA). Clique aqui para ver maior figura .
Discussão
1. Modificações protocolo atual
1.1 dispositivos de gravação e considerações de rendimento
Devido à sua disponibilidade comercial, a LumiCycle (Actimetrics) tornou-se o dispositivo de luminimetro mais comumente utilizado automatizado para gravação em tempo real 4,5,9,19,29-31. O LumiCycle emprega tubos fotomultiplicadores (PMT), como detectores de luz, que proporcionam sensibilidade extremamente elevada e baixo ruído 14, e, por consegui...
Divulgações
Não há conflitos de interesse declarados.
Agradecimentos
Este trabalho foi financiado em parte pela National Science Foundation (IOS-0920417) (ACL).
Materiais
| Nome do reagente | Companhia | Número de catálogo | Comentários |
| DMEM | HyClone | SH30243FS | Para o crescimento celular normal |
| DMEM | Invitrogen | 12100-046 | Para luminometria |
| FBS | HyClone | SH3091003 | |
| Pen / Strep / Gln (100x) | HyClone | SV3008201 | |
| B-27 | Invitrogen | 17504-044 | |
| D-luciferina | Biosynth | L-8220 | |
| Poli-L-lisina | Sigma | P4707 | |
| Polybrene | Millipore | TR-1003-G | |
| Forskolin | Sigma | F6886 | |
| Todos os outros produtos químicos | Sigma | ||
| Equipamento | |||
| De cultura de tecidos incubadora | 5% de CO2 a 37 ° C | ||
| Capuz de cultura de tecidos | BSL-2 certificado | ||
| Light & fluorescente microscópio | O contraste de fase opcional | ||
| LumiCycle | Actimetrics |
Referências
- Reppert, S. M., Weaver, D. R. Coordination of circadian timing in mammals. Nature. 418, 935-941 (2002).
- Hastings, M. H., Reddy, A. B., Maywood, E. S. A clockwork web: circadian timing in brain and periphery, in health and disease. Nat. Rev. Neurosci. 4, 649-661 (2003).
- Nagoshi, E. Circadian gene expression in individual fibroblasts: cell-autonomous and self-sustained oscillators pass time to daughter cells. Cell. 119, 693-705 (2004).
- Welsh, D. K. Bioluminescence imaging of individual fibroblasts reveals persistent, independently phased circadian rhythms of clock gene expression. Curr. Biol. 14, 2289-2295 (2004).
- Liu, A. C. Intercellular coupling confers robustness against mutations in the SCN circadian clock network. Cell. 129, 605-616 (2007).
- Kornmann, B. System-driven and oscillator-dependent circadian transcription in mice with a conditionally active liver clock. PLoS Biol. 5, e34 (2007).
- Hogenesch, J. B., Herzog, E. D. Intracellular and intercellular processes determine robustness of the circadian clock. FEBS Lett. 585, 1427-1434 (2011).
- DeBruyne, J. P., Weaver, D. R., Reppert, S. M. Peripheral circadian oscillators require CLOCK. Curr. Biol. 17, 538-539 (2007).
- Liu, A. C. Redundant function of REV-ERBalpha and beta and non-essential role for Bmal1 cycling in transcriptional regulation of intracellular circadian rhythms. PLoS Genet. 4, e1000023 (2008).
- Zhang, E. E. A genome-wide RNAi screen for modifiers of the circadian clock in human cells. Cell. 139, 199-210 (2009).
- Baggs, J. E. Network features of the mammalian circadian clock. PLoS Biol. 7, e52 (2009).
- Hirota, T. High-throughput chemical screen identifies a novel potent modulator of cellular circadian rhythms and reveals CKIalpha as a clock regulatory kinase. PLoS Biol. 8, e1000559 (2010).
- Ukai-Tadenuma, M. Delay in feedback repression by cryptochrome 1 is required for circadian clock function. Cell. 144, 268-281 (2011).
- Yamazaki, S., Takahashi, J. S. Real-time luminescence reporting of circadian gene expression in mammals. Methods Enzymol. 393, 288-301 (2005).
- Welsh, D. K., Imaizumi, T., Kay, S. A. Real-time reporting of circadian-regulated gene expression by luciferase imaging in plants and mammalian cells. Methods Enzymol. 393, 269-288 (2005).
- Sato, T. K. Feedback repression is required for mammalian circadian clock function. Nat. Genet. 38, 312-319 (2006).
- Ueda, H. R. System-level identification of transcriptional circuits underlying mammalian circadian clocks. Nat. Genet. 37, 187-192 (2005).
- Brown, S. A. The period length of fibroblast circadian gene expression varies widely among human individuals. PLoS Biol. 3, e338 (2005).
- Hirota, T. A chemical biology approach reveals period shortening of the mammalian circadian clock by specific inhibition of GSK-3beta. Proc. Natl. Acad. Sci. U.S.A. 105, 20746-20751 (2008).
- Tiscornia, G., Singer, O., Verma, I. M. Production and purification of lentiviral vectors. Nat. Protoc. 1, 241-245 (2006).
- Ueda, H. R. A transcription factor response element for gene expression during circadian night. Nature. 418, 534-539 (2002).
- Zufferey, R., Donello, J. E., Trono, D., Hope, T. J. Woodchuck hepatitis virus posttranscriptional regulatory element enhances expression of transgenes delivered by retroviral vectors. J. Virol. 73, 2886-2892 (1999).
- Buhr, E. D., Yoo, S. H., Takahashi, J. S. Temperature as a universal resetting cue for mammalian circadian oscillators. Science. 330, 379-385 (2010).
- Balsalobre, A., Damiola, F., Schibler, . U.A serum shock induces circadian gene expression in mammalian tissue culture cells. Cell. 93, 929-937 (1998).
- Savelyev, S. A., Larsson, K. C., Johansson, A., Lundkvist, G. B. S. Slice Preparation, Organotypic Tissue Culturing and Luciferase Recording of Clock Gene Activity in the Suprachiasmatic Nucleus. J. Vis. Exp. (48), e2439 (2011).
- Akashi, M., Ichise, T., Mamine, T., Takumi, T. Molecular mechanism of cell-autonomous circadian gene expression of Period2, a crucial regulator of the mammalian circadian clock. Mol. Biol. Cell. 17, 555-565 (2006).
- Ohno, T., Onishi, Y., Ishida, N. A novel E4BP4 element drives circadian expression of mPeriod2. Nucleic Acids Res. 35, 648-655 (2007).
- Maier, B. A large-scale functional RNAi screen reveals a role for CK2 in the mammalian circadian clock. Genes Dev. 23, 708-718 (2009).
- Yoo, S. H. PERIOD2::LUCIFERASE real-time reporting of circadian dynamics reveals persistent circadian oscillations in mouse peripheral tissues. Proc. Natl. Acad. Sci. U.S.A. 101, 5339-5346 (2004).
- Liu, A. C., Lewis, W. G., Kay, S. A. Mammalian circadian signaling networks and therapeutic targets. Nat. Chem. Biol. 3, 630-639 (2007).
- Ko, C. H. Emergence of noise-induced oscillations in the central circadian pacemaker. PLoS Biol. 8, e1000513 (2010).
- Izumo, M., Johnson, C. H., Yamazaki, S. Circadian gene expression in mammalian fibroblasts revealed by real-time luminescence reporting: temperature compensation and damping. Proc. Natl. Acad. Sci. U.S.A. 100, 16089-16094 (2003).
- Izumo, M., Sato, T. R., Straume, M., Johnson, C. H. Quantitative analyses of circadian gene expression in mammalian cell cultures. PLoS Comput. Biol. 2, e136 (2006).
- Chen, Z. Identification of diverse modulators of central and peripheral circadian clocks by high-throughput chemical screening. Proc. Natl. Acad. Sci. U.S.A. 109, 101-106 (2011).
- Yamaguchi, S. Synchronization of cellular clocks in the suprachiasmatic nucleus. Science. 302, 1408-1412 (2003).
- Akashi, M., Hayasaka, N., Yamazaki, S., Node, K. Mitogen-activated protein kinase is a functional component of the autonomous circadian system in the suprachiasmatic nucleus. J. Neurosci. 28, 4619-4623 (2008).
- Hoshino, H., Nakajima, Y., Ohmiya, Y. Luciferase-YFP fusion tag with enhanced emission for single-cell luminescence imaging. Nat. Methods. 4, 637-639 (2007).
- Asai, M. Visualization of mPer1 transcription in vitro: NMDA induces a rapid phase shift of mPer1 gene in cultured SCN. Curr. Biol. 11, 1524-1527 (2001).
- Wilsbacher, L. D. Photic and circadian expression of luciferase in mPeriod1-luc transgenic mice in vivo. Proc. Natl. Acad. Sci. U.S.A. 99, 489-494 (2002).
- Yamazaki, S. Resetting central and peripheral circadian oscillators in transgenic rats. Science. 288, 682-685 (2000).
- Welsh, D. K., Noguchi, T., Yuste, R. Cellular bioluminescence imaging. Imaging: A Laboratory Manual. , 369-385 (2011).
- Nakajima, Y. Enhanced beetle luciferase for high-resolution bioluminescence imaging. PLoS One. 5, e10011 (2010).
- Guilding, C. A riot of rhythms: neuronal and glial circadian oscillators in the mediobasal hypothalamus. Mol. Brain. 2, 28 (2009).
- O'Neill, J. S. cAMP-dependent signaling as a core component of the mammalian circadian pacemaker. Science. 320, 949-953 (2008).
- Fuller, P. M., Lu, J., Saper, C. B. Differential rescue of light- and food-entrainable circadian rhythms. Science. 320, 1074-1077 (2008).
- Mukherjee, S. Knockdown of Clock in the ventral tegmental area through RNA interference results in a mixed state of mania and depression-like behavior. Biol. Psychiatry. 68, 503-511 (2010).
- Saijo, K. A Nurr1/CoREST pathway in microglia and astrocytes protects dopaminergic neurons from inflammation-induced death. Cell. 137, 47-59 (2009).
- Elias, G. M. Synapse-specific and developmentally regulated targeting of AMPA receptors by a family of MAGUK scaffolding proteins. Neuron. 52, 307-320 (2006).
- Isojima, Y. CKIepsilon/delta-dependent phosphorylation is a temperature-insensitive, period-determining process in the mammalian circadian clock. Proc. Natl. Acad. Sci. U.S.A. 106, 15744-15749 (2009).
- Bucan, M., Abel, T. The mouse: genetics meets behaviour. Nat. Rev. Genet. 3, 114-123 (2002).
- Hughes, M. E. Harmonics of circadian gene transcription in mammals. PLoS Genet. 5, e1000442 (2009).
- Atwood, A. Cell-autonomous circadian clock of hepatocytes drives rhythms in transcription and polyamine synthesis. Proc. Natl. Acad. Sci. U.S.A. 108, 18560-18565 (2011).
- Panda, S. Coordinated transcription of key pathways in the mouse by the circadian clock. Cell. 109, 307-320 (2002).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados