JoVE 비디오를 활용하시려면 도서관을 통한 기관 구독이 필요합니다. 전체 비디오를 보시려면 로그인하거나 무료 트라이얼을 시작하세요.
Method Article
루시 페라 제 Bioluminescence 기자를 사용하여 유전자 발현의 셀 자치 Circadian 시계 리듬을 모니터링
요약
Circadian 시계는 개별 세포 내 기능, 즉, 그들은 세포 자율적 있습니다. 여기, 우리는 비 침습적, 루시 페라 기반의 실시간 bioluminescence 기술을 사용하여 셀 자치 클럭 모델을 생성하는 방법을 설명합니다. 리포터 전지는 circadian 생물학을 공부를위한 다루기 쉬운, 기능 모델 시스템을 제공합니다.
초록
포유 동물에서 행동과 수면 각성주기와 간 대사 등 생리학의 여러 측면은 내생 circadian 시계 (1,2 검토)에 의해 규제됩니다. circadian 시간 유지 시스템은 동기화와 다른 1,2 추가 - SCN 및 주변 장치 클럭을 조정 suprachiasmatic 핵 (SCN)에있는 중앙 시계와 함께 계층 멀티 발진기 네트워크입니다. 개인 세포가 생성 및 circadian 리듬 3,4, 그리고 유기체 공유에서 다른 조직 유형과 매우 비슷 생화학 부정적인 피드백 메커니즘의 이러한 발진기의 유지 관리를위한 기능 단위입니다. 그러나, SCN의 neuronal 네트워크 수준과 organismal 수준의 리듬, 전신 신호를 통해 상호 작용으로 인해, organismal 수준의 circadian 리듬은 세포 자율적는 5-7 일 필요는 없습니다. 생체 및 SCN explants 예 생체, C의 전위의 활동의 전통 연구에 비해예쁜 기반 세포 자율적 circadian 결함 5,8의 검색을 허용 체외 assays 인치 전략적으로, 셀 기반 모델은 phenotypic 특성화 및 기본 클럭 메커니즘 5,8-13의 급속한 발견에 대한 더 많은 실험적으로 다루기 쉬운 수 있습니다.
circadian 리듬는 동적이기 때문에, 높은 시간적 해상도 길이 측정은 시계 기능을 평가하기 위해 필요합니다. 최근에는 기자로 반딧불의 루시 페라 제를 사용하여 실시간 bioluminescence 기록은 분자 리듬의 인내와 역학의 시험을 수 있으므로, 포유 동물 14,15에 circadian 리듬을 공부를위한 공통의 기술이되었습니다. 유전자 표현의 세포 자치 circadian 리듬을 모니터링하려면 루시 페라 제 기자 과도 transfection 13,16,17 또는 안정적으로 도입 5,10,18,19를 통해 세포에 도입 할 수 있습니다. 여기 lentivirus로 인한 유전자 전달을 사용하여 안정적인 전달 프로토콜을 설명합니다. 티그는 lentiviral 벡터 시스템은 때문에 그 효율성과 기능성의 과도 transfection 및 germline 전송 등의 전통적인 방법으로 우수합니다 : 그것은 분리와 세포 (20)를 비 나누어 모두의 호스트 게놈에 효율적으로 전달하고 안정적인 통합을 할 수 있습니다. 기자 셀 라인이 설정되면, 클럭 기능의 역학은 bioluminescence 기록을 통해 검사 할 수 있습니다. 우리가 처음 P (Per2)-D 루크의 기자 라인, 다음이 및 기타 circadian 기자의 현재 데이터의 생성을 설명합니다. 이러한 assays에서 3T3 마우스 섬유 아세포와 U2OS 인간의 osteosarcoma 세포는 세포 모델로 사용됩니다. 우리는 또한 circadian 연구에서 이러한 클럭 모델을 사용하여 다양한 방법에 대해 논의합니다. 여기에 설명 된 방법은 circadian 시계의 세포와 분자 기초를 공부하는 세포 유형의 훌륭한 다양한에 적용 할 수 있으며, 다른 생물 시스템의 문제를 태클에 유용 할 수 있습니다.
프로토콜
1. Lentiviral 루시 페라 제 기자의 건설
포유류의 circadian 기자 구조는 일반적으로 circadian 발기인이 루시 페라 제 유전자와 융합되는 표현 카세트가 포함되어 있습니다. 모두 결합 및 재결합 기반의 전략은 일반적으로 DNA의 복제에 사용됩니다. 예를 들어, 여기에 우리는 불안정한 루시 페라 제 (D 루크)은 마우스 Per2 발기인의 통제하에 있습니다있는 P (Per2)-D 루크 lentiviral 기자를 생성하기위한 재조합 기반의 게이트웨이 복제 방법을 설명합니다.
- Per2 발기인의 복제. 앞으로 입문서 (5'-CTCGAGCGGATTACCGAGGCTGGTCACG TC-3 ')와 역 프라이머 (5'를 사용하여 상류 마우스 Per2 BAC 클론 9-13에서 전사 시작 사이트의, 526 BP의 Per2 프로모터 DNA 조각을 증폭하는 PCR를 사용하여 - CTCGAGTCCCTTGCTCGGCCCGTCAC TTGG-3 ')와 pENTR5'-TOPO 벡터 (Invitrogen)에 클론 생성하기pENTR5'-P (Per2).
- D 루크의 복제. D 루크가 반딧불의 루시 페라 제 유전자와 이전에 21에 설명 된대로 빠른 단백질 저하를위한 C-터미널 해충 순서가 포함되어 있습니다. pENTR / DD 루크을 생성 할 pENTR / D-TOPO 벡터 (Invitrogen)에 D 루크 DNA 조각 및 복제를 증폭 PCR을 사용합니다.
- 기자 벡터의 건설. lentiviral 대상 벡터 pLV7 - BSD (BSD, blasticidin 저항 유전자)와 두 pENTR의 plasmids, pENTR5'-P (Per2)와 pENTR / DD 루크, 혼합, 그리고 pLV7-BSD-P를 생성하는 Clonase를 사용하여 재조합 반응을 수행 (Per2)-D 루크 기자 (그림 1). pLV7-BSD는 마못 간염 바이러스가 포스트 전사 규제 요소 (WPRE) 시퀀스 (22)가 표현 CA의 하류 즉시 삽입 된에 pLenti6/R4R2/V5-DEST (Invitrogen)의 수정 버전 (우리가 실험실에서 만든)입니다 유전자 발현을 향상시킬 수 ssette.
2. Lentiviral 입자의 생산
1. 종자 293T 세포 (일 1)
- 10% FBS와 1X 10cm 문화 요리에 페니실린 - 스트렙토 마이신-글루타민 (PSG)와 보충 정기적 DMEM의 90-100%의 합류로 인간 배아 신장 (HEK) 293T 세포를 성장. (낮은 통로 번호가 빠르게 성장하는 세포는 효율적인 transfection에 중요하다.)
- 이전 각도에 PBS에서 0.001 % 폴리-L-라이신의 1 ML를 추가하고 20 분 동안 실온에서 배양하여 transfection, 코트 6도 문화 판의 셀을 퍼 뜨리고 있습니다. 솔루션을 기음하고 사용하기 전에 1X PBS로 한 번 씻어.
- 트립신과 씨앗을 0.75로 293T 세포를 해리 X 10 2 ML 정기적 DMEM으로 사전 코팅 플레이트의 각도로 6 셀. 접시 철저하게 소용돌이 각 우물에 세포의도 분포를 얻을 수 있습니다. 37 ° C 하룻밤에 보육에 세포를 성장.
- 일 1 번 시드의 세포를 관찰. 세포는 80-90%의 합류에 도달해야합니다.
- 그리고 3 포장 벡터 (1.3 μg의 개그 / 폴, 0.5 μg, lentiviral 기자 플라스미드 DNA의 2 μg을 (류 연구실 예를 들어, pLV7-P (Per2)-D 루크)를 추가하여 1.5 ML의 microcentrifuge 관 플라스미드 transfection 믹스를 준비 레브, 그리고 0.7 μg의 VSVG, Invitrogen). transfection 및 그 이후의 감염 모두 제어, 우리는 일반적으로 CMV 프로모터의 제어에 따라 강화 된 녹색 형광 단백질 (EGFP)를 품고 lentiviral GFP의 발현 벡터에 대한 transfection에 추가 잘 pLV156-CMV-EGFP (그림 1A)를 포함 이전과 같이 20 설명했다.
- 2 단계에서 플라스미드 믹스에 0.25 M CaCl 2의 100 μl를 (2.5 M 주식에서 DNase / RNase 무료 ddH 2 O로 희석) 추가하고 철저하게 섞는다. 그런 다음 배 BBS 솔루션 100 μl (50 MM은 BE를 추가S, 280 MM NaCl, 1.5 2 HPO 4, 산도 6.95) 오세영 MM 부드럽게하지만 철저하게 섞는다. 15 분 동안 실온에서 DNA 믹스를 품다.
- 기다리는 동안, 293T 세포에서 미디어를 대기음 2 ML 신선한 매체로 변경합니다. transfection 전에 중간 pH를 평형 적어도 10 분 동안 인큐베이터에 판을 반환합니다.
- 드롭하여 293T 세포 드롭 3 단계에서 transfection 믹스를 추가합니다. 소용돌이 부드럽게 플레이트와 현미경 입자 형성을 관찰합니다. 5 % ° C 야간 CO 2, 37 품다. (카포 4 / DNA의 석출물의 미세 입자 형성, 효율적인 transfection에 중요합니다.)
3. 수확 바이러스 입자 (일 3-4)
- 정보 16시간 후 transfection (주 3) 어떤하여 시간 세포는 100 % 합류에 도달해야 세포에서 미디어를 대기음 2 ML 신선한 정기적 DMEM으로 교체하십시오. 37 ° C 하룻밤에 품다.
- 일 4 일 트란에 EGFP 표정을 관찰하여 transfection 효율을 평가sfection 제어 세포 (높은 EGFP 발현과 90-100%의 Transfection 효율이 좋은 바이러스 사립의 믿을 수 예측기입니다.)
- 분비 전염성 바이러스 입자를 포함하는 매체를 수집합니다. 잔여 293T 세포를 제거하고 바이러스가 포함 된 표면에 뜨는를 수집하는 5 분에> 2,000 XG에 원심 분리기. 또는 매체는 0.45 μm 멤브레인 필터로 삭제 될 수 있습니다. 바이러스 입자는 감염에 사용하기위한 준비가되어 있습니다.
3. 3T3 세포의 감염
1. 종자 3T3 세포 (3 일)
12 잘 접시에 플리트 (Split)와 3T3 세포의 씨앗이 적절한 수의 (~ 12,000) 다음 날인 20-30%의 합류를 얻을 수 있습니다. 37 ° C 하룻밤에 품다.
2. 3T3 세포를 (일 4) 감염
- 시드 세포를 관찰. 20~30% (50 % 미만)의 합류는 감염 원하는 있습니다.
- 바이러스를 포함하는 수집 된 매체에 5 μg / ML의 최종 농도에 polybrene을 추가입자. pipetting으로 잘 섞는다.
- 3T3 세포에서 미디어를 대기음, 잘 당 위의 바이러스 혼합물의 1 ML를 추가합니다. 37 ° C 하룻밤에 품다. (Polybrene는 감염 효율을 향상하는 데 사용됩니다,하지만 절대적으로 필요하지 않습니다. 그 어떤 세포에 독성이 될 수 있으므로, 사전에 테스트를 권장합니다.)
3. 감염된 세포를 (일 5 이후)을 선택
- 스물 네 시간 후 감염, 감염된 세포에서 바이러스와 polybrene를 포함하는 매체를 대기음, 1X PBS로 한 번 씻고, 신선한 매체에 변경합니다. 37 복구 및 성장 ° C 하룻밤에 품다.
- 언제 합류하면 (보통 1-2일 이상), 37 ° C 하룻밤에 세포와 배양을 분할.
- 다음 날, 기음 세포 (<50 % 합류가 원하는 경우)에서 매체와 안정적를 위해 선택할 수 10 μg / ML의 Blasticidin를 포함하는 새로운 매체로 교체가 세포를 transduced. (Blasticidin이 죽일 곡선 경험적으로 특정 세포 라인에 대한 결정해야합니다.)
- 는 클럭 기자 (일반적으로 4~6일 총) 표현 항생제에 저항력이 세포의 지속적인 선택에 대한 모든 2-3일 Blasticidin를 포함하는 새로운 매체로 변경합니다.
4. 리포터 세포 Bioluminescence 기록
1. 시드 기자 세포
Blasticidin 방지 기자 세포를 전파하고 35 mm 문화 요리에 분할. 합류 할 때까지 37 ° C에서 알을 품다. 우리는 보통 circadian phenotyping 각 조건에서 각각 기자 셀 라인 ≥ 3 요리를 준비합니다.
2. 동기화 및 기록 매체로 변경
- 합류 기자 세포에서 미디어를 대기음, PBS로 한 번 씻고, 그리고 DMEM 10 μM forskolin (또는 200 nm의 dexamethasone)을 포함하는으로 대체합니다. 세포를 동기화 1 시간 37 ° C에서 알을 품다. (또는 셀은 온도 사이클 23 또는 혈청 충격 (24)에 의해 동기화 할 수 있습니다.)
- 기다리는 동안 기록을 준비다음과 같이 3T3 세포에 대한 ING 매체 : FBS, 1X 펜 / 패 혈성 / Gln, 1 μM forskolin, 1 MM luciferin, 25 MM HEPES, pH를 7.4 10 %를 포함 1X DMEM (HyClone). 세럼과 forskolin 농도는 경험적으로 결정될 수있다. 매우 희미한 세포를 들어, 페놀 붉은 무료 매체를 사용할 수 있습니다.
- forskolin 치료의 끝에서, 미디어를 기음과 갓 만든 기록 매체를 교체하십시오.
3. 기자 세포의 Bioluminescence 기록
- 중간 변경 후, 증발을 방지하기 위해 진공 그리스와 장소에서 40mm 멸균 coverslips 및 인감과 문화 요리를 다룹니다.
- H 2 O 나 CO 2가 36 ° C에서 보육 세트 내부에 보관되어 LumiCycle의 luminometer,에 요리를로드합니다.
- 실시간 bioluminescence 녹화를 시작합니다. 우리는 보통 두 번째 주에 대한 중간 변화와 연속 녹화 (Savelyev 외를 참조하십시오. 자세한 내용은) 25 다음 1 주일 동안 리듬을 기록합니다. (96 - 우리의 기록을 위해붙인다 접시, 시너지 SL2는 녹음 장치로 사용되었다가, 세부 사항에 대한 토론 1.1을 참조).
5. 데이터 분석 및 프리젠 테이션
리포터 전지는 circadian 시계 기능에 phenotypic 효과를 결정하는 중요한 고해상도 정량 발광 녹음을 용이하게합니다. 상 기간 길이, 리듬 진폭과 감쇠 속도 등의 circadian 매개 변수를 얻으려면, 우리는 bioluminescence 데이터에게 5,14를 분석 할 수있는 LumiCycle 분석 프로그램 (Actimetrics)을 사용합니다. 간단히, 원시 데이터는 기준선 먼저 장착되어 있습니다, 그리고 기본-빼는 데이터는 매개 변수가 결정되는에서 사인 웨이브에 장착되어 있습니다. 영구적 인 리듬,> 90 % 이내 달성의 신 -의 - 피팅을 보여 샘플하십시오. 중간 변경시 높은 과도 bioluminescence로 인해, 우리는 보통 분석에서 데이터의 첫 번째 사이클을 제외 할 수 있습니다.
데이터 프리젠 테이션 위해, 우리는 일반적으로 TI에 대한 원시 데이터 (bioluminescence, 카운트 / 초)를 타게나 (일). 필요한 경우 기본 - 공제 데이터는 진폭 및 위상을 비교 해본 할 수 있습니다.
6. 대표 결과
1. 단계 별 circadian 기자
circadian 시계는 생화학 부정적인 피드백 메커니즘 하나에 기반을두고 있습니다. 핵심 피드백 루프는 리듬 유전자 발현을 (아침 단계, 예를 들어, 레브-erb α 포함) 생산 circadian E / E' 박스 증강 요소에 따라 행동 전사 activators BMAL1와 시계, 그리고 repressors 인용과 CRYs로 구성되어 있습니다. 핵심 루프는 적어도 두 개의 다른 circadian CIS 요소, DBP/E4BP4 바인딩 요소 조절과 통합 (D-상자를, 일 단계, 예를 들어, Per3 용), 밤 단계에 있으며, ROR / 레브-ERB 바인딩 요소 (RRE 예를 들어, Bmal1) 17. 여러 circadian 요소의 조합 규제 소설 중간 단계를 생성 할 수 있습니다. 예를 들어, Cry1 transcrip기는 별개의 Cry1 저녁 시간 단계 13 상승을주는 세 가지 circadian 요소 (Cry1 유전자의 첫 번째 인트론의 발기인 및 RREs에서 즉, E / E'-상자 및 D-상자 요소)에 의해 중재된다.
유전자 규정의 이러한 메커니즘을 바탕으로, 우리는 네 가지 기자 구조 생성 : P (Per2)-D 루크와 P (Cry1)를 E / E'-상자 및 규제 지역 17 D-상자 요소를 모두 포함하는-D 루크 기자, 26,27, P (Cry1) - 모든 세 가지 요소에 의해 조합 규정 (즉, E / E' 박스, D-상자, RRE) 13,17을 대표하는 인트론-D 루크, 그리고 P (Bmal1)-D 루크 규제 독점적으로 RRE 9,17,19,21으로. 우리는 기자 식의 예상 독특한 단계 (그림 2)을 생산하기 위해 3T3 세포로이 기자를 소개했다.
2. RNAi와 pharmacologically acti을 통해 유전자 최저화합물이 있어요
transfection 효율이 높은 경우, 합성 siRNA는 transiently 유전자 발현을 노크 셀로 transfected 수 있습니다. transfection이 기술적으로 어려운 경우, shRNA의 발현 벡터는 세포에 의해 생성 shRNA이 유전자 최저 (KD)에 대한 siRNA를 처리되므로, 안정적으로 lentiviral 감염을 통해 세포에 transduced 할 수 있습니다. 여기 pLL3.7 게이트웨이 발현 벡터 9 (그림 3B)를 사용하여 3T3 세포에 U2OS 세포에서 siRNA (그림 3A)과 shRNA를 사용하여 Cry1과 Cry2 유전자의 KD 효과를 제시한다. 3T3 세포뿐만 아니라, U2OS 모델은 상용화 인간의 siRNA 라이브러리 높은 처리량 검사 (예를 들어, 인간의 기원, 강력한 circadian 리듬의 검증 기능을 생성 할 수에 대한 주요 요구 사항을 충족 크게하기 때문에 또 다른 걸출한 세포의 시계 모델이되었습니다 모든 알려진 시계 유전자, 그리고 고효율 transfection 및 의무양적 발광 기록). 두 세포 유형에서 RNAi로 인한 KD은 이전 마우스 녹아웃 (KO)와 셀룰러 KD 연구 5,10,11,28과 일치 클럭 phenotypes하게되었습니다. 예를 들어, Cry1 KD 단축 기간의 길이와 리듬 지속성을 감소는 Cry2 KD는 기간을 길게 반면. 또한, 선택 작은 분자 pharmacologically 표적을 교란 단백질 기능 (그림 3C)에 사용할 수 있습니다.
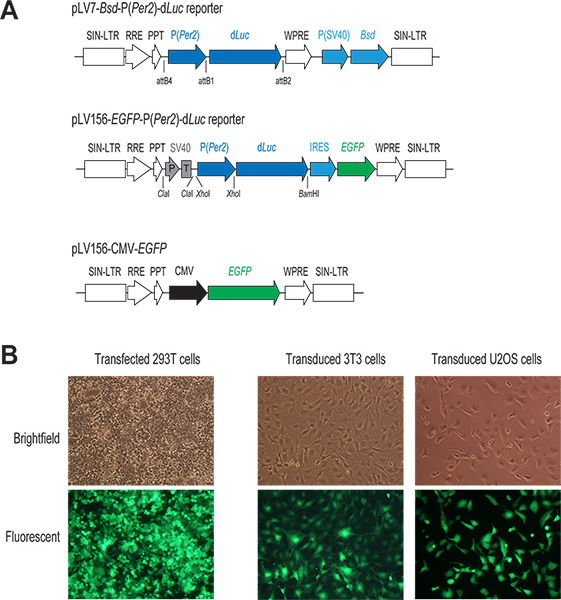
1 그림. lentivirus로 인한 유전자 전달 시스템. 이 lentiviral P (Per2)-D 루크의 기자 벡터와 CMV-EGFP의 (A) 도식 다이어그램 구성합니다. 숙주 세포의 게놈에 통합 만이 지역이 표시됩니다. 두 기자 구조에서 D 루크의 전사는 Per2 발기인 직접 통제하에 있습니다. pLV7-BSD-P (Per2)-D 루크 벡터 (재에서조합 기반의 복제), coexpressed Blasticidin 저항 유전자 (BSD)는 감염된 세포의 선택을 용이하게합니다. pLV156-P (Per2)에서-D 루크 벡터 (결합 기반 복제)은 EGFP 번역은 시각적 관찰과 감염된 세포의 분류 FACS을 허용, 내부 ribosome 항목 사이트 (IRES) D 루크의 하류에 의해 중재됩니다. 또한,이 SV40 프로모터 / 터미네이터 (P / T)은 절연체 (토론 1.3 참조)로 사용됩니다. CMV-EGFP 제어 벡터에서 EGFP 표현은 강한 CMV 프로모터의 제어 아래에 있습니다. transfected하고 감염된 GFP-표현 세포의 (B) 형광 이미지. 일반적으로, 우리는 293T 세포의 과도 transfection과 이러한 셀에 GFP 표현으로 표시 우리의 관심의 세포 라인의 lentiviral 감염 모두에서 높은 효율을 얻을 수 있습니다. 더 큰 그림을 보려면 여기를 클릭하십시오 .
그림 2. 3T3 세포의 bioluminescence 기자의 단계 별 표현이 실험에 사용 된 lentiviral 기자 벡터가 pLV7-BSD-P (Per2)-D 루크, P (Cry1)-D 루크, P (Cry1) -. 인트론-D 루크, 와 P (Bmal1)-D 루크. 각 기자는 화살표로 표시 진동의 고유 한 위상을 전시하고 있습니다. Per2 및 아침 일 단계 밤 단계에서 Bmal1 발기인, P (Cry1)에 의한 조합 규정에 Cry1 추구하는 드라이브 피크 bioluminescence는 있지만 - 인트론은 E-상자를 품고, D-상자, RRE 요소는 최대 bioluminescence의 저녁 단계를 수여 . 큰 그림을 보려면 여기를 클릭하십시오 .
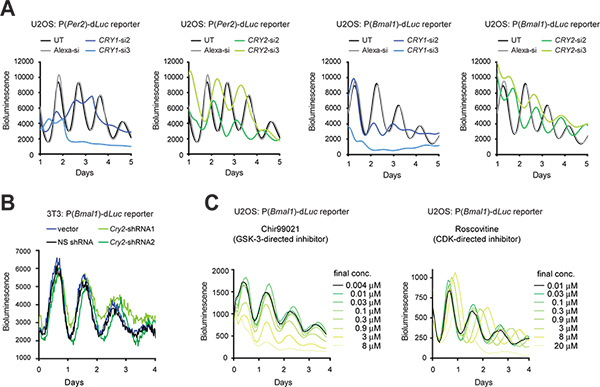
그림 3. 기자 세포의 circadian bioluminescence 리듬의 유전자와 약리 섭동. (A) Cry1의 효과와 U2OS 기자 세포의 세포 리듬에 siRNAs로 Cry2 최저. LumiCycle의 luminometer는 35mm 요리 세포의 bioluminescence 기록을 위해 사용되었다. 그림이 구성된다 참조 Elsevier (2009 년)의 허가와 # 10. 3T3 기자 세포의 세포 리듬에 shRNAs의 Cry2 최저의 (B) 효과.에서 U6-shRNA 카세트를 포함하는 pLL3.7 게이트웨이 벡터는 Cry2 유전자 최저 사용되었다. shRNA2 서양 얼룩 분석 (데이터가 표시되지 않음)에 의해 결정으로 shRNA1보다 최저 효율을 갖추고 있습니다. 시너지 luminometer는 96 - 웰 플레이트에 세포의 bioluminescence 기록을 위해 사용되었다. 레코딩을위한 설정은 다음과 같습니다. 보육 온도 33 ° C, 적분 시간, 15 초, cel에 작은 분자 억제제의 간격 시간, 30 분 (C) 효과U2OS 기자 세포의 lular 리듬. Chir99021 및 Roscovitine는 각각, GSK-3와 CDK에 대한 감독 억제제입니다. ViewLux 시스템 (Chir99021 검정)과 Tecan의 luminometer (Roscovitine 검정)는 384 잘 접시에 세포의 bioluminescence 녹음을 위해 사용되었습니다. 그림은 참조 # 19 (과학 저작권 2008 국립 아카데미, USA)에서 구성된다. 큰 그림을 보려면 여기를 클릭하십시오 .
토론
1. 현재 프로토콜에 대한 수정
1.1 녹화 장치 및 처리량을 고려
때문에 그 상업적 가용성, LumiCycle (Actimetrics)는 실시간 녹화 4,5,9,19,29-31에 가장 일반적으로 사용되는 자동화 된 luminometer 장치가되었습니다. LumiCycle은 매우 높은 감도와 낮은 노이즈 14 제공 빛 감지기로 광전자 증 튜브를 (PMTs) 고용, 따라서 매우 희미 루시 페라 기반 bioluminescence의...
공개
관심 없음 충돌이 선언 없습니다.
감사의 말
이 작품은 국립 과학 재단 (National Science Foundation) (IOS-0920417) (ACL)에 의해 부분적으로 지원되었다.
자료
| 시약의 이름 | 회사 | 카탈로그 번호 | 코멘트 |
| DMEM | HyClone | SH30243FS | 일반 세포의 성장을 위해 |
| DMEM | Invitrogen | 12100-046 | luminometry에 대한 |
| FBS | HyClone | SH3091003 | |
| 펜 / 패 혈성 / Gln (100x) | HyClone | SV3008201 | |
| B-27 | Invitrogen | 17504-044 | |
| D-Luciferin | Biosynth | L-8220 | |
| 폴리-L-라이신 | 시그마 | P4707 | |
| Polybrene | Millipore | TR-1003-G | |
| Forskolin | 시그마 | F6886 | |
| 다른 모든 화학 물질 | 시그마 | ||
| 장비 | |||
| 조직 문화 인큐베이터 | 37 ° C에서 5 % CO2 | ||
| 조직 문화 후드 | BSL-2 인증 | ||
| 형광 및 라이트 현미경 | 선택 단계 대비 | ||
| LumiCycle | Actimetrics |
참고문헌
- Reppert, S. M., Weaver, D. R. Coordination of circadian timing in mammals. Nature. 418, 935-941 (2002).
- Hastings, M. H., Reddy, A. B., Maywood, E. S. A clockwork web: circadian timing in brain and periphery, in health and disease. Nat. Rev. Neurosci. 4, 649-661 (2003).
- Nagoshi, E. Circadian gene expression in individual fibroblasts: cell-autonomous and self-sustained oscillators pass time to daughter cells. Cell. 119, 693-705 (2004).
- Welsh, D. K. Bioluminescence imaging of individual fibroblasts reveals persistent, independently phased circadian rhythms of clock gene expression. Curr. Biol. 14, 2289-2295 (2004).
- Liu, A. C. Intercellular coupling confers robustness against mutations in the SCN circadian clock network. Cell. 129, 605-616 (2007).
- Kornmann, B. System-driven and oscillator-dependent circadian transcription in mice with a conditionally active liver clock. PLoS Biol. 5, e34 (2007).
- Hogenesch, J. B., Herzog, E. D. Intracellular and intercellular processes determine robustness of the circadian clock. FEBS Lett. 585, 1427-1434 (2011).
- DeBruyne, J. P., Weaver, D. R., Reppert, S. M. Peripheral circadian oscillators require CLOCK. Curr. Biol. 17, 538-539 (2007).
- Liu, A. C. Redundant function of REV-ERBalpha and beta and non-essential role for Bmal1 cycling in transcriptional regulation of intracellular circadian rhythms. PLoS Genet. 4, e1000023 (2008).
- Zhang, E. E. A genome-wide RNAi screen for modifiers of the circadian clock in human cells. Cell. 139, 199-210 (2009).
- Baggs, J. E. Network features of the mammalian circadian clock. PLoS Biol. 7, e52 (2009).
- Hirota, T. High-throughput chemical screen identifies a novel potent modulator of cellular circadian rhythms and reveals CKIalpha as a clock regulatory kinase. PLoS Biol. 8, e1000559 (2010).
- Ukai-Tadenuma, M. Delay in feedback repression by cryptochrome 1 is required for circadian clock function. Cell. 144, 268-281 (2011).
- Yamazaki, S., Takahashi, J. S. Real-time luminescence reporting of circadian gene expression in mammals. Methods Enzymol. 393, 288-301 (2005).
- Welsh, D. K., Imaizumi, T., Kay, S. A. Real-time reporting of circadian-regulated gene expression by luciferase imaging in plants and mammalian cells. Methods Enzymol. 393, 269-288 (2005).
- Sato, T. K. Feedback repression is required for mammalian circadian clock function. Nat. Genet. 38, 312-319 (2006).
- Ueda, H. R. System-level identification of transcriptional circuits underlying mammalian circadian clocks. Nat. Genet. 37, 187-192 (2005).
- Brown, S. A. The period length of fibroblast circadian gene expression varies widely among human individuals. PLoS Biol. 3, e338 (2005).
- Hirota, T. A chemical biology approach reveals period shortening of the mammalian circadian clock by specific inhibition of GSK-3beta. Proc. Natl. Acad. Sci. U.S.A. 105, 20746-20751 (2008).
- Tiscornia, G., Singer, O., Verma, I. M. Production and purification of lentiviral vectors. Nat. Protoc. 1, 241-245 (2006).
- Ueda, H. R. A transcription factor response element for gene expression during circadian night. Nature. 418, 534-539 (2002).
- Zufferey, R., Donello, J. E., Trono, D., Hope, T. J. Woodchuck hepatitis virus posttranscriptional regulatory element enhances expression of transgenes delivered by retroviral vectors. J. Virol. 73, 2886-2892 (1999).
- Buhr, E. D., Yoo, S. H., Takahashi, J. S. Temperature as a universal resetting cue for mammalian circadian oscillators. Science. 330, 379-385 (2010).
- Balsalobre, A., Damiola, F., Schibler, . U.A serum shock induces circadian gene expression in mammalian tissue culture cells. Cell. 93, 929-937 (1998).
- Savelyev, S. A., Larsson, K. C., Johansson, A., Lundkvist, G. B. S. Slice Preparation, Organotypic Tissue Culturing and Luciferase Recording of Clock Gene Activity in the Suprachiasmatic Nucleus. J. Vis. Exp. (48), e2439 (2011).
- Akashi, M., Ichise, T., Mamine, T., Takumi, T. Molecular mechanism of cell-autonomous circadian gene expression of Period2, a crucial regulator of the mammalian circadian clock. Mol. Biol. Cell. 17, 555-565 (2006).
- Ohno, T., Onishi, Y., Ishida, N. A novel E4BP4 element drives circadian expression of mPeriod2. Nucleic Acids Res. 35, 648-655 (2007).
- Maier, B. A large-scale functional RNAi screen reveals a role for CK2 in the mammalian circadian clock. Genes Dev. 23, 708-718 (2009).
- Yoo, S. H. PERIOD2::LUCIFERASE real-time reporting of circadian dynamics reveals persistent circadian oscillations in mouse peripheral tissues. Proc. Natl. Acad. Sci. U.S.A. 101, 5339-5346 (2004).
- Liu, A. C., Lewis, W. G., Kay, S. A. Mammalian circadian signaling networks and therapeutic targets. Nat. Chem. Biol. 3, 630-639 (2007).
- Ko, C. H. Emergence of noise-induced oscillations in the central circadian pacemaker. PLoS Biol. 8, e1000513 (2010).
- Izumo, M., Johnson, C. H., Yamazaki, S. Circadian gene expression in mammalian fibroblasts revealed by real-time luminescence reporting: temperature compensation and damping. Proc. Natl. Acad. Sci. U.S.A. 100, 16089-16094 (2003).
- Izumo, M., Sato, T. R., Straume, M., Johnson, C. H. Quantitative analyses of circadian gene expression in mammalian cell cultures. PLoS Comput. Biol. 2, e136 (2006).
- Chen, Z. Identification of diverse modulators of central and peripheral circadian clocks by high-throughput chemical screening. Proc. Natl. Acad. Sci. U.S.A. 109, 101-106 (2011).
- Yamaguchi, S. Synchronization of cellular clocks in the suprachiasmatic nucleus. Science. 302, 1408-1412 (2003).
- Akashi, M., Hayasaka, N., Yamazaki, S., Node, K. Mitogen-activated protein kinase is a functional component of the autonomous circadian system in the suprachiasmatic nucleus. J. Neurosci. 28, 4619-4623 (2008).
- Hoshino, H., Nakajima, Y., Ohmiya, Y. Luciferase-YFP fusion tag with enhanced emission for single-cell luminescence imaging. Nat. Methods. 4, 637-639 (2007).
- Asai, M. Visualization of mPer1 transcription in vitro: NMDA induces a rapid phase shift of mPer1 gene in cultured SCN. Curr. Biol. 11, 1524-1527 (2001).
- Wilsbacher, L. D. Photic and circadian expression of luciferase in mPeriod1-luc transgenic mice in vivo. Proc. Natl. Acad. Sci. U.S.A. 99, 489-494 (2002).
- Yamazaki, S. Resetting central and peripheral circadian oscillators in transgenic rats. Science. 288, 682-685 (2000).
- Welsh, D. K., Noguchi, T., Yuste, R. Cellular bioluminescence imaging. Imaging: A Laboratory Manual. , 369-385 (2011).
- Nakajima, Y. Enhanced beetle luciferase for high-resolution bioluminescence imaging. PLoS One. 5, e10011 (2010).
- Guilding, C. A riot of rhythms: neuronal and glial circadian oscillators in the mediobasal hypothalamus. Mol. Brain. 2, 28 (2009).
- O'Neill, J. S. cAMP-dependent signaling as a core component of the mammalian circadian pacemaker. Science. 320, 949-953 (2008).
- Fuller, P. M., Lu, J., Saper, C. B. Differential rescue of light- and food-entrainable circadian rhythms. Science. 320, 1074-1077 (2008).
- Mukherjee, S. Knockdown of Clock in the ventral tegmental area through RNA interference results in a mixed state of mania and depression-like behavior. Biol. Psychiatry. 68, 503-511 (2010).
- Saijo, K. A Nurr1/CoREST pathway in microglia and astrocytes protects dopaminergic neurons from inflammation-induced death. Cell. 137, 47-59 (2009).
- Elias, G. M. Synapse-specific and developmentally regulated targeting of AMPA receptors by a family of MAGUK scaffolding proteins. Neuron. 52, 307-320 (2006).
- Isojima, Y. CKIepsilon/delta-dependent phosphorylation is a temperature-insensitive, period-determining process in the mammalian circadian clock. Proc. Natl. Acad. Sci. U.S.A. 106, 15744-15749 (2009).
- Bucan, M., Abel, T. The mouse: genetics meets behaviour. Nat. Rev. Genet. 3, 114-123 (2002).
- Hughes, M. E. Harmonics of circadian gene transcription in mammals. PLoS Genet. 5, e1000442 (2009).
- Atwood, A. Cell-autonomous circadian clock of hepatocytes drives rhythms in transcription and polyamine synthesis. Proc. Natl. Acad. Sci. U.S.A. 108, 18560-18565 (2011).
- Panda, S. Coordinated transcription of key pathways in the mouse by the circadian clock. Cell. 109, 307-320 (2002).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유