需要订阅 JoVE 才能查看此. 登录或开始免费试用。
Method Article
通过通过纳米孔可调纳米易位流速Zeta电位测定:利用DNA修饰颗粒为例
摘要
在这里,我们使用集成到一个电阻脉冲感测技术经由粒子易位速度的测量,这可以被用来确定各个纳米粒子的ζ电位来表征纳米颗粒表面化学的聚氨酯可调谐纳米孔。
摘要
纳米孔技术,统称为电阻脉冲传感器(RPS),被用来检测,量化和表征的蛋白质,分子和纳米颗粒。可调电阻脉冲感测(TRPS)是一个相对较新的适应RPS并入,可以实时地改变的可调孔径。这里,我们使用TRPS监测DNA改性的纳米粒子的易位倍,因为它们穿过可调孔膜作为DNA浓度和结构的一个功能( 即 ,单链到双链DNA)。
TRPS是基于两个的Ag / AgCl电极,通过它建立在所施加的电场产生稳定的离子电流的弹性体孔膜分离。与各种基于光学颗粒表征技术,TRPS可以表征单个颗粒之间的样本人口,允许能够轻松进行分析多式联运样品。这里,我们证明zeta电位测量通过已知的标准粒子易位速度和应用这些样品的分析物易位倍,从而导致测量这些分析物的Zeta电位。
以及获得的平均ζ电位的值,将样品全部采用的粒子由粒子立体显示通过样品人口分布的给定样品的详细信息,例如测量的。这样,这种方法演示了医疗和环境等领域的传感应用中的潜力。
引言
官能化的纳米颗粒变得如在医疗和环境领域的生物传感器日益流行。的能力,以改变纳米颗粒的表面化学,用DNA,例如,被证明是用于靶向药物递送系统1和监测DNA-蛋白质相互作用2-4是有用的。被利用在生物测定一个越来越普遍的纳米颗粒性质和在治疗剂的递送是超顺磁性5。超顺磁性粒子(SPP的)是在确定和从复杂混合物中去除特定分析物是非常有用的,可以用简单的使用单个磁体的这样做。一旦移除,所述分析物结合的颗粒可以被表征和分析适合目的。
用于纳米颗粒的检测和表征以前的方法包括光学技术,诸如动态光散射(DLS),或称为光子相关光谱法。虽然一喜GH可以通过技术,DLS被限定于一个平均为基础的技术,并没有加入专业软件分析多峰样品时,较大的颗粒将产生一个更主导信号,留下一些较小的颗粒完全被忽视6,7。粒子按颗粒表征技术,因此更利于分析和纳米功能化纳米粒子系统。
基于RPS技术基于周围施加电场到样品,并通过合成或生物纳米孔监测粒子的输送机构。根据RPS相对近期的纳米检测与表征技术是可调电阻脉冲感应(TRPS)8-16。 TRPS是由弹性,可调谐孔隙膜分开的两个电极系统。的可调孔径的方法允许在各种形状17和大小的分析物通过其反待测量通过毛孔口的机制。可调孔隙先前已经用于生产比较的结果,其他技术如透射电子光谱法(TEM)10检测的小颗粒(70-95纳米直径)。当施加电场时,离子电流被观察到并作为颗粒/分子穿过孔,它们暂时阻止孔径,导致在当前的减少,可以被定义为一个"封锁事件'。每个封锁事件是代表单个颗粒,使得样本内的每个颗粒可以表征单独基于所述封锁幅度,Δ 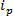 和全宽度半最大,FWHM,以及其他封锁性能。判断单个颗粒,因为他们通过纳米孔为多峰的样品有利的,因为TRPS可以成功地和有效区分的范围内的颗粒尺寸阿蒙GST一个样本。可调电阻脉冲感测完成大小10,同时ζ电位12,18和浓度15测量在单次运行,因此,仍可以区分的类似的样品,如果不通过表面电荷19相同的大小;在施胶替代技术的优势。
和全宽度半最大,FWHM,以及其他封锁性能。判断单个颗粒,因为他们通过纳米孔为多峰的样品有利的,因为TRPS可以成功地和有效区分的范围内的颗粒尺寸阿蒙GST一个样本。可调电阻脉冲感测完成大小10,同时ζ电位12,18和浓度15测量在单次运行,因此,仍可以区分的类似的样品,如果不通过表面电荷19相同的大小;在施胶替代技术的优势。
ζ电位是指在剪切20的平面上的静电势,并作为它们穿过的孔19从粒子速度计算。单个颗粒的ζ电位测量值从而给出洞察易位机制和在溶液中的纳米颗粒系统中,纳米颗粒测定设计未来的有价值的信息用于一系列应用的行为。粒子通过粒子这种性质的分析,还允许之间的样本人群中蔓延,zeta电位值的分布加以探讨,使邻更多信息要达到n个反应动力学(单链到双链DNA,例如)和颗粒稳定性在溶液中。
在这里,我们描述了检测和表征未修饰和DNA修饰SPP表面的技术。本文描述的协议是适用于一系列无机和生物纳米颗粒,但我们证明用DNA修饰表面的方法,由于其广泛的应用。该技术允许用户在纳米颗粒表面的单链和双链DNA靶区分基础上,通过一个孔体系粒子易位速度和因此它们的ζ电势。
研究方案
1.用吐温20(PBST)缓冲使磷酸盐缓冲液
- 溶解1的PBS片剂(0.01M磷酸盐缓冲液,0.0027 1M氢氯化物,0.137氯化钠,pH值7.4)在200毫升的去离子水(18.2兆欧厘米)。
- 加入100微升(0.05(V / V)%)吐温20至200毫升缓冲液溶液作为表面活性剂。
2.准备羧基聚苯乙烯颗粒标准
- 涡旋超声2分钟前30秒的校准颗粒在80瓦到创建粒子的单分散性。
- 稀释校准颗粒在100 1至在PBST缓冲液和涡旋1×10 10个粒子/ ml的浓度为30秒。
3.准备链亲和素包覆颗粒
- 涡旋超声2分钟前30秒的颗粒在80瓦的单分散性保证。
- 稀释链霉包衣颗粒1 100在PBST缓冲到交流hieve的1×10 9个粒子/ ml,30秒所得浓度和旋涡。
注:一个典型的样本量为200微升。例如,如果调查不同的DNA的浓度制备1ml适当稀释的链霉亲涂覆颗粒。
4.寡核苷酸的制备
- 重构用去离子水的寡核苷酸到所得100μM的浓度。
5.加成捕获探针(CP)的DNA的链亲和素包被的颗粒
- 之前DNA结合,涡流链霉包衣颗粒(200微升样品体积)30秒,随后通过一个2分钟超声处理在80瓦。
- 基于由供应商(4352皮摩尔/毫克)所提供的结合能力,加DNA的适当的浓度在颗粒为10,20,30,40,47,95,140,和210纳米的DNA得到的浓度。
- 涡旋10秒并置于样品上在室温下在旋转轮30分钟以允许将DNA通过链霉抗生物素相互作用结合到粒子表面。
- 一旦捕获的DNA已被添加,并与链霉亲包衣颗粒孵育,通过将试样放在一个磁架30分钟除去在经由磁分离溶液中的过量的DNA。
- 除去上清液,注意不要扰乱最接近磁铁粒子的新形成的群集,并用相同体积的新的PBST缓冲液的更换。
6.杂交的互补DNA到CP颗粒
- 添加所需的靶DNA(超过500纳米)的量,以确保最大可能的靶结合为止。
- 涡旋样品10秒和地点上在室温下在旋转轮30分钟。
- 一旦杂交完成后,通过将样品上的磁架30分钟除去通过磁分离过量的靶DNA。
- 除去上清液,注意不要扰乱最接近磁铁粒子的新形成的群集,并用相同体积的新的PBST缓冲液的更换。
- 重复步骤重复样本6.1至6.4,并放置这些样品,在旋转轮在室温下16小时,以研究DNA杂交时间。
7. TRPS设置
- 插入仪器到计算机系统中以代替软件。
- 校准用卡尺的初始拉伸。
- 测量两个平行夹爪的外侧之间的距离。
- 输入通过在"仪器设置"选项卡中输入在"拉伸"字段拉伸,然后点击"校准舒展"的标签下的软件。
- 横向适合分析适当大小的聚氨酯纳米孔膜上朝上纳米孔ID号钳口。然后,伸展钳口用于使用分析所需的拉伸在仪器的一侧伸展调节手柄。伸展43和48毫米的下颌。
注意:使校准微粒封锁是至少0.3 nA的尺寸是一起施加电压所确定的拉伸的精确值。拉伸已经输入到软件在步骤7.2和作为钳口被拉伸会自动调整。 - 放置80微升PBST缓冲液中的下部流体细胞,纳米孔的下方,以确保不存在本气泡可能影响测量。如果有气泡看出,取出并更换缓冲。
- 点击上方的流体细胞到位,并把40微升缓冲进去,从而也确保没有泡沫存在。如果气泡存在于上部流体细胞,通过更换液体移除。
- 当可再现的基线电流已从用缓冲液更换上部流体细胞达到,通过点击"ST添加40微升样品到上部流体细胞和测量的"中的"数据采集"软件屏幕上标签的艺术。
注:数据采集完成后,在50千赫的0.05呐封锁幅度下限的频率,虽然可以使用通过"分析数据"选项卡中的软件进行更改(在"分析设置"和"电阻式封锁") 。 - 放置一个法拉第笼在流体电池系统的顶部,以减少对测量电背景噪声。
- 使用一个可变压力模块(VPM)来的压力或真空施加到样品。
- 施加的外部压力,喷嘴连接到该上部流体细胞,然后旋转压力臂和卡入到位(取决于是否将被施加正压力(PRE)或真空(VAC))。
- 在使用位于VPM顶部的压力级旋钮'厘米'或'毫米'刻度施加压力。按下旋钮,适用于"厘米"的规模压力,向上拉Ťo在"毫米"规模施加压力。
8.准备的样品分析TRPS
- 涡旋样品在TRPS分析前80瓦特30秒,超声2分钟。
9.校准纳米孔的泽塔分析
- 将40微升校准颗粒(1×10粒/ ml)倒入上层液体电池后,完成TRPS测量(设置为在第7)在3施加的电压。 " - "按钮上的电压规模在"仪器设置"中的软件选项卡中点击"+"和改变电压。
- 检查3电压回到约140,110背景电流和80 nA的。保证在中等电压的校准微粒产生至少0.3的平均阻滞大小nA的。
- 施加压力,使所述校准微粒的平均半峰全宽(FWHM)的持续时间至少是0.15毫秒。为此使用连接到可变压力模块的压力臂手动。选择压力(PRE)或真空(VAC),直到它卡在期望的位置,并且在步骤7.8.2所适用相应以下设置指示所述臂旋转。一旦这些条件已经实现,通过点击"数据采集"选项卡中的软件'开始'开始运行。
- 当至少有500个颗粒进行了测量(见"粒子计数"在软件屏幕在测量过程中底部),运行已超过30秒,由"数据采集"标签按下"停止"完成运行(参见"运行时间'也朝向屏幕的底部)。
- 通过描述每一种新的纳米孔引入时间或分析每一个新的一天完成步骤9.1-9.4完成校准运行校准系统。
10.运行样品
- 运行在最高的样品或第二高电压作为确保类似的(±10 NA)的校准样品,如果不相同,基线电流。
- 一旦适当的基准电流来实现,用40微升样品的替换在上部流体单元中的电解质。当引入的样品,封锁将看到的信号路径。开始通过点击"开始"中的"数据采集"选项卡中运行样品并记录最低500粒子(检查'粒子计数"信号走线下位于),并确保运行时间最少30秒(见"运行时代"也位于信号迹线下方)。
- 要完成测量,点击"停止"中的"数据采集卡"并保存数据文件。
- 要保存文件,输入如下格式的文件信息; '调查'是文件将保存在该文件夹,"纳米孔ID'正在被使用,'部分#'我毛孔的序列号š孔的类型( 即 ,NP150 / NP200),'样品ID'是样品的名字,"校准或样品"的信息是否是一个校准或测量样品,"稀释",如果样品稀释被使用(键入100如果样品稀释100倍),"压力"是所施加的压力到样品(以厘米 - 见第7.8节),'电解质ID"是的缓冲样品中由姓名,和'注意事项'是有关样本或运行任何个人笔记。
- 每个样品运行之间,通过将40微升的PBST缓冲液到上部流体细胞几次并施加各种压力(在-10通常,-5厘米(真空),以及5和10厘米(正压力)),直到洗系统没有更多的阻断事件都存在,确保有残留在系统中没有残留的粒子和样品之间,因此没有交叉污染。在这种洗涤步骤每个重复样品之间完成一式三份样品运行身份运行WELl由于不同样本之间。
结果
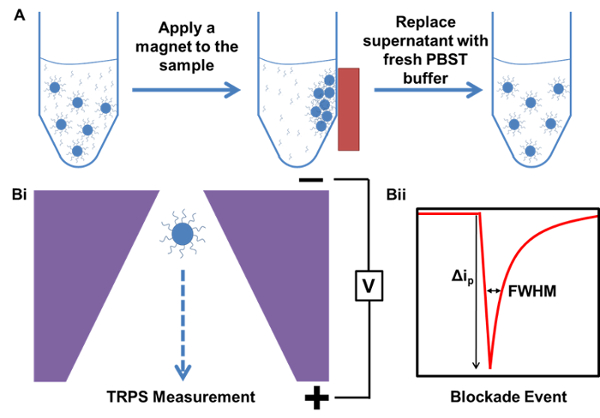
图1. 磁性纯化和TRPS测量过程的示意图。 A)的开始与含有样品过量,未结合的捕获探针的DNA。B)的样本的磁性纯化的实施例的TRP测定例ⅰ)粒子穿过纳米孔和ii)从在孔粒子暂时阻塞离子封锁事件产生使暂时降低在目前的;从被用来计算粒子易位速度的信息。
讨论
为ζ电势用于计算由Arjmandi 等人 21的工作有关的校准基础的方法。因为它们穿过纳米孔被测量作为所施加电压的函数,使用平均电场和粒子速度定期锥形孔的整体上颗粒的易位持续时间。的电泳迁移率是1 / T(其中T是封锁持续时间)相对于电压的导数,乘以感应区长度的平方, 湖在通过传感区域的多个基准点的平均速度测量以允许最小的误差在使用这种方法计算ζ电位。...
披露声明
ELCJB由IZON科技有限公司的支持
致谢
作者感谢IZON科技有限公司的支持。这项工作是由欧洲委员会研究(PCIG11-GA-2012-321836 Nano4Bio)的支持。
材料
| Name | Company | Catalog Number | Comments |
| Phosphate buffered Saline (PBS) | Sigma Aldrich, UK | P4417 | 1 tablet dissolved in 200 ml deionized water to make buffer solution. |
| Tween-20 | Sigma Aldrich, UK | P1379 | 0.05% (v/v) in PBS buffer as a surfactant |
| Carboxyl polystyrene nanoparticles | Bangs Laboratories, US | CPC200 | Nominal diamter of 220 nm, raw concentration of 1 x 1012 particles/ml, specific surface charge of 86 µeq/g (equivalent to a surface charge density of 3.2 x 1019 C/nm2. |
| Streptavidin coated nanoparticles | Ademtech, France | 3121 | Batch had binding capacity of 4,352 pmol/mg (188 nM theoretical DNA binding capacity) at a raw concentration of 1.1 x 1011 particles/ml. |
| Biotinylated oligonucleotides | Sigma Aldrich, UK | VC00001 | Supplier spec: Reverse Phase 1 purification (0.05 Scale); Biotin modification at 3' end; Lyophilized powders reconstituted to 100 µM using deionized water, and diluted as required. Sequences: CP 5'ATGGTTAAACCTCACTAC GCGTGGC[Btn]3' |
| Standard olignonucleotides | Sigma Aldrich, UK | VC00001 | Supplier spec: Reverse Phase 1 purification (0.05 Scale); Lyophilized powders reconstituted to 100 µM using deionized water, and diluted as required. Sequences of DNA targets: Fully complementary - 5'GCCACGCGTAGTGA GGTTTAACCAT3', Middle binding - 5'GTAGTGAGGT3', End binding - 5'GTTTAACCAT3', Partially complementary overhanging - 5'GTGAGGTTTAACCAT TTTTTTTTTTTTTTT3'. |
| Izon qNano | Izon Science, NZ | Inherent pressure on system of 47 Pa | |
| Izon Variable Pressure Module (VPM) | Izon Science, NZ | Each 'cm' of pressure is equivalent to approximately 100 Pa. | |
| Polyurethane nanopore membranes | Izon Science, NZ | NP150 | Analyte size range 60-480 nm, pore diameter of calculated to be 799 nm at a 45 mm stretch. |
| Magrack 6 | GE Healthcare, UK | 28-9489-64 | |
| Sonic Bath | Fisher Scientific, UK | 10692353 | 80 Watts |
| Vortexer | IKA, Germany | 0003365000 | |
| Rotary Wheel | Labnet International, US | H5500-230 V |
参考文献
- Alexander, C. M., Maye, M. M., Dabrowiak, J. C. DNA-capped nanoparticles designed for doxorubicin drug delivery. Chem Commun. 47 (12), 3418-3420 (2011).
- Billinge, E. R., Platt, M. Aptamer based dispersion assay using tunable resistive pulse sensing (TRPS). Anal Methods. 7 (20), 8534-8538 (2015).
- Bulyk, M. L. Protein Binding Microarrays for the Characterization of Protein-DNA Interactions. Adv Biochem Eng Biotechnol. 104, 65-85 (2007).
- Platt, M., Rowe, W., Knowles, J., Day, P. J., Kell, D. B. Analysis of aptamer sequence activity relationships. Integr Biol. 1 (1), 116-122 (2009).
- Ruiz-Hernández, E., Baeza, A., Vallet-Regí, M. Smart Drug Delivery through DNA/Magnetic Nanoparticle Gates. ACS Nano. 5 (2), 1259-1266 (2011).
- Murdock, R. C., Braydich-stolle, L., Schrand, A. M., Schlager, J. J., Hussain, S. M. Characterization of Nanomaterial Dispersion in Solution Prior to In Vitro Exposure Using Dynamic Light Scattering Technique. Toxicol Sci. 101 (2), 239-253 (2008).
- Hupfield, S., Holsaeter, A. M., Skar, M., Frantzen, C. B., Brandl, M. Liposome size analysis by dynamic/static light scattering upon size exclusion-/field flow fractionation. J Nanosci Nanotechnol. 6 (7), 3025-3031 (2006).
- Roberts, G. S., et al. Tunable pores for measuring concentrations of synthetic and biological nanoparticle dispersions. Biosens Bioelectron. 31 (1), 17-25 (2012).
- Roberts, G. S., Kozak, D., Anderson, W., Broom, M. F., Vogel, R., Trau, M. Tunable nano/micropores for particle detection and discrimination: scanning ion occlusion spectroscopy. Small. 6 (23), 2653-2658 (2010).
- Vogel, R., et al. Quantitative sizing of nano/microparticles with a tunable elastomeric pore sensor. Anal Chem. 83 (9), 3499-3506 (2011).
- Booth, M. A., Vogel, R., Curran, J. M., Harbison, S., Travas-Sejdic, J. Detection of target-probe oligonucleotide hybridization using synthetic nanopore resistive pulse sensing. Biosens Bioelectron. 45, 136-140 (2013).
- Kozak, D., Anderson, W., Vogel, R., Chen, S. Simultaneous size and ζ-potential measurements of individual nanoparticles in dispersion using size-tunable pore sensors. ACS Nano. 6 (8), 6990-6997 (2012).
- Kozak, D., Anderson, W., Vogel, R., Trau, M. Advances in Resistive Pulse Sensors: Devices bridging the void between molecular and microscopic detection. Nano Today. 6 (5), 531-545 (2011).
- Weatherall, E., Willmott, G. R. Applications of tunable resistive pulse sensing. Analyst. 140, 3318-3334 (2015).
- Willmott, G. R., et al. Use of tunable nanopore blockade rates to investigate colloidal dispersions. J Phys Condens Matter. 22 (45), 454116 (2010).
- Blundell, E. L. C. J., Mayne, L. J., Billinge, E. R., Platt, M. Emergence of tunable resistive pulse sensing as a biosensor. Anal Methods. 7, 7055-7066 (2015).
- Platt, M., Willmott, G. R., Lee, G. U. Resistive Pulse Sensing of Analyte-Induced Multicomponent Rod Aggregation Using Tunable Pores. Small. 8 (15), 2436-2444 (2012).
- Vogel, R., Anderson, W., Eldridge, J., Glossop, B., Willmott, G. A variable pressure method for characterizing nanoparticle surface charge using pore sensors. Anal Chem. 84 (7), 3125-3131 (2012).
- Blundell, E. L. C. J., Vogel, R., Platt, M. Particle-by-Particle Charge Analysis of DNA-Modified Nanoparticles Using Tunable Resistive Pulse Sensing. Langmuir. 32 (4), (2016).
- Hunter, R. J. . Zeta Potential in Colloid Science: Principles and Applications. , (1981).
- Arjmandi, N., Van Roy, W., Lagae, L., Borghs, G. Measuring the electric charge and zeta potential of nanometer-sized objects using pyramidal-shaped nanopores. Anal Chem. 84 (20), 8490-8496 (2012).
- Bacri, L., et al. Dynamics of colloids in single solid-state nanopores. J Phys Chem B. 115 (12), 2890-2898 (2011).
- Cabello-Aguilar, S., et al. Dynamics of polymer nanoparticles through a single artificial nanopore with a high-aspect-ratio. Soft Matter. 10 (42), 8413-8419 (2014).
- Billinge, E. R., Muzard, J., Platt, M. Tunable resistive pulse sensing as a tool to monitor analyte induced particle aggregation. Nanomater Nanosci. 1 (1), 11 (2013).
- Li, J., Fan, C., Pei, H., Shi, J., Huang, Q. Smart Drug Delivery Nanocarriers with Self-Assembled DNA Nanostructures. Adv Mater. 25 (32), 4386-4396 (2013).
- Billinge, E. R., Broom, M., Platt, M. Monitoring aptamer-protein interactions using tunable resistive pulse sensing. Anal Chem. 86 (2), 1030-1037 (2014).
- Gold, L., et al. Aptamer-Based Multiplexed Proteomic Technology for Biomarker Discovery. PLoS One. 5 (12), e15004 (2010).
- Park, S. -. J., Taton, T. A., Mirkin, C. A. Array-Based Electrical Detection of DNA with Nanoparticle Probes. Science. 295 (5559), 1503-1506 (2002).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。