È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Determinazione della Zeta Potenziale tramite nanoparticelle Traslocazione Le velocità attraverso una Tunable Nanopore: usando le particelle di DNA modificato come esempio
In questo articolo
Riepilogo
Qui usiamo un nanoporo sintonizzabile poliuretano integrato in una tecnica di rilevamento di impulsi resistivo caratterizzare nanoparticelle chimica di superficie tramite la misura della velocità di traslocazione delle particelle, che può essere utilizzato per determinare il potenziale zeta delle singole nanoparticelle.
Abstract
tecnologie nanoporo, noti collettivamente come sensori resistivi Pulse (RPS), vengono utilizzati per rilevare, quantificare e caratterizzare le proteine, molecole e nanoparticelle. sensing sintonizzabile impulso resistivo (TRPS) è relativamente recente adattamento di RPS che incorpora un poro sintonizzabile che può essere alterato in tempo reale. Qui, usiamo TRPS di monitorare i tempi di traslocazione di nanoparticelle DNA modificato, e attraversa la membrana pori sintonizzabile in funzione della concentrazione di DNA e struttura (cioè, a singolo filamento di DNA a doppio filamento).
TRPS si basa su due elettrodi Ag / AgCl, separate da una membrana elastomerica pori che stabilisce una corrente ionica stabile su un campo elettrico applicato. A differenza di varie tecnologie di caratterizzazione delle particelle ottiche a base, TRPS possono caratterizzare le singole particelle, tra un campione di popolazione, permettendo per i campioni multimodali da analizzare con facilità. Qui, dimostriamo misurazioni potenziale zetatramite velocità traslocazione delle particelle di standard noti e applicarle per assaggiare i tempi traslocazione analiti, con il risultato di misura del potenziale zeta di questi analiti.
Oltre ad acquisire valori di potenziale zeta medi, i campioni sono tutti misurati utilizzando una prospettiva particelle-by-particella esibendo ulteriori informazioni su un determinato campione attraverso distribuzioni di popolazione campione, per esempio. Di conseguenza, il metodo illustrato potenziale all'interno applicazioni di rilevamento per entrambi i campi medico e ambientale.
Introduzione
nanoparticelle funzionalizzate stanno diventando sempre più popolare come biosensori in entrambi i campi medico e ambientale. La capacità di alterare la chimica di superficie di una nanoparticella, con il DNA, per esempio, si sta rivelando utile per i sistemi di somministrazione di farmaci mirati 1 e monitoraggio interazioni DNA-proteina 2-4. Una proprietà di nanoparticelle sempre più comune di essere utilizzato nel test biologici e nella consegna di terapie è superparamagnetismo 5. particelle superparamagnetiche (SPP) sono estremamente utili per identificare e rimuovere analiti specifici da miscele complesse e possono farlo con il semplice uso di un unico magnete. Una volta rimosse, le particelle di analiti-bound possono essere caratterizzate e analizzati adatto allo scopo.
I metodi precedenti utilizzati per la rilevazione e la caratterizzazione di nanoparticelle comprendono tecniche ottiche quali light scattering dinamico (DLS), altrimenti noti come la spettroscopia di correlazione di fotoni. Anche se un hitecnica di throughput gh, DLS si limita ad essere una tecnica basata compensazione e nell'analisi di campioni multimodali senza l'aggiunta di software specializzato, le particelle più grandi produrranno un segnale molto più dominante, lasciando alcune delle particelle più piccole completamente inosservato 6,7. Particle-by-particella tecniche di caratterizzazione sono quindi molto più favorevole per analizzare i sistemi di nanoparticelle funzionalizzati nanoparticelle e.
tecnologie basate RPS sono basati su applicazione di un campo elettrico per un campione e monitorare il meccanismo di trasporto delle particelle attraverso un nanoporo sintetico o biologico. Un relativamente recente tecnica di rilevazione e la caratterizzazione di nanoparticelle sulla base di RPS è sintonizzabile resistivo impulso di rilevamento (TRPS) 8-16. TRPS è un sistema a due elettrodi separati da una membrana elastomerica pori sintonizzabile. Metodo pori sintonizzabile permette di analiti di una serie di forma 17 e la dimensione deve essere misurata tramite il loro transmeccanismi di porta attraverso il poro. Pori sintonizzabili sono stati precedentemente utilizzati per la rilevazione di piccole particelle (70-95 nm di diametro) producendo risultati paragonabili a altre tecniche come la spettroscopia elettronica a trasmissione (TEM) 10. Quando un campo elettrico viene applicato, una corrente ionica viene osservata e come particelle / molecole passano attraverso il poro, essi bloccare temporaneamente il poro, provocando una riduzione della corrente che può essere definita come un 'evento blocco'. Ogni evento blocco è rappresentativo di una singola particella in modo che ogni particella di un campione può essere caratterizzato individualmente in base alla grandezza blocco, Δ 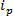 E larghezza a metà massimo FWHM, così come altre proprietà del blocco. Analizzando le singole particelle che passano attraverso un nanoporo è vantaggioso per i campioni multimodali come TRPS grado di distinguere correttamente ed efficacemente una gamma di dimensioni delle particelle amonGST un singolo campione. Sintonizzabile rilevamento impulso resistivo completa dimensione 10, potenziale zeta 12,18 e la concentrazione di 15 misurazioni simultaneamente in un unico passaggio e quindi può ancora distinguere campioni di simile, se non la stessa dimensione con la loro carica di superficie 19; un vantaggio rispetto alle tecniche dimensionali alternative.
E larghezza a metà massimo FWHM, così come altre proprietà del blocco. Analizzando le singole particelle che passano attraverso un nanoporo è vantaggioso per i campioni multimodali come TRPS grado di distinguere correttamente ed efficacemente una gamma di dimensioni delle particelle amonGST un singolo campione. Sintonizzabile rilevamento impulso resistivo completa dimensione 10, potenziale zeta 12,18 e la concentrazione di 15 misurazioni simultaneamente in un unico passaggio e quindi può ancora distinguere campioni di simile, se non la stessa dimensione con la loro carica di superficie 19; un vantaggio rispetto alle tecniche dimensionali alternative.
Potenziale Zeta è definito come il potenziale elettrostatico in corrispondenza del piano di taglio 20, e viene calcolato le velocità delle particelle, e attraversa un poro 19. Zeta potenziali misure delle singole particelle dà quindi informazioni sui meccanismi di traslocazione e il comportamento dei sistemi di nanoparticelle in soluzione, informazioni preziose per il futuro dei disegni di analisi nanoparticelle per una gamma di applicazioni. l'analisi delle particelle-by-particella di natura tale permette anche per la diffusione e la distribuzione dei valori potenziale zeta tra un campione di popolazione da esplorare, consentendo per ulteriori informazioni ocinetica di reazione n (single-stranded di DNA a doppia elica, per esempio) e stabilità di particelle in soluzione da raggiungere.
Qui, descriviamo una tecnica che individua e caratterizza entrambe le superfici modificate e DNA modificato spp. Il protocollo qui descritto è applicabile ad una gamma di nanoparticelle inorganiche e biologiche, ma dimostrano la procedura utilizzando superfici DNA modificato per la loro vasta gamma di applicazioni. La tecnica consente all'utente di distinguere tra obiettivi a filamento singolo e doppio filamento di DNA su una superficie di nanoparticelle, sulla base delle velocità di traslocazione particelle attraverso un sistema di pori e quindi i loro potenziali zeta.
Protocollo
1. Rendere il tampone fosfato con Tween-20 (PBST) Buffer
- Sciogliere un PBS tablet (0,01 tampone M di fosfato, 0,0027 M cloruro di potassio, 0,137 M di sodio cloruro, pH 7,4) in 200 ml di acqua deionizzata (18,2 MW cm).
- Aggiungere 100 pl (0.05 (v / v)%) Tween-20 per la soluzione di 200 ml di buffer come un tensioattivo.
2. Preparazione La particella Standards Carboxyl polistirolo
- Agitare le particelle di calibrazione per 30 sec prima sonicazione per 2 min a 80 watt per creare monodispersity delle particelle.
- Diluire le particelle di calibrazione 1 in 100 ad una concentrazione di 1x10 10 particelle / ml in tampone PBST e vortex per 30 sec.
3. Preparazione particelle rivestite di streptavidina
- Agitare le particelle per 30 sec prima sonicazione per 2 min a 80 watt per garantire monodispersity.
- Diluire le ricoperto streptavidina particelle 1 su 100 in PBST buffer per achieve una concentrazione risultante di 1x10 9 particelle / ml e vortex per 30 sec.
Nota: Un volume tipico del campione è di 200 ml. Ad esempio, se indagare cinque concentrazioni di DNA preparare 1 ml di particelle rivestite con streptavidina diluiti.
4. Preparazione di oligonucleotidi
- Ricostituire oligonucleotidi con acqua deionizzata ad una conseguente concentrazione di 100 pM.
5. L'aggiunta di sonda DNA Capture (CP) alle particelle rivestite di streptavidina
- Prima di legame al DNA, vortex i rivestito streptavidina particelle (volume di 200 ml del campione) per 30 secondi seguiti da 2 minuti a ultrasuoni a 80 watt.
- Sulla base della capacità di legame fornito dal fornitore (4.352 pmol / mg), aggiungere la concentrazione appropriata di DNA alle particelle per risultanti concentrazioni di 10, 20, 30, 40, 47, 95, 140, e 210 nM DNA.
- Vortice i campioni per 10 sec e posto su una ruota girevole a temperatura ambienteper 30 minuti per consentire il DNA di legarsi alle superfici delle particelle tramite un'interazione streptavidina-biotina.
- Una volta che il DNA di cattura è stato aggiunto e incubato con le particelle rivestite di streptavidina, rimuovere l'eccesso DNA in soluzione tramite separazione magnetica ponendo i campioni su un sostegno magnetico per 30 min.
- Rimuovere il surnatante, facendo attenzione a non disturbare il cluster di nuova formazione di particelle più vicini al magnete, e sostituirlo con lo stesso volume di nuovo buffer PBST.
6. Ibridazione DNA complementare al CP-particelle
- Aggiungere la quantità di DNA bersaglio (superiori a 500 nM) per garantire la massima possibile target vincolante è stato raggiunto.
- Vortex i campioni per 10 sec e posto su una ruota rotante a temperatura ambiente per 30 min.
- Dopo l'ibridazione, rimuovere il DNA bersaglio eccesso tramite separazione magnetica ponendo i campioni su un sostegno magnetico per 30 min.
- Rimuovere il surnatante, Facendo attenzione a non disturbare il cluster di nuova formazione di particelle più vicini al magnete, e sostituirlo con lo stesso volume di nuovo buffer PBST.
- Ripetere i punti da 6.1 a 6.4 per i campioni duplicati e mettere questi campioni su una ruota girevole a temperatura ambiente per 16 ore per studiare i tempi di ibridazione del DNA.
Setup 7. TRPS
- Collegare lo strumento in un sistema di computer con software a posto.
- Calibrare il tratto iniziale utilizzando una pinza.
- Misurare la distanza tra l'esterno di due ganasce parallele.
- Ingresso nel software digitando il tratto nel campo 'stretch' nella scheda 'Instrument Impostazioni' e cliccando su 'Calibra tratto' sotto la scheda.
- Lateralmente montare una membrana poliuretanica nanoporo di adeguato dimensionamento per l'analisi sulle mascelle con il numero ID nanoporo rivolto verso l'alto. Poi, allungare le ganasce per il tratto necessario per l'analisi utilizzando lamanopola di regolazione tratto sul lato dello strumento. Allungare le ganasce tra i 43 ei 48 mm.
Nota: Il valore esatto del tratto è determinato a fianco di tensione applicata in modo che i blocchi di particelle di taratura sono almeno 0,3 nA in termini di dimensioni. Il tratto è già immesso nel software nel passo 7.2 e regola automaticamente le ganasce sono allungati. - Mettere 80 ml di tampone PBST nella cella inferiore fluida, sotto il nanoporo, garantire che vi siano bolle presenti che possono influenzare la misura. Se ci sono bolle visibili, rimuovere e sostituire il buffer.
- Fare clic sulla cella fluido superiore in posizione e mettere 40 ml di tampone in esso, ancora una volta garantire che vi siano bolle presenti. Se le bolle sono presenti nella cellula fluido superiore, rimuoverli sostituendo il liquido.
- Quando una corrente di riferimento riproducibile è stato raggiunto dalla sostituzione della cella di fluido superiore con tampone, aggiungere 40 ml di campione alla cella fluido superiore e misura cliccando 'stl'arte 'in la' scheda di acquisizione dati 'sullo schermo del software.
Nota: L'acquisizione dei dati è stata completata ad una frequenza di 50 kHz con il limite di un blocco grandezza inferiore di 0,05 nA, anche se questo può essere modificato tramite il software tramite la scheda 'Analyse dati' (sotto 'impostazioni di analisi' e 'resistivo Blocchi') . - Inserire una gabbia di Faraday sopra la parte superiore del sistema cellulare di liquidi per ridurre il rumore di fondo elettrico sulle misurazioni.
- Utilizzare un modulo di pressione variabile (VPM) per applicare una pressione o vuoto per i campioni.
- Per applicare una pressione esterna collegare l'ugello alla cella di fluido superiore, poi ruotare il braccio di pressione e agganciarla (a seconda che verranno applicate una pressione positiva (PRE) o vuoto (VAC)).
- Applicare pressione in un 'cm' o scala 'mm' utilizzando la manopola stadio di pressione situata sulla sommità della VPM. Premere la manopola verso il basso per fare pressione sulla scala 'cm' e tirarlo verso l'alto to applicare una pressione sulla scala 'mm'.
8. preparazione dei campioni per l'analisi TRPS
- campioni Vortex per 30 secondi e ultrasuoni per 2 min a 80 watt prima dell'analisi TRPS.
9. Calibrazione del nanoporo per l'analisi Zeta
- Dopo aver posizionato 40 particelle di calibrazione microlitri (1x10 10 particelle / ml) nella cella fluido superiore, completare una misurazione TRPS (messa a punto come nella sezione 7) a 3 tensioni applicate. Modificare la tensione facendo clic sul '+' e '-' pulsanti sulla scala di tensione nella scheda 'Instrument Settings' sul software.
- Verificare che le 3 tensioni tornano correnti di circa 140, 110 di fondo, e 80 nA. Assicurarsi che alla tensione media delle particelle di calibrazione producono una magnitudine media blocco di almeno 0,3 nA.
- Applicare una pressione in modo che la media ampiezza metà massimo (FWHM) durate delle particelle di taratura sono almeno0.15 msec. Fare questo manualmente utilizzando il braccio di pressione collegato al modulo di pressione variabile. Selezionare pressione (PRE) o vuoto (VAC) ruotando il braccio fino a farlo scattare nella posizione desiderata e applicano di conseguenza seguente set up istruzioni al punto 7.8.2. Una volta che queste condizioni sono stati raggiunti, inizia la corsa facendo clic su 'Start' sul software nella scheda 'acquisizione dati'.
- Completa la corsa premendo il tasto 'stop' nella scheda 'Data Acquisition' quando almeno 500 particelle sono state misurate (vedi 'Conte di particelle' nella parte inferiore della schermata del software durante la misura) e la pista ha superato 30 sec (vedere ' Run Time 'anche verso la parte inferiore dello schermo).
- Calibrare il sistema completando una corsa di taratura come descritto ogni volta che viene introdotto un nuovo nanoporo o per ogni nuovo giorno di analisi completando passo 9,1-9,4.
10. L'esecuzione di un campione
- Eseguire i campioni al più alto osecondo più alto a tensione di campioni di calibrazione a garantire un simile (± 10 nA), se non la stessa corrente di base.
- Una volta che la corrente di base del caso si ottiene, sostituire l'elettrolita nella cella di fluido superiore con 40 ml di campione. Quando viene introdotto un campione, i blocchi saranno visibili sulla traccia del segnale. Avviare il campione di eseguire cliccando su 'Start' nella scheda 'acquisizione dati' e registrare un minimo di 500 particelle (controllare 'Conte di particelle' situata sotto la traccia del segnale) e di garantire il tempo di esecuzione è un minimo di 30 secondi (vedere 'Run tempo 'situato anche sotto la traccia del segnale).
- Per completare la misurazione, cliccare su 'stop' in la 'scheda di acquisizione dati' e salvare il file di dati.
- Per salvare il file, inserire le informazioni sul file nel seguente formato; 'Indagine' è la cartella il file verrà salvato in, 'Nanopore ID' è il numero di serie del poro in uso, 'Parte #' is il tipo di pori (cioè, NP150 / NP200), 'ID campione' è il nome del campione, 'calibrazione o campione' dettagli se si tratta di una misura di calibrazione o campione, viene utilizzato 'diluizione' se il campione è stato diluito ( digitare 100 se il campione è stato diluito 100 volte), 'Pressure' è la pressione applicata al campione (in cm - vedere paragrafo 7.8), 'elettrolito ID' è il nome del tampone campione è costituito, e ' Note 'sono delle note personali sul campione o correre.
- Tra ogni corsa del campione, lavare il sistema, ponendo 40 ml di PBST tampone nella cella fluido superiore diverse volte e l'applicazione di varie pressioni (di solito a -10, -5 cm (a vuoto), e 5 e 10 cm (pressione positiva)) fino a altri eventi del blocco sono presenti, garantendo l'assenza di particelle residue rimangono nel sistema e quindi nessuna contaminazione incrociata tra i campioni. campioni analizzato in triplicato con questa fase di lavaggio completato tra ogni campione di ripetizione corrono come well come tra i diversi campioni.
Risultati
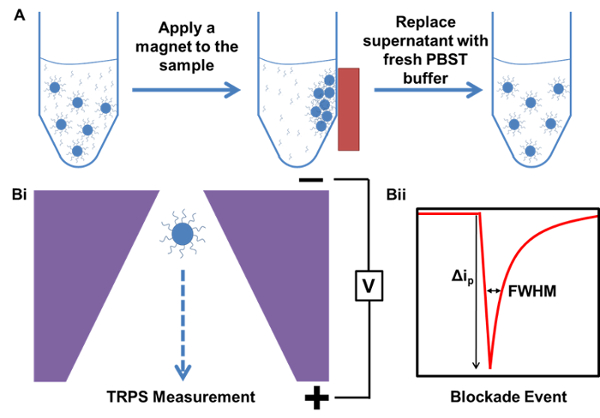
Figura 1. Rappresentazione schematica dei processi di purificazione magnetica e una misurazione TRPS. A) Esempio di purificazione magnetica del campione a partire da un campione contenente eccesso, non legato DNA sonda di cattura. B) TrPs esempio di misura i) di particelle che passa attraverso il nanoporo e ii) evento blocco prodotta da particelle occlusione temporan...
Discussione
Il calcolo per il potenziale zeta ha utilizzato un metodo basato calibrazione relativi al lavoro in Arjmandi et al. 21. La durata della traslocazione di particelle, e attraversa un nanoporo viene misurata in funzione della tensione applicata, utilizzando un campo e particella media velocità elettrici sulla totalità di un poro conica regolare. La mobilità elettroforetica è la derivata di 1 / T (dove T è la durata blocco) rispetto alla tensione, moltiplicata per il quadrato della lunghezza d...
Divulgazioni
ELCJB è supportato da Izon Science Ltd.
Riconoscimenti
Gli autori ringraziano Izon Science Ltd per il loro sostegno. Il lavoro è stato sostenuto dalla Commissione Europea per la Ricerca (PCIG11-GA-2012-321.836 Nano4Bio).
Materiali
| Name | Company | Catalog Number | Comments |
| Phosphate buffered Saline (PBS) | Sigma Aldrich, UK | P4417 | 1 tablet dissolved in 200 ml deionized water to make buffer solution. |
| Tween-20 | Sigma Aldrich, UK | P1379 | 0.05% (v/v) in PBS buffer as a surfactant |
| Carboxyl polystyrene nanoparticles | Bangs Laboratories, US | CPC200 | Nominal diamter of 220 nm, raw concentration of 1 x 1012 particles/ml, specific surface charge of 86 µeq/g (equivalent to a surface charge density of 3.2 x 1019 C/nm2. |
| Streptavidin coated nanoparticles | Ademtech, France | 3121 | Batch had binding capacity of 4,352 pmol/mg (188 nM theoretical DNA binding capacity) at a raw concentration of 1.1 x 1011 particles/ml. |
| Biotinylated oligonucleotides | Sigma Aldrich, UK | VC00001 | Supplier spec: Reverse Phase 1 purification (0.05 Scale); Biotin modification at 3' end; Lyophilized powders reconstituted to 100 µM using deionized water, and diluted as required. Sequences: CP 5'ATGGTTAAACCTCACTAC GCGTGGC[Btn]3' |
| Standard olignonucleotides | Sigma Aldrich, UK | VC00001 | Supplier spec: Reverse Phase 1 purification (0.05 Scale); Lyophilized powders reconstituted to 100 µM using deionized water, and diluted as required. Sequences of DNA targets: Fully complementary - 5'GCCACGCGTAGTGA GGTTTAACCAT3', Middle binding - 5'GTAGTGAGGT3', End binding - 5'GTTTAACCAT3', Partially complementary overhanging - 5'GTGAGGTTTAACCAT TTTTTTTTTTTTTTT3'. |
| Izon qNano | Izon Science, NZ | Inherent pressure on system of 47 Pa | |
| Izon Variable Pressure Module (VPM) | Izon Science, NZ | Each 'cm' of pressure is equivalent to approximately 100 Pa. | |
| Polyurethane nanopore membranes | Izon Science, NZ | NP150 | Analyte size range 60-480 nm, pore diameter of calculated to be 799 nm at a 45 mm stretch. |
| Magrack 6 | GE Healthcare, UK | 28-9489-64 | |
| Sonic Bath | Fisher Scientific, UK | 10692353 | 80 Watts |
| Vortexer | IKA, Germany | 0003365000 | |
| Rotary Wheel | Labnet International, US | H5500-230 V |
Riferimenti
- Alexander, C. M., Maye, M. M., Dabrowiak, J. C. DNA-capped nanoparticles designed for doxorubicin drug delivery. Chem Commun. 47 (12), 3418-3420 (2011).
- Billinge, E. R., Platt, M. Aptamer based dispersion assay using tunable resistive pulse sensing (TRPS). Anal Methods. 7 (20), 8534-8538 (2015).
- Bulyk, M. L. Protein Binding Microarrays for the Characterization of Protein-DNA Interactions. Adv Biochem Eng Biotechnol. 104, 65-85 (2007).
- Platt, M., Rowe, W., Knowles, J., Day, P. J., Kell, D. B. Analysis of aptamer sequence activity relationships. Integr Biol. 1 (1), 116-122 (2009).
- Ruiz-Hernández, E., Baeza, A., Vallet-Regí, M. Smart Drug Delivery through DNA/Magnetic Nanoparticle Gates. ACS Nano. 5 (2), 1259-1266 (2011).
- Murdock, R. C., Braydich-stolle, L., Schrand, A. M., Schlager, J. J., Hussain, S. M. Characterization of Nanomaterial Dispersion in Solution Prior to In Vitro Exposure Using Dynamic Light Scattering Technique. Toxicol Sci. 101 (2), 239-253 (2008).
- Hupfield, S., Holsaeter, A. M., Skar, M., Frantzen, C. B., Brandl, M. Liposome size analysis by dynamic/static light scattering upon size exclusion-/field flow fractionation. J Nanosci Nanotechnol. 6 (7), 3025-3031 (2006).
- Roberts, G. S., et al. Tunable pores for measuring concentrations of synthetic and biological nanoparticle dispersions. Biosens Bioelectron. 31 (1), 17-25 (2012).
- Roberts, G. S., Kozak, D., Anderson, W., Broom, M. F., Vogel, R., Trau, M. Tunable nano/micropores for particle detection and discrimination: scanning ion occlusion spectroscopy. Small. 6 (23), 2653-2658 (2010).
- Vogel, R., et al. Quantitative sizing of nano/microparticles with a tunable elastomeric pore sensor. Anal Chem. 83 (9), 3499-3506 (2011).
- Booth, M. A., Vogel, R., Curran, J. M., Harbison, S., Travas-Sejdic, J. Detection of target-probe oligonucleotide hybridization using synthetic nanopore resistive pulse sensing. Biosens Bioelectron. 45, 136-140 (2013).
- Kozak, D., Anderson, W., Vogel, R., Chen, S. Simultaneous size and ζ-potential measurements of individual nanoparticles in dispersion using size-tunable pore sensors. ACS Nano. 6 (8), 6990-6997 (2012).
- Kozak, D., Anderson, W., Vogel, R., Trau, M. Advances in Resistive Pulse Sensors: Devices bridging the void between molecular and microscopic detection. Nano Today. 6 (5), 531-545 (2011).
- Weatherall, E., Willmott, G. R. Applications of tunable resistive pulse sensing. Analyst. 140, 3318-3334 (2015).
- Willmott, G. R., et al. Use of tunable nanopore blockade rates to investigate colloidal dispersions. J Phys Condens Matter. 22 (45), 454116 (2010).
- Blundell, E. L. C. J., Mayne, L. J., Billinge, E. R., Platt, M. Emergence of tunable resistive pulse sensing as a biosensor. Anal Methods. 7, 7055-7066 (2015).
- Platt, M., Willmott, G. R., Lee, G. U. Resistive Pulse Sensing of Analyte-Induced Multicomponent Rod Aggregation Using Tunable Pores. Small. 8 (15), 2436-2444 (2012).
- Vogel, R., Anderson, W., Eldridge, J., Glossop, B., Willmott, G. A variable pressure method for characterizing nanoparticle surface charge using pore sensors. Anal Chem. 84 (7), 3125-3131 (2012).
- Blundell, E. L. C. J., Vogel, R., Platt, M. Particle-by-Particle Charge Analysis of DNA-Modified Nanoparticles Using Tunable Resistive Pulse Sensing. Langmuir. 32 (4), (2016).
- Hunter, R. J. . Zeta Potential in Colloid Science: Principles and Applications. , (1981).
- Arjmandi, N., Van Roy, W., Lagae, L., Borghs, G. Measuring the electric charge and zeta potential of nanometer-sized objects using pyramidal-shaped nanopores. Anal Chem. 84 (20), 8490-8496 (2012).
- Bacri, L., et al. Dynamics of colloids in single solid-state nanopores. J Phys Chem B. 115 (12), 2890-2898 (2011).
- Cabello-Aguilar, S., et al. Dynamics of polymer nanoparticles through a single artificial nanopore with a high-aspect-ratio. Soft Matter. 10 (42), 8413-8419 (2014).
- Billinge, E. R., Muzard, J., Platt, M. Tunable resistive pulse sensing as a tool to monitor analyte induced particle aggregation. Nanomater Nanosci. 1 (1), 11 (2013).
- Li, J., Fan, C., Pei, H., Shi, J., Huang, Q. Smart Drug Delivery Nanocarriers with Self-Assembled DNA Nanostructures. Adv Mater. 25 (32), 4386-4396 (2013).
- Billinge, E. R., Broom, M., Platt, M. Monitoring aptamer-protein interactions using tunable resistive pulse sensing. Anal Chem. 86 (2), 1030-1037 (2014).
- Gold, L., et al. Aptamer-Based Multiplexed Proteomic Technology for Biomarker Discovery. PLoS One. 5 (12), e15004 (2010).
- Park, S. -. J., Taton, T. A., Mirkin, C. A. Array-Based Electrical Detection of DNA with Nanoparticle Probes. Science. 295 (5559), 1503-1506 (2002).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon