Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Determinación del Potencial Zeta a través de nanopartículas de translocación velocidades a través de un sintonizable nanoporos: El uso de partículas de ADN modificado como ejemplo
En este artículo
Resumen
Aquí se utiliza un nanopore sintonizable de poliuretano integrado en una técnica de detección de impulsos resistiva para caracterizar las nanopartículas química de la superficie a través de la medición de las velocidades de translocación de partículas, que se puede utilizar para determinar el potencial zeta de las nanopartículas individuales.
Resumen
tecnologías de nanoporos, conocidos colectivamente como sensores de pulso resistivas (RPS), están siendo utilizados para detectar, cuantificar y caracterizar las proteínas, las moléculas y nanopartículas. detección de pulso resistiva sintonizable (TRPS) es una adaptación relativamente reciente al PRC que incorpora un poro ajustable que puede ser alterado en tiempo real. Aquí, nosotros usamos TRPS para controlar los tiempos de translocación de las nanopartículas de ADN modificado a medida que atraviesan la membrana de poro sintonizable como una función de la concentración de ADN y la estructura (es decir, de una sola cadena en ADN de doble cadena).
TRPS se basa en dos electrodos de Ag / AgCl, separadas por una membrana de poro de elastómero que establece una corriente iónica estable a un campo eléctrico aplicado. A diferencia de las diversas tecnologías de caracterización de partículas basado en la óptica, TRPS pueden caracterizar partículas individuales en una población de la muestra, lo que permite muestras multimodales para ser analizados con facilidad. Aquí, demostramos medidas de potencial zetaa través de las velocidades de translocación de partículas de estándares conocidos y aplicar estos a la muestra tiempos de translocación de analito, lo que resulta en la medición del potencial zeta de los analitos.
Así como la adquisición de valores del potencial zeta medias, las muestras se miden utilizando una perspectiva de partícula por partícula que exhibe más información sobre una muestra dada por medio de distribuciones de la población de la muestra, por ejemplo. De tal, este método demuestra potencial dentro de aplicaciones de detección para ambos campos de la medicina y ambientales.
Introducción
nanopartículas funcionalizadas se están convirtiendo cada vez más popular como biosensores en ambos campos de la medicina y ambientales. La capacidad de alterar la química de la superficie de una nanopartícula, con el ADN, por ejemplo, está demostrando útil para sistemas de administración de fármacos dirigidos 1 y monitoreo interacciones ADN-proteína 2-4. Una propiedad de nanopartículas cada vez más común siendo utilizado en los bioensayos y en la entrega de la terapéutica es superparamagnetismo 5. partículas superparamagnéticas (SPP) son extremadamente útiles en la identificación y eliminación de sustancias específicas de análisis a partir de mezclas complejas y pueden hacerlo con el simple uso de un solo imán. Una vez extraídas, las partículas de analito unido se pueden caracterizar y analizar adecuado para el propósito.
Los métodos anteriores utilizados para la detección y caracterización de nanopartículas incluyen técnicas ópticas tales como la dispersión dinámica de luz (DLS), también conocida como espectroscopía de correlación de fotones. Aunque un hitécnica de rendimiento gh, DLS se limita a ser una técnica basada promediado y en el análisis de muestras multimodales sin la adición de un software especializado, las partículas más grandes producirán una señal mucho más dominante, dejando algunos de las partículas más pequeñas totalmente desapercibido 6,7. Partícula por partícula técnicas de caracterización, por tanto, son mucho más favorables para analizar los sistemas de nanopartículas funcionalizadas de nanopartículas y.
tecnologías basadas RPS se basan en la aplicación de un campo eléctrico a una muestra y el seguimiento del mecanismo de transporte de las partículas a través de un nanopore sintético o biológico. Una técnica de detección y caracterización de nanopartículas relativamente reciente basado en RPS es la detección de pulso resistiva sintonizable (TRPS) 8-16. TRPS es un sistema de dos electrodos separados por una membrana de poros elastomérico, sintonizable. Un método sintonizable de poros permite analitos de una gama de 17 forma y tamaño para ser medido a través de su transmecanismos de puerto a través del poro. Poros sintonizables anteriormente se han utilizado para la detección de partículas pequeñas (70 a 95 nm de diámetro) que produce resultados comparables a otras técnicas como la espectroscopía electrónica de transmisión (TEM) 10. Cuando se aplica un campo eléctrico, se observa una corriente iónica y como partículas / moléculas pasan a través del poro, que bloquean temporalmente el poro, causando una reducción en la corriente que se puede definir como un "evento bloqueo '. Cada evento bloqueo es representante de una sola partícula de manera que cada partícula dentro de una muestra se pueden caracterizar individualmente basa en la magnitud bloqueo, Δ 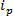 , Y la anchura total media-máxima, FWHM, así como otras propiedades del bloqueo. El análisis de las partículas individuales a medida que pasan a través de un nanopore es ventajoso para muestras multimodales como TRPS con éxito y eficacia pueden distinguir una gama de tamaños de partícula amonGST una sola muestra. Detección de pulso resistiva sintonizable completa tamaño 10, potencial zeta 12,18 y la concentración de 15 mediciones simultáneamente en una única ejecución y por lo tanto puede todavía diferenciar muestras de similar, si no el mismo tamaño por su carga de superficie 19; una ventaja sobre las técnicas de encolado alternativos.
, Y la anchura total media-máxima, FWHM, así como otras propiedades del bloqueo. El análisis de las partículas individuales a medida que pasan a través de un nanopore es ventajoso para muestras multimodales como TRPS con éxito y eficacia pueden distinguir una gama de tamaños de partícula amonGST una sola muestra. Detección de pulso resistiva sintonizable completa tamaño 10, potencial zeta 12,18 y la concentración de 15 mediciones simultáneamente en una única ejecución y por lo tanto puede todavía diferenciar muestras de similar, si no el mismo tamaño por su carga de superficie 19; una ventaja sobre las técnicas de encolado alternativos.
El potencial zeta se define como el potencial electrostático en el plano de cizallamiento 20, y se calcula a partir de velocidades de las partículas a medida que atraviesan un poro 19. mediciones de potencial zeta de las partículas individuales de este modo da una idea de los mecanismos y comportamiento de los sistemas de nanopartículas en solución, información valiosa para el futuro de los diseños de ensayo de nanopartículas para una gama de aplicaciones de translocación. análisis de partículas por partícula de tal naturaleza también permite la propagación y la distribución de los valores de potencial zeta entre una población de muestra para ser explorado, lo que permite más información ocinética de la reacción n (monocatenario a ADN de doble cadena, por ejemplo) y estabilidades de partículas en solución que hay que alcanzar.
A continuación, describimos una técnica que detecta y caracteriza a ambas superficies modificadas y no modificadas de ADN SPP. El protocolo descrito en el presente documento es aplicable a una gama de nanopartículas inorgánicas y biológicas, pero se demuestra el procedimiento de uso de superficies de ADN modificado debido a su amplia gama de aplicaciones. La técnica permite al usuario distinguir entre dianas de ADN de una sola hebra y de doble cadena en una superficie de las nanopartículas, basado en las velocidades de translocación de partículas a través de un sistema de poros y por lo tanto sus potenciales zeta.
Protocolo
1. Hacer la solución tampón fosfato con Tween-20 (PBST) Buffer
- Disolver un comprimido de PBS (tampón fosfato 0,01 M, 0,0027 M cloruro de potasio, 0,137 M de cloruro de sodio, pH 7,4) en 200 ml de agua desionizada (18,2 mO cm).
- Añadir 100 ml (0,05 (v / v)%) de Tween-20 a la solución 200 ml de tampón como un agente tensioactivo.
2. Preparación de las Normas de partículas de poliestireno carboxilo
- Vortex las partículas de calibración para 30 segundos antes de la sonicación durante 2 minutos a 80 vatios para crear monodispersidad de las partículas.
- Diluir las partículas de calibración 1 en 100 a una concentración de 1x10 10 partículas / ml en tampón PBST y agitar durante 30 seg.
3. Preparación de partículas recubiertas con estreptavidina
- Vortex las partículas durante 30 segundos antes de la sonicación durante 2 minutos a 80 vatios para asegurar monodispersidad.
- Diluir las partículas recubiertas con estreptavidina 1 en 100 en tampón PBST para achieve como resultado una concentración de 1x10 9 partículas / ml y agitar durante 30 segundos.
Nota: Un volumen de muestra típica es de 200 l. Por ejemplo, si la investigación de cinco concentraciones de ADN se preparan 1 ml de partículas recubiertas con estreptavidina diluidas.
4. Preparación de oligonucleótidos
- Reconstituir oligonucleótidos con agua desionizada a una concentración resultante de 100 mM.
5. La adición de ADN de captura de sonda (CP) a las partículas recubiertas con estreptavidina
- Antes de la unión a ADN, vórtice las partículas recubiertas con estreptavidina (200 l de volumen de la muestra) durante 30 segundos seguido de un tratamiento con ultrasonidos 2 min a 80 vatios.
- Sobre la base de la capacidad de unión proporcionada por el proveedor (4352 pmol / mg), añadir la concentración apropiada de ADN a las partículas para las concentraciones resultantes de, DNA 10, 20, 30, 40, 47 95, 140, y 210 nM.
- Vortex las muestras durante 10 segundos y colocar en una rueda giratoria a temperatura ambientedurante 30 min para permitir el ADN se una a las superficies de las partículas a través de una interacción de estreptavidina-biotina.
- Una vez que el ADN de captura se ha añadido y se incubaron con las partículas recubiertas con estreptavidina, eliminar el exceso de ADN en solución a través de la separación magnética mediante la colocación de las muestras sobre una rejilla magnética durante 30 min.
- Eliminar el sobrenadante, teniendo cuidado de no alterar el clúster recién formado de partículas más cercanos al imán, y reemplazar con el mismo volumen de tampón PBST nueva.
6. Hibridación de ADN complementario al CP-partículas
- Añadir la cantidad requerida de ADN diana (en exceso a 500 nm) para asegurar el objetivo máximo posible la unión fue alcanzado.
- Vortex las muestras durante 10 segundos y colocar en una rueda giratoria a temperatura ambiente durante 30 min.
- Una vez que la hibridación es completa, eliminar el exceso de ADN diana mediante separación magnética mediante la colocación de las muestras sobre una rejilla magnética durante 30 minutos.
- Eliminar el sobrenadante, Teniendo cuidado de no alterar el clúster recién formado de partículas más cercanos al imán, y reemplazar con el mismo volumen de tampón PBST nueva.
- Repetir los pasos 6.1 a 6.4 para muestras duplicadas y colocar estas muestras en una rueda giratoria a temperatura ambiente durante 16 horas para investigar tiempos de hibridación de ADN.
7. Configuración TRPS
- Enchufe el instrumento en un sistema informático con el software en su lugar.
- Calibrar el tramo inicial utilizando un calibrador.
- Medir la distancia entre la parte exterior de dos mordazas paralelas.
- De entrada en el software escribiendo el estiramiento en el campo 'estiramiento' en la pestaña "Configuración del instrumento" y hacer clic en "Calibrar tramo 'debajo de la lengüeta.
- ajustarse lateralmente una membrana de poliuretano nanoporos de dimensionamiento adecuado para el análisis en las mandíbulas con el número de identificación de nanoporos hacia arriba. A continuación, se extienden las mordazas para el estiramiento necesario para el análisis utilizando lamanija de ajuste de estiramiento en el lado del instrumento. Estirar las mandíbulas entre 43 y 48 mm.
Nota: El valor exacto de la recta se determina junto con la tensión aplicada de modo que los bloqueos de partículas de calibración son al menos 0,3 nA en tamaño. El tramo ya se introduce en el software en el paso 7.2 y ajustará automáticamente a medida que las mandíbulas se estiran. - Colocar 80 l de tampón PBST en la celda de fluido inferior, debajo de la nanoporos, asegurando que no hay burbujas presentes que pueden afectar a la medición. Si hay burbujas vistos, remover y reemplazar el tampón.
- Haga clic en la celda de fluido superior en su lugar y colocar 40 l de tampón en ella, asegurando de nuevo que no hay burbujas presentes. Si hay burbujas presentes en la célula fluido superior, eliminarlos mediante la sustitución del líquido.
- Cuando una corriente de línea de base reproducible se ha alcanzado de la sustitución de la célula de fluido superior con tampón, añadir 40 l de la muestra a la celda de fluido superior y la medida haciendo clic 'starte "de la pestaña" Adquisición de datos "en la pantalla del software.
Nota: La adquisición de datos se completa con una frecuencia de 50 kHz con límite de una magnitud bloqueo inferior de 0,05 nA, aunque esto puede ser alterado mediante el software a través de la pestaña "Análisis de Datos" (bajo "Configuración de análisis" y "resistente bloqueos ') . - Colocar una jaula de Faraday sobre la parte superior del sistema de pila de fluido para reducir el ruido de fondo eléctrico en las mediciones.
- Utilice un módulo de presión variable (VPM) para aplicar una presión o vacío a las muestras.
- Para aplicar una presión externa conectar la boquilla a la celda de fluido superior, a continuación, girar el brazo de presión y haga clic en su lugar (dependiendo de si se aplica una presión positiva (PRE) o un vacío (VAC)).
- Aplicar presión en un 'cm' o la escala "mm" con el mando de nivel de presión situado en la parte superior de la VPM. Presione el botón de abajo para aplicar presión sobre la escala 'cm' y tire de él hacia arriba to aplicar presión en la escala de "mm".
8. Preparación de muestras para el análisis de TRPS
- muestras de vórtice durante 30 segundos y se somete a ultrasonidos durante 2 minutos a 80 vatios antes del análisis TRPS.
9. Calibración de la nanoporos para el Análisis Zeta
- Después de colocar 40 l partículas de calibración (1x10 10 partículas / ml) en la célula fluido superior, completar una medición TRPS (instalación como en la sección 7) a las 3 tensiones aplicadas. Alterar el voltaje haciendo clic en los botones "+" y "-" situados en la escala de tensión en la pestaña "Configuración del instrumento" en el software.
- Compruebe que las 3 tensiones vuelven corrientes de fondo de aproximadamente 140, 110 y 80 nA. Asegúrese de que en la media tensión las partículas de calibración producen una magnitud media bloqueo de al menos 0,3 nA.
- Aplicar una presión por lo que el promedio de ancho total a media altura (FWHM) duraciones de las partículas de calibración son, al menos,0,15 mseg. Hacer esto de forma manual mediante el brazo de presión unida al módulo de presión variable. Seleccione la presión (PRE) o vacío (VAC) haciendo girar el brazo hasta que encaje en la posición deseada y aplicar en consecuencia siguiente configuración instrucciones en el paso 7.8.2. Una vez que se han alcanzado estas condiciones, iniciar el proceso haciendo clic en "inicio" en el software en la pestaña 'Adquisición de Datos'.
- Completar la ejecución pulsando 'parada' en la pestaña 'Adquisición de Datos "cuando al menos 500 partículas se han medido (ver" El recuento de partículas' en la parte inferior de la pantalla del software durante la medición) y la carrera ha superado los 30 segundos (ver " Run Time 'también hacia la parte inferior de la pantalla).
- Calibrar el sistema completando una fase de calibración como se describe cada vez que se introduce un nuevo nanoporos o para cada nuevo día de análisis por parte de completar el paso 9.1-9.4.
10. La ejecución de una Muestra
- Ejecutar las muestras al más alto osegundo voltaje más alto que las muestras de calibración a garantizar un similar (± 10 nA), si no la misma corriente, la línea de base.
- Una vez que se consigue la corriente de base de referencia, sustituir el electrolito en la celda de fluido superior con 40 l de muestra. Cuando se introduce una muestra, los bloqueos se pueden ver en la traza de la señal. Comience la muestra ejecutar haciendo clic en "Inicio" en la pestaña "Adquisición de Datos 'y registrar un mínimo de 500 partículas (marque' recuento de partículas 'situado debajo de la traza de la señal) y asegurar el tiempo de ejecución es de un mínimo de 30 segundos (véase" Ejecutar tiempo también situada por debajo de la traza de la señal).
- Para completar la medición, haga clic en "stop" en la pestaña "Data Acquisition" y guardar el archivo de datos.
- Para guardar el archivo, introduzca la información del archivo en el siguiente formato; 'Investigación' es la carpeta del archivo se guardará en "nanoporos ID 'es el número de serie del poro siendo utilizado,' Parte # 'iEs el tipo de poros (es decir, NP150 / NP200), 'ID de la muestra' es el nombre de la muestra, 'calibración o de la muestra' detalles sobre si se trata de una calibración o muestra de medición, "dilución" se utiliza si se diluyó la muestra ( escriba 100 si la muestra se diluyó 100 veces), "presión" es la presión aplicada a la muestra (en cm - ver sección 7.8), 'el electrolito ID' es el nombre de la memoria intermedia de la muestra se compone de, y ' notas 'son las notas personales sobre la muestra o correr.
- Entre cada muestra de ejecución, se lava el sistema mediante la colocación de 40 l de PBST tampón en la célula fluido superior varias veces y la aplicación de varias presiones (generalmente a -10, -5 cm (de vacío), y 5 y 10 cm (presión positiva)) hasta hay más eventos de bloqueo están presentes, asegurando que no hay partículas residuales que quedan en el sistema y, por tanto, no haya contaminación cruzada entre las muestras. muestras por triplicado con este paso de lavado completado entre cada repetición de la muestra se ejecutan como well como entre diferentes muestras.
Resultados
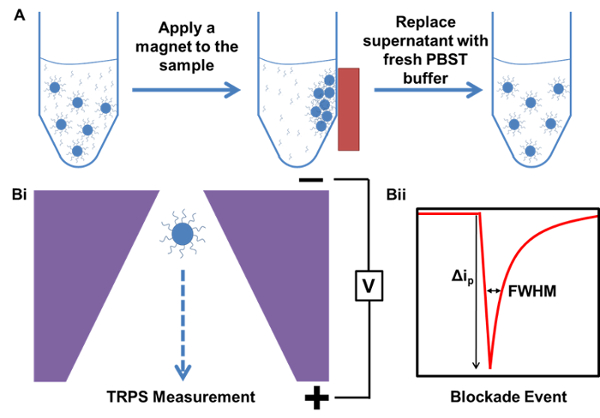
Figura 1. Representación esquemática de los procesos de purificación magnética y una medición TRPS. A) Ejemplo de purificación magnética de la muestra a partir de una muestra que contiene exceso, sin unir ADN sonda de captura. B) TRPS ejemplo de medición i) de partículas que pasa por el nanoporos y ii) evento bloqueo producido a partir de partículas de ione...
Discusión
El cálculo para el potencial zeta utiliza un método basado calibración relacionados con el trabajo por Arjmandi et al. 21. La duración de la translocación de las partículas a medida que atraviesan un nanopore se mide como una función de la tensión aplicada, utilizando un campo y de partículas promedio de las velocidades eléctricos sobre la totalidad de un poro cónica regular. La movilidad electroforética es el derivado de 1 / T (donde T es la duración del bloqueo) con respecto al v...
Divulgaciones
ELCJB se apoya en Izon Science Ltd.
Agradecimientos
Los autores agradecen a Izon Science Ltd por su apoyo. El trabajo fue apoyado por la Comisión Europea para la Investigación (PCIG11-GA-2012-321836 Nano4Bio).
Materiales
| Name | Company | Catalog Number | Comments |
| Phosphate buffered Saline (PBS) | Sigma Aldrich, UK | P4417 | 1 tablet dissolved in 200 ml deionized water to make buffer solution. |
| Tween-20 | Sigma Aldrich, UK | P1379 | 0.05% (v/v) in PBS buffer as a surfactant |
| Carboxyl polystyrene nanoparticles | Bangs Laboratories, US | CPC200 | Nominal diamter of 220 nm, raw concentration of 1 x 1012 particles/ml, specific surface charge of 86 µeq/g (equivalent to a surface charge density of 3.2 x 1019 C/nm2. |
| Streptavidin coated nanoparticles | Ademtech, France | 3121 | Batch had binding capacity of 4,352 pmol/mg (188 nM theoretical DNA binding capacity) at a raw concentration of 1.1 x 1011 particles/ml. |
| Biotinylated oligonucleotides | Sigma Aldrich, UK | VC00001 | Supplier spec: Reverse Phase 1 purification (0.05 Scale); Biotin modification at 3' end; Lyophilized powders reconstituted to 100 µM using deionized water, and diluted as required. Sequences: CP 5'ATGGTTAAACCTCACTAC GCGTGGC[Btn]3' |
| Standard olignonucleotides | Sigma Aldrich, UK | VC00001 | Supplier spec: Reverse Phase 1 purification (0.05 Scale); Lyophilized powders reconstituted to 100 µM using deionized water, and diluted as required. Sequences of DNA targets: Fully complementary - 5'GCCACGCGTAGTGA GGTTTAACCAT3', Middle binding - 5'GTAGTGAGGT3', End binding - 5'GTTTAACCAT3', Partially complementary overhanging - 5'GTGAGGTTTAACCAT TTTTTTTTTTTTTTT3'. |
| Izon qNano | Izon Science, NZ | Inherent pressure on system of 47 Pa | |
| Izon Variable Pressure Module (VPM) | Izon Science, NZ | Each 'cm' of pressure is equivalent to approximately 100 Pa. | |
| Polyurethane nanopore membranes | Izon Science, NZ | NP150 | Analyte size range 60-480 nm, pore diameter of calculated to be 799 nm at a 45 mm stretch. |
| Magrack 6 | GE Healthcare, UK | 28-9489-64 | |
| Sonic Bath | Fisher Scientific, UK | 10692353 | 80 Watts |
| Vortexer | IKA, Germany | 0003365000 | |
| Rotary Wheel | Labnet International, US | H5500-230 V |
Referencias
- Alexander, C. M., Maye, M. M., Dabrowiak, J. C. DNA-capped nanoparticles designed for doxorubicin drug delivery. Chem Commun. 47 (12), 3418-3420 (2011).
- Billinge, E. R., Platt, M. Aptamer based dispersion assay using tunable resistive pulse sensing (TRPS). Anal Methods. 7 (20), 8534-8538 (2015).
- Bulyk, M. L. Protein Binding Microarrays for the Characterization of Protein-DNA Interactions. Adv Biochem Eng Biotechnol. 104, 65-85 (2007).
- Platt, M., Rowe, W., Knowles, J., Day, P. J., Kell, D. B. Analysis of aptamer sequence activity relationships. Integr Biol. 1 (1), 116-122 (2009).
- Ruiz-Hernández, E., Baeza, A., Vallet-Regí, M. Smart Drug Delivery through DNA/Magnetic Nanoparticle Gates. ACS Nano. 5 (2), 1259-1266 (2011).
- Murdock, R. C., Braydich-stolle, L., Schrand, A. M., Schlager, J. J., Hussain, S. M. Characterization of Nanomaterial Dispersion in Solution Prior to In Vitro Exposure Using Dynamic Light Scattering Technique. Toxicol Sci. 101 (2), 239-253 (2008).
- Hupfield, S., Holsaeter, A. M., Skar, M., Frantzen, C. B., Brandl, M. Liposome size analysis by dynamic/static light scattering upon size exclusion-/field flow fractionation. J Nanosci Nanotechnol. 6 (7), 3025-3031 (2006).
- Roberts, G. S., et al. Tunable pores for measuring concentrations of synthetic and biological nanoparticle dispersions. Biosens Bioelectron. 31 (1), 17-25 (2012).
- Roberts, G. S., Kozak, D., Anderson, W., Broom, M. F., Vogel, R., Trau, M. Tunable nano/micropores for particle detection and discrimination: scanning ion occlusion spectroscopy. Small. 6 (23), 2653-2658 (2010).
- Vogel, R., et al. Quantitative sizing of nano/microparticles with a tunable elastomeric pore sensor. Anal Chem. 83 (9), 3499-3506 (2011).
- Booth, M. A., Vogel, R., Curran, J. M., Harbison, S., Travas-Sejdic, J. Detection of target-probe oligonucleotide hybridization using synthetic nanopore resistive pulse sensing. Biosens Bioelectron. 45, 136-140 (2013).
- Kozak, D., Anderson, W., Vogel, R., Chen, S. Simultaneous size and ζ-potential measurements of individual nanoparticles in dispersion using size-tunable pore sensors. ACS Nano. 6 (8), 6990-6997 (2012).
- Kozak, D., Anderson, W., Vogel, R., Trau, M. Advances in Resistive Pulse Sensors: Devices bridging the void between molecular and microscopic detection. Nano Today. 6 (5), 531-545 (2011).
- Weatherall, E., Willmott, G. R. Applications of tunable resistive pulse sensing. Analyst. 140, 3318-3334 (2015).
- Willmott, G. R., et al. Use of tunable nanopore blockade rates to investigate colloidal dispersions. J Phys Condens Matter. 22 (45), 454116 (2010).
- Blundell, E. L. C. J., Mayne, L. J., Billinge, E. R., Platt, M. Emergence of tunable resistive pulse sensing as a biosensor. Anal Methods. 7, 7055-7066 (2015).
- Platt, M., Willmott, G. R., Lee, G. U. Resistive Pulse Sensing of Analyte-Induced Multicomponent Rod Aggregation Using Tunable Pores. Small. 8 (15), 2436-2444 (2012).
- Vogel, R., Anderson, W., Eldridge, J., Glossop, B., Willmott, G. A variable pressure method for characterizing nanoparticle surface charge using pore sensors. Anal Chem. 84 (7), 3125-3131 (2012).
- Blundell, E. L. C. J., Vogel, R., Platt, M. Particle-by-Particle Charge Analysis of DNA-Modified Nanoparticles Using Tunable Resistive Pulse Sensing. Langmuir. 32 (4), (2016).
- Hunter, R. J. . Zeta Potential in Colloid Science: Principles and Applications. , (1981).
- Arjmandi, N., Van Roy, W., Lagae, L., Borghs, G. Measuring the electric charge and zeta potential of nanometer-sized objects using pyramidal-shaped nanopores. Anal Chem. 84 (20), 8490-8496 (2012).
- Bacri, L., et al. Dynamics of colloids in single solid-state nanopores. J Phys Chem B. 115 (12), 2890-2898 (2011).
- Cabello-Aguilar, S., et al. Dynamics of polymer nanoparticles through a single artificial nanopore with a high-aspect-ratio. Soft Matter. 10 (42), 8413-8419 (2014).
- Billinge, E. R., Muzard, J., Platt, M. Tunable resistive pulse sensing as a tool to monitor analyte induced particle aggregation. Nanomater Nanosci. 1 (1), 11 (2013).
- Li, J., Fan, C., Pei, H., Shi, J., Huang, Q. Smart Drug Delivery Nanocarriers with Self-Assembled DNA Nanostructures. Adv Mater. 25 (32), 4386-4396 (2013).
- Billinge, E. R., Broom, M., Platt, M. Monitoring aptamer-protein interactions using tunable resistive pulse sensing. Anal Chem. 86 (2), 1030-1037 (2014).
- Gold, L., et al. Aptamer-Based Multiplexed Proteomic Technology for Biomarker Discovery. PLoS One. 5 (12), e15004 (2010).
- Park, S. -. J., Taton, T. A., Mirkin, C. A. Array-Based Electrical Detection of DNA with Nanoparticle Probes. Science. 295 (5559), 1503-1506 (2002).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados