需要订阅 JoVE 才能查看此. 登录或开始免费试用。
Method Article
通过显微注射卵母细胞生成转基因小鼠
摘要
小鼠卵母细胞的显微注射通常用于经典转基因( 即转基因的随机整合)和CRISPR介导的基因靶向。该协议审查显微注射的最新发展,特别强调质量控制和基因分型策略。
摘要
使用转基因小鼠对生理和病理体内过程的研究有显着的贡献。将DNA表达构建体的原核注射到受精卵中仍然是产生用于过表达的转基因小鼠的最常用技术。随着用于基因靶向的CRISPR技术的引入,原核注入受精卵母细胞已经扩展到敲除和敲入小鼠的产生。这项工作描述了用于注射的DNA的制备和用于基因靶向的CRISPR指南的产生,特别强调质量控制。识别潜在创始人所需的基因分型程序至关重要。本文介绍了利用CRISPR"复用"能力的创新型基因分型策略。还概述了外科手术。一起,协议的步骤将允许生成gen以及随后建立的大量研究领域的小鼠集落,包括免疫学,神经科学,癌症,生理学,发育等。
引言
在脊椎动物和无脊椎动物中的动物模型已经有助于检查人类病情如阿尔茨海默病1,2的病理生理学。他们也是寻找疾病修饰剂的最宝贵的工具,并最终开发出新的治疗策略,希望得到治愈。虽然每个模型都有固有的局限性,但是使用动物作为整个系统模型对于生物医学研究至关重要。这是因为组织培养不能完全模拟代谢和复杂的生理环境。
迄今为止,鼠标仍然是用于遗传操作的最常见的哺乳动物物种,因为它具有几个优点。与疾病相关的生理过程和基因在小鼠和人之间是高度保守的。小鼠是第一个拥有全基因组测序的哺乳动物(2002年),人类基因组前一年我(2003)。除了丰富的遗传信息,小鼠具有良好的育种能力,快速的开发周期(从受精到断奶6周)和合理的大小。所有这些优点,加上生理指标,如独特的外套颜色(交叉策略所需),使得鼠标成为遗传操作的有吸引力的模型。值得注意的是,在现代遗传学的很早的时代,格雷戈尔·门德尔(Gregor Mendel)开始对老鼠进行植物移植3 。
基因转移技术导致在三十年前第一代转基因小鼠的产生4 ,最初使用病毒递送。然而,研究人员很快意识到,小鼠转基因的主要挑战之一是无法控制外源DNA的命运。因为转基因向小鼠卵母细胞的病毒递送导致随机地整合到基因组中的多个拷贝,这是可能的建立后续转基因株系的限制。
当Gordon 等人通过显微注射产生第一个转基因小鼠系列5,6 。这开始了重组DNA技术的时代,影响显微注射结局的参数已被广泛研究7 。虽然显微注射不允许控制转基因的整合位点(最终导致每个创始人小鼠的特异性表达水平),但是原核显微注射的主要优点仍然是形成连续体( 即,转基因的多个拷贝的阵列,在基因组整合前5) 。多年来已经使用这种特征来建立数千个过表达感兴趣的基因的转基因小鼠品系。从那时起,转基因,a生物体基因组的人工修饰已广泛用于鉴定单一基因在疾病发生中的作用。
当马里奥·卡佩奇奇成功地破坏了小鼠中的单个基因时,达到了操纵小鼠基因组的另一个关键成果,打开了基因靶向时代8 。然而,基于ES细胞的基因靶向快速出现主要缺点,包括培养ES细胞的挑战,嵌合体的程度有所不同,以及进程的长度( 即 12-18个月,最小化获得小鼠) 。
最近,已经出现了诸如工程内切核酸酶( 例如锌指核酸酶(ZFN),转录激活物样效应核酸酶(TALEN))和聚集的定期交织的短回文重复序列(CRISPR / Cas9)的新技术的进展,作为替代方法加速麦克风中基因靶向过程e 9,10 。这些核酸内切酶可以通过显微注射容易地注入小鼠卵母细胞,从而在短短6周内产生基因靶向的小鼠。
自从关于使用CRISPR进行基因组编辑的第一份报告11以来,这种细菌适应性免疫系统由于具有许多优点而取代了ZFN和TALEN,包括易于合成和一次靶向多个基因座的能力(简称"复用" ")。 CRISPR首先用于小鼠12中的基因靶向,并已被应用于从植物到人类的无数种类13,14 。迄今为止,还没有关于抗CRISPR基因组编辑的单一物种的报道。
产生转基因小鼠的两个主要限制步骤是注射卵母细胞和再植入的这些卵母细胞变成假怀孕的雌性。尽管我们已经描述了这种技术15和其他16 ,但是近来在小鼠胚胎学和基因转移技术方面的技术改进已经改变了生产转基因小鼠的过程。这里将描述这些改进。
Access restricted. Please log in or start a trial to view this content.
研究方案
所有程序已获得新南威尔士大学动物护理与伦理委员会的批准。
1.准备转基因(随机整合)
- 分析琼脂糖凝胶电泳。
- 根据制造商的建议,使用合适的酶(1小时孵育)或快速消化酶(15至30分钟孵育)在热循环仪中消化质粒以切割转基因(参见图2A及其图例)。
- 加入1%三醋酸乙二胺四乙酸(EDTA)(TEA)琼脂糖凝胶,用0.5-1.0μg/ mL溴化乙锭(EtBr)染色。
注意:使用EtBr时要小心;它是一种有效的诱变剂。请使用适当的个人防护装备(参见材料安全数据表)。 - 加载1 kb分子量标记。
- 加载线性化片段( 即转基因和骨架)。
- 在100V下运行电泳45分钟。
- 可视化紫外线(UV)透射仪上的凝胶,以检查消化是否完整并确认转基因的正确大小( 即 7,338 bp; 见图2A )。
- 制备琼脂糖凝胶电泳。
- 注射1%TAE琼脂糖凝胶,用低毒凝胶染色剂染色。加载不含分子量标记物的8等份(每个1μg)线性化转基因,并在100V下运行45分钟。使用手术刀切除对应于线性化转基因的所有8个带。
- 根据制造商的建议,使用凝胶提取试剂盒提取DNA(参见材料表 )。
- 将凝胶碎片在50℃在1.5-mL管中熔化至少15分钟。用异丙醇沉淀,然后加入结合缓冲液。
- 结合整个DNA将其全部移入一列。洗脱到不含核酸酶的显微注射缓冲液(8mM Tris-HCl和0.15mM EDTA)中。通过0.22微米离心过滤器过滤并以12,000 x g离心60秒。
- 使用分光光度计测量DNA的浓度和质量。检查A260 / A280的比例在1.8左右( 即没有蛋白质污染),A260 / A230的比例至少为2.0( 即没有有机溶剂污染)。在无核酸酶的显微注射缓冲液中稀释至3ng /μL,等分(20-50μL),并在-20℃下冷冻。
2. CRISPR成分的合成(基因靶向)
- Cas9 mRNA。
- 在无核酸酶的显微注射缓冲液中,将Cas9 mRNA(从商业来源获得,参见材料表)稀释至1μg/μL的浓度。
- 等份在2μL和免费在-80°C。
- 单向导向RNA(SgRNA)。
- 使用计算设计工具识别所需的双靶基因组序列(指南),允许最小的潜在脱靶活动17 。
- 对于基因敲除,系统地选择位于相反方向上几百个碱基对(bp)的两个指南,并且包括感兴趣的基因的起始密码子。对于同源性直接修复,选择两个重叠的引导(尽可能),在相反的方向。
注意:例如,以下指南最近已成功地共同注射以破坏TPM4.2基因的第一个外显子:
指南1:5'CGCCATCCAGTTCGCGCTGC 3';
指南2:5'CAGAACGATTGAGCTATGGC 3'
- 对于基因敲除,系统地选择位于相反方向上几百个碱基对(bp)的两个指南,并且包括感兴趣的基因的起始密码子。对于同源性直接修复,选择两个重叠的引导(尽可能),在相反的方向。
- 订购一组如表1所示的引物。
- 合成线性DNA模板。
- 稀释px330不含核酸酶的水中的质粒为10ng /μL。
- 准备主混合物,如表1所示 。最后添加聚合酶,并将主混合物保持在冰上。
- 混合并分成8个PCR管(19μL/管)。每管加入1μL的px330,并在以下条件下进行PCR:98℃1分钟; 98℃10秒,64℃30秒和72℃15秒的40个循环;最后在72℃延伸5分钟。保持在4°C。
- 使用PCR纯化试剂盒(参见材料表 ),歧管和真空源(用于更快的处理)来纯化PCR产物。应用最大真空强度( 即 8毫巴)。
- 向1体积的PCR样品中加入5体积(100μL)的结合缓冲液并混合。
- 将(提供的)二氧化硅膜旋转柱放置在(提供的)2-mL收集管中用于离心,或在歧管上进行真空处理ING。为了结合DNA,将所有8个样品连续施加到柱上,并对每个负载以12,000 xg进行真空或离心60秒。
- 为了洗涤,向柱中加入0.75mL洗涤缓冲液(缓冲液PE),真空或以12,000xg离心60秒。以12,000 xg离心柱60 s,以消除残留的乙醇。
- 将色谱柱置于干净的1.5 mL微量离心管中。为了洗脱DNA,将30μL无核酸酶的水加入到膜的中心,让柱静置1分钟,并以12,000g离心60秒。
- 使用分光光度计测量浓度;通常浓度应为100ng /μL或更高。检查A260 / A280的比例在1.8左右( 即没有蛋白质污染),A260 / A230的比例至少为2.0( 即没有有机溶剂污染)。
- 使用T7 RNA合成试剂盒进行体外转录。
- 准备t他的主要混合物,如表1所示 ,并在37℃下在热循环仪中孵育3小时。加入28μL无核酸酶水和2μLDNA酶I,并在37℃下在热循环仪中孵育另外15分钟。
- 使用旋转柱进行RNA纯化。
- 将所提供的旋转柱中包含的粉末溶解在650μL不含核酸酶的显微注射缓冲液中,小心地清除所有气泡。在室温下盖上管子并水化5-15分钟。
- 取出底部的蓝色帽子,并将色谱柱放入2-mL管中。以750 xg和室温离心2分钟。将色谱柱放入新鲜的1.5 mL管中,并将50μLRNA溶液逐滴加入中心,而不要接触柱壁。在750 x g下旋转色谱柱2分钟。
- 使用分光光度计测量RNA浓度(一个反应的典型产率为30 - 50μg)。检查A260 / A280比率是否为( 即没有蛋白污染),A260 / A230比例至少为2.0( 即没有有机溶剂污染)。将sgRNA存储在-80°C直到使用。
- 使用常规用于DNA电泳的1%TAE凝胶评估RNA的质量。运行200-400 ng的DNA模板和相应的sgRNA并行。 RNA带(≈100bp)应略大于DNA条带( 见图3A )。
- 使用计算设计工具识别所需的双靶基因组序列(指南),允许最小的潜在脱靶活动17 。
捐助者模板
- 订购商业合成的单链寡核苷酸(ssOligos,参见材料表 ),用于点突变或用于整合至50 bp的小序列;同源臂通常为60-90 bp长。
- 对于较大的插入,使用供体质粒作为模板。使用经典的克隆方法或从头孢菌素合成生成合适的质粒商业来源。
注意:建议每个同源臂至少800 bp。主干的大小对同源直接修复(HDR)的效率没有影响。
4.注射混合
- 使用不含核酸酶的显微注射缓冲液进行基因敲除实验,将Cas9 mRNA稀释至50ng /μL,sgRNA至12.5ng /μL(总共为25ng /μL)。添加浓度为200 ng /μL的供体模板(ssOligo或质粒),用于基于同源性直接修复的基因组编辑实验。
注意:可以使用在线工具(如重悬浮计算器18)轻松确定稀释量。 - 在显微注射会话期间将每个等分试样保持在冰上,然后丢弃(不要重新冷冻注射组合)。
5.阴道输精管切除术
注意:两种类型的输精管切除术通常在小鼠中进行:腹部和阴囊。后者是较少侵入性的,之前已经描述过15 。
- 高压灭菌所有不锈钢手术器械。
- 使用称重确定小鼠的体重,并用注射麻醉药(氯胺酮100mg / kg,赛拉嗪10mg / kg)腹膜内(ip)注射麻醉麻醉男性。
- 监测脚趾反射的损失,一旦鼠标被镇静放置在加热垫的顶部。
- 用局部消毒剂如氯己定和70%乙醇的交替擦拭来消毒睾丸周围的皮肤。
- 将睾丸推入阴囊,并用手术刀进行皮肤切口。
- 可视化睾丸,睾丸附睾和输精管。
- 用镊子抓住输精管,握住镊子并在镊子的两侧灼烧,以移除3 mm的输精管。
- 对第二睾丸执行相同的程序。
- 用伤口夹闭合皮肤切口并监视鼠标紧密直到完全恢复。
6.超排卵(卵母细胞供体)和时间交配(假怀孕女性)
注意:在其他地方已经描述了产生合适数量的受精卵母细胞和插入寄生雌虫进行再植入的技术。
- 在第1天下午12点左右,注射10只雌性ip与5IU的怀孕母马血清促性腺激素(PMSG;100μL)。
- 在46-48小时后(第3天上午11点左右),用5IU的人绒毛膜促性腺激素(hCG;100μL)注射雌性ip。
- 在一夜之间立即与单身男性交配的女性。
原核(随机整合)和细胞质(基因靶向)注射
- 如先前所述,通过颈部脱位剔除超排卵小鼠,暴露腹部,并进入卵巢和输卵管。解剖o导管并在立体构架仪15下收获卵丘 - 卵母细胞复合物(COCs)。将它们放入一滴补充有氨基酸(KSOMaa)的单纯疱疹优化培养基,按照传统方法15 。
- 通过使用吸气嘴口将近似的1μL透明质酸酶(10mg / mL)添加到KSOMAA培养基的滴中并将培养皿放置在37℃/ 5%CO 2培养箱中30秒至1分钟来净化卵母细胞。
- 使用吸气嘴口用4个新鲜的KSOMAA培养基洗涤卵母细胞,并将其转移到用矿物油(约2mL)覆盖的最后一滴KSOMaa培养基中。将盘放在37°C / 5%CO 2培养箱中,直至卵母细胞准备注射。
- 原核注射(随机整合)。
- 在立体显微镜下可视化鸡蛋,并将大约50个鸡蛋转移到注射室p的M2介质用矿物油覆盖)使用吸气嘴。
- 将注射室转移到倒置显微镜,并注射受精的卵母细胞(两个可见原核)与几个注射液混合物,靶向原核(任何原核可以靶向)。通过观察原核的肿胀来观察成功的注射。
- 细胞质注射(基因靶向)。
- 使用口器将大约50个卵转移到含有5μg/ mL细胞色素B的KSOMaa的一滴,并在37℃/ 5%CO 2培养箱中孵育5分钟。
- 使用嘴巴将鸡蛋转移到注射室。
- 使用自动化显微注射器的补偿压力尽可能在极低压(50-100hPa))将注射混合物的少量皮下注射入细胞质。
- 注射后,将卵母细胞转移到一滴KSO中哇哇哇哇哇哇哇哇哇哇咔咔咔咔咔咔咔咔咔咔咔咔咔咔咔咔咔咔咔咔咔咔咔咔咔咔咔咔咔咔咔咔咔
8.重新植入
- 高压灭菌所有不锈钢手术器械。
- 使用称重确定小鼠的体重,并通过注射麻醉剂(氯胺酮100mg / kg,赛拉嗪10mg / kg)腹膜内(ip)注射雌性麻醉。
- 监测脚趾反射的损失,一旦鼠标被镇静放置在加热垫的顶部。
- 将大部分皮毛围绕在雌鼠的背部中线上。
- 用局部消毒剂如氯己定和70%乙醇的交替擦拭来消毒暴露的皮肤。
- 按照传统方法15暴露生殖道。做一个1厘米长的皮肤切口平行于背部中线,切割肌肉,并抓住脂肪垫wi镊子,然后轻轻拉出卵巢,直到其附着的输卵管和子宫清晰可见。
- 用容器夹固定脂肪垫。在立体显微镜下可视化输卵管,并使用一把微型剪刀在壶腹上游几毫米的输卵管壁上进行切口。
- 在立体显微镜下,将25个微注射的卵装入连接到口部的玻璃毛细管(毛细管的内径应为120μm宽)。将玻璃毛细管引入输卵管并排出直到壶腹内可见气泡。
- 轻轻取出玻璃毛细管并将生殖道放回腹部。用3-0不可吸收的外科缝合线缝合切口,然后用伤口夹闭合。密切监视鼠标直到完全恢复。
基因分型策略/排序
注意:隔离基因组根据相关动物伦理规定,从2毫米尾部或耳朵活组织检查DNA。
- 快速基因组DNA提取(随机整合)。
- 在100μL碱性裂解试剂(25mM氢氧化钠和0.2mM EDTA,pH = 12)中将组织样品(〜2mm)在95℃下裂解1小时。
- 加入100μL中和剂(40 mM Tris-HCl,pH = 5)。
- 在12,000g和4℃下离心5分钟。
- 高质量的基因组DNA提取(基因靶向)。
- 加入500μL尾缓冲液(50mM Tris,pH = 8; 100mM EDTA,pH = 8; 100mM NaCl; 1%SDS和0.5mg / mL蛋白酶K,新鲜加入)。
- 在55℃下孵育过夜。
- 通过反转混合5分钟(不要旋涡)。
- 加入250μL饱和NaCl(6 M),倒置搅拌5分钟(不要旋涡)。
- 在12,000 xg和4℃下旋转5-10分钟,将上清液倒入新管中。
- 加入500μL异丙醇,倒置搅拌5 min(不要旋涡)。
- 在12,000 xg和室温下旋转10分钟。
- 倾倒上清液(颗粒不可见,粘在管上)。
- 用1 mL 70%乙醇洗涤。
- 在12,000 xg和室温下旋转5-10分钟。去除上清液,小心,因为白色颗粒不粘,可以丢失。
- 空气干燥〜1 h。
- 加入400μLTE缓冲液(10mM Tris和1mM EDTA,pH = 8)。
- 将DNA在55-60℃溶解2小时。
- 底漆设计。
- 使用计算工具设计引物至少20 bp长。
注意:对于随机整合,引物应与转基因杂交,产生200-800bp的不同片段。为了基因靶向,设计引物使其与基因组DNA杂交,产生不同的f在切割地点周围数百bp的碎片。对于大的插入,引物可以位于转基因上,但是随后使用引物步行或类似技术来进行靶向整合的确认。
- 使用计算工具设计引物至少20 bp长。
- PCR基因分型。
- 对于每个基因组修饰,设计一个新的PCR方案并经验检验。考虑产生的片段的长度和每对引物的解链温度(Tm)。
- 测序。
- 对于基因定位,将PCR产物发送给Sanger测序服务提供商。
Access restricted. Please log in or start a trial to view this content.
结果
下面描述了在随机整合和CRISPR介导的基因靶向的情况下显微注射的工作流程( 图1 )。
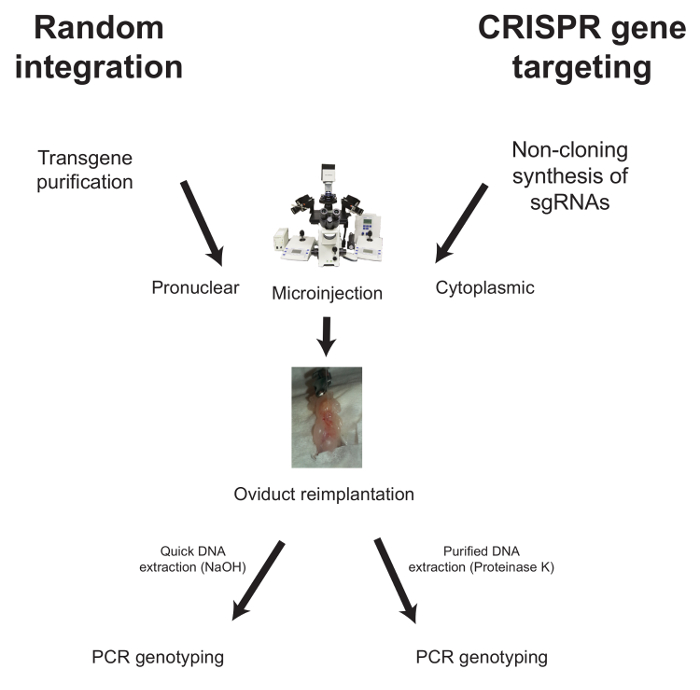
图1:产生转基因小鼠的典型工作流程。对于随机整合,将纯化的转基因注射入受精卵母细胞的原核,然后输卵管转移到插入的寄养雌性中。?...
Access restricted. Please log in or start a trial to view this content.
讨论
协议中的关键步骤
已知基因修饰的小鼠的产生在技术上是具有挑战性的。然而,这里提出的协议是一种优化和简化的方法,可以在记录时间内掌握和排除故障。成功完成技术需要两个步骤。首先,不用氯化镁(MgCl 2 )可以实现线性DNA模板的合成(用于合成sgRNA)。然而,强烈建议系统地将MgCl 2加入到主混合物中,因为在没有MgCl 2的情况下经常防止正向引...
Access restricted. Please log in or start a trial to view this content.
披露声明
作者通过新南威尔士大学Mark Wainwright分析中心为小鼠提供学术转基因服务。
致谢
作者感谢动物设施(BRC)的工作人员不断的支持。这项工作由国家卫生和医学研究理事会和澳大利亚研究理事会资助。
Access restricted. Please log in or start a trial to view this content.
材料
| Name | Company | Catalog Number | Comments |
| Micropipette 0.1-2.5 μL | Eppendorf | 4920000016 | |
| Micropipette 2 - 20 μL | Eppendorf | 4920000040 | |
| Micropipette 20 - 200 μL | Eppendorf | 4920000067 | |
| Micropipette 100 - 1,000 μL | Eppendorf | 4920000083 | |
| Molecular weight marker | Bioline | BIO-33025 | HyperLadder 1 kb |
| Molecular weight marker | Bioline | BIO-33056 | HyperLadder 100 bp |
| Agarose | Bioline | BIO-41025 | |
| EDTA buffer | Sigma-Aldrich | 93296 | 10x - Dilute to 1x |
| Ethidium bromide | Thermo Fisher Scientific | 15585011 | |
| SYBR Safe gel stain | Invitrogen | S33102 | |
| Gel extraction kit | Qiagen | 28706 | |
| PCR purification kit (Qiaquick) | Qiagen | 28106 | |
| Vacuum system (Manifold) | Promega | A7231 | |
| Nuclease-free microinjection buffer | Millipore | MR-095-10F | |
| Ultrafree-MC microcentrifuge filter | Millipore | UFC30GV00 | |
| Cas9 mRNA | Sigma-Aldrich | CAS9MRNA | |
| CRISPR expressing plasmid (px330) | Addgene | 42230 | |
| Nuclease free water | Sigma-Aldrich | W4502 | |
| Phusion polymerase | New England Biolabs | M0530L | |
| T7 Quick High Yield RNA kit | New England Biolabs | E2050S | |
| RNA purification spin columns (NucAway) | Thermo Fisher Scientific | AM10070 | |
| ssOligos | Sigma-Aldrich | OLIGO STANDARD | |
| Donor plasmid | Thermo Fisher Scientific | GeneArt | |
| Hyaluronidase | Sigma-Aldrich | H3884 | |
| KSOMaa embryo culture medium | Zenith Biotech | ZEKS-100 | |
| Mineral oil | Zenith Biotech | ZSCO-100 | |
| M2 Medium | Sigma-Aldrich | M7167 | |
| Cytochalasin B | Sigma-Aldrich | C6762 | |
| Mouthpiece | Sigma-Aldrich | A5177 | |
| Glass microcapillaries | Sutter Instrument | BF100-78-10 | |
| Proteinase K | Applichem | A3830.0100 | |
| Dumont #5 forceps | Fine Science Tools | 91150-20 | |
| Iris scissors | Fine Science Tools | 91460-11 | |
| Vessel clamp | Fine Science Tools | 18374-43 | |
| Wound clips | Fine Science Tools | 12040-01 | |
| Clips applier | Fine Science Tools | 12018-12 | |
| Micro-scissors | Fine Science Tools | 15000-03 | |
| Cauterizer | Fine Science Tools | 18000-00 | |
| Non-absorbable surgical sutures (Ethilon 3-0) | Ethicon | 1691H | |
| 5% CO2 incubator | MG Scientific | Galaxy 14S | |
| Spectrophotometer | Thermo Fisher Scientific | Nanodrop 2000c | |
| Thermocycler | Eppendorf | 6321 000.515 | |
| Electrophoresis set up | BioRad | 1640300 | |
| UV Transilluminator | BioRad | 1708110EDU | |
| Thermocycler | Eppendorf | 6334000069 | |
| Stereoscopic microscope | Olympus | SZX7 | |
| Inverted microscope | Olympus | IX71 | |
| 2x Micromanipulators | Eppendorf | 5188000.012 | |
| Oocytes manipulator | Eppendorf | 5176000.025 | |
| Microinjector (Femtojet) | Eppendorf | 5247000.013 | |
| Mice C57BL/6J strain | Australian BioResources | C57BL/6JAusb |
参考文献
- Ittner, L. M. Dendritic function of tau mediates amyloid-beta toxicity in Alzheimer's disease mouse models. Cell. 142 (3), 387-397 (2010).
- Ittner, A. Site-specific phosphorylation of tau inhibits amyloid-beta toxicity in Alzheimer's mice. Science. 354 (6314), 904-908 (2016).
- Marantz Henig, R. The Monk in the Garden. , Mariner books. (2000).
- Jaenisch, R., Mintz, B. Simian virus 40 DNA sequences in DNA of healthy adult mice derived from preimplantation blastocysts injected with viral DNA. Proc Natl Acad Sci U S A. 71 (4), 1250-1254 (1974).
- Gordon, J. W., Ruddle, F. H. Integration and stable germ line transmission of genes injected into mouse pronuclei. Science. 214 (4526), 1244-1246 (1981).
- Gordon, J. W., Scangos, G. A., Plotkin, D. J., Barbosa, J. A., Ruddle, F. H. Genetic transformation of mouse embryos by microinjection of purified DNA. Proc Natl Acad Sci U S A. 77 (12), 7380-7384 (1980).
- Brinster, R. L., Chen, H. Y., Trumbauer, M. E., Yagle, M. K., Palmiter, R. D. Factors affecting the efficiency of introducing foreign DNA into mice by microinjecting eggs. Proc Natl Acad Sci U S A. 82 (13), 4438-4442 (1985).
- Capecchi, M. R. Gene targeting in mice: functional analysis of the mammalian genome for the twenty-first century. Nat Rev Genet. 6 (6), 507-512 (2005).
- Carbery, I. D. Targeted genome modification in mice using zinc-finger nucleases. Genetics. 186 (2), 451-459 (2010).
- Sung, Y. H. Knockout mice created by TALEN-mediated gene targeting. Nat Biotechnol. 31 (1), 23-24 (2013).
- Jinek, M. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science. 337 (6096), 816-821 (2012).
- Wang, H. One-step generation of mice carrying mutations in multiple genes by CRISPR/Cas-mediated genome engineering. Cell. 153 (4), 910-918 (2013).
- Kang, X. Introducing precise genetic modifications into human 3PN embryos by CRISPR/Cas-mediated genome editing. J Assist Reprod Genet. 33 (5), 581-588 (2016).
- Liang, P. CRISPR/Cas9-mediated gene editing in human tripronuclear zygotes. Protein Cell. 6 (5), 363-372 (2015).
- Ittner, L. M., Götz, J. Pronuclear injection for the production of transgenic mice. Nat Protoc. 2 (5), 1206-1215 (2007).
- Yang, H., Wang, H., Jaenisch, R. Generating genetically modified mice using CRISPR/Cas-mediated genome engineering. Nat Protoc. 9 (8), 1956-1968 (2014).
- CRISPR DESIGN. , Available from: http://crispr.mit.edu (2017).
- Integrated DNA Technologies. Resuspension Calculator. , Available from: https://sg.idtdna.com/calc/resuspension/ (2017).
- BioTools at UMass Medical School. Primer3: WWW primer tool. , Available from: http://biotools.umassmed.edu/bioapps/primer3_www.cgi (2017).
- Auerbach, A. B. Strain-dependent differences in the efficiency of transgenic mouse production. Transgenic Res. 12 (1), 59-69 (2003).
- Merriman, J. A., Jennings, P. C., McLaughlin, E. A., Jones, K. T. Effect of aging on superovulation efficiency, aneuploidy rates, and sister chromatid cohesion in mice aged up to 15 months. Biol Reprod. 86 (2), 49(2012).
- Nakagawa, Y., et al. Ultra-superovulation for the CRISPR-Cas9-mediated production of gene-knockout, single-amino-acid-substituted, and floxed mice. Biol Open. 5 (8), 1142-1148 (2016).
- Byers, S. L., Wiles, M. V., Dunn, S. L., Taft, R. A. Mouse estrous cycle identification tool and images. PLoS One. 7 (4), e35538(2012).
- Whitten, W. K. Modification of the oestrous cycle of the mouse by external stimuli associated with the male. J Endocrinol. 13 (4), 399-404 (1956).
- Ye, S., Dhillon, S., Ke, X., Collins, A. R., Day, I. N. An efficient procedure for genotyping single nucleotide polymorphisms. Nucleic Acids Res. 29 (17), E88(2001).
- Yoshimi, K., et al. ssODN-mediated knock-in with CRISPR-Cas for large genomic regions in zygotes. Nat Commun. 7, 10431(2016).
- Suzuki, K., et al. In vivo genome editing via CRISPR/Cas9 mediated homology-independent targeted integration. Nature. , (2016).
- Soriano, P. Generalized lacZ expression with the ROSA26 Cre reporter strain. Nat Genet. 21, 70-71 (1999).
- Liu, X., et al. A targeted mutation at the known collagenase cleavage site in mouse type I collagen impairs tissue remodeling. J Cell Biol. 130 (1), 227-237 (1995).
- Ke, Y. D. Short-term suppression of A315T mutant human TDP-43 expression improves functional deficits in a novel inducible transgenic mouse model of FTLD-TDP and ALS. Acta Neuropathol. 130 (5), 661-678 (2015).
- Auerbach, A. B. Production of functional transgenic mice by DNA pronuclear microinjection. Acta Biochim Pol. 51 (1), 9-31 (2004).
- Iyer, V., et al. Off-target mutations are rare in Cas9-modified mice. Nat Methods. 12 (6), 479(2015).
- Zhou, Y. High-throughput screening of a CRISPR/Cas9 library for functional genomics in human cells. Nature. 509 (7501), 487-491 (2014).
- Brinster, R. L., Chen, H. Y., Trumbauer, M. E., Avarbock, M. R. Translation of globin messenger RNA by the mouse ovum. Nature. 283 (5746), 499-501 (1980).
- Pinkert, C. A., Irwin, M. H., Johnson, L. W., Moffatt, R. J. Mitochondria transfer into mouse ova by microinjection. Transgenic Res. 6 (6), 379-383 (1997).
- Biggers, J. D., Summers, M. C. Choosing a culture medium: making informed choices. Fertil Steril. 90 (3), 473-483 (2008).
- Nakagata, N. Embryo transfer through the wall of the fallopian tube in mice. Jikken Dobutsu. 41 (3), 387-388 (1992).
- Truett, G. E. Preparation of PCR-quality mouse genomic DNA with hot sodium hydroxide and tris (HotSHOT). Biotechniques. 29 (1), 52-54 (2000).
- Richardson, C. D., Ray, G., DeWitt, M. A., Curie, G. L., Corn, J. E. Enhancing homology-directed genome editing by catalytically active and inactive CRISPR-Cas9 using asymmetric donor DNA. Nat Biotechnol. 34 (3), 339-344 (2016).
- Delerue, F., Ittner, L. M. Genome Editing in Mice Using CRISPR/Cas9: Achievements and Prospects. Clon. Transgen. 4, (2015).
Access restricted. Please log in or start a trial to view this content.
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。