Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Production de souris génétiquement modifiées par micro-injection d'ovocytes
Dans cet article
Résumé
La micro-injection d'ovocytes de souris est couramment utilisée à la fois pour la transgénèse classique ( c.-à-d. L'intégration aléatoire des transgènes) et le ciblage des gènes médié par CRISPR. Ce protocole examine les derniers développements en micro-injection, avec un accent particulier sur le contrôle de la qualité et les stratégies de génotypage.
Résumé
L'utilisation de souris génétiquement modifiées a contribué de manière significative aux études sur les processus physiologiques et pathologiques in vivo . L'injection pronucléaire des constructions d'expression d'ADN dans les ovocytes fécondés reste la technique la plus couramment utilisée pour générer des souris transgéniques pour une surexpression. Avec l'introduction de la technologie CRISPR pour le ciblage des gènes, l'injection pronucléaire dans les ovocytes fécondés a été étendue à la génération de souris knock-out et knockin. Ce travail décrit la préparation de l'ADN pour l'injection et la génération de guides CRISPR pour le ciblage des gènes, en mettant l'accent sur le contrôle de la qualité. Les procédures de génotypage nécessaires à l'identification des fondateurs potentiels sont essentielles. Des stratégies novatrices de génotypage qui profitent des capacités de "multiplexage" de CRISPR sont présentées ici. Les procédures chirurgicales sont également décrites. Ensemble, les étapes du protocole permettront la génération de genDes souris modifiées de manière sélective et pour l'établissement ultérieur de colonies de souris pour une pléthore de domaines de recherche, y compris l'immunologie, les neurosciences, le cancer, la physiologie, le développement et d'autres.
Introduction
Les modèles animaux, chez les vertébrés et les invertébrés, ont contribué à l'examen de la pathophysiologie des affections humaines telles que la maladie d'Alzheimer 1 , 2 . Ils sont également des outils précieux pour rechercher des modificateurs de maladies et pour finalement développer de nouvelles stratégies de traitement dans l'espoir d'un remède. Bien que chaque modèle ait des limites intrinsèques, l'utilisation des animaux comme modèles systémiques complets est essentielle à la recherche biomédicale. C'est parce que l'environnement physiologique métabolique et complexe ne peut pas être entièrement simulé dans la culture tissulaire.
À ce jour, la souris reste l'espèce de mammifère la plus commune utilisée pour la manipulation génétique car elle présente plusieurs avantages. Les processus physiologiques et les gènes associés aux maladies sont fortement conservés chez les souris et les humains. La souris a été le premier mammifère à avoir son génome complet séquencé (2002), un an avant le génome humainMoi (2003). Outre cette richesse de l'information génétique, la souris possède de bonnes capacités d'élevage, un cycle de développement rapide (6 semaines après la fertilisation jusqu'au sevrage) et une taille raisonnable. Tous ces avantages, couplés à des indicateurs physiologiques, tels que des couleurs de revêtement distinctes (requis pour les stratégies de croisement), ont fait de la souris un modèle attrayant pour la manipulation génétique. Notamment, au début de la génétique moderne, Gregor Mendel a commencé à travailler sur des souris avant de passer à des plantes 3 .
Les techniques de transfert de gènes ont entraîné la génération de la première souris transgénique il y a trois décennies 4 , initialement créée à l'aide de l'administration virale. Cependant, les chercheurs ont rapidement compris que l'un des principaux défis de la transgénèse de la souris était l'incapacité de contrôler le devenir de l'ADN exogène. Parce que l'administration virale de transgènes dans des ovocytes de souris a abouti à des copies multiples intégrées au hasard dans le génome, la possibilitéY d'établir des lignes transgéniques ultérieures était limité.
Une telle limitation a été surmontée lorsque Gordon et al. A généré la première ligne de souris transgénique par microinjection 5 , 6 . Cela a commencé l'ère de la technologie de l'ADN recombinant, et les paramètres influençant le résultat d'une session de micro-injection ont été largement étudiés 7 . Bien que la micro-injection ne permette pas le contrôle sur le site d'intégration du transgène (qui aboutit finalement à des niveaux d'expression spécifiques pour chaque souris fondatrice), l'avantage principal de la microinjection pronucléaire reste la formation de concatemers ( c.-à-d. Des tableaux de multiples copies du transgène, Liés en série) avant intégration génomique 5 . Cette caractéristique a été utilisée au cours des années pour établir des milliers de lignes de souris transgéniques qui surexpriment un gène d'intérêt. Depuis lors, la transgénèse, la aModification artificielle du génome d'un organisme, a été largement utilisée pour identifier le rôle des gènes simples dans l'apparition de maladies.
Une autre réalisation clé dans la manipulation du génome de la souris a été atteinte lorsque Mario Capecchi a perturbé avec succès un seul gène à la souris, ouvrant l'ère du ciblage des gènes 8 . Cependant, des inconvénients majeurs ont rapidement émergé du ciblage génétique basé sur les cellules ES, y compris les défis de la culture des cellules ES, le degré de chimérisme quelque peu variable et la durée du processus ( c'est-à-dire 12-18 mois minimum pour obtenir la souris) .
Récemment, les progrès dans les nouvelles technologies, telles que les endonucléases modifiées ( p. Ex., Les nucléases de doigts de zinc (ZFN), les nucléases effectrices de l'activateur de la transcription (TALEN) et les répétitions palindromiques courtes (CRISPR / Cas9) classées régulièrement sont apparues comme des méthodes alternatives Accélérer le processus de ciblage des gènes dans le microE 9 , 10 . Ces endonucléases peuvent être facilement injectées dans des ovocytes de souris par micro-injection, ce qui permet de générer des souris ciblées par gène en 6 semaines seulement.
Depuis le premier rapport sur l'utilisation de CRISPR pour l'édition génomique 11 , ce système immunitaire adaptatif bactérien a remplacé ZFN et TALEN en raison de ses nombreux avantages, y compris la facilité de la synthèse et la capacité de cibler de multiples loci à la fois (appelés "multiplexage" "). Le CRISPR a d'abord été utilisé pour le ciblage des gènes chez la souris 12 et a depuis été appliqué à d'innombrables espèces, des plantes aux humains 13 , 14 . À ce jour, aucun rapport d'une seule espèce n'est résistant à l'édition du génome CRISPR.
Les deux principales étapes limitantes de la génération de souris transgéniques sont l'injection d'ovocytes et la réimplantationDe ces ovocytes chez des femelles pseudo-grossières. Bien que cette technique ait été décrite par nous 15 et d'autres 16 , des améliorations techniques récentes dans l'embryologie de souris et les techniques de transfert de gènes ont révolutionné le processus de génération de souris génétiquement modifiées. Ces améliorations seront décrites ici.
Protocole
Toutes les procédures ont été approuvées par le Comité des soins et de l'éthique des animaux de l'Université de New-Galles du Sud.
1. Préparation du transgène (intégration aléatoire)
- Électrophorèse analytique en gel d'agarose.
- Décrivez le plasmide pour acciser le transgène en utilisant des enzymes appropriées (1 heure d'incubation) ou des enzymes à digestion rapide (15 à 30 minutes d'incubation) dans un thermocycleur en suivant les recommandations du fabricant (voir la figure 2A et sa légende).
- Mélanger un gel d'agarose à 1% tris-acétate-éthylènediaminetétraacétique (EDTA) (TEA), coloré avec 0,5 à 1,0 μg / ml de bromure d'éthidium (EtBr).
REMARQUE: faites preuve de prudence lorsque vous utilisez EtBr; C'est un puissant mutagène. Utilisez un équipement de protection individuelle approprié (reportez-vous à la fiche technique de sécurité). - Chargez un marqueur de masse moléculaire de 1 kb.
- Chargez les fragments linéarisés ( c'est-à-dire le transgène et le squelette).
- Faire fonctionner l'électrophorèse à 100 V pendant 45 minutes.
- Visualisez le gel sur un transilluminateur ultraviolet (UV) pour vérifier que la digestion est terminée et pour confirmer la taille correcte du transgène ( c.-à-d., 7 338 pb, voir Figure 2A ).
- Électrophorèse préparative en gel d'agarose.
- Lancer un gel d'agarose TAE à 1% coloré avec une tache de gel à faible toxicité. Chargez 8 portions aliquotes (1 μg chacune) de transgène linéarisé sans marqueur de poids moléculaire et effectuez l'électrophorèse à 100 V pendant 45 minutes. Utilisez un scalpel pour acciser toutes les 8 bandes correspondant au transgène linéarisé.
- Extrayez l'ADN en utilisant un kit d'extraction de gel en suivant les recommandations du fabricant (voir la Table des matériaux ).
- Faire fondre les fragments de gel à 50 ° C dans des tubes de 1,5 mL pendant au moins 15 minutes. Précipiter avec de l'isopropanol puis ajouter le tampon de liaison.
- Combinez l'ensemble de la DNA en pipettant tout dans une colonne. Eluer dans un tampon de microinjection sans nucléase (Tris-HCl 8 mM et EDTA 0,15 mM). Filtrer par un filtre à microcentrifugeuse de 0,22 μm et centrifuger pendant 60 s à 12 000 x g.
- Mesurer la concentration et la qualité de l'ADN à l'aide d'un spectrophotomètre. Vérifiez que le rapport A260 / A280 est d'environ 1.8 ( c.-à-d., Pas de contamination par les protéines) et le rapport A260 / A230 est d'au moins 2,0 ( c.-à-d. Pas de contamination par les solvants organiques). Diluez jusqu'à 3 ng / μL dans un tampon de microinjection sans nucléase, une aliquote (20-50 μL) et gèlez à -20 ° C.
2. Synthèse des composants CRISPR (ciblage des gènes)
- Cas9 mRNA.
- Diluer l'ARNm Cas9 (obtenu à partir d'une source commerciale, voir la Table des matériaux ) à une concentration de 1 μg / μL dans un tampon de microinjection sans nucléase.
- Aliquote dans 2 μL et libre Ze à -80 ° C.
- RNA à guide unique (SgRNA).
- Identifiez les séquences génomiques à deux cibles souhaitées (guides) en utilisant un outil de conception de calcul permettant une activité potentiellement hors cible hors potentiel 17 .
- Pour le knockout génique, choisissez systématiquement deux guides situés à quelques centaines de paires de bases (bp), dans des directions opposées, et incluent le codon de départ du gène d'intérêt. Pour une réparation directe d'homologie, sélectionnez deux guides qui se chevauchent (dans la mesure du possible), dans des directions opposées.
REMARQUE: à titre d'exemple, les guides suivants ont récemment été co-injectés avec succès pour perturber le premier exon du gène TPM4.2 :
Guide 1: 5 'CGCCATCCAGTTCGCGCTGC 3';
Guide 2: 5 'CAGAACGATTGAGCTATGGC 3'
- Pour le knockout génique, choisissez systématiquement deux guides situés à quelques centaines de paires de bases (bp), dans des directions opposées, et incluent le codon de départ du gène d'intérêt. Pour une réparation directe d'homologie, sélectionnez deux guides qui se chevauchent (dans la mesure du possible), dans des directions opposées.
- Commandez un ensemble d'amorces comme décrit dans le tableau 1 .
- Synthèse d'un modèle d'ADN linéaire.
- Diluer px330Ref "> 12 plasmide à 10 ng / μL dans de l'eau exempte de nuclease.
- Préparez le mélange maître comme indiqué dans le tableau 1 . Ajouter la polymérase à la fin et garder le mélange maître sur de la glace.
- Mélanger et diviser cela en 8 tubes PCR (19 μL / tube). Ajouter 1 μL de px330 par tube et exécuter la PCR dans les conditions suivantes: 1 min à 98 ° C; 40 cycles de 98 ° C pendant 10 s, 64 ° C pendant 30 s et 72 ° C pendant 15 s; Et un allongement final à 72 ° C pendant 5 min. Maintenir à 4 ° C.
- Purifier les produits de PCR en utilisant un kit de purification par PCR (voir la Table des matériaux ), un collecteur et une source de vide (pour un traitement plus rapide). Appliquer la force maximale de l'aspirateur ( c'est-à-dire 8 mbar).
- Ajouter 5 volumes (100 μL) de tampon de liaison à 1 volume de l'échantillon de PCR et mélanger.
- Placez une colonne de spin de la membrane de silice (fournie) dans un tube de collecte (fourni) de 2 ml pour la centrifugation ou sur le collecteur pour le processus sous videIng. Pour lier l'ADN, appliquer successivement tous les 8 échantillons à la colonne et aspirer ou centrifuger pendant 60 s à 12 000 xg pour chaque charge.
- Pour laver, ajouter 0,75 ml de tampon de lavage (tampon PE) à la colonne et aspirater ou centrifuger pendant 60 s à 12 000 x g. Centrifuger la colonne pendant 60 s à 12 000 xg pour éliminer l'éthanol résiduel.
- Placez la colonne dans un tube de microcentrifugeuse propre de 1,5 ml. Pour éluer l'ADN, ajouter 30 μL d'eau exempte de nuclease au centre de la membrane, laisser reposer la colonne pendant 1 min et centrifuger pendant 60 s à 12 000 g.
- Mesurer la concentration en utilisant un spectrophotomètre; Généralement, la concentration devrait être de 100 ng / μL ou plus. Vérifiez que le rapport A260 / A280 est d'environ 1.8 ( c.-à-d., Pas de contamination par les protéines) et le rapport A260 / A230 est d'au moins 2,0 ( c.-à-d. Pas de contamination par les solvants organiques).
- Transcription in vitro utilisant un kit de synthèse d'ARN T7.
- Préparez-vousIl maîtrise le mélange, comme indiqué dans le tableau 1 , et incube pendant 3 h à 37 ° C dans un thermocycleur. Ajouter 28 μL d'eau libre de nuclease et 2 μL de DNase I et incuber pendant encore 15 minutes à 37 ° C dans un thermocycleur.
- Purification d'ARN à l'aide de colonnes de spin.
- Dissoudre la poudre contenue dans la colonne de spin fournie dans 650 μL de tampon de microinjection sans nuclease, en éliminant soigneusement toutes les bulles d'air. Capter le tube et hydrater pendant 5 à 15 min à température ambiante.
- Retirez le capuchon bleu en bas et placez la colonne dans un tube de 2 ml. Centrifuger pendant 2 min à 750 xg et la température ambiante. Placez la colonne dans un tube frais de 1,5 ml et appliquez 50 μL de la solution d'ARN dans le centre, sans toucher la paroi de la colonne. Faire tourner la colonne pendant 2 min à 750 x g.
- Mesurer la concentration d'ARN à l'aide d'un spectrophotomètre (le rendement typique d'une réaction est de 30 à 50 μg). Vérifiez que le rapport A260 / A280 est unLe cycle 2.0 ( c.-à-d. Pas de contamination par les protéines) et le rapport A260 / A230 est d'au moins 2,0 ( c.-à-d. Pas de contamination par les solvants organiques). Rangez les sgRNA à -80 ° C jusqu'à l'utilisation.
- Évaluer la qualité de l'ARN en utilisant les gels TAE à 1% habituellement utilisés pour l'électrophorèse de l'ADN. Exécutez 200-400 ng du modèle d'ADN et le sgRNA correspondant en parallèle. La bande d'ARN (≈ 100 pb) devrait être légèrement plus grande que la bande d'ADN (voir la figure 3A ).
- Identifiez les séquences génomiques à deux cibles souhaitées (guides) en utilisant un outil de conception de calcul permettant une activité potentiellement hors cible hors potentiel 17 .
3. Modèle de donateur
- Commander des oligonucleotides monocaténaires synthétisés dans le commerce (ssOligos, voir la Table des matériaux ) pour des mutations ponctuelles ou pour l'intégration de petites séquences jusqu'à 50 pb; Les bras d'homologie sont généralement de 60 à 90 paires de long.
- Pour des insertions plus importantes, utilisez un plasmide donneur comme modèle. Générer un plasmide approprié en utilisant soit la méthode classique de clonage, soit la synthèse de novo à partir d'unSource commerciale.
REMARQUE: Un minimum de 800 pb par bras d'homologie est recommandé. La taille de l'épine dorsale n'a aucune influence sur l'efficacité de la réparation directe d'homologie (HDR).
4. Mélange d'injection
- Diluer l'ARNm de Cas9 à 50 ng / μL et les sgRNAs à 12,5 ng / μL (25 ng / μL au total) en utilisant un tampon de microinjection sans nucléase pour les expériences de knock-out du gène. Ajouter le modèle de donneur (ssOligo ou plasmide) à une concentration de 200 ng / μL pour des expériences d'édition de génome basées sur la réparation directe d'homologie.
REMARQUE: Les volumes de dilution peuvent être facilement déterminés à l'aide d'outils en ligne, tels qu'un calculateur de remise en suspension 18 . - Conservez chaque aliquote sur la glace lors de la séance de microinjection et jettez ensuite (ne pas congeler le mélange d'injection).
5. Vasectomie scrotale
NOTE: Deux types de vasectomie sont fréquemment utilisés chez les souris: abdominaux et scrotales. Le dernierEst moins invasive et a déjà été décrite 15 .
- Autoclave tous les instruments chirurgicaux en acier inoxydable.
- Déterminer le poids de la souris à l'aide d'une balance de pesée et anesthésier les mâles par injection intrapéritonéale (IP) avec des anesthésiques injectables (kétamine 100 mg / kg, xylazine 10 mg / kg).
- Surveillez la perte de réflexe de pincement et une fois que la souris est sédée, placez-la sur un coussin chauffant.
- Désinfectez la peau autour des testicules avec des lingettes alternées d'un antiseptique topique tel que la chlorhexidine et 70% d'éthanol.
- Empêcher les testicules dans le sac scrotale et faire une incision cutanée avec un scalpel.
- Visualisez les testicules, la cauda epididyme et le canal déférent.
- Prenez le canal déférent avec une pince, maintenez-le et cautérisez de chaque côté de la pince pour enlever 3 mm du canal déférent.
- Effectuez la même procédure sur le deuxième testicule.
- Fermez les incisions cutanées avec des clips enroulés et surveillez lesLa souris étroitement jusqu'à la récupération complète.
6. Superovulation (donneurs d'ovocytes) et accouplement temporel (femelles pseudo-enceintes)
NOTE: La technique pour générer un nombre approprié d'ovocytes fécondés et de femelles adoptives branchées pour la réimplantation a été décrite ailleurs 15 .
- Injecter 10 femelles ip avec 5 UI de gonadotrophine sérique de jument enceinte (PMSG, dans 100 μL) vers 12 heures le jour 1.
- Injecter les femelles ip avec 5 UI de gonadotrophine chorionique humaine (hCG, dans 100 μL) 46-48 h plus tard (environ 11 heures le jour 3).
- Entraînez immédiatement les femelles avec des mâles à un seul logement pendant la nuit.
7. Injections pronucléaires (intégration aléatoire) et cytoplasmiques (ciblage par voie génétique)
- Culler les souris superovulées par dislocation cervicale, exposer l'abdomen et accéder aux ovaires et oviductes, comme décrit précédemment 15 . Dissectionnez le oÉvacue et récolte les cumulus-ovocytes-complexes (COC) sous un stéréomicorscope 15 . Placez-les dans une goutte de milieu d'optimisation de potassium simplex complétée par des acides aminés (KSOMaa) selon la méthode traditionnelle 15 .
- Purifier les ovocytes en ajoutant approximativement 1 μl d'hyaluronidase (10 mg / mL) à la goutte du milieu KSOMaa en utilisant une embouchure aspirante et placer le plat dans un incubateur de CO 2 à 37 ° C / 5% pendant 30 s à 1 min.
- Lavez les ovocytes avec 4 gouttes fraîches de milieu KSOMaa à l'aide de la bouche aspirante et transférez-les à une dernière goutte de milieu KSOMaa recouvert d'huile minérale (environ 2 ml). Placez le plat dans l'incubateur à 37 ° C / 5% CO 2 jusqu'à ce que les ovocytes soient prêts à l'injection.
- Injection pronucléaire (intégration aléatoire).
- Visualiser les œufs sous microscope stéréoscopique et transférer environ 50 oeufs dans la chambre d'injection (une droP de milieu M2 recouvert d'huile minérale) à l'aide de la bouche aspirante.
- Transférer la chambre d'injection au microscope inversé et injecter les ovocytes fertilisés (deux pronucléés visibles) avec quelques picolitres du mélange d'injection, ciblant un pronucleus (l'un des pronuclei peut être ciblé). Visualisez une injection réussie en observant l'enflure du pronucleus.
- Injection cytoplasmique (ciblage des gènes).
- Transférer environ 50 oeufs à une goutte de KSOMaa contenant 5 μg / mL de cytochalasine B en utilisant la bouche et incuber pendant 5 min dans l'incubateur à 37 ° C / 5% CO 2 .
- Transférer les oeufs dans la chambre d'injection à l'aide de la bouche.
- Injecter quelques picolitres du mélange d'injection dans le cytoplasme à très basse pression (50-100 hPa), en utilisant la pression de compensation du micro-injecteur automatisé dans la mesure du possible.
- Après l'injection, transférez les ovocytes dans une goutte de KSOMaa (à l'aide de la bouche) et les garder dans l'incubateur à 37 ° C / 5% de CO 2 , jusqu'à ce qu'ils soient chargés pour la réimplantation dans l'oviducte de femelles pseudo-enceintes.
8. Réimplantation
- Autoclave tous les instruments chirurgicaux en acier inoxydable.
- Déterminer le poids de la souris à l'aide de l'échelle de pesée et anesthésier les femelles par injection intrapéritonéale (IP) avec des anesthésiques injectables (kétamine 100 mg / kg, xylazine 10 mg / kg).
- Surveillez la perte de réflexe de pincement et une fois que la souris est sédée, placez-la sur un coussin chauffant.
- Accrochez une grande zone de la fourrure autour de la ligne médiane dorsale de la souris femelle.
- Désinfectez la peau exposée avec des lingettes alternées d'un antiseptique topique tel que la chlorhexidine et l'éthanol à 70%.
- Exposer le tractus reproducteur selon la méthode traditionnelle 15 . Faire une incision de la peau de 1 cm de long parallèlement à la ligne médiane dorsale, couper le muscle et saisir le coussinet grasUne pince, puis tirez doucement l'ovaire jusqu'à ce que son oviduct et son utérus soient clairement visibles.
- Fixez le coussinet gras avec une pince à vaisseau. Visualiser l'oviducte sous microscope stéréoscopique et utiliser une paire de micro-ciseaux font une incision dans la paroi de l'oviduct quelques millimètres en amont de l'ampoule.
- Sous le microscope stéréoscopique, charger 25 oeufs micro-injectés dans le capillaire en verre relié à la bouche (le diamètre intérieur du capillaire devrait avoir une largeur d'environ 120 μm). Introduire le capillaire en verre dans l'oviducte et expulser jusqu'à ce qu'une bulle d'air soit visible à l'intérieur de l'ampoule.
- Retirez doucement le capillaire en verre et placez le tractus reproducteur dans l'abdomen. Suture l'incision avec des sutures chirurgicales 3-0 non absorbables, puis fermez-les avec des clips enroulés. Surveillez la souris de près jusqu'à la récupération complète.
9. Stratégies de génotypage / séquençage
REMARQUE: Isoler le génomiqueL'ADN provenant de biopsies de queue ou d'oreille de 2 mm, conformément à la réglementation sur l'éthique des animaux.
- Extraction rapide d'ADN génomique (intégration aléatoire).
- Lyse les échantillons de tissus (~ 2 mm) à 95 ° C pendant 1 h dans 100 μl de réactif de lyse alcalin (hydroxyde de sodium 25 mM et EDTA 0,2 mM, pH = 12).
- Ajouter 100 μl de réactif neutralisant (Tris-HCl 40 mM, pH = 5).
- Centrifugez pendant 5 min à 12 000 g et 4 ° C.
- Extraction d'ADN génomique de haute qualité (ciblage des gènes).
- Ajouter 500 μl de tampon de queue (50 mM de Tris, pH = 8, 100 mM d'EDTA, pH = 8, 100 mM de NaCl, 1% de SDS et 0,5 mg / mL de protéinase K, fraîchement ajoutée).
- Incuber pendant une nuit à 55 ° C.
- Mélanger pendant 5 minutes en inversant (pas de vortex).
- Ajouter 250 μl de NaCl saturé (6 M) et mélanger pendant 5 minutes en inversant (ne pas tourbillonner).
- Faire tourner pendant 5 à 10 min à 12 000 xg et 4 ° C et verser le surnageant dans un nouveau tube.
- Ajouter 500 μl d'isopropanol et mélanger pendant 5 minutes en inversant (ne pas tourbillonner).
- Faire tourner pendant 10 min à 12 000 xg et température ambiante.
- Décanter le surnageant (la pastille est invisible et colle au tube).
- Laver avec 1 ml d'éthanol à 70%.
- Tournez pendant 5-10 min à 12 000 xg et température ambiante. Retirez le surnageant avec précaution, car la pastille blanche n'est plus collante et peut être perdue.
- Séchez l'air pendant environ 1 h.
- Ajouter 400 μl de tampon TE (Tris 10 mM et EDTA 1 mM, pH = 8).
- Dissoudre l'ADN à 55-60 ° C pendant 2 h.
- Design d'amorçage.
- Conception d'amorces d'un minimum de 20 pb de long à l'aide d'un outil de calcul 19 .
REMARQUE: pour l'intégration aléatoire, les amorces doivent s'hybrider avec le transgène, générant un fragment distinct de 200 à 800 pb. Pour le ciblage des gènes, concevez les amorces afin qu'elles s'hybrident avec l'ADN génomique, générant une f distincteRamenant quelques centaines de pixels autour des sites coupés. Pour les grandes insertions, les amorces peuvent s'asseoir sur le transgène, mais la confirmation de l'intégration ciblée devrait ensuite être effectuée à l'aide de la marche à l'avant ou d'une technique similaire.
- Conception d'amorces d'un minimum de 20 pb de long à l'aide d'un outil de calcul 19 .
- Génotypage par PCR.
- Pour chaque modification génomique, concevez un nouveau protocole de PCR et testez-le de manière empirique. Considérons la longueur du fragment généré et la température de fusion (Tm) de chaque paire d'amorces.
- Séquençage.
- Pour le ciblage des gènes, envoyez des produits de PCR à un fournisseur de services de séquençage Sanger.
Résultats
Ci-dessous, les flux de travail pour la micro-injection dans le cas de l'intégration aléatoire et le ciblage des gènes médiés par CRISPR sont décrits ( Figure 1 ).
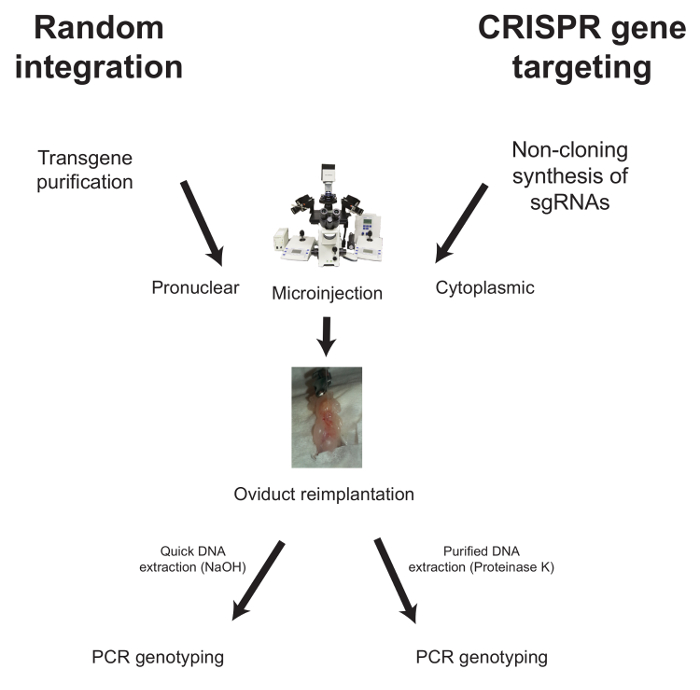
Figure 1: Flux de travail typique pour la génération de souris génétiquement modifiées. Pour une intégration alé...
Discussion
Etapes critiques dans le protocole
On sait que la génération de souris génétiquement modifiées présente un défi technique. Cependant, le protocole présenté ici est une méthode optimisée et simplifiée qui permet de maîtriser et de dépanner la technique en un temps record. Il faut deux étapes pour réussir la technique. Tout d'abord, la synthèse de modèles d'ADN linéaires (pour la synthèse des sgRNA) peut être obtenue sans chlorure de magnésium (MgCl 2 ). C...
Déclarations de divulgation
Les auteurs fournissent des services de transgénèse académique chez la souris via le centre d'analyse Mark Wainwright de l'Université de New South Wales.
Remerciements
Les auteurs remercient le personnel de l'établissement animal (BRC) pour leur soutien continu. Ce travail a été financé par le National Health and Medical Research Council et le Australian Research Council.
matériels
| Name | Company | Catalog Number | Comments |
| Micropipette 0.1-2.5 ul | Eppendorf | 4920000016 | |
| Micropipette 2-20 ul | Eppendorf | 4920000040 | |
| Micropipette 20-200 ul | Eppendorf | 4920000067 | |
| Micropipette 100-1000 ul | Eppendorf | 4920000083 | |
| Molecular weight marker | Bioline | BIO-33025 | HyperLadder 1kb |
| Molecular weight marker | Bioline | BIO-33056 | HyperLadder 100 bp |
| Agarose | Bioline | BIO-41025 | |
| EDTA buffer | Sigma-Aldrich | 93296 | 10x - Dilute to 1x |
| Ethidium bromide | Thermo Fisher Scientific | 15585011 | |
| SYBR Safe gel stain | Invitrogen | S33102 | |
| Gel extraction kit | Qiagen | 28706 | |
| PCR purification kit (Qiaquick) | Qiagen | 28106 | |
| Vacuum system (Manifold) | Promega | A7231 | |
| Nuclease-free microinjection buffer | Millipore | MR-095-10F | |
| Ultrafree-MC microcentrifuge filter | Millipore | UFC30GV00 | |
| Cas9 mRNA | Sigma-Aldrich | CAS9MRNA | |
| CRISPR expressing plasmid (px330) | Addgene | 42230 | |
| Nuclease free water | Sigma-Aldrich | W4502 | |
| Phusion polymerase | New England Biolabs | M0530L | |
| T7 Quick High Yield RNA kit | New England Biolabs | E2050S | |
| RNA purification spin columns (NucAway) | Thermo Fisher Scientific | AM10070 | |
| ssOligos | Sigma-Aldrich | OLIGO STANDARD | |
| Donor plasmid | Thermo Fisher Scientific | GeneArt | |
| Hyaluronidase | Sigma-Aldrich | H3884 | |
| KSOMaa embryo culture medium | Zenith Biotech | ZEKS-100 | |
| Mineral oil | Zenith Biotech | ZSCO-100 | |
| M2 Medium | Sigma-Aldrich | M7167 | |
| Cytochalasin B | Sigma-Aldrich | C6762 | |
| Mouthpiece | Sigma-Aldrich | A5177 | |
| Glass microcapillaries | Sutter Instrument | BF100-78-10 | |
| Proteinase K | Applichem | A3830.0100 | |
| Dumont #5 forceps | Fine Science Tools | 91150-20 | |
| Iris scissors | Fine Science Tools | 91460-11 | |
| Vessel clamp | Fine Science Tools | 18374-43 | |
| Wound clips | Fine Science Tools | 12040-01 | |
| Clips applier | Fine Science Tools | 12018-12 | |
| Micro-scissors | Fine Science Tools | 15000-03 | |
| Cauterizer | Fine Science Tools | 18000-00 | |
| Non-absorbable surgical sutures (Ethilon 3-0) | Ethicon | 1691H | |
| 5% CO2 incubator | MG Scientific | Galaxy 14S | |
| Spectrophotometer | Thermo Fisher Scientific | Nanodrop 2000c | |
| Thermocycler | Eppendorf | 6321 000.515 | |
| Electrophoresis set up | BioRad | 1640300 | |
| UV Transilluminator | BioRad | 1708110EDU | |
| Thermocycler | Eppendorf | 6334000069 | |
| Stereoscopic microscope | Olympus | SZX7 | |
| Inverted microscope | Olympus | IX71 | |
| 2x Micromanipulators | Eppendorf | 5188000.012 | |
| Oocytes manipulator | Eppendorf | 5176000.025 | |
| Microinjector (Femtojet) | Eppendorf | 5247000.013 | |
| Mice C57BL/6J strain | Australian BioResources | C57BL/6JAusb |
Références
- Ittner, L. M. Dendritic function of tau mediates amyloid-beta toxicity in Alzheimer's disease mouse models. Cell. 142 (3), 387-397 (2010).
- Ittner, A. Site-specific phosphorylation of tau inhibits amyloid-beta toxicity in Alzheimer's mice. Science. 354 (6314), 904-908 (2016).
- Marantz Henig, R. . The Monk in the Garden. , (2000).
- Jaenisch, R., Mintz, B. Simian virus 40 DNA sequences in DNA of healthy adult mice derived from preimplantation blastocysts injected with viral DNA. Proc Natl Acad Sci U S A. 71 (4), 1250-1254 (1974).
- Gordon, J. W., Ruddle, F. H. Integration and stable germ line transmission of genes injected into mouse pronuclei. Science. 214 (4526), 1244-1246 (1981).
- Gordon, J. W., Scangos, G. A., Plotkin, D. J., Barbosa, J. A., Ruddle, F. H. Genetic transformation of mouse embryos by microinjection of purified DNA. Proc Natl Acad Sci U S A. 77 (12), 7380-7384 (1980).
- Brinster, R. L., Chen, H. Y., Trumbauer, M. E., Yagle, M. K., Palmiter, R. D. Factors affecting the efficiency of introducing foreign DNA into mice by microinjecting eggs. Proc Natl Acad Sci U S A. 82 (13), 4438-4442 (1985).
- Capecchi, M. R. Gene targeting in mice: functional analysis of the mammalian genome for the twenty-first century. Nat Rev Genet. 6 (6), 507-512 (2005).
- Carbery, I. D. Targeted genome modification in mice using zinc-finger nucleases. Genetics. 186 (2), 451-459 (2010).
- Sung, Y. H. Knockout mice created by TALEN-mediated gene targeting. Nat Biotechnol. 31 (1), 23-24 (2013).
- Jinek, M. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science. 337 (6096), 816-821 (2012).
- Wang, H. One-step generation of mice carrying mutations in multiple genes by CRISPR/Cas-mediated genome engineering. Cell. 153 (4), 910-918 (2013).
- Kang, X. Introducing precise genetic modifications into human 3PN embryos by CRISPR/Cas-mediated genome editing. J Assist Reprod Genet. 33 (5), 581-588 (2016).
- Liang, P. CRISPR/Cas9-mediated gene editing in human tripronuclear zygotes. Protein Cell. 6 (5), 363-372 (2015).
- Ittner, L. M., Götz, J. Pronuclear injection for the production of transgenic mice. Nat Protoc. 2 (5), 1206-1215 (2007).
- Yang, H., Wang, H., Jaenisch, R. Generating genetically modified mice using CRISPR/Cas-mediated genome engineering. Nat Protoc. 9 (8), 1956-1968 (2014).
- . Resuspension Calculator Available from: https://sg.idtdna.com/calc/resuspension/ (2017)
- Auerbach, A. B. Strain-dependent differences in the efficiency of transgenic mouse production. Transgenic Res. 12 (1), 59-69 (2003).
- Merriman, J. A., Jennings, P. C., McLaughlin, E. A., Jones, K. T. Effect of aging on superovulation efficiency, aneuploidy rates, and sister chromatid cohesion in mice aged up to 15 months. Biol Reprod. 86 (2), 49 (2012).
- Nakagawa, Y., et al. Ultra-superovulation for the CRISPR-Cas9-mediated production of gene-knockout, single-amino-acid-substituted, and floxed mice. Biol Open. 5 (8), 1142-1148 (2016).
- Byers, S. L., Wiles, M. V., Dunn, S. L., Taft, R. A. Mouse estrous cycle identification tool and images. PLoS One. 7 (4), e35538 (2012).
- Whitten, W. K. Modification of the oestrous cycle of the mouse by external stimuli associated with the male. J Endocrinol. 13 (4), 399-404 (1956).
- Ye, S., Dhillon, S., Ke, X., Collins, A. R., Day, I. N. An efficient procedure for genotyping single nucleotide polymorphisms. Nucleic Acids Res. 29 (17), E88 (2001).
- Yoshimi, K., et al. ssODN-mediated knock-in with CRISPR-Cas for large genomic regions in zygotes. Nat Commun. 7, 10431 (2016).
- Suzuki, K., et al. In vivo genome editing via CRISPR/Cas9 mediated homology-independent targeted integration. Nature. , (2016).
- Soriano, P. Generalized lacZ expression with the ROSA26 Cre reporter strain. Nat Genet. 21, 70-71 (1999).
- Liu, X., et al. A targeted mutation at the known collagenase cleavage site in mouse type I collagen impairs tissue remodeling. J Cell Biol. 130 (1), 227-237 (1995).
- Ke, Y. D. Short-term suppression of A315T mutant human TDP-43 expression improves functional deficits in a novel inducible transgenic mouse model of FTLD-TDP and ALS. Acta Neuropathol. 130 (5), 661-678 (2015).
- Auerbach, A. B. Production of functional transgenic mice by DNA pronuclear microinjection. Acta Biochim Pol. 51 (1), 9-31 (2004).
- Iyer, V., et al. Off-target mutations are rare in Cas9-modified mice. Nat Methods. 12 (6), 479 (2015).
- Zhou, Y. High-throughput screening of a CRISPR/Cas9 library for functional genomics in human cells. Nature. 509 (7501), 487-491 (2014).
- Brinster, R. L., Chen, H. Y., Trumbauer, M. E., Avarbock, M. R. Translation of globin messenger RNA by the mouse ovum. Nature. 283 (5746), 499-501 (1980).
- Pinkert, C. A., Irwin, M. H., Johnson, L. W., Moffatt, R. J. Mitochondria transfer into mouse ova by microinjection. Transgenic Res. 6 (6), 379-383 (1997).
- Biggers, J. D., Summers, M. C. Choosing a culture medium: making informed choices. Fertil Steril. 90 (3), 473-483 (2008).
- Nakagata, N. Embryo transfer through the wall of the fallopian tube in mice. Jikken Dobutsu. 41 (3), 387-388 (1992).
- Truett, G. E. Preparation of PCR-quality mouse genomic DNA with hot sodium hydroxide and tris (HotSHOT). Biotechniques. 29 (1), 52-54 (2000).
- Richardson, C. D., Ray, G., DeWitt, M. A., Curie, G. L., Corn, J. E. Enhancing homology-directed genome editing by catalytically active and inactive CRISPR-Cas9 using asymmetric donor DNA. Nat Biotechnol. 34 (3), 339-344 (2016).
- Delerue, F., Ittner, L. M. Genome Editing in Mice Using CRISPR/Cas9: Achievements and Prospects. Clon. Transgen. 4, (2015).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon