Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Erzeugung von genetisch modifizierten Mäusen durch die Mikroinjektion von Oozyten
In diesem Artikel
Zusammenfassung
Die Mikroinjektion von Maus-Oozyten wird üblicherweise sowohl für die klassische Transgenese ( dh die zufällige Integration von Transgenen) als auch für das CRISPR-vermittelte Gen-Targeting verwendet. Dieses Protokoll überprüft die neuesten Entwicklungen in der Mikroinjektion, mit einem besonderen Schwerpunkt auf Qualitätskontrolle und Genotypisierung Strategien.
Zusammenfassung
Die Verwendung von genetisch veränderten Mäusen hat wesentlich zu Studien an physiologischen und pathologischen In-vivo- Prozessen beigetragen. Die nukleare Injektion von DNA-Expressionskonstrukten in befruchtete Oozyten bleibt die am häufigsten verwendete Technik, um transgene Mäuse für die Überexpression zu erzeugen. Mit der Einführung der CRISPR-Technologie für das Gen-Targeting wurde die nukleare Injektion in befruchtete Oozyten auf die Erzeugung von Knockout- und Knockin-Mäusen ausgedehnt. Diese Arbeit beschreibt die Herstellung von DNA für die Injektion und die Generierung von CRISPR-Guides für das Gen-Targeting mit besonderem Schwerpunkt auf Qualitätskontrolle. Die für die Identifikation von potentiellen Gründern erforderlichen Genotypisierungsverfahren sind entscheidend. Innovative Genotypisierungsstrategien, die die "Multiplexing" -Fähigkeiten von CRISPR nutzen, werden hier vorgestellt. Auch chirurgische Verfahren werden skizziert. Gemeinsam werden die Schritte des Protokolls die Erzeugung von Gen ermöglichenEtikodisch modifizierte Mäuse und für die anschließende Etablierung von Mauskolonien für eine Fülle von Forschungsfeldern, einschließlich Immunologie, Neurowissenschaften, Krebs, Physiologie, Entwicklung und andere.
Einleitung
Tiermodelle, sowohl bei Wirbeltieren als auch bei Wirbellosen, waren maßgeblich für die Untersuchung der Pathophysiologie menschlicher Zustände wie der Alzheimer-Krankheit 1 , 2 . Sie sind auch unschätzbare Werkzeuge, um nach Krankheitsmodifikatoren zu suchen und letztlich neue Behandlungsstrategien in der Hoffnung auf eine Heilung zu entwickeln. Obwohl jedes Modell intrinsische Einschränkungen hat, ist die Verwendung von Tieren als ganze systemische Modelle für die biomedizinische Forschung unerlässlich. Denn die metabolische und komplexe physiologische Umgebung kann nicht vollständig in der Gewebekultur simuliert werden.
Bis heute bleibt die Maus die häufigste Säugetierart, die für die genetische Manipulation verwendet wird, da sie mehrere Vorteile aufweist. Die physiologischen Prozesse und Gene, die mit Krankheiten assoziiert sind, sind zwischen Mäusen und Menschen hoch konserviert. Die Maus war der erste Säugetier, um sein volles Genom sequenziert zu haben (2002), ein Jahr vor dem menschlichen GenoIch (2003). Abgesehen von diesem Reichtum an genetischer Information hat die Maus gute Zuchtkapazitäten, einen schnellen Entwicklungszyklus (6 Wochen von der Befruchtung bis zur Entwöhnung) und eine vernünftige Größe. Alle diese Vorteile, verbunden mit physiologischen Indikatoren, wie unterschiedliche Fellfarben (erforderlich für Crossing-Strategien), machte die Maus ein attraktives Modell für die genetische Manipulation. Vor allem in der frühen Zeit der modernen Genetik begann Gregor Mendel, an Mäusen zu arbeiten, bevor er zu Pflanzen ging 3 .
Gentransfer-Techniken führten zur Erzeugung der ersten transgenen Maus über drei Jahrzehnte her 4 , die anfänglich mit viraler Verabreichung hergestellt wurde. Allerdings erkannten die Forscher bald, dass eine der Hauptaufgaben der Maus-Transgenese die Unfähigkeit war, das Schicksal der exogenen DNA zu kontrollieren. Weil die virale Abgabe von Transgenen in Maus-Oozyten dazu geführt hat, dass mehrere Kopien zufällig in das Genom integriert wurden, die MöglichkeitY der Herstellung nachfolgender transgener Linien war begrenzt.
Eine solche Einschränkung wurde überwunden, als Gordon et al. Erzeugte die erste transgene Mauslinie durch Mikroinjektion 5 , 6 . Dies begann die Ära der rekombinanten DNA-Technologie, und die Parameter, die das Ergebnis einer Mikroinjektionssitzung beeinflussen, wurden weitgehend untersucht 7 . Obwohl die Mikroinjektion keine Kontrolle über die Integrationsstelle des Transgens erlaubt (was schließlich zu spezifischen Expressionsniveaus für jede Gründermaus führt) bleibt der Hauptvorteil der vorkernigen Mikroinjektion die Bildung von Concatemern ( dh Arrays von mehreren Kopien des Transgens, Verknüpft in Serie) vor der genomischen Integration 5 . Dieses Merkmal wurde im Laufe der Jahre verwendet, um Tausende von transgenen Mauslinien zu etablieren, die ein Gen von Interesse überexprimieren. Seitdem, transgenese, die aRtificiale Modifikation des Genoms eines Organismus wurde weitgehend verwendet, um die Rolle einzelner Gene beim Auftreten von Krankheiten zu identifizieren.
Eine weitere wichtige Errungenschaft bei der Manipulation des Mausgenoms wurde erreicht, als Mario Capecchi erfolgreich ein einziges Gen in der Maus unterbrach und die Ära des Gen-Targeting 8 öffnete. Allerdings traten grundsätzliche Nachteile schnell aus dem ES-zellbasierten Gen-Targeting hervor, einschließlich der Herausforderungen der Kultivierung von ES-Zellen, des etwas variablen Chimäritätsgrades und der Länge des Prozesses ( dh 12-18 Monate, minimal, um die Maus zu erhalten) .
In jüngster Zeit haben sich Fortschritte bei neuen Technologien wie z. B. konstruierte Endonukleasen ( z. B. Zinkfinger-Nukleasen (ZFN), Transkriptionsaktivator-ähnliche Effektor-Nukleasen (TALEN) und gruppierten regelmäßig zusammengesetzten kurzen palindromischen Wiederholungen (CRISPR / Cas9) als alternative Methoden ergeben Beschleunigen den Prozess der Gen-Targeting in micE 9 , 10 Diese Endonukleasen können durch Mikroinjektion leicht in Mausozyten injiziert werden, was die Erzeugung von Gen-gezielten Mäusen in nur 6 Wochen ermöglicht.
Seit dem ersten Bericht über die Verwendung von CRISPR für die Genombearbeitung 11 hat dieses bakterielle adaptive Immunsystem ZFN und TALEN aufgrund seiner vielen Vorteile, einschließlich der Leichtigkeit der Synthese und der Fähigkeit, mehrere Loci auf einmal (als "Multiplexing" bezeichnet) ersetzt "). CRISPR wurde zuerst für Gen-Targeting in Mäusen 12 verwendet und ist seitdem auf unzählige Arten angewendet worden, von Pflanzen auf Menschen 13 , 14 . Bisher gibt es keinen Bericht über eine einzige Art, die gegen CRISPR-Genom-Bearbeitung resistent ist.
Die beiden Hauptbegrenzungsschritte der Erzeugung von transgenen Mäusen sind die Injektion von Oozyten und die ReimplantationDieser Oozyten zu pseudo-schwangeren Frauen. Obwohl diese Technik von uns 15 und anderen 16 beschrieben worden ist, haben die jüngsten technischen Verbesserungen bei der Mausembryologie und den Gentransfertechniken den Prozess der Erzeugung von genetisch modifizierten Mäusen revolutioniert. Diese Verbesserungen werden hier beschrieben.
Access restricted. Please log in or start a trial to view this content.
Protokoll
Alle Verfahren wurden von der University of New South Wales Animals Care und Ethics Committee genehmigt.
1. Vorbereitung des Transgens (Random Integration)
- Analytische Agarose-Gelelektrophorese.
- Das Plasmid wird unter Verwendung geeigneter Enzyme (1 h Inkubation) oder Fast-Digest-Enzymen (15 bis 30 min Inkubation) in einem Thermocycler nach den Empfehlungen des Herstellers untersucht (siehe Abbildung 2A und seine Legende).
- Es wurde ein 1% iges Trisacetat-Ethylendiamintetraessigsäure- (EDTA) (TEA) -Affarosegel gegossen, das mit 0,5-1,0 μg / ml Ethidiumbromid (EtBr) gefärbt ist.
HINWEIS: Bei der Verwendung von EtBr Vorsicht walten lassen. Es ist ein starkes mutagen. Bitte geeignete Schutzausrüstung verwenden (siehe Sicherheitsdatenblatt). - Laden Sie eine 1 kb Molekulargewichtsmarkierung.
- Laden Sie die linearisierten Fragmente ( dh Transgen und Backbone).
- Führen Sie die Elektrophorese bei 100 V für 45 min.
- Visualisieren Sie das Gel auf einem Ultraviolett- (UV) Transilluminator, um zu überprüfen, ob die Verdauung vollständig ist, und um die korrekte Größe des Transgens zu bestätigen ( dh 7,338 bp, siehe Abbildung 2A ).
- Präparative Agarose-Gelelektrophorese
- Gießen Sie ein 1% TAE-Agarosegel, gefärbt mit einem niedrigen Toxizitätsgelfleck. Laden Sie 8 Aliquots (je 1 μg) linearisiertes Transgen ohne Molekulargewichtsmarker und führen Sie die Elektrophorese bei 100 V für 45 min durch. Verwenden Sie ein Skalpell, um alle 8 Bänder, die dem linearisierten Transgen entsprechen, zu exzimieren.
- Extrahieren Sie die DNA mit einem Gel-Extraktionskit, indem Sie den Empfehlungen des Herstellers folgen (siehe Tabelle der Materialien ).
- Die Gelfragmente bei 50 ° C in 1,5-ml-Röhrchen für mindestens 15 min schmelzen. Mit Isopropanol abfließen und dann den Bindungspuffer zugeben.
- Kombiniere die gesamte DNA durch Pipettieren alles in eine Spalte. Eluieren in nukleasefreien Mikroinjektionspuffer (8 mM Tris-HCl und 0,15 mM EDTA). Durch einen 0,22 μm Mikrozentrifugenfilter filtrieren und 60 s bei 12.000 x g zentrifugieren.
- Messen Sie die Konzentration und die Qualität der DNA mit einem Spektrophotometer. Prüfen Sie, ob das A260 / A280-Verhältnis etwa 1,8 beträgt ( dh keine Verunreinigung durch Proteine) und das A260 / A230-Verhältnis mindestens 2,0 ( dh keine Kontamination durch organische Lösungsmittel). Bis zu 3 ng / μl in nukleasefreiem Mikroinjektionspuffer, Aliquot (20-50 μl) und bei -20 ° C einfrieren.
2. Synthese von CRISPR-Komponenten (Gen-Targeting)
- Cas9-mRNA
- Verdünnen Sie die Cas9-mRNA (erhalten aus einer kommerziellen Quelle, siehe Tabelle der Materialien ) bis zu einer Konzentration von 1 μg / μL in nukleasefreiem Mikroinjektionspuffer.
- Aliquot in 2 μl und frei Ze bei -80 ° C.
- Single-Guide-RNA (SGRNA).
- Identifizieren Sie die gewünschten Zwei-Ziel-Genom-Sequenzen (Guides) mit einem rechnerischen Design-Tool ermöglicht minimale potenzielle Off-target-Aktivität 17 .
- Für Gen-Knockout, systematisch wählen Sie zwei Führer, die ein paar hundert Basenpaare (bp) auseinander, in entgegengesetzte Richtungen, und enthalten die Start-Codon des Gens von Interesse. Für die Homologie direkt reparieren, wählen Sie zwei überlappende Führungen (wann immer möglich), in entgegengesetzte Richtungen.
HINWEIS: Als Beispiel wurden die folgenden Guides vor kurzem erfolgreich zusammengespritzt, um das erste Exon des TPM4.2- Gens zu stören:
Leitfaden 1: 5 'CGCCATCCAGTTCGCGCTGC 3';
Leitfaden 2: 5 'CAGAACGATTGAGCTATGGC 3'
- Für Gen-Knockout, systematisch wählen Sie zwei Führer, die ein paar hundert Basenpaare (bp) auseinander, in entgegengesetzte Richtungen, und enthalten die Start-Codon des Gens von Interesse. Für die Homologie direkt reparieren, wählen Sie zwei überlappende Führungen (wann immer möglich), in entgegengesetzte Richtungen.
- Bestellen Sie einen Satz von Primern wie in Tabelle 1 beschrieben.
- Synthese einer linearen DNA-Matrize.
- Verdünnen px330Ref "> 12 Plasmid auf 10 ng / μL in nukleasefreiem Wasser.
- Bereiten Sie den Master-Mix wie in Tabelle 1 angegeben vor. Füge die Polymerase am Ende hinzu und halte die Mastermischung auf Eis.
- Mischen und auf 8 PCR-Röhrchen (19 μl / Tube) aufteilen. Füge 1 μl px330 pro Röhrchen hinzu und laufe die PCR unter folgenden Bedingungen: 1 min bei 98 ° C; 40 Zyklen von 98 ° C für 10 s, 64 ° C für 30 s und 72 ° C für 15 s; Und eine Enddehnung bei 72 ° C für 5 min. Bei 4 ° C halten.
- Reinigen Sie die PCR-Produkte mit einem PCR-Reinigungskit (siehe Tabelle der Materialien ), einer Mannigfaltigkeit und einer Vakuumquelle (für eine schnellere Verarbeitung). Die maximale Vakuumstärke ( dh 8 mbar) auftragen.
- Füge 5 Volumina (100 μl) Bindungspuffer zu 1 Volumen der PCR-Probe hinzu und vermische.
- Legen Sie eine (vorgesehene) Siliciumdioxid-Membran-Spin-Säule in ein (mitgeliefertes) 2-ml-Sammelrohr für die Zentrifugation oder auf den Verteiler für Vakuumverfahren einIng. Um DNA zu binden, sukzessive alle 8 Proben auf die Säule auftragen und vakuumieren oder für 60 s bei 12.000 xg für jede Ladung zentrifugieren.
- Zum Waschen werden 0,75 ml Waschpuffer (Puffer PE) in die Säule gegeben und für 60 s bei 12.000 x g vakuumiert oder zentrifugiert. Die Säule für 60 s bei 12.000 xg zentrifugieren, um restliches Ethanol zu entfernen.
- Legen Sie die Säule in ein sauberes 1,5-ml-Mikrozentrifugenröhrchen. Um die DNA zu eluieren, füge 30 μl nukleasefreies Wasser in die Mitte der Membran hinzu, lasse die Säule 1 min stehen und zentrisch für 60 s bei 12.000 g
- Messen Sie die Konzentration mit einem Spektrophotometer; Typischerweise sollte die Konzentration 100 ng / μL oder höher betragen. Prüfen Sie, ob das A260 / A280-Verhältnis etwa 1,8 beträgt ( dh keine Verunreinigung durch Proteine) und das A260 / A230-Verhältnis mindestens 2,0 ( dh keine Kontamination durch organische Lösungsmittel).
- In-vitro- Transkription unter Verwendung eines T7-RNA-Synthesekits.
- Vorbereitung tEr mischte, wie in Tabelle 1 angegeben, und inkubieren für 3 h bei 37 ° C in einem Thermocycler. Füge 28 μl nukleasefreies Wasser und 2 μl DNase I hinzu und inkubiere weitere 15 min bei 37 ° C in einem Thermocycler.
- RNA-Reinigung mit Spin-Säulen.
- Lösen Sie das in der bereitgestellten Spinsäule enthaltene Pulver in 650 μl nukleasefreiem Mikroinjektionspuffer, wobei alle Luftblasen sorgfältig entfernt werden. Das Röhrchen umhüllen und 5 bis 15 min bei Raumtemperatur hydratisieren.
- Entfernen Sie die blaue Kappe an der Unterseite und legen Sie die Säule in ein 2-ml-Rohr. 2 min bei 750 xg und Raumtemperatur zentrifugieren. Die Säule in ein frisches 1,5-ml-Röhrchen geben und 50 μl der RNA-Lösung tropfenweise in die Mitte geben, ohne die Säulenwand zu berühren. Spin die Säule für 2 min bei 750 x g.
- Messen Sie die RNA-Konzentration mit einem Spektrophotometer (die typische Ausbeute einer Reaktion beträgt 30 - 50 μg). Prüfen Sie, ob das A260 / A280-Verhältnis a istRunde 2.0 ( dh keine Verunreinigung durch Proteine) und das A260 / A230-Verhältnis beträgt mindestens 2,0 ( dh keine Verunreinigung durch organische Lösungsmittel). Die sgRNAs bei -80 ° C bis zur Verwendung aufbewahren.
- Beurteilen Sie die Qualität der RNA unter Verwendung der 1% TAE-Gele, die routinemäßig für die DNA-Elektrophorese verwendet werden. Führen Sie 200-400 ng der DNA-Matrize und die entsprechende sgRNA parallel aus. Die RNA-Bande (≈ 100 bp) sollte etwas größer sein als die DNA-Bande (siehe Abbildung 3A ).
- Identifizieren Sie die gewünschten Zwei-Ziel-Genom-Sequenzen (Guides) mit einem rechnerischen Design-Tool ermöglicht minimale potenzielle Off-target-Aktivität 17 .
3. Spendervorlage
- Bestellen von kommerziell synthetisierten einzelsträngigen Oligonukleotiden (ssOligos, siehe Tabelle der Materialien ) für Punktmutationen oder für die Integration von kleinen Sequenzen bis zu 50 bp; Homologiearme sind typischerweise 60-90 bp lang.
- Für größere Insertionen verwenden Sie ein Spenderplasmid als Vorlage. Erzeugung eines geeigneten Plasmids unter Verwendung der klassischen Klonierungsmethode oder der de novo - Synthese aus einemKommerzielle Quelle.
HINWEIS: Es werden mindestens 800 bp pro Homologiearm empfohlen. Die Größe des Rückgrats hat keinen Einfluss auf die Effizienz der Homologie-Direktreparatur (HDR).
4. Injektionsmischung
- Verdünnen Sie die Cas9-mRNA auf 50 ng / μl und die sgRNAs auf 12,5 ng / μL (insgesamt 25 ng / μL) unter Verwendung von nukleasefreiem Mikroinjektionspuffer für Gen-Knockout-Experimente. Fügen Sie die Spender-Matrize (ssOligo oder Plasmid) in einer Konzentration von 200 ng / μL für Genom-Editing-Experimente auf der Grundlage von Homologie direkte Reparatur.
HINWEIS: Verdünnungsvolumina können mit Hilfe von Online-Tools, wie z. B. einem Resuspensionsrechner, leicht ermittelt werden. - Halten Sie jedes Aliquot auf Eis während der Mikroinjektionssitzung und verwerfen Sie danach (nicht wieder einfrieren die Einspritzung Mischung).
5. Skrotal-Vasektomie
ANMERKUNG: Zwei Arten von Vasektomie werden häufig bei Mäusen durchgeführt: Bauch und Skrotal. LetztereIst weniger invasiv und wurde zuvor beschrieben 15 .
- Autoklav alle chirurgischen Instrumente aus rostfreiem Stahl.
- Bestimmen Sie das Gewicht der Maus mit einer Waage und betäubt die Männchen durch intraperitoneale (ip) Injektion mit injizierbarem Anästhetikum (Ketamin 100 mg / kg, Xylazin 10 mg / kg).
- Überwachen Sie den Verlust des Zehen-Quetsch-Reflexes und sobald die Maus sediert ist, legen Sie sie auf ein Heizkissen.
- Desinfizieren Sie die Haut um die Hoden mit wechselnden Tüchern eines topischen Antiseptikums wie Chlorhexidin und 70% Ethanol.
- Schieben Sie die Hoden in den Skrotalsack und machen Sie einen Hautschnitt mit einem Skalpell.
- Visualisierung der Hoden, Cauda-Nebenhoden und Vas deferens.
- Greife die Vas deferens mit Pinzette, halte sie und kauterisiere auf jeder Seite der Pinzette, um 3 mm der Vas deferens zu entfernen.
- Führen Sie das gleiche Verfahren auf dem zweiten Testis durch.
- Schließen Sie die Hautinzisionen mit Wundclips und überwachen Sie dieMaus bis zur vollständigen Erholung.
6. Superovulation (Oozytenspender) und Zeitmessung (Pseudo-schwangere Frauen)
HINWEIS: Die Technik zur Erzeugung einer geeigneten Anzahl von befruchteten Oozyten und verstopfte Pflegefrauen zur Reimplantation wurde an anderer Stelle beschrieben 15 .
- Injizieren Sie 10 Frauen mit 5 IE des Serum Gonadotropins der schwangeren Stute (PMSG; in 100 μl) um 12 Uhr am Tag 1.
- Injizieren Sie die Weibchen ip mit 5 IE menschlichem Choriongonadotropin (hCG, in 100 μl) 46-48 h später (um 11 Uhr am Tag 3).
- Sofort beruhigen die Weibchen mit Eingeborenen über Nacht.
7. Pronukleare (zufällige Integration) und zytoplasmatische (Gene-Targeting) Injektionen
- Cull die superovulierten Mäuse durch zervikale Versetzung, entlasten den Bauch und greifen auf die Eierstöcke und Ovidukte, wie zuvor beschrieben 15 . Dissect die oViducts und ernten die Cumulus-Oozyten-Komplexe (COCs) unter einem Stereomikroskop 15 . Legen Sie sie in einen Tropfen Kalium-Simplex-Optimierungsmedium, ergänzt mit Aminosäuren (KSOMaa) nach dem traditionellen Verfahren 15 .
- Die Oozyten werden durch Zugabe von ca. 1 & mgr; l Hyaluronidase (10 mg / ml) zum Tropfen KSOMaa-Medium unter Verwendung eines Aspirationsmundstücks zugegeben und die Schale in einen 37 ° C / 5% CO 2 -Inkubator für 30 s bis 1 min gegeben.
- Die Oozyten mit 4 frischen Tropfen KSOMaa-Medium mit dem Aspirator-Mundstück waschen und auf einen endgültigen Tropfen KSOMaa-Medium überführen, der mit Mineralöl (ca. 2 ml) überzogen ist. Legen Sie die Schale in den 37 ° C / 5% CO 2 -Inkubator, bis die Oozyten zur Injektion bereit sind.
- Pronukleare Injektion (zufällige Integration).
- Die Eier unter dem stereoskopischen Mikroskop visualisieren und ca. 50 Eier in die Injektionskammer bringen (a droP von M2 Medium mit Mineralöl überlagert) mit dem Aspirator Mundstück.
- Übertragen Sie die Injektionskammer auf das invertierte Mikroskop und injizieren Sie die befruchteten Oozyten (zwei sichtbare Vorkerne) mit einigen Picolitern der Injektionsmischung, die auf einen Vorkern abgestimmt sind (jeder der Vorkern kann gezielt werden). Manifestiere eine erfolgreiche Injektion durch Beobachtung der Schwellung des Vorkerns.
- Zytoplasmatische Injektion (Gen-Targeting).
- Übertragen Sie etwa 50 Eier zu einem Tropfen KSOMaa mit 5 μg / ml Cytochalasin B mit dem Mundstück und inkubieren für 5 min im 37 ° C / 5% CO 2 -Inkubator.
- Übertragen Sie die Eier mit dem Mundstück in die Injektionskammer.
- In den Zytoplasma bei sehr niedrigem Druck (50-100 hPa) wenige Pikoliter der Injektionsmischung einspritzen, wobei der Kompensationsdruck des automatisierten Mikroinjektors nach Möglichkeit möglich ist.
- Nach der Injektion die Oozyten wieder in einen Tropfen KSO überführenMaa (mit dem Mundstück) und halten sie im 37 ° C / 5% CO 2 -Inkubator, bis zur Reimplantation in den Eileiter der pseudo-schwangeren Frauen geladen.
8. Reimplantation
- Autoklav alle chirurgischen Instrumente aus rostfreiem Stahl.
- Bestimmen Sie das Gewicht der Maus mit der Waage und betäubt die Weibchen durch intraperitoneale (ip) Injektion mit injizierbarem Anästhetikum (Ketamin 100 mg / kg, Xylazin 10 mg / kg).
- Überwachen Sie den Verlust des Zehen-Quetsch-Reflexes und sobald die Maus sediert ist, legen Sie sie auf ein Heizkissen.
- Clip eine große Fläche des Pelzes um die dorsale Mittellinie der weiblichen Maus.
- Desinfizieren Sie die exponierte Haut mit abwechselnden Tüchern eines topischen Antiseptikums wie Chlorhexidin und 70% Ethanol.
- Exponieren Sie den Fortpflanzungstrakt nach der traditionellen Methode 15 . Machen Sie einen 1 cm langen Hautschnitt parallel zur dorsalen Mittellinie, schneiden Sie den Muskel ab und packen Sie den fetten Pad wiTh eine Pinzette, dann sanft ziehen die Eierstöcke aus, bis seine beigefügten Eileiter und Uterus deutlich sichtbar sind.
- Fix das Fett-Pad mit einer Gefäßklemme. Visualisieren Sie den Eileiter unter dem stereoskopischen Mikroskop und verwenden Sie ein Paar Mikro-Schere machen einen Einschnitt in die Wand des Ovidukt wenige Millimeter stromaufwärts der Ampulle.
- Unter dem stereoskopischen Mikroskop laden 25 mikroinjizierte Eier in die Glaskapillare, die mit dem Mundstück verbunden ist (der Innendurchmesser der Kapillare sollte etwa 120 μm breit sein). Füge die Glaskapillare in den Eileiter ein und vertreibe, bis eine Luftblase in der Ampulle sichtbar ist.
- Entfernen Sie vorsichtig die Glaskapillare und legen Sie den Fortpflanzungstrakt wieder in den Bauch. Naht den Einschnitt mit 3-0 nicht resorbierbaren chirurgischen Nähten, dann schließen mit Wundclips. Überwachen Sie die Maus genau bis zur vollständigen Wiederherstellung.
9. Genotypisierungsstrategien / Sequenzierung
HINWEIS: Das Genom isolierenDNA aus 2-mm-Schwanz- oder Ohrenbiopsien, nach relevanten Tierethikvorschriften.
- Schnelle genomische DNA-Extraktion (zufällige Integration).
- Lyse die Gewebeproben (~ 2 mm) bei 95 ° C für 1 h in 100 μl alkalischem Lyse-Reagenz (25 mM Natriumhydroxid und 0,2 mM EDTA, pH = 12).
- 100 μl Neutralisationsreagenz (40 mM Tris-HCl, pH = 5) zugeben.
- 5 min bei 12.000 g und 4 ° C zentrifugieren.
- Hochwertige genomische DNA-Extraktion (Gen-Targeting).
- Fügen Sie 500 μl Schwanzpuffer (50 mM Tris, pH = 8, 100 mM EDTA, pH = 8, 100 mM NaCl, 1% SDS und 0,5 mg / ml Proteinase K, frisch zugesetzt) hinzu.
- Inkubieren über Nacht bei 55 ° C.
- Mischen für 5 min durch Invertieren (nicht vortexen).
- Zugabe von 250 μl gesättigtem NaCl (6 M) und Mischen für 5 min durch Invertieren (nicht vortexen).
- Für 5-10 min bei 12.000 xg und 4 ° C drehen und den Überstand in neues Röhrchen geben.
- Fügen Sie 500 μl Isopropanol hinzu und mischen für 5 min durch Invertieren (nicht vortexen).
- Für 10 min bei 12.000 xg und Raumtemperatur drehen.
- Dekantieren den Überstand (das Pellet ist unsichtbar und haftet an der Tube).
- Mit 1 ml 70% igem Ethanol waschen.
- Spin für 5-10 min bei 12.000 xg und Raumtemperatur. Entfernen Sie den Überstand sorgfältig, da das weiße Pellet nicht mehr klebrig ist und verloren gehen kann.
- Luft trocknen diese für ~ 1 h.
- Man gibt 400 μl TE-Puffer (10 mM Tris und 1 mM EDTA, pH = 8) zu.
- Die DNA bei 55-60 ° C für 2 h auflösen.
- Grundierung Design.
- Entwerfen Sie Primer ein Minimum von 20 bp lang mit einem Rechenwerkzeug 19 .
HINWEIS: Für die zufällige Integration sollten die Primer mit dem Transgen hybridisieren und ein unterschiedliches Fragment von 200-800 bp erzeugen. Für die Gen-Targeting, die Primer so zu gestalten, dass sie mit der genomischen DNA hybridisieren, wodurch eine deutliche fRennen ein paar hundert Bp um die geschnittenen Seiten. Für große Insertionen können die Primer auf dem Transgen sitzen, aber die Bestätigung der gezielten Integration sollte anschließend mit Primer Walking oder einer ähnlichen Technik durchgeführt werden.
- Entwerfen Sie Primer ein Minimum von 20 bp lang mit einem Rechenwerkzeug 19 .
- PCR-Genotypisierung.
- Für jede genomische Modifikation, entwerfen Sie ein neues PCR-Protokoll und testen Sie es empirisch. Man betrachte die Länge des erzeugten Fragments und die Schmelztemperatur (Tm) jedes Paares von Primern.
- Sequenzierung.
- Für Gen-Targeting, senden PCR-Produkte an einen Sanger Sequenzierung Dienstleister.
Access restricted. Please log in or start a trial to view this content.
Ergebnisse
Im Folgenden werden die Arbeitsabläufe für die Mikroinjektion im Falle der zufälligen Integration und des CRISPR-vermittelten Gen-Targeting beschrieben (Abbildung 1 ).
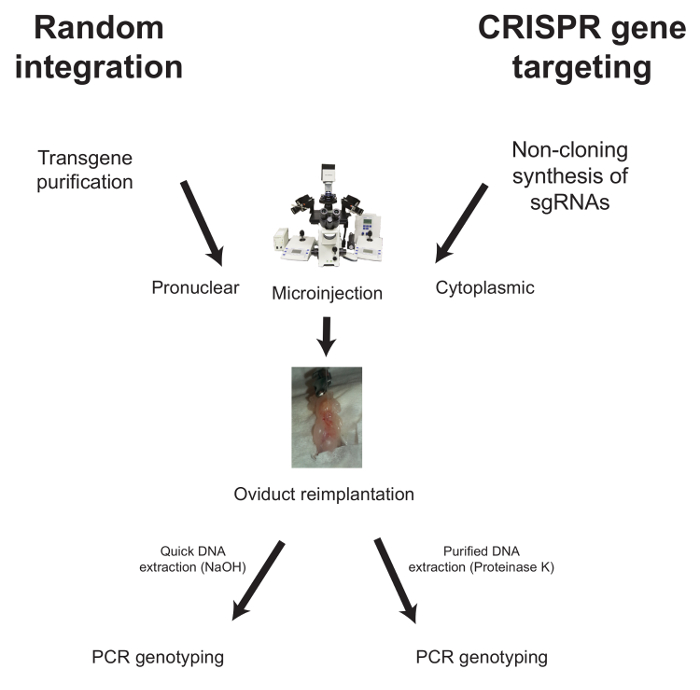
Abbildung 1: Typischer Workflow zur Erzeugung von genetisch modifizierten Mäusen. Für die zufällige Integration wird...
Access restricted. Please log in or start a trial to view this content.
Diskussion
Kritische Schritte im Protokoll
Die Erzeugung von genetisch veränderten Mäusen ist technisch anspruchsvoll. Allerdings ist das hier vorgestellte Protokoll eine optimierte und vereinfachte Methode, die es ermöglicht, die Technik in Rekordzeit zu beherrschen und zu beheben. Für den erfolgreichen Abschluss der Technik sind zwei Schritte erforderlich. Zuerst kann die Synthese von linearen DNA-Matrizen (für die Synthese von sgRNAs) ohne Magnesiumchlorid (MgCl 2 ) erreicht werden. Es ...
Access restricted. Please log in or start a trial to view this content.
Offenlegungen
Die Autoren bieten akademische Transgenese Dienstleistungen in Mäusen über die University of New South Wales Mark Wainwright Analytical Center.
Danksagungen
Die Autoren danken den Mitarbeitern der Tierfabrik (BRC) für ihre laufende Unterstützung. Diese Arbeit wurde vom National Health and Medical Research Council und dem Australian Research Council finanziert.
Access restricted. Please log in or start a trial to view this content.
Materialien
| Name | Company | Catalog Number | Comments |
| Micropipette 0.1-2.5 μL | Eppendorf | 4920000016 | |
| Micropipette 2 - 20 μL | Eppendorf | 4920000040 | |
| Micropipette 20 - 200 μL | Eppendorf | 4920000067 | |
| Micropipette 100 - 1,000 μL | Eppendorf | 4920000083 | |
| Molecular weight marker | Bioline | BIO-33025 | HyperLadder 1 kb |
| Molecular weight marker | Bioline | BIO-33056 | HyperLadder 100 bp |
| Agarose | Bioline | BIO-41025 | |
| EDTA buffer | Sigma-Aldrich | 93296 | 10x - Dilute to 1x |
| Ethidium bromide | Thermo Fisher Scientific | 15585011 | |
| SYBR Safe gel stain | Invitrogen | S33102 | |
| Gel extraction kit | Qiagen | 28706 | |
| PCR purification kit (Qiaquick) | Qiagen | 28106 | |
| Vacuum system (Manifold) | Promega | A7231 | |
| Nuclease-free microinjection buffer | Millipore | MR-095-10F | |
| Ultrafree-MC microcentrifuge filter | Millipore | UFC30GV00 | |
| Cas9 mRNA | Sigma-Aldrich | CAS9MRNA | |
| CRISPR expressing plasmid (px330) | Addgene | 42230 | |
| Nuclease free water | Sigma-Aldrich | W4502 | |
| Phusion polymerase | New England Biolabs | M0530L | |
| T7 Quick High Yield RNA kit | New England Biolabs | E2050S | |
| RNA purification spin columns (NucAway) | Thermo Fisher Scientific | AM10070 | |
| ssOligos | Sigma-Aldrich | OLIGO STANDARD | |
| Donor plasmid | Thermo Fisher Scientific | GeneArt | |
| Hyaluronidase | Sigma-Aldrich | H3884 | |
| KSOMaa embryo culture medium | Zenith Biotech | ZEKS-100 | |
| Mineral oil | Zenith Biotech | ZSCO-100 | |
| M2 Medium | Sigma-Aldrich | M7167 | |
| Cytochalasin B | Sigma-Aldrich | C6762 | |
| Mouthpiece | Sigma-Aldrich | A5177 | |
| Glass microcapillaries | Sutter Instrument | BF100-78-10 | |
| Proteinase K | Applichem | A3830.0100 | |
| Dumont #5 forceps | Fine Science Tools | 91150-20 | |
| Iris scissors | Fine Science Tools | 91460-11 | |
| Vessel clamp | Fine Science Tools | 18374-43 | |
| Wound clips | Fine Science Tools | 12040-01 | |
| Clips applier | Fine Science Tools | 12018-12 | |
| Micro-scissors | Fine Science Tools | 15000-03 | |
| Cauterizer | Fine Science Tools | 18000-00 | |
| Non-absorbable surgical sutures (Ethilon 3-0) | Ethicon | 1691H | |
| 5% CO2 incubator | MG Scientific | Galaxy 14S | |
| Spectrophotometer | Thermo Fisher Scientific | Nanodrop 2000c | |
| Thermocycler | Eppendorf | 6321 000.515 | |
| Electrophoresis set up | BioRad | 1640300 | |
| UV Transilluminator | BioRad | 1708110EDU | |
| Thermocycler | Eppendorf | 6334000069 | |
| Stereoscopic microscope | Olympus | SZX7 | |
| Inverted microscope | Olympus | IX71 | |
| 2x Micromanipulators | Eppendorf | 5188000.012 | |
| Oocytes manipulator | Eppendorf | 5176000.025 | |
| Microinjector (Femtojet) | Eppendorf | 5247000.013 | |
| Mice C57BL/6J strain | Australian BioResources | C57BL/6JAusb |
Referenzen
- Ittner, L. M. Dendritic function of tau mediates amyloid-beta toxicity in Alzheimer's disease mouse models. Cell. 142 (3), 387-397 (2010).
- Ittner, A. Site-specific phosphorylation of tau inhibits amyloid-beta toxicity in Alzheimer's mice. Science. 354 (6314), 904-908 (2016).
- Marantz Henig, R. The Monk in the Garden. , Mariner books. (2000).
- Jaenisch, R., Mintz, B. Simian virus 40 DNA sequences in DNA of healthy adult mice derived from preimplantation blastocysts injected with viral DNA. Proc Natl Acad Sci U S A. 71 (4), 1250-1254 (1974).
- Gordon, J. W., Ruddle, F. H. Integration and stable germ line transmission of genes injected into mouse pronuclei. Science. 214 (4526), 1244-1246 (1981).
- Gordon, J. W., Scangos, G. A., Plotkin, D. J., Barbosa, J. A., Ruddle, F. H. Genetic transformation of mouse embryos by microinjection of purified DNA. Proc Natl Acad Sci U S A. 77 (12), 7380-7384 (1980).
- Brinster, R. L., Chen, H. Y., Trumbauer, M. E., Yagle, M. K., Palmiter, R. D. Factors affecting the efficiency of introducing foreign DNA into mice by microinjecting eggs. Proc Natl Acad Sci U S A. 82 (13), 4438-4442 (1985).
- Capecchi, M. R. Gene targeting in mice: functional analysis of the mammalian genome for the twenty-first century. Nat Rev Genet. 6 (6), 507-512 (2005).
- Carbery, I. D. Targeted genome modification in mice using zinc-finger nucleases. Genetics. 186 (2), 451-459 (2010).
- Sung, Y. H. Knockout mice created by TALEN-mediated gene targeting. Nat Biotechnol. 31 (1), 23-24 (2013).
- Jinek, M. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science. 337 (6096), 816-821 (2012).
- Wang, H. One-step generation of mice carrying mutations in multiple genes by CRISPR/Cas-mediated genome engineering. Cell. 153 (4), 910-918 (2013).
- Kang, X. Introducing precise genetic modifications into human 3PN embryos by CRISPR/Cas-mediated genome editing. J Assist Reprod Genet. 33 (5), 581-588 (2016).
- Liang, P. CRISPR/Cas9-mediated gene editing in human tripronuclear zygotes. Protein Cell. 6 (5), 363-372 (2015).
- Ittner, L. M., Götz, J. Pronuclear injection for the production of transgenic mice. Nat Protoc. 2 (5), 1206-1215 (2007).
- Yang, H., Wang, H., Jaenisch, R. Generating genetically modified mice using CRISPR/Cas-mediated genome engineering. Nat Protoc. 9 (8), 1956-1968 (2014).
- CRISPR DESIGN. , Available from: http://crispr.mit.edu (2017).
- Integrated DNA Technologies. Resuspension Calculator. , Available from: https://sg.idtdna.com/calc/resuspension/ (2017).
- BioTools at UMass Medical School. Primer3: WWW primer tool. , Available from: http://biotools.umassmed.edu/bioapps/primer3_www.cgi (2017).
- Auerbach, A. B. Strain-dependent differences in the efficiency of transgenic mouse production. Transgenic Res. 12 (1), 59-69 (2003).
- Merriman, J. A., Jennings, P. C., McLaughlin, E. A., Jones, K. T. Effect of aging on superovulation efficiency, aneuploidy rates, and sister chromatid cohesion in mice aged up to 15 months. Biol Reprod. 86 (2), 49(2012).
- Nakagawa, Y., et al. Ultra-superovulation for the CRISPR-Cas9-mediated production of gene-knockout, single-amino-acid-substituted, and floxed mice. Biol Open. 5 (8), 1142-1148 (2016).
- Byers, S. L., Wiles, M. V., Dunn, S. L., Taft, R. A. Mouse estrous cycle identification tool and images. PLoS One. 7 (4), e35538(2012).
- Whitten, W. K. Modification of the oestrous cycle of the mouse by external stimuli associated with the male. J Endocrinol. 13 (4), 399-404 (1956).
- Ye, S., Dhillon, S., Ke, X., Collins, A. R., Day, I. N. An efficient procedure for genotyping single nucleotide polymorphisms. Nucleic Acids Res. 29 (17), E88(2001).
- Yoshimi, K., et al. ssODN-mediated knock-in with CRISPR-Cas for large genomic regions in zygotes. Nat Commun. 7, 10431(2016).
- Suzuki, K., et al. In vivo genome editing via CRISPR/Cas9 mediated homology-independent targeted integration. Nature. , (2016).
- Soriano, P. Generalized lacZ expression with the ROSA26 Cre reporter strain. Nat Genet. 21, 70-71 (1999).
- Liu, X., et al. A targeted mutation at the known collagenase cleavage site in mouse type I collagen impairs tissue remodeling. J Cell Biol. 130 (1), 227-237 (1995).
- Ke, Y. D. Short-term suppression of A315T mutant human TDP-43 expression improves functional deficits in a novel inducible transgenic mouse model of FTLD-TDP and ALS. Acta Neuropathol. 130 (5), 661-678 (2015).
- Auerbach, A. B. Production of functional transgenic mice by DNA pronuclear microinjection. Acta Biochim Pol. 51 (1), 9-31 (2004).
- Iyer, V., et al. Off-target mutations are rare in Cas9-modified mice. Nat Methods. 12 (6), 479(2015).
- Zhou, Y. High-throughput screening of a CRISPR/Cas9 library for functional genomics in human cells. Nature. 509 (7501), 487-491 (2014).
- Brinster, R. L., Chen, H. Y., Trumbauer, M. E., Avarbock, M. R. Translation of globin messenger RNA by the mouse ovum. Nature. 283 (5746), 499-501 (1980).
- Pinkert, C. A., Irwin, M. H., Johnson, L. W., Moffatt, R. J. Mitochondria transfer into mouse ova by microinjection. Transgenic Res. 6 (6), 379-383 (1997).
- Biggers, J. D., Summers, M. C. Choosing a culture medium: making informed choices. Fertil Steril. 90 (3), 473-483 (2008).
- Nakagata, N. Embryo transfer through the wall of the fallopian tube in mice. Jikken Dobutsu. 41 (3), 387-388 (1992).
- Truett, G. E. Preparation of PCR-quality mouse genomic DNA with hot sodium hydroxide and tris (HotSHOT). Biotechniques. 29 (1), 52-54 (2000).
- Richardson, C. D., Ray, G., DeWitt, M. A., Curie, G. L., Corn, J. E. Enhancing homology-directed genome editing by catalytically active and inactive CRISPR-Cas9 using asymmetric donor DNA. Nat Biotechnol. 34 (3), 339-344 (2016).
- Delerue, F., Ittner, L. M. Genome Editing in Mice Using CRISPR/Cas9: Achievements and Prospects. Clon. Transgen. 4, (2015).
Access restricted. Please log in or start a trial to view this content.
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten