需要订阅 JoVE 才能查看此. 登录或开始免费试用。
Methods Article
子结构分析仪:用于荧光显微镜图像中细胞体快速探索和精确分析的用户友好型工作流程
摘要
我们提出了一个自由可用的工作流程,用于在荧光显微镜图像中快速探索和准确分析特定细胞腔中的细胞体。此用户友好的工作流在开源软件 Icy 上设计,并且还使用 ImageJ 功能。在没有图像分析知识的情况下,管道是负担得起的。
摘要
过去十年的特点是荧光显微镜技术的突破,表现为空间分辨率的提高,但也表现在活细胞成像和高通量显微镜技术。这导致单个实验的显微镜数据的数量和复杂性不断增加。由于对显微镜数据的手动分析非常耗时、主观,并且禁止定量分析,生物图像分析的自动化几乎不可避免。我们构建了一个名为子结构分析器的信息学工作流程,以完全自动化荧光显微镜生物图像中的信号分析。此工作流是在用户友好的开源平台 Icy 上开发的,由 ImageJ 的功能完成。它包括图像的预处理,以提高信号与噪声比,细胞的单独分割(细胞边界的检测)和检测/定量的细胞体丰富在特定的细胞隔间。此工作流程的主要优点是通过用户友好的界面向没有图像分析专业知识的用户提出复杂的生物成像功能。此外,它具有高度模块化,适应几个问题,从核/细胞质转移的特征到不同细胞亚结构中不同细胞体的比较分析。通过研究氧化应激(OS)条件下的Cajal(线圈)体,可以说明此工作流程的功能。来自荧光显微镜的数据表明,在进入操作系统几小时后,它们在人体细胞中的完整性会受到影响。这种效应的特点是线圈核化减少为特征的Cajal体,与线圈素的核质再分配成增加的较小 foci 的数量有关。线圈在CB组件与周围核质之间的交换中的核心作用表明,OS诱导的线圈的再分配可能会影响Cajal实体的组成和功能。
引言
光显微镜,特别是荧光显微镜是生物科学中常用的坚固和通用的技术。它们通过特定的荧光标签,获得各种生物分子(如蛋白质或RNA)的精确定位。过去十年的特点是显微镜和成像技术的飞速发展,2014年诺贝尔化学奖授予埃里克·贝齐格、斯特凡·赫尔和威廉·默纳,用于开发超解析荧光显微镜(SRFM)1。SFRM绕过传统光学显微镜的衍射极限,将它引入纳米测量。现场成像或高吞吐量筛选方法等技术的改进也增加了每个实验要处理的数据的数量和复杂性。大多数时候,研究人员面临着细胞的高异质群体,并且想要在单细胞水平上分析表型。
最初,分析,如foci计数是由眼睛执行,这是一些研究人员的首选,因为它提供了对计数过程的完全视觉控制。然而,对此类数据的手动分析过于耗时,导致观察者之间的变异,并且不能访问更复杂的功能,因此计算机辅助方法正在得到广泛使用,几乎不可避免。生物图像信息学方法大大提高了数据分析的效率,没有人工计数分析不可避免的操作主体性和潜在偏差。这一领域需求的增加和计算机功率的提高,导致了大量图像分析平台的发展。其中一些是免费提供的,并允许访问各种工具,以执行分析与个人电脑。开放访问工具的分类最近已建立3,并提出了Icy4作为一个强大的软件结合可用性和功能。此外,冰具有与 ImageJ 通信的优势。
对于没有图像分析专业知识的用户,主要障碍是根据有问题和正确调整通常不被很好地理解的参数选择适当的工具。此外,设置时间通常很长。Icy 提出了一个名为"协议"的用户友好的点击界面,通过组合在详尽的集合 4 中的一些插件来开发工作流。灵活的模块化设计和点击式界面使得非程序员的分析变得可行。在这里,我们提出了一个名为"子结构分析仪"的工作流程,该分析器是在冰的界面中开发的,其功能是分析特定蜂窝隔间中的荧光信号,并测量亮度、foci编号、foci 大小和空间分布等不同要素。此工作流程解决了几个问题,如信号转移的量化、表达荧光报告器的转染细胞分析,或分析单个细胞中不同细胞子结构的 foci。它允许同时处理多个图像,输出结果将导出到选项卡分隔工作表,该工作表可以在常用的电子表格程序中打开。
子结构分析器管道如图1 所示。首先,对指定文件夹中包含的所有图像进行预处理,以提高其信噪比。此步骤可提高以下步骤的效率并缩短运行时间。然后,对与应检测荧光信号的图像区域对应的感兴趣区域 (RO) 进行标识和分段。最后,对荧光信号进行分析,结果导出到选项卡分隔工作表中。
对象分割(边界检测)是图像分析中最具挑战性的一步,其效率决定了生成的细胞测量的准确性。在图像中识别的第一个对象(称为原对象)通常是DNA染色图像(DAPI或霍奇斯特染色)的核,尽管原对象也可以是整个细胞、珠子、斑点、肿瘤或任何染色物体。在大多数生物图像中,细胞或核相互接触或重叠,导致简单而快速的算法失效。迄今为止,没有通用算法能够对所有对象执行完美的分割,主要是因为它们的特性(大小、形状或纹理)调节了分割效率5。通常与显微镜软件一起分发的分段工具(如分子设备变形成像软件6,或尼康7的NIS-元素推进研究软件)通常基于标准技术,如相关匹配、阈值或形态操作。虽然在基本系统中效率很高,但这些过度泛化的方法在更具挑战性和特定性的情况下使用时会迅速产生局限性。事实上,分割对实验参数(如细胞类型、细胞密度或生物标志物)非常敏感,并且经常需要对大型数据集进行重复调整。子结构分析器工作流集成了简单和更复杂的算法,以提出适合图像复杂性和用户需求的不同替代方案。它特别提出了基于标记的流域算法8,用于高聚类对象。此分段方法的效率取决于每个对象上各个标记的选择。这些标记是大多数时间手动选择的,以获得正确的参数进行完全分段,当用户面对大量对象时,这非常耗时。子结构分析仪建议自动检测这些标记,提供高效的分段过程。分割是图像分析的极限步骤,可以根据图像的分辨率、每个图像的对象数和对象的聚类级别大幅修改处理时间。在标准台式计算机上,每个映像需要几秒钟到 5 分钟。分析更复杂的图像可能需要更强大的计算机和图像分析的一些基本知识。
此工作流的灵活性和功能在代表性结果中用各种示例进行了说明。通过研究氧化应激(OS)条件下的核子结构,可以显著地展示这一工作流程的优点。OS 对应于氧化剂的氧化平衡不平衡,并且与高活性氧物种 (ROS) 相关。由于ROS作为信号分子,其浓度和亚细胞定位的变化对无数调节生理功能的通路和网络产生正或负影响,包括信号转导、修复机制、基因表达、细胞死亡和增殖9,9、10。因此,操作系统直接参与各种疾病(神经退行性疾病和心血管疾病,癌症,糖尿病等),但也细胞老化。因此,破译操作系统对人体细胞组织和功能的影响是理解操作系统在人类病理发病和发育中作用的关键一步。已经确定,OS通过调节转录因子(p53,Nrf2,FOXO3A)11来调节基因表达,但也通过影响几个共同和后转录过程的调节,如前RNA12,13,14,13,14的替代拼接(AS)。11主编码和非编码转录的替代方案拼接是一种基本机制,通过生成转录器等构形式来提高基因组的编码能力。AS由一个巨大的核糖核蛋白复合物称为拼接体,包含近300蛋白质和5个U丰富的小型核RNA(UsnRNA)15。拼接体组装和 AS 在细胞中受到严格控制,拼接体成熟的一些步骤发生在名为 Cajal 实体的无膜核隔间内。这些核子结构的特点是其结构及其组成的动态性质,主要通过RNA和蛋白质成分与线圈蛋白的多价相互作用进行。使用子结构分析器工作流程对数千个细胞进行分析,可以对从未描述的操作系统对 Cajal 实体的影响进行表征。事实上,获得的数据表明,OS修改了Cajal实体的核,诱导线圈蛋白的核质再分配到许多较小的核源。卡哈尔体结构的这种变化可能会影响拼接体的成熟,并参与OS的 AS 调制。
研究方案
注:用户友好的教程可在 Icy 的网站http://icy.bioimageanalysis.org 。
1. 下载"结冰"和"子结构分析器"协议
- 从结冰网站http://icy.bioimageanalysis.org/download(http://icy.bioimageanalysis.org/download)下载冰,下载子结构分析器协议:http://icy.bioimageanalysis.org/protocols?sort=latest。.
注意:如果使用 64 位操作系统,请务必使用 64 位版本的 Java。此版本允许增加分配给 Icy 的内存(首选项 |一般 |最大内存)。
2. 打开协议
- 打开"结冰"并单击功能区菜单中的"工具"。
- 单击协议以打开协议编辑器界面。
- 单击"加载"并打开协议子结构分析器。协议加载可能需要几秒钟。在使用协议之前,请确保协议的打开已完成。
注:工作流由图2a 中的 13个常规块组成。每个块都用作由多个执行特定子任务框组成的管道。
3. 与结冰的工作流交互
注:每个块或框都有编号,并且在工作流中具有特定的排名(图 2b)。通过单击此数字,将最接近第一个块/框的位置分配给所选块/框,然后重新组织其他块/框的位置。在准备工作流时,尊重块的右顺序。例如,Spot 检测器块需要预定义的 PI,以便分段块必须先于点检测器块之前运行。不要修改框的位置。不要在图像名称中使用"."
- 通过单击左上角图标,折叠、展开、放大、缩小或删除块(图 2b)。
- 工作流的每个管道的特点是通过输入和输出连接的盒子网络(图2b)。要创建连接,请单击"输出"并维护,直到光标达到输入。可以通过单击输出标记来删除连接。
4. 图像通道的合并
- 使用块合并通道生成合并的图像。如有必要,重命名文件,以便要合并的序列具有相同名称的前缀,后跟一个不同的分隔符。例如,来自图像 A 的单个通道序列名为:ImageA_red,ImageA_blue。
注:对于分隔符,不要使用图像名称中已经存在的字符。 - 在同一文件夹中,每个通道创建一个新文件夹进行合并。例如,要合并红色、绿色和蓝色通道,请创建 3 个文件夹,并将相应的序列存储在这些文件夹中。
- 仅使用块合并通道,删除其他块,并将协议另存为合并通道。
- 访问框以设置参数。对于每个通道,分别填充通道号 X(框1、5 或 9)、文件夹通道号 X(框 2、6 或 10)、分隔通道号 X(框 3、7 或 11)和Colormap 通道 nb X(框4、8 和 12)。
注:这些框按四个水平分组,每行对应于同一通道。在每行中,还提供一个显示(框 23、24 或 25),以直接可视化相应通道的顺序。- 在通道号X 框中,选择要提取的通道(在经典 RGB 图像中,0=红色,1=绿色,2=蓝色)。用户在"序列"选项卡的"检查器"窗口中快速Inspector访问图像的不同通道。 将Sequence最小通道的值写在上行中,在底线中写入最高的通道值。
- 在"文件夹通道编号 X"框中,写入包含通道 X 图像的文件夹的\Name。
- 在分隔符通道编号 X 框中,编写用于图像名称的分隔符(在上一个示例中:"_red","_green"和"_blue")。
- 在"颜色图"通道 nb X 框中,用数字指示要使用哪个颜色图模型来可视化 Icy 中的相应通道。可用颜色图在检查器窗口的"序列"选项卡中可见。
- 在合并图像的"格式"(框 28)中,编写扩展以保存合并图像:.tif、.gif、.jpg、.bmp 或 .png。
注:要仅合并 2 个通道,请勿填充与第三个通道对应的四个框。
- 在"合并通道"块的左上角,单击直接指向文件夹右侧的链接。在显示的"打开"对话框中,双击包含"文件夹通道 1"框(框2)中定义的第一个通道序列的文件夹。然后,单击"打开"。
- 通过单击合并通道块左上角的黑色箭头来运行协议(有关详细信息,请参阅第 7 部分)。合并的图像与单个通道的文件夹保存在同一目录中的合并文件夹中。
- 访问框以设置参数。对于每个通道,分别填充通道号 X(框1、5 或 9)、文件夹通道号 X(框 2、6 或 10)、分隔通道号 X(框 3、7 或 11)和Colormap 通道 nb X(框4、8 和 12)。
5. 感兴趣区域的划分
注:子结构分析器集成了简单和更复杂的算法,以提出适应图像复杂性和用户需求的不同替代方案。
- 选择已调整的块。
- 如果对象不相互接触,或者用户不需要单独区分聚类对象,请使用块分割 A:非聚类对象。
- 当对象不互相接触,但其中一些是接近的时,请使用块分割 B:群集不佳的对象。
- 对于具有高聚类级别和凸形的对象,请使用块分割 C:具有凸形的聚类对象。
- 如果对象呈现高聚类级别且具有不规则形状,请使用块分割D:具有不规则形状的聚类对象。
- 使用块分割E:聚类细胞质,使用分段核作为标记单独分割接触细胞质。此块必须需要分段核进行处理。
注:独立适应主对象分割过程的块,以便在同一运行中可以使用多个块来比较其特定子结构的效率或对不同类型的子结构进行分段。如果聚类级别在同一组图像中是异构的,则分别在调整的块中处理小型和高度聚类对象。
- 将块的输出 0(文件)链接到所选分段块的文件夹输入。
- 设置所选分段块的参数。
- 分割 A:非聚类对象和分段 C:具有凸形的聚类对象
- 在框通道信号(框 1)中,将对象的通道设置为分段。
- 作为选项,在框高斯滤波器(框 2)中,如果对象内部的信号是异构的,则增加 X 和 Y 西格玛值。高斯滤镜平滑纹理,以获得更均匀的区域,并提高核分割的速度和效率。对象越小,西格玛值越低。避免高西格玛值。将默认值设置为 0。
- 在框HK-Means(框 3)中,设置"强度"类参数以及要检测的对象的近似最小大小和最大大小(以像素为单位)。
注:对于强度类,值 2 对 2 类中的像素进行分类:背景和前景。因此,当对象和背景之间的对比度很高时,它被调整。如果前景对象具有不同强度,或者与背景的对比度较低,则增加类数。默认设置为 2。可以通过围绕感兴趣的对象手动绘制 ROI 来快速评估对象大小。ROI 的大小(内部(以像素为单位)在用光标指向图像时直接显示在图像上,或者可以在ROI 统计窗口中访问(从搜索栏中打开)。最佳参数在单个 ROI 中检测每个前景对象。它们可以在"冰"(检测和跟踪 | |HK-表示)。 - 在"活动轮廓"(框 4)框中,优化对象边框的检测。此插件的详尽文档可在线提供:http://icy.bioimageanalysis.org/plugin/Active_Contours。也可以在"结冰"(检测和跟踪||活动轮廓)。
- 在这个过程中,会自动创建一个文件夹来保存分段对象的图像。在"文本"(框 6)框中,请命名此文件夹(例如:分段核)。要设置保存分段对象(Tiff、Gif、Jpeg、BMP、PNG)图像的格式,请填充分段对象图像的框格式。该文件夹在包含合并图像的文件夹中创建。
- 运行工作流(有关详细信息,请参阅第 7 部分)。
- 分段 B:聚类较差的对象
- 按照与 5.3.1 相同的步骤设置框通道信号、HK-Means、活动轮廓、扩展的参数以保存分段对象和文本(框等级与步骤 5.3.1 中不同)。 Channel signal
- 在"调用IJ 插件"(框 4)框中,将滚动参数设置为控制背景减法。将此参数设置为至少为不是背景一部分的最大对象的大小。减小此值会增加背景移除,但也会导致前景信号丢失。
- 在"自适应直方图均衡"(框 6)框中,改进前景对象和背景之间的对比度。增加斜率可生成更多对比度序列。
- 运行工作流(有关详细信息,请参阅第 7 部分)。
- 分割 D:形状不规则的聚类对象
注:三种不同的分割方法适用于每个图像:首先,HK-意味着聚类结合活动轮廓方法应用。然后,经典流域算法(使用 Euclidian 距离图)应用于以前分段错误的对象。最后,基于标记的流域算法使用。只有HK手段和基于标记的流域方法需要用户干预。对于这两种方法,可以应用于所有图像(全自动版本)或更改每个图像(半自动版本)的相同参数。如果用户未接受这些分段方法的训练,则强烈建议进行半自动处理。在处理此块期间,需要手动干预。完成分段方法后,用户必须在下一个分段方法的开头之前手动删除错误分割的对象。成功分割的对象将保存,并且不会在下一步中考虑。此块必须与块连接聚类/异构形状主对象分割对话框正常工作。- 在 11 月 11 日下载 ImageJ https://github.com/ijpb/MorphoLibJ/releases。此协议使用 MorphoLibJ 1.4.0 版本。将文件MorphoLibJ_-1.4.0.jar放在文件夹中。有关此集合内容的更多信息,请访问https://imagej.net/MorphoLibJ。
- 按照步骤5.3.1中相同的步骤设置框通道信号、高斯滤波器、活动轮廓、扩展的参数,以保存分段对象和文本。框等级与步骤 5.3.1 中不一样。
- 设置框自适应直方图均衡的参数(参见步骤 5.3.2.3)。
- 要激活"减法背景",请写"应用减法背景"(方框 5)。否则,写否。如果插件已激活,请设置滚动参数(参见步骤 5.3.2),在"减去背景"参数(框7)中。
- 香港手段的自动化:要对所有图像应用相同的参数(全自动处理),请设置类的 Nb(框 11)、最小大小(框 12)和最大大小(框 13)(参见步骤 5.3.1)。必须设置这些参数以选择最大前景像素并优化前景对象的个性化。对于半自动处理版本,无需干预。
- 标记提取的自动化:对于全自动版本,展开框内部标记提取(框 27),并在脚本第 13 行中设置"动态"参数的值。对于半自动处理版本,无需干预。
注:通过对由"动态"参数控制的输入图像应用扩展-最小变换来提取标记。在基于标记的流域算法中,模拟来自这些标记的泛洪以执行对象分割。为了成功分割前景对象,应提取每个前景对象的单一标记。最佳标记提取的"动态"参数的设置主要取决于图像的分辨率。因此,如果不熟悉此参数,请使用半自动版本。 - 运行工作流(有关详细信息,请参阅第 7 部分)。
- 在处理开始时,对话框HK-意味着参数和基于标记的流域相继打开。要对所有图像(全自动版本)应用相同的参数,请单击"是"。否则,请单击"否"。打开一个信息框,要求"使用 HK-Means 插件确定最佳 ROIS 并关闭图像"。点击确定并手动应用 HK-Means 插件(检测和跟踪|HK-means)在图像上,它会自动打开。在HK-Means 插件框中选择导出 RBI 选项。应用最佳参数以使 PI 包含最大前景像素,并优化前景对象的个性化。找到最佳 PI 时,直接关闭图像。
- 在第一个分段方法的末尾,将打开一个信息框,要求"删除不需要的 ROIS 并关闭图像"。这些 PI 对应于分段对象的边框。选择"确定"并删除图像中错误分段对象的 PI,该对象将自动打开。通过将光标放在其边框并使用键盘的"删除"按钮,可以轻松删除 ROI。关闭图像。完成第二个分段步骤后,重复相同的过程。
- 在此阶段,如果选择 YES 按钮实现基于标记的流域算法的完全自动化,则以前设置的参数将应用于所有图像。
- 如果选择了 NO 按钮,将打开一个信息框,要求"确定和调整内部标记"。单击"确定",在 Icy 的 ImageJ 界面中,转到插件 |莫福利比 |迷你和马克西玛|扩展最小值和最大值。在"操作"中,选择"扩展最小"。
- 选择"预览"可在自动打开的图像上预可视化转换结果。移动动态,直到观察到最佳标记。标记是值为 255(不一定是白色像素)的像素组。最佳参数导致每个对象一个标记。重点关注未使用前两种分段方法进行良好分段的其余对象。
- 如有必要,通过应用其他形态操作(如"打开"或"关闭")(插件 |莫福利比 |形态过滤器)。获取标记的最终图像时,请将其保持打开状态并关闭以图像结尾的所有其他图像,该图像最初用作扩展 Minima 操作的输入。如果ImageJ框要求保存此图像上的更改,请单击"否"。
- 在包含信息框的图像的 Nb 框中(框 14),确定应显示多少具有信息框的图像。
- 分割 E:聚类细胞质
注:此块使用以前分段的核作为单个标记来启动细胞质分割。在使用之前,请确保已处理过核分割块。- 在盒通道细胞质(框 1)中,设置细胞质信号的通道。
- 在"扩展分段核"(框2)框中,编写用于保存分段核图像的格式(tif、jpeg、bmp、png)。默认格式为 tif。
- 在"文本"(框 3)框中,写入包含分段核的文件夹的 \Name。
- 在分段细胞质图像的框中格式(框 4),设置用于保存分段对象图像的格式(Tiff、Gif、Jpeg、BMP、PNG)。
- 在这个过程中,会自动创建一个文件夹来保存分段细胞质的图像。在"文本"(框 5)框中,命名此文件夹(例如:分段细胞质)。该文件夹在包含合并图像的文件夹中创建。
- 按照步骤 5.3.1 中的步骤设置高斯过滤器和主动轮廓框的参数(小心,框等级与步骤 5.3.1 中的步骤不一样)。
- 运行工作流(有关详细信息,请参阅第 7 部分)。
- 分割 A:非聚类对象和分段 C:具有凸形的聚类对象
6. 荧光信号检测与分析
- 选择已调整的块。
- 在块荧光分析 A: 1 通道中,对一种分段物体内一个通道中的 foci 进行检测和分析:检测原子核内的线圈 foci(红色通道)。
- 在块荧光分析B:同一隔间中的2个通道中,对一种分段物体内的两个通道中的 foci 进行检测和分析:在原子核内检测线圈(红色通道)和53BP1(绿色通道)foci。
- 在块荧光分析C:2通道在两个隔间中,执行检测和分析在一个或两个通道,特别是在核及其相应的细胞质内:检测细胞核内及其相应的细胞质,或检测细胞核内的线圈叶(红通道)和G3BP因子(绿色通道)在相应的细胞质内。
- 在块荧光分析 D: 全局位位中,计算来自两个蜂窝隔间(a 和 b)中一个信道的信号百分比。例如,在细胞质/核易位测定中,在最终的"结果"电子表格中导出每个图像的核信号和细胞质信号的计算百分比。用于计算核信号百分比的公式如下所示。此块可用于任何亚细胞隔间:
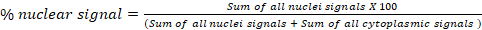
- 在块荧光分析E:单个细胞转移中,计算每个细胞两个细胞隔间中一个信道的信号百分比。该块特别针对单细胞级别的细胞核/细胞质转移测定进行了优化。
注:由于块荧光分析E:单个细胞转移在单细胞水平上执行分析,因此需要有效分割细胞核和细胞质。
- 将块的输出 0(文件)选择文件夹(块 1)链接到所选块的文件夹输入(黑色圆圈中的白色箭头)。
- 设置所选块的参数。
- 荧光分析 A: 1 通道, 荧光分析 B: 同一隔间中的 2 个通道和荧光分析 C: 两个隔间中的 2 个通道
- 在"文件夹图像 ROI"框中,将包含分段对象图像的文件夹名称写在反斜杠之前。(例如: +分段核)。
- 在分段对象图像的框中格式(框 2),编写用于保存分段对象(tif、jpeg、bmp、png)图像的格式。默认格式为 tif。
- 在"杀死边框?"框中,写"是"以删除边框对象。否则,请写否。需要安装 ImageJ 的 MorphoLibJ 集合才能使用此功能(请参见步骤 5.3.3)。
- 在框(es)通道点信号中,设置必须检测到斑点的通道。在经典 RGB 图像中,0=红色、1=绿色和 2=蓝色。
- 在局部分子名称的框中,将分子的名称写在斑点中。要进入的字段数取决于分子的数量。
- 在"框(es)小波点检测器块"中,设置每个通道的点检测参数。设置刻度(指点大小)和检测的灵敏度(灵敏度越小,检测点数越小,默认值为 100,最小值为 0)。此插件的详尽文档可在线提供:http://icy.bioimageanalysis.org/plugin/Spot_Detector。参数也可以在"冰"(检测和跟踪 | |点检测器)。
- 作为选项,在"按大小筛选 ROI"框中,通过设置大小间隔(以像素为单位)筛选检测到斑点的分段对象。此步骤对于删除分段不足或过段的对象特别有用。若要手动估计对象的大小,请参阅步骤 5.3.1。默认参数不包括按大小筛选 ROIS。两个隔间中的块 2 通道包含两个框:按大小过滤核(框 19)和按大小筛选细胞质(框 46)。
- 可选地,在"按大小筛选点"框中,根据检测到的斑点的大小(以像素为单位)筛选斑点以删除不需要的伪影。要手动估计点大小,请单击检测和跟踪并打开Spot 探测器插件。在"输出"选项中,选择"导出到 ROI"。请注意,默认参数不包括按大小筛选斑点,分析时未考虑筛选的斑点。要输入的字段数取决于通道数。
- 可选地,在"滤波点"框中,对检测到的点应用额外的滤镜(对比度、均匀性、周长度、圆度)。请注意,默认参数不包括点滤波,分析中未考虑筛选的斑点。要输入的字段数取决于通道数。
- 可选地,在"点"尺寸阈值框中,为分析点的面积(以像素为单位)设置阈值。低于此阈值的计数点数在最终结果电子表格中导出。要通知的框数取决于通道数。
- 运行工作流(有关详细信息,请参阅第 7 部分)。数据导出到保存在包含合并图像的文件夹中的结果电子表格中。
- 荧光分析 D:全球转移和荧光分析 E:单个细胞转移:
- 在文件夹图像(框1 和 2)框中,写入包含分段对象图像的文件夹的 \Name。在荧光分析D:全球转移块中,这两种类型的ROI被确定为ROI a和ROI b。对于块荧光分析E:单个细胞转移,在文件夹图像分段核和文件夹图像分段细胞质框,分别写包含分段核和细胞质的文件夹的名称。
- 在框通道信号(框 3)中,输入信号的通道。
- 在分段对象图像的框中格式(方框 4),编写用于保存分段对象图像(tif、jpeg、bmp、png)的格式。默认格式为 tif。"终止边框"选项也可用于删除边框对象(请参阅步骤 6.3.1)。
- 可选地,在框中按大小筛选 ROI,通过设置大小间隔(以像素为单位)来筛选分段对象。此步骤可用于删除分段不足或过段的对象。若要手动估计对象大小,请参阅步骤 5.3.1。有两个字段要输入,每个通道一个。默认参数不包括按大小筛选 ROI。
- 运行工作流(有关详细信息,请参阅第 7 部分)。导出电子表格中保存在包含合并图像的文件夹中的结果中的数据。
- 荧光分析 A: 1 通道, 荧光分析 B: 同一隔间中的 2 个通道和荧光分析 C: 两个隔间中的 2 个通道
7. 运行协议
- 若要在运行中处理一个块,请删除所选块和块"选择文件夹"之间的连接。将通缉区放在第 1级。在通缉块的左上角,单击直接指向文件夹右侧的链接。在显示的"打开"对话框中,双击包含合并图像的文件夹。然后,单击"打开"。单击"运行"以启动工作流。可以通过单击"停止"按钮来停止处理。
- 若要在运行中处理不同的块,请使用块选择文件夹(块1)保留所选块的连接。确保其排名允许对工作流进行良好的处理。例如,如果特定块需要处理分段对象,请确保分段块以前处理过。在运行工作流之前,请删除未使用的块,并保存使用另一个名称的新协议。
- 单击"运行"以启动工作流。当打开的对话框出现时,双击包含合并图像的文件夹。然后,单击"打开"。工作流自动运行。如有必要,单击"停止"按钮停止处理。
- 在处理结束时,检查执行的工作流是否成功出现在右下角,以及所有块都用绿色符号标记(图 2b)。如果没有,则显示错误符号的块和内部框指示要更正的元素(图 2b)。
注意:工作流成功执行后,无法直接启动新的运行,并且要再次处理工作流,应至少标记一个块,并标记其符号为"准备处理"。若要更改块的状态,请删除并重新创建此块内两个框之间的链接,或者只需关闭并重新打开协议。如果在处理过程中发生错误,可以直接启动新的运行。在新运行期间,将处理管道的所有块,即使其中一些块用绿色符号标记。
结果
所有描述的分析都在标准笔记本电脑(64 位四核处理器,2.80 GHz,16 GB 随机访问内存 (RAM))上执行,使用 64 位版本的 Java。随机访问内存是需要考虑的重要参数,具体取决于要分析的图像的数量和分辨率。使用 32 位版本的 Java 将内存限制在 1300 MB,这可能不适合大数据分析,而 64 位版本允许增加分配给 Icy 的内存。图 3报告不同类型图像和不同分辨率的分割所需时间。它?...
讨论
越来越多的自由软件工具可用于分析荧光细胞图像。用户必须根据问题的复杂性、图像处理知识以及在分析中花费的时间正确选择适当的软件。冰,细胞产品,或图像J/斐济是强大的工具,结合了可用性和功能3。Icy 是一个独立工具,它呈现清晰的图形用户界面 (GUI),特别是其"协议"点和点击界面,通过该界面可以轻松设计或操作工作流 4。该软件的功能通过?...
披露声明
作者没有什么可透露的。
致谢
G.H. 获得来自微型德莱古埃和辅助技术的研究生奖学金的支持。L.H. 获得洛林大学(ICL)研究生研究金的支持,而Q.T.则由法国国家研究机构(ANR)监督的公共赠款支持,作为第二个"Avenir投资"方案的一部分,FIGHT-HF(参考:ANR-15-RHU457004)。这项工作由全国抵抗军和洛林大学(UMR 7365)资助。
材料
| Name | Company | Catalog Number | Comments |
| 16% Formaldehyde solution (w/v) methanol free | Thermo Fisher Scientific | 28908 | to fix the cells |
| Alexa Fluor 488 of goat anti-rabbit | Thermo Fisher Scientific | A-11008 | fluorescent secondary antibody |
| Alexa Fluor 555 of goat anti-mouse | Thermo Fisher Scientific | A-21425 | fluorescent secondary antibody |
| Alexa Fluor 555 Phalloidin | Thermo Fisher Scientific | A34055 | fluorescent secondary antibody |
| Bovine serum albumin standard (BSA) | euromedex | 04-100-812-E | |
| DMEM | Sigma-Aldrich | D5796-500ml | cell culture medium |
| Duolink In Situ Mounting Medium with DAPI | Sigma-Aldrich | DUO82040-5ML | mounting medium |
| Human: HeLa S3 cells | IGBMC, Strasbourg, France | cell line used to perform the experiments | |
| Hydrogen peroxide solution 30% (H2O2) | Sigma-Aldrich | H1009-100ml | used as a stressing agent |
| Lipofectamine 2000 Reagent | Thermo Fisher Scientific | 11668-019 | transfection reagent |
| Mouse monoclonal anti-coilin | abcam | ab11822 | Coilin-specific antibody |
| Nikon Optiphot-2 fluorescence microscope | Nikon | epifluoresecence microscope | |
| Opti-MEM I Reduced Serum Medium | Thermo Fisher Scientific | 31985062 | transfection medium |
| PBS pH 7.4 (10x) | gibco | 70011-036 | to wash the cells |
| Rabbit polyclonal anti-53BP1 | Thermo Fisher Scientific | PA1-16565 | 53BP1-specific antibody |
| Rabbit polyclonal anti-EDC4 | Sigma-Aldrich | SAB4200114 | EDC4-specific antibody |
| Triton X-100 | Roth | 6683 | to permeabilize the cells |
参考文献
- Möckl, L., Lamb, D. C., Bräuchle, C. Super-resolved fluorescence microscopy: Nobel Prize in Chemistry 2014 for Eric Betzig, Stefan Hell, and William E. Moerner. Angewandte Chemie. 53 (51), 13972-13977 (2014).
- Meijering, E., Carpenter, A. E., Peng, H., Hamprecht, F. A., Olivo-Marin, J. -. C. Imagining the future of bioimage analysis. Nature Biotechnology. 34 (12), 1250-1255 (2016).
- Wiesmann, V., Franz, D., Held, C., Münzenmayer, C., Palmisano, R., Wittenberg, T. Review of free software tools for image analysis of fluorescence cell micrographs. Journal of Microscopy. 257 (1), 39-53 (2015).
- de Chaumont, F., et al. Icy: an open bioimage informatics platform for extended reproducible research. Nature Methods. 9 (7), 690-696 (2012).
- Girish, V., Vijayalakshmi, A. Affordable image analysis using NIH Image/ImageJ. Indian J Cancer. 41 (1), 47 (2004).
- Zaitoun, N. M., Aqel, M. J. Survey on image segmentation techniques. Procedia Computer Science. 65, 797-806 (2015).
- . MetaMorph Microscopy Automation and Image Analysis Software Available from: https://www.moleculardevices.com/products/cellular-imaging-systems/acquisition-and-analysis-software/metamorph-microscopy (2018)
- . NIS-Elements Imaging Software Available from: https://www.nikon.com/products/microscope-solutions/lineup/img_soft/nis-element (2014)
- Meyer, F., Beucher, S. Morphological segmentation. Journal of Visual Communication and Image Representation. 1 (1), 21-46 (1990).
- Schieber, M., Chandel, N. S. ROS Function in Redox Signaling and Oxidative Stress. Current Biology. 24 (10), 453-462 (2014).
- D'Autréaux, B., Toledano, M. B. ROS as signalling molecules: mechanisms that generate specificity in ROS homeostasis. Nature Reviews. Molecular Cell Biology. 8 (10), 813-824 (2007).
- Davalli, P., Mitic, T., Caporali, A., Lauriola, A., D'Arca, D. ROS, Cell Senescence, and Novel Molecular Mechanisms in Aging and Age-Related Diseases. Oxidative Medicine and Cellular Longevity. 2016, 3565127 (2016).
- Disher, K., Skandalis, A. Evidence of the modulation of mRNA splicing fidelity in humans by oxidative stress and p53. Genome. 50 (10), 946-953 (2007).
- Takeo, K., et al. Oxidative stress-induced alternative splicing of transformer 2β (SFRS10) and CD44 pre-mRNAs in gastric epithelial cells. American Journal of Physiology - Cell Physiology. 297 (2), 330-338 (2009).
- Seo, J., et al. Oxidative Stress Triggers Body-Wide Skipping of Multiple Exons of the Spinal Muscular Atrophy Gene. PLOS ONE. 11 (4), 0154390 (2016).
- Will, C. L., Luhrmann, R. Spliceosome Structure and Function. Cold Spring Harbor Perspectives in Biology. 3 (7), 003707 (2011).
- Ljosa, V., Sokolnicki, K. L., Carpenter, A. E. Annotated high-throughput microscopy image sets for validation. Nature Methods. 9 (7), 637-637 (2012).
- Wang, Q., et al. Cajal bodies are linked to genome conformation. Nature Communications. 7, (2016).
- Carpenter, A. E., et al. CellProfiler: image analysis software for identifying and quantifying cell phenotypes. Genome Biology. 7, 100 (2006).
- McQuin, C., et al. CellProfiler 3.0: Next-generation image processing for biology. PLoS Biology. 16 (7), 2005970 (2018).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。