このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Methods Article
サブ構造アナライザー:蛍光顕微鏡画像における細胞体の迅速な探査と正確な分析のためのユーザフレンドリーなワークフロー
要約
蛍光顕微鏡画像で特定の細胞区画内の細胞体を迅速に探査し、正確に分析するために構築された自由に利用可能なワークフローを提示します。このユーザーフレンドリーなワークフローはオープンソースソフトウェアのIcy上で設計され、また、ImageJの機能を使用しています。パイプラインは、画像解析に関する知識なしに手頃な価格です。
要約
過去10年間は、空間分解能の向上だけでなく、ライブセルイメージングおよびハイスループット顕微鏡技術においても示された蛍光顕微鏡技術のブレークスルーによって特徴付けられています。これにより、1回の実験での顕微鏡データの量と複雑さが一定に増加しました。顕微鏡データの手動分析は非常に時間がかかり、主観的で、定量的な分析が禁止されているため、バイオイメージ解析の自動化はほとんどやむを得なくなっています。蛍光顕微鏡によるバイオイメージのシグナル解析を完全に自動化するサブストラクチャアナライザと呼ばれる情報学ワークフローを構築しました。このワークフローは、ユーザーフレンドリーなオープンソースプラットフォームIcy上で開発され、ImageJの機能によって完了されます。信号対雑音比を改善するための画像の前処理、細胞の個別のセグメンテーション(細胞境界の検出)、特定の細胞コンパートメントに富んだ細胞体の検出/定量化が含まれます。このワークフローの主な利点は、ユーザーフレンドリーなインターフェースを介して、画像解析の専門知識を持たないユーザーに複雑なバイオイメージング機能を提案することです。さらに、それは非常にモジュール化され、異なる細胞のサブ構造の異なる細胞体の比較分析に核/細胞質転位の特徴付けからいくつかの問題に適応する。このワークフローの機能は酸化ストレス(OS)条件下でのカハル(コイル状)体の研究を通して示される。蛍光顕微鏡のデータは、ヒト細胞におけるその完全性がOSの誘導の数時間後に影響を受けることを示している。この効果は、コイリンの核分裂が特徴カジャール体に減少し、小さな病巣の増加にコイリンの核内再分配に関連することを特徴とする。CB成分と周囲核内核形成との交換におけるコイリンの中心的な役割は、OSによるコイリンの再分配がカハル体の組成および機能に影響を及ぼす可能性があることを示唆している。
概要
光顕微鏡、そしてより具体的には、蛍光顕微鏡は、生物科学で一般的に使用される堅牢で汎用性の高い技術です。タンパク質やRNAなどの様々な生体分子の特異的蛍光標識を通じて、正確に局在化できます。過去10年間は、超解決蛍光顕微鏡(SRFM)の開発のためにエリック・ベッツィヒ、ステファン・W・ヘル、ウィリアム・E・モアナーを受賞した2014年のノーベル化学賞によって証明された顕微鏡および画像技術の急速な進歩によって特徴付けられています。SFRMは、従来の光学顕微鏡の回折限界をバイパスしてナノ次元に持ち込みます。ライブイメージングやハイスループットスクリーニングアプローチなどの技術の改善は、各実験で扱うデータの量と複雑度も増加します。ほとんどの場合、研究者は細胞の高い不均一な集団に直面しており、単一細胞レベルでの発現型の分析を望んでいます。
当初、病巣計数などの分析は目で行われ、カウントプロセスを完全に視覚的に制御できるため、一部の研究者が好む。しかし、このようなデータの手動分析は時間がかかりすぎて、観察者間の変動を招き、より複雑な機能にアクセスすることは不可能であり、コンピュータ支援アプローチが広く使用されるようになり、ほとんどやむを得ない2。バイオイメージ情報学的手法は、データ分析の効率を大幅に向上させ、手動計数解析の不可避的なオペレータの主観性と潜在的バイアスを含まない。この分野での需要の増加とコンピュータのパワーの向上は、多数の画像解析プラットフォームの開発につながりました。一部は自由に利用でき、パーソナルコンピュータで分析を行うための様々なツールにアクセスできます。オープンアクセスツールの分類は最近確立されています3そして、使いやすさと機能を組み合わせた強力なソフトウェアとしてIcy4を提示.さらに、アイシーはImageJと通信する利点があります。
サブ構造アナライザーパイプラインは図 1に示されています。まず、指定されたフォルダに含まれるすべての画像が事前処理され、信号とノイズの比率が向上します。この手順により、次の手順の効率が向上し、実行時間が短縮されます。次に、蛍光シグナルが検出されるべき画像領域に対応する対象領域(ROI)を特定し、セグメント化する。最後に蛍光シグナルを分析し、その結果をタブ区切りのワークシートにエクスポートします。
オブジェクトのセグメンテーション(境界の検出)は、画像解析の中で最も困難なステップであり、その効率によって得られる細胞測定の精度が決まります。画像内で最初に同定された物体(主体物体と呼ばれる)は、しばしばDNA染色画像(DAPIまたはHoechst染色)からの核であるが、主な物体は細胞全体、ビーズ、斑点、腫瘍、または染色された物体である可能性もある。ほとんどの生物学的画像では、細胞または核が互いに接触したり、重なり合ったりして、単純で速いアルゴリズムが失敗します。現在までに、ユニバーサルアルゴリズムは、すべてのオブジェクトの完全なセグメンテーションを実行することはできません。5一般的に顕微鏡ソフトウェア(分子デバイス6によるMetaMorphイメージングソフトウェア、ニコン7によるNIS-Elements Advances Researchソフトウェアなど)で配布されるセグメンテーションツールは、一般的に相関マッチング、閾値、形態演算などの標準的な手法に基づいています。基本的なシステムでは効率的ですが、これらの一般化された方法は、より困難で特定のコンテキストで使用すると、急速に限界を提示します。実際、セグメンテーションは、細胞タイプ、細胞密度、バイオマーカーなどの実験パラメータに対して高感度であり、大きなデータセットに対して繰り返し調整を必要とする場合が多い。Substructure Analyzerワークフローは、シンプルで洗練されたアルゴリズムの両方を統合し、画像の複雑さとユーザーのニーズに合わせたさまざまな選択肢を提案します。特に、高度にクラスター化されたオブジェクトに対するマーカーベースの流域アルゴリズム8を提案しています。このセグメンテーション方法の効率は、各オブジェクトの個々のマーカーの選択に依存します。これらのマーカーは、ほとんどの場合、ユーザーが多数のオブジェクトに直面する場合に非常に時間がかかる、完全なセグメンテーションのための正しいパラメータを取得するために手動で選択されます。サブストラクチャアナライザは、これらのマーカーの自動検出を提案し、高効率のセグメンテーションプロセスを提供します。セグメンテーションは、ほとんどの場合、画像解析の制限ステップであり、画像の解像度、画像あたりのオブジェクト数、およびオブジェクトのクラスタリングのレベルに応じて処理時間を大幅に変更できます。標準的なパイプラインでは、標準のデスクトップ コンピューターでイメージあたり数秒から 5 分を必要とします。より複雑な画像の解析には、より強力なコンピュータと画像解析の基本的な知識が必要になる場合があります。
このワークフローの柔軟性と機能性は、代表的な結果にさまざまな例を示しています。このワークフローの利点は、酸化ストレス(OS)条件下での核下構造の研究を通じて顕著に表示されます。OSは酸化剤を支持する酸化還元恒常性の不均衡に対応し、高レベルの活性酸素種(ROS)に関連付けられている。ROSはシグナル伝達分子として作用するので、それらの濃度および細胞内局在の変化は、シグナル伝達、修復機構、遺伝子発現、細胞死、増殖を含む生理機能を調節する無数の経路およびネットワークにプラスまたは負の影響を及9,ぼす。OSは、様々な病理(神経変性、心血管疾患、癌、糖尿病など)に直接関与するが、細胞老化にも関与している。したがって、ヒト細胞の組織と機能に及ぼすOSの結果を解読することは、ヒト病理の発症および発達におけるOSの役割を理解する上で重要なステップとなる。OSは、いくつかの転写因子(p53、Nrf2、FOXO3A)11を介して転写を調節することによって遺伝子発現を調節することが確立されている11が、また、RNA12、13、14,13,14の代替スプライシング(AS)などのいくつかの共転写および転写後プロセスの調節に影響を与えることによっても確立されている。一次符号化および非コード転写物の代替スプライシングは、トランスクリプトアイソフォームを産生することによってゲノムのコード容量を増加させる不可欠なメカニズムである。ASは、スプライセオソームと呼ばれる巨大なリボヌクレオタンパク質複合体によって行われ、ほぼ300個のタンパク質および5つのUリッチ15な小型核RNA(UsnRNA)15を含む。スプライセオソームアセンブリとASは細胞内で厳しく制御されており、スプライセサム成熟のいくつかのステップは、カハルボディと名付けられた膜のない核コンパートメント内で起こる。これらの核下構造は、その構造の動的性質とそれらの組成によって特徴付けられるもので、主にそれらのRNAとタンパク質成分とコイリンタンパク質との多価相互作用によって行われる。Substructure Analyzerワークフローを使用して何千ものセルを分析すると、CAJALボディに対するOSの影響を説明したことがないという特徴付けが可能になりました。実際、得られたデータは、OSがカハル体の核形成を変え、コイリンタンパク質の核内再分配を多数の小さな核病巣に誘導することを示唆している。カハルボディの構造のこのような変化は、スプライセソームの成熟に影響を与え、OSによるAS変調に参加する可能性があります。
プロトコル
注:ユーザーフレンドリーなチュートリアルは、Icyのウェブサイトhttp://icy.bioimageanalysis.orgで入手できます。
1. 氷とサブ構造アナライザプロトコルをダウンロード
- Icy の Web サイト (http://icy.bioimageanalysis.org/download)から Icy をダウンロードし、サブ構造アナライザー プロトコルhttp://icy.bioimageanalysis.org/protocols?sort=latestをダウンロードします。
注: 64 ビット OS を使用する場合は、必ず 64 ビットバージョンの Java を使用してください。このバージョンでは、ICYに割り当てられたメモリを増やすことができます (環境設定 |一般 |最大メモリ)。
2. プロトコルを開く
- [ICY] を開き、リボンメニューの[ツール] をクリックします。
- [プロトコル] をクリックして、[プロトコル エディタ]インターフェイスを開きます。
- [読み込み] をクリックし、プロトコルのサブ構造アナライザを開きます。プロトコルの読み込みには数秒かかることがあります。プロトコルを使用する前に、プロトコルのオープンが完了していることを確認してください。
注: ワークフローは、図 2aに示す 13 個の一般ブロックで構成されています。各ブロックは、特定のサブタスクを実行する複数のボックスで構成されるパイプラインとして機能します。
3. Icy 上のワークフローとの対話
注: 各ブロックまたはボックスには番号が付けられており、ワークフロー内の特定のランクが付けられます (図 2b)。この番号をクリックすると、最初のブロック/ボックスに最も近い位置が割り当てられ、他のブロック/ボックスの位置が再編成されます。ワークフローを準備する際には、ブロックの正しい順序を尊重します。たとえば、スポット ディテクター ブロックには、スポット ディテクター ブロックの前にセグメンテーション ブロックを実行する必要があるため、事前に定義された ROI が必要です。ボックスの位置を変更しないでください。画像名に "." を使用しないでください。
- 左上隅のアイコンをクリックすると、ブロックを折りたたんだり、展開したり、拡大したり、狭めたり、削除したりします(図2b)。
- ワークフローの各パイプラインは、入力と出力を介して接続されたボックスのネットワークによって特徴付けられます(図 2b)。接続を作成するには、[出力]をクリックし、カーソルが入力に到達するまで維持します。接続は、Outputタグをクリックして削除できます。
4. 画像のチャンネルの結合
- [チャネルのマージ]ブロックを使用して、マージされたイメージを生成します。必要に応じて、マージするシーケンスの名前に、同じ名前の接頭辞の後に別の区切り文字が付くようにファイル名を変更します。たとえば、イメージ A の個々のチャネルのシーケンスには、ImageA_red、ImageA_blueという名前が付けられます。
注: 区切り記号については、イメージ名に既に含まれる文字を使用しないでください。 - 同じフォルダに、マージするチャネルごとに 1 つの新しいフォルダを作成します。たとえば、赤、緑、青のチャンネルを結合するには、3 つのフォルダを作成し、対応するシーケンスをこれらのフォルダに保存します。
- [マージ チャネル] ブロックのみを使用し、他のブロックを削除し、プロトコルを[チャネルのマージ]として保存します。
- パラメータを設定するには、ボックスにアクセスします。各チャネルについて、チャネル番号 X (ボックス 1、5、または 9)、フォルダー チャネル番号 X (ボックス 2、6、または 10)、区切りチャネル番号 X (ボックス 3、7、または 11)、カラーマップ チャネル nb X (ボックス 4、8、12) をそれぞれ入力します。
注: これらのボックスは、同じチャネルに対応する各行を 4 つ分ずつ横にグループ化しています。各行には、対応するチャンネルのシーケンスを直接可視化するためのディスプレイ(ボックス23、24、または25)も利用できます。- [チャンネル番号 X]ボックスで、抽出するチャンネルを選択します (クラシック RGB 画像では、0 =赤、1 = 緑、2=青)。「シーケンス」タブの「Icy」インスペクタ・ウィンドウ内の画像のさまざまなチャンネルに、ユーザーがすばやくアクセスします。
- [フォルダチャネル番号 X]ボックスに、チャンネル X のイメージを含むフォルダの \Name を入力します。
- [区切りチャネル番号 X]ボックスに、イメージの名前に使用する区切り記号を入力します (前の例では、"_red"、"_green"、"_blue" を入力します)。
- [カラーマップ チャネル nb X]ボックスで、対応するチャネルを Icy で視覚化するために使用するカラーマップ モデルを数値で示します。使用可能なカラーマップは、「インスペクタ」ウィンドウの「シーケンス」タブに表示されます。
- [マージされたイメージの形式] ボックス (ボックス 28) に、マージされたイメージを保存する拡張子を書き込みます。
注: 2 つのチャンネルのみをマージするには、3 番目のチャネルに対応する 4 つのボックスを埋めないでください。
- [チャンネルの結合] ブロックの左上隅にある [フォルダ] の右にあるリンクをクリックします。表示される [開く] ダイアログ ボックスで、[フォルダ チャネル番号 1 (ボックス 2) で定義されている最初のチャネルのシーケンスを含むフォルダーをダブルクリックします。次に、[開く] をクリックします。
- [チャネルのマージ] ブロックの左上隅にある黒い矢印をクリックして、プロトコルを実行します (詳細については、パート 7 を参照してください)。マージされた画像は、個々のチャンネルのフォルダと同じディレクトリのMergeフォルダに保存されます。
- パラメータを設定するには、ボックスにアクセスします。各チャネルについて、チャネル番号 X (ボックス 1、5、または 9)、フォルダー チャネル番号 X (ボックス 2、6、または 10)、区切りチャネル番号 X (ボックス 3、7、または 11)、カラーマップ チャネル nb X (ボックス 4、8、12) をそれぞれ入力します。
5. 関心領域のセグメンテーション
注: Substructure Analyzer は、シンプルで洗練されたアルゴリズムを統合し、画像の複雑さとユーザーのニーズに合わせたさまざまな選択肢を提案します。
- 適合ブロックを選択します。
- オブジェクトが互いに接触しない場合、またはユーザーがクラスタ化されたオブジェクトを個別に区別する必要がない場合は、ブロックセグメンテーション A: 非クラスタ化オブジェクトを使用します。
- オブジェクトが互いに接触せず、オブジェクトの一部が近い場合は、ブロックセグメンテーション B: クラスタ化が不十分なオブジェクトを使用します。
- 高いクラスタ化レベルと凸形状のオブジェクトの場合は、ブロックセグメンテーション C: 凸形状のクラスタ化オブジェクトを使用します。
- オブジェクトが高いクラスタ レベルを持ち、不規則な形状を持つ場合は、ブロックセグメンテーション D: 不規則な形状を持つクラスタ化オブジェクトを使用します。
- ブロックセグメンテーションE:クラスター化された細胞質を使用して、セグメント化された核をマーカーとして使用して個別に触れるサイトプラズムをセグメント化します。このブロックは、処理するためにセグメント化された核を必要とします。
注: 一次オブジェクトセグメンテーション プロセスに個別に適合したブロックは、複数のブロックを同じ実行で使用して、特定のサブ構造の効率を比較したり、異なるタイプのサブ構造をセグメント化したりできるようにします。同じイメージセット内でクラスタリングレベルが異種である場合は、適合ブロック内で小さくて高度にクラスタ化されたオブジェクトを別々に処理します。
- ブロックのoutput0 (ファイル)を[フォルダを選択]を、選択したセグメンテーション ブロックのフォルダ入力にリンクします。
- 選択したセグメンテーション ブロックのパラメータを設定します。
- セグメンテーション A: 非クラスタ化オブジェクトとセグメンテーション C: 凸形状を持つクラスタ化オブジェクト
- ボックスチャネル信号(ボックス1)で、オブジェクトのチャンネルをセグメントに設定します。
- オプションとして、ボックスガウスフィルタ(ボックス2)で、オブジェクト内の信号が不均一な場合は、XおよびYシグマの値を増やします。ガウスフィルタは、テクスチャを滑らかにして、より均一な領域を得て、核のセグメンテーションの速度と効率を向上させます。オブジェクトが小さいほど、シグマ値は低くなります。高いシグマ値は避けてください。既定値を 0 に設定します。
- ボックスHK-Means (ボックス 3)で、[強度クラス] パラメーターと、検出するオブジェクトの最小サイズと最大サイズ (ピクセル単位) を設定します。
注: 強度クラスの場合、値 2 は、背景と前景の 2 つのクラスのピクセルを分類します。このように、オブジェクトと背景のコントラストが高い場合に適応されます。前景オブジェクトの強度が異なる場合や、背景とのコントラストが低い場合は、クラス数を増やします。デフォルト設定は 2 です。対象のオブジェクトの周囲に手動で ROI を描画することで、オブジェクトのサイズを簡単に評価できます。ROI のサイズ (ピクセル単位) は、カーソルでイメージをポイントすると画像に直接表示されるか、ROI 統計情報ウィンドウでアクセスできます(検索バーから開きます)。最適パラメータは、各前景オブジェクトを単一のROIで検出します。これらは、ICY (検出と追跡 |HK-Means)。 - [アクティブコンター ](ボックス 4)で、オブジェクトの境界線の検出を最適化します。このプラグインの包括的なドキュメントはオンラインで入手できます: http://icy.bioimageanalysis.org/plugin/Active_Contours.正しいパラメータは、Icy (検出とトラッキング |アクティブな輪郭。
- このプロセス中に、セグメント化されたオブジェクトのイメージを保存するフォルダが自動的に作成されます。テキスト (ボックス6) ボックスに、このフォルダー (例: セグメント化された核) に名前を付けます。セグメント化されたオブジェクトのイメージ (Tiff、Gif、Jpeg、BMP、PNG) の保存形式を設定するには、セグメント化されたオブジェクトのイメージのボックス形式を入力します。マージされたイメージを含むフォルダにフォルダが作成されます。
- ワークフローを実行します (詳細については、パート 7 を参照してください)。
- セグメンテーション B: クラスター化が不十分なオブジェクト
- 5.3.1 と同じ手順で、セグメント化されたオブジェクトとテキストを保存するための、ボックスチャネル信号、HK-平均、アクティブコンター、拡張のパラメータを設定します (ボックスのランクはステップ 5.3.1 と同じではありません)。
- 「IJ プラグインを呼び出す」(ボックス 4)のボックスで、バックグラウンド減算を制御するRollingパラメーターを設定します。このパラメーターは、背景の一部ではない最大のオブジェクトのサイズを少なくとも設定します。この値を小さくすると、バックグラウンドの削除が増加しますが、フォアグラウンド信号の損失を引き起こす可能性もあります。
- [アダプティブ ヒストグラムイコライゼーション]ボックス(ボックス 6)で、前景オブジェクトと背景のコントラストを改善します。傾斜を大きくすると、より対照的なシーケンスが表示されます。
- ワークフローを実行します (詳細については、パート 7 を参照してください)。
- セグメンテーション D: 不規則な形状を持つクラスタ化されたオブジェクト
注: セグメンテーションの 3 つの異なる方法は、各イメージに適用されます: まず、HK 平均クラスタリングと組み合わさアクティブコンター法が適用されます。次に、古典的な流域アルゴリズム(ユークリッド距離マップを使用して) は、以前に誤ってセグメント化されたオブジェクトに適用されます。最後に、マーカーベースの流域アルゴリズムが使用されます。ユーザーの介入が必要なのは、HK 平均法とマーカーベースの流域メソッドだけです。どちらの方法でも、すべての画像に同じパラメータを適用することも(完全に自動化されたバージョン)、または各画像に対して変更することもできます(半自動バージョン)。ユーザーがこれらのセグメンテーション方法でトレーニングを受けていない場合は、半自動処理を強くお勧めします。このブロックの処理中は、手動による介入が必要です。セグメンテーションメソッドが終了したら、ユーザーは次のセグメンテーションメソッドの開始前に、誤ったセグメント化されたオブジェクトを手動で削除する必要があります。正常にセグメント化されたオブジェクトは保存され、次のステップでは考慮されません。このブロックはブロックと接続する必要があります[クラスタ化/異種図形] プライマリ オブジェクトの [セグメント化] ダイアログ ボックスをクリックして正しく動作します。- https://github.com/ijpb/MorphoLibJ/releasesで ImageJ コレクション MorphoLibJをダウンロードします。このプロトコルでは、MorphoLibJ 1.4.0 バージョンが使用されています。ファイルをMorphoLibJ_-1.4.0.jarフォルダー icy/ij/plugins に配置します。このコレクションのコンテンツの詳細については、https://imagej.net/MorphoLibJを参照してください。
- ステップ 5.3.1 と同じ手順に従って、セグメント化されたオブジェクトと Text を保存するための、チャネル信号、ガウスフィルタ、アクティブコンター、拡張のパラメータを設定します。ボックスのランクは、ステップ 5.3.1 とは同じではありません。
- [適応型ヒストグラムイコライゼーション]ボックスのパラメータを設定します(ステップ5.3.2.3を参照)。
- [背景を減算] をアクティブにするには、[減算背景を適用しますか?] (ボックス 5) に「はい」と入力します。それ以外の場合は、いいえ を書き込みます。プラグインがアクティブになっている場合は、ローリングパラメータ(ステップ 5.3.2 を参照)を設定し、ボックスの背景パラメータを減算(ボックス 7) で。
- HK手段の自動化:すべてのイメージに同じパラメータを適用するには(完全に自動化された処理)、クラスのNb(ボックス11)、最小サイズ(ボックス12)、最大サイズ(box13)を設定します(ステップ5.3.1を参照)。これらのパラメータは、最大前景ピクセルを選択し、前景オブジェクトの個別化を最適化するために設定する必要があります。半自動処理バージョンでは、介入は必要ありません。
- マーカー抽出の自動化:完全自動化バージョンの場合は、「内部マーカー抽出(ボックス 27)」ボックスを展開し、スクリプトの 13 行目に「動的」パラメーターの値を設定します。半自動処理バージョンでは、介入は必要ありません。
注: マーカーは、「動的」パラメータによって制御される入力イメージに拡張ミニマ変換を適用することによって抽出されます。マーカーベースの流域アルゴリズムでは、これらのマーカーからのフラッディングがシミュレートされ、オブジェクトのセグメンテーションが実行されます。前景オブジェクトのセグメンテーションが成功するためには、フォアグラウンドオブジェクトごとに1つのマーカーを抽出する必要があります。最適なマーカー抽出のための「動的」パラメータの設定は、主に画像の解像度に依存します。したがって、このパラメータに慣れていない場合は、半自動化バージョンを使用してください。 - ワークフローを実行します (詳細については、パート 7 を参照してください)。
- 処理の開始時に、ダイアログ ボックスHK 平均パラメータとマーカー ベースの集水域が連続して開きます。すべてのイメージに同じパラメータを適用するには(完全に自動化されたバージョン)、[はい]をクリックします。それ以外の場合は、[いいえ] をクリックします。「HK-Meansプラグインで最適なROIを決定し、画像を閉じる」ことを求める情報ボックスが開きます。OKをクリックして、手動でHK-Meansプラグインを適用します (検出とトラッキング|HK-平均)が自動的に開きます。HK-Means プラグインボックスで「ROIをエクスポート」オプションを選択します。最大前景ピクセルを含む ROI を持ち、フォアグラウンド オブジェクトの個別化を最適化するには、最適なパラメータを適用します。最適な ROI が見つかったら、画像を直接閉じます。
- 最初のセグメンテーション方法の最後に、情報ボックスが開き、「不要なROIを削除して画像を閉じる」と尋ねられます。これらの ROI は、セグメント化されたオブジェクトの境界に対応します。[OK]を選択し、イメージ内の誤ったセグメントオブジェクトの ROI を削除すると、自動的に開きます。ROI は、その境界線上にカーソルを置いて、キーボードの「削除」ボタンを使用して簡単に削除できます。画像を閉じます。2 番目のセグメンテーション ステップが完了した後も同じ手順を繰り返します。
- この段階で、マーカーベースの流域アルゴリズムのフルオートマタイズに YES ボタンが選択されている場合、以前に設定したパラメータがすべての画像に適用されます。
- NOボタンを選択すると、情報ボックスが開き、「内部マーカーを決定して調整する」ことを求める。OKをクリックし、IcyのImageJインターフェイス内で、プラグインに移動|モーフォリブJ |ミニマとマキシマ|拡張最小と最大.[操作] で 、[拡張ミニマ] を選択します。
- [プレビュー]を選択すると、自動的に開かれた画像に変換結果が事前に表示されます。最適なマーカーが観察されるまで、ダイナミックを移動します。マーカーは、値が 255 (必ずしも白いピクセルではない) のピクセルのグループです。最適なパラメータは、オブジェクトごとに1つのマーカーにつながります。前の 2 つのセグメンテーション メソッドで適切にセグメント化されていない残りのオブジェクトに注目します。
- 必要に応じて、「開く」や「閉じる」などの追加の形態学的操作を適用してマーカーを改善する(プラグイン|モーフォリブJ |形態素フィルター)。マーカーの最終的な画像を取得する場合は、それを開いたままにして、拡張ミニマ操作の入力として最初に使用された画像で終わる他のすべての画像を閉じます。ImageJ ボックスで、このイメージに変更を保存するように求める場合は、[いいえ] をクリックします。
- 情報ボックス (ボックス 14)を持つ画像のボックス Nbで、情報ボックスを持つ画像の数を決定します。
- セグメンテーション E: クラスター化細胞質
注: このブロックは、以前にセグメント化された核を個別のマーカーとして使用して、細胞質セグメンテーションを開始します。核セグメントのブロックが使用する前に処理されていることを確認してください。- ボックスチャネル細胞質(ボックス1)で、細胞質信号のチャネルを設定します。
- [拡張セグメント化された核](ボックス 2)に、セグメント化された核のイメージを保存するために使用される形式を書き込みます(tif、jpeg、bmp、png)。デフォルトのフォーマットは tif です。
- テキスト(ボックス 3) ボックスに、セグメント化された核を含むフォルダーの \Name を書き込みます。
- [セグメント化されたサイトプラクムのイメージの形式] ボックス (ボックス 4) で、セグメント化されたオブジェクトのイメージ (Tiff、GIF、Jpeg、BMP、PNG) の保存に使用する形式を設定します。
- このプロセス中に、セグメント化されたサイトプラズムのイメージを保存するフォルダが自動的に作成されます。テキスト(ボックス 5) ボックスに、このフォルダーに名前を付けます (例: セグメント化サイトプラズム)。マージされたイメージを含むフォルダにフォルダが作成されます。
- ステップ 5.3.1 と同じ手順に従って、ボックスのガウス フィルタとアクティブ コンターのパラメータを設定します (ボックスのランクは手順 5.3.1 と同じではありません)。
- ワークフローを実行します (詳細については、パート 7 を参照してください)。
- セグメンテーション A: 非クラスタ化オブジェクトとセグメンテーション C: 凸形状を持つクラスタ化オブジェクト
6. 蛍光シグナル検出と分析
- 適合ブロックを選択します。
- ブロック蛍光分析A:1チャネルで、1つのタイプのセグメント化されたオブジェクトの中の1つのチャネルでの病巣の検出および分析を行う:核内のコイリン病巣(赤チャネル)の検出。
- ブロック蛍光分析B:同じコンパートメント内の2チャンネルで、1つのタイプのセグメント化されたオブジェクトの中の2つのチャネルでのフォチの検出と分析を行う:核内のコイリン(赤チャネル)および53BP1(緑チャネル)病巣の検出。
- ブロック蛍光分析C:2つのコンパートメント内の2つのチャネルは、核内の特異的に1つまたは2つのチャネルで、核内の特異的な検出および対応する細胞質(赤チャネル)の両方のコイリン病巣(赤チャネル)の検出、または対応する細胞質内のコイリン病巣(赤チャネル)の検出および対応する細胞内のコイリン病巣(緑色チャネル)の検出を行う。
- ブロック蛍光分析D:グローバル転位で、2つのセルラーコンパートメント(aとb)の1つのチャネルからの信号の割合を計算します。例えば、細胞質/核転座アッセイでは、最終的な「結果」スプレッドシートの各画像に対して、計算された核信号と細胞質信号の割合をエクスポートします。核信号の割合を計算するために使用される式を以下に示します。このブロックは、任意の細胞内区画に使用できます。
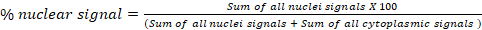
- ブロック蛍光分析 E: 個々の細胞転位で、各セルの 2 つのセルラーコンパートメント内の 1 つのチャネルからの信号の割合を計算します。このブロックは単一細胞レベルの核/細胞質転座アッセイのために特別に最適化される。
注:ブロック蛍光分析E:個々の細胞転位は単一細胞レベルで分析を行うので、核と細胞質の効率的なセグメンテーションが必要です。
- ブロック選択フォルダ(ブロック1)のoutput0(ファイル)を、選択したブロックのフォルダ入力(黒丸の白い矢印)にリンクします。
- 選択したブロックのパラメータを設定します。
- 蛍光分析A:1チャネル、蛍光分析B:同じコンパートメント内の2チャンネルと蛍光分析C:2つのコンパートメント内の2チャンネル
- [フォルダ イメージの ROI] ボックスに、円記号が付いたセグメント化されたオブジェクトのイメージを含むフォルダの名前を入力します。(例: \セグメント化された核)。
- セグメント化されたオブジェクトのイメージのフォーマット(ボックス 2) に、セグメント化されたオブジェクトのイメージを保存するために使用する形式 (tif、jpeg、bmp、png) を書き込みます。デフォルトのフォーマットは tif です。
- [罫線を殺す]ボックスに [はい] と入力して、枠線オブジェクトを削除します。それ以外の場合は、Noと書き込みます。この関数を使用するには、ImageJ の MorphoLibJ コレクションのインストールが必要です (ステップ 5.3.3 を参照)。
- ボックスのチャンネルスポット信号で、スポットを検出する必要があるチャンネルを設定します。従来の RGB イメージでは、0 =赤、1 =緑、および 2=青。
- [局所的な分子の名前] ボックスに、スポットに局所化する分子の名前を書きます。入力するフィールドの数は、分子の数によって異なります。
- ボックスで、ウェーブレットスポット検出器ブロックで、各チャンネルのスポット検出パラメータを設定します。スケール(スポットサイズと呼ばれる)と検出の感度を設定します(感度が小さいほど検出されたスポットの数が減少し、デフォルト値は100、最小値は0)。このプラグインの包括的なドキュメントはオンラインで入手できます: http://icy.bioimageanalysis.org/plugin/Spot_Detector.パラメータは、ICY (検出とトラッキング |スポット検出器)。
- オプションとして、[サイズで ROI をフィルター]ボックスで、サイズの間隔を設定して、スポットが検出されるセグメント化されたオブジェクトをフィルター処理します (ピクセル単位)。この手順は、セグメントが少ないオブジェクトやセグメント分割されていないオブジェクトを削除する場合に特に便利です。オブジェクトのサイズを手動で見積もる方法については、ステップ 5.3.1 を参照してください。デフォルトのパラメータには、ROI のサイズによるフィルタリングは含まれません。2つのコンパートメントのブロック2チャンネルには、サイズ別フィルター核(ボックス19)とフィルター細胞質(ボックス46)の2つのボックスが含まれています。
- 必要に応じて、[サイズでフィルター]ボックスで、検出されたスポットをサイズ (ピクセル単位) に従ってフィルター処理し、不要なアーティファクトを削除します。スポットサイズを手動で推定するには、[検出と追跡]をクリックしてスポット検出プラグインを開きます。[出力オプション] で、[ROI にエクスポート]を選択します。既定のパラメータには、サイズによるスポットのフィルタリングが含まれていないこと、およびフィルタリングされたスポットが解析に考慮されないことに注意してください。入力するフィールドの数は、チャネルの数によって異なります。
- 必要に応じて、[フィルター スポット] ボックスで、検出されたスポットに追加のフィルター (コントラスト、均質性、境界、丸み) を適用します。デフォルトのパラメータにはスポット フィルタリングが含まれず、フィルタリングされたスポットは解析に考慮されないことに注意してください。入力するフィールドの数は、チャネルの数によって異なります。
- 必要に応じて、[スポット サイズのしきい値] ボックスで、分析されたスポットの領域 (ピクセル単位) のしきい値を設定します。このしきい値を下回る数としきい値を超えるカウントされたスポットの数は、最終結果スプレッドシートにエクスポートされます。通知されるボックスの数は、チャネルの数によって異なります。
- ワークフローを実行します (詳細については、パート 7 を参照してください)。データは、マージされたイメージを含むフォルダに保存された結果スプレッドシートにエクスポートされます。
- 蛍光分析D:グローバル転位・蛍光分析E:個々の細胞転位:
- [フォルダ イメージ](ボックス 1 および 2)に、セグメント化されたオブジェクトのイメージを含むフォルダの \Name を入力します。「蛍光分析 D: グローバル転位」では、ROI の 2 つのタイプが ROI a および ROI b と識別されます。ブロック蛍光分析 E: 個々のセルの転位のフォルダー 画像セグメント化された核とフォルダー 画像セグメント化されたサイトプラズムボックスで、それぞれセグメント化された核とサイトプラズムを含むフォルダーの名前を書きます。
- ボックスチャンネル信号(ボックス3)に、信号のチャンネルを入力します。
- セグメント化されたオブジェクトのイメージのフォーマット(ボックス 4) に、セグメント化されたオブジェクトのイメージを保存するために使用する形式 (tif、jpeg、bmp、png) を書き込みます。デフォルトのフォーマットは tif です。[境界を強制終了]オプションは、境界オブジェクトを削除するためにも使用できます(ステップ6.3.1を参照)。
- 必要に応じて、[サイズで ROI をフィルター]ボックスで、サイズの間隔を設定してセグメント化されたオブジェクトをフィルター処理します (ピクセル単位)。この手順は、セグメントが少ないオブジェクトやセグメント分割されていないオブジェクトを削除する場合に役立ちます。オブジェクトサイズを手動で見積もる方法については、ステップ 5.3.1 を参照してください。入力するフィールドは 2 つあり、チャネルごとに 1 つ。デフォルトのパラメータには、サイズによる ROI フィルタリングは含まれません。
- ワークフローを実行します (詳細については、パート 7 を参照してください)。スプレッドシート内のデータをエクスポート結果は、マージされた画像を含むフォルダーに保存されます。
- 蛍光分析A:1チャネル、蛍光分析B:同じコンパートメント内の2チャンネルと蛍光分析C:2つのコンパートメント内の2チャンネル
7. プロトコルを実行する
- 実行で 1 つのブロックを処理するには、選択したブロックとブロックの間の接続を削除します。1stランクに指名手配ブロックをst配置します。目的のブロックの左上隅にあるフォルダの右にあるリンクをクリックします。表示される [開く] ダイアログ ボックスで、結合したイメージを含むフォルダをダブルクリックします。次に、[開く] をクリックします。[実行] をクリックしてワークフローを開始します。処理は、停止ボタンをクリックして停止することができます。
- 実行で異なるブロックを処理するには、選択したブロックの接続をブロックの[フォルダを選択](ブロック1)に保持します。ランクがワークフローの良好な処理を許可していることを確認します。たとえば、特定のブロックでセグメント化されたオブジェクトを処理する必要がある場合は、セグメント化ブロックが処理される前に必ず行ってください。ワークフローを実行する前に、使用されていないブロックを削除し、新しいプロトコルを別の名前で保存します。
- [実行] をクリックしてワークフローを開始します。[開く] ダイアログ ボックスが表示されたら、結合したイメージを含むフォルダをダブルクリックします。次に、[開く] をクリックします。ワークフローが自動的に実行されます。必要に応じて、停止ボタンをクリックして処理を停止します。
- 処理の最後に、ワークフロー実行のメッセージが右下隅に表示され、すべてのブロックに緑色のフラグが付いているかどうかを確認します(図2b)。見えない場合は、エラー記号を示すブロックと内側のボックスが、修正する要素を示します (図 2b)。
注: ワークフローが正常に実行された後、新しい実行を直接開始することはできず、ワークフローを再度処理するには、少なくとも 1 つのブロックに「処理準備完了」というフラグが付く必要があります。ブロックの状態を変更するには、このブロック内の 2 つのボックス間のリンクを削除して再作成するか、プロトコルを閉じて再度開きます。処理中にエラーが発生した場合は、新しい実行を直接開始できます。新しい実行中に、パイプラインの一部に緑色の記号が付いている場合でも、パイプラインのすべてのブロックが処理されます。
結果
説明されたすべての分析は、Javaの64ビットバージョンで動作する標準ラップトップ(2.80 GHzの64ビットクアッドコアプロセッサと16 GBのランダムアクセスメモリ(RAM))で実行されています。ランダム アクセス メモリは、分析する画像の量と解像度に応じて考慮すべき重要なパラメーターです。32 ビットバージョンの Java を使用すると、メモリは約 1300 MB に制限されますが、これはビッグデータ分?...
ディスカッション
蛍光細胞画像の解析には、ますます多くのフリーソフトウェアツールが利用できます。ユーザーは、問題の複雑さ、画像処理の知識、および分析に費やす時間に応じて適切なソフトウェアを正しく選択する必要があります。アイシー、セルプロファイル、または ImageJ/フィジーは、ユーザビリティと機能性の両方を組み合わせた強力なツールです 3.Icy は、明確なグラフィカ?...
開示事項
著者らは開示するものは何もない。
謝辞
G.H.は、ミニステール・デレジェ・ア・ラ・レシェルシュ・エ・オ・テクノロジーズの大学院フェローシップによって支えられていました。L.H.はロレーヌ研究所(ICL)の大学院フェローシップによって支えられましたが、Q.T.は第2回「投資ダベニール」プログラムFIGHT-HF(参照:ANR-15-RHU457000)の一環としてフランス国家研究庁(ANR)が監督する公的助成金によって支援されました。この作品は、CNRSとロレーヌ大学(UMR 7365)によって資金提供されました。
資料
| Name | Company | Catalog Number | Comments |
| 16% Formaldehyde solution (w/v) methanol free | Thermo Fisher Scientific | 28908 | to fix the cells |
| Alexa Fluor 488 of goat anti-rabbit | Thermo Fisher Scientific | A-11008 | fluorescent secondary antibody |
| Alexa Fluor 555 of goat anti-mouse | Thermo Fisher Scientific | A-21425 | fluorescent secondary antibody |
| Alexa Fluor 555 Phalloidin | Thermo Fisher Scientific | A34055 | fluorescent secondary antibody |
| Bovine serum albumin standard (BSA) | euromedex | 04-100-812-E | |
| DMEM | Sigma-Aldrich | D5796-500ml | cell culture medium |
| Duolink In Situ Mounting Medium with DAPI | Sigma-Aldrich | DUO82040-5ML | mounting medium |
| Human: HeLa S3 cells | IGBMC, Strasbourg, France | cell line used to perform the experiments | |
| Hydrogen peroxide solution 30% (H2O2) | Sigma-Aldrich | H1009-100ml | used as a stressing agent |
| Lipofectamine 2000 Reagent | Thermo Fisher Scientific | 11668-019 | transfection reagent |
| Mouse monoclonal anti-coilin | abcam | ab11822 | Coilin-specific antibody |
| Nikon Optiphot-2 fluorescence microscope | Nikon | epifluoresecence microscope | |
| Opti-MEM I Reduced Serum Medium | Thermo Fisher Scientific | 31985062 | transfection medium |
| PBS pH 7.4 (10x) | gibco | 70011-036 | to wash the cells |
| Rabbit polyclonal anti-53BP1 | Thermo Fisher Scientific | PA1-16565 | 53BP1-specific antibody |
| Rabbit polyclonal anti-EDC4 | Sigma-Aldrich | SAB4200114 | EDC4-specific antibody |
| Triton X-100 | Roth | 6683 | to permeabilize the cells |
参考文献
- Möckl, L., Lamb, D. C., Bräuchle, C. Super-resolved fluorescence microscopy: Nobel Prize in Chemistry 2014 for Eric Betzig, Stefan Hell, and William E. Moerner. Angewandte Chemie. 53 (51), 13972-13977 (2014).
- Meijering, E., Carpenter, A. E., Peng, H., Hamprecht, F. A., Olivo-Marin, J. -. C. Imagining the future of bioimage analysis. Nature Biotechnology. 34 (12), 1250-1255 (2016).
- Wiesmann, V., Franz, D., Held, C., Münzenmayer, C., Palmisano, R., Wittenberg, T. Review of free software tools for image analysis of fluorescence cell micrographs. Journal of Microscopy. 257 (1), 39-53 (2015).
- de Chaumont, F., et al. Icy: an open bioimage informatics platform for extended reproducible research. Nature Methods. 9 (7), 690-696 (2012).
- Girish, V., Vijayalakshmi, A. Affordable image analysis using NIH Image/ImageJ. Indian J Cancer. 41 (1), 47 (2004).
- Zaitoun, N. M., Aqel, M. J. Survey on image segmentation techniques. Procedia Computer Science. 65, 797-806 (2015).
- . MetaMorph Microscopy Automation and Image Analysis Software Available from: https://www.moleculardevices.com/products/cellular-imaging-systems/acquisition-and-analysis-software/metamorph-microscopy (2018)
- . NIS-Elements Imaging Software Available from: https://www.nikon.com/products/microscope-solutions/lineup/img_soft/nis-element (2014)
- Meyer, F., Beucher, S. Morphological segmentation. Journal of Visual Communication and Image Representation. 1 (1), 21-46 (1990).
- Schieber, M., Chandel, N. S. ROS Function in Redox Signaling and Oxidative Stress. Current Biology. 24 (10), 453-462 (2014).
- D'Autréaux, B., Toledano, M. B. ROS as signalling molecules: mechanisms that generate specificity in ROS homeostasis. Nature Reviews. Molecular Cell Biology. 8 (10), 813-824 (2007).
- Davalli, P., Mitic, T., Caporali, A., Lauriola, A., D'Arca, D. ROS, Cell Senescence, and Novel Molecular Mechanisms in Aging and Age-Related Diseases. Oxidative Medicine and Cellular Longevity. 2016, 3565127 (2016).
- Disher, K., Skandalis, A. Evidence of the modulation of mRNA splicing fidelity in humans by oxidative stress and p53. Genome. 50 (10), 946-953 (2007).
- Takeo, K., et al. Oxidative stress-induced alternative splicing of transformer 2β (SFRS10) and CD44 pre-mRNAs in gastric epithelial cells. American Journal of Physiology - Cell Physiology. 297 (2), 330-338 (2009).
- Seo, J., et al. Oxidative Stress Triggers Body-Wide Skipping of Multiple Exons of the Spinal Muscular Atrophy Gene. PLOS ONE. 11 (4), 0154390 (2016).
- Will, C. L., Luhrmann, R. Spliceosome Structure and Function. Cold Spring Harbor Perspectives in Biology. 3 (7), 003707 (2011).
- Ljosa, V., Sokolnicki, K. L., Carpenter, A. E. Annotated high-throughput microscopy image sets for validation. Nature Methods. 9 (7), 637-637 (2012).
- Wang, Q., et al. Cajal bodies are linked to genome conformation. Nature Communications. 7, (2016).
- Carpenter, A. E., et al. CellProfiler: image analysis software for identifying and quantifying cell phenotypes. Genome Biology. 7, 100 (2006).
- McQuin, C., et al. CellProfiler 3.0: Next-generation image processing for biology. PLoS Biology. 16 (7), 2005970 (2018).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved