È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Methods Article
Substructure Analyzer: un flusso di lavoro facile da usare per l'esplorazione rapida e l'analisi accurata dei corpi cellulari nelle immagini di microscopia a fluorescenza
In questo articolo
Riepilogo
Vi presentiamo un flusso di lavoro liberamente disponibile costruito per una rapida esplorazione e analisi accurata dei corpi cellulari in specifici compartimenti cellulari in immagini di microscopia a fluorescenza. Questo flusso di lavoro user-friendly è progettato sul software open-source E utilizza anche funzionalità ImageJ. La pipeline è conveniente senza conoscere l'analisi delle immagini.
Abstract
L'ultimo decennio è stato caratterizzato da scoperte nelle tecniche di microscopia a fluorescenza illustrate dal miglioramento della risoluzione spaziale, ma anche nell'imaging a cellule vive e nelle tecniche di microscopia ad alto consumo. Ciò ha portato ad un costante aumento della quantità e della complessità dei dati di microscopia per un singolo esperimento. Poiché l'analisi manuale dei dati di microscopia richiede molto tempo, soggettiva e proibisce l'analisi quantitativa, l'automazione dell'analisi delle immagini biologiche sta diventando quasi inevitabile. Abbiamo costruito un flusso di lavoro informatico chiamato Substructure Analyzer per automatizzare completamente l'analisi del segnale in bioimmagini dalla microscopia fluorescente. Questo flusso di lavoro è sviluppato sulla piattaforma open source user-friendly Icy ed è completato da funzionalità da ImageJ. Esso comprende la pre-elaborazione delle immagini per migliorare il rapporto segnale-rumore, la segmentazione individuale delle cellule (rilevamento dei confini cellulari) e la rilevazione/quantificazione dei corpi cellulari arricchiti in specifici compartimenti cellulari. Il vantaggio principale di questo flusso di lavoro è quello di proporre complesse funzionalità di bio-imaging agli utenti senza esperienza di analisi delle immagini attraverso un'interfaccia user-friendly. Inoltre, è altamente modulare e adattato a diverse questioni, dalla caratterizzazione della traslocazione nucleare/citoplasmatica all'analisi comparativa di diversi corpi cellulari in diverse sottostrutture cellulari. La funzionalità di questo flusso di lavoro è illustrata attraverso lo studio dei corpi Cajal (coiled) in condizioni di sollecitazione ossidativa (OS). I dati della microscopia a fluorescenza mostrano che la loro integrità nelle cellule umane è influenzata poche ore dopo l'induzione del sistema operativo. Questo effetto è caratterizzato da una diminuzione della nucleazione della bobina nei corpi cajal caratteristici, associata a una ridistribuzione nucleoplasmica della bobina in un numero maggiore di focolai più piccoli. Il ruolo centrale della bobina nello scambio tra i componenti CB e il nucleoplasmo circostante suggerisce che la ridistribuzione della bobina indotta dall'OS potrebbe influenzare la composizione e la funzionalità dei corpi cajal.
Introduzione
La microscopia leggera e, più in particolare, la microscopia a fluorescenza sono tecniche robuste e versatili comunemente utilizzate nelle scienze biologiche. Danno accesso alla localizzazione precisa di varie biomolecole come proteine o RNA attraverso la loro specifica etichettatura fluorescente. L'ultimo decennio è stato caratterizzato da rapidi progressi nelle tecnologie di microscopia e imaging, come dimostra il Premio Nobel 2014 per la chimica che ha assegnato Eric Betzig, Stefan W. Hell e William E. Moerner per lo sviluppo della microscopia a fluorescenza super risolta (SRFM)1. SFRM bypassa il limite di diffrazione della microscopia ottica tradizionale per portarla nella nanodimensione. Il miglioramento di tecniche come l'imaging live o gli approcci di screening ad alta velocità effettiva aumenta anche la quantità e la complessità dei dati da trattare per ogni esperimento. La maggior parte del tempo, i ricercatori si trovano di fronte ad alte popolazioni eterogenee di cellule e vogliono analizzare i fenotipi a livello di singola cellula.
Inizialmente, analisi come il conteggio dei foci sono state eseguite a occhio, che è preferito da alcuni ricercatori in quanto fornisce il pieno controllo visivo sul processo di conteggio. Tuttavia, l'analisi manuale di tali dati richiede troppo tempo, porta a variabilità tra gli osservatori e non consente l'accesso a funzionalità più complesse in modo che gli approcci assistiti da computer stiano diventando ampiamente utilizzati e quasi inevitabili2. I metodi di bioimmagine informatica aumentano notevolmente l'efficienza dell'analisi dei dati e sono liberi dall'inevitabile soggettività dell'operatore e dalla potenziale distorsione dell'analisi di conteggio manuale. L'aumento della domanda in questo campo e il miglioramento della potenza informatica hanno portato allo sviluppo di un gran numero di piattaforme di analisi delle immagini. Alcuni di loro sono liberamente disponibili e danno accesso a vari strumenti per eseguire analisi con personal computer. Una classificazione degli strumenti ad accesso aperto è stata recentemente stabilita3 e presenta Icy4 come un potente software che combina usabilità e funzionalità. Inoltre, Icy ha il vantaggio di comunicare con ImageJ.
Per gli utenti senza esperienza nell'analisi delle immagini, gli ostacoli principali sono scegliere lo strumento appropriato in base ai parametri problematici e sintonizzati correttamente che spesso non sono ben compresi. Inoltre, i tempi di installazione sono spesso lunghi. Icy propone un'interfaccia user-and-click denominata "Protocols" per sviluppare il flusso di lavoro combinando alcuni plugin che si trovano all'interno di una raccolta esaustiva4. Il design modulare flessibile e l'interfaccia point-and-click rendono possibile l'impostazione di un'analisi per i non programmatori. Qui presentiamo un flusso di lavoro chiamato Substructure Analyzer, sviluppato nell'interfaccia di Icy, la cui funzione è quella di analizzare i segnali fluorescenti in specifici compartimenti cellulari e misurare diverse caratteristiche come luminosità, numero foci, dimensione del foci e distribuzione spaziale. Questo flusso di lavoro affronta diversi problemi come la quantificazione della traslocazione del segnale, l'analisi delle cellule trasfette che esprimono un reporter fluorescente o l'analisi dei foci da diverse sottostrutture cellulari nelle singole cellule. Consente l'elaborazione simultanea di più immagini e i risultati di output vengono esportati in un foglio di lavoro delimitato da tabulazioni che può essere aperto in programmi di fogli di calcolo di uso comune.
La pipeline dell'analizzatore di sottostruttura è illustrata nella Figura 1. In primo luogo, tutte le immagini contenute in una cartella specificata vengono pre-elaborate per migliorare il loro rapporto segnale-rumore. Questo passaggio aumenta l'efficienza dei passaggi seguenti e riduce il tempo di esecuzione. Quindi, le regioni di interesse (ROI), corrispondenti alle aree dell'immagine in cui il segnale fluorescente deve essere rilevato, vengono identificate e segmentate. Infine, il segnale fluorescente viene analizzato e i risultati vengono esportati in un foglio di lavoro delimitato da tabulazioni.
La segmentazione degli oggetti (rilevamento dei confini) è il passo più impegnativo nell'analisi delle immagini e la sua efficienza determina l'accuratezza delle misurazioni delle celle risultanti. I primi oggetti identificati in un'immagine (chiamati oggetti primari) sono spesso nuclei da immagini macchiate di DNA (colorazione DAPI o Hoechst), anche se gli oggetti primari possono anche essere cellule intere, perline, macchie, tumori o qualsiasi altro oggetto macchiato. Nella maggior parte delle immagini biologiche, cellule o nuclei si toccano o si sovrappongono causando il fallimento degli algoritmi semplici e veloci. Ad oggi, nessun algoritmo universale può eseguire una segmentazione perfetta di tutti gli oggetti, soprattutto perché le loro caratteristiche (dimensioni, forma o trama) modulano l'efficienza della segmentazione5. Gli strumenti di segmentazione comunemente distribuiti con il software di microscopia (come il MetaMorph Imaging Software di Molecular Devices6o il software NIS-Elements Advances Research di Nikon7) sono generalmente basati su tecniche standard come la corrispondenza di correlazione, la soglia o le operazioni morfologiche. Sebbene efficienti nei sistemi di base, questi metodi sovrageneralizzati presentano rapidamente limitazioni se utilizzati in contesti più impegnativi e specifici. In effetti, la segmentazione è altamente sensibile ai parametri sperimentali come il tipo di cellula, la densità delle cellule o i biomarcatori e spesso richiede una regolazione ripetuta per un set di dati di grandi dimensioni. Il flusso di lavoro Substructure Analyzer integra algoritmi semplici e più sofisticati per proporre diverse alternative adattate alla complessità dell'immagine e alle esigenze degli utenti. Propone in particolare l'algoritmo del bacino idrografico basato su marcatori8 per gli oggetti altamente raggruppati. L'efficienza di questo metodo di segmentazione si basa sulla selezione di singoli marcatori su ogni oggetto. Questi marcatori vengono scelti manualmente la maggior parte del tempo per ottenere i parametri corretti per la segmentazione completa, che richiede molto tempo quando gli utenti devono affrontare un numero elevato di oggetti. Substructure Analyzer propone un rilevamento automatico di questi marcatori, fornendo un processo di segmentazione altamente efficiente. La segmentazione è, nella maggior parte dei tempi, la fase limitante dell'analisi dell'immagine e può modificare notevolmente il tempo di elaborazione a seconda della risoluzione dell'immagine, del numero di oggetti per immagine e del livello di clustering degli oggetti. Le pipeline tipiche richiedono da pochi secondi a 5 minuti per immagine in un computer desktop standard. L'analisi di immagini più complesse può richiedere un computer più potente e alcune conoscenze di base nell'analisi delle immagini.
La flessibilità e la funzionalità di questo flusso di lavoro sono illustrate con vari esempi nei risultati rappresentativi. I vantaggi di questo flusso di lavoro sono mostrati in particolare attraverso lo studio di sottostrutture nucleari in condizioni di stress ossidativo (OS). OS corrisponde a uno squilibrio dell'omeostasi redox a favore di ossidanti ed è associato ad alti livelli di specie reattive dell'ossigeno (ROS). Poiché il ROS agisce come molecole di segnalazione, i cambiamenti nella loro concentrazione e localizzazione subcellulare influenzano positivamente o negativamente una miriade di percorsi e reti che regolano le funzioni fisiologiche, tra cui la trasduzione del segnale, i meccanismi di riparazione, l'espressione genica, la morte cellulare e la proliferazione9,10. OS è quindi direttamente coinvolto in varie patologie (malattie neurodegenerative e cardiovascolari, tumori, diabete, ecc),), ma anche l'invecchiamento cellulare. Pertanto, decifrare le conseguenze del sistema operativo sull'organizzazione e la funzione della cellula umana costituisce un passo cruciale nella comprensione dei ruoli del sistema operativo nell'insorgenza e nello sviluppo delle patologie umane. È stato stabilito che l'OS regola l'espressione genica modulando la trascrizione attraverso diversi fattori di trascrizione (p53, Nrf2, FOXO3A)11, ma anche influenzando la regolazione di diversi processi co- e post-trascrizione come lo splicing alternativo (AS) dei pre-RNA12,13,14. Lo splicing alternativo delle trascrizioni primarie e non codificanti è un meccanismo essenziale che aumenta la capacità di codifica dei genomi producendo isoforme di trascrizione. AS è eseguita da un enorme complesso di ribonucleoproteina chiamato spliceosome, contenente quasi 300 proteine e 5 RNA nucleari piccoli ricchi di U (UsnRNA)15. L'assemblaggio spliceo e l'AS sono strettamente controllati nelle cellule e alcuni passi della maturazione sillimitata si verificano all'interno di compartimenti nucleari senza membrana chiamati Cajal Bodies. Queste sottostrutture nucleari sono caratterizzate dalla natura dinamica della loro struttura e dalla loro composizione, che sono principalmente condotte da interazioni multivalenti del loro RNA e componenti proteici con la proteina della coilina. L'analisi di migliaia di celle con il flusso di lavoro Substructure Analyzer ha consentito la caratterizzazione degli effetti mai descritti del sistema operativo sui corpi di Cajal. Infatti, i dati ottenuti suggeriscono che l'OS modifica la nucleazione dei corpi di Cajal, inducendo una ridistribuzione nucleoplasmica della proteina coilina in numerosi focolai nucleari più piccoli. Tale cambiamento della struttura dei corpi cajal potrebbe influenzare la maturazione dello spliceo some e partecipare alla modulazione AS da OS.
Protocollo
NOTA: tutorial user-friendly sono disponibili sul sito web di Icy http://icy.bioimageanalysis.org.
1. Scaricare Icy e il protocollo Substructure Analyzer
- Scaricare Icy dal sito Web Icy (http://icy.bioimageanalysis.org/download) e scaricare il protocollo Substructure Analyzer: http://icy.bioimageanalysis.org/protocols?sort=latest.
NOTA: se si utilizza un sistema operativo a 64 bit, assicurarsi di utilizzare la versione a 64 bit di Java. Questa versione consente di aumentare la memoria allocata a Icy (Preferenze Proprietà General . Memoria massima).
2. Apertura del protocollo
- Aprire Icy e fare clic su Strumenti nel menu della barra multifunzione.
- Fare clic su Protocolli per aprire l'interfaccia dell'Editor dei protocolli.
- Fare clic su Carica e aprire il protocollo Substructure Analyzer. Il caricamento del protocollo può richiedere alcuni secondi. Assicurarsi che l'apertura del protocollo sia stata completata prima di utilizzarlo.
NOTA: il flusso di lavoro è composto da 13 blocchi generali presentati nella figura 2a. Ogni blocco funziona come una pipeline composta da diverse caselle che eseguono sottoattività specifiche.
3. Interagire con il flusso di lavoro su Icy
NOTA: ogni blocco o casella è numerato e ha un rango specifico all'interno del flusso di lavoro(Figura 2b). Cliccando su questo numero, la posizione più vicina possibile alla prima viene assegnata al blocco/scatola selezionata, quindi la posizione degli altri blocchi/caselle viene riorganizzata. Rispettare l'ordine corretto dei blocchi durante la preparazione del flusso di lavoro. Ad esempio, il blocco Rilevatore spot deve utilizzare ROI predefiniti in modo che i blocchi di segmentazione vengano eseguiti prima dei blocchi di Rilevamento macchie. Non modificare la posizione delle caselle. Non utilizzare "." nel nome dell'immagine.
- Facendo clic sull'icona dell'angolo superiore sinistro, comprimere, espandere, ingrandire, restringere o rimuovere il blocco (Figura 2b).
- Ogni pipeline del flusso di lavoro è caratterizzata da una rete di caselle connesse tramite input e output (Figura 2b). Per creare una connessione, fare clic su Output e mantenere fino a quando il cursore non raggiunge un input. Le connessioni possono essere rimosse facendo clic sul tag Output.
4. Unione dei canali di un'immagine
- Utilizzare il blocco Unisci canali per generare immagini unite. Se necessario, rinominare i file in modo che le sequenze da unire abbiano lo stesso nome prefisso seguito da un separatore distinto. Ad esempio, le sequenze di singoli canali da un'immagine A sono denominate: ImageA_red, ImageA_blue.
NOTA: per il separatore, non utilizzare caratteri già presenti nel nome dell'immagine. - Nella stessa cartella, creare una nuova cartella per canale da unire. Ad esempio, per unire i canali rosso, verde e blu, creare 3 cartelle e memorizzare le sequenze corrispondenti in queste cartelle.
- Utilizzare solo il blocco Unisci canali, rimuovere gli altri blocchi e salvare il protocollo come Unisci canali.
- Accedere alle caselle per impostare i parametri. Per ogni canale, riempire le caselle Numero canale X (caselle 1, 5 o 9), Numero canale cartella X (caselle 2, 6 o 10), Numero di canale separatore X (caselle 3, 7 o 11) e Canale mappa colori nb X (caselle 4, 8 e 12) rispettivamente.
NOTA: queste caselle sono raggruppate orizzontalmente per quattro, ciascuna delle quali corrisponde allo stesso canale. In ogni riga è disponibile anche un display (caselle 23, 24 o 25) per visualizzare direttamente la sequenza del canale corrispondente.- Nella casella Numero di canale X, scegliere il canale da estrarre (nelle immagini RGB classiche, 0 -Rosso, 1 -Verde, 2-Blu). L'utente accede rapidamente ai diversi canali di un'immagine all'interno della finestra Di impostazioni di Icy, nella scheda Sequenza. Scrivi il valore del canale più piccolo nella riga superiore e quello più alto nella riga inferiore.
- Nella casella Numero canale cartella X, scrivere il nome della cartella contenente le immagini del canale X.
- Nella casella Numero di canale separatore X, scrivere il separatore utilizzato per il nome dell'immagine (nell'esempio precedente: "_red", "_green" e "_blue").
- Nella casella Colormap channel nb X, indicare con un numero quale modello di mappa colori utilizzare per visualizzare il canale corrispondente in Icy. Le mappe colore disponibili sono visibili nella scheda Sequenza della finestra Impostazioni.
- Nella casella Formato delle immagini unite (casella 28), scrivere l'estensione per salvare le immagini unite: .tif, .gif, .jpg, .bmp o .png.
NOTA: per unire solo 2 canali, non riempire le quattro caselle corrispondenti al terzo canale.
- Nell'angolo in alto a sinistra del blocco Unisci canali, fare clic sul collegamento direttamente a destra di Cartella. Nella finestra di dialogo Apri visualizzata, fare doppio clic sulla cartella contenente le sequenze del primo canale che è stato definito nella casella Numero canale cartella 1 (casella 2). Quindi, fare clic su Apri.
- Eseguire il protocollo facendo clic sulla freccia nera nell'angolo in alto a sinistra del blocco Unisci canali (vedere la parte 7 per ulteriori dettagli). Le immagini unite vengono salvate in una cartella Unisci nella stessa directory delle cartelle dei singoli canali.
- Accedere alle caselle per impostare i parametri. Per ogni canale, riempire le caselle Numero canale X (caselle 1, 5 o 9), Numero canale cartella X (caselle 2, 6 o 10), Numero di canale separatore X (caselle 3, 7 o 11) e Canale mappa colori nb X (caselle 4, 8 e 12) rispettivamente.
5. Segmentazione delle regioni di interesse
NOTA: Substructure Analyzer integra algoritmi semplici e più sofisticati per proporre diverse alternative adattate alla complessità dell'immagine e alle esigenze degli utenti.
- Selezionare il blocco adattato.
- Se gli oggetti non si toccano o l'utente non deve differenziare singolarmente gli oggetti cluster, utilizzare il blocco Segmentazione A: Oggetti non raggruppati.
- Quando gli oggetti non si toccano, ma alcuni di essi sono vicini, utilizzare il blocco Segmentazione B: Oggetti raggruppati in modo inadeguato .
- Per gli oggetti con un livello di clustering elevato e una forma convessa, utilizzare il blocco Segmentazione C: Oggetti raggruppati con forme convesse.
- Se gli oggetti presentano un livello di clustering elevato e hanno forme irregolari, utilizzare il blocco Segmentazione D: Oggetti raggruppati con forme irregolari.
- Utilizzare il blocco Segmentazione E: citoplasma raggruppato per segmentare i citolasmi che toccano individualmente utilizzando i nuclei segmentati come marcatori. Questo blocco ha bisogno imperativamente di nuclei segmentati per la processo.
NOTA: blocchi adattati per il processo di segmentazione degli oggetti primari in modo indipendente in modo che diversi blocchi possano essere utilizzati nella stessa congestione per confrontarne l'efficienza per una particolare sottostruttura o per segmentare diversi tipi di sottostrutture. Se il livello di clustering è eterogeneo all'interno dello stesso set di immagini, elaborare gli oggetti piccoli e altamente raggruppati separatamente nei blocchi adattati.
- Collegare l'output0 (File) del blocco Seleziona cartella all'input della cartella del blocco di segmentazione scelto.
- Impostare i parametri del blocco di segmentazione scelto.
- Segmentazione A: oggetti non raggruppati e segmentazione C: oggetti raggruppati con forme convesse
- Nella casella Segnale canale (casella 1), impostare il canale degli oggetti sul segmento.
- Come opzione, nella casella Filtro gaussiano (casella 2), aumentare i valori di sigma X e Y se il segnale all'interno degli oggetti è eterogeneo. Il filtro gaussiano leviga le texture per ottenere regioni più uniformi e aumenta la velocità e l'efficienza della segmentazione dei nuclei. Più piccoli sono gli oggetti, minore è il valore sigma. Evitare valori sigma elevati. Impostare i valori predefiniti su 0.Set default values to 0.
- Nella casella HK-Means (casella 3), impostare il parametro Classi di intensità e le dimensioni minime e massime approssimative (in pixel) degli oggetti da rilevare.
NOTA: per le classi di intensità, un valore pari a 2 classifica i pixel in 2 classi: sfondo e primo piano. Viene quindi adattato quando il contrasto tra gli oggetti e lo sfondo è elevato. Se gli oggetti in primo piano hanno intensità diverse o se il contrasto con lo sfondo è basso, aumentare il numero di classi. L'impostazione predefinita è 2. Le dimensioni dell'oggetto possono essere valutate rapidamente disegnando un ROI manualmente intorno all'oggetto di interesse. Le dimensioni del ROI (Interno in pixel) vengono visualizzate direttamente sull'immagine quando la si posiziona con il cursore o sono accessibili nella finestra delle statistiche del ROI (aprirla dalla barra di ricerca). I parametri ottimali rilevano ogni oggetto in primo piano in un singolo ROI. Possono essere definiti manualmente in Icy(Rilevamento e Tracciamento) | HK-Means). - Nella casella Contorni attivi (casella 4), ottimizzare il rilevamento dei bordi degli oggetti. La documentazione esaustiva per questo plugin è disponibile online: http://icy.bioimageanalysis.org/plugin/Active_Contours. I parametri corretti possono anche essere definiti manualmente in Icy(Detection and Tracking | Contorni attivi).
- Durante il processo, viene creata automaticamente una cartella per salvare le immagini degli oggetti segmentati. Nella casella Testo (casella 6), denominare questa cartella (ad es. nuclei segmentati). Per impostare il formato per il salvataggio delle immagini di oggetti segmentati (Tiff, Gif, Jpeg, BMP, PNG), riempire il formato casella delle immagini degli oggetti segmentati. La cartella viene creata nella cartella contenente le immagini unite.
- Eseguire il flusso di lavoro (per i dettagli, vedere la parte 7).
- Segmentazione B: oggetti raggruppati in modo inadeguato
- Seguire la stessa procedura descritta nella 5.3.1 per impostare i parametri delle caselle Segnalecanale , HK-Means, Contorni attivi, Estensione per salvare gli oggetti segmentati e Text (le classificazioni delle caselle non sono gli stessi del passaggio 5.3.1).
- Nella casella Chiama il plug-in IJ (casella 4), impostare il parametro Rolling per controllare la sottrazione in background. Impostare questo parametro almeno sulla dimensione dell'oggetto più grande che non fa parte dello sfondo. Diminuendo questo valore aumenta la rimozione dello sfondo, ma può anche indurre la perdita del segnale in primo piano.
- Nella casella Equalizzazione istogramma adattivo (casella 6), migliora i contrasti tra gli oggetti in primo piano e lo sfondo. Aumentando la pendenza si ottiene più sequenze contrastate.
- Eseguire il flusso di lavoro (per i dettagli, vedere la parte 7).
- Segmentazione D: oggetti raggruppati con forme irregolari
NOTA: per ogni immagine si applicano tre diversi metodi di segmentazione: in primo luogo,Clustering HK-mezziin combinazione con ilMetodo Active Contoursviene applicato. Quindi, ilalgoritmo spartiacque classico(utilizzando la mappa della distanza euclidea) viene applicato agli oggetti precedentemente segmentati in modo non corretto. Infine, unAlgoritmo spartiacque basato su marcatoriviene utilizzato. Solo i metodi hK-means e marker-based spartiacque richiedono l'intervento dell'utente. Per entrambi i metodi, gli stessi parametri possono essere applicati per tutte le immagini (versione completamente automatizzata) o essere modificati per ogni immagine (versione semi-automatica). Se l'utente non viene addestrato in questi metodi di segmentazione, l'elaborazione semiautomatica è altamente consigliata. Durante l'elaborazione di questo blocco, è necessario un intervento manuale. Al termine di un metodo di segmentazione, l'utente deve rimuovere manualmente gli oggetti segmentati in modo non corretto prima dell'inizio del metodo di segmentazione successivo. Gli oggetti segmentati correttamente vengono salvati e non considerati nei passaggi successivi. Questo blocco deve essere collegato con il bloccoFinestra di dialogo Segmentazione oggetti primari di forme raggruppate/eterogeneeper funzionare correttamente.- Scaricare la raccolta ImageJ MorphoLibJ su https://github.com/ijpb/MorphoLibJ/releases. In questo protocollo viene utilizzata la versione MorphoLibJ 1.4.0. Inserire il file MorphoLibJ_-1.4.0.jar nella cartella ghiacciata/ij/plugins. Ulteriori informazioni sul contenuto di questa raccolta sono disponibili su https://imagej.net/MorphoLibJ.
- Seguire la stessa procedura descritta nel passaggio 5.3.1 per impostare i parametri delle caselle Segnalecanale , Filtro gaussiano, Contorni attivi, Estensione per salvare gli oggetti segmentati e Testo. I ranghi delle scatole non sono gli stessi del passaggio 5.3.1.
- Impostare i parametri della casella Equalizzazione istogramma adattiva (vedere il passaggio 5.3.2.3).
- Per attivare Sottrai sfondo, scrivere sì in Applica sottrazione sfondo? (casella 5). Altrimenti, scrivere No. Se il plugin è attivato, impostare il parametro rolling (vedere il passaggio 5.3.2), nella casella Sottrai parametro Sfondo (casella 7).
- Automatizzazione dei mezzi HK: Per applicare gli stessi parametri per tutte le immagini (elaborazione completamente automatizzata), impostare nb delle classi (casella 11), Dimensioni minime (casella 12) e Dimensioni massime (casella13) (vedere il passaggio 5.3.1). Questi parametri devono essere impostati per selezionare un massimo di pixel in primo piano e per ottimizzare l'individualizzazione degli oggetti in primo piano. Per la versione di elaborazione semi-automatica, non è necessario alcun intervento.
- Automatizzazione delle estrazioni dei marcatori: per la versione completamente automatizzata, espandere la casella Estrazione marcatori interni (casella 27) e impostare il valore del parametro "dinamico" nella riga 13 dello script. Per la versione di elaborazione semi-automatica, non è necessario alcun intervento.
NOTA: i marcatori vengono estratti applicando una trasformazione a minima minima estesa su un'immagine di input controllata da un parametro "dinamico". Nell'algoritmo dello spartiacque basato su marker, l'inondazione da questi marcatori viene simulata per eseguire la segmentazione degli oggetti. Per la segmentazione corretta degli oggetti in primo piano, deve essere estratto un singolo marcatore per ogni oggetto in primo piano. L'impostazione del parametro "dinamico" per l'estrazione ottimale dei marcatori dipende principalmente dalla risoluzione delle immagini. Pertanto, se non si ha familiarità con questo parametro, utilizzare la versione semiautomatica. - Eseguire il flusso di lavoro (per i dettagli, vedere la parte 7).
- All'inizio dell'elaborazione, le finestre di dialogo HK-means parametri e Marker-based spartiacque successivamente aperto. Per applicare gli stessi parametri per tutte le immagini (versione completamente automatizzata), fare clic su SI. In caso contrario, fare clic su NO. Si apre una finestra di informazioni in cui viene chiesto di "Determinare i ROM ottimali con il plug-in HK-Means e chiudere l'immagine". Fare clic su OK e applicare manualmente il plug-in HK-Means (Rilevamento e tracciamento. HK-Means) sull'immagine, che si apre automaticamente. Selezionare l'opzione Esporta ROI nella casella del plug-in HK-Means. Applicare i parametri migliori per avere ROI contenenti un massimo di pixel in primo piano e per ottimizzare l'individualizzazione degli oggetti in primo piano. Quando vengono trovate ROI ottimali, chiudere direttamente l'immagine.
- Alla fine del primo metodo di segmentazione, si apre una finestra di informazioni che chiede di "Rimuovere le ROI indesiderate e chiudere l'immagine". Questi ROI corrispondono ai bordi degli oggetti segmentati. Selezionare OK e rimuovere i ROI degli oggetti segmentati in modo non corretto nell'immagine, che si apre automaticamente. Un ROI può essere facilmente rimosso posizionando il cursore sul suo bordo e utilizzando il pulsante "Elimina" della tastiera. Chiudere l'immagine. Ripetere la stessa procedura dopo il completamento del secondo passaggio di segmentazione.
- In questa fase, se il pulsante YES è stato selezionato per l'automazione completa dell'algoritmo spartiacque basato su marcatori, i parametri impostati in precedenza verranno applicati a tutte le immagini.
- Se è stato selezionato il pulsante NO, si apre una finestra di informazioni in cui viene chiesto di "determinare e regolare i marcatori interni". Fare clic su OK e all'interno dell'interfaccia ImageJ di Icy, andare a Plugins | Proprietà MorphoLibJ . Minima e Maxima| Esteso Min & Max. In Operazione, selezionare Minima estesa.
- Selezionare Anteprima per previsualizzare nell'immagine aperta automaticamente il risultato della trasformazione. Spostare la dinamica finché non vengono osservati i marcatori ottimali. I marcatori sono gruppi di pixel con un valore di 255 (non necessariamente pixel bianchi). I parametri ottimali portano a un marcatore per oggetto. Concentrarsi sugli oggetti rimanenti che non sono stati segmentati bene con i due metodi di segmentazione precedenti.
- Se necessario, migliorare i marcatori applicando ulteriori operazioni morfologiche come "Apertura" o "Chiusura"(Plugins | Proprietà MorphoLibJ . Filtri morfologici). Quando si ottiene l'immagine finale dei marcatori, tenerla aperta e chiudere tutte le altre immagini che terminano con l'immagine inizialmente utilizzata come input per l'operazione Extended Minima. Fare clic su No se una casella ImageJ richiede di salvare le modifiche su questa immagine.
- Nella casella Nb delle immagini con Information Box (casella 14), determinare quante immagini con le caselle informazioni devono essere visualizzate.
- Segmentazione E: citoplasma a grappolo
NOTA: Questo blocco utilizza nuclei precedentemente segmentati come singoli marcatori per avviare la segmentazione del citoplasma. Assicurarsi che il blocco di segmentazione dei nuclei sia elaborato prima di utilizzarlo.- Nella scatola Ctoplasma canale (casella 1), impostare il canale del segnale citoplasmico.
- Nella casella Estensione nuclei segmentati (casella 2), scrivere il formato utilizzato per salvare le immagini dei nuclei segmentati (tif, jpeg, bmp, png). Il formato predefinito è tif.
- Nella casella Testo (casella 3), scrivere il nome della cartella contenente i nuclei segmentati.
- Nella casella Formato delle immagini di citoplasmi segmentati (casella 4), impostare il formato da utilizzare per il salvataggio di immagini di oggetti segmentati (Tiff, Gif, Jpeg, BMP, PNG).
- Durante il processo, viene creata automaticamente una cartella per salvare le immagini dei citoplami segmentati. Nella casella Testo (casella 5), denominare questa cartella (ad es. citoplasmi segmentati). La cartella viene creata nella cartella contenente le immagini unite.
- Seguire la stessa procedura descritta nel passaggio 5.3.1 per impostare i parametri delle caselle Filtro gaussiano e Contorni attivi (attenzione, i ranghi delle caselle non sono gli stessi del passaggio 5.3.1).
- Eseguire il flusso di lavoro (per i dettagli, vedere la parte 7).
- Segmentazione A: oggetti non raggruppati e segmentazione C: oggetti raggruppati con forme convesse
6. Rilevamento e analisi del segnale fluorescente
- Selezionare il blocco adattato.
- Nel blocco Fluorescence Analysis A: 1 Channel, eseguire il rilevamento e l'analisi dei foci in un canale all'interno di un tipo di oggetto segmentato: rilevamento di foci di bobina (canale rosso) all'interno del nucleo.
- Nel blocco Fluorescence Analysis B: 2 Channels in the same compartment, eseguire il rilevamento e l'analisi dei foci in due canali all'interno di un tipo di oggetto segmentato: rilevamento di bobina (canale rosso) e 53BP1 (canale verde) foci all'interno del nucleo.
- Nel blocco Fluorescence Analysis C: 2 Channels in two compartments, eseguire il rilevamento e l'analisi dei focolai in uno o due canali, in particolare all'interno dei nuclei e del corrispondente citoplasma: rilevamento di Coilin foci (canale rosso) sia all'interno del nucleo e del corrispondente citoplasma o rilevamento del foci Coilin (canale rosso) all'interno del nucleo e g3BP foci (canale verde) all'interno del citom corrispondente.
- Nel blocco Fluorescence Analysis D: Translocation globale, calcolare la percentuale di segnale da un canale in due compartimenti cellulari (a e b). Ad esempio, in un test di traslocazione del citoplasma/nucleo, esportare percentuali calcolate di segnali nucleari e citoplasmici per ogni immagine nel foglio di calcolo finale "Risultati". La formula utilizzata per calcolare la percentuale del segnale nucleare è mostrata di seguito. Questo blocco può essere utilizzato per qualsiasi compartimento subcellulare:
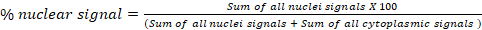
- Nel blocco Fluorescence Analysis E: Individual Cell Translocation, calcola la percentuale di segnale da un canale in due compartimenti cellulari per ogni cella. Questo blocco è appositamente ottimizzato per il saggio di traslocazione del nucleo/citoplasma a livello di singola cellula.
NOTA: poiché il blocco Fluorescence Analysis E: Single Cell Translocation esegue un'analisi a livello di singola cellula, è necessaria una segmentazione efficiente del nucleo e del citoplasma.
- Collegare l'output0 (File) del blocco Seleziona cartella (blocco 1) all'input della cartella (frecce bianche in cerchi neri) del blocco scelto.
- Impostare i parametri del blocco scelto.
- Analisi della fluorescenza A: 1 Canale, Fluorescenza Analisi B: 2 Canali nello stesso vano e Fluorescenza Analisi C: 2 Canali in due scomparti
- Nella casella Cartelle immagini ROI, scrivere il nome della cartella contenente le immagini di oggetti segmentati preceduti da una barra rovesciata. (Ad esempio: nuclei segmentati).
- Nella casella Formato delle immagini di oggetti segmentati (casella 2), scrivere il formato utilizzato per salvare le immagini degli oggetti segmentati (tif, jpeg, bmp, png). Il formato predefinito è tif.
- Nella casella Kill Borders?, scrivere Sì per rimuovere gli oggetti bordo. In caso contrario, scrivere No. L'installazione della raccolta MorphoLibJ di ImageJ è necessaria per utilizzare questa funzione (vedere il passaggio 5.3.3).
- Nella casella (es) Segnale macchie canale, impostare il canale in cui devono essere rilevati i punti. Nelle immagini RGB classiche, 0- Rosso, 1 - Verde e 2 - Blu.
- Nelle caselle Nome della molecola localizzata, scrivere il nome della molecola localizzando negli spot. Il numero di campi da inserire dipende dal numero di molecole.
- Nella casella (es) Wavelet Spot Detector Block, impostare i parametri di rilevamento spot per ogni canale. Impostare le scale (riferite alla dimensione del punto) e la sensibilità del rilevamento (una sensibilità minore riduce il numero di punti rilevati, il valore predefinito è 100 e il valore minimo è 0). La documentazione esaustiva di questo plugin è disponibile online: http://icy.bioimageanalysis.org/plugin/Spot_Detector. I parametri possono anche essere definiti manualmente in Icy(Detection and Tracking | Rilevatore spot).
- Come opzione, nella casella Filtra ROI per dimensione, filtra gli oggetti segmentati in cui vengono rilevate macchie impostando un intervallo di dimensioni (in pixel). Questo passaggio è particolarmente utile per rimuovere oggetti sotto o sovrasegmentati. Per stimare manualmente le dimensioni degli oggetti, vedere il passaggio 5.3.1. I parametri predefiniti non includono il filtro dei ROI in base alla dimensione. Il blocco 2 Canali in due scomparti contiene due scatole: Filtrare i nuclei per dimensione (scatola 19) e Filtrare il citoplasma per dimensione (casella 46).
- Facoltativamente, nella casella Filtra i punti per dimensione,filtra i punti rilevati in base alle dimensioni (in pixel) per rimuovere artefatti indesiderati. Per stimare manualmente le dimensioni spot, fare clic su Rilevamento e monitoraggio e aprire il plugin rilevatore Spot. In Opzioni di output, selezionare Esporta in ROI. Prestare attenzione che i parametri predefiniti non includano il filtraggio dei punti in base alle dimensioni e che i punti filtrati non vengano presi in considerazione per l'analisi. Il numero di campi da immettere dipende dal numero di canali.
- Facoltativamente, nella casella Punti filtro, applicare un filtro aggiuntivo (contrasto, omogeneità, perimetro, rotondità) sui punti rilevati. Prestare attenzione che i parametri predefiniti non includano il filtraggio dei punti e che i punti filtrati non vengano presi in considerazione per l'analisi. Il numero di campi da immettere dipende dal numero di canali.
- Facoltativamente, nelle caselle Dimensione tinta (Spot size threshold),impostare una soglia per l'area (in pixel) dei punti analizzati. Il numero di punti conteggiati al di sotto e al di sopra di questa soglia viene esportato nel foglio di calcolo Risultati finale. Il numero di caselle da informare dipende dal numero di canali.
- Eseguire il flusso di lavoro (per i dettagli, vedere la parte 7). I dati vengono esportati in un foglio di calcolo Risultati salvato nella cartella contenente le immagini unite.
- Analisi della fluorescenza D: Analisi di traslocazione globale e fluorescenza E: Traslocazione cellulare individuale:
- Nelle caselle Immagini cartella (caselle 1 e 2), scrivere il nome della cartella contenente le immagini degli oggetti segmentati. Nel blocco Fluorescence Analysis D: Global Translocation, i due tipi di ROI sono identificati come ROI a e ROI b. Per il blocco Fluorescence Analysis E: Individual Cell Translocation, in Folder images segmented nuclei and Folder images segmented cytoplas box, write the name of the folder containing segmented nuclei and cytoplais, respectively.
- Nella casella Segnale canale (casella 3), immettere il canale del segnale.
- Nella casella Formato delle immagini di oggetti segmentati (casella 4), scrivere il formato utilizzato per salvare le immagini degli oggetti segmentati (tif, jpeg, bmp, png). Il formato predefinito è tif. L'opzione Uccidi bordi è disponibile anche per rimuovere gli oggetti bordo (vedere il passaggio 6.3.1).
- Facoltativamente, nelle caselle Filtra ROI per dimensione, filtra gli oggetti segmentati impostando un intervallo di dimensioni (in pixel). Questo passaggio può essere utile per rimuovere oggetti sotto o sovrasegmentati. Per stimare manualmente le dimensioni dell'oggetto, vedere il passaggio 5.3.1. Ci sono due campi da inserire, uno per canale. I parametri predefiniti non includono il filtro ROI in base alla dimensione.
- Eseguire il flusso di lavoro (per i dettagli, vedere la parte 7). Esportare i dati in un foglio di calcolo Risultati salvati nella cartella contenente le immagini unite.
- Analisi della fluorescenza A: 1 Canale, Fluorescenza Analisi B: 2 Canali nello stesso vano e Fluorescenza Analisi C: 2 Canali in due scomparti
7. Eseguire il protocollo
- Per elaborare un blocco in un'esecuzione, rimuovere la connessione tra il blocco selezionato e il blocco Seleziona cartella. Posizionare il blocco desiderato al1st rank. Nell'angolo in alto a sinistra del blocco desiderato, fare clic sul collegamento direttamente a destra della cartella. Nella finestra di dialogo Apri visualizzata, fare doppio clic sulla cartella contenente le immagini unite. Quindi, fare clic su Apri. Fare clic su Esegui per avviare il flusso di lavoro. L'elaborazione può essere interrotta facendo clic sul pulsante Stop.
- Per elaborare blocchi diversi in un'esecuzione, mantenere le connessioni dei blocchi scelti con il blocco Seleziona cartella (blocco 1). Assicurarsi che il loro rango consenta una buona elaborazione del flusso di lavoro. Ad esempio, se un blocco specifico necessita di oggetti segmentati da elaborare, assicurarsi che il blocco di segmentazione elabori prima. Prima di eseguire il flusso di lavoro, rimuovere i blocchi inutilizzati e salvare il nuovo protocollo con un altro nome.
- Fare clic su Esegui per avviare il flusso di lavoro. Quando viene visualizzata la finestra di dialogo Apri, fare doppio clic sulla cartella contenente le immagini unite. Quindi, fare clic su Apri. Il flusso di lavoro viene eseguito automaticamente. Se necessario, interrompere l'elaborazione facendo clic sul pulsante Interrompi.
- Al termine dell'elaborazione, verificare che il messaggio Il flusso di lavoro eseguito correttamente sia visualizzato correttamente nell'angolo inferiore destro e che tutti i blocchi siano contrassegnati con un segno verde ( Figura2b). In caso contrario, il blocco e la casella interna che presentano il simbolo di errore indicano l'elemento da correggere (Figura 2b).
NOTA: dopo che il flusso di lavoro è stato eseguito correttamente, non è possibile avviata direttamente una nuova esecuzione e per elaborare nuovamente il flusso di lavoro, almeno un blocco deve essere contrassegnato con il segno "pronto per l'elaborazione". Per modificare lo stato di un blocco, eliminare e ricreare un collegamento tra due caselle all'interno di questo blocco oppure semplicemente chiudere e riaprire il protocollo. Se si verifica un errore durante l'elaborazione, è possibile avviata direttamente una nuova esecuzione. Durante una nuova esecuzione, tutti i blocchi della pipeline vengono elaborati, anche se alcuni di essi sono contrassegnati con il segno verde.
Risultati
Tutte le analisi descritte sono state eseguite su un computer portatile standard (processore quad-core a 64 bit a 2,80 GHz con 16 GB di memoria ad accesso casuale (RAM)) che funziona con la versione a 64 bit di Java. La memoria ad accesso casuale è un parametro importante da considerare, a seconda della quantità e della risoluzione delle immagini da analizzare. L'utilizzo della versione a 32 bit di Java limita la memoria a circa 1300 MB, il che potrebbe non essere adatto per l'analisi dei Big Data, mentre la versione a...
Discussione
Un numero crescente di strumenti software liberi è disponibile per l'analisi delle immagini delle cellule a fluorescenza. Gli utenti devono scegliere correttamente il software adeguato in base alla complessità del loro problema, alle loro conoscenze nell'elaborazione delle immagini e al tempo che vogliono trascorrere nella loro analisi. Icy, CellProfiler o ImageJ/Fiji sono potenti strumenti che combinano sia l'usabilità che la funzionalità3. Icy è uno strumento autonomo che presenta una chiar...
Divulgazioni
Gli autori non hanno nulla da rivelare.
Riconoscimenti
G.H. è stato sostenuto da una borsa di studio della Ministère Délégué à la Recherche et aux Technologies. L.H. è stata sostenuta da una borsa di studio universitaria dell'Institut de Cancérologie de Lorraine (ICL), mentre Q.T. è stata sostenuta da una sovvenzione pubblica supervisionata dall'Agenzia nazionale francese di ricerca (ANR) come parte del secondo programma "Investissements d'Avenir" FIGHT-HF (riferimento: ANR-15-RHU4570004). Questo lavoro è stato finanziato dal CNRS e dall'Università di Lorraine (UMR 7365).
Materiali
| Name | Company | Catalog Number | Comments |
| 16% Formaldehyde solution (w/v) methanol free | Thermo Fisher Scientific | 28908 | to fix the cells |
| Alexa Fluor 488 of goat anti-rabbit | Thermo Fisher Scientific | A-11008 | fluorescent secondary antibody |
| Alexa Fluor 555 of goat anti-mouse | Thermo Fisher Scientific | A-21425 | fluorescent secondary antibody |
| Alexa Fluor 555 Phalloidin | Thermo Fisher Scientific | A34055 | fluorescent secondary antibody |
| Bovine serum albumin standard (BSA) | euromedex | 04-100-812-E | |
| DMEM | Sigma-Aldrich | D5796-500ml | cell culture medium |
| Duolink In Situ Mounting Medium with DAPI | Sigma-Aldrich | DUO82040-5ML | mounting medium |
| Human: HeLa S3 cells | IGBMC, Strasbourg, France | cell line used to perform the experiments | |
| Hydrogen peroxide solution 30% (H2O2) | Sigma-Aldrich | H1009-100ml | used as a stressing agent |
| Lipofectamine 2000 Reagent | Thermo Fisher Scientific | 11668-019 | transfection reagent |
| Mouse monoclonal anti-coilin | abcam | ab11822 | Coilin-specific antibody |
| Nikon Optiphot-2 fluorescence microscope | Nikon | epifluoresecence microscope | |
| Opti-MEM I Reduced Serum Medium | Thermo Fisher Scientific | 31985062 | transfection medium |
| PBS pH 7.4 (10x) | gibco | 70011-036 | to wash the cells |
| Rabbit polyclonal anti-53BP1 | Thermo Fisher Scientific | PA1-16565 | 53BP1-specific antibody |
| Rabbit polyclonal anti-EDC4 | Sigma-Aldrich | SAB4200114 | EDC4-specific antibody |
| Triton X-100 | Roth | 6683 | to permeabilize the cells |
Riferimenti
- Möckl, L., Lamb, D. C., Bräuchle, C. Super-resolved fluorescence microscopy: Nobel Prize in Chemistry 2014 for Eric Betzig, Stefan Hell, and William E. Moerner. Angewandte Chemie. 53 (51), 13972-13977 (2014).
- Meijering, E., Carpenter, A. E., Peng, H., Hamprecht, F. A., Olivo-Marin, J. -. C. Imagining the future of bioimage analysis. Nature Biotechnology. 34 (12), 1250-1255 (2016).
- Wiesmann, V., Franz, D., Held, C., Münzenmayer, C., Palmisano, R., Wittenberg, T. Review of free software tools for image analysis of fluorescence cell micrographs. Journal of Microscopy. 257 (1), 39-53 (2015).
- de Chaumont, F., et al. Icy: an open bioimage informatics platform for extended reproducible research. Nature Methods. 9 (7), 690-696 (2012).
- Girish, V., Vijayalakshmi, A. Affordable image analysis using NIH Image/ImageJ. Indian J Cancer. 41 (1), 47 (2004).
- Zaitoun, N. M., Aqel, M. J. Survey on image segmentation techniques. Procedia Computer Science. 65, 797-806 (2015).
- . MetaMorph Microscopy Automation and Image Analysis Software Available from: https://www.moleculardevices.com/products/cellular-imaging-systems/acquisition-and-analysis-software/metamorph-microscopy (2018)
- . NIS-Elements Imaging Software Available from: https://www.nikon.com/products/microscope-solutions/lineup/img_soft/nis-element (2014)
- Meyer, F., Beucher, S. Morphological segmentation. Journal of Visual Communication and Image Representation. 1 (1), 21-46 (1990).
- Schieber, M., Chandel, N. S. ROS Function in Redox Signaling and Oxidative Stress. Current Biology. 24 (10), 453-462 (2014).
- D'Autréaux, B., Toledano, M. B. ROS as signalling molecules: mechanisms that generate specificity in ROS homeostasis. Nature Reviews. Molecular Cell Biology. 8 (10), 813-824 (2007).
- Davalli, P., Mitic, T., Caporali, A., Lauriola, A., D'Arca, D. ROS, Cell Senescence, and Novel Molecular Mechanisms in Aging and Age-Related Diseases. Oxidative Medicine and Cellular Longevity. 2016, 3565127 (2016).
- Disher, K., Skandalis, A. Evidence of the modulation of mRNA splicing fidelity in humans by oxidative stress and p53. Genome. 50 (10), 946-953 (2007).
- Takeo, K., et al. Oxidative stress-induced alternative splicing of transformer 2β (SFRS10) and CD44 pre-mRNAs in gastric epithelial cells. American Journal of Physiology - Cell Physiology. 297 (2), 330-338 (2009).
- Seo, J., et al. Oxidative Stress Triggers Body-Wide Skipping of Multiple Exons of the Spinal Muscular Atrophy Gene. PLOS ONE. 11 (4), 0154390 (2016).
- Will, C. L., Luhrmann, R. Spliceosome Structure and Function. Cold Spring Harbor Perspectives in Biology. 3 (7), 003707 (2011).
- Ljosa, V., Sokolnicki, K. L., Carpenter, A. E. Annotated high-throughput microscopy image sets for validation. Nature Methods. 9 (7), 637-637 (2012).
- Wang, Q., et al. Cajal bodies are linked to genome conformation. Nature Communications. 7, (2016).
- Carpenter, A. E., et al. CellProfiler: image analysis software for identifying and quantifying cell phenotypes. Genome Biology. 7, 100 (2006).
- McQuin, C., et al. CellProfiler 3.0: Next-generation image processing for biology. PLoS Biology. 16 (7), 2005970 (2018).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon