Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Methods Article
Analyseur de sous-structure : Un flux de travail convivial pour l’exploration rapide et l’analyse précise des corps cellulaires dans les images de microscopie de fluorescence
Dans cet article
Résumé
Nous présentons un flux de travail librement disponible construit pour l’exploration rapide et l’analyse précise des corps cellulaires dans des compartiments cellulaires spécifiques dans des images de microscopie de fluorescence. Ce flux de travail convivial est conçu sur le logiciel open-source Icy et utilise également les fonctionnalités ImageJ. Le pipeline est abordable sans connaissance dans l’analyse d’image.
Résumé
La dernière décennie a été caractérisée par des percées dans les techniques de microscopie de fluorescence illustrées par l’amélioration de la résolution spatiale, mais aussi dans l’imagerie à cellules vivantes et les techniques de microscopie à haut débit. Cela a conduit à une augmentation constante de la quantité et de la complexité des données de microscopie pour une seule expérience. Étant donné que l’analyse manuelle des données de microscopie prend beaucoup de temps, qu’elle est subjective et qu’elle interdit les analyses quantitatives, l’automatisation de l’analyse de la bioimage devient presque inévitable. Nous avons construit un flux de travail informatique appelé Substructure Analyzer pour automatiser entièrement l’analyse du signal dans les bioimages à partir de la microscopie fluorescente. Ce flux de travail est développé sur la plate-forme open-source iux conviviale et est complété par des fonctionnalités d’ImageJ. Il comprend le pré-traitement des images pour améliorer le rapport signal/bruit, la segmentation individuelle des cellules (détection des limites cellulaires) et la détection/quantification des corps cellulaires enrichis dans des compartiments cellulaires spécifiques. Le principal avantage de ce flux de travail est de proposer des fonctionnalités complexes de bio-imagerie aux utilisateurs sans expertise en analyse d’image via une interface conviviale. En outre, il est très modulaire et adapté à plusieurs questions allant de la caractérisation de la translocation nucléaire/cytoplasmique à l’analyse comparative de différents corps cellulaires dans différentes sous-structures cellulaires. La fonctionnalité de ce flux de travail est illustrée par l’étude des corps Cajal (enroulés) dans des conditions de stress oxydatif (OS). Les données de la microscopie de fluorescence montrent que leur intégrité dans les cellules humaines est affectée quelques heures après l’induction de l’OS. Cet effet est caractérisé par une diminution de la nucléation de la coiline dans les corps cajal caractéristiques, associée à une redistribution nucléoplasmique de la coiline dans un nombre accru de foyers plus petits. Le rôle central de la coiline dans l’échange entre les composants CB et le nucléoplasme environnant suggère que la redistribution induite par l’OS de la coiline pourrait affecter la composition et la fonctionnalité des corps de Cajal.
Introduction
La microscopie légère et, plus particulièrement, la microscopie de fluorescence sont des techniques robustes et polyvalentes couramment utilisées en sciences biologiques. Ils donnent accès à la localisation précise de diverses biomolécules comme les protéines ou l’ARN par leur étiquetage fluorescent spécifique. La dernière décennie a été caractérisée par des progrès rapides dans les technologies de microscopie et d’imagerie comme en témoigne le prix Nobel de chimie 2014 qui récompense Eric Betzig, Stefan W. Hell et William E. Moerner pour le développement de la microscopie de fluorescence super-résolue (SRFM)1. SFRM contourne la limite de diffraction de la microscopie optique traditionnelle pour l’introduire dans la nanodimension. L’amélioration des techniques comme l’imagerie vivante ou les approches de dépistage à haut débit augmente également la quantité et la complexité des données à traiter pour chaque expérience. La plupart du temps, les chercheurs sont confrontés à des populations hétérogènes élevées de cellules et veulent analyser les phénotypes au niveau unicellulaire.
Au départ, des analyses telles que le comptage des foyers ont été effectuées par l’œil, ce qui est préféré par certains chercheurs car il fournit un contrôle visuel complet sur le processus de comptage. Toutefois, l’analyse manuelle de ces données prend trop de temps, entraîne une variabilité entre les observateurs et ne donne pas accès à des fonctionnalités plus complexes de sorte que les approches assistées par ordinateur sont de plus en plus largement utilisées et presqueinévitables 2. Les méthodes informatiques de Bioimage augmentent considérablement l’efficacité de l’analyse des données et sont exemptes de la subjectivité inévitable de l’opérateur et du biais potentiel de l’analyse manuelle de comptage. L’augmentation de la demande dans ce domaine et l’amélioration de la puissance informatique ont conduit au développement d’un grand nombre de plates-formes d’analyse d’images. Certains d’entre eux sont disponibles gratuitement et donnent accès à divers outils pour effectuer l’analyse avec des ordinateurs personnels. Une classification des outils d’accès libre a été récemment établie3 et présente Icy4 comme un logiciel puissant combinant la facilité d’utilisation et la fonctionnalité. De plus, Icy a l’avantage de communiquer avec ImageJ.
Pour les utilisateurs qui n’ont pas d’expertise en analyse d’image, les principaux obstacles sont de choisir l’outil approprié en fonction des paramètres problématiques et correctement régler qui sont souvent mal compris. De plus, les temps d’installation sont souvent longs. Icy propose une interface point-and-click conviviale nommée « Protocoles » pour développer le flux de travail en combinant certains plugins trouvés dans une collection exhaustive4. La conception modulaire flexible et l’interface point-and-click rendent la mise en place d’une analyse réalisable pour les non-programmeurs. Nous présentons ici un flux de travail appelé Substructure Analyzer, développé dans l’interface de Icy, dont la fonction est d’analyser les signaux fluorescents dans des compartiments cellulaires spécifiques et de mesurer différentes caractéristiques comme la luminosité, le nombre de foyers, la taille des foyers et la distribution spatiale. Ce flux de travail aborde plusieurs questions telles que la quantification de la translocation du signal, l’analyse des cellules transfectées exprimant un journaliste fluorescent, ou l’analyse des foyers de différentes sous-structures cellulaires dans les cellules individuelles. Il permet le traitement simultané de plusieurs images, et les résultats de sortie sont exportés vers une feuille de calcul délimitée par onglets qui peut être ouverte dans les programmes de feuille de calcul couramment utilisés.
Le pipeline De substructure Analyzer est présenté à la figure 1. Tout d’abord, toutes les images contenues dans un dossier spécifié sont pré-traitées pour améliorer leur rapport signal/bruit. Cette étape augmente l’efficacité des étapes suivantes et diminue le temps de fonctionnement. Ensuite, les régions d’intérêt (ROV), correspondant aux zones d’image où le signal fluorescent doit être détecté, sont identifiées et segmentées. Enfin, le signal fluorescent est analysé et les résultats sont exportés dans une feuille de calcul délimitée par onglets.
La segmentation des objets (détection des limites) est l’étape la plus difficile dans l’analyse d’image, et son efficacité détermine la précision des mesures cellulaires résultantes. Les premiers objets identifiés dans une image (appelés objets primaires) sont souvent des noyaux d’images tachées d’ADN (taches DAPI ou Hoechst), bien que les objets primaires puissent aussi être des cellules entières, des perles, des taches, des tumeurs ou tout autre objet taché. Dans la plupart des images biologiques, les cellules ou les noyaux se touchent ou se chevauchent, ce qui provoque l’échec des algorithmes simples et rapides. À ce jour, aucun algorithme universel ne peut effectuer une segmentation parfaite de tous les objets, principalement parce que leurs caractéristiques (taille, forme ou texture) modulent l’efficacité de la segmentation5. Les outils de segmentation couramment distribués avec des logiciels de microscopie (tels que le logiciel d’imagerie MetaMorph par Molecular Devices6, ou le logiciel NIS-Elements Advances Research par Nikon7) sont généralement basés sur des techniques standard telles que l’appariement de corrélation, le seuil ou les opérations morphologiques. Bien qu’efficaces dans les systèmes de base, ces méthodes surgénéralisées présentent rapidement des limites lorsqu’elles sont utilisées dans des contextes plus difficiles et spécifiques. En effet, la segmentation est très sensible aux paramètres expérimentaux tels que le type de cellule, la densité cellulaire ou les biomarqueurs, et nécessite souvent des ajustements répétés pour un grand ensemble de données. Le flux de travail Substructure Analyzer intègre des algorithmes simples et plus sophistiqués pour proposer différentes alternatives adaptées à la complexité de l’image et aux besoins des utilisateurs. Il propose notamment l’algorithme de bassin versant8 basé sur des marqueurs pour les objets fortement groupés. L’efficacité de cette méthode de segmentation repose sur la sélection de marqueurs individuels sur chaque objet. Ces marqueurs sont choisis manuellement la plupart du temps pour obtenir des paramètres corrects pour la segmentation complète, ce qui prend beaucoup de temps lorsque les utilisateurs font face à un grand nombre d’objets. Substructure Analyzer propose une détection automatique de ces marqueurs, offrant un processus de segmentation très efficace. La segmentation est, la plupart du temps, l’étape limitante de l’analyse d’image et peut modifier considérablement le temps de traitement en fonction de la résolution de l’image, le nombre d’objets par image, et le niveau de regroupement des objets. Les pipelines typiques nécessitent de quelques secondes à 5 minutes par image sur un ordinateur de bureau standard. L’analyse d’images plus complexes peut nécessiter un ordinateur plus puissant et des connaissances de base dans l’analyse d’images.
La flexibilité et la fonctionnalité de ce flux de travail sont illustrées par divers exemples dans les résultats représentatifs. Les avantages de ce flux de travail sont notamment affichés par l’étude des sous-structures nucléaires dans des conditions de stress oxydatif (OS). L’OS correspond à un déséquilibre de l’homéostasie redox en faveur des oxydants et est associé à des niveaux élevés d’espèces réactives d’oxygène (ROS). Puisque ros agissent comme molécules de signalisation, les changements dans leur concentration et la localisation subcellulaire affectent positivement ou négativement une myriade de voies et de réseaux qui régulent les fonctions physiologiques, y compris la transduction du signal, les mécanismes de réparation, l’expression des gènes, la mort cellulaire, et la prolifération9,10. OS est donc directement impliqué dans diverses pathologies (maladies neurodégénératives et cardiovasculaires, cancers, diabète, etc.), mais aussi dans le vieillissement cellulaire. Par conséquent, déchiffrer les conséquences de l’OS sur l’organisation et la fonction de la cellule humaine constitue une étape cruciale dans la compréhension des rôles de l’OS dans le début et le développement des pathologies humaines. Il a été établi que l’OS régule l’expression des gènes en modulant la transcription à travers plusieurs facteurs de transcription (p53, Nrf2, FOXO3A)11, mais aussi en affectant la régulation de plusieurs processus co- et post-transcriptionnels tels que l’épissage alternatif (AS) des pré-ARN12,13,14. L’épissage alternatif des transcriptions primaires de codage et de non-codage est un mécanisme essentiel qui augmente la capacité d’encodage des génomes en produisant des isoformes de transcription. AS est effectuée par un énorme complexe de ribonucléoprotéines appelé splicéosome, contenant près de 300 protéines et 5 RN nucléaires riches en U (UsnARN)15. L’assemblage splicéosome et l’AS sont étroitement contrôlés dans les cellules et certaines étapes de la maturation splicéosome se produisent dans les compartiments nucléaires sans membrane nommés Cajal Bodies. Ces sous-structures nucléaires se caractérisent par la nature dynamique de leur structure et leur composition, qui sont principalement menées par des interactions multivalentes de leur ARN et des composants protéiques avec la protéine coiline. L’analyse de milliers de cellules avec le flux de travail de l’analyseur de sous-structure a permis la caractérisation des effets jamais décrits de l’OS sur les corps de Cajal. En effet, les données obtenues suggèrent que l’OS modifie la nucléation des corps de Cajal, induisant une redistribution nucléoplasmique de la protéine de coiline dans de nombreux foyers nucléaires plus petits. Un tel changement de la structure des corps de Cajal pourrait affecter la maturation du splicéosome et participer à la modulation AS par OS.
Protocole
REMARQUE : Des didacticiels convivial sont disponibles sur le site web de Icy http://icy.bioimageanalysis.org.
1. Télécharger icy et le protocole d’analyseur de sous-structure
- Téléchargez Icy à partir du site Icy (http://icy.bioimageanalysis.org/download) et téléchargez le protocole Substructure Analyzer : http://icy.bioimageanalysis.org/protocols?sort=latest.
REMARQUE : Si vous utilisez un système d’exploitation 64 bits, assurez-vous d’utiliser la version 64 bits de Java. Cette version permet d’augmenter la mémoire allouée à Icy (Préférences | Généralités | Mémoire maximale).
2. Ouverture du protocole
- Ouvrez Icy et cliquez sur Outils dans le menu Ruban.
- Cliquez sur Protocoles pour ouvrir l’interface Éditeur de protocoles.
- Cliquez sur Charger et ouvrez l’analyseur de sous-structure deprotocole . Le chargement du protocole peut prendre quelques secondes. Assurez-vous que l’ouverture du protocole est terminée avant de l’utiliser.
REMARQUE : Le flux de travail est composé de 13 blocs généraux présentés à la figure 2a. Chaque bloc fonctionne comme un pipeline composé de plusieurs boîtes effectuant des sous-tâches spécifiques.
3. Interagir avec le flux de travail sur Icy
REMARQUE : Chaque bloc ou boîte est numéroté et a un rang spécifique dans le flux de travail (Figure 2b). En cliquant sur ce numéro, la position la plus proche possible de la première est attribuée au bloc/boîte sélectionné, puis la position des autres blocs/cases est réorganisée. Respectez le bon ordre des blocs lors de la préparation du flux de travail. Par exemple, le bloc de détection de taches a besoin d’vois prédéfinis de sorte que les blocs de segmentation doivent s’exécuter avant les blocs de détecteur de tache. Ne modifiez pas la position des cases. N’utilisez pas « » dans le nom de l’image.
- En cliquant sur l’icône du coin supérieur gauche, effondrez, élargissez, agrandissez, rétrécissez ou supprimez le bloc (Figure 2b).
- Chaque pipeline du flux de travail est caractérisé par un réseau de boîtes connectées via leur entrée et leur sortie (Figure 2b). Pour créer une connexion, cliquez sur Sortie et maintenez jusqu’à ce que le curseur atteigne une entrée. Les connexions peuvent être supprimées en cliquant sur la balise Sortie.
4. Fusion des canaux d’une image
- Utilisez le bloc Merge Channels pour générer des images fusionnées. Si nécessaire, renommez les fichiers de sorte que les séquences à fusionner ont le préfixe du même nom suivi d’un séparateur distinct. Par exemple, les séquences de canaux individuels d’une image A sont nommées : ImageA_red, ImageA_blue.
REMARQUE : Pour le séparateur, n’utilisez pas les caractères déjà présents dans le nom de l’image. - Dans le même dossier, créez un nouveau dossier par canal pour fusionner. Par exemple, pour fusionner les canaux rouges, verts et bleus, créez 3 dossiers et stockez les séquences correspondantes dans ces dossiers.
- Utilisez uniquement le bloc Merge Channels, supprimez les autres blocs et enregistrez le protocole en tant que canaux de fusion.
- Accédez aux cases pour définir des paramètres. Pour chaque canal, remplissez les cases Numéro de canal X (cases 1, 5 ou 9), Numéro de canal dossier X (boîtes 2, 6 ou 10), numéro de canal séparateur X (boîtes 3, 7 ou 11) et canal Colormap nb X (boîtes 4, 8 et 12) respectivement.
REMARQUE : Ces boîtes sont regroupées horizontalement par quatre, chaque ligne correspondant au même canal. Dans chaque ligne, un affichage est également disponible (cases 23, 24 ou 25) pour visualiser directement la séquence du canal correspondant.- Dans la zone Numéro de canal X, choisissez le canal à extraire (dans les images RGB classiques, 0=Rouge, 1=Vert, 2=Bleu). L’utilisateur accède rapidement aux différents canaux d’une image dans la fenêtre Inspecteur de Icy, dans l’onglet Séquence. Écrivez la valeur du plus petit canal dans la ligne supérieure et la plus élevée de la ligne de fond.
- Dans la zone Dossier numéro de canal X, écrivez le \Nom du dossier contenant des images du canal X.
- Dans la boîte Numéro de canal séparateur X, écrivez le séparateur utilisé pour le nom de l’image (dans l’exemple précédent : « _red », « _green » et « _blue »).
- Dans la zone Colormap channel nb X, indiquez avec un nombre quel modèle colormap utiliser pour visualiser le canal correspondant dans Icy. Les colormaps disponibles sont visibles dans l’onglet Séquence de la fenêtre Inspecteur.
- Dans la zone Format des images fusionnées (encadré 28), écrivez l’extension pour enregistrer les images fusionnées : .tif, .gif, .jpg, .bmp ou .png.
REMARQUE : Pour fusionner seulement 2 canaux, ne remplissez pas les quatre cases correspondant au troisième canal.
- Dans le coin supérieur gauche du bloc Canaux de fusion, cliquez sur le lien directement à droite du dossier. Dans la boîte de dialogue Ouvrir qui s’affiche, double-cliquez sur le dossier contenant des séquences du premier canal qui a été défini dans la zone Dossier numéro 1 (encadré 2). Ensuite, cliquez sur Ouvrir.
- Exécutez le protocole en cliquant sur la flèche noire dans le coin supérieur gauche du bloc Canaux de fusion (voir la partie 7 pour plus de détails). Les images fusionnées sont enregistrées dans un dossier Merge dans le même répertoire que les dossiers des canaux individuels.
- Accédez aux cases pour définir des paramètres. Pour chaque canal, remplissez les cases Numéro de canal X (cases 1, 5 ou 9), Numéro de canal dossier X (boîtes 2, 6 ou 10), numéro de canal séparateur X (boîtes 3, 7 ou 11) et canal Colormap nb X (boîtes 4, 8 et 12) respectivement.
5. Segmentation des régions d’intérêt
REMARQUE : Substructure Analyzer intègre des algorithmes simples et plus sophistiqués pour proposer différentes alternatives adaptées à la complexité de l’image et aux besoins des utilisateurs.
- Sélectionnez le bloc adapté.
- Si les objets ne se touchent pas ou si l’utilisateur n’a pas besoin de différencier individuellement les objets en cluster, utilisez le bloc Segmentation A : Objets non groupés.
- Lorsque les objets ne se touchent pas, mais que certains d’entre eux sont proches, utilisez le bloc Segmentation B : objets mal regroupés.
- Pour les objets ayant un niveau de clustering élevé et une forme convexe, utilisez le bloc Segmentation C : objets groupés avec des formes convexes.
- Si les objets présentent un niveau de clustering élevé et ont des formes irrégulières, utilisez le bloc Segmentation D : objets groupés de formes irrégulières.
- Utilisez le bloc Segmentation E : Cytoplasme groupé pour segmenter les cytoplasmes en touchant individuellement en utilisant des noyaux segmentés comme marqueurs. Ce bloc a un besoin impératif de noyaux segmentés pour le traiter.
REMARQUE : Blocs adaptés au processus de segmentation des objets primaires indépendamment afin que plusieurs blocs puissent être utilisés dans la même course pour comparer leur efficacité pour une sous-structure particulière ou pour segmenter différents types de sous-structures. Si le niveau de clustering est hétérogène dans le même ensemble d’images, traitez les objets petits et fortement groupés séparément dans les blocs adaptés.
- Reliez la sortie0 (Fichier) du bloc Sélectionner le dossier à l’entrée de dossier du bloc de segmentation choisi.
- Définissez les paramètres du bloc de segmentation choisi.
- Segmentation A : objets non groupés et segmentation C : objets groupés avec des formes convexes
- Dans la boîte Signal de canal (encadré 1), définissez le canal des objets sur segment.
- En option, dans la boîte filtre gaussien (encadré 2), augmentez les valeurs sigma X et Y si le signal à l’intérieur des objets est hétérogène. Le filtre gaussien lisse les textures pour obtenir des régions plus uniformes et augmente la vitesse et l’efficacité de la segmentation des noyaux. Plus les objets sont petits, plus la valeur sigma est faible. Évitez les valeurs sigma élevées. Définissez les valeurs par défaut sur 0.
- Dans la case HK-Means (encadré 3), définissez le paramètre Intensité des classes et les tailles minimales et maximales approximatives (en pixels) des objets à détecter.
REMARQUE : Pour les classes d’intensité, une valeur de 2 classes classe en 2 classes : arrière-plan et premier plan. Il est ainsi adapté lorsque le contraste entre les objets et l’arrière-plan est élevé. Si les objets de premier plan ont des intensités différentes ou si le contraste avec l’arrière-plan est faible, augmentez le nombre de classes. Le paramètre par défaut est 2. La taille de l’objet peut être rapidement évaluée en dessinant manuellement un retour sur investissement autour de l’objet d’intérêt. La taille du ROI (Intérieur en pixels) apparaît directement sur l’image lorsque vous la pointez avec le curseur ou peut être consultée dans la fenêtre statistiques de retour sur investissement (ouvrez-la à partir de la barre de recherche). Les paramètres optimaux détectent chaque objet de premier plan dans un seul roi. Ils peuvent être définis manuellement dans Icy(Détection et suivi | HK-Means). - Dans la zone Contours actifs (encadré 4), optimisez la détection des bordures d’objet. Une documentation exhaustive pour ce plugin est disponible en ligne: http://icy.bioimageanalysis.org/plugin/Active_Contours. Les paramètres corrects peuvent également être définis manuellement dans Icy (Détection et suivi | Contours actifs).
- Pendant le processus, un dossier est automatiquement créé pour enregistrer des images d’objets segmentés. Dans la zone Texte (zone 6), nommez ce dossier (ex : noyaux segmentés). Pour définir le format d’enregistrement des images d’objets segmentés (Tiff, Gif, Jpeg, BMP, PNG), remplissez le format de boîte d’images d’objets segmentés. Le dossier est créé dans le dossier contenant des images fusionnées.
- Exécutez le flux de travail (pour plus de détails, voir la partie 7).
- Segmentation B : objets mal regroupés
- Suivez les mêmes étapes que dans 5.3.1 pour définir les paramètres des cases Signal de canal, HK-Means, Active Contours , Extension pour enregistrer les objets segmentés et le texte (Les rangs des boîtes ne sont pas les mêmes qu’à l’étape 5.3.1). Active Contours
- Dans la zone Appelez le plugin IJ (encadré 4), définissez le paramètre Rolling pour contrôler la soustraction d’arrière-plan. Définissez ce paramètre sur au moins la taille de l’objet le plus grand qui ne fait pas partie de l’arrière-plan. La diminution de cette valeur augmente la suppression de l’arrière-plan, mais peut également induire la perte du signal de premier plan.
- Dans la zone D’égalisation d’histogramme adaptatif (encadré 6), améliorez les contrastes entre les objets de premier plan et l’arrière-plan. L’augmentation de la pente donne des séquences plus contrastées.
- Exécutez le flux de travail (pour plus de détails, voir la partie 7).
- Segmentation D : objets groupés de formes irrégulières
REMARQUE : Trois méthodes différentes de segmentation s’appliquent à chaque image : premièrement,Clustering hk-meanscombiné avec leActive Contours, méthodeest appliquée. Ensuite, lealgorithme classique de bassin versant(à l’aide de la carte de distance Euclidian) est appliqué aux objets précédemment mal segmentés. Enfin, unalgorithme de bassin versant basé sur les marqueursest utilisé. Seules les méthodes de pointe hk et de bassins versants basées sur les marqueurs doivent être interventions de l’utilisateur. Pour les deux méthodes, les mêmes paramètres peuvent être appliqués pour toutes les images (version entièrement automatisée) ou être modifiés pour chaque image (version semi-automatisée). Si l’utilisateur n’est pas formé à ces méthodes de segmentation, le traitement semi-automatisé est fortement recommandé. Pendant le traitement de ce bloc, une intervention manuelle est nécessaire. Lorsqu’une méthode de segmentation est terminée, l’utilisateur doit supprimer manuellement les objets mal segmentés avant le début de la méthode de segmentation suivante. Les objets segmentés avec succès sont enregistrés et non pris en compte à l’étape suivante. Ce bloc doit être connecté au blocBoîte de dialogue Segmentation des objets primaires de formes groupées/hétérogènesfonctionner correctement.- Téléchargez la collection ImageJ MorphoLibJ sur https://github.com/ijpb/MorphoLibJ/releases. La version MorphoLibJ 1.4.0 est utilisée dans ce protocole. Placez le fichier MorphoLibJ_-1.4.0.jar dans le dossier icy/ij/plugins. Plus d’informations sur le contenu de cette collection sont disponibles sur https://imagej.net/MorphoLibJ.
- Suivez les mêmes étapes que dans l’étape 5.3.1 pour définir les paramètres des boîtes Signal de canal, Filtre gaussien, Contours actifs , Extension pour enregistrer les objets segmentés et Texte. Active Contours Les rangs des boîtes ne sont pas les mêmes qu’à l’étape 5.3.1.
- Définir les paramètres de la boîte D’égalisation d’histogramme adaptatif (voir étape 5.3.2.3).
- Pour activer Subtract Background, écrivez oui dans Appliquer l’arrière-plan de soustrait ? (encadré 5). Sinon, écrivez No. Si le plugin est activé, définissez le paramètre de roulement (voir l’étape 5.3.2), dans la zone Soustraire le paramètre Background (encadré 7).
- Automatisation des moyens HK : Pour appliquer les mêmes paramètres pour toutes les images (traitement entièrement automatisé), définissez le Nb des classes (encadré 11), la taille minimale (encadré 12) et la taille maximale (encadré13) (voir étape 5.3.1). Ces paramètres doivent être configurés pour sélectionner un maximum de pixels de premier plan et pour optimiser l’individualisation des objets de premier plan. Pour la version de traitement semi-automatisée, aucune intervention n’est nécessaire.
- Automatisation des extractions de marqueurs : pour la version entièrement automatisée, développez la zone Extraction de marqueurs internes (encadré 27) et définissez la valeur du paramètre « dynamique » dans la ligne 13 du script. Pour la version de traitement semi-automatisée, aucune intervention n’est nécessaire.
REMARQUE : Les marqueurs sont extraits en appliquant une transformation de minima étendus sur une image d’entrée contrôlée par un paramètre « dynamique ». Dans l’algorithme de bassin versant basé sur les marqueurs, l’inondation de ces marqueurs est simulée pour effectuer la segmentation des objets. Pour la segmentation réussie des objets de premier plan, un seul marqueur par objet de premier plan doit être extrait. Le réglage du paramètre « ypiqu » pour l’extraction optimale des marqueurs dépend principalement de la résolution des images. Ainsi, si vous n’êtes pas familier avec ce paramètre, utilisez la version semi-automatisée. - Exécutez le flux de travail (pour plus de détails, voir la partie 7).
- Au début du traitement, les boîtes de dialogue HK-means paramètres et le bassin versant basé sur les marqueurs s’ouvrent successivement. Pour appliquer les mêmes paramètres pour toutes les images (version entièrement automatisée), cliquez sur OUI. Sinon, cliquez sur NO. Une boîte d’information s’ouvre, demandant de « Déterminer les ROI optimaux avec le plugin HK-Means et l’image étroite ». Cliquez sur OK et appliquez manuellement le plugin HK-Means(Détection et suivi| HK-Means) sur l’image, qui s’ouvre automatiquement. Sélectionnez l’option Exporter des ROV dans la zone plugin HK-Means. Appliquez les meilleurs paramètres pour avoir des ROI contenant un maximum de pixels de premier plan et pour optimiser l’individualisation des objets de premier plan. Lorsque des VOI optimaux sont trouvés, fermez directement l’image.
- À la fin de la première méthode de segmentation, une boîte d’information s’ouvre et demande de « Supprimer les ORI indésirables et fermer l’image ». Ces VOI correspondent aux bordures des objets segmentés. Sélectionnez OK et supprimez les VOI des objets mal segmentés dans l’image, qui s’ouvre automatiquement. Un retour sur investissement peut être facilement supprimé en plaçant le curseur sur sa bordure et en utilisant le bouton « upprime » du clavier. Fermez l’image. Répétez la même procédure après l’achèvement de la deuxième étape de segmentation.
- À ce stade, si le bouton OUI a été sélectionné pour l’automatisation complète de l’algorithme de bassin versant basé sur les marqueurs, les paramètres précédemment fixés seront appliqués à toutes les images.
- Si le bouton NO a été sélectionné, une zone d’information s’ouvre, demandant de « Déterminer et ajuster les marqueurs internes ». Cliquez sur OK et dans l’interface ImageJ de Icy, accédez à Plugins | MorphoLibJ | Minima et Maxima| Min et Max étendus. Dans Operation, sélectionnez Minima étendu.
- Sélectionnez Aperçu pour pré-visualiser sur l’image ouverte automatiquement le résultat de la transformation. Déplacez la dynamique jusqu’à ce que des marqueurs optimaux soient observés. Les marqueurs sont des groupes de pixels d’une valeur de 255 (pas nécessairement des pixels blancs). Les paramètres optimaux conduisent à un marqueur par objet. Concentrez-vous sur les objets restants qui n’ont pas été bien segmentés avec les deux méthodes de segmentation précédentes.
- Si nécessaire, améliorer les marqueurs en appliquant des opérations morphologiques supplémentaires comme « unt » ou « oute » (Plugins | MorphoLibJ | Filtres morphologiques). Lorsque vous obtenez l’image finale des marqueurs, gardez-la ouverte et fermez toutes les autres images se terminant par l’image initialement utilisée comme entrée pour l’opération Minima étendu. Cliquez sur Non si une zone ImageJ demande à enregistrer les modifications sur cette image.
- Dans la zone Nb des images avec la zone d’information (encadré 14), déterminez le nombre d’images avec les boîtes d’information à afficher.
- Segmentation E : cytoplasme groupé
REMARQUE : Ce bloc utilise des noyaux segmentés précédemment comme marqueurs individuels pour initier la segmentation du cytoplasme. Assurez-vous que le bloc de segmentation des noyaux a été traité avant de l’utiliser.- Dans la boîte Canal cytoplasme (encadré 1), définissez le canal du signal cytoplasmique.
- Dans la boîte Extension segmenté noyaux (encadré 2), écrivez le format utilisé pour enregistrer des images de noyaux segmentés (tif, jpeg, bmp, png). Le format par défaut est tif.
- Dans la zone Texte (zone 3), écrivez le \Nom du dossier contenant des noyaux segmentés.
- Dans la boîte Format d’images de cytoplasmes segmentés (encadré 4), définissez le format à utiliser pour enregistrer des images d’objets segmentés (Tiff, Gif, Jpeg, BMP, PNG).
- Pendant le processus, un dossier est automatiquement créé pour enregistrer des images de cytoplasmes segmentés. Dans la zone Texte (zone 5), nommez ce dossier (ex : cytoplasmes segmentés). Le dossier est créé dans le dossier contenant des images fusionnées.
- Suivez les mêmes étapes qu’à l’étape 5.3.1 pour définir les paramètres des cases Filtre gaussien et Contours actifs (Attention, les classements des cases ne sont pas les mêmes qu’à l’étape 5.3.1).
- Exécutez le flux de travail (pour plus de détails, voir la partie 7).
- Segmentation A : objets non groupés et segmentation C : objets groupés avec des formes convexes
6. Détection et analyse des signaux fluorescents
- Sélectionnez le bloc adapté.
- Dans le bloc Fluorescence Analysis A: 1 Channel, effectuer la détection et l’analyse des foyers dans un canal à l’intérieur d’un type d’objet segmenté: détection des foyers de coilin (canal rouge) dans le noyau.
- Dans le bloc Fluorescence Analysis B: 2 Canaux dans le même compartiment, effectuer la détection et l’analyse des foyers dans deux canaux à l’intérieur d’un type d’objet segmenté: détection de la coiline (canal rouge) et 53BP1 (canal vert) foyers dans le noyau.
- Dans le bloc Fluorescence Analysis C: 2 Canaux dans deux compartiments, effectuer la détection et l’analyse des foyers dans un ou deux canaux, en particulier à l’intérieur des noyaux et de leur cytoplasme correspondant: détection de Coilin foyers (canal rouge) à la fois dans le noyau et son cytoplasme correspondant ou la détection de Coilin foyers (canal rouge) dans le noyau et G3BP foyers (canal vert) dans le cytoplasme correspondant.
- Dans le bloc Fluorescence Analysis D: Global Translocation, calculer le pourcentage de signal à partir d’un canal dans deux compartiments cellulaires (a et b). Par exemple, dans un essai de translocation cytoplasme/noyau, exporter des pourcentages calculés de signaux nucléaires et cytoplasmiques pour chaque image de la feuille de calcul finale « Résultats ». La formule utilisée pour calculer le pourcentage de signal nucléaire est indiquée ci-dessous. Ce bloc peut être utilisé pour n’importe quel compartiment subcellulaire :
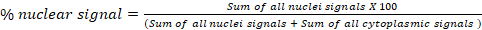
- Dans le bloc Fluorescence Analysis E: Individual Cell Translocation, calculer le pourcentage de signal à partir d’un canal dans deux compartiments cellulaires pour chaque cellule. Ce bloc est spécialement optimisé pour l’essai de translocation du noyau/cytoplasme au niveau unicellulaire.
REMARQUE : Étant donné que le bloc De fluorescence Analyse E : La translocation individuelle des cellules effectue une analyse au niveau unicellulaire, une segmentation efficace du noyau et du cytoplasme est nécessaire.
- Reliez la sortie0 (Fichier) du bloc Sélectionner le dossier (bloc 1) à l’entrée de dossier (flèches blanches dans les cercles noirs) du bloc choisi.
- Définissez les paramètres du bloc choisi.
- Analyse de fluorescence A : 1 canal, analyse de fluorescence B : 2 canaux dans le même compartiment et analyse de fluorescence C : 2 canaux dans deux compartiments
- Dans la zone Images du dossier ROI, écrivez le nom du dossier contenant des images d’objets segmentés précédés d’une barre oblique inverse. (Par exemple : \Noyaux segmentés).
- Dans la case Format d’images d’objets segmentés (encadré 2), écrivez le format utilisé pour enregistrer des images d’objets segmentés (tif, jpeg, bmp, png). Le format par défaut est tif.
- Dans la zone Kill Borders?, écrire Oui pour supprimer les objets de bordure. Sinon, écrivez No. L’installation de la collection MorphoLibJ d’ImageJ est nécessaire pour utiliser cette fonction (voir l’étape 5.3.3).
- Dans la boîte(es) Signal de taches de canal, définissez le canal où les taches doivent être détectées. Dans les images RGB classiques, 0=Rouge, 1=Vert et 2=Bleu.
- Dans les cases Nom de la molécule localisée, écrire le nom de la molécule qui se localise dans les taches. Le nombre de champs à entrer dépend du nombre de molécules.
- Dans la zone(es) Wavelet Spot Detector Block, définissez les paramètres de détection de tache pour chaque canal. Définissez l’échelle (s) (référence à la taille du spot) et la sensibilité de la détection (une sensibilité plus faible diminue le nombre de taches détectées, la valeur par défaut étant de 100 et la valeur minimale étant 0). La documentation exhaustive de ce plugin est disponible en ligne: http://icy.bioimageanalysis.org/plugin/Spot_Detector. Les paramètres peuvent également être définis manuellement dans Icy (Détection et suivi | Détecteur de taches).
- En option, dans la zone Filtrer le retour sur investissement par taille, filtrez les objets segmentés où les taches sont détectées en définissant un intervalle de taille (en pixels). Cette étape est particulièrement utile pour supprimer les objets sous-segmentés ou sur segmentés. Pour estimer manuellement la taille des objets, voir étape 5.3.1. Les paramètres par défaut n’incluent pas le filtrage des ROV par taille. Le bloc 2 Canaux dans deux compartiments contient deux cases : les noyaux de filtre par taille (encadré 19) et le cytoplasme de filtre par taille (encadré 46).
- En option, dans la boîte Filtrer les taches par taille, filtrer les taches détectées en fonction de leur taille (en pixels) pour supprimer les artefacts indésirables. Pour estimer manuellement la taille des taches, cliquez sur Détection et suivi et ouvrez le plugin du détecteur spot. Dans les options De sortie, sélectionnez Exporter vers le retour sur investissement. Veillez à ce que les paramètres par défaut n’incluent pas le filtrage des taches par taille et que les taches filtrées ne soient pas prises en compte pour l’analyse. Le nombre de champs à entrer dépend du nombre de canaux.
- En option, dans la boîte Taches de filtre, appliquez un filtre supplémentaire (contraste, homogénéité, périmètre, rondeur) sur les taches détectées. Veillez à ce que les paramètres par défaut n’incluent pas le filtrage des taches et que les taches filtrées ne soient pas prises en compte pour l’analyse. Le nombre de champs à entrer dépend du nombre de canaux.
- En option, dans les cases Seuil de taille de tache, définissez un seuil pour la zone (en pixels) des taches analysées. Le nombre de taches comptées en dessous et au-dessus de ce seuil est exporté dans la feuille de calcul final des résultats. Le nombre de cases à informer dépend du nombre de canaux.
- Exécutez le flux de travail (pour plus de détails, voir la partie 7). Les données sont exportées dans une feuille de calcul Résultats enregistrée dans le dossier contenant des images fusionnées.
- Analyse de fluorescence D : Analyse globale de translocation et de fluorescence E : Translocation individuelle des cellules :
- Dans les cases Images du dossier (cases 1 et 2), écrivez le \Nom du dossier contenant des images d’objets segmentés. Dans le bloc Fluorescence Analysis D: Global Translocation, les deux types de retour sur investissement sont identifiés comme roi a et roi b. Pour le bloc Fluorescence Analysis E: Individual Cell Translocation, dans les images de dossier segmentés noyaux et dossiers images segmentées cytoplasmes boîtes, écrire le nom du dossier contenant des noyaux segmentés et cytoplasmes, respectivement.
- Dans la zone Signal de canal (encadré 3), entrez le canal du signal.
- Dans la case Format d’images d’objets segmentés (encadré 4), écrivez le format utilisé pour enregistrer des images d’objets segmentés (tif, jpeg, bmp, png). Le format par défaut est tif. L’option Kill Borders est également disponible pour supprimer les objets de bordure (voir l’étape 6.3.1).
- En option, dans les cases Filtrer le retour sur investissement par taille, filtrer les objets segmentés en définissant un intervalle de taille (en pixels). Cette étape peut être utile pour supprimer les objets sous ou sur segmentés. Pour estimer manuellement la taille des objets, voir étape 5.3.1. Il y a deux champs à entrer, un par canal. Les paramètres par défaut n’incluent pas le filtrage du retour sur investissement par taille.
- Exécutez le flux de travail (pour plus de détails, voir la partie 7). Exporter des données dans une feuille de calcul Résultats enregistrés dans le dossier contenant des images fusionnées.
- Analyse de fluorescence A : 1 canal, analyse de fluorescence B : 2 canaux dans le même compartiment et analyse de fluorescence C : 2 canaux dans deux compartiments
7. Exécuter le protocole
- Pour traiter un bloc dans une course, supprimez la connexion entre le bloc sélectionné et le bloc Sélectionner un dossier. Placez le bloc recherché au1er rang. Dans le coin supérieur gauche du bloc recherché, cliquez sur le lien directement à droite du dossier. Dans la boîte de dialogue Ouvrir qui s’affiche, double-cliquez sur le dossier contenant les images fusionnées. Ensuite, cliquez sur Ouvrir. Cliquez sur Exécuter pour démarrer le flux de travail. Le traitement peut être arrêté en cliquant sur le bouton Arrêter.
- Pour traiter différents blocs dans une course, conservez les connexions des blocs choisis avec le bloc Sélectionner le dossier (bloc 1). Assurez-vous que leur rang permet le bon traitement du flux de travail. Par exemple, si un bloc spécifique a besoin d’objets segmentés pour traiter, assurez-vous que le bloc de segmentation traite avant. Avant d’exécuter le flux de travail, supprimez les blocs inutilisés et enregistrez le nouveau protocole avec un autre nom.
- Cliquez sur Exécuter pour démarrer le flux de travail. Lorsque la boîte de dialogue ouverte s’affiche, double-cliquez sur le dossier contenant les images fusionnées. Ensuite, cliquez sur Ouvrir. Le flux de travail s’exécute automatiquement. Si nécessaire, arrêtez le traitement en cliquant sur le bouton Arrêter.
- À la fin du traitement, vérifiez que le message Le flux de travail exécuté avec succès est apparu dans le coin inférieur droit et que tous les blocs sont marqués avec un signe vert ( Figure2b). Si ce n’est pas le cas, le bloc et la boîte intérieure présentant le signe d’erreur indiquent l’élément à corriger (Figure 2b).
REMARQUE : Une fois le flux de travail exécuté avec succès, une nouvelle exécution ne peut pas être démarrée directement et pour traiter à nouveau le flux de travail, au moins un bloc doit être marqué avec le signe « prêt à traiter ». Pour modifier l’état d’un bloc, supprimez et créez à nouveau un lien entre deux cases à l’intérieur de ce bloc, soit fermez et réal ouverts le protocole. Si une erreur se produit pendant le traitement, une nouvelle exécution peut être démarrée directement. Au cours d’une nouvelle course, tous les blocs du pipeline sont traités, même si certains d’entre eux sont signalés avec le signe vert.
Résultats
Toutes les analyses décrites ont été effectuées sur un ordinateur portable standard (processeur quad-core 64 bits à 2,80 GHz avec mémoire d’accès aléatoire (RAM) de 16 Go fonctionnant avec la version 64 bits de Java. La mémoire à accès aléatoire est un paramètre important à considérer, en fonction de la quantité et de la résolution des images à analyser. L’utilisation de la version 32 bits de Java limite la mémoire à environ 1300 Mo, ce qui pourrait être inapproprié pour l’analyse big data, ta...
Discussion
Un nombre croissant d’outils logiciels libres sont disponibles pour l’analyse des images des cellules de fluorescence. Les utilisateurs doivent choisir correctement le logiciel adéquat en fonction de la complexité de leur problématique, de leurs connaissances dans le traitement d’image, et au temps qu’ils veulent passer dans leur analyse. Icy, CellProfiler, ou ImageJ / Fidji sont des outils puissants combinant à la fois la facilité d’utilisation et la fonctionnalité3. Icy est un ou...
Déclarations de divulgation
Les auteurs n’ont rien à divulguer.
Remerciements
G.H. a reçu une bourse d’études supérieures du Ministère délégué à la Recherche et aux Technologies. L.H. a reçu une bourse d’études supérieures de l’Institut de Cédrologie de Lorraine (ICL), tandis que Q.T. a bénéficié d’une subvention publique supervisée par l’Agence nationale de la recherche Français (ANR) dans le cadre du deuxième programme « Investissements d’Avenir » FIGHT-HF (référence : ANR-15-RHU4570004). Ces travaux ont été financés par le CNRS et l’Université de Lorraine (UMR 7365).
matériels
| Name | Company | Catalog Number | Comments |
| 16% Formaldehyde solution (w/v) methanol free | Thermo Fisher Scientific | 28908 | to fix the cells |
| Alexa Fluor 488 of goat anti-rabbit | Thermo Fisher Scientific | A-11008 | fluorescent secondary antibody |
| Alexa Fluor 555 of goat anti-mouse | Thermo Fisher Scientific | A-21425 | fluorescent secondary antibody |
| Alexa Fluor 555 Phalloidin | Thermo Fisher Scientific | A34055 | fluorescent secondary antibody |
| Bovine serum albumin standard (BSA) | euromedex | 04-100-812-E | |
| DMEM | Sigma-Aldrich | D5796-500ml | cell culture medium |
| Duolink In Situ Mounting Medium with DAPI | Sigma-Aldrich | DUO82040-5ML | mounting medium |
| Human: HeLa S3 cells | IGBMC, Strasbourg, France | cell line used to perform the experiments | |
| Hydrogen peroxide solution 30% (H2O2) | Sigma-Aldrich | H1009-100ml | used as a stressing agent |
| Lipofectamine 2000 Reagent | Thermo Fisher Scientific | 11668-019 | transfection reagent |
| Mouse monoclonal anti-coilin | abcam | ab11822 | Coilin-specific antibody |
| Nikon Optiphot-2 fluorescence microscope | Nikon | epifluoresecence microscope | |
| Opti-MEM I Reduced Serum Medium | Thermo Fisher Scientific | 31985062 | transfection medium |
| PBS pH 7.4 (10x) | gibco | 70011-036 | to wash the cells |
| Rabbit polyclonal anti-53BP1 | Thermo Fisher Scientific | PA1-16565 | 53BP1-specific antibody |
| Rabbit polyclonal anti-EDC4 | Sigma-Aldrich | SAB4200114 | EDC4-specific antibody |
| Triton X-100 | Roth | 6683 | to permeabilize the cells |
Références
- Möckl, L., Lamb, D. C., Bräuchle, C. Super-resolved fluorescence microscopy: Nobel Prize in Chemistry 2014 for Eric Betzig, Stefan Hell, and William E. Moerner. Angewandte Chemie. 53 (51), 13972-13977 (2014).
- Meijering, E., Carpenter, A. E., Peng, H., Hamprecht, F. A., Olivo-Marin, J. -. C. Imagining the future of bioimage analysis. Nature Biotechnology. 34 (12), 1250-1255 (2016).
- Wiesmann, V., Franz, D., Held, C., Münzenmayer, C., Palmisano, R., Wittenberg, T. Review of free software tools for image analysis of fluorescence cell micrographs. Journal of Microscopy. 257 (1), 39-53 (2015).
- de Chaumont, F., et al. Icy: an open bioimage informatics platform for extended reproducible research. Nature Methods. 9 (7), 690-696 (2012).
- Girish, V., Vijayalakshmi, A. Affordable image analysis using NIH Image/ImageJ. Indian J Cancer. 41 (1), 47 (2004).
- Zaitoun, N. M., Aqel, M. J. Survey on image segmentation techniques. Procedia Computer Science. 65, 797-806 (2015).
- . MetaMorph Microscopy Automation and Image Analysis Software Available from: https://www.moleculardevices.com/products/cellular-imaging-systems/acquisition-and-analysis-software/metamorph-microscopy (2018)
- . NIS-Elements Imaging Software Available from: https://www.nikon.com/products/microscope-solutions/lineup/img_soft/nis-element (2014)
- Meyer, F., Beucher, S. Morphological segmentation. Journal of Visual Communication and Image Representation. 1 (1), 21-46 (1990).
- Schieber, M., Chandel, N. S. ROS Function in Redox Signaling and Oxidative Stress. Current Biology. 24 (10), 453-462 (2014).
- D'Autréaux, B., Toledano, M. B. ROS as signalling molecules: mechanisms that generate specificity in ROS homeostasis. Nature Reviews. Molecular Cell Biology. 8 (10), 813-824 (2007).
- Davalli, P., Mitic, T., Caporali, A., Lauriola, A., D'Arca, D. ROS, Cell Senescence, and Novel Molecular Mechanisms in Aging and Age-Related Diseases. Oxidative Medicine and Cellular Longevity. 2016, 3565127 (2016).
- Disher, K., Skandalis, A. Evidence of the modulation of mRNA splicing fidelity in humans by oxidative stress and p53. Genome. 50 (10), 946-953 (2007).
- Takeo, K., et al. Oxidative stress-induced alternative splicing of transformer 2β (SFRS10) and CD44 pre-mRNAs in gastric epithelial cells. American Journal of Physiology - Cell Physiology. 297 (2), 330-338 (2009).
- Seo, J., et al. Oxidative Stress Triggers Body-Wide Skipping of Multiple Exons of the Spinal Muscular Atrophy Gene. PLOS ONE. 11 (4), 0154390 (2016).
- Will, C. L., Luhrmann, R. Spliceosome Structure and Function. Cold Spring Harbor Perspectives in Biology. 3 (7), 003707 (2011).
- Ljosa, V., Sokolnicki, K. L., Carpenter, A. E. Annotated high-throughput microscopy image sets for validation. Nature Methods. 9 (7), 637-637 (2012).
- Wang, Q., et al. Cajal bodies are linked to genome conformation. Nature Communications. 7, (2016).
- Carpenter, A. E., et al. CellProfiler: image analysis software for identifying and quantifying cell phenotypes. Genome Biology. 7, 100 (2006).
- McQuin, C., et al. CellProfiler 3.0: Next-generation image processing for biology. PLoS Biology. 16 (7), 2005970 (2018).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon