É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Methods Article
Analisador de subestrutura: Um fluxo de trabalho fácil de usar para exploração rápida e análise precisa de corpos celulares em imagens de microscopia de fluorescência
Neste Artigo
Resumo
Apresentamos um fluxo de trabalho livremente disponível construído para rápida exploração e análise precisa de corpos celulares em compartimentos celulares específicos em imagens de microscopia de fluorescência. Este fluxo de trabalho fácil de usar é projetado no software de código aberto Icy e também usa funcionalidades ImageJ. O gasoduto é acessível sem conhecimento em análise de imagens.
Resumo
A última década tem sido caracterizada por avanços em técnicas de microscopia de fluorescência ilustradas pela melhoria da resolução espacial, mas também em imagens de células vivas e técnicas de microscopia de alto rendimento. Isso levou a um aumento constante na quantidade e complexidade dos dados de microscopia para um único experimento. Como a análise manual dos dados de microscopia é muito demorada, subjetiva e proíbe análises quantitativas, a automação da análise de bioimagem está se tornando quase inevitável. Construímos um fluxo de trabalho de informática chamado Substructure Analyzer para automatizar totalmente a análise de sinais em bioimagems de microscopia fluorescente. Este fluxo de trabalho é desenvolvido na plataforma de código aberto Icy e é concluído por funcionalidades do ImageJ. Inclui o pré-processamento de imagens para melhorar a relação sinal/ruído, a segmentação individual das células (detecção de limites celulares) e a detecção/quantificação de corpos celulares enriquecidos em compartimentos celulares específicos. A principal vantagem deste fluxo de trabalho é propor funcionalidades complexas de bioimagem aos usuários sem experiência em análise de imagem por meio de uma interface fácil de usar. Além disso, é altamente modular e adaptado a várias questões desde a caracterização da translocação nuclear/citoplasmática até a análise comparativa de diferentes corpos celulares em diferentes subescobrições celulares. A funcionalidade deste fluxo de trabalho é ilustrada através do estudo dos Corpos de Cajal (enrolados) sob condições de estresse oxidativo (OS). Dados da microscopia de fluorescência mostram que sua integridade em células humanas é impactada poucas horas após a indução do SO. Este efeito é caracterizado pela diminuição da nucleação de coilina em corpos característicos de Cajal, associados a uma redistribuição nucleoplasmática de coilina em um número aumentado de focos menores. O papel central da coilina na troca entre os componentes do CB e o nucleoplasma circundante sugere que a redistribuição induzida pelo SO do coilin poderia afetar a composição e a funcionalidade dos Corpos de Cajal.
Introdução
Microscopia leve e, mais particularmente, microscopia de fluorescência são técnicas robustas e versáteis comumente utilizadas em ciências biológicas. Eles dão acesso à localização precisa de várias biomoléculas como proteínas ou RNA através de sua rotulagem fluorescente específica. A última década foi caracterizada por rápidos avanços em tecnologias de microscopia e imagem, evidenciados pelo Prêmio Nobel de Química de 2014, premiando Eric Betzig, Stefan W. Hell e William E. Moerner pelo desenvolvimento de microscopia de fluorescência super resolvida (SRFM)1. O SFRM contorna o limite de difração da microscopia óptica tradicional para trazê-lo para a nanodimensional. A melhoria de técnicas como imagens ao vivo ou abordagens de triagem de alto rendimento também aumenta a quantidade e a complexidade dos dados para tratar para cada experimento. Na maioria das vezes, os pesquisadores se deparam com altas populações heterogêneas de células e querem analisar fenótipos no nível unicelular.
Inicialmente, análises como a contagem de focos foram realizadas por olho, o que é preferido por alguns pesquisadores, uma vez que fornece controle visual total sobre o processo de contagem. No entanto, a análise manual desses dados é muito demorada, leva à variabilidade entre observadores e não dá acesso a recursos mais complexos para que abordagens assistidas por computador estejam se tornando amplamente utilizadas e quase inevitáveis2. Os métodos de informática da bioimagem aumentam substancialmente a eficiência da análise de dados e estão livres da subjetividade inevitável do operador e do viés potencial da análise de contagem manual. O aumento da demanda neste campo e a melhoria do poder computacional levaram ao desenvolvimento de um grande número de plataformas de análise de imagens. Alguns deles estão disponíveis gratuitamente e dão acesso a várias ferramentas para realizar análises com computadores pessoais. Uma classificação de ferramentas de acesso aberto foi recentemente estabelecida3 e apresenta o Icy4 como um software poderoso que combina usabilidade e funcionalidade. Além disso, Icy tem a vantagem de se comunicar com ImageJ.
Para usuários sem experiência em análise de imagem, os principais obstáculos são escolher a ferramenta apropriada de acordo com os parâmetros problemáticos e de sintonia correta que muitas vezes não são bem compreendidos. Além disso, os tempos de configuração são muitas vezes longos. A Icy propõe uma interface de ponto e clique amigável chamada "Protocolos" para desenvolver fluxo de trabalho, combinando alguns plugins encontrados dentro de uma coleção exaustiva4. O design modular flexível e a interface point-and-click tornam a configuração uma análise viável para não-programadores. Aqui apresentamos um fluxo de trabalho chamado Substructure Analyzer,desenvolvido na interface de Icy, cuja função é analisar sinais fluorescentes em compartimentos celulares específicos e medir diferentes características como brilho, número de focos, tamanho de focos e distribuição espacial. Este fluxo de trabalho aborda várias questões como quantificação da translocação de sinal, análise de células transfeminadas expressando um repórter fluorescente ou análise de focos de diferentes subestruturas celulares em células individuais. Ele permite o processamento simultâneo de várias imagens e os resultados de saída são exportados para uma planilha delimitada por guias que podem ser abertas em programas de planilha comumente usados.
O gasoduto Analisador de Subestrutura é apresentado na Figura 1. Primeiro, todas as imagens contidas em uma pasta especificada são pré-processadas para melhorar sua relação sinal/ruído. Esta etapa aumenta a eficiência das etapas seguintes e diminui o tempo de execução. Em seguida, são identificadas e segmentadas as Regiões de Interesse (ROIs), correspondentes às áreas de imagem onde o sinal fluorescente deve ser detectado, são identificadas e segmentadas. Finalmente, o sinal fluorescente é analisado e os resultados são exportados para uma planilha delimitada por guias.
A segmentação de objetos (detecção de limites) é o passo mais desafiador na análise de imagens, e sua eficiência determina a precisão das medições celulares resultantes. Os primeiros objetos identificados em uma imagem (chamados objetos primários) são frequentemente núcleos de imagens manchadas de DNA (coloração DAPI ou Hoechst), embora objetos primários também possam ser células inteiras, contas, manchas, tumores ou quaisquer objetos manchados. Na maioria das imagens biológicas, células ou núcleos se tocam ou se sobrepõem fazendo com que os algoritmos simples e rápidos falhem. Até o momento, nenhum algoritmo universal pode realizar a segmentação perfeita de todos os objetos, principalmente porque suas características (tamanho, forma ou textura) modulam a eficiência da segmentação5. As ferramentas de segmentação comumente distribuídas com software de microscopia (como o MetaMorph Imaging Software by Molecular Devices6, ou o NIS-Elements Advances Research software by Nikon7) são geralmente baseadas em técnicas padrão, como correspondência de correlação, limiar ou operações morfológicas. Embora eficientes em sistemas básicos, esses métodos supergeralizados rapidamente apresentam limitações quando utilizados em contextos mais desafiadores e específicos. De fato, a segmentação é altamente sensível a parâmetros experimentais, como tipo de célula, densidade celular ou biomarcadores, e frequentemente requer ajuste repetido para um grande conjunto de dados. O fluxo de trabalho do Analisador de Subestrutura integra algoritmos simples e mais sofisticados para propor diferentes alternativas adaptadas à complexidade da imagem e às necessidades do usuário. Ele propõe notavelmente o algoritmo de bacia hidrográfica baseado em marcador8 para objetos altamente agrupados. A eficiência deste método de segmentação depende da seleção de marcadores individuais em cada objeto. Esses marcadores são escolhidos manualmente na maior parte do tempo para obter parâmetros corretos para a segmentação completa, o que é altamente demorado quando os usuários enfrentam um alto número de objetos. O Analisador de Subestrutura propõe uma detecção automática desses marcadores, proporcionando um processo de segmentação altamente eficiente. A segmentação é, na maioria das vezes, a etapa limitante da análise de imagens e pode modificar consideravelmente o tempo de processamento dependendo da resolução da imagem, do número de objetos por imagem e do nível de agrupamento de objetos. Os dutos típicos requerem alguns segundos a 5 minutos por imagem em um computador de mesa padrão. A análise de imagens mais complexas pode exigir um computador mais poderoso e alguns conhecimentos básicos em análise de imagens.
A flexibilidade e funcionalidade deste fluxo de trabalho são ilustradas com vários exemplos nos resultados representativos. As vantagens deste fluxo de trabalho são notavelmente demonstradas através do estudo de subestruturas nucleares sob condições de estresse oxidativo (OS). O OS corresponde a um desequilíbrio da homeostase redox em favor dos oxidantes e está associado a altos níveis de espécies reativas de oxigênio (ROS). Uma vez que a ROS atua como moléculas de sinalização, as mudanças em sua concentração e localização subcelular afetam positiva ou negativamente uma miríade de vias e redes que regulam funções fisiológicas, incluindo transdução de sinais, mecanismos de reparação, expressão genética, morte celular e proliferação9,,10. Assim, a OS está diretamente envolvida em diversas patologias (doenças neurodegenerativas e cardiovasculares, cânceres, diabetes, etc.), mas também no envelhecimento celular. Portanto, decifrar as consequências da OS na organização e função da célula humana constitui um passo crucial na compreensão dos papéis da OS no início e desenvolvimento das patologias humanas. Foi estabelecido que o OS regula a expressão genética através da modulação da transcrição através de vários fatores de transcrição (p53, Nrf2, FOXO3A)11, mas também afetando a regulação de vários processos co e pós-transcrição, como o emenda alternativa (AS) das pré-RNAs12,,13,,14. O splicing alternativo de codificação primária e transcrições não codificadas é um mecanismo essencial que aumenta a capacidade de codificação dos genomas produzindo isoformas de transcrição. O AS é realizado por um enorme complexo de ribonucleoproteína chamado emenda ao esfarmato, contendo quase 300 proteínas e 5 pequenas RNAs nucleares (UsnRNAs)15. A montagem de spliceosome e as são fortemente controladas em células e alguns passos da maturação de emendas ocorrem dentro de compartimentos nucleares sem membrana chamados Corpos de Cajal. Essas subestruturas nucleares são caracterizadas pela natureza dinâmica de sua estrutura e sua composição, que são conduzidas principalmente por interações multivalentes de seus componentes de RNA e proteína com a proteína coilina. A análise de milhares de células com o fluxo de trabalho do Analisador de Subestrutura permitiu a caracterização de efeitos nunca descritos do SO em Corpos de Cajal. De fato, dados obtidos sugerem que o OS modifica a nucleação dos Corpos de Cajal, induzindo uma redistribuição nucleoplasmática da proteína coilin em numerosos focos nucleares menores. Tal mudança na estrutura dos Corpos de Cajal pode afetar o amadurecimento do emenda e participar da modulação de AS pela OS.
Protocolo
NOTA: Tutoriais fáceis de usar estão disponíveis no site da Icy http://icy.bioimageanalysis.org.
1. Baixe O Gelo e o protocolo Analisador de Subestrutura
- Baixe Icy no site da Icy(http://icy.bioimageanalysis.org/download) e baixe o protocolo Substructure Analyzer: http://icy.bioimageanalysis.org/protocols?sort=latest.
NOTA: Se usar um SO de 64 bits, certifique-se de usar a versão de 64 bits do Java. Esta versão permite aumentar a memória alocada ao Gelo (Preferências | Geral | Memória máxima).
2. Abertura do protocolo
- Abra o Gelo e clique em Ferramentas no menu Fita.
- Clique em Protocolos para abrir a interface do Editor de Protocolos.
- Clique em Carregar e abra o protocolo Subestrutura Analisador. O carregamento do protocolo pode levar alguns segundos. Certifique-se de que a abertura do protocolo está completa antes de usá-lo.
NOTA: O fluxo de trabalho é composto por 13 blocos gerais apresentados na Figura 2a. Cada bloco funciona como um pipeline composto por várias caixas executando subtarefas específicas.
3. Interagindo com o fluxo de trabalho em Icy
NOTA: Cada bloco ou caixa é numerado e tem uma classificação específica dentro do fluxo de trabalho (Figura 2b). Ao clicar neste número, a posição mais próxima possível da primeira é atribuída ao bloco/caixa selecionado, em seguida, a posição dos outros blocos/caixas é reorganizada. Respeite a ordem certa dos blocos ao preparar o fluxo de trabalho. Por exemplo, o bloco Spot Detector precisa de ROIs pré-definidos para que os blocos de segmentação tenham que ser executados antes dos blocos do Detector de Manchas. Não modifique a posição das caixas. Não use "." no nome da imagem.
- Clicando no ícone do canto superior esquerdo, desabar, expandir, ampliar, estreitar ou remover o bloco(Figura 2b).
- Cada pipeline do fluxo de trabalho é caracterizado por uma rede de caixas conectadas através de sua entrada e saída(Figura 2b). Para criar uma conexão, clique em Saída e mantenha até que o cursor atinja uma entrada. As conexões podem ser removidas clicando na tag Saída.
4. Fusão dos canais de uma imagem
- Use o bloco Mesclar canais para gerar imagens mescladas. Se necessário, renomeie os arquivos para que as sequências a serem mescladas tenham o prefixo do mesmo nome seguido por um separador distinto. Por exemplo, sequências de canais individuais de uma imagem A são nomeadas: ImageA_red, ImageA_blue.
NOTA: Para o separador, não utilize caracteres já presentes no nome da imagem. - Na mesma pasta, crie uma nova pasta por canal para mesclar. Por exemplo, para mesclar canais vermelhos, verdes e azuis, crie 3 pastas e armazene as sequências correspondentes nessas pastas.
- Use apenas os canais de mesclagemdo bloco, remova os outros blocos e salve o protocolo como Canais de Mesclagem.
- Acesse as caixas para definir parâmetros. Para cada canal, preencha as caixas Canal X (caixas 1, 5 ou 9), pasta do canal número X (caixas 2, 6 ou 10), separador número X (caixas 3, 7 ou 11) e canal Colormap nb X (caixas 4, 8 e 12) respectivamente.
NOTA: Estas caixas são agrupadas horizontalmente por quatro, cada linha correspondente ao mesmo canal. Em cada linha, um display também está disponível (caixas 23, 24 ou 25) para visualizar diretamente a sequência do canal correspondente.- Na caixa Canal X,escolha qual canal extrair (em imagens RGB clássicas, 0=Vermelho, 1=Verde, 2=Azul). O usuário acessa rapidamente os diferentes canais de uma imagem dentro da janela Inspetor de Gelo, na guia Sequência. Escreva o menor valor do canal na linha superior e o mais alto na linha de fundo.
- Na caixa Pasta do canal X,escreva o \Nome da pasta contendo imagens do canal X.
- Na caixa Separador do canal X,escreva o separador usado para o nome da imagem (no exemplo anterior: "_red", "_green" e "_blue").
- Na caixa Colormap canal nb X,indique com um número qual modelo colormap usar para visualizar o canal correspondente em Gelo. As imagens coloridas disponíveis são visíveis na guia Sequência da janela Inspetor.
- Na caixa Formato de imagens mescladas (caixa 28), escreva a extensão para salvar imagens mescladas: .tif, .gif, .jpg, .bmp ou .png.
NOTA: Para mesclar apenas 2 canais, não preencha as quatro caixas correspondentes ao terceiro canal.
- No canto superior esquerdo do bloco Canais de Mesclagem, clique no link diretamente à direita da Pasta. Na caixa de diálogo Abrir que aparece, clique duas vezes na pasta contendo sequências do primeiro canal que foi definido na caixa Pasta do canal número 1 (caixa 2). Em seguida, clique em Abrir.
- Execute o protocolo clicando na seta preta no canto superior esquerdo do bloco Canais de Mesclagem (consulte a parte 7 para obter mais detalhes). As imagens mescladas são salvas em uma pasta 'Mescla' no mesmo diretório das pastas de canais individuais.
- Acesse as caixas para definir parâmetros. Para cada canal, preencha as caixas Canal X (caixas 1, 5 ou 9), pasta do canal número X (caixas 2, 6 ou 10), separador número X (caixas 3, 7 ou 11) e canal Colormap nb X (caixas 4, 8 e 12) respectivamente.
5. Segmentação das regiões de interesse
NOTA: O Analisador de Subestrutura integra algoritmos simples e mais sofisticados para propor diferentes alternativas adaptadas à complexidade da imagem e às necessidades do usuário.
- Selecione o bloco adaptado.
- Se os objetos não se tocarem, ou o usuário não precisar diferenciar objetos agrupados individualmente, use o bloco Segmentação A: Objetos não agrupados.
- Quando os objetos não se tocam, mas alguns deles estão próximos, use o bloco Segmentação B: Objetos mal agrupados.
- Para objetos com alto nível de agrupamento e forma convexa, use o bloco Segmentação C: Objetos agrupados com formas convexas.
- Se os objetos apresentarem um alto nível de agrupamento e tiverem formas irregulares, use o bloco Segmentação D: Objetos agrupados com formas irregulares.
- Use o bloco Segmentação E: Citoplasma agrupado para segmentar citoplasmas tocantes individualmente usando núcleos segmentados como marcadores. Este bloco precisa imperativamente de núcleos segmentados para processar.
NOTA: Blocos adaptados para o processo de segmentação de objetos primários de forma independente para que vários blocos possam ser usados na mesma execução para comparar sua eficiência com uma subestrutura específica ou para segmentar diferentes tipos de subestruturas. Se o nível de agrupamento for heterogêneo dentro do mesmo conjunto de imagens, então processe objetos pequenos e altamente agrupados separadamente nos blocos adaptados.
- Vincule a saída0 (Arquivo) do bloco Selecione pasta à entrada da pasta do bloco de segmentação escolhido.
- Definir parâmetros do bloco de segmentação escolhido.
- Segmentação A: Objetos não agrupados e Segmentação C: Objetos agrupados com formas convexas
- Na caixa Sinal do canal (caixa 1), defina o canal dos objetos para segmentar.
- Como opção, na caixa filtro gaussiano (caixa 2), aumente os valores X e Y sigma se o sinal dentro dos objetos for heterogêneo. O filtro gaussiano suaviza texturas para obter regiões mais uniformes e aumenta a velocidade e eficiência da segmentação dos núcleos. Quanto menor os objetos, menor é o valor sigma. Evite valores sigma altos. Defina os valores padrão para 0.
- Na caixa HK-Means (caixa 3), defina o parâmetro de classes de intensidade e os tamanhos mínimos e máximos aproximados (em pixels) dos objetos a serem detectados.
NOTA: Para classes de intensidade, um valor de 2 classifica pixels em 2 classes: fundo e primeiro plano. Assim, é adaptado quando o contraste entre os objetos e o fundo é alto. Se os objetos em primeiro plano tiverem intensidades diferentes ou se o contraste com o fundo for baixo, aumente o número de classes. A configuração padrão é 2. O tamanho do objeto pode ser rapidamente avaliado desenhando um ROI manualmente em torno do objeto de interesse. O tamanho do ROI (Interior em pixels) aparece diretamente na imagem ao apontá-lo com o cursor ou pode ser acessado na janela de estatísticas do ROI (abra-a da barra de pesquisa). Parâmetros ideais detectam cada objeto em primeiro plano em um único ROI. Eles podem ser definidos manualmente em Gelo(Detecção e Rastreamento | HK-Means). - Na caixa Contornos Ativos (caixa 4), otimize a detecção de bordas de objeto. A documentação exaustiva para este plugin está disponível online: http://icy.bioimageanalysis.org/plugin/Active_Contours. Parâmetros corretos também podem ser definidos manualmente em Gelo (Detecção e Rastreamento | Contornos Ativos).
- Durante o processo, uma pasta é criada automaticamente para salvar imagens de objetos segmentados. Na caixa Texto (caixa 6), nomeie esta pasta (ex: Núcleos segmentados). Para definir o formato para salvar imagens de objetos segmentados (Tiff, Gif, Jpeg, BMP, PNG), preencha o formato de caixa de imagens de objetos segmentados. A pasta é criada na pasta contendo imagens mescladas.
- Execute o fluxo de trabalho (para mais detalhes, consulte a parte 7).
- Segmentação B: Objetos mal agrupados
- Siga os mesmos passos do 5.3.1 para definir parâmetros de caixas Sinal de canal, HK-Means, Contornos Ativos, Extensão para salvar objetos segmentados e Texto (As fileiras das caixas não são as mesmas da etapa 5.3.1).
- Na caixa Ligue para o plugin IJ (caixa 4), defina o parâmetro Rolamento para controlar a subtração de fundo. Defina este parâmetro para pelo menos o tamanho do maior objeto que não faz parte do fundo. A diminuição desse valor aumenta a remoção de fundo, mas também pode induzir a perda de sinal em primeiro plano.
- Na caixa Equalização de histograma adaptativo (caixa 6), melhore os contrastes entre os objetos do primeiro plano e o fundo. Aumentar a inclinação dá sequências mais contrastadas.
- Execute o fluxo de trabalho (para mais detalhes, consulte a parte 7).
- Segmentação D: Objetos agrupados com formas irregulares
NOTA: Três métodos diferentes de segmentação se aplicam a cada imagem: em primeiro lugar,Agrupamento de meios HKcombinado com oMétodo de contornos ativosé aplicado. Então, oalgoritmo divisor de águas clássico(usando o mapa de distância euclidiano) é aplicado a objetos segmentados anteriormente. Finalmente, umalgoritmo de bacia hidrográfica baseado em marcadoré usado. Apenas os métodos de bacia hidrográfica baseados em HK e marcadores precisam de intervenção do usuário. Para ambos os métodos, os mesmos parâmetros podem ser aplicados para todas as imagens (versão totalmente automatizada) ou ser alterados para cada imagem (versão semi-automatizada). Se o usuário não for treinado nesses métodos de segmentação, o processamento semi-automatizado é altamente recomendado. Durante o processamento deste bloco, é necessária intervenção manual. Quando um método de segmentação é concluído, o usuário deve remover manualmente objetos segmentados incorretamente antes do início do próximo método de segmentação. Objetos segmentados com sucesso são salvos e não considerados na próxima etapa(s). Este bloco tem que ser conectado com o blocoAgrupado/heterogêneo formas de segmentação de objetos primários Caixa de diálogopara trabalhar corretamente.- Baixe a coleção ImageJ MorphoLibJ em https://github.com/ijpb/MorphoLibJ/releases. A versão MorphoLibJ 1.4.0 é usada neste protocolo. Coloque o arquivo MorphoLibJ_-1.4.0.jar na pasta icy/ij/plugins. Mais informações sobre o conteúdo desta coleção estão disponíveis na https://imagej.net/MorphoLibJ.
- Siga os mesmos passos da etapa 5.3.1 para definir parâmetros de caixas Sinal de canal, filtro gaussiano, Contornos Ativos, Extensão para salvar objetos segmentados e Texto. Active Contours As caixas não são as mesmas da etapa 5.3.1.
- Definir parâmetros da caixa Equalização do histograma adaptativo (ver passo 5.3.2.3).
- Para ativar o fundo subtraído,escreva sim em Aplicar fundo subtraído? (caixa 5). Senão, escreva "Não". Se o plugin estiver ativado, defina o parâmetro de rolamento (ver o passo 5.3.2), na caixa Subtrair parâmetro de fundo (caixa 7).
- Automatização de hk-significa: Para aplicar os mesmos parâmetros para todas as imagens (processamento totalmente automatizado), defina o Nb de classes (caixa 11), o tamanho mínimo (caixa 12) e o tamanho máximo (box13) (ver etapa 5.3.1). Esses parâmetros devem ser definidos para selecionar um máximo de pixels em primeiro plano e otimizar a individualização de objetos em primeiro plano. Para a versão de processamento semi-automatizada, não é necessária nenhuma intervenção.
- Automatização de extrações de marcadores: para a versão totalmente automatizada, expanda a caixa Extração de Marcadores Internos (caixa 27) e defina o valor do parâmetro "dinâmico" na linha 13 do script. Para a versão de processamento semi-automatizada, não é necessária nenhuma intervenção.
NOTA: Os marcadores são extraídos aplicando uma transformação de minima estendida em uma imagem de entrada controlada por um parâmetro "dinâmico". No algoritmo da bacia hidrográfica baseado em marcadores, a inundação desses marcadores é simulada para realizar a segmentação de objetos. Para a segmentação bem sucedida de objetos em primeiro plano, um único marcador por objeto em primeiro plano deve ser extraído. A configuração do parâmetro "dinâmico" para extração de marcadores ideais depende principalmente da resolução de imagens. Assim, se não estiver familiarizado com este parâmetro, use a versão semi-automatizada. - Execute o fluxo de trabalho (para mais detalhes, consulte a parte 7).
- No início do processamento, as caixas de diálogo HK-means parâmetros e a bacia hidrográfica baseada em marcadores são abertas sucessivamente. Para aplicar os mesmos parâmetros para todas as imagens (versão totalmente automatizada), clique em YES. Caso contrário, clique em NO. Uma caixa de informações é aberta, pedindo para "Determinar ROIs ideais com plugin HK-Means e imagem fechada". Clique em OK e aplique manualmente o plugin HK-Means (Detecção e Rastreamento| HK-Means) na imagem, que se abre automaticamente. Selecione a opção ROIs de exportação na caixa de plugin HK-Means. Aplique os melhores parâmetros para ter ROIs contendo um máximo de pixels em primeiro plano e para otimizar a individualização de objetos em primeiro plano. Quando forem encontrados ROIs ideais, feche diretamente a imagem.
- No final do primeiro método de segmentação, uma caixa de informações é aberta e pede para "Remover ROIs indesejados e fechar imagem". Esses ROIs correspondem às bordas dos objetos segmentados. Selecione OK e remova ROIs de objetos segmentados errados na imagem, que abre automaticamente. Um ROI pode ser facilmente removido colocando o cursor em sua borda e usando o botão "Excluir" do teclado. Feche a imagem. Repita o mesmo procedimento após a conclusão da segunda etapa de segmentação.
- Nesta fase, se o botão YES for selecionado para a automatização completa do algoritmo de bacia hidrográfica baseado em marcadores, os parâmetros previamente definidos serão aplicados a todas as imagens.
- Se o botão NO for selecionado, uma caixa de informações será aberta, pedindo para "Determinar e ajustar marcadores internos". Clique em OK e dentro da interface ImageJ do Icy, vá para Plugins | MorphoLibJ | Minima e Maxima| Min & Max estendido. Em Operação, selecione Minima Estendida.
- Selecione Visualizar para visualizar previamente a imagem aberta automaticamente, o resultado da transformação. Mova a dinâmica até que os marcadores ideais sejam observados. Marcadores são grupos de pixels com um valor de 255 (não necessariamente pixels brancos). Os parâmetros ideais levam a um marcador por objeto. Concentre-se nos objetos remanescentes que não foram bem segmentados com os dois métodos de segmentação anteriores.
- Se necessário, melhore os marcadores aplicando operações morfológicas adicionais como "Abertura" ou "Fechamento"(Plugins | MorphoLibJ | Filtros Morfológicos). Ao obter a imagem final dos marcadores, mantenha-a aberta e feche todas as outras imagens terminando com a imagem inicialmente usada como entrada para a operação Minima Estendida. Clique em Não se uma caixa ImageJ pedir para salvar alterações nesta imagem.
- Na caixa Nb de imagens com Caixa de Informações (caixa 14), determine quantas imagens com caixas de informação devem aparecer.
- Segmentação E: Citoplasma agrupado
NOTA: Este bloco usa núcleos segmentados anteriormente como marcadores individuais para iniciar a segmentação do citoplasma. Certifique-se de que o bloco de segmentação de núcleos tenha sido processado antes de usá-lo.- Na caixa Canal cytoplasm (caixa 1), defina o canal do sinal citoplasmizado.
- Na caixa Extensão de núcleos segmentados (caixa 2), escreva o formato utilizado para salvar imagens de núcleos segmentados (tif, jpeg, bmp, png). O formato padrão é tif.
- Na caixa Texto (caixa3), escreva o \Nome da pasta contendo núcleos segmentados.
- Na caixa Formato de imagens de citoplasmas segmentados (caixa 4), defina o formato para ser usado para salvar imagens segmentadas (Tiff, Gif, Jpeg, BMP, PNG).
- Durante o processo, uma pasta é criada automaticamente para salvar imagens de citoplasmas segmentados. Na caixa Texto (caixa 5), nomeie esta pasta (ex: citoplasmas segmentados). A pasta é criada na pasta contendo imagens mescladas.
- Siga os mesmos passos da etapa 5.3.1 para definir parâmetros de caixas filtro gaussiano e Contornos Ativos (Tenha cuidado, as classificações da caixa não são as mesmas da etapa 5.3.1).
- Execute o fluxo de trabalho (para mais detalhes, consulte a parte 7).
- Segmentação A: Objetos não agrupados e Segmentação C: Objetos agrupados com formas convexas
6. Detecção e análise de sinal fluorescente
- Selecione o bloco adaptado.
- No bloco Análise de Fluorescência A: 1 Canal,realize a detecção e análise de focos em um canal dentro de um tipo de objeto segmentado: detecção de focos de coilin (canal vermelho) dentro do núcleo.
- No bloco Análise de Fluorescência B: 2 Canais no mesmo compartimento,realizam detecção e análise de focos em dois canais dentro de um tipo de objeto segmentado: detecção de coilin (canal vermelho) e 53BP1 (canal verde) focos dentro do núcleo.
- No bloco Análise de Fluorescência C: 2 Canais em dois compartimentos,realizam detecção e análise de focos em um ou dois canais, especificamente dentro dos núcleos e seu citoplasma correspondente: detecção de focos de coilin (canal vermelho) tanto dentro do núcleo quanto de seu citoplasma correspondente ou detecção de focos de coilin (canal vermelho) dentro do núcleo e foci G3BP (canal verde) dentro do citoplasma correspondente.
- No bloco Análise de Fluorescência D: Translocação Global,calcule a porcentagem de sinal de um canal em dois compartimentos celulares (a e b). Por exemplo, em um ensaio de translocação de citoplasma/núcleo, exportar percentuais calculados de sinais nucleares e citoplasmáticos para cada imagem na planilha final de "Resultados". A fórmula usada para calcular a porcentagem de sinal nuclear é mostrada abaixo. Este bloco pode ser usado para qualquer compartimento subcelular:
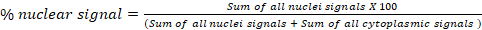
- No bloco Análise de Fluorescência E: Translocação celular individual,calcule a porcentagem de sinal de um canal em dois compartimentos celulares para cada célula. Este bloco é especialmente otimizado para ensaio de translocação de núcleo/citoplasma no nível de célula única.
NOTA: Como é necessária a análise de fluorescência do bloco E: Translocação celular individual realiza análises no nível unicelular, é necessária uma segmentação eficiente do núcleo e do citoplasma.
- Vincule a saída0 (Arquivo) do bloco Selecionar pasta (bloco 1) à entrada da pasta (setas brancas em círculos pretos) do bloco escolhido.
- Defina os parâmetros do bloco escolhido.
- Análise de Fluorescência A: 1 Canal, Análise de Fluorescência B: 2 Canais no mesmo compartimento e Análise de Fluorescência C: 2 Canais em dois compartimentos
- Na caixa Imagens de pasta ROI, escreva o nome da pasta contendo imagens de objetos segmentados precedidos por uma barra traseira. (Por exemplo: \Núcleos segmentados).
- Na caixa Formato de imagens de objetos segmentados (caixa 2), escreva o formato usado para salvar imagens de objetos segmentados (tif, jpeg, bmp, png). O formato padrão é tif.
- Na caixa Kill Borders?, escreva Sim para remover objetos de fronteira. Caso contrário, escreva "Não". A instalação da coleção MorphoLibJ de ImageJ é necessária para usar esta função (ver passo 5.3.3).
- Na caixa(es) Sinal de manchas do canal,defina o canal onde as manchas devem ser detectadas. Em imagens RGB clássicas, 0=Vermelho, 1=Verde e 2=Azul.
- Nas caixas Nome da molécula localizada,escreva o nome da molécula localizado nos pontos. O número de campos para entrar depende do número de moléculas.
- Na caixa(es) Bloco do detector de pontos de onda,defina parâmetros de detecção de ponto para cada canal. Defina a escala (referida para o tamanho da mancha) e a sensibilidade da detecção (uma sensibilidade menor diminui o número de pontos detectados, o valor padrão é 100 e o valor mínimo é 0). A documentação exaustiva deste plugin está disponível online: http://icy.bioimageanalysis.org/plugin/Spot_Detector. Os parâmetros também podem ser definidos manualmente em Gelo(Detecção e Rastreamento | Detector de manchas).
- Como opção, na caixa Filtro ROI por tamanho,filtrar os objetos segmentados onde as manchas são detectadas definindo um intervalo de tamanho (em pixels). Esta etapa é especialmente útil para remover objetos subous ou sobre-segmentados. Para estimar manualmente o tamanho dos objetos, consulte o passo 5.3.1. Os parâmetros padrão não incluem a filtragem de ROIs por tamanho. O bloco 2 Canais em dois compartimentos contém duas caixas: Núcleos de filtro por tamanho (caixa 19) e citoplasma do filtro por tamanho (caixa 46).
- Opcionalmente, na caixa O filtro mancha por tamanho,filtra as manchas detectadas de acordo com seu tamanho (em pixels) para remover artefatos indesejados. Para estimar manualmente o tamanho do local, clique em Detecção e Rastreamento e abra o plugin do detector Spot. Nas opções de saída, selecione Exportar para ROI. Tenha cuidado para que os parâmetros padrão não incluam filtragem de manchas por tamanho, e que as manchas filtradas não são levadas em conta para a análise. O número de campos para entrar depende do número de canais.
- Opcionalmente, na caixa Manchas de filtro,aplique um filtro adicional (contraste, homogeneidade, perímetro, arredondamento) nos pontos detectados. Tenha cuidado para que os parâmetros padrão não incluam filtragem de manchas e que as manchas filtradas não sejam levadas em conta para a análise. O número de campos para entrar depende do número de canais.
- Opcionalmente, nas caixas Limite de tamanho spot, defina um limite para a área (em pixels) de pontos analisados. O número de pontos contados abaixo e acima desse limite é exportado na planilha de resultados finais. O número de caixas a serem informadas depende do número de canais.
- Execute o fluxo de trabalho (para mais detalhes, consulte a parte 7). Os dados são exportados em uma planilha de resultados salva na pasta contendo imagens mescladas.
- Análise de Fluorescência D: Análise global de translocação e fluorescência E: Translocação celular individual:
- Nas caixas Imagens da pasta (caixas 1 e 2), escreva o \Nome da pasta contendo imagens de objetos segmentados. No bloco Análise de Fluorescência D: Translocação Global,os dois tipos de ROI são identificados como ROI a e ROI b. Para o bloco Análise de Fluorescência E: Translocação celular individual,em imagens de pasta segmentadas núcleos e caixas de pasta segmentadas de citoplasmas, escreva o nome da pasta contendo núcleos segmentados e citoplasmas segmentados, respectivamente.
- Na caixa Sinal do canal (caixa 3), digite o canal do sinal.
- Na caixa Formato de imagens de objetos segmentados (caixa 4), escreva o formato usado para salvar imagens de objetos segmentados (tif, jpeg, bmp, png). O formato padrão é tif. A opção Kill Borders também está disponível para remover objetos de borda (ver passo 6.3.1).
- Opcionalmente, nas caixas Filtro ROI por tamanho,filtrar os objetos segmentados definindo um intervalo de tamanho (em pixels). Esta etapa pode ser útil para remover objetos subous ou super-segmentados. Para estimar manualmente o tamanho do objeto, consulte o passo 5.3.1. Há dois campos para entrar, um por canal. Os parâmetros padrão não incluem filtragem de ROI por tamanho.
- Execute o fluxo de trabalho (para mais detalhes, consulte a parte 7). Exportar dados em uma planilha Resultados salvos na pasta contendo imagens mescladas.
- Análise de Fluorescência A: 1 Canal, Análise de Fluorescência B: 2 Canais no mesmo compartimento e Análise de Fluorescência C: 2 Canais em dois compartimentos
7. Execute o protocolo
- Para processar um bloco em uma execução, remova a conexão entre o bloco selecionado e a pasta de seleçãodo bloco . Coloque o bloco procurado na1ª fila. No canto superior esquerdo do bloco procurado, clique no link diretamente para a direita da pasta. Na caixa de diálogo Abrir que aparece, clique duas vezes na pasta contendo as imagens mescladas. Em seguida, clique em Abrir. Clique em Executar para iniciar o fluxo de trabalho. O processamento pode ser interrompido clicando no botão Parar.
- Para processar diferentes blocos em uma execução, mantenha conexões de blocos escolhidos com o bloco Selecionar pasta (bloco 1). Certifique-se de que sua classificação permite o bom processamento do fluxo de trabalho. Por exemplo, se um bloco específico precisar de objetos segmentados para processar, certifique-se de que o bloco de segmentação processe antes. Antes de executar o fluxo de trabalho, remova blocos nãousados e salve o novo protocolo com outro nome.
- Clique em Executar para iniciar o fluxo de trabalho. Quando a caixa de diálogo aberta for exibida, clique duas vezes na pasta que contém as imagens mescladas. Em seguida, clique em Abrir. O fluxo de trabalho é executado automaticamente. Se necessário, pare o processamento clicando no botão Parar.
- No final do processamento, verifique se a mensagem O fluxo de trabalho executado com sucesso apareceu no canto inferior direito e que todos os blocos são sinalizados com um sinal verde(Figura 2b). Caso não, o bloco e a caixa interna que apresenta o sinal de erro indicam o elemento para corrigir(Figura 2b).
NOTA: Após o fluxo de trabalho executado com sucesso, uma nova execução não pode ser iniciada diretamente e, para processar novamente o fluxo de trabalho, pelo menos um bloco deve ser sinalizado com o sinal "pronto para processar". Para alterar o estado de um bloco, exclua e rees crie um link entre duas caixas dentro deste bloco ou simplesmente feche e reassua o protocolo. Se ocorrer um erro durante o processamento, uma nova execução pode ser iniciada diretamente. Durante uma nova corrida, todos os blocos do gasoduto são processados, mesmo que alguns deles sejam sinalizados com o sinal verde.
Resultados
Todas as análises descritas foram realizadas em um laptop padrão (processador quad-core de 64 bits a 2,80 GHz com memória de acesso aleatório (RAM) de 16 GB, trabalhando com a versão de 64 bits do Java. A memória de acesso aleatório é um parâmetro importante a ser considerado, dependendo da quantidade e da resolução das imagens para analisar. O uso da versão de 32 bits do Java limita a memória a cerca de 1300 MB, o que pode ser inadequado para análise de big data, enquanto a versão de 64 bits permite aumen...
Discussão
Um número crescente de ferramentas de software livre estão disponíveis para a análise de imagens celulares de fluorescência. Os usuários devem escolher corretamente o software adequado de acordo com a complexidade de sua problemática, com seus conhecimentos em processamento de imagem e com o tempo que desejam passar em suas análises. Icy, CellProfiler ou ImageJ/Fiji são ferramentas poderosas que combinam usabilidade e funcionalidade3. O Icy é uma ferramenta autônoma que apresenta uma in...
Divulgações
Os autores não têm nada a revelar.
Agradecimentos
G.H. foi apoiado por uma bolsa de pós-graduação da Ministère Délégué à la Recherche et aux Technologies. L.H. foi apoiado por uma bolsa de pós-graduação do Institut de Cancérologie de Lorraine (ICL), enquanto Q.T. foi apoiado por uma bolsa pública supervisionada pela Agência Nacional de Pesquisa Francesa (ANR) como parte do segundo programa "Investissements d'Avenir" FIGHT-HF (referência: ANR-15-RHU4570004). Este trabalho foi financiado pelo CNRS e pela Université de Lorraine (UMR 7365).
Materiais
| Name | Company | Catalog Number | Comments |
| 16% Formaldehyde solution (w/v) methanol free | Thermo Fisher Scientific | 28908 | to fix the cells |
| Alexa Fluor 488 of goat anti-rabbit | Thermo Fisher Scientific | A-11008 | fluorescent secondary antibody |
| Alexa Fluor 555 of goat anti-mouse | Thermo Fisher Scientific | A-21425 | fluorescent secondary antibody |
| Alexa Fluor 555 Phalloidin | Thermo Fisher Scientific | A34055 | fluorescent secondary antibody |
| Bovine serum albumin standard (BSA) | euromedex | 04-100-812-E | |
| DMEM | Sigma-Aldrich | D5796-500ml | cell culture medium |
| Duolink In Situ Mounting Medium with DAPI | Sigma-Aldrich | DUO82040-5ML | mounting medium |
| Human: HeLa S3 cells | IGBMC, Strasbourg, France | cell line used to perform the experiments | |
| Hydrogen peroxide solution 30% (H2O2) | Sigma-Aldrich | H1009-100ml | used as a stressing agent |
| Lipofectamine 2000 Reagent | Thermo Fisher Scientific | 11668-019 | transfection reagent |
| Mouse monoclonal anti-coilin | abcam | ab11822 | Coilin-specific antibody |
| Nikon Optiphot-2 fluorescence microscope | Nikon | epifluoresecence microscope | |
| Opti-MEM I Reduced Serum Medium | Thermo Fisher Scientific | 31985062 | transfection medium |
| PBS pH 7.4 (10x) | gibco | 70011-036 | to wash the cells |
| Rabbit polyclonal anti-53BP1 | Thermo Fisher Scientific | PA1-16565 | 53BP1-specific antibody |
| Rabbit polyclonal anti-EDC4 | Sigma-Aldrich | SAB4200114 | EDC4-specific antibody |
| Triton X-100 | Roth | 6683 | to permeabilize the cells |
Referências
- Möckl, L., Lamb, D. C., Bräuchle, C. Super-resolved fluorescence microscopy: Nobel Prize in Chemistry 2014 for Eric Betzig, Stefan Hell, and William E. Moerner. Angewandte Chemie. 53 (51), 13972-13977 (2014).
- Meijering, E., Carpenter, A. E., Peng, H., Hamprecht, F. A., Olivo-Marin, J. -. C. Imagining the future of bioimage analysis. Nature Biotechnology. 34 (12), 1250-1255 (2016).
- Wiesmann, V., Franz, D., Held, C., Münzenmayer, C., Palmisano, R., Wittenberg, T. Review of free software tools for image analysis of fluorescence cell micrographs. Journal of Microscopy. 257 (1), 39-53 (2015).
- de Chaumont, F., et al. Icy: an open bioimage informatics platform for extended reproducible research. Nature Methods. 9 (7), 690-696 (2012).
- Girish, V., Vijayalakshmi, A. Affordable image analysis using NIH Image/ImageJ. Indian J Cancer. 41 (1), 47 (2004).
- Zaitoun, N. M., Aqel, M. J. Survey on image segmentation techniques. Procedia Computer Science. 65, 797-806 (2015).
- . MetaMorph Microscopy Automation and Image Analysis Software Available from: https://www.moleculardevices.com/products/cellular-imaging-systems/acquisition-and-analysis-software/metamorph-microscopy (2018)
- . NIS-Elements Imaging Software Available from: https://www.nikon.com/products/microscope-solutions/lineup/img_soft/nis-element (2014)
- Meyer, F., Beucher, S. Morphological segmentation. Journal of Visual Communication and Image Representation. 1 (1), 21-46 (1990).
- Schieber, M., Chandel, N. S. ROS Function in Redox Signaling and Oxidative Stress. Current Biology. 24 (10), 453-462 (2014).
- D'Autréaux, B., Toledano, M. B. ROS as signalling molecules: mechanisms that generate specificity in ROS homeostasis. Nature Reviews. Molecular Cell Biology. 8 (10), 813-824 (2007).
- Davalli, P., Mitic, T., Caporali, A., Lauriola, A., D'Arca, D. ROS, Cell Senescence, and Novel Molecular Mechanisms in Aging and Age-Related Diseases. Oxidative Medicine and Cellular Longevity. 2016, 3565127 (2016).
- Disher, K., Skandalis, A. Evidence of the modulation of mRNA splicing fidelity in humans by oxidative stress and p53. Genome. 50 (10), 946-953 (2007).
- Takeo, K., et al. Oxidative stress-induced alternative splicing of transformer 2β (SFRS10) and CD44 pre-mRNAs in gastric epithelial cells. American Journal of Physiology - Cell Physiology. 297 (2), 330-338 (2009).
- Seo, J., et al. Oxidative Stress Triggers Body-Wide Skipping of Multiple Exons of the Spinal Muscular Atrophy Gene. PLOS ONE. 11 (4), 0154390 (2016).
- Will, C. L., Luhrmann, R. Spliceosome Structure and Function. Cold Spring Harbor Perspectives in Biology. 3 (7), 003707 (2011).
- Ljosa, V., Sokolnicki, K. L., Carpenter, A. E. Annotated high-throughput microscopy image sets for validation. Nature Methods. 9 (7), 637-637 (2012).
- Wang, Q., et al. Cajal bodies are linked to genome conformation. Nature Communications. 7, (2016).
- Carpenter, A. E., et al. CellProfiler: image analysis software for identifying and quantifying cell phenotypes. Genome Biology. 7, 100 (2006).
- McQuin, C., et al. CellProfiler 3.0: Next-generation image processing for biology. PLoS Biology. 16 (7), 2005970 (2018).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados