JoVE 비디오를 활용하시려면 도서관을 통한 기관 구독이 필요합니다. 전체 비디오를 보시려면 로그인하거나 무료 트라이얼을 시작하세요.
Methods Article
하위 구조 분석기: 형광 현미경 이미지에서 세포 체의 신속한 탐색 및 정확한 분석을 위한 사용자 친화적인 워크플로우
요약
우리는 형광 현미경 이미지의 특정 세포 구획에서 세포 체의 신속한 탐사와 정확한 분석을 위해 구축 된 자유롭게 사용할 수있는 워크플로우를 제시합니다. 이 사용자 친화적인 워크플로우는 오픈 소스 소프트웨어 Icy에서 설계되었으며 ImageJ 기능을 사용합니다. 파이프라인은 이미지 분석에 대한 지식없이 저렴합니다.
초록
지난 10년 동안 공간 해상도 개선뿐만 아니라 살아있는 세포 이미징 및 고처리량 현미경 기술에서도 도시된 형광 현미경 기술의 돌파구가 특징입니다. 이로 인해 단일 실험에 대한 현미경 데이터의 양과 복잡성이 지속적으로 증가했습니다. 현미경 데이터의 수동 분석은 매우 시간이 많이 걸리고 주관적이며 정량적 분석을 금지하기 때문에 생체 이미지 분석의 자동화는 거의 피할 수 없게되고 있습니다. 우리는 형광 현미경 검사법에서 생체 이미지에서 신호 분석을 완전히 자동화하기 위해 하위 구조 분석기라는 정보학 워크플로우를 구축했습니다. 이 워크플로우는 사용자 친화적인 오픈 소스 플랫폼 Icy에서 개발되었으며 ImageJ의 기능으로 완료됩니다. 그것은 소음 비에 신호, 세포의 개별 세분화 (세포 경계의 검출) 및 특정 세포 구획에서 농축 된 세포 체의 검출 /정량화를 개선하기 위해 이미지의 사전 처리를 포함한다. 이 워크플로우의 주요 장점은 사용자 친화적인 인터페이스를 통해 이미지 분석 전문 지식 없이 사용자에게 복잡한 바이오 이미징 기능을 제안하는 것입니다. 더욱이, 그것은 매우 모듈식이며 핵 /세포 분석 전대의 특성화에서 다른 세포 하위 구조에서 다른 세포 체의 비교 분석에 이르기까지 여러 문제에 적응한다. 이 워크플로우의 기능은 산화 스트레스(OS) 조건 하에서 Cajal(코일) 바디의 연구를 통해 설명됩니다. 형광 현미경 검사법의 데이터는 인간 세포에 있는 그들의 무결성이 OS의 유도 후에 몇 시간 영향을 받았다는 것을 보여줍니다. 이러한 효과는 코일린의 핵재분배와 연관된 코일린 핵형성의 감소를 특성형 카잘 체체로 감소시켜 더 작은 포시 수로 재분배하는 것을 특징으로 한다. CB 성분과 주변 뉴클레오플라즘 사이의 교환에서 코일린의 중심 역할은 코일린의 OS 유도 재분배가 Cajal Body의 조성 및 기능에 영향을 미칠 수 있음을 시사한다.
서문
가벼운 현미경 검사법과 특히 형광 현미경 검사는 생물 과학에서 일반적으로 사용되는 견고하고 다재다능한 기술입니다. 그(것)들은 그들의 특정 형광 표시를 통해 단백질 또는 RNA 같이 각종 생체 분자의 정확한 현지화에 접근을 줍니다. 지난 10년 동안 에릭 베치그, 스테판 W. 지옥, 윌리엄 E. 모너가 슈퍼 해결형 형광 현미경 검사법(SRFM)1의개발을 수여한 2014년 노벨 화학상에서 입증된 바와 같이 현미경 및 이미징 기술의 급속한 발전이 특징지어졌습니다. SFRM은 나노 크기로 가져오기 위해 기존의 광학 현미경 검사법의 회절 한계를 우회합니다. 라이브 이미징 또는 높은 처리량 스크리닝 접근법과 같은 기술의 개선은 또한 각 실험에 대해 치료할 데이터의 양과 복잡성을 증가시킵니다. 대부분의 경우 연구자들은 세포의 이질적인 집단에 직면하고 단일 세포 수준에서 표현형을 분석하고자 합니다.
처음에, foci 계산과 같은 분석은 계산 프로세스를 완전히 시각적인 통제를 제공하기 때문에 몇몇 연구원에 의해 선호되는 눈에 의해 수행되었습니다. 그러나 이러한 데이터의 수동 분석은 너무 오래 걸리고 관찰자 간의 가변성을 유발하며 컴퓨터 지원 접근 방식이 널리 사용되고 거의 피할 수 없는2가되도록 보다 복잡한 기능에 대한 액세스를 제공하지 않습니다. 생체 이미지 정보학 방법은 데이터 분석의 효율성을 크게 향상시키고 수동 계수 분석의 피할 수 없는 작업자 주관성과 잠재적 편향이 없습니다. 이 분야의 수요 증가와 컴퓨터 전력의 개선으로 인해 많은 수의 이미지 분석 플랫폼이 개발되었습니다. 그들 중 일부는 자유롭게 사용할 수 있으며 개인용 컴퓨터로 분석을 수행하기 위해 다양한 도구에 액세스 할 수 있습니다. 오픈 액세스 도구의 분류는 최근 설립되었습니다3 유용성과 기능을 결합하는 강력한 소프트웨어로 얼음4를 제시한다. 또한, 얼음은 ImageJ와 통신의 장점이있다.
이미지 분석 전문 지식이 없는 사용자의 경우 주요 장애물은 문제가 있는 매개 변수에 따라 적절한 도구를 선택하고 잘 이해하지 못하는 매개 변수를 올바르게 조정하는 것입니다. 또한 설정 시간은 종종 깁니다. Icy는 "프로토콜"이라는 사용자 친화적인 포인트 앤 클릭 인터페이스를 제안하여 전체 컬렉션4내에서 발견되는 일부 플러그인을 결합하여 워크플로우를 개발할 수 있습니다. 유연한 모듈식 설계와 포인트 앤 클릭 인터페이스를 통해 프로그래머가 아닌 사람들에게 가능한 분석을 설정할 수 있습니다. 여기서 우리는 특정 셀룰러 구획에서 형광 신호를 분석하고 밝기, foci 수, foci 크기 및 공간 분포와 같은 다양한 기능을 측정하는 Icy의 인터페이스에서 개발 된 하위 구조 분석기라는워크플로우를 제공합니다. 이 워크플로우는 신호 전좌의 정량화, 형광 기자를 발현하는 전감염된 세포의 분석, 또는 개별 세포에서 다른 세포 하위 구조에서 의 초점 분석과 같은 몇 가지 문제를 해결합니다. 여러 이미지를 동시에 처리할 수 있으며, 출력 결과를 일반적으로 사용되는 스프레드시트 프로그램에서 열 수 있는 탭 구분워크시트로 내보낼 수 있습니다.
하위 구조 분석기 파이프라인은 그림 1에표시됩니다. 첫째, 지정된 폴더에 포함된 모든 이미지는 신호 대 노이즈 비율을 개선하기 위해 미리 처리됩니다. 이 단계는 다음 단계의 효율성을 높이고 실행 시간을 줄입니다. 그런 다음 형광 신호를 감지해야 하는 이미지 영역에 대응하는 관심 영역(ROI)이 식별되고 세분화됩니다. 마지막으로 형광 신호를 분석하고 결과는 탭 구분 워크시트로 내보냅니다.
물체 세분화(경계 감지)는 이미지 분석에서 가장 까다로운 단계이며, 그 효율성은 결과 셀 측정의 정확도를 결정합니다. 이미지(1차 개체라고 함)에서 확인된 첫 번째 개체는 DNA 염색 된 이미지 (DAPI 또는 Hoechst 염색)에서 종종 핵이지만 기본 개체는 전체 세포, 구슬, 반점, 종양 또는 얼룩이있는 물체일 수 있습니다. 대부분의 생물학적 이미지에서 세포 또는 핵은 서로 접촉하거나 중첩되어 간단하고 빠른 알고리즘이 실패합니다. 현재까지, 어떤 범용 알고리즘도 모든 객체의 완벽한 세분화를 수행할 수 없으며, 대부분 특성(크기, 모양 또는 텍스처)이 분할5의효율성을 조절하기 때문입니다. 현미경 소프트웨어(예: 분자 장치에 의한 변형 이미징 소프트웨어6또는 Nikon7의NIS-Elements Advances 연구 소프트웨어)와 함께 일반적으로 배포되는 세분화 도구는 일반적으로 상관 관계 일치, 임계값 또는 형태학적 작업과 같은 표준 기술을 기반으로 합니다. 기본 시스템에서는 효율적이지만 이러한 보다 일반화된 메서드는 보다 까다롭고 특정한 컨텍스트에서 사용할 때 한계를 빠르게 제시합니다. 실제로, 세분화는 세포 유형, 세포 밀도 또는 바이오마커와 같은 실험 파라미터에 매우 민감하며, 종종 큰 데이터 세트에 대한 반복적인 조정이 필요하다. 하위 구조 분석기 워크플로는 간단하고 정교한 알고리즘을 통합하여 이미지 복잡성및 사용자 요구에 맞게 다양한 대안을 제안합니다. 특히 고클러스터된 개체에 대한 마커 기반 유역 알고리즘8을 제안합니다. 이 세분화 방법의 효율성은 각 개체의 개별 마커 선택에 의존합니다. 이러한 마커는 사용자가 많은 수의 객체에 직면할 때 매우 많은 시간이 소요되는 전체 세분화에 대한 올바른 매개 변수를 얻기 위해 대부분의 시간을 수동으로 선택합니다. 하위 구조 분석기는 이러한 마커를 자동으로 감지하여 매우 효율적인 세분화 프로세스를 제공합니다. 세분화는, 대부분의 경우 이미지 분석의 제한 단계이며 이미지의 해상도, 이미지당 개체 수 및 개체 의 클러스터링 수준에 따라 처리 시간을 상당히 수정할 수 있습니다. 일반적인 파이프라인은 표준 데스크톱 컴퓨터에서 이미지당 몇 초에서 5분이 필요합니다. 더 복잡한 이미지를 분석하려면 보다 강력한 컴퓨터와 이미지 분석에 대한 몇 가지 기본 지식이 필요할 수 있습니다.
이 워크플로의 유연성과 기능은 대표 결과의 다양한 예제와 함께 설명되어 있습니다. 이 워크플로우의 장점은 특히 산화 스트레스 (OS) 조건하에서 핵 하위 구조의 연구를 통해 표시됩니다. OS는 산화제에 찬성하는 레독스 항상성의 불균형에 대응하고 반응성 산소 종 (ROS)의 높은 수준과 연관된다. ROS는 신호 분자로서 작용하기 때문에, 그들의 농도 및 세포 세포 소소화의 변화는 신호 변환, 수리 메커니즘, 유전자 발현, 세포 사멸 및 증식9,,10을포함하여 생리적 기능을 조절하는 수많은 경로 및 네트워크에 긍정적 또는 부정적으로 영향을 미친다. 따라서 OS는 다양한 병리학 (신경 퇴행성 및 심혈관 질환, 암, 당뇨병 등)뿐만 아니라 세포 노화에도 직접 관여합니다. 따라서 인간 세포의 조직과 기능에 대한 OS의 결과를 해독하는 것은 인간 병리학의 발병 및 개발에서 OS의 역할을 이해하는 데 중요한 단계입니다. OS는 여러 전사 인자(p53, Nrf2, FOXO3A)11을통해 전사를 조절함으로써 유전자 발현을 조절하는 것으로 확립되었지만, 또한 전RNA12,,13,14의대체 접합(AS)과 같은 여러 공동 및 전사 후 과정의조절에영향을 미치는 것으로 확립되었다. 1차 코딩 및 비코딩 전사체의 대체 접합은 전사동형태를 생성하여 게놈의 인코딩 능력을 증가시키는 필수적인 메커니즘이다. AS는 거의 300 단백질과 5 U-풍부한 작은 핵 RNA (UsnRNAs)15를포함하는 spliceosome에게 불린 거대한 ribonucleoprotein 복합체에 의해 수행됩니다. Spliceosome 조립 및 AS는 세포에서 단단히 통제되고 화려한 성숙의 몇몇 단계는 Cajal 바디라는 막없는 핵 구획 내에서 생깁니다. 이러한 핵 하위 구조는 주로 코일린 단백질을 가진 RNA 및 단백질 성분의 다원적 상호 작용에 의해 수행되는 구조와 그 조성물의 동적 특성을 특징으로 합니다. 하위 구조 분석기 워크플로우를 가진 수천 개의 셀을 분석하여 OS가 Cajal Body에 미치는 영향을 설명하지 않은 특성화를 허용했습니다. 실제로, 얻은 데이터는 OS가 카잘 바디의 핵을 수정하고, 수많은 작은 핵 포시로 코일린 단백질의 핵 소플라즈마 재분배를 유도한다는 것을 건의합니다. Cajal 바디의 구조의 이러한 변화는 화려한의 성숙에 영향을 미치고 OS에 의한 AS 변조에 참여할 수 있습니다.
프로토콜
참고 : 사용자 친화적 인 튜토리얼은 http://icy.bioimageanalysis.orgIcy의 웹 사이트에서 사용할 수 있습니다.
1. 얼음 과 하위 구조 분석기 프로토콜을 다운로드
- 얼음 웹 사이트에서 얼음을 다운로드(http://icy.bioimageanalysis.org/download) 하위 구조 분석기 프로토콜을 다운로드 : http://icy.bioimageanalysis.org/protocols?sort=latest.
참고: 64비트 OS를 사용하는 경우 64비트 버전의 Java를 사용해야 합니다. 이 버전은 얼음에 할당 된 메모리를 증가 할 수있습니다 (환경 설정 | 일반 | 최대 메모리).
2. 프로토콜 열기
- 얼음을 열고 리본 메뉴의 도구를 클릭합니다.
- 프로토콜을 클릭하여 프로토콜 편집기 인터페이스를 엽니다.
- 로드를 클릭하고 프로토콜 하위 구조 분석기를 엽니다. 프로토콜 로드는 몇 초 정도 걸릴 수 있습니다. 프로토콜을 사용하기 전에 프로토콜의 열기가 완료되었는지 확인합니다.
참고: 워크플로는 그림 2a에제시된 13개의 일반 블록으로 구성됩니다. 각 블록은 특정 하위 작업을 수행하는 여러 상자로 구성된 파이프라인으로 작동합니다.
3. 얼음에 워크플로우와 상호 작용
참고: 각 블록 또는 상자에 번호가 매겨지며워크플로(그림 2b)내에서 특정 랭크가 있습니다. 이 번호를 클릭하면 첫 번째 에 가장 가까운 위치가 선택한 블록/상자에 할당된 다음 다른 블록/상자의 위치가 다시 구성됩니다. 워크플로를 준비할 때 블록의 올바른 순서를 존중합니다. 예를 들어 스팟 검출기 블록은 스팟 검출기 블록 전에 세분화 블록을 실행해야 하므로 미리 정의된 ROI가 필요합니다. 상자의 위치를 수정하지 마십시오. 이미지 이름에 ""를 사용하지 마십시오.
- 왼쪽 상단 모서리 아이콘을 클릭하여블록(그림 2b)을축소, 확장, 확대, 확대 또는 제거합니다.
- 워크플로의 각 파이프라인은 입력 및출력(그림 2b)을통해 연결된 상자 네트워크를 특징으로 합니다. 연결을 만들려면 출력을 클릭하고 커서가 입력에 도달할 때까지 유지 관리합니다. 출력 태그를 클릭하여 연결을 제거할 수 있습니다.
4. 이미지 채널 병합
- 블록 병합 채널을 사용하여 병합된 이미지를 생성합니다. 필요한 경우 병합할 시퀀스가 동일한 이름의 접두사를 갖도록 파일이름을 변경한 다음 고유한 분리기입니다. 예를 들어 이미지 A의 개별 채널 시퀀스의 이름이 ImageA_red ImageA_blue.
참고: 분리기의 경우 이미지 이름에 이미 있는 문자를 사용하지 마십시오. - 동일한 폴더에서 병합할 채널당 새 폴더 를 만듭니다. 예를 들어 빨간색, 녹색 및 파란색 채널을 병합하고 3개의 폴더를 만들고 해당 시퀀스를 이러한 폴더에 저장합니다.
- 블록 병합 채널만사용하고, 다른 블록을 제거하고, 프로토콜을 병합 채널로 저장합니다.
- 상자에 액세스하여 매개 변수를 설정합니다. 각 채널에 대해 상자 채널 번호 X(상자 1, 5 또는 9), 폴더 채널 번호 X(상자 2, 6 또는 10), 분리기 채널 번호 X(상자 3, 7 또는 11) 및 Colormap 채널 nb X(상자 4, 8 및 12)를 각각 채웁니다.
참고: 이 상자는 동일한 채널에 해당하는 각 줄의 4개줄로 가로로 그룹화됩니다. 각 선에서 디스플레이(상자 23, 24 또는 25)도 사용하여 해당 채널의 시퀀스를 직접 시각화할 수 있습니다.- 상자 채널 번호 X에서추출할 채널을 선택합니다(클래식 RGB 이미지, 0=빨간색, 1=녹색, 2=파란색). 사용자는 시퀀스 탭에서 Icy의 검사기 창 내에서 이미지의 다른 채널에 빠르게 액세스합니다.
- 상자 폴더 채널 번호 X에서채널 X의 이미지가 포함된 폴더의 \Name을 작성합니다.
- 상자 분리기 채널 번호 X에서이미지 이름에 사용되는 분리기(이전 예: "_red", "_green" 및 "_blue")를 작성합니다.
- 상자 Colormap 채널 nb X에서Icy에서 해당 채널을 시각화하는 데 사용할 컬러맵 모델을 숫자로 나타냅니다. 사용 가능한 색상 맵은 검사기 창의 시퀀스 탭에 표시됩니다.
- 병합된 이미지의 상자 형식(상자 28)에서 병합된 이미지를 저장하기 위해 확장을 작성합니다.
참고: 2개의 채널만 병합하려면 세 번째 채널에 해당하는 네 개의 상자를 채우지 마십시오.
- 병합 채널 블록의 왼쪽 위 모서리에서 폴더오른쪽에 있는 링크를 직접 클릭합니다. 표시되는 열린 대화 상자에서 상자 폴더 채널 번호 1(상자 2)에 정의된 첫 번째 채널의 시퀀스가 포함된 폴더를 두 번 클릭합니다. 그런 다음 열기를 클릭합니다.
- 병합 채널 블록의 왼쪽 위 모서리에 있는 검은 색 화살표를 클릭하여 프로토콜을 실행합니다(자세한 내용은 7부 참조). 병합된 이미지는 개별 채널의 폴더와 동일한 디렉토리의 병합 폴더에 저장됩니다.
- 상자에 액세스하여 매개 변수를 설정합니다. 각 채널에 대해 상자 채널 번호 X(상자 1, 5 또는 9), 폴더 채널 번호 X(상자 2, 6 또는 10), 분리기 채널 번호 X(상자 3, 7 또는 11) 및 Colormap 채널 nb X(상자 4, 8 및 12)를 각각 채웁니다.
5. 관심 영역의 세분화
참고: 하위 구조 분석기는 단순하고 정교한 알고리즘을 통합하여 이미지 복잡성 및 사용자 요구에 맞게 다양한 대안을 제안합니다.
- 적응된 블록을 선택합니다.
- 개체가 서로 만지지 않거나 사용자가 클러스터된 개체를 개별적으로 구분할 필요가 없는 경우 블록 세분화 A: 클러스터된 개체가 아닌 객체를사용합니다.
- 개체가 서로 만지지 않지만 그 중 일부가 가까이있는 경우 블록 세분화 B: 클러스터된 개체를사용합니다.
- 클러스터링 레벨과 볼록 모양이 높은 객체의 경우 볼록 모양이 있는 클러스터된 객체인 블록 세분화 C: 클러스터된 객체를사용합니다.
- 개체가 높은 클러스터링 레벨을 표시하고 불규칙한 셰이프가 있는 경우 블록 세분화 D: 불규칙한 셰이프가 있는 클러스터된 객체를사용합니다.
- 블록 세분화 E를 사용하십시오: 클러스터된 세포질은 분할된 핵을 마커로 사용하여 개별적으로 접촉하는 세포질을 분할합니다. 이 블록은 반드시 공정하기 위해 분할 된 핵이 필요합니다.
참고: 기본 개체 세분화 프로세스에 맞게 조정된 블록은 동일한 실행에서 여러 블록을 사용하여 특정 하위 구조에 대한 효율성을 비교하거나 다양한 유형의 하위 구조를 분할하는 데 사용할 수 있습니다. 클러스터링 수준이 동일한 이미지 집합 내에서 이질적인 경우 적응된 블록에서 작고 고도로 클러스터된 개체를 별도로 처리합니다.
- 블록 선택 폴더의 출력0(파일)을 선택한 분할 블록의 폴더 입력에 연결합니다.
- 선택한 분할 블록의 매개 변수를 설정합니다.
- 분할 A: 클러스터되지 않은 개체 및 분할 C: 볼록 모양이 있는 클러스터된 개체
- 상자 채널 신호(상자 1)에서 오브젝트의 채널을 세그먼트로 설정합니다.
- 옵션으로, 상자 가우시안 필터 (상자 2)에서, 객체 내부의 신호가 이질적 인 경우 X와 Y 시그마 값을 증가. 가우시안 필터는 질감을 부드럽게 하여 더 균일한 영역을 확보하고 핵 분단의 속도와 효율성을 높입니다. 개체가 작을수록 시그마 값이 낮습니다. 높은 시그마 값을 피하십시오. 기본 값을 0으로 설정합니다.
- 상자 HK-Means(상자 3)에서 강도 클래스 매개 변수와 감지할 개체의 최소 및 최대 크기(픽셀)를 설정합니다.
참고: 강도 클래스의 경우 2값은 배경 과 전경의 2개 클래스에서 픽셀을 분류합니다. 따라서 개체와 배경 사이의 대비가 높을 때 조정됩니다. 전경 오브젝트의 강도가 다르거나 배경과의 대비가 낮은 경우 클래스 수를 늘립니다. 기본 설정은 2입니다. 개체 크기를 관심 있는 개체 주위에 수동으로 ROI를 그리면 개체 크기를 신속하게 평가할 수 있습니다. 커서로 가리키는 경우 ROI 크기(픽셀내 내부)가 이미지에 직접 나타나거나 ROI 통계 창에서 액세스할 수 있습니다(검색 표시줄에서 열림). 최적의 매개 변수는 단일 ROI에서 각 전경 오브젝트를감지합니다. 그들은 수동으로 얼음(탐지 및 추적 | HK-Means). - 상자에서 활성 윤곽(상자 4)에서 개체 테두리 의 검색을 최적화합니다. 이 플러그인에 대 한 철저한 설명서 온라인 사용할 수 있습니다.: http://icy.bioimageanalysis.org/plugin/Active_Contours. 올바른 매개 변수는 Icy(탐지 및 추적]에서 수동으로 정의할 수도 있습니다. 활성 윤곽).
- 이 과정에서 세그먼트된 개체의 이미지를 저장하기 위해 폴더가 자동으로 만들어집니다. 상자 텍스트(상자 6)에서 이 폴더의 이름을 지정합니다(예: 분할된 핵). 분할된 개체(Tiff, GIF, Jpeg, BMP, PNG)의 이미지를 저장하는 형식을 설정하려면 분할된 개체의 이미지의상자 형식을 채웁니다. 폴더는 병합된 이미지가 포함된 폴더에 만들어집니다.
- 워크플로를 실행합니다(자세한 내용은 7부 참조).
- 분할 B: 클러스터가 제대로 클러스터된 개체
- 상자 채널 신호, HK-Means, 활성 윤곽선,분할 된 개체 및 텍스트를 저장하는 확장의 매개 변수를 설정하려면 5.3.1에서와 동일한 단계를 따르십시오 (상자 순위는 5.3.1 단계와 동일하지 않습니다).
- 상자 에서 IJ 플러그인 (상자 4)를 호출, 배경 뺄셈을 제어하기 위해 롤링 매개 변수를 설정합니다. 이 매개 변수를 배경의 일부가 아닌 가장 큰 개체의 크기로 설정합니다. 이 값을 줄이면 배경 제거가 증가하지만 전경 신호의 손실을 유발할 수도 있습니다.
- 상자 적응 히스토그램 균등화(상자 6)에서 전경 오브젝트와 배경 사이의 대비를 개선합니다. 경사를 늘리면 더 대조적인 시퀀스가 됩니다.
- 워크플로를 실행합니다(자세한 내용은 7부 참조).
- 분할 D: 불규칙한 모양이 있는 클러스터된 개체
참고: 각 이미지에 세 가지 세분화 방법이 적용됩니다.HK-수단 클러스터링액티브 컨투어 방법적용됩니다. 그런 다음,고전 분수령 알고리즘(유클리디아 거리 맵사용)은 이전에 잘못 분할된 개체에 적용됩니다. 마지막으로,마커 기반 분수령 알고리즘사용됩니다. HK-means 및 마커 기반 유역 방법만 사용자 개입이 필요합니다. 두 방법 모두 모든 이미지(완전 자동화된 버전)에 동일한 매개 변수를 적용하거나 각 이미지(반자동 버전)에 대해 변경할 수 있습니다. 사용자가 이러한 세분화 방법에 대해 교육을 받지 않은 경우 반자동 처리를 권장합니다. 이 블록을 처리하는 동안 수동 개입이 필요합니다. 세분화 메서드가 완료되면 사용자는 다음 분할 메서드가 시작되기 전에 잘못 분할된 개체를 수동으로 제거해야 합니다. 성공적으로 분할된 개체는 저장되며 다음 단계에서 고려되지 않습니다. 이 블록은 블록과 연결되어야 합니다.클러스터/이기종 모양 기본 개체 분할 대화 상자제대로 작동합니다.- https://github.com/ijpb/MorphoLibJ/releases이미지J 컬렉션 MorphoLibJ를 다운로드합니다. MorphoLibJ 1.4.0 버전은 이 프로토콜에 사용됩니다. 폴더 얼음 / ij / 플러그인에 파일 MorphoLibJ_-1.4.0.jar을 놓습니다. 이 컬렉션의 내용에 대한 자세한 내용은 https://imagej.net/MorphoLibJ 확인할 수 있습니다.
- 5.3.1 단계와 동일한 단계를 수행하여 상자 채널 신호, 가우시안 필터, 액티브 컨투어,확장의 매개 변수를 설정하여 분할된 개체 및 텍스트를저장합니다. 상자 순위는 5.3.1 단계와 동일하지 않습니다.
- 상자 적응 히스토그램 균등화의 매개 변수를 설정합니다(5.3.2.3 단계 참조).
- 빼기 배경을활성화하려면, 선택 서적 배경 적용에 예 쓰기? (상자 5). 다른, 아니오를 작성합니다. 플러그인이 활성화되면 롤링 매개 변수(단계 5.3.2 참조)를 상자에 배경 매개 변수(상자 7)를 뺍니다.
- HK 수단의 자동화: 모든 이미지(완전 자동화처리)에 동일한 매개변수를 적용하려면 클래스 의 Nb(상자 11), 최소 크기(상자 12), 최대 크기(box13)를 설정합니다(단계 5.3.1 참조). Minimum size 이러한 매개 변수는 최대 전경 픽셀을 선택하고 전경 객체의 개별화를 최적화하도록 설정해야 합니다. 반자동 처리 버전의 경우 개입이 필요하지 않습니다.
- 마커 추출의 자동화: 완전 자동화된 버전의 경우 내부 마커 추출(box 27)을 확장하고 스크립트의 13줄에서 "동적" 매개 변수값을 설정합니다. 반자동 처리 버전의 경우 개입이 필요하지 않습니다.
참고: 마커는 "동적" 매개 변수에 의해 제어되는 입력 이미지에 확장 된 미니마 변환을 적용하여 추출됩니다. 마커 기반 분수령 알고리즘에서 이러한 마커의 플러드는 개체 세분화를 수행하기 위해 시뮬레이션됩니다. 전경 오브젝트의 성공적인 세분화를 위해 전경 오브젝트당 하나의 마커를 추출해야 합니다. 최적의 마커 추출을 위한 "동적" 매개 변수의 설정은 주로 이미지의 해상도에 따라 달라집니다. 따라서 이 매개 변수에 익숙하지 않은 경우 반자동 버전을 사용합니다. - 워크플로를 실행합니다(자세한 내용은 7부 참조).
- 처리 초기에 대화 상자는 HK-means 매개 변수와 마커 기반 유역이 연속적으로 열립니다. 모든 이미지(완전 자동화된 버전)에 동일한 매개 변수를 적용하려면 YES를 클릭합니다. 그렇지 않으면 아니오를 클릭합니다. 정보 상자가 열리며 "HK-Means 플러그인으로 최적의 ROI를 결정하고 이미지를 닫아야 합니다." 확인을 클릭하고 수동으로 HK-수단 플러그인을 적용(탐지 및 추적| 홍콩-수단) 자동으로 열리는 이미지에서. HK-Means 플러그인 상자에서 내보내기 ROI 옵션을 선택합니다. 최대 전경 픽셀을 포함하는 ROI를 적용하고 전경 객체의 개별화를 최적화합니다. 최적의 ROI가 발견되면 이미지를 직접 닫습니다.
- 첫 번째 세분화 메서드가 끝나면 정보 상자가 열리고 "원치 않는 ROI 제거 및 이미지 닫기"를 요청합니다. 이러한 ROI는 분할된 개체의 테두리에 해당합니다. 확인을 선택하고 이미지에서 잘못 분할된 개체의 ROI를 제거하여 자동으로 열립니다. ROI는 커서를 테두리에 배치하고 키보드의 "삭제" 버튼을 사용하여 쉽게 제거할 수 있습니다. 이미지를 닫습니다. 두 번째 세분화 단계가 완료된 후 동일한 절차를 반복합니다.
- 이 단계에서 마커 기반 분수령 알고리즘의 전체 자동화를 위해 YES 버튼을 선택한 경우 이전에 설정된 매개 변수가 모든 이미지에 적용됩니다.
- NO 버튼을 선택하면 정보 상자가 열리며 "내부 마커를 결정하고 조정"하라는 요청이 표시됩니다. 확인을 클릭하고 얼음의 ImageJ 인터페이스 내에서, 플러그인으로 이동 | 모포리브제이 | 미니마와 맥시마| 확장 된 Min & 최대. 작업 중 확장 된 미니마를 선택합니다.
- 미리 보기를 선택하여 자동으로 열린 이미지에서 변환 결과를 미리 시각화합니다. 최적의 마커가 관찰될 때까지 동적을 이동합니다. 마커는 값이 255(반드시 흰색 픽셀은 아님)의 픽셀 그룹입니다. 최적의 매개 변수는 개체당 하나의 마커로 연결됩니다. 이전 두 분할 메서드로 잘 분할되지 않은 나머지 개체에 초점을 맞춥니다.
- 필요한 경우 "열기" 또는 "닫기"와 같은 추가 형태학적 작업을 적용하여마커를 개선합니다(플러그인 | 모포리브제이 | 형태학적 필터). 마커의 최종 이미지를 얻을 때, 그것을 열고 처음에 확장 미니마 작업에 대한 입력으로 사용되는 이미지로 끝나는 다른 모든 이미지를 닫습니다. ImageJ 상자가 이 이미지의 변경 내용을 저장하도록 요청하는 경우 아니오를 클릭합니다.
- 정보 상자(상자 14)가 있는 이미지의 상자 Nb에서 정보 상자가 있는 이미지 수를 결정합니다.
- 세분화 E: 클러스터된 세포질
참고: 이 블록은 세포질 분할을 시작하기 위해 이전에 분할된 핵을 개별 마커로 사용합니다. 핵 분할의 블록은 그것을 사용하기 전에 처리되었는지 확인하십시오.- 상자 채널 세포질 (상자 1)에서, 세포질 신호의 채널을 설정합니다.
- 상자 확장 분할 핵 (상자 2)에서, 분할 된 핵의 이미지를 저장하는 데 사용되는 형식을 작성 (tif, jpeg, bmp, png). 기본 형식은 tif입니다.
- 상자 텍스트(box3)에 분할된 핵을 포함하는 폴더의 \Name을 작성합니다. Text
- 세분화된 세포질(상자 4)의 이미지 상자 형식에서 분할된 개체 이미지(Tiff, GIF, Jpeg, BMP, PNG)를 저장하는 데 사용할 형식을 설정합니다.
- 이 과정에서 세그먼트된 세포질의 이미지를 저장하기 위해 폴더가 자동으로 만들어집니다. 상자 텍스트(상자 5)에서 이 폴더의 이름을 지정합니다(예: 분할된 사이토플라솜). 폴더는 병합된 이미지가 포함된 폴더에 만들어집니다.
- 5.3.1 단계와 동일한 단계를 수행하여 상자 가우시안 필터 및 활성 윤곽의 매개 변수를 설정합니다(주의해야, 상자 랭크는 5.3.1 단계와 동일하지 않음).
- 워크플로를 실행합니다(자세한 내용은 7부 참조).
- 분할 A: 클러스터되지 않은 개체 및 분할 C: 볼록 모양이 있는 클러스터된 개체
6. 형광 신호 감지 및 분석
- 적응된 블록을 선택합니다.
- 블록 형광 분석 A: 1 채널에서,하나의 유형의 분할 된 개체 내부의 한 채널에서 초점의 검출 및 분석을 수행 : 핵 내에서 코일린 포시 (적색 채널)의 검출.
- 블록 형광 분석 B: 2 채널은 동일한 구획에서,하나의 유형의 분할 된 개체 내부의 두 채널에서 포시의 검출 및 분석을 수행합니다 : 핵 내코일린 (빨간 채널) 및 53BP1 (녹색 채널) 초점의 검출.
- 블록 형광 분석 C: 2 개의 구획에서 2 채널,하나 또는 두 개의 채널에서 초점의 검출 및 분석을 수행, 특히 핵 과 해당 세포질 내부: 코일린 포시 (적색 채널)의 검출 모두 핵 과 그 해당 세포질 내의 검출 (적색 채널) 핵 및 G3BP 내의 코일린 포시 (적색 채널)
- 블록 형광 분석 D: 글로벌 전좌에서두 셀룰러 구획(a 및 b)에서 한 채널의 신호 비율을 계산합니다. 예를 들어, 세포질/핵 전좌 분석에서 최종 "결과" 스프레드시트에서 각 이미지에 대해 계산된 백분율의 핵 및 세포질 신호를 내보냅니다. 핵 신호 백분율을 계산하는 데 사용되는 공식은 다음과 같습니다. 이 블록은 모든 셀룰러 구획에 사용할 수 있습니다.
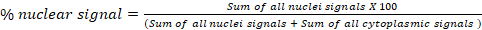
- 블록 형광 분석 E: 개별 세포 전좌에서각 셀룰러 구획에 하나의 채널에서 신호의 백분율을 계산합니다. 이 블록은 단일 세포 수준에서 핵/세포질 전좌 분석에 특별히 최적화되어 있습니다.
참고: 블록 형광 분석 E: 개별 세포 전좌는 단일 세포 수준에서 분석을 수행하기 때문에 핵과 세포질의 효율적인 세분화가 필요합니다.
- 블록 선택 폴더(블록 1)의 출력0(파일)을 선택한 블록의 폴더 입력(검은색 원의 흰색 화살표)에 연결합니다.
- 선택한 블록의 매개 변수를 설정합니다.
- 형광 분석 A: 1 채널, 형광 분석 B: 동일한 구획 및 형광 분석 C의 2 채널: 2 개의 구획에 2 개의 채널
- 상자 폴더 이미지 ROI에서백슬래시 앞에 분할된 개체의 이미지가 포함된 폴더의 이름을 작성합니다. (예를 들어: \분할된 핵).
- 분할된 개체의 이미지 형식(상자 2)에서 분할된 개체의 이미지를 저장하는 데 사용되는 형식을 작성합니다(tif, jpeg, bmp, png). 기본 형식은 tif입니다.
- 상자에서 국경을 죽여? Yes 그렇지 않으면 아니오를 작성합니다. ImageJ의 MorphoLibJ 컬렉션의 설치는이 기능을 사용 하 여 필요 (단계 참조 5.3.3).
- 상자(es) 채널 스팟 신호에서반점을 감지해야 하는 채널을 설정합니다. 클래식 RGB 이미지에서 0=빨간색, 1=녹색 및 2=파란색입니다.
- 상자에 국화 된 분자의 이름,반점에 국소화 분자의 이름을 작성합니다. 입력할 필드 수는 분자 수에 따라 다릅니다.
- 상자(es) 웨이블렛 스팟 검출기 블록에서각 채널에 대한 스팟 감지 매개 변수를 설정합니다. 배율(들)을 설정하고(스팟 크기라고 함) 및 검출의 감도(감도가 작을수록 감지된 스팟 의 수가 감소하고 기본 값이 100이고 최소 값이 0이 됨). 이 플러그인의 철저한 문서는 온라인으로 사용할 수 있습니다 : http://icy.bioimageanalysis.org/plugin/Spot_Detector. 매개 변수는 얼음(탐지 및 추적 |에서 수동으로 정의 할 수 있습니다. 스팟 검출기).
- 옵션으로 상자에서 크기별로 ROI를 필터링하여크기 간격(픽셀)을 설정하여 반점이 감지되는 세그먼트 객체를 필터링합니다. 이 단계는 세분화된 아래 또는 과도하게 분할된 개체를 제거하는 데 특히 유용합니다. 개체 크기를 수동으로 추정하려면 5.3.1 단계를 참조하십시오. 기본 매개 변수에는 크기별로 ROI 필터링이 포함되지 않습니다. 두 구획의 블록 2 채널에는 크기별로 핵 필터(상자 19) 및 크기별 필터 세포질(상자 46)의 두 개의 상자가 포함되어 있습니다.
- 선택적으로 상자에서 크기별로 반점을 필터링하여크기(픽셀)에 따라 감지된 반점을 필터링하여 원치 않는 아티팩트를 제거합니다. 스팟 크기를 수동으로 추정하려면 감지 및 추적을 클릭하고 스팟 감지기 플러그인을 엽니다. 출력 옵션에서 ROI로 내보내기를 선택합니다. 기본 매개 변수에는 크기별로 스팟 필터링이 포함되지 않으며 필터링된 반점이 분석을 고려하지 않도록 주의해야 합니다. 입력할 필드 수는 채널 수에 따라 다릅니다.
- 선택적으로, 상자 필터 반점에,감지 된 반점에 추가 필터 (대조, 균일성, 둘레, 둥근)를 적용합니다. 기본 매개 변수에는 스팟 필터링이 포함되지 않으며 필터링된 스팟이 분석을 고려하지 않도록 주의해야 합니다. 입력할 필드 수는 채널 수에 따라 다릅니다.
- 선택적으로, 상자 스팟 크기 임계값에서분석된 반점의 영역(픽셀)에 대한 임계값을 설정합니다. 이 임계값 이하의 계산된 스팟 수는 최종 결과 스프레드시트에서 내보냅니다. 정보에 입각할 상자 수는 채널 수에 따라 다릅니다.
- 워크플로를 실행합니다(자세한 내용은 7부 참조). 데이터는 병합된 이미지가 포함된 폴더에 저장된 결과 스프레드시트에 내보내지 않습니다.
- 형광 분석 D: 글로벌 전좌 및 형광 분석 E: 개별 세포 전좌:
- 상자 폴더 이미지(상자 1 및 2)에 분할된 개체의 이미지가 포함된 폴더의 \Name을 작성합니다. 블록 형광 분석 D: 글로벌 전좌에서두 가지 유형의 ROI는 ROI a 및 ROI b로 식별됩니다. 블록 형광 분석 E: 개별 세포 전좌,폴더 이미지 분할 핵 및 폴더 이미지 분할 세포질 상자를, 분할 된 핵과 세포질을 포함하는 폴더의 이름을 각각 작성합니다.
- 상자 채널 신호(상자 3)에서 신호의 채널을 입력합니다.
- 분할된 개체의 이미지 형식(상자 4)에서 분할된 개체의 이미지를 저장하는 데 사용되는 형식을 작성합니다(tif, jpeg, bmp, png). 기본 형식은 tif입니다. 테두리 개체를 제거할 수 있는 데 보더 테두리 제거 옵션도 있습니다(6.3.1 단계 참조).
- 선택적으로, 상자에서 크기별로 ROI를 필터링하고,크기 간격(픽셀)을 설정하여 분할된 객체를 필터링합니다. 이 단계는 세분화된 아래 또는 과도하게 분할된 개체를 제거하는 데 유용할 수 있습니다. 개체 크기를 수동으로 추정하려면 5.3.1 단계를 참조하십시오. 입력할 필드가 두 개 있습니다. 기본 매개 변수에는 크기별 ROI 필터링이 포함되지 않습니다.
- 워크플로를 실행합니다(자세한 내용은 7부 참조). 병합된 이미지가 포함된 폴더에 저장된 스프레드시트 결과에서 데이터를 내보냅니다.
- 형광 분석 A: 1 채널, 형광 분석 B: 동일한 구획 및 형광 분석 C의 2 채널: 2 개의 구획에 2 개의 채널
7. 프로토콜 실행
- 실행에서 한 블록을 처리하려면 선택한 블록과 블록 선택 폴더 사이의 연결을 제거합니다. 원하는 블록을 1등급에 놓습니다. 원하는 블록의 왼쪽 위 모서리에서 폴더오른쪽에 있는 링크를 직접 클릭합니다. 표시되는 열린 대화 상자에서 병합된 이미지가 포함된 폴더를 두 번 클릭합니다. 그런 다음 열기를 클릭합니다. 실행 실행을 클릭하여 워크플로를 시작합니다. 중지 버튼을 클릭하여 처리를 중지할 수 있습니다.
- 실행에서 다른 블록을 처리하려면 선택한 블록의 연결을 블록 선택 폴더(블록 1)로 유지합니다. 해당 순위에서 워크플로를 잘 처리할 수 있는지 확인합니다. 예를 들어 특정 블록에서 처리하려면 분할된 개체가 필요한 경우 세분화 블록이 이전에 처리해야 합니다. 워크플로를 실행하기 전에 사용하지 않은 블록을 제거하고 새 프로토콜을 다른 이름으로 저장합니다.
- 실행 실행을 클릭하여 워크플로를 시작합니다. 열린 대화 상자가 나타나면 병합된 이미지가 포함된 폴더를 두 번 클릭합니다. 그런 다음 열기를 클릭합니다. 워크플로가 자동으로 실행됩니다. 필요한 경우 중지 버튼을 클릭하여 처리를 중지합니다.
- 처리가 끝나면 실행된 워크플로가 오른쪽 아래 모서리에 성공적으로 나타나고 모든 블록에 녹색기호(그림 2b)로플래그가 지정된지 확인합니다. 그렇지 않은 경우 오류 기호를 표시하는 블록 과 내부 상자는 올바른 요소를나타냅니다(그림 2b).
참고: 워크플로를 성공적으로 실행한 후에는 새 실행을 직접 시작할 수 없으며 워크플로를 다시 처리하려면 "처리할 준비가 됨"이라는 기호로 하나 이상의 블록에 플래그를 지정해야 합니다. 블록의 상태를 변경하려면 이 블록 내부의 두 상자 사이의 링크를 삭제하고 다시 만들거나 프로토콜을 닫고 다시 엽니다. 처리 중에 오류가 발생하면 새 실행을 직접 시작할 수 있습니다. 새 실행 중에 파이프라인의 모든 블록이 처리됩니다.
결과
설명된 모든 분석은 64비트 버전의 Java와 함께 작동하는 표준 랩톱(2.80GHz의 쿼드 코어 프로세서)에서 수행되었습니다. 랜덤 액세스 메모리는 분석할 이미지의 양과 해상도에 따라 고려해야 할 중요한 매개 변수입니다. 자바의 32 비트 버전을 사용 하 여 약 1300 MB에 메모리를 제한, 빅 데이터 분석에 적합 할 수 있는 반면, 반면 64 비트 버전 아이시에 할당 된 메모리를 증가 수 있습니다.
토론
형광 세포 이미지의 분석에 사용할 수있는 무료 소프트웨어 도구의 증가가 사용할 수 있습니다. 사용자는 문제가 있는 복잡성, 이미지 처리에 대한 지식 및 분석에 지출하려는 시간에 따라 적절한 소프트웨어를 올바르게 선택해야 합니다. 얼음, 셀 프로파일러, 또는 ImageJ / 피지는 유용성과 기능 모두를 결합하는 강력한도구입니다 3. Icy는 명확한 그래픽 사용자 인터페이스(GUI)를...
공개
저자는 공개 할 것이 없습니다.
감사의 말
G.H.는 미니슈르 델레구에 아 라 레쉐 에 오 테크놀로지스의 대학원 펠로우십에 의해 지원되었다. L.H.는 인스티투트 드 칸세로로지 드 로렌(ICL)의 대학원 펠로우십의 지원을 받았고, Q.T.는 프랑스 국립연구청(ANR)이 감독하는 공공 보조금으로 지원받았고, 두 번째 "인베스트먼트 다베니르" 프로그램 파이트-HF(참조: ANR-15-RHU40)의 일환으로 지원되었다. 이 작품은 CNRS와 유니버시테 드 로렌 (UMR 7365)에 의해 투자되었다.
자료
| Name | Company | Catalog Number | Comments |
| 16% Formaldehyde solution (w/v) methanol free | Thermo Fisher Scientific | 28908 | to fix the cells |
| Alexa Fluor 488 of goat anti-rabbit | Thermo Fisher Scientific | A-11008 | fluorescent secondary antibody |
| Alexa Fluor 555 of goat anti-mouse | Thermo Fisher Scientific | A-21425 | fluorescent secondary antibody |
| Alexa Fluor 555 Phalloidin | Thermo Fisher Scientific | A34055 | fluorescent secondary antibody |
| Bovine serum albumin standard (BSA) | euromedex | 04-100-812-E | |
| DMEM | Sigma-Aldrich | D5796-500ml | cell culture medium |
| Duolink In Situ Mounting Medium with DAPI | Sigma-Aldrich | DUO82040-5ML | mounting medium |
| Human: HeLa S3 cells | IGBMC, Strasbourg, France | cell line used to perform the experiments | |
| Hydrogen peroxide solution 30% (H2O2) | Sigma-Aldrich | H1009-100ml | used as a stressing agent |
| Lipofectamine 2000 Reagent | Thermo Fisher Scientific | 11668-019 | transfection reagent |
| Mouse monoclonal anti-coilin | abcam | ab11822 | Coilin-specific antibody |
| Nikon Optiphot-2 fluorescence microscope | Nikon | epifluoresecence microscope | |
| Opti-MEM I Reduced Serum Medium | Thermo Fisher Scientific | 31985062 | transfection medium |
| PBS pH 7.4 (10x) | gibco | 70011-036 | to wash the cells |
| Rabbit polyclonal anti-53BP1 | Thermo Fisher Scientific | PA1-16565 | 53BP1-specific antibody |
| Rabbit polyclonal anti-EDC4 | Sigma-Aldrich | SAB4200114 | EDC4-specific antibody |
| Triton X-100 | Roth | 6683 | to permeabilize the cells |
참고문헌
- Möckl, L., Lamb, D. C., Bräuchle, C. Super-resolved fluorescence microscopy: Nobel Prize in Chemistry 2014 for Eric Betzig, Stefan Hell, and William E. Moerner. Angewandte Chemie. 53 (51), 13972-13977 (2014).
- Meijering, E., Carpenter, A. E., Peng, H., Hamprecht, F. A., Olivo-Marin, J. -. C. Imagining the future of bioimage analysis. Nature Biotechnology. 34 (12), 1250-1255 (2016).
- Wiesmann, V., Franz, D., Held, C., Münzenmayer, C., Palmisano, R., Wittenberg, T. Review of free software tools for image analysis of fluorescence cell micrographs. Journal of Microscopy. 257 (1), 39-53 (2015).
- de Chaumont, F., et al. Icy: an open bioimage informatics platform for extended reproducible research. Nature Methods. 9 (7), 690-696 (2012).
- Girish, V., Vijayalakshmi, A. Affordable image analysis using NIH Image/ImageJ. Indian J Cancer. 41 (1), 47 (2004).
- Zaitoun, N. M., Aqel, M. J. Survey on image segmentation techniques. Procedia Computer Science. 65, 797-806 (2015).
- . MetaMorph Microscopy Automation and Image Analysis Software Available from: https://www.moleculardevices.com/products/cellular-imaging-systems/acquisition-and-analysis-software/metamorph-microscopy (2018)
- . NIS-Elements Imaging Software Available from: https://www.nikon.com/products/microscope-solutions/lineup/img_soft/nis-element (2014)
- Meyer, F., Beucher, S. Morphological segmentation. Journal of Visual Communication and Image Representation. 1 (1), 21-46 (1990).
- Schieber, M., Chandel, N. S. ROS Function in Redox Signaling and Oxidative Stress. Current Biology. 24 (10), 453-462 (2014).
- D'Autréaux, B., Toledano, M. B. ROS as signalling molecules: mechanisms that generate specificity in ROS homeostasis. Nature Reviews. Molecular Cell Biology. 8 (10), 813-824 (2007).
- Davalli, P., Mitic, T., Caporali, A., Lauriola, A., D'Arca, D. ROS, Cell Senescence, and Novel Molecular Mechanisms in Aging and Age-Related Diseases. Oxidative Medicine and Cellular Longevity. 2016, 3565127 (2016).
- Disher, K., Skandalis, A. Evidence of the modulation of mRNA splicing fidelity in humans by oxidative stress and p53. Genome. 50 (10), 946-953 (2007).
- Takeo, K., et al. Oxidative stress-induced alternative splicing of transformer 2β (SFRS10) and CD44 pre-mRNAs in gastric epithelial cells. American Journal of Physiology - Cell Physiology. 297 (2), 330-338 (2009).
- Seo, J., et al. Oxidative Stress Triggers Body-Wide Skipping of Multiple Exons of the Spinal Muscular Atrophy Gene. PLOS ONE. 11 (4), 0154390 (2016).
- Will, C. L., Luhrmann, R. Spliceosome Structure and Function. Cold Spring Harbor Perspectives in Biology. 3 (7), 003707 (2011).
- Ljosa, V., Sokolnicki, K. L., Carpenter, A. E. Annotated high-throughput microscopy image sets for validation. Nature Methods. 9 (7), 637-637 (2012).
- Wang, Q., et al. Cajal bodies are linked to genome conformation. Nature Communications. 7, (2016).
- Carpenter, A. E., et al. CellProfiler: image analysis software for identifying and quantifying cell phenotypes. Genome Biology. 7, 100 (2006).
- McQuin, C., et al. CellProfiler 3.0: Next-generation image processing for biology. PLoS Biology. 16 (7), 2005970 (2018).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유