Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Methods Article
Unterstrukturanalysator: Ein benutzerfreundlicher Workflow für die schnelle Erforschung und genaue Analyse von Zellkörpern in Fluoreszenzmikroskopiebildern
In diesem Artikel
Zusammenfassung
Wir präsentieren einen frei verfügbaren Workflow für die schnelle Erforschung und genaue Analyse von Zellkörpern in bestimmten Zellkompartimenten in Fluoreszenzmikroskopiebildern. Dieser benutzerfreundliche Workflow wurde auf der Open-Source-Software Icy entwickelt und verwendet auch ImageJ-Funktionalitäten. Die Pipeline ist ohne Kenntnisse in der Bildanalyse erschwinglich.
Zusammenfassung
Das letzte Jahrzehnt war durch Durchbrüche in Fluoreszenzmikroskopie-Techniken gekennzeichnet, die durch die Verbesserung der räumlichen Auflösung, aber auch in Live-Zell-Bildgebung und Hochdurchsatzmikroskopie-Techniken veranschaulicht wurden. Dies führte zu einer stetigen Zunahme der Menge und Komplexität der Mikroskopiedaten für ein einzelnes Experiment. Da die manuelle Analyse von Mikroskopiedaten sehr zeitaufwändig, subjektiv und quantitative Analysen verbietet, wird die Automatisierung der Biobildanalyse nahezu unvermeidbar. Wir haben einen Informatik-Workflow namens Substructure Analyzer entwickelt, um die Signalanalyse in Biobildern aus der fluoreszierenden Mikroskopie vollständig zu automatisieren. Dieser Workflow wird auf der benutzerfreundlichen Open-Source-Plattform Icy entwickelt und durch Funktionalitäten von ImageJ ergänzt. Es umfasst die Vorverarbeitung von Bildern zur Verbesserung des Signal-Rausch-Verhältnisses, die individuelle Segmentierung von Zellen (Erkennung von Zellgrenzen) und die Erkennung/Quantifizierung von Zellkörpern, die in bestimmten Zellkompartimen angereichert sind. Der Hauptvorteil dieses Workflows besteht darin, Benutzern komplexe Bio-Imaging-Funktionalitäten ohne Bildanalyse-Expertise über eine benutzerfreundliche Oberfläche vorzuschlagen. Darüber hinaus ist es hochmodular und an mehrere Themen angepasst, von der Charakterisierung der kern-zytoplasmatischen Translokation bis hin zur vergleichenden Analyse verschiedener Zellkörper in verschiedenen zellulären Substrukturen. Die Funktionalität dieses Workflows wird durch die Untersuchung der Cajal (gewickelt) Körper unter oxidativen Stressbedingungen (OS) veranschaulicht. Daten aus der Fluoreszenzmikroskopie zeigen, dass ihre Integrität in menschlichen Zellen wenige Stunden nach der Induktion des Betriebssystems beeinträchtigt wird. Dieser Effekt ist durch eine Abnahme der Coilin-Kernbildung in charakteristische Cajal-Körper gekennzeichnet, verbunden mit einer nukleoplasmatischen Umverteilung von Coilin in eine erhöhte Anzahl kleinerer Brennpunkte. Die zentrale Rolle von Coilin beim Austausch zwischen CB-Komponenten und dem umgebenden Nukleoplasma legt nahe, dass OS-induzierte Umverteilung von Coilin die Zusammensetzung und Die Funktionalität von Cajal Bodies beeinflussen könnte.
Einleitung
Die Lichtmikroskopie und insbesondere die Fluoreszenzmikroskopie sind robuste und vielseitige Techniken, die in den biologischen Wissenschaften häufig eingesetzt werden. Sie geben Zugang zur präzisen Lokalisierung verschiedener Biomoleküle wie Proteine oder RNA durch ihre spezifische fluoreszierende Kennzeichnung. Das letzte Jahrzehnt war geprägt von rasanten Fortschritten in der Mikroskopie und Bildgebungstechnologien, wie der Nobelpreis für Chemie 2014 zeigt, der Eric Betzig, Stefan W. Hell und William E. Moerner für die Entwicklung der superaufgelösten Fluoreszenzmikroskopie (SRFM)1vergibt. SFRM umgeht die Beugungsgrenze der traditionellen optischen Mikroskopie, um sie in die Nanodimension zu bringen. Die Verbesserung von Techniken wie Live-Imaging oder Screening-Ansätzen mit hohem Durchsatz erhöht auch die Menge und Komplexität der daten, die für jedes Experiment behandelt werden sollen. Meistens sind Forscher mit hohen heterogenen Zellpopulationen konfrontiert und wollen Phänotypen auf einzelliger Ebene analysieren.
Zunächst wurden Analysen wie die Brennweite mit dem Auge durchgeführt, was von einigen Forschern bevorzugt wird, da es eine vollständige visuelle Kontrolle über den Zählprozess bietet. Die manuelle Analyse solcher Daten ist jedoch zu zeitaufwändig, führt zu Einer Variabilität zwischen den Beobachtern und bietet keinen Zugriff auf komplexere Merkmale, so dass computergestützte Ansätze weit verbreitet und nahezu unvermeidbar werden2. Bioimage-Informatik-Methoden erhöhen die Effizienz der Datenanalyse erheblich und sind frei von der unvermeidbaren Bedienersubjektivität und potenziellen Verzerrung der manuellen Zählanalyse. Die gestiegene Nachfrage in diesem Bereich und die Verbesserung der Computerleistung führten zur Entwicklung einer vielzahl von Bildanalyseplattformen. Einige von ihnen sind frei verfügbar und ermöglichen den Zugriff auf verschiedene Tools, um Analysen mit PCs durchzuführen. Eine Klassifizierung von Open-Access-Tools wurde vor kurzem3 eingeführt und präsentiert Icy4 als leistungsstarke Software, die Benutzerfreundlichkeit und Funktionalität kombiniert. Darüber hinaus hat Eis den Vorteil, mit ImageJ zu kommunizieren.
Für Anwender ohne Bildanalyse-Expertise besteht das Haupthindernis darin, das passende Werkzeug entsprechend den problematischen und korrekt abgestimmten Parametern auszuwählen, die oft nicht gut verstanden werden. Darüber hinaus sind die Rüstzeiten oft lang. Icy schlägt eine benutzerfreundliche Point-and-Click-Schnittstelle namens "Protokolle" vor, um den Workflow zu entwickeln, indem einige Plugins kombiniert werden, die in einer erschöpfenden Sammlung gefunden wurden4. Der flexible modulare Aufbau und die Point-and-Click-Schnittstelle machen die Einrichtung einer Analyse für Nicht-Programmierer möglich. Hier stellen wir einen Workflow namens Substructure Analyzervor, der in der Icy-Schnittstelle entwickelt wurde und dessen Funktion darin besteht, fluoreszierende Signale in bestimmten zellulären Kompartimenten zu analysieren und verschiedene Merkmale wie Helligkeit, Brennziffer, Brennpunktegröße und räumliche Verteilung zu messen. Dieser Workflow befasst sich mit mehreren Fragen wie der Quantifizierung der Signaltranslokation, der Analyse transfizierter Zellen, die einen fluoreszierenden Reporter exdrücken, oder der Analyse von Brennpunkten aus verschiedenen zellulären Unterstrukturen in einzelnen Zellen. Es ermöglicht die gleichzeitige Verarbeitung mehrerer Bilder, und Ausgabeergebnisse werden in ein von Registerkarten getrenntes Arbeitsblatt exportiert, das in häufig verwendeten Tabellenkalkulationsprogrammen geöffnet werden kann.
Die Substructure Analyzer-Pipeline ist in Abbildung 1dargestellt. Erstens werden alle Bilder, die in einem angegebenen Ordner enthalten sind, vorverarbeitet, um ihr Signal-Rausch-Verhältnis zu verbessern. Dieser Schritt erhöht die Effizienz der folgenden Schritte und verringert die Laufzeit. Dann werden die Regionen des Interesses (ROIs), die den Bildbereichen entsprechen, in denen das Fluoreszenzsignal nachgewiesen werden soll, identifiziert und segmentiert. Schließlich wird das Fluoreszenzsignal analysiert, und die Ergebnisse werden in ein durch Tabstopps getrenntes Arbeitsblatt exportiert.
Die Objektsegmentierung (Erkennung von Grenzen) ist der anspruchsvollste Schritt in der Bildanalyse, und ihre Effizienz bestimmt die Genauigkeit der resultierenden Zellmessungen. Die ersten Objekte, die in einem Bild identifiziert werden (primäre Objekte genannt) sind oft Kerne aus DNA-befleckten Bildern (DAPI- oder Hoechst-Färbung), obwohl primäre Objekte auch ganze Zellen, Perlen, Flecken, Tumoren oder was auch immer gefärbte Objekte sein können. In den meisten biologischen Bildern berühren sich Zellen oder Kerne oder überlappen sich, wodurch die einfachen und schnellen Algorithmen versagen. Bis heute kann kein universeller Algorithmus eine perfekte Segmentierung aller Objekte durchführen, vor allem, weil ihre Eigenschaften (Größe, Form oder Textur) die Effizienz der Segmentierung5modulieren. Die Segmentierungstools, die üblicherweise mit Mikroskopiesoftware vertrieben werden (z. B. die MetaMorph Imaging Software von Molecular Devices6oder die NIS-Elements Advances Research Software von Nikon7), basieren im Allgemeinen auf Standardtechniken wie Korrelationsabgleich, Schwellenwertierung oder morphologischen Operationen. Obwohl diese übergeneralisierten Methoden in grundlegenden Systemen effizient sind, stellen sie schnell Einschränkungen dar, wenn sie in anspruchsvolleren und spezifischeren Kontexten eingesetzt werden. Tatsächlich ist die Segmentierung sehr empfindlich gegenüber experimentellen Parametern wie Zelltyp, Zelldichte oder Biomarkern und erfordert häufig eine wiederholte Anpassung für einen großen Datensatz. Der Substructure Analyzer-Workflow integriert sowohl einfache als auch anspruchsvollere Algorithmen, um verschiedene Alternativen vorzuschlagen, die an die Bildkomplexität und die Benutzeranforderungen angepasst sind. Sie schlägt insbesondere den markerbasierten Wasserscheidealgorithmus8 für stark gruppierte Objekte vor. Die Effizienz dieser Segmentierungsmethode hängt von der Auswahl der einzelnen Marker für jedes Objekt ab. Diese Marker werden die meiste Zeit manuell ausgewählt, um korrekte Parameter für die vollständige Segmentierung zu erhalten, was sehr zeitaufwändig ist, wenn Benutzer mit einer hohen Anzahl von Objekten konfrontiert sind. Substructure Analyzer schlägt eine automatische Erkennung dieser Marker vor und bietet so einen hocheffizienten Segmentierungsprozess. Die Segmentierung ist meistens der einschränkende Schritt der Bildanalyse und kann die Verarbeitungszeit je nach Auflösung des Bildes, der Anzahl der Objekte pro Bild und der Ebene des Clusterings von Objekten erheblich ändern. Typische Pipelines benötigen auf einem Standarddesktopcomputer einige Sekunden bis 5 Minuten pro Bild. Die Analyse komplexerer Bilder kann einen leistungsfähigeren Computer und einige Grundlegendes in der Bildanalyse erfordern.
Die Flexibilität und Funktionalität dieses Workflows wird anhand verschiedener Beispiele in den repräsentativen Ergebnissen veranschaulicht. Die Vorteile dieses Workflows werden vor allem durch die Untersuchung nuklearer Unterstrukturen unter oxidativen Stressbedingungen (OS) deutlich. OS entspricht einem Ungleichgewicht der Redoxhomöostase zugunsten von Oxidationsmitteln und ist mit hohen Konzentrationen an reaktiven Sauerstoffspezies (ROS) verbunden. Da ROS als Signalmoleküle fungieren, wirken sich Veränderungen in ihrer Konzentration und subzellulären Lokalisation positiv oder negativ auf eine Vielzahl von Signalwegen und Netzwerken aus, die physiologische Funktionen regulieren, einschließlich Signaltransduktion, Reparaturmechanismen, Genexpression, Zelltod und Proliferation9,10. OS ist somit direkt an verschiedenen Pathologien (neurodegenerative und herzkarvaskuläre Erkrankungen, Krebs, Diabetes, etc.), aber auch zelluläreAlterung beteiligt. Daher stellt die Entschlüsselung der Folgen von OS für die Organisation und Funktion der menschlichen Zelle einen entscheidenden Schritt im Verständnis der Rolle von OS beim Beginn und der Entwicklung menschlicher Pathologien dar. Es wurde festgestellt, dass OS die Genexpression reguliert, indem es die Transkription durch mehrere Transkriptionsfaktoren moduliert (p53, Nrf2, FOXO3A)11, aber auch durch die Beeinflussung mehrerer ko- und posttranskriptionaler Prozesse wie alternatives Spleißen (AS) von Pre-RNAs12,13,14. Das alternative Spleißen von primären Kodierungs- und Nicht-Codierungs-Transkripten ist ein wesentlicher Mechanismus, der die Kodierungskapazität der Genome durch die Produktion von Transkript-Isoformen erhöht. AS wird von einem riesigen Ribonukleoprotein-Komplex namens Spliceoom durchgeführt, der fast 300 Proteine und 5 U-reiche kleine nukleare RNAs (UsnRNAs)15enthält. Spliceosome Montage und AS werden in den Zellen streng kontrolliert und einige Schritte der spliceosome Reifung treten in membranlosen Kernräumen namens Cajal Bodies. Diese kerntechnischen Unterstrukturen zeichnen sich durch die Dynamik ihrer Struktur und ihre Zusammensetzung aus, die hauptsächlich durch multivalente Wechselwirkungen ihrer RNA- und Proteinkomponenten mit dem Coilin-Protein erfolgen. Die Analyse von Tausenden von Zellen mit dem Substructure Analyzer-Workflow ermöglichte die Charakterisierung nie beschriebener Os-Effekte auf Cajal Bodies. Tatsächlich deuten die erhaltenen Daten darauf hin, dass OS die Keimbildung von Cajal Bodies modifiziert und eine nukleoplasmatische Umverteilung des Coilin-Proteins in zahlreiche kleinere Kernbrennpunkte induzieren. Eine solche Änderung der Struktur von Cajal Bodies könnte die Reifung des Spliceooms beeinflussen und an der AS-Modulation durch OS teilnehmen.
Protokoll
HINWEIS: Benutzerfreundliche Tutorials sind auf der Website von Icy http://icy.bioimageanalysis.orgverfügbar.
1. Herunterladen von Eis und dem Substructure Analyzer-Protokoll
- Laden Sie Icy von der Icy-Website (http://icy.bioimageanalysis.org/download) herunter und laden Sie das Substructure Analyzer-Protokoll herunter: http://icy.bioimageanalysis.org/protocols?sort=latest.
HINWEIS: Wenn Sie ein 64-Bit-Betriebssystem verwenden, sollten Sie die 64-Bit-Version von Java verwenden. Diese Version ermöglicht die Erhöhung des Speichers, der Icy zugewiesen ist(Einstellungen | Allgemeines | Max. Speicher).
2. Öffnen des Protokolls
- Öffnen Sie Eis und klicken Sie im Menü "Menüleiste" auf Extras.
- Klicken Sie auf Protokolle, um die Protokoll-Editor-Schnittstelle zu öffnen.
- Klicken Sie auf Laden und öffnen Sie den Protokoll-Substructure Analyzer. Das Laden des Protokolls kann einige Sekunden dauern. Stellen Sie sicher, dass das Öffnen des Protokolls abgeschlossen ist, bevor Sie es verwenden.
HINWEIS: Der Workflow besteht aus 13 allgemeinen Blöcken, die in Abbildung 2adargestellt sind. Jeder Block funktioniert als Pipeline, die aus mehreren Feldern besteht, die bestimmte Teilaufgaben ausführen.
3. Interaktion mit dem Workflow auf Eis
HINWEIS: Jeder Block oder jedes Feld ist nummeriert und hat einen bestimmten Rang innerhalb des Workflows (Abbildung 2b). Durch Anklicken dieser Nummer wird die nächstgelegene Position zum ersten dem ausgewählten Block/Feld zugewiesen, dann wird die Position der anderen Blöcke/Boxen neu organisiert. Beachten Sie bei der Vorbereitung des Workflows die richtige Reihenfolge der Blöcke. Beispielsweise benötigt der Spot Detector-Block vordefinierte ROIs, sodass Segmentierungsblöcke vor Spot Detector-Blöcken ausgeführt werden müssen. Ändern Sie die Position der Felder nicht. Verwenden Sie "." nicht im Namen des Bildes.
- Durch Klicken auf das symbol "obere linke Ecke" wird der Block reduziert, erweitert, vergrößert, verengt oder entfernt (Abbildung 2b).
- Jede Pipeline des Workflows zeichnet sich durch ein Netz von Boxen aus, die über deren Ein- und Ausgabe verbunden sind (Abbildung 2b). Um eine Verbindung herzustellen, klicken Sie auf Ausgabe und halten Sie, bis der Cursor eine Eingabe erreicht. Verbindungen können entfernt werden, indem Sie auf das Ausgabe-Tag klicken.
4. Zusammenführen der Kanäle eines Bildes
- Verwenden Sie den Block Merge Channels, um zusammengeführte Bilder zu generieren. Benennen Sie die Dateien bei Bedarf so um, dass Sequenzen, die zusammengeführt werden sollen, das Präfix des gleichen Namens haben, gefolgt von einem separaten Trennzeichen. Sequenzen einzelner Kanäle aus einem Bild A werden beispielsweise benannt: ImageA_red, ImageA_blue.
HINWEIS: Verwenden Sie für das Trennzeichen keine Zeichen, die bereits im Namen des Bildes vorhanden sind. - Erstellen Sie im gleichen Ordner einen neuen Ordner pro Kanal, der zusammengeführt werden soll. Um beispielsweise rote, grüne und blaue Kanäle zusammenzuführen, erstellen Sie 3 Ordner, und speichern Sie die entsprechenden Sequenzen in diesen Ordnern.
- Verwenden Sie nur den Block Merge Channels, entfernen Sie die anderen Blöcke, und speichern Sie das Protokoll als Merge Channels.
- Greifen Sie auf die Felder zu, um Parameter festzulegen. Füllen Sie für jeden Kanal die Felder Kanalnummer X (Felder 1, 5 oder 9), Ordnerkanalnummer X (Felder 2, 6 oder 10), Trennkanalnummer X (Felder 3, 7 oder 11) und Colormap-Kanal nb X (Felder 4, 8 und 12).
HINWEIS: Diese Felder sind horizontal durch vier gruppiert, jede Zeile entspricht demselben Kanal. In jeder Zeile ist auch ein Display (Felder 23, 24 oder 25) verfügbar, um die Reihenfolge des entsprechenden Kanals direkt zu visualisieren.- Wählen Sie im Feld Kanalnummer Xaus, welchen Kanal extrahiert werden soll (in klassischen RGB-Bildern, 0=Rot, 1=Grün, 2=Blau). Der Benutzer greift schnell auf die verschiedenen Kanäle eines Bildes im Inspector-Fenster von Eis, in der Registerkarte Sequenz. Schreiben Sie den Wert des kleinsten Kanals in der oberen Zeile und den höchsten in der unteren Zeile.
- Schreiben Sie in das Feld Ordnerkanalnummer Xden Namen des Ordners, der Bilder von Kanal X enthält.
- Schreiben Sie in das Feld Trennkanalnummer Xdas Trennzeichen, das für den Namen des Bildes verwendet wird (im vorherigen Beispiel: "_red", "_green" und "_blue").
- Geben Sie im Feld Colormap-Kanal nb Xmit einer Zahl an, welches Colormap-Modell verwendet werden soll, um den entsprechenden Kanal in Icy zu visualisieren. Die verfügbaren Farbkarten sind auf der Registerkarte Sequenz des Inspektorfensters sichtbar.
- Schreiben Sie in das Feld Format der zusammengeführten Bilder (Feld 28) die Erweiterung, um zusammengeführte Bilder zu speichern: .tif, .gif, .jpg, .bmp oder .png.
HINWEIS: Um nur 2 Kanäle zusammenzuführen, füllen Sie nicht die vier Felder, die dem dritten Kanal entsprechen.
- Klicken Sie in der oberen linken Ecke des Blocks Kanäle zusammenführen auf den Link direkt rechts von Folder. Doppelklicken Sie im dialogoffenen Dialogfeld auf den Ordner mit Sequenzen des ersten Kanals, der im Feld Ordnerkanal Nummer 1 (Feld 2) definiert wurde. Klicken Sie dann auf Öffnen.
- Führen Sie das Protokoll aus, indem Sie auf den schwarzen Pfeil in der oberen linken Ecke des Merge Channels-Blocks klicken (weitere Details finden Sie in Teil 7). Zusammengeführte Bilder werden in einem Mergeordner im selben Verzeichnis wie die Ordner der einzelnen Kanäle gespeichert.
- Greifen Sie auf die Felder zu, um Parameter festzulegen. Füllen Sie für jeden Kanal die Felder Kanalnummer X (Felder 1, 5 oder 9), Ordnerkanalnummer X (Felder 2, 6 oder 10), Trennkanalnummer X (Felder 3, 7 oder 11) und Colormap-Kanal nb X (Felder 4, 8 und 12).
5. Segmentierung der Interessengebiete
HINWEIS: Substructure Analyzer integriert sowohl einfache als auch anspruchsvollere Algorithmen, um verschiedene Alternativen vorzuschlagen, die an die Bildkomplexität und die Benutzeranforderungen angepasst sind.
- Wählen Sie den angepassten Block aus.
- Wenn sich Objekte nicht berühren oder der Benutzer gruppierte Objekte nicht einzeln unterscheiden muss, verwenden Sie den Block Segmentierung A: Nicht gruppierte Objekte.
- Wenn sich Objekte nicht berühren, aber einige nahe sind, verwenden Sie den Block Segmentierung B: Schlecht gruppierte Objekte.
- Verwenden Sie für Objekte mit einer hohen Clusterebene und einer konvexen Form den Block Segmentierung C: Gruppierte Objekte mit konvexen Formen.
- Wenn Objekte eine hohe Clusterebene aufweisen und unregelmäßige Formen aufweisen, verwenden Sie den Block Segmentierung D: Gruppierte Objekte mit unregelmäßigen Formen.
- Verwenden Sie den Block Segmentierung E: Clustered Cytoplasma, um berührende Zytoplasmen einzeln unter Verwendung segmentierter Kerne als Marker zu segmentieren. Dieser Block muss unbedingt segmentierte Kerne verarbeiten.
ANMERKUNG: Blöcke, die unabhängig für den primären Objektsegmentierungsprozess angepasst sind, sodass mehrere Blöcke in derselben Ausführung verwendet werden können, um ihre Effizienz für eine bestimmte Unterstruktur zu vergleichen oder verschiedene Arten von Unterstrukturen zu segmentieren. Wenn die Clusterebene innerhalb desselben Satzes von Bildern heterogen ist, verarbeiten Sie kleine und stark gruppierte Objekte separat in den angepassten Blöcken.
- Verknüpfen Sie die Ausgabe0 (Datei) des Blocks Ordner auswählen mit der Ordnereingabe des gewählten Segmentierungsblocks.
- Legen Sie die Parameter des gewählten Segmentierungsblocks fest.
- Segmentierung A: Nicht gruppierte Objekte und Segmentierung C: Gruppierte Objekte mit konvexen Formen
- Legen Sie im Box-Kanal-Signal (Box 1) den Kanal der Objekte auf Segment fest.
- Optional erhöhen Sie in Kasten Gaußsche Filter (Box 2) die X- und Y-Sigma-Werte, wenn das Signal in Objekten heterogen ist. Der Gaußsche Filter glättet Texturen, um gleichmäßigere Regionen zu erhalten und erhöht die Geschwindigkeit und Effizienz der Kernsegmentierung. Je kleiner die Objekte, desto niedriger ist der Sigma-Wert. Vermeiden Sie hohe Sigma-Werte. Legen Sie die Standardwerte auf 0 fest.
- Legen Sie in Feld HK-Means (Feld 3) den Parameter Intensitätsklassen und die ungefähren minimalen und maximalen Größen (in Pixel) der zu erkennenden Objekte fest.
HINWEIS: Für Intensitätsklassen klassifiziert ein Wert von 2 Pixel in 2 Klassen: Hintergrund und Vordergrund. Es wird somit angepasst, wenn der Kontrast zwischen den Objekten und dem Hintergrund hoch ist. Wenn Vordergrundobjekte unterschiedliche Intensitäten haben oder der Kontrast zum Hintergrund gering ist, erhöhen Sie die Anzahl der Klassen. Die Standardeinstellung ist 2. Die Objektgröße kann schnell ausgewertet werden, indem ein ROI manuell um das Objekt von Interesse gezeichnet wird. Die Größe des ROI (Interior in Pixel) wird direkt auf dem Bild angezeigt, wenn er mit dem Cursor zeigt, oder kann im ROI-Statistikfenster aufgerufen werden (öffnen Sie ihn über die Suchleiste). Optimale Parameter erfassen jedes Vordergrundobjekt in einem einzigen ROI. Sie können manuell in Icy (Erkennung und Nachverfolgung | HK-Mittel). - Optimieren Sie im Feld Aktive Konturen (Feld 4) die Erkennung von Objektgrenzen. Umfangreiche Dokumentation für dieses Plugin ist online verfügbar: http://icy.bioimageanalysis.org/plugin/Active_Contours. Korrekte Parameter können auch manuell in Icy(Erkennung und Nachverfolgung | Aktive Konturen).
- Während des Vorgangs wird automatisch ein Ordner erstellt, um Bilder segmentierter Objekte zu speichern. Benennen Sie diesen Ordner im Feld Text (Feld 6) (z. B. Segmentierte Kerne). Um das Format zum Speichern von Bildern segmentierter Objekte (Tiff, Gif, Jpeg, BMP, PNG) festzulegen, füllen Sie das Feldformat von Bildern segmentierter Objekteaus. Der Ordner wird in dem Ordner erstellt, der zusammengeführte Bilder enthält.
- Führen Sie den Workflow aus (Details finden Sie in Teil 7).
- Segmentierung B: Schlecht gruppierte Objekte
- Führen Sie die gleichen Schritte wie in 5.3.1 aus, um Parameter von Boxen Kanalsignal, HK-Means, Active Contours, Extension zum Speichern segmentierter Objekte und Text festzulegen (Boxen sind nicht die gleichen wie in Schritt 5.3.1).
- Legen Sie im Feld Anruf IJ Plugin (Feld 4) den Rolling-Parameter fest, um die Hintergrundsubtraktion zu steuern. Legen Sie diesen Parameter auf mindestens die Größe des größten Objekts fest, das nicht Teil des Hintergrunds ist. Das Verringern dieses Werts erhöht die Hintergrundentfernung, kann aber auch den Verlust des Vordergrundsignals verursachen.
- Verbessern Sie im Feld Adaptivehistogrammentzerrung (Feld 6) die Kontraste zwischen den Vordergrundobjekten und dem Hintergrund. Durch das Erhöhen der Neigung werden mehr kontrastreiche Sequenzen angezeigt.
- Führen Sie den Workflow aus (Details finden Sie in Teil 7).
- Segmentierung D: Gruppierte Objekte mit unregelmäßigen Formen
ANMERKUNG: Für jedes Bild gelten drei verschiedene Segmentierungsmethoden:HK-means Clusteringkombiniert mit derActive Contours-Methodeangewendet wird. Dann wird dieklassischer Wasserscheidealgorithmus(mit der euklidischen Entfernungskarte) wird auf zuvor falsch segmentierte Objekte angewendet. Schließlich wird einMarker-basierter Wassereinzugsgebietsalgorithmusverwendet wird. Nur HK-Mittel und markerbasierte Wassereinzugsgebietsmethoden müssen von der Nutzer eingreifen. Für beide Methoden können für alle Bilder (vollautomatische Version) die gleichen Parameter angewendet oder für jedes Bild geändert werden (halbautomatische Version). Wenn der Benutzer in diesen Segmentierungsmethoden nicht geschult ist, wird die halbautomatische Verarbeitung dringend empfohlen. Bei der Bearbeitung dieses Blocks ist ein manuelles Eingreifen erforderlich. Wenn eine Segmentierungsmethode abgeschlossen ist, muss der Benutzer falsch segmentierte Objekte vor dem Beginn der nächsten Segmentierungsmethode manuell entfernen. Erfolgreich segmentierte Objekte werden gespeichert und im nächsten Schritt nicht berücksichtigt. Dieser Block muss mit dem Block verbunden werdenClustered/Heterogene Shapes primäre Objekte Segmentierung DialogFeldkorrekt zu funktionieren.- Laden Sie die ImageJ-Kollektion MorphoLibJ auf https://github.com/ijpb/MorphoLibJ/releases herunter. Die MorphoLibJ 1.4.0 Version wird in diesem Protokoll verwendet. Platzieren Sie die Datei MorphoLibJ_-1.4.0.jar im Ordner eis/ij/plugins. Weitere Informationen zum Inhalt dieser Sammlung finden Sie auf https://imagej.net/MorphoLibJ.
- Führen Sie die gleichen Schritte wie in Schritt 5.3.1 aus, um Parameter von Boxen Kanalsignal, Gaußsche Filter, Aktive Konturen, Erweiterung, um segmentierte Objekte und Textzu speichern . Die Boxensindsind sind nicht die gleichen wie in Schritt 5.3.1.
- Legen Sie die Parameter des Feldes Adaptiver Histogrammausgleich fest (siehe Schritt 5.3.2.3).
- Um Den Hintergrund subtrahierenzu aktivieren, schreiben Sie ja in Apply Subtrahierhintergrund? (Feld 5). Andernfalls, schreiben Sie Nein. Wenn das Plugin aktiviert ist, setzen Sie den Rolling-Parameter (siehe Schritt 5.3.2), im Feld Hintergrund subtrahieren (Feld 7).
- Automatisierung von HK-Mitteln: Um die gleichen Parameter für alle Bilder anzuwenden (vollautomatische Verarbeitung), legen Sie den Nb der Klassen (Feld 11), die Mindestgröße (Feld 12) und die maximale Größe (Feld13) fest (siehe Schritt 5.3.1). Diese Parameter müssen so eingestellt werden, dass ein Maximum an Vordergrundpixeln ausgewählt und die Individualisierung von Vordergrundobjekten optimiert wird. Für die halbautomatische Verarbeitungsversion ist kein Eingriff erforderlich.
- Automatisierung von Markerextraktionen: Erweitern Sie für die vollautomatische Version die Box Interne Markerextraktion (Box 27) und legen Sie den Wert des Parameters "dynamisch" in Zeile 13 des Skripts fest. Für die halbautomatische Verarbeitungsversion ist kein Eingriff erforderlich.
HINWEIS: Marker werden extrahiert, indem eine Extended-Minima-Transformation auf ein Eingabebild angewendet wird, das durch einen "dynamischen" Parameter gesteuert wird. Im markerbasierten Wassereinzugsgebietsalgorithmus wird das Überfluten von diesen Markern simuliert, um die Objektsegmentierung durchzuführen. Für die erfolgreiche Segmentierung von Vordergrundobjekten sollte ein einzelner Marker pro Vordergrundobjekt extrahiert werden. Die Einstellung des Parameters "dynamisch" für eine optimale Markerextraktion hängt meist von der Auflösung von Bildern ab. Wenn Sie also mit diesem Parameter nicht vertraut sind, verwenden Sie die halbautomatische Version. - Führen Sie den Workflow aus (Details finden Sie in Teil 7).
- Zu Beginn der Verarbeitung öffnen sich die Dialogfelder HK-means Parameter und Marker-basierte Wasserscheide sukzessive. Um die gleichen Parameter für alle Bilder anzuwenden (vollautomatische Version), klicken Sie auf JA. Andernfalls klicken Sie auf NEIN. Es öffnet sich ein Informationsfeld mit der Bitte: "Bestimmen Sie optimale ROIs mit HK-Means Plugin und Close Image". Klicken Sie auf OK und wenden Sie das HK-Means Plugin(Erkennung und Tracking| HK-Mittel) auf dem Bild, das sich automatisch öffnet. Wählen Sie die Option ROIs exportieren im MODULfeld HK-Means aus. Wenden Sie die besten Parameter an, um ROIs mit maximal einem Vordergrundpixel zu verwenden und die Individualisierung von Vordergrundobjekten zu optimieren. Wenn optimale ROIs gefunden werden, schließen Sie das Bild direkt.
- Am Ende der ersten Segmentierungsmethode öffnet sich ein Informationsfeld und fordert "Unerwünschte ROIs entfernen und Bild schließen". Diese ROIs entsprechen den Rändern der segmentierten Objekte. Wählen Sie OK aus, und entfernen Sie ROIs von falsch segmentierten Objekten im Bild, das automatisch geöffnet wird. Ein ROI kann leicht entfernt werden, indem Sie den Cursor auf den Rand setzen und die Schaltfläche "Löschen" der Tastatur verwenden. Schließen Sie das Bild. Wiederholen Sie das gleiche Verfahren nach Abschluss des zweiten Segmentierungsschritts.
- Wenn in diesem Stadium die YES-Schaltfläche für die vollständige Automatisierung des markerbasierten Wassereinzugsgebietsalgorithmus ausgewählt wurde, werden die zuvor festgelegten Parameter auf alle Bilder angewendet.
- Wenn die Schaltfläche NO ausgewählt wurde, wird ein Informationsfeld geöffnet, in dem Sie aufgefordert werden, "interne Markierungen zu bestimmen und anzupassen". Klicken Sie auf OK und gehen Sie innerhalb der ImageJ-Schnittstelle von Icy zu Plugins | MorphoLibJ | Minima und Maxima| Erweiterte Min & Max. Wählen Sie im Betrieb Extended Minimaaus.
- Wählen Sie Vorschau aus, um das Ergebnis der Transformation auf dem automatisch geöffneten Bild vorab zu visualisieren. Verschieben Sie die Dynamik, bis optimale Marker beobachtet werden. Marker sind Pixelgruppen mit einem Wert von 255 (nicht unbedingt weiße Pixel). Optimale Parameter führen zu einem Marker pro Objekt. Konzentrieren Sie sich auf die verbleibenden Objekte, die mit den beiden vorherigen Segmentierungsmethoden nicht gut segmentiert wurden.
- Verbessern Sie ggf. die Marker, indem Sie zusätzliche morphologische Operationen wie "Öffnen" oder "Schließen"(Plugins | MorphoLibJ | Morphologische Filter). Wenn Sie das endgültige Bild der Marker erhalten, halten Sie es offen und schließen Sie alle anderen Bilder, die mit dem Bild enden, das ursprünglich als Eingabe für den Extended Minima-Vorgang verwendet wurde. Klicken Sie auf Nein, wenn ein ImageJ-Feld darum bittet, Änderungen an diesem Bild zu speichern.
- Bestimmen Sie im Feld Nb von Bildern mit Informationsbox (Feld 14), wie viele Bilder mit Informationsfeldern angezeigt werden sollen.
- Segmentierung E: Clustered Cytoplasma
HINWEIS: Dieser Block verwendet zuvor segmentierte Kerne als einzelne Marker, um die Zytoplasmasegmentierung zu initiieren. Stellen Sie sicher, dass der Block der Kernsegmentierung verarbeitet wurde, bevor Sie ihn verwenden.- Legen Sie in der Box Kanalzytoplasma (Box 1) den Kanal des zytoplasmatischen Signals ein.
- Schreiben Sie in das Feld Extension segmentierte Kerne (Feld 2), das Format verwendet, um Bilder von segmentierten Kernen zu speichern (tif, jpg, bmp, png). Das Standardformat ist tif.
- Schreiben Sie in das Feld Text (Feld3) den Namen des Ordners, der segmentierte Kerne enthält.
- Legen Sie im Feld Format der Bilder segmentierter Zytoplasmen (Feld 4) das Format fest, das zum Speichern segmentierter Objekte (Tiff, Gif, Jpeg, BMP, PNG) verwendet werden soll.
- Während des Prozesses wird automatisch ein Ordner erstellt, um Bilder von segmentierten Zytoplasmen zu speichern. Benennen Sie diesen Ordner im Feld Text (Feld 5) (z. B. segmentierte Zytoplasmen). Der Ordner wird in dem Ordner erstellt, der zusammengeführte Bilder enthält.
- Führen Sie die gleichen Schritte wie in Schritt 5.3.1 aus, um Parameter von Boxen Gaußsche Filter und Aktive Konturen (Vorsicht, Box Ränge sind nicht die gleichen wie in Schritt 5.3.1).
- Führen Sie den Workflow aus (Details finden Sie in Teil 7).
- Segmentierung A: Nicht gruppierte Objekte und Segmentierung C: Gruppierte Objekte mit konvexen Formen
6. Fluoreszierende Signalerkennung und -analyse
- Wählen Sie den angepassten Block aus.
- Im Block Fluoreszenzanalyse A: 1 Kanalführen Sie die Erkennung und Analyse von Brennpunkten in einem Kanal innerhalb eines Segmentobjekts durch: Detektion von Coilin-Brennpunkten (roter Kanal) innerhalb des Kerns.
- Im Block Fluoreszenzanalyse B: 2 Kanäle im selben Fachführen Sie die Erkennung und Analyse von Brennpunkten in zwei Kanälen innerhalb eines Segmentobjekttyps durch: Erkennung von Coilin (roter Kanal) und 53BP1 (grüner Kanal) im Kern.
- Im Block Fluoreszenzanalyse C: 2 Kanäle in zwei Kompartimentenführen Die Detektion und Analyse von Brennpunkten in einem oder zwei Kanälen, insbesondere innerhalb der Kerne und des entsprechenden Zytoplasmas, durch: Detektion von Coilin-Brennpunkten (roter Kanal) sowohl innerhalb des Kerns als auch des entsprechenden Zytoplasmas oder Nachweis von Coilin-Brennpunkten (roter Kanal) innerhalb des Kerns und G3BP-Foci (grüner Kanal) innerhalb des entsprechenden Zytoplasmas.
- Im Block Fluoreszenzanalyse D: Globale Translokationberechnen Sie den Prozentsatz des Signals von einem Kanal in zwei Zellkompartimen (a und b). Exportieren Sie beispielsweise in einem Zytoplasma/Kerntranslokationstest berechnete Prozentsätze nuklearer und zytoplasmatischer Signale für jedes Bild in der endgültigen Tabelle "Ergebnisse". Die Formel zur Berechnung des Kernsignalprozentsatzes ist unten dargestellt. Dieser Block kann für jedes subzelluläre Fach verwendet werden:
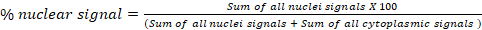
- Im Block Fluoreszenzanalyse E: Individuelle Zelltranslokationberechnen Sie den Prozentsatz des Signals aus einem Kanal in zwei Zellkompartimenten für jede Zelle. Dieser Block ist speziell für den Kern-/Zytoplasma-Translokationstest auf einzelliger Ebene optimiert.
HINWEIS: Da der Block Fluoreszenzanalyse E: Individuelle Zelltranslokation Analysen auf einzelzeller Ebene durchführt, ist eine effiziente Segmentierung von Kern und Zytoplasma erforderlich.
- Verknüpfen Sie die Ausgabe0 (Datei) des Blocks Ordner auswählen (Block 1) mit der Ordnereingabe (weiße Pfeile in schwarzen Kreisen) des gewählten Blocks.
- Legen Sie die Parameter des gewählten Blocks fest.
- Fluoreszenzanalyse A: 1 Kanal, Fluoreszenzanalyse B: 2 Kanäle im selben Fach und Fluoreszenzanalyse C: 2 Kanäle in zwei Fächern
- Schreiben Sie im Feld Ordnerbilder ROIden Namen des Ordners, der Bilder von segmentierten Objekten enthält, denen ein umgekehrter Schrägstrich vorangestellt ist. (Zum Beispiel: "Segmentierte Kerne").
- Schreiben Sie im Feld Format der Bilder segmentierter Objekte (Feld 2) das Format, das zum Speichern von Bildern segmentierter Objekte (tif, jpg, bmp, png) verwendet wird. Das Standardformat ist tif.
- In das Feld "Grenzen töten? ,schreiben Sie Ja, um Rahmenobjekte zu entfernen. Andernfalls schreiben Sie Nr. Die Installation der MorphoLibJ-Sammlung von ImageJ ist erforderlich, um diese Funktion zu verwenden (siehe Schritt 5.3.3).
- In der box(es) Kanalspots Signal,stellen Sie den Kanal, wo Flecken erkannt werden müssen. In klassischen RGB-Bildern, 0=Rot, 1=Grün und 2=Blau.
- In die Felder Name des lokalisierten Molekülsschreiben Sie den Namen des Moleküls, das sich in die Flecken lokalisiert. Die Anzahl der einzugebenden Felder hängt von der Anzahl der Moleküle ab.
- Legen Sie im box(es) Wavelet Spot Detector BlockSpot-Erkennungsparameter für jeden Kanal fest. Legen Sie die Skala(n) (bezogen auf die Spotgröße) und die Empfindlichkeit der Erkennung fest (eine kleinere Empfindlichkeit verringert die Anzahl der erkannten Flecken, wobei der Standardwert 100 und der Mindestwert 0 beträgt). Die ausführliche Dokumentation dieses Plugins ist online verfügbar: http://icy.bioimageanalysis.org/plugin/Spot_Detector. Parameter können auch manuell in Icy definiert werden(Erkennung und Nachverfolgung | Spotdetektor).
- Optional filtern Sie im Feld ROI nach Größefiltern die segmentierten Objekte, bei denen Flecken erkannt werden, indem Sie ein Intervall der Größe (in Pixel) festlegen. Dieser Schritt ist besonders nützlich, um unter- oder übersegmentierte Objekte zu entfernen. Informationen zur manuellen Schätzung der Größe von Objekten finden Sie in Schritt 5.3.1. Die Standardparameter enthalten nicht die Filterung von ROIs nach Größe. Der Block 2 Kanäle in zwei Fächern enthält zwei Boxen: Filterkerne nach Größe (Box 19) und Filterzytoplasma nach Größe (Box 46).
- Optional filtern Sie im Feld Spots nach Größefiltern die erkannten Flecken nach ihrer Größe (in Pixel), um unerwünschte Artefakte zu entfernen. Um die Spotgröße manuell zu schätzen, klicken Sie auf Erkennung und Tracking und öffnen Sie das Spot-Detektor-Plugin. Wählen Sie unter Ausgabeoptionen Die Option Exportieren in ROIaus. Achten Sie darauf, dass Standardparameter keine Filterung von Flecken nach Größe enthalten und gefilterte Flecken bei der Analyse nicht berücksichtigt werden. Die Anzahl der einzugebenden Felder hängt von der Anzahl der Kanäle ab.
- Optional wird in der Box Filterspotsein zusätzlicher Filter (Kontrast, Homogenität, Umfang, Rundheit) auf die erkannten Stellen angewendet. Achten Sie darauf, dass Standardparameter keine Punktfilterung enthalten und gefilterte Flecken bei der Analyse nicht berücksichtigt werden. Die Anzahl der einzugebenden Felder hängt von der Anzahl der Kanäle ab.
- Optional legen Sie in den Feldern Spot-Größenschwelleeinen Schwellenwert für den Bereich (in Pixel) der analysierten Punkte fest. Die Anzahl der gezählten Punkte unterhalb und oberhalb dieses Schwellenwerts wird in der endgültigen Ergebnistabelle exportiert. Die Anzahl der zu informierenden Felder hängt von der Anzahl der Kanäle ab.
- Führen Sie den Workflow aus (Details finden Sie in Teil 7). Daten werden in einer Ergebnistabelle exportiert, die in dem Ordner mit zusammengeführten Bildern gespeichert ist.
- Fluoreszenzanalyse D: Globale Translokation und Fluoreszenzanalyse E: Individuelle Zelltranslokation:
- Schreiben Sie in die Felder Ordnerbilder (Felder 1 und 2) den Namen des Ordners, der Bilder von segmentierten Objekten enthält. Im Block Fluoreszenzanalyse D: Global Translocationwerden die beiden ROI-Typen als ROI a und ROI b identifiziert. Für den Block Fluoreszenzanalyse E: Individuelle Zelltranslokation, in Ordnerbildern segmentierte Kerne und Ordnerbilder segmentierte Zytoplasmen-Boxen, schreiben Sie den Namen des Ordners, der segmentierte Kerne bzw. Zytoplasmen enthält.
- Geben Sie im Kasten Kanalsignal (Box 3) den Kanal des Signals ein.
- Schreiben Sie im Feld Format der Bilder segmentierter Objekte (Feld 4) das Format, das zum Speichern von Bildern segmentierter Objekte (tif, jpg, bmp, png) verwendet wird. Das Standardformat ist tif. Die Option "Rahmen töten" ist auch zum Entfernen von Rahmenobjekten verfügbar (siehe Schritt 6.3.1).
- Optional filtern Sie in Boxen den ROInach Größe , filtern Sie die segmentierten Objekte, indem Sie ein Intervall der Größe (in Pixel) festlegen. Dieser Schritt kann nützlich sein, um unter- oder übersegmentierte Objekte zu entfernen. Informationen zur manuellen Schätzung der Objektgröße finden Sie unter Schritt 5.3.1. Es gibt zwei Felder, die eingegeben werden müssen, eines pro Kanal. Standardparameter enthalten keine ROI-Filterung nach Größe.
- Führen Sie den Workflow aus (Details finden Sie in Teil 7). Exportieren von Daten in einer Kalkulationstabelle Ergebnisse, die in dem Ordner mit zusammengeführten Bildern gespeichert sind.
- Fluoreszenzanalyse A: 1 Kanal, Fluoreszenzanalyse B: 2 Kanäle im selben Fach und Fluoreszenzanalyse C: 2 Kanäle in zwei Fächern
7. Führen Sie das Protokoll aus
- Um einen Block in einer Ausführung zu verarbeiten, entfernen Sie die Verbindung zwischen dem ausgewählten Block und dem Block Ordner auswählen. Platzieren Sie den gewünschten Block auf dem1. Rang. Klicken Sie in der oberen linken Ecke des gewünschten Blocks auf den Link direkt rechts vom Ordner. Doppelklicken Sie im Dialogfeld Öffnen auf den Ordner mit den zusammengeführten Bildern. Klicken Sie dann auf Öffnen. Klicken Sie auf Ausführen, um den Workflow zu starten. Die Verarbeitung kann durch Klicken auf die Stopp-Taste gestoppt werden.
- Um verschiedene Blöcke in einem Durchlauf zu verarbeiten, behalten Sie Verbindungen ausgewählter Blöcke mit dem Block Ordner auswählen (Block 1). Stellen Sie sicher, dass ihr Rang die gute Verarbeitung des Workflows ermöglicht. Wenn z. B. ein bestimmter Block segmentierte Objekte verarbeiten muss, stellen Sie sicher, dass der Segmentierungsblock zuvor verarbeitet wird. Entfernen Sie vor dem Ausführen des Workflows nicht verwendete Blöcke, und speichern Sie das neue Protokoll unter einem anderen Namen.
- Klicken Sie auf Ausführen, um den Workflow zu starten. Wenn das geöffnete Dialogfeld angezeigt wird, doppelklicken Sie auf den Ordner, der die zusammengeführten Bilder enthält. Klicken Sie dann auf Öffnen. Der Workflow wird automatisch ausgeführt. Beenden Sie ggf. die Verarbeitung, indem Sie auf die Schaltfläche Stopp klicken.
- Überprüfen Sie am Ende der Verarbeitung, ob die Nachricht Der ausgeführte Workflow erfolgreich in der unteren rechten Ecke angezeigt wurde und dass alle Blöcke mit einem grünen Vorzeichen gekennzeichnet sind ( Abbildung2b). Ist dies nicht der Fall, geben der Block und das Innere des Fehlerzeichens das zu korrigierende Element an (Abbildung 2b).
HINWEIS: Nachdem der Workflow erfolgreich ausgeführt wurde, kann eine neue Ausführung nicht direkt gestartet werden, und um den Workflow erneut zu verarbeiten, sollte mindestens ein Block mit dem Zeichen "bereit zu verarbeiten" gekennzeichnet werden. Um den Status eines Blocks zu ändern, löschen und erstellen Sie entweder eine Verknüpfung zwischen zwei Feldern innerhalb dieses Blocks, oder schließen Und öffnen Sie das Protokoll einfach. Wenn während der Verarbeitung ein Fehler auftritt, kann eine neue Ausführung direkt gestartet werden. Während einer neuen Ausführung werden alle Blöcke der Pipeline verarbeitet, auch wenn einige von ihnen mit dem grünen Vorzeichen gekennzeichnet sind.
Ergebnisse
Alle beschriebenen Analysen wurden auf einem Standard-Laptop (64-Bit- Quad-Core-Prozessor bei 2,80 GHz mit 16 GB Ram-Speicher (Random-Access Memory) durchgeführt, der mit der 64-Bit-Version von Java arbeitet. Der Arbeitsspeicher mit zufälligem Zugriff ist ein wichtiger Parameter, der je nach Menge und Auflösung der zu analysierenden Bilder zu berücksichtigen ist. Die Verwendung der 32-Bit-Version von Java beschränkt den Speicher auf ca. 1300 MB, was für big Data-Analysen ungeeignet sein könnte, während die 64-Bit...
Diskussion
Für die Analyse von Fluoreszenzzellbildern stehen immer mehr freie Software-Tools zur Verfügung. Benutzer müssen die geeignete Software entsprechend der Komplexität ihres Problems, ihrem Wissen in der Bildverarbeitung und der Zeit, die sie in ihrer Analyse verbringen möchten, richtig auswählen. Icy, CellProfiler oder ImageJ/Fiji sind leistungsstarke Tools, die sowohl Benutzerfreundlichkeit als auch Funktionalität kombinieren3. Icy ist ein eigenständiges Tool, das eine klare grafische Benut...
Offenlegungen
Die Autoren haben nichts zu verraten.
Danksagungen
G.H. wurde durch ein Graduiertenstipendium des Ministére Délégué é la Recherche et aux Technologies unterstützt. L.H. wurde durch ein Graduiertenstipendium des Institut de Cancérologie de Lorraine (ICL) unterstützt, während Q.T. durch ein öffentliches Stipendium unterstützt wurde, das von der französischen Nationalen Forschungsagentur (ANR) im Rahmen des zweiten Programms "Investissements d'Avenir" FIGHT-HF (Referenz: ANR-15-RHU4570004) überwacht wurde. Diese Arbeit wurde von CNRS und der Université de Lorraine (UMR 7365) finanziert.
Materialien
| Name | Company | Catalog Number | Comments |
| 16% Formaldehyde solution (w/v) methanol free | Thermo Fisher Scientific | 28908 | to fix the cells |
| Alexa Fluor 488 of goat anti-rabbit | Thermo Fisher Scientific | A-11008 | fluorescent secondary antibody |
| Alexa Fluor 555 of goat anti-mouse | Thermo Fisher Scientific | A-21425 | fluorescent secondary antibody |
| Alexa Fluor 555 Phalloidin | Thermo Fisher Scientific | A34055 | fluorescent secondary antibody |
| Bovine serum albumin standard (BSA) | euromedex | 04-100-812-E | |
| DMEM | Sigma-Aldrich | D5796-500ml | cell culture medium |
| Duolink In Situ Mounting Medium with DAPI | Sigma-Aldrich | DUO82040-5ML | mounting medium |
| Human: HeLa S3 cells | IGBMC, Strasbourg, France | cell line used to perform the experiments | |
| Hydrogen peroxide solution 30% (H2O2) | Sigma-Aldrich | H1009-100ml | used as a stressing agent |
| Lipofectamine 2000 Reagent | Thermo Fisher Scientific | 11668-019 | transfection reagent |
| Mouse monoclonal anti-coilin | abcam | ab11822 | Coilin-specific antibody |
| Nikon Optiphot-2 fluorescence microscope | Nikon | epifluoresecence microscope | |
| Opti-MEM I Reduced Serum Medium | Thermo Fisher Scientific | 31985062 | transfection medium |
| PBS pH 7.4 (10x) | gibco | 70011-036 | to wash the cells |
| Rabbit polyclonal anti-53BP1 | Thermo Fisher Scientific | PA1-16565 | 53BP1-specific antibody |
| Rabbit polyclonal anti-EDC4 | Sigma-Aldrich | SAB4200114 | EDC4-specific antibody |
| Triton X-100 | Roth | 6683 | to permeabilize the cells |
Referenzen
- Möckl, L., Lamb, D. C., Bräuchle, C. Super-resolved fluorescence microscopy: Nobel Prize in Chemistry 2014 for Eric Betzig, Stefan Hell, and William E. Moerner. Angewandte Chemie. 53 (51), 13972-13977 (2014).
- Meijering, E., Carpenter, A. E., Peng, H., Hamprecht, F. A., Olivo-Marin, J. -. C. Imagining the future of bioimage analysis. Nature Biotechnology. 34 (12), 1250-1255 (2016).
- Wiesmann, V., Franz, D., Held, C., Münzenmayer, C., Palmisano, R., Wittenberg, T. Review of free software tools for image analysis of fluorescence cell micrographs. Journal of Microscopy. 257 (1), 39-53 (2015).
- de Chaumont, F., et al. Icy: an open bioimage informatics platform for extended reproducible research. Nature Methods. 9 (7), 690-696 (2012).
- Girish, V., Vijayalakshmi, A. Affordable image analysis using NIH Image/ImageJ. Indian J Cancer. 41 (1), 47 (2004).
- Zaitoun, N. M., Aqel, M. J. Survey on image segmentation techniques. Procedia Computer Science. 65, 797-806 (2015).
- . MetaMorph Microscopy Automation and Image Analysis Software Available from: https://www.moleculardevices.com/products/cellular-imaging-systems/acquisition-and-analysis-software/metamorph-microscopy (2018)
- . NIS-Elements Imaging Software Available from: https://www.nikon.com/products/microscope-solutions/lineup/img_soft/nis-element (2014)
- Meyer, F., Beucher, S. Morphological segmentation. Journal of Visual Communication and Image Representation. 1 (1), 21-46 (1990).
- Schieber, M., Chandel, N. S. ROS Function in Redox Signaling and Oxidative Stress. Current Biology. 24 (10), 453-462 (2014).
- D'Autréaux, B., Toledano, M. B. ROS as signalling molecules: mechanisms that generate specificity in ROS homeostasis. Nature Reviews. Molecular Cell Biology. 8 (10), 813-824 (2007).
- Davalli, P., Mitic, T., Caporali, A., Lauriola, A., D'Arca, D. ROS, Cell Senescence, and Novel Molecular Mechanisms in Aging and Age-Related Diseases. Oxidative Medicine and Cellular Longevity. 2016, 3565127 (2016).
- Disher, K., Skandalis, A. Evidence of the modulation of mRNA splicing fidelity in humans by oxidative stress and p53. Genome. 50 (10), 946-953 (2007).
- Takeo, K., et al. Oxidative stress-induced alternative splicing of transformer 2β (SFRS10) and CD44 pre-mRNAs in gastric epithelial cells. American Journal of Physiology - Cell Physiology. 297 (2), 330-338 (2009).
- Seo, J., et al. Oxidative Stress Triggers Body-Wide Skipping of Multiple Exons of the Spinal Muscular Atrophy Gene. PLOS ONE. 11 (4), 0154390 (2016).
- Will, C. L., Luhrmann, R. Spliceosome Structure and Function. Cold Spring Harbor Perspectives in Biology. 3 (7), 003707 (2011).
- Ljosa, V., Sokolnicki, K. L., Carpenter, A. E. Annotated high-throughput microscopy image sets for validation. Nature Methods. 9 (7), 637-637 (2012).
- Wang, Q., et al. Cajal bodies are linked to genome conformation. Nature Communications. 7, (2016).
- Carpenter, A. E., et al. CellProfiler: image analysis software for identifying and quantifying cell phenotypes. Genome Biology. 7, 100 (2006).
- McQuin, C., et al. CellProfiler 3.0: Next-generation image processing for biology. PLoS Biology. 16 (7), 2005970 (2018).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten