Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Methods Article
Analizador de Subestructuras: Un flujo de trabajo amigable para el usuario para la exploración rápida y el análisis preciso de los cuerpos celulares en imágenes de microscopía de fluorescencia
En este artículo
Resumen
Presentamos un flujo de trabajo de libre disposición construido para la exploración rápida y el análisis preciso de cuerpos celulares en compartimentos celulares específicos en imágenes de microscopía de fluorescencia. Este flujo de trabajo fácil de usar está diseñado en el software de código abierto Icy y también utiliza funcionalidades ImageJ. La canalización es asequible sin conocimientos en el análisis de imágenes.
Resumen
La última década se ha caracterizado por avances en las técnicas de microscopía de fluorescencia ilustradas por la mejora de la resolución espacial, pero también en las técnicas de imagen de células vivas y microscopía de alto rendimiento. Esto condujo a un aumento constante en la cantidad y complejidad de los datos de la microscopía para un solo experimento. Debido a que el análisis manual de los datos de la microscopía consume mucho tiempo, es subjetivo y prohíbe los análisis cuantitativos, la automatización del análisis de bioimagen se está volviendo casi inevitable. Construimos un flujo de trabajo informático llamado Substructure Analyzer para automatizar completamente el análisis de señales en bioimagens a partir de microscopía fluorescente. Este flujo de trabajo se desarrolla en la plataforma de código abierto fácil de usar Icy y se completa con las funcionalidades de ImageJ. Incluye el preprocesamiento de imágenes para mejorar la relación señal-ruido, la segmentación individual de celdas (detección de límites celulares) y la detección/cuantificación de cuerpos celulares enriquecidos en compartimentos celulares específicos. La principal ventaja de este flujo de trabajo es proponer complejas funcionalidades de bioimagen a los usuarios sin experiencia en análisis de imágenes a través de una interfaz fácil de usar. Además, es altamente modular y se adapta a varias cuestiones desde la caracterización de la translocación nuclear/citoplasmático hasta el análisis comparativo de diferentes cuerpos celulares en diferentes subefilas celulares. La funcionalidad de este flujo de trabajo se ilustra a través del estudio de los cuerpos Deles (bobinados) en condiciones de estrés oxidativo (OS). Los datos de la microscopía de fluorescencia muestran que su integridad en las células humanas se ve afectada unas horas después de la inducción del sistema operativo. Este efecto se caracteriza por una disminución de la nucleación de bobina en cuerpos típicos de Cajal, asociado con una redistribución nucleoplasmica de bobina en un mayor número de focos más pequeños. El papel central de la bobina en el intercambio entre los componentes CB y el nucleoplasmo circundante sugiere que la redistribución inducida por el sistema operativo de bobinado podría afectar la composición y la funcionalidad de Los cuerpos de Cajal.
Introducción
La microscopía ligera y, más particularmente, la microscopía de fluorescencia son técnicas robustas y versátiles comúnmente utilizadas en las ciencias biológicas. Dan acceso a la localización precisa de varias biomoléculas como proteínas o ARN a través de su etiquetado fluorescente específico. La última década se ha caracterizado por rápidos avances en las tecnologías de microscopía e imagen, como lo demuestra el Premio Nobel de Química 2014 que otorga a Eric Betzig, Stefan W. Hell y William E. Moerner por el desarrollo de la microscopía de fluorescencia superconsuelta (SRFM)1. SFRM evita el límite de difracción de la microscopía óptica tradicional para incorporarlo a la nanodimensión. La mejora de técnicas como la imagen en vivo o los enfoques de detección de alto rendimiento también aumenta la cantidad y la complejidad de los datos que se deben tratar para cada experimento. La mayoría de las veces, los investigadores se enfrentan a altas poblaciones heterogéneas de células y quieren analizar fenotipos a nivel de una sola célula.
Inicialmente, análisis como el conteo de focos fueron realizados a simple vista, lo que es preferido por algunos investigadores ya que proporciona un control visual completo sobre el proceso de conteo. Sin embargo, el análisis manual de estos datos consume demasiado tiempo, conduce a la variabilidad entre los observadores y no da acceso a características más complejas para que los enfoques asistidos por ordenador se estén volviendo ampliamente utilizados y casi inevitables2. Los métodos informáticos bioimagen aumentan sustancialmente la eficiencia del análisis de datos y están libres de la subjetividad inevitable del operador y el sesgo potencial del análisis de recuento manual. El aumento de la demanda en este campo y la mejora de la potencia informática llevaron al desarrollo de un gran número de plataformas de análisis de imágenes. Algunos de ellos están disponibles de forma gratuita y dan acceso a varias herramientas para realizar análisis con ordenadores personales. Recientemente se ha establecido una clasificación de herramientas de acceso abierto3 y presenta Icy4 como un potente software que combina facilidad de uso y funcionalidad. Además, Icy tiene la ventaja de comunicarse con ImageJ.
Para los usuarios sin experiencia en análisis de imágenes, los principales obstáculos son elegir la herramienta adecuada de acuerdo con los parámetros problemáticos y de ajuste correcto que a menudo no se entienden bien. Además, los tiempos de configuración suelen ser largos. Icy propone una interfaz fácil de usar de apuntar y hacer clic llamada "Protocolos" para desarrollar el flujo de trabajo mediante la combinación de algunos plugins que se encuentran dentro de una colección exhaustiva4. El diseño modular flexible y la interfaz de apuntar y hacer clic hacen que la configuración de un análisis sea factible para los no programadores. Aquí presentamos un flujo de trabajo llamado Substructure Analyzer,desarrollado en la interfaz de Icy, cuya función es analizar señales fluorescentes en compartimentos celulares específicos y medir diferentes características como brillo, número de focos, tamaño de los focos y distribución espacial. Este flujo de trabajo aborda varios problemas como la cuantificación de la translocación de la señal, el análisis de células trans infectadas que expresan a un reportero fluorescente o el análisis de focos de diferentes subestructuras celulares en células individuales. Permite el procesamiento simultáneo de varias imágenes, y los resultados de salida se exportan a una hoja de trabajo delimitada por tabulaciones que se puede abrir en programas de hoja de cálculo de uso común.
La canalización Analizador de subestructuras se presenta en la Figura 1. En primer lugar, todas las imágenes contenidas en una carpeta especificada se procesan previamente para mejorar su relación señal/ruido. Este paso aumenta la eficiencia de los pasos siguientes y disminuye el tiempo de ejecución. A continuación, se identifican y segmentan las Regiones de Interés (ROI), correspondientes a las áreas de imagen donde se debe detectar la señal fluorescente. Por último, se analiza la señal fluorescente y los resultados se exportan a una hoja de cálculo delimitada por tabulaciones.
La segmentación de objetos (detección de límites) es el paso más difícil en el análisis de imágenes, y su eficiencia determina la precisión de las mediciones de celda resultantes. Los primeros objetos identificados en una imagen (llamados objetos primarios) son a menudo núcleos de imágenes manchadas de ADN (tinción DAPI o Hoechst), aunque los objetos primarios también pueden ser células enteras, cuentas, motas, tumores o cualquier objeto manchado. En la mayoría de las imágenes biológicas, las células o núcleos se tocan entre sí o se superponen haciendo que los algoritmos simples y rápidos fallen. Hasta la fecha, ningún algoritmo universal puede realizar una segmentación perfecta de todos los objetos, principalmente porque sus características (tamaño, forma o textura) modulan la eficiencia de la segmentación5. Las herramientas de segmentación comúnmente distribuidas con software de microscopía (como MetaMorph Imaging Software de Molecular Devices6o el software NIS-Elements Advances Research de Nikon7)se basan generalmente en técnicas estándar como la coincidencia de correlaciones, umbrales u operaciones morfológicas. Aunque son eficientes en los sistemas básicos, estos métodos sobregeneracionalizados presentan rápidamente limitaciones cuando se utilizan en contextos más desafiantes y específicos. De hecho, la segmentación es muy sensible a parámetros experimentales como el tipo de celda, la densidad de celdas o los biomarcadores, y con frecuencia requiere un ajuste repetido para un conjunto de datos grande. El flujo de trabajo de Substructure Analyzer integra algoritmos simples y más sofisticados para proponer diferentes alternativas adaptadas a la complejidad de la imagen y a las necesidades del usuario. En particular, propone el algoritmo de cuenca hidrográfica basada en marcadores8 para objetos altamente agrupados. La eficiencia de este método de segmentación se basa en la selección de marcadores individuales en cada objeto. Estos marcadores se eligen manualmente la mayor parte del tiempo para obtener los parámetros correctos para la segmentación completa, lo que consume mucho tiempo cuando los usuarios se enfrentan a un gran número de objetos. Substructure Analyzer propone una detección automática de estos marcadores, proporcionando un proceso de segmentación altamente eficiente. La segmentación es, la mayor parte del tiempo, el paso limitante del análisis de imágenes y puede modificar considerablemente el tiempo de procesamiento en función de la resolución de la imagen, el número de objetos por imagen y el nivel de agrupación en clústeres de objetos. Las canalizaciones típicas requieren de unos segundos a 5 minutos por imagen en un equipo de escritorio estándar. El análisis de imágenes más complejas puede requerir un ordenador más potente y algunos conocimientos básicos en el análisis de imágenes.
La flexibilidad y la funcionalidad de este flujo de trabajo se ilustran con varios ejemplos en los resultados representativos. Las ventajas de este flujo de trabajo se muestran notablemente a través del estudio de subestructuras nucleares en condiciones de estrés oxidativo (OS). El sistema operativo corresponde a un desequilibrio de la homeostasis redox en favor de los oxidantes y se asocia con altos niveles de especies reactivas de oxígeno (ROS). Dado que ROS actúa como moléculas de señalización, los cambios en su concentración y localización subcelular afectan positiva o negativamente a una miríada de vías y redes que regulan las funciones fisiológicas, incluyendo la transducción de señales, los mecanismos de reparación, la expresión génica, la muerte celular y la proliferación9,,10. Por lo tanto, el sistema operativo participa directamente en diversas patologías (enfermedades neurodegenerativas y cardiovasculares, cánceres, diabetes, etc.), pero también en el envejecimiento celular. Por lo tanto, descifrar las consecuencias del sistema operativo en la organización y función de la célula humana constituye un paso crucial en la comprensión de las funciones del sistema operativo en la aparición y desarrollo de patologías humanas. Se ha establecido que el sistema operativo regula la expresión génica mediante la modulación de la transcripción a través de varios factores de transcripción (p53, Nrf2, FOXO3A)11,pero también afectando a la regulación de varios procesos co- y post-transcripción como el empalme alternativo (AS) de pre-RNAs12,,13,14. El empalme alternativo de transcripciones de codificación primaria y no codificación es un mecanismo esencial que aumenta la capacidad de codificación de los genomas mediante la producción de isoformas de transcripción. As es realizado por un enorme complejo de ribonucleoproteína llamado spliceosoma, que contiene casi 300 proteínas y 5 PEQUEÑOS ARN nucleares (UsnRNAs) ricos en U15. El ensamblaje de empalmes y AS están estrechamente controlados en las células y algunos pasos de la maduración del empalme ocurren dentro de compartimentos nucleares sin membrana llamados Cuerpos Cajales. Estas subestructuras nucleares se caracterizan por la naturaleza dinámica de su estructura y su composición, que se llevan a cabo principalmente por interacciones multivalentes de sus componentes de ARN y proteína con la proteína coilin. El análisis de miles de celdas con el flujo de trabajo de Substructure Analyzer permitió caracterizar los efectos nunca descritos del sistema operativo en Los cuerpos de Cajal. De hecho, los datos obtenidos sugieren que el sistema operativo modifica la nucleación de los cuerpos de Cajal, induciendo una redistribución nucleoplasmica de la proteína de bobina en numerosos focos nucleares más pequeños. Tal cambio de la estructura de los Cuerpos de Cajales podría afectar a la maduración del empalme y participar en la modulación AS por el sistema operativo.
Protocolo
NOTA: Tutoriales fáciles de usar están disponibles en el sitio web de Icy http://icy.bioimageanalysis.org.
1. Descargue Icy y el protocolo Substructure Analyzer
- Descargue Icy desde el sitio web de Icy (http://icy.bioimageanalysis.org/download) y descargue el protocolo Substructure Analyzer: http://icy.bioimageanalysis.org/protocols?sort=latest.
NOTA: Si utiliza un sistema operativo de 64 bits, asegúrese de utilizar la versión de 64 bits de Java. Esta versión permite aumentar la memoria asignada a Icy (Preferencias ? General ? Memoria máxima).
2. Apertura del protocolo
- Abra Icy y haga clic en Herramientas en el menú de la cinta de opciones.
- Haga clic en Protocolos para abrir la interfaz del Editor de protocolos.
- Haga clic en Cargar y abra el protocolo Substructure Analyzer. La carga del protocolo puede tardar unos segundos. Asegúrese de que la apertura del protocolo se ha completado antes de usarlo.
NOTA: El flujo de trabajo se compone de 13 bloques generales presentados en la Figura 2a. Cada bloque funciona como una canalización compuesta por varios cuadros que realizan subtareas específicas.
3. Interactuar con el flujo de trabajo en Icy
NOTA: Cada bloque o cuadro está numerado y tiene un rango específico dentro del flujo de trabajo (Figura 2b). Al hacer clic en este número, la posición más cercana posible a la primera se asigna al bloque/cuadro seleccionado y, a continuación, se reorganiza la posición de los otros bloques/cajas. Respete el orden correcto de los bloques al preparar el flujo de trabajo. Por ejemplo, el bloque Detector de spot necesita ROI predefinidos para que los bloques de segmentación tengan que ejecutarse antes de los bloques de Detector de spot. No modifique la posición de los cuadros. No utilice "." en el nombre de la imagen.
- Haciendo clic en el icono de la esquina superior izquierda, contraiga, expanda, amplíe, acote o elimine el bloque (Figura 2b).
- Cada canalización del flujo de trabajo se caracteriza por una red de cajas conectadas a través de su entrada y salida(Figura 2b). Para crear una conexión, haga clic en Salida y mantenga hasta que el cursor alcance una entrada. Las conexiones se pueden eliminar haciendo clic en la etiqueta Salida.
4. Combinación de los canales de una imagen
- Utilice el bloque Canales de combinación para generar imágenes combinadas. Si es necesario, cambie el nombre de los archivos para que las secuencias que se van a combinar tengan el mismo prefijo de nombre seguido de un separador distinto. Por ejemplo, las secuencias de canales individuales de una imagen A se denominan: ImageA_red ImageA_blue.
NOTA: Para el separador, no utilice caracteres ya presentes en el nombre de la imagen. - En la misma carpeta, cree una nueva carpeta por canal para combinar. Por ejemplo, para combinar canales rojos, verdes y azules, cree 3 carpetas y almacene las secuencias correspondientes en estas carpetas.
- Utilice solamente el bloque Fusionar canales, elimine los otros bloques y guarde el protocolo como Canales de fusión.
- Acceda a las casillas para establecer parámetros. Para cada canal, rellene las casillas Número de canal X (cuadros 1, 5 o 9), Número de canal de carpeta X (cuadros 2, 6 o 10), Número de canal separador X (cuadros 3, 7 o 11) y Canal de mapa de color nb X (cuadros 4, 8 y 12) respectivamente.
NOTA: Estos cuadros se agrupan horizontalmente por cuatro, cada línea correspondiente al mismo canal. En cada línea, también está disponible una pantalla (cuadros 23, 24 o 25) para visualizar directamente la secuencia del canal correspondiente.- En el cuadro Número de canal X, elija qué canal extraer (en imágenes RGB clásicas, 0,rojo, 1, verde, 2, azul). El usuario accede rápidamente a los diferentes canales de una imagen dentro de la ventana Inspector de Icy, en la pestaña Secuencia.
- En el cuadro Número de canal de carpeta X, escriba el nombre de la carpeta que contiene imágenes del canal X.
- En el cuadro Separador número de canal X, escriba el separador utilizado para el nombre de la imagen (en el ejemplo anterior: "_red", "_green" y "_blue").
- En el cuadro Canal de mapa de colores nb X, indique con un número qué modelo de mapa de colores utilizar para visualizar el canal correspondiente en Icy. Los mapas de colores disponibles están visibles en la pestaña Secuencia de la ventana Inspector.
- En el cuadro Formato de imágenes combinadas (cuadro 28), escriba la extensión para guardar imágenes combinadas: .tif, .gif, .jpg, .bmp o .png.
NOTA: Para fusionar solo 2 canales, no llene los cuatro cuadros correspondientes al tercer canal.
- En la esquina superior izquierda del bloque Combinar canales, haga clic en el vínculo directamente a la derecha de Carpeta. En el cuadro de diálogo Abrir que aparece, haga doble clic en la carpeta que contiene secuencias del primer canal que se ha definido en el cuadro Canal de carpeta número 1 (cuadro 2). A continuación, haga clic en Abrir.
- Ejecute el protocolo haciendo clic en la flecha negra en la esquina superior izquierda del bloque Canales de fusión (consulte la parte 7 para obtener más detalles). Las imágenes combinadas se guardan en una carpeta Combinar en el mismo directorio que las carpetas de canales individuales.
- Acceda a las casillas para establecer parámetros. Para cada canal, rellene las casillas Número de canal X (cuadros 1, 5 o 9), Número de canal de carpeta X (cuadros 2, 6 o 10), Número de canal separador X (cuadros 3, 7 o 11) y Canal de mapa de color nb X (cuadros 4, 8 y 12) respectivamente.
5. Segmentación de las regiones de interés
NOTA: Substructure Analyzer integra algoritmos simples y más sofisticados para proponer diferentes alternativas adaptadas a la complejidad de la imagen y a las necesidades del usuario.
- Seleccione el bloque adaptado.
- Si los objetos no se tocan entre sí o el usuario no necesita diferenciar los objetos agrupados individualmente, utilice el bloque Segmentación A: Objetos no agrupados.
- Cuando los objetos no se toquen entre sí, pero algunos de ellos están cerca, utilice el bloque Segmentación B: Objetos mal agrupados.
- Para objetos con un nivel de agrupación en clústeres alto y una forma convexa, utilice el bloque Segmentación C: Objetos agrupados con formas convexas.
- Si los objetos presentan un nivel de agrupación en clústeres alto y tienen formas irregulares, utilice el bloque Segmentación D: Objetos agrupados con formas irregulares.
- Utilice el bloque Segmentación E: citoplasma agrupado para segmentar los citoplasmas que tocan individualmente utilizando núcleos segmentados como marcadores. Este bloque necesita imperativamente núcleos segmentados para procesar.
NOTA: Bloques adaptados para el proceso de segmentación de objetos primarios de forma independiente para que se puedan utilizar varios bloques en la misma ejecución para comparar su eficiencia para una subestructura determinada o para segmentar diferentes tipos de subestructuras. Si el nivel de agrupación en clústeres es heterogéneo dentro del mismo conjunto de imágenes, procese objetos pequeños y altamente agrupados por separado en los bloques adaptados.
- Vincule la salida0 (Archivo) del bloque Seleccionar carpeta a la entrada de carpeta del bloque de segmentación elegido.
- Establezca los parámetros del bloque de segmentación elegido.
- Segmentación A: Objetos no agrupados y Segmentación C: Objetos agrupados con formas convexas
- En el cuadro Señal de canal (cuadro 1), establezca el canal de los objetos en segmentar.
- Como opción, en el cuadro Filtro gaussiano (caja 2), aumente los valores de sigma X e Y si la señal dentro de los objetos es heterogénea. El filtro gaussiano suaviza las texturas para obtener regiones más uniformes y aumenta la velocidad y la eficiencia de la segmentación de núcleos. Cuanto más pequeños son los objetos, menor es el valor sigma. Evite valores de sigma altos. Establezca los valores predeterminados en 0.
- En el cuadro HK-Means (cuadro 3), establezca el parámetro Intensity classes y los tamaños mínimo y máximo aproximados (en píxeles) de los objetos que se van a detectar.
NOTA: Para las clases de intensidad, un valor de 2 clasifica los píxeles en 2 clases: fondo y primer plano. Por lo tanto, se adapta cuando el contraste entre los objetos y el fondo es alto. Si los objetos de primer plano tienen intensidades diferentes o si el contraste con el fondo es bajo, aumente el número de clases. El valor predeterminado es 2. El tamaño del objeto se puede evaluar rápidamente dibujando un ROI manualmente alrededor del objeto de interés. El tamaño del ROI (Interior en píxeles) aparece directamente en la imagen al apuntarla con el cursor o se puede acceder a ella en la ventana De estadísticas de ROI (ábrala desde la barra de búsqueda). Los parámetros óptimos detectan cada objeto en primer plano en un únicoROI. Se pueden definir manualmente en Icy (Detección y seguimiento de la tecnología de la aplicación ( HK-Means). - En el cuadro Contornos activos (cuadro 4), optimice la detección de bordes de objeto. La documentación exhaustiva de este plugin está disponible en línea: http://icy.bioimageanalysis.org/plugin/Active_Contours. Los parámetros correctos también se pueden definir manualmente en Icy (Detección y seguimiento de la función . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . . Contornos activos).
- Durante el proceso, se crea automáticamente una carpeta para guardar imágenes de objetos segmentados. En el cuadro Texto (cuadro 6), asigne un nombre a esta carpeta (por ejemplo: Núcleos segmentados). Para establecer el formato para guardar imágenes de objetos segmentados (Tiff, Gif, Jpeg, BMP, PNG), rellene el formato de cuadro de las imágenes de objetos segmentados. La carpeta se crea en la carpeta que contiene imágenes combinadas.
- Ejecute el flujo de trabajo (para obtener más información, consulte la parte 7).
- Segmentación B: Objetos mal agrupados
- Siga los mismos pasos que en 5.3.1 para establecer los parámetros de las cajas Señal de canal, HK-Means, Contornos activos, Extensión para guardar objetos segmentados y Texto (los rangos de cuadros no son los mismos que en el paso 5.3.1).
- En el cuadro Llamar al plugin IJ (cuadro 4), establezca el parámetro Rolling para controlar la resta de fondo. Establezca este parámetro en al menos el tamaño del objeto más grande que no forma parte del fondo. La reducción de este valor aumenta la eliminación de fondo, pero también puede inducir la pérdida de señal de primer plano.
- En el cuadro Ecualización adaptativa del histograma (cuadro 6), mejore los contrastes entre los objetos de primer plano y el fondo. Aumentar la pendiente da secuencias más contrastadas.
- Ejecute el flujo de trabajo (para obtener más información, consulte la parte 7).
- Segmentación D: Objetos agrupados con formas irregulares
NOTA: Se aplican tres métodos diferentes de segmentación a cada imagen: en primer lugar,HK-means clusteringcombinado con elMétodo de contornos activosse aplica. Entonces, elalgoritmo clásico de cuenca(mediante el mapa de distancia euclidiana) se aplica a objetos previamente mal segmentados. Por último, unalgoritmo de cuenca hidrográfica basado en marcadoresse utiliza. Solo los métodos de cuenca hidrográfica basados en marcadores y medios HK necesitan la intervención del usuario. Para ambos métodos, se pueden aplicar los mismos parámetros para todas las imágenes (versión totalmente automatizada) o cambiarse para cada imagen (versión semiautomática). Si el usuario no está entrenado en estos métodos de segmentación, se recomienda encarecidamente el procesamiento semiautomático. Durante el procesamiento de este bloque, se necesita la intervención manual. Cuando finaliza un método de segmentación, el usuario debe quitar manualmente los objetos mal segmentados antes del comienzo del siguiente método de segmentación. Los objetos segmentados correctamente se guardan y no se consideran en los pasos siguientes. Este bloque tiene que estar conectado con el bloqueCuadro de diálogo de segmentación de objetos primarios de formas agrupadas/heterogéneaspara funcionar correctamente.- Descargue la colección ImageJ MorphoLibJ en https://github.com/ijpb/MorphoLibJ/releases. La versión MorphoLibJ 1.4.0 se utiliza en este protocolo. Coloque el archivo MorphoLibJ_-1.4.0.jar en la carpeta icy/ij/plugins. Más información sobre el contenido de esta colección está disponible en https://imagej.net/MorphoLibJ.
- Siga los mismos pasos que en el paso 5.3.1 para establecer parámetros de cuadros Señal de canal, Filtro gaussiano, Contornos activos, Extensión para guardar objetos segmentados y Texto. Los rangos de las cajas no son los mismos que en el paso 5.3.1.
- Establezca los parámetros del cuadro Ecualización adaptativa del histograma (consulte el paso 5.3.2.3).
- Para activar Restar fondo, escriba sí en Aplicar fondo de resta? (cuadro 5). De lo contrario, escriba No. Si el plugin está activado, establezca el parámetro de balanceo (consulte el paso 5.3.2), en el cuadro Restar fondo parámetro (cuadro 7).
- Automatización de HK-means: Para aplicar los mismos parámetros para todas las imágenes (procesamiento totalmente automatizado), establezca el Nb de las clases (cuadro 11), el tamaño mínimo (cuadro 12) y el tamaño máximo (cuadro13) (consulte el paso 5.3.1). Estos parámetros deben establecerse para seleccionar un máximo de píxeles de primer plano y para optimizar la individualización de los objetos de primer plano. Para la versión de procesamiento semiautomática, no se necesita intervención.
- Automatización de extracciones de marcadores: para la versión totalmente automatizada, expanda la caja Extracción de marcadores internos (caja 27) y establezca el valor del parámetro "dinámico" en la línea 13 del script. Para la versión de procesamiento semiautomática, no se necesita intervención.
NOTA: Los marcadores se extraen aplicando una transformación de mínimo extendido en una imagen de entrada controlada por un parámetro "dinámico". En el algoritmo de cuenca hidrográfica basada en marcadores, se simula la inundación de estos marcadores para realizar la segmentación de objetos. Para la segmentación correcta de objetos de primer plano, se debe extraer un único marcador por objeto de primer plano. La configuración del parámetro "dinámico" para la extracción óptima de marcadores depende principalmente de la resolución de imágenes. Por lo tanto, si no está familiarizado con este parámetro, utilice la versión semiautomática. - Ejecute el flujo de trabajo (para obtener más información, consulte la parte 7).
- Al principio del procesamiento, los cuadros de diálogo HK-means parámetros y la cuenca hidrográfica basada en marcadores se abren sucesivamente. Para aplicar los mismos parámetros para todas las imágenes (versión totalmente automatizada), haga clic en Si. De lo contrario, haga clic en NO. Se abre un cuadro de información, que pide "Determinar ROI óptimos con el plugin HK-Means y cerrar la imagen". Haga clic en Aceptar y aplique manualmente el plugin HK-Means (Detección y seguimientode lafunción de seguimiento ( HK-Means) en la imagen, que se abre automáticamente. Seleccione la opción Exportar ROI en el cuadro del complemento HK-Means. Aplique los mejores parámetros para que los ROI contengan un máximo de píxeles de primer plano y para optimizar la individualización de los objetos en primer plano. Cuando se encuentren ROI óptimos, cierre directamente la imagen.
- Al final del primer método de segmentación, se abre un cuadro de información que pide "Eliminar ROI no deseados y cerrar la imagen". Estos ROI corresponden a los bordes de los objetos segmentados. Seleccione Aceptar y elimine los ROI de los objetos mal segmentados en la imagen, que se abre automáticamente. Un ROI se puede eliminar fácilmente colocando el cursor en su borde y usando el botón "Eliminar" del teclado. Cierre la imagen. Repita el mismo procedimiento después de completar el segundo paso de segmentación.
- En esta etapa, si se seleccionó el botón YES para la automatización completa del algoritmo de cuenca hidrográfica basado en marcadores, los parámetros establecidos anteriormente se aplicarán a todas las imágenes.
- Si se ha seleccionado el botón NO, se abre un cuadro de información que pide "Determinar y ajustar los marcadores internos". Haga clic en OK y dentro de la interfaz ImageJ de Icy, vaya a Plugins . MorphoLibJ ? Minima y Maxima| Extended Min & Max. En Operación, seleccione Mínimo extendido.
- Seleccione Vista previa para previsualizar en la imagen abierta automáticamente el resultado de la transformación. Mueva la dinámica hasta que se observen los marcadores óptimos. Los marcadores son grupos de píxeles con un valor de 255 (no necesariamente píxeles blancos). Los parámetros óptimos conducen a un marcador por objeto. Céntrese en los objetos restantes que no se han segmentado bien con los dos métodos de segmentación anteriores.
- Si es necesario, mejore los marcadores aplicando operaciones morfológicas adicionales como "Apertura" o "Cierre"(Plugins ? MorphoLibJ ? Filtros Morfológicos). Al obtener la imagen final de los marcadores, manténgala abierta y cierre todas las demás imágenes que terminan con la imagen utilizada inicialmente como entrada para la operación De minima extendida. Haga clic en No si un cuadro ImageJ pide guardar los cambios en esta imagen.
- En el cuadro Nb de imágenes con cuadro de información (cuadro 14), determine cuántas imágenes con cuadros de información deben aparecer.
- Segmentación E: citoplasma agrupado
NOTA: Este bloque utiliza núcleos segmentados previamente como marcadores individuales para iniciar la segmentación del citoplasma. Asegúrese de que el bloque de segmentación de núcleos se ha procesado antes de usarlo.- En la caja citoplasma de canal (caja 1), ajuste el canal de la señal citoplasma.
- En el cuadro Extension segmented nuclei (cuadro 2), escriba el formato utilizado para guardar imágenes de núcleos segmentados (tif, jpeg, bmp, png). El formato predeterminado es tif.
- En el cuadro Texto (cuadro3), escriba el nombre de la carpeta que contiene núcleos segmentados.
- En el cuadro Formato de imágenes de citoplasmas segmentadas (cuadro 4), establezca el formato que se utilizará para guardar imágenes de objetos segmentados (Tiff, Gif, Jpeg, BMP, PNG).
- Durante el proceso, se crea automáticamente una carpeta para guardar imágenes de citoplasmas segmentados. En el cuadro Texto (cuadro 5), asigne un nombre a esta carpeta (por ejemplo: citoplasmas segmentados). La carpeta se crea en la carpeta que contiene imágenes combinadas.
- Siga los mismos pasos que en el paso 5.3.1 para establecer los parámetros de los cuadros Filtro gaussiano y Contornos activos (Tenga cuidado, los rangos de caja no son los mismos que en el paso 5.3.1).
- Ejecute el flujo de trabajo (para obtener más información, consulte la parte 7).
- Segmentación A: Objetos no agrupados y Segmentación C: Objetos agrupados con formas convexas
6. Detección y análisis de señal fluorescente
- Seleccione el bloque adaptado.
- En el bloque Análisis de fluorescencia A: 1 Canal,realizar la detección y análisis de focos en un canal dentro de un tipo de objeto segmentado: detección de focos de bobina (canal rojo) dentro del núcleo.
- En el bloque Análisis de Fluorescencia B: 2 Canales en el mismo compartimiento,realizar la detección y análisis de focos en dos canales dentro de un tipo de objeto segmentado: detección de bobinado (canal rojo) y 53BP1 (canal verde) focos dentro del núcleo.
- En el bloque Análisis de Fluorescencia C: 2 Canales en dos compartimentos,realizar la detección y análisis de focos en uno o dos canales, específicamente dentro de los núcleos y su correspondiente citoplasma: detección de foci coilin (canal rojo) tanto dentro del núcleo como en su correspondiente citoplasma o detección de focos de Coilin (canal rojo) dentro del núcleo y foci G3BP (canal verde) dentro del citoplasma correspondiente.
- En el bloque Análisis de fluorescencia D: Translocación global,calcule el porcentaje de señal de un canal en dos compartimentos celulares (a y b). Por ejemplo, en un ensayo de translocación de citoplasma/núcleo, exporte porcentajes calculados de señales nucleares y citoplasmáticos para cada imagen en la hoja de cálculo final "Resultados". La fórmula utilizada para calcular el porcentaje de señal nuclear se muestra a continuación. Este bloque se puede utilizar para cualquier compartimento subcelular:
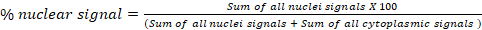
- En el bloque Análisis de fluorescencia E: Translocación celular individual,calcule el porcentaje de señal de un canal en dos compartimentos celulares para cada celda. Este bloque está especialmente optimizado para el ensayo de translocación de núcleo/citoplasma a nivel de una sola célula.
NOTA: Debido a que el bloque Análisis de fluorescencia E: La translocación celular individual realiza análisis a nivel de una sola célula, se necesita una segmentación eficiente del núcleo y del citoplasma.
- Vincule la salida0 (Archivo) del bloque Seleccionar carpeta (bloque 1) a la entrada de carpeta (flechas blancas en círculos negros) del bloque elegido.
- Establezca los parámetros del bloque elegido.
- Análisis de fluorescencia A: 1 canal, Análisis de fluorescencia B: 2 canales en el mismo compartimiento y Análisis de fluorescencia C: 2 canales en dos compartimentos
- En el cuadro Roi de imágenesde carpeta , escriba el nombre de la carpeta que contiene imágenes de objetos segmentados precedidos por una barra diagonal invertida. (Por ejemplo: núcleos segmentados).
- En el cuadro Formato de imágenes de objetos segmentados (cuadro 2), escriba el formato utilizado para guardar imágenes de objetos segmentados (tif, jpeg, bmp, png). El formato predeterminado es tif.
- En el cuadro Kill Borders ?, escriba Sí para eliminar objetos de borde. De lo contrario, escriba No. La instalación de la colección MorphoLibJ de ImageJ es necesaria para utilizar esta función (consulte el paso 5.3.3).
- En la(s) señal de puntos de canalde la caja, ajuste el canal donde se deben detectar los puntos. En las imágenes RGB clásicas, 0-Rojo, 1-Verde, y 2-Azul.
- En las cajas Nombre de la molécula localizada,escriba el nombre de la molécula que se localiza en las manchas. El número de campos a introducir depende del número de moléculas.
- En la(s) caja(s) Wavelet Spot Detector Block, establezca los parámetros de detección de punto para cada canal. Establezca la(s) escala(s) (referida(s) al tamaño del punto) y la sensibilidad de la detección (una sensibilidad menor disminuye el número de puntos detectados, el valor predeterminado es 100 y el valor mínimo es 0). La documentación exhaustiva de este plugin está disponible en línea: http://icy.bioimageanalysis.org/plugin/Spot_Detector. Los parámetros también se pueden definir manualmente en Icy (Detección y seguimiento de la función de seguimiento ( Detector de puntos).
- Como opción, en el cuadro Filtrar ROI por tamaño, filtre los objetos segmentados donde se detectan puntos estableciendo un intervalo de tamaño (en píxeles). Este paso es especialmente útil para eliminar objetos subs segmentados o subs segmentados. Para estimar manualmente el tamaño de los objetos, consulte el paso 5.3.1. Los parámetros predeterminados no incluyen el filtrado de ROI por tamaño. El bloque 2 canales en dos compartimentos contiene dos cajas: núcleos de filtro por tamaño (caja 19) y citoplasma de filtro por tamaño (caja 46).
- Opcionalmente, en el cuadro Filtrar manchas por tamaño, filtre los puntos detectados según su tamaño (en píxeles) para eliminar artefactos no deseados. Para estimar manualmente el tamaño del spot, haga clic en Detección y seguimiento y abra el plugin Detector de spot. En Opciones de salida, seleccione Exportar a ROI. Tenga cuidado de que los parámetros predeterminados no incluyan el filtrado de puntos por tamaño y que los puntos filtrados no se tengan en cuenta para el análisis. El número de campos a introducir depende del número de canales.
- Opcionalmente, en la caja Puntos de filtro,aplique un filtro adicional (contraste, homogeneidad, perímetro, redondez) en las manchas detectadas. Tenga cuidado de que los parámetros predeterminados no incluyan el filtrado de puntos y que los puntos filtrados no se tengan en cuenta para el análisis. El número de campos a introducir depende del número de canales.
- Opcionalmente, en las casillas Umbral de tamaño de spot, establezca un umbral para el área (en píxeles) de los puntos analizados. El número de puntos contados por debajo y por encima de este umbral se exporta en la hoja de cálculo Resultados final. El número de cajas a informar depende del número de canales.
- Ejecute el flujo de trabajo (para obtener más información, consulte la parte 7). Los datos se exportan en una hoja de cálculo Resultados guardada en la carpeta que contiene imágenes combinadas.
- Análisis de fluorescencia D: Análisis global de translocación y fluorescencia E: Translocación celular individual:
- En los cuadros Imágenes de carpeta (cuadros 1 y 2), escriba el nombre de la carpeta que contiene imágenes de objetos segmentados. En el bloque Análisis de fluorescencia D: Translocación global, los dos tipos de ROI se identifican como ROI a y ROI b. Para el bloque Análisis de fluorescencia E: Translocación de celda individual, en imágenes de carpetas segmentadas núcleos y imágenes de carpeta segmentadas citoplasmas, escriba el nombre de la carpeta que contiene núcleos segmentados y citoplasmas, respectivamente.
- En la caja Señal de canal (cuadro 3), ingrese el canal de la señal.
- En el cuadro Formato de imágenes de objetos segmentados (cuadro 4), escriba el formato utilizado para guardar imágenes de objetos segmentados (tif, jpeg, bmp, png). El formato predeterminado es tif. La opción Matar bordes también está disponible para eliminar objetos de borde (consulte el paso 6.3.1).
- Opcionalmente, en los cuadros Filtrar ROI por tamaño, filtre los objetos segmentados estableciendo un intervalo de tamaño (en píxeles). Este paso podría ser útil para eliminar objetos sub-o sobres segmentados. Para estimar manualmente el tamaño del objeto, consulte el paso 5.3.1. Hay dos campos para entrar, uno por canal. Los parámetros predeterminados no incluyen el filtrado de ROI por tamaño.
- Ejecute el flujo de trabajo (para obtener más información, consulte la parte 7). Exportar datos en una hoja de cálculo Resultados guardados en la carpeta que contiene imágenes combinadas.
- Análisis de fluorescencia A: 1 canal, Análisis de fluorescencia B: 2 canales en el mismo compartimiento y Análisis de fluorescencia C: 2 canales en dos compartimentos
7. Ejecute el protocolo
- Para procesar un bloque en una ejecución, elimine la conexión entre el bloque seleccionado y el bloque Seleccionar carpeta. Coloque el bloque deseado en elrango 1. En la esquina superior izquierda del bloque deseado, haga clic en el enlace directamente a la derecha de la carpeta. En el cuadro de diálogo Abrir que aparece, haga doble clic en la carpeta que contiene las imágenes combinadas. A continuación, haga clic en Abrir. Haga clic en Ejecutar para iniciar el flujo de trabajo. El procesamiento se puede detener haciendo clic en el botón Detener.
- Para procesar diferentes bloques en una ejecución, mantenga las conexiones de los bloques elegidos con el bloque Seleccionar carpeta (bloque 1). Asegúrese de que su rango permite el buen procesamiento del flujo de trabajo. Por ejemplo, si un bloque específico necesita objetos segmentados para procesar, asegúrese de que el bloque de segmentación se procesa antes. Antes de ejecutar el flujo de trabajo, quite los bloques no utilizados y guarde el nuevo protocolo con otro nombre.
- Haga clic en Ejecutar para iniciar el flujo de trabajo. Cuando aparezca el cuadro de diálogo abierto, haga doble clic en la carpeta que contiene las imágenes combinadas. A continuación, haga clic en Abrir. El flujo de trabajo se ejecuta automáticamente. Si es necesario, detenga el procesamiento haciendo clic en el botón Detener.
- Al final del procesamiento, compruebe que el mensaje El flujo de trabajo ejecutado apareció correctamente en la esquina inferior derecha y que todos los bloques se marcan con un signo verde ( Figura2b). Si no es así, el bloque y el cuadro interior que presenta el signo de error indican el elemento que se va a corregir(Figura 2b).
NOTA: Una vez que el flujo de trabajo se ejecutó correctamente, no se puede iniciar una nueva ejecución directamente y, para procesar el flujo de trabajo de nuevo, se debe marcar al menos un bloque con el signo "listo para procesar". Para cambiar el estado de un bloque, elimine y vuelva a crear un vínculo entre dos cuadros dentro de este bloque o simplemente cierre y vuelva a abrir el protocolo. Si se produce un error durante el procesamiento, se puede iniciar directamente una nueva ejecución. Durante una nueva ejecución, se procesan todos los bloques de la canalización, incluso si algunos de ellos se marcan con el signo verde.
Resultados
Todos los análisis descritos se han realizado en un portátil estándar (procesador de cuatro núcleos de 64 bits a 2,80 GHz con memoria de acceso aleatorio (RAM) de 16 GB que trabaja con la versión de 64 bits de Java. La memoria de acceso aleatorio es un parámetro importante a tener en cuenta, dependiendo de la cantidad y la resolución de las imágenes a analizar. El uso de la versión de 32 bits de Java limita la memoria a unos 1300 MB, lo que podría no ser adecuado para el análisis de big data, mientras que la v...
Discusión
Un número cada vez mayor de herramientas de software libre están disponibles para el análisis de imágenes de celdas de fluorescencia. Los usuarios deben elegir correctamente el software adecuado de acuerdo con la complejidad de su problema, a su conocimiento en el procesamiento de imágenes, y al tiempo que quieren pasar en su análisis. Icy, CellProfiler o ImageJ/Fiji son potentes herramientas que combinan facilidad de uso y funcionalidad3. Icy es una herramienta independiente que presenta un...
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
G.H. fue apoyado por una beca de posgrado de la Ministére Délégué a la Recherche et aux Technologies. L.H. fue apoyado por una beca de posgrado del Institut de Cancérologie de Lorraine (ICL), mientras que Q.T. fue apoyado por una subvención pública supervisada por la Agencia Nacional de Investigación de Francia (ANR) como parte del segundo programa "Investissements d'Avenir" FIGHT-HF (referencia: ANR-15-RHU4570004). Esta obra fue financiada por el CNRS y la Universidad de Lorena (UMR 7365).
Materiales
| Name | Company | Catalog Number | Comments |
| 16% Formaldehyde solution (w/v) methanol free | Thermo Fisher Scientific | 28908 | to fix the cells |
| Alexa Fluor 488 of goat anti-rabbit | Thermo Fisher Scientific | A-11008 | fluorescent secondary antibody |
| Alexa Fluor 555 of goat anti-mouse | Thermo Fisher Scientific | A-21425 | fluorescent secondary antibody |
| Alexa Fluor 555 Phalloidin | Thermo Fisher Scientific | A34055 | fluorescent secondary antibody |
| Bovine serum albumin standard (BSA) | euromedex | 04-100-812-E | |
| DMEM | Sigma-Aldrich | D5796-500ml | cell culture medium |
| Duolink In Situ Mounting Medium with DAPI | Sigma-Aldrich | DUO82040-5ML | mounting medium |
| Human: HeLa S3 cells | IGBMC, Strasbourg, France | cell line used to perform the experiments | |
| Hydrogen peroxide solution 30% (H2O2) | Sigma-Aldrich | H1009-100ml | used as a stressing agent |
| Lipofectamine 2000 Reagent | Thermo Fisher Scientific | 11668-019 | transfection reagent |
| Mouse monoclonal anti-coilin | abcam | ab11822 | Coilin-specific antibody |
| Nikon Optiphot-2 fluorescence microscope | Nikon | epifluoresecence microscope | |
| Opti-MEM I Reduced Serum Medium | Thermo Fisher Scientific | 31985062 | transfection medium |
| PBS pH 7.4 (10x) | gibco | 70011-036 | to wash the cells |
| Rabbit polyclonal anti-53BP1 | Thermo Fisher Scientific | PA1-16565 | 53BP1-specific antibody |
| Rabbit polyclonal anti-EDC4 | Sigma-Aldrich | SAB4200114 | EDC4-specific antibody |
| Triton X-100 | Roth | 6683 | to permeabilize the cells |
Referencias
- Möckl, L., Lamb, D. C., Bräuchle, C. Super-resolved fluorescence microscopy: Nobel Prize in Chemistry 2014 for Eric Betzig, Stefan Hell, and William E. Moerner. Angewandte Chemie. 53 (51), 13972-13977 (2014).
- Meijering, E., Carpenter, A. E., Peng, H., Hamprecht, F. A., Olivo-Marin, J. -. C. Imagining the future of bioimage analysis. Nature Biotechnology. 34 (12), 1250-1255 (2016).
- Wiesmann, V., Franz, D., Held, C., Münzenmayer, C., Palmisano, R., Wittenberg, T. Review of free software tools for image analysis of fluorescence cell micrographs. Journal of Microscopy. 257 (1), 39-53 (2015).
- de Chaumont, F., et al. Icy: an open bioimage informatics platform for extended reproducible research. Nature Methods. 9 (7), 690-696 (2012).
- Girish, V., Vijayalakshmi, A. Affordable image analysis using NIH Image/ImageJ. Indian J Cancer. 41 (1), 47 (2004).
- Zaitoun, N. M., Aqel, M. J. Survey on image segmentation techniques. Procedia Computer Science. 65, 797-806 (2015).
- . MetaMorph Microscopy Automation and Image Analysis Software Available from: https://www.moleculardevices.com/products/cellular-imaging-systems/acquisition-and-analysis-software/metamorph-microscopy (2018)
- . NIS-Elements Imaging Software Available from: https://www.nikon.com/products/microscope-solutions/lineup/img_soft/nis-element (2014)
- Meyer, F., Beucher, S. Morphological segmentation. Journal of Visual Communication and Image Representation. 1 (1), 21-46 (1990).
- Schieber, M., Chandel, N. S. ROS Function in Redox Signaling and Oxidative Stress. Current Biology. 24 (10), 453-462 (2014).
- D'Autréaux, B., Toledano, M. B. ROS as signalling molecules: mechanisms that generate specificity in ROS homeostasis. Nature Reviews. Molecular Cell Biology. 8 (10), 813-824 (2007).
- Davalli, P., Mitic, T., Caporali, A., Lauriola, A., D'Arca, D. ROS, Cell Senescence, and Novel Molecular Mechanisms in Aging and Age-Related Diseases. Oxidative Medicine and Cellular Longevity. 2016, 3565127 (2016).
- Disher, K., Skandalis, A. Evidence of the modulation of mRNA splicing fidelity in humans by oxidative stress and p53. Genome. 50 (10), 946-953 (2007).
- Takeo, K., et al. Oxidative stress-induced alternative splicing of transformer 2β (SFRS10) and CD44 pre-mRNAs in gastric epithelial cells. American Journal of Physiology - Cell Physiology. 297 (2), 330-338 (2009).
- Seo, J., et al. Oxidative Stress Triggers Body-Wide Skipping of Multiple Exons of the Spinal Muscular Atrophy Gene. PLOS ONE. 11 (4), 0154390 (2016).
- Will, C. L., Luhrmann, R. Spliceosome Structure and Function. Cold Spring Harbor Perspectives in Biology. 3 (7), 003707 (2011).
- Ljosa, V., Sokolnicki, K. L., Carpenter, A. E. Annotated high-throughput microscopy image sets for validation. Nature Methods. 9 (7), 637-637 (2012).
- Wang, Q., et al. Cajal bodies are linked to genome conformation. Nature Communications. 7, (2016).
- Carpenter, A. E., et al. CellProfiler: image analysis software for identifying and quantifying cell phenotypes. Genome Biology. 7, 100 (2006).
- McQuin, C., et al. CellProfiler 3.0: Next-generation image processing for biology. PLoS Biology. 16 (7), 2005970 (2018).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados