需要订阅 JoVE 才能查看此. 登录或开始免费试用。
Method Article
LTE 的生产和优化,一种用于重组蛋白生产的利 什曼原虫 衍生的无细胞蛋白表达系统
摘要
利什曼原虫翻译提取物 (LTE) 是一种源自单细胞寄生虫利 什曼原虫的真核无细胞蛋白表达系统。这种优化的协议使 LTE 的制造变得简单且具有成本效益。它适用于专注于复杂真核蛋白及其相互作用的多平行表达和研究的各种应用。
摘要
该方案概述了源自单细胞鞭毛利 什曼原虫 tarentolae 的真核无细胞蛋白表达系统 (CFPS) 的生产和优化,称为利什曼原虫翻译提取物或 LTE。虽然这种生物最初是作为壁虎的寄生虫进化而来的,但它可以在烧瓶或生物反应器中轻松且廉价地培养。与 重型利什曼原虫不同,它对人类无致病性,不需要特殊的实验室预防措施。将利什曼原虫用于 CFPS 的另一个优点是,向 CFPS 中添加单个反义寡核苷酸,靶向所有蛋白质编码 RNA 的 5' 端的保守剪接前导序列,可以抑制内源性蛋白质表达。我们提供细胞破碎和裂解物处理的程序,与以前的版本相比,这些程序已得到简化和改进。这些程序从简单的培养瓶培养开始。此外,我们还解释了如何使用包含物种非依赖性翻译起始位点 (SITS) 的载体引入遗传信息,以及如何进行简单的批次优化和质量控制以确保一致的蛋白质表达质量。
引言
在 1960 年代,无细胞蛋白表达系统在揭示遗传密码方面发挥了关键作用1。然而,主要基于大肠杆菌的原核无细胞蛋白表达系统目前在实验室和商业应用中占主导地位。虽然基于大肠杆菌的系统具有成本效益、可扩展性和高表达产量等优势,但它们在生产活性形式的多结构域蛋白和促进蛋白质复合物的组装时面临挑战 2,3。如今,真核生物无细胞蛋白合成 (CFPS) 的常用形式包括小麦胚芽提取物 (WGE)、兔网织红细胞裂解物 (RRL) 和昆虫细胞裂解物 (ICL)4,5,6。这项工作介绍了一种基于单细胞鞭毛寄生虫 Leishmania tarentolae 的替代真核无细胞系统,该系统既简单又可扩展。
利什曼原虫 可以使用经济高效的培养基在培养瓶中轻松培养,也可以在生物反应器中扩大规模以实现更高的细胞密度。细胞裂解物中存在的内源性 mRNA(否则可能会与引入的信息竞争)可以使用靶向保守利什曼原虫 mRNA 剪接前导序列的反义寡核苷酸7 进行中和。与导致人类疾病的大 利什曼原虫不同, L. tarentolae 会感染沼泽壁虎 (Tarentolae mauritanica),使其适合在 PC2 实验室环境中培养,无需特殊预防措施。它以前曾被用作体内蛋白表达的转基因生物8。
为了促进无细胞系统中的模板引发,基于聚合物 RNA 结构设计了通用序列,以增强翻译起始9。这些不依赖物种的翻译序列 (SITS) 适用于原核和真核无细胞系统,并且适合将遗传信息引入 LTE。虽然该方案没有提供 LTE 无细胞蛋白表达载体构建的详细解释,但优化和质量控制需要合适的载体,其中包含 SITS 位点下游所需目标蛋白质的荧光团融合。为此,已将适当的 LTE 载体存放在 Addgene 基因存储库中,例如 pCellFree_G03 载体,它使用 Gateway 克隆位点编码 N 端 eGFP 融合到所需的目标蛋白质。
LTE 已在需要蛋白质表达的广泛应用中证明了其价值,包括蛋白质自组装分析 10,16、人整合膜蛋白的生产17、抗病毒候选药物的研究18、生物技术有用的酶的开发19、蛋白质生物传感器的原型设计20,21 以及钩虫生物制剂的研究22.LTE 还有助于绘制病毒学和细胞结构领域的蛋白质-蛋白质相互作用网络21,32。LTE 的基准性能与其他真核无细胞系统相似,可表达全长、单分散和非聚集蛋白33,同时提供更具成本效益和可扩展性的生产。
该方案提供了培养和破坏宿主生物体、制备裂解物和补充用于偶联转录/翻译蛋白表达的补料溶液 (FS) 的技术。此外,它还包括用于优化生产批次的协议。在利什曼原虫无细胞系统的初始版本中,在表达水平、全长蛋白质的分数和蛋白质聚集体的存在方面观察到不希望的批次间变化,导致批次34 的处理。随后对协议进行了改进以解决此问题25。当前的方案建立在这些改进的基础上,允许针对单个批次的峰值蛋白质表达和大小进行优化。它通过密切控制细胞破坏物负载(以 600 nm 处的光密度测量;OD600 nm),并使用 280 nm 处的吸光度 (Abs280 nm) 对所得裂解物输出进行归一化。此外,它还结合了一种在制造过程中用 rNTP 和镁部分补充裂解物的方法,随后在测试表达期间优化这些补料溶液组分。尽管此优化在协议中作为一个选项提供,但作者强烈建议这样做。
研究方案
该方案包括详细的培养基配方和步骤,包括培养、离心、使用多功能酶标仪测量 GFP 荧光、测量培养物 OD600 nm 和评估裂解物 Abs280 nm。它还涵盖了 SDS-PAGE 蛋白凝胶的设置和成像。该实验方案所需或建议的材料列在材料电子表格中。需要注意的是,除非另有说明,否则典型的实验室资源(如培养基组件、离心机、试管、分光光度计和凝胶电泳装置)可以互换使用。 图 1 提供了 LTE 制造过程的摘要。
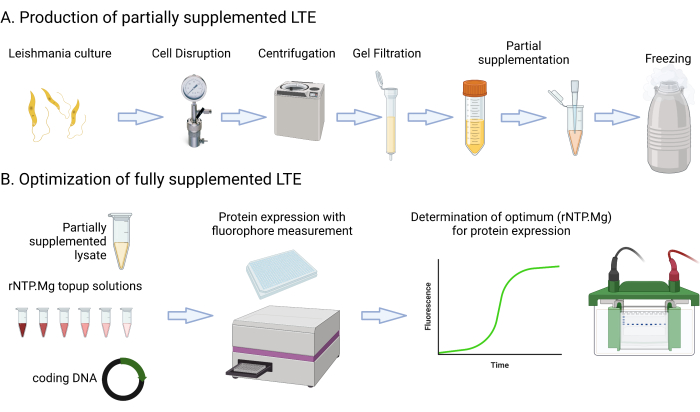
图 1:LTE 制造协议概述。 此漫画简要概述了 LTE 制造协议。 请单击此处查看此图的较大版本。
1. 利什曼原虫培养物的生长
- 准备至少 3 L TBGG 生长培养基(细菌蛋白胨 12 g/L、酵母提取物 24 g/L、甘油 8 mL/L、葡萄糖 1 g/L、KH2PO4 2.3 g/L、K2HPO4 2.5 g/L,参见 材料表)。在真空或类似设置下使用 0.22 μm 过滤器对培养基进行消毒。
- 将培养基储存在室温 (RT) 下,在接种 L. tarentolae 之前添加最终添加物(血红素、抗生素)。以 0.2% v/v 的浓度加入血红素(0.25% v/v 的 50% 三乙醇胺溶液中),以 0.5% v/v 的浓度加入青霉素(10,000 单位/mL)加链霉素(10,000 μg/mL)混合物。
注意:该方案的起点是野生型 L. tarentolae 的维持 2 x 10 mL 培养物。维持培养物在 27 °C 下在标准 50 mL 组织培养瓶中以低振荡 (75 rpm) 生长。这种 10 mL 培养物可以每 2-3 天在无菌(TBGG + 血红素、青霉素、链霉素)中以 ~1/20 稀释液无限期维持。建议在 PC2 实验室中使用标准生物安全柜;然而,添加的抗生素往往可以防止细菌污染,而真菌污染通常可以通过 L. tarentolae 来消除。 - 在两天内,通过 1:10 稀释液将 L. tarentolae 维持培养物扩增至 200 mL(第 1 天),然后通过 2 L(第 2 天),每天增加 TBGG + 血红素/抗生素的体积。在高压灭菌的带挡板的 5 L 玻璃瓶(最大填充至 1 L)中进行两次稀释。第二次稀释必须在下午 3 点至 6 点之间进行,目的是在第二天上午 8 点至 11 点之间开始裂解液生产。
注意:该方案使用 LTE 生产的最小起始体积(2 x 1 L 培养物)。还可以通过加入额外的扩增步骤(例如,第 1 天:100 mL;第 2 天:1 升;第 3 天:10 升)。尽管该方案使用带挡板的烧瓶(参见 材料表)来培养 L. tarentolae,但可以选择使用设计用于带有 Rushton 叶轮的细菌生长的常规生物反应器,前提是搅拌速率保持在 100 rpm 以下。生物反应器中改进的通气和 pH 控制通常会延长 L. tarentolae 培养物的对数期生长,从而允许在步骤 1.4 中使用更高的收获 OD600 nm (10)。 - 通过在 TBGG 中以 1:10 稀释度直接在分光光度计比色皿中记录培养物的 OD600nm,一式三份。制备裂解物的合适起始范围是 OD600nm = 4.0-8.0。
- 如果 OD为 600 nm ,则提供额外的孵育时间< 4.0。OD600 nm > 8 的培养物是可用的,并且会产生更大体积的无细胞表达裂解物,但由于对数较晚生长期开始,因此质量较低。将培养瓶放在冰上,等待后续步骤。
注:准确测量最终培养物的 OD600nm 至关重要,因为它用于计算破碎前浓缩细胞的最终体积。该计算取代了早期版本的 LTE 制造中使用的颗粒称重方法,用于在破碎34 之前校准细胞浓度,以简化协议。确保在 TBGG 中稀释以进行 OD600nm 测量,否则渗透压休克会改变电池形状,从而导致测量误差。在进行分光光度读数前几秒钟,移液器将 1:10 稀释液混合用于 OD600nm 测量(直接在比色皿中),因为 L. tarentolae 细胞迅速沉淀并呈现独特的混浊外观。如果认为表达培养物的最终体积是近似的,则还建议在收获时称量培养瓶(使用合适的空培养瓶皮重)以获得更好的估计体积(1 g = 1 mL)。L. tarentolae 在挡板培养瓶中生长的最大OD 600nm 为 15-20,但由于达到固定相,这不适合裂解物生产。
2. L. tarentolae 培养物的浓度
- 利 什曼原虫 细胞必须在破碎前洗涤和浓缩约 60 倍。根据最终浓缩液的 OD600nm = 300 计算细胞浓度的目标体积。方程式为 V = 收获体积 (mL) x(收获 OD600nm/300)。例如,使用收获物 OD600 = 5 的 2 L 培养物,目标体积为 33 mL。
注意:可以修改 300 的 OD600nm 靶标;以前的 LTE 产品使用 150-350 范围内的值。较高浓度的细胞进入破碎状态往往会产生最终的无细胞表达反应,蛋白质产量更高,但脆弱蛋白质聚集的趋势增加。OD600nm = 300 代表适合 LTE 生产的默认目标。 - 将收获的培养物转移到合适的离心瓶中,并在 4 °C 下以 2500 x g 旋转 10 分钟。 小心地将上清液倒入培养废液中。
注:尽量减少细胞损失到丢弃的上清液中非常重要,因为它会影响破坏负荷的计算。在以前的 LTE 生产方案中,通过在测试微量离心管中旋转细胞浓缩物并测量沉淀重量与总重量34 来校准用于破碎的 L. tarentolae 细胞的浓度。相反,这种简化的方案基于测得的收获 OD600 nm 对浓缩物使用理论 OD600nm 靶标,并假设在细胞浓缩和洗涤过程中细胞损失较低。 - 在 SEB 缓冲液(45 mM HEPES-KOH pH 7.6、250 mM 蔗糖、100 mM KOAc、3 mM Mg(OAc)2,保持在冰上)中洗涤细胞沉淀 3 次,每次在 4 °C 下以 2500 x g 离心 10 分钟。 第一次洗涤时,将每 1 L 沉淀培养物重悬于 100 mL SEB 缓冲液中,然后将它们合并到单个离心瓶中。对于第二次洗涤,每 1 L 原始培养物也使用 100 mL 的 SEB。
注:对于最终沉淀重悬,将 SEB 缓冲液添加至最终目标重悬体积的 50%(步骤 2.1)。这样就可以在步骤 2.4 中小心地将混合浓缩物加注至最终目标体积。每次重悬必须尽可能温和,以避免 L. tarentolae 过早裂解,例如,将添加的 SEB 轻轻旋转在倾析的沉淀上,或在粘附在离心管壁上的沉淀上移液 SEB。在最后一步中,将上清液转移到较小的离心管中可能更方便。 - 将重悬的浓缩物倒入合适的清洗玻璃容量圆筒中,然后使用额外的冷 SEB 将体积加满至目标体积(步骤 2.1)并轻轻混合。
3. L. tarentolae 浓缩物的裂解
- 将细胞浓缩物转移到预冷至 4 °C 的氮气空化装置(参见 材料表)中,加压至 70 bar 氮气,并在冰上孵育 45 分钟。
注意:虽然氮气空化干扰器不是常见的实验室物品,但建议将其用于 LTE 生产。已经尝试了细胞冻融和 French Press 型干扰剂等替代方法;然而,与使用氮空化方法相比,蛋白质表达活性为 <50%。氮气空化装置在使用前和运行之间必须彻底清洁,类似于从此步骤开始与细胞裂解物接触的所有重复使用的容器(例如接收瓶)。合适的清洁方案包括用实验室清洁剂清洗,然后用去离子水彻底冲洗。 - 打开氮气空化装置上的通风口,将所得裂解物排放到适当坚固的容器中,例如冰上的真空接收瓶。倾斜接收瓶,确保所有得到的裂解物都沉降,并可以移液到新的离心管或类似容器中。
注意:氮气空化破坏剂依赖于细胞浓缩物从 70 bar 氮气到环境压力的突然转变,这是通过首先液体的强烈流动,然后是氮气通过设备的出口阀来实现的。必须在化学安全罩中使用适当的个人防护设备 (PPE) 进行通风。存在破坏目标容器和丢失裂解物的风险,这就是我们使用坚固的真空接收器而不是通用培养瓶的原因。如果设备的出口阀是管子,请避免将管子直接放在接收器内,以防止在排气点积聚过大的压力。
4. 细胞裂解物的离心
- 将裂解物转移到合适的额定 g 力离心管中,并在 4 °C 下以 10,000 x g 离心 15 分钟。 将上清液去除到新鲜的类似离心管中。
- 将裂解物在 4 °C 下以 30,000 x g 离心 15 分钟,然后将最终上清液除去到放置在冰上的新鲜离心管或类似容器中。估计总体积。
5. 细胞裂解物的凝胶过滤
注:凝胶过滤用于去除 SEB 缓冲液中包含的蔗糖。虽然蔗糖有助于在细胞破碎过程中稳定细胞机制,但如果保留在蛋白质表达反应中,则会降低产量。
- 以机架形式设置足够数量的 PD-10 重力进料凝胶过滤柱(参见 材料表),使其能够滴入收集盘或下面的类似容器中,确保它们可以以每根柱 2.5 mL 的速度过滤整个裂解物体积。通过预先通过 10 mL 4 °C EB 缓冲液(45 mM HEPES-KOH pH 7.6、100 mM KOAc、3 mM Mg(OAc)2)来预平衡色谱柱。
注意:从这一点开始的所有步骤都受益于在 4 °C 冷藏室中进行。但是,将所有裂解物和试剂保存在台式冰盘上也是合适的。一个例外是凝胶过滤步骤,其中作者在重新缓冲时将一架柱放入 4 °C 冰箱中。在该方案的原始版本中,新的凝胶过滤柱通过最初缓冲裂解物并丢弃第一个输出而被“阻塞”。虽然这被认为没有必要,但必须用 EB 缓冲液冲洗色谱柱,并在两次裂解物之间储存在 4 °C 下。由于裂解物组分在新色谱柱上保留了一些背景,因此第一个输出裂解物的蛋白表达活性可能低于后续输出。 - 向每根柱中加入 2.5 mL 裂解物,等待其进入柱中。再加入 0.5 mL EB,将裂解物沉降到色谱柱中,同时丢弃洗脱液。
- 向每根色谱柱中额外添加 2.5 mL EB 来洗脱凝胶过滤的裂解物,通过在色谱柱下方放置一个新鲜、干净的托盘或其他容器来收集结果。
6. 补充细胞裂解物
- 使用 nanodrop 分光光度计(参见 材料表)测量凝胶过滤裂解物的 Abs280nm 。如果超过 60,则使用额外的 4 °C EB 缓冲液将其稀释至达到 Abs280nm = 60。
注:虽然使用 OD600 nm 控制输入到破碎中的细胞密度大约决定了裂解物输出强度,但在裂解物破碎和处理后对 Abs280 nm 进行归一化可进一步提高裂解物性能的批次一致性。裂解物 Abs280 nm 可以上下调整,从而影响蛋白质表达产量和聚集(参见“讨论”部分)。如果未补充的裂解物表明 Abs280 nm < 60,则可能需要在破碎步骤中加入更多的利什曼原虫生物量,即在步骤 2.1 中将细胞破碎物负载增加到 OD600 nm > 300。 - 以 2:5 的比例向裂解物中加入 5x 补料溶液(5x FS, 表 1),并彻底涡旋混合。将其分装到合适的容器中(例如,1.5 mL 微量离心管),并在液氮中快速冷冻。如果按照下面的可选步骤 7.1-7.3 进行 LTE 表达式优化,请使用 表 1 中降低的 rNTP.Mg 5x FS,而不是默认的 5x FS。包括 5 x 100 μL 等分试样用于冷冻,以用于优化实验。
注:使用默认的 5x FS 以 2:5 的比例冷冻可产生表达即用型 LTE,以 7 μL/10 μL 表达使用(因此,5x FS 在最终反应中变为 1x FS)。然而,作者建议遵循进一步的可选步骤,其中 5x FS 中提供 0.6 倍的默认 rNTP 和镁量。接下来是一个优化步骤,其中添加两者的等摩尔混合物(称为 rNTP.Mg)以将测试反应补充到优化值。部分 5x FS 还包含一个寡核苷酸,可关闭内源性 mRNA 表达(参见“简介”部分)。寡核苷酸的序列是 CAATAAAGTACAGAAACTGATACTTATAGCGTT。
7. 最终补充 LTE 的 QC 和优化
注:确定向还原的 rNTP 和补充镁的裂解物中适当“补充”添加 rNTP.Mg 的最少必要步骤包括在没有融合伴侣的情况下表达 eGFP 或类似的荧光团(例如,sfGFP)。向反应中加入浓度递增的 rNTP.Mg 以确定表达水平( 通过 多功能酶板机以 eGFP RFU 测量)优化的点。eGFP 的过早终止(不发出荧光)在过高的 eGFP rNTP.Mg 浓度下降低会变得明显。然而,LTE 的短产物功能障碍在较大表达的蛋白 (>50 kDa) 中更频繁地发生。因此,可以使用比 eGFP 更大的模板来执行此优化,特别是如果模板可用于合适的表达载体,从而提供 LTE 为特定应用或研究所需的荧光团融合(参见代表性结果部分)。
- 解冻 100 μL 等分试样并设置六个 10 μL 表达反应,每个反应由步骤 6.2 中部分补充的 7 μL 裂解物、1 μL 补充溶液(如 表 2)和 2 μL 超纯水组成,其中含有足够的 DNA 对照模板,可在反应中达到 50 ng/μL 的最终浓度。
- 将反应物在25°C下孵育2小时,并使用多功能读板器监测GFP荧光的增加。
注意:GFP 的合适配置值为 485 nm(带宽 5 nm)激发,516 nm(带宽 5 nm)发射,读数间隔为 1 分钟,持续 2 小时。 - 对最终表达值进行排名,以确定对应于最高 eGFP RFU 的 rNTP.Mg 浓度。如果动力学数据可用,则在 2 小时表达期间 eGFP RFU 的双相增加也将表明过量 rNTP.Mg(参见代表性结果部分)。
- 一旦确定了优化的补充 rNTP.Mg 浓度,使用在前面步骤中部分补充的 LTE 批次将其添加到所有进一步的蛋白质表达中。
注意:如果步骤 6.2 中的等分试样是用固定体积小心完成的,则可以在不解冻的情况下回顾性地将加注添加到每个等分试样中,例如,将等分试样置于干冰上。这些等分试样现在已完全补充,因为当它们解冻和混合以供使用时,正确的 rNTP.Mg 填充物将通过每个等分试样混合。
结果
无细胞蛋白表达的目的是以折叠的活性形式产生全长蛋白,适用于广泛的应用。LTE(利什曼原虫 提取物)之前已与其他原核和真核无细胞表达系统进行了比较,在最佳操作时表现出避免截断和聚集的高能力,特别是与基于 大肠杆菌的无细胞表达相比33。然而,这在以前伴随着输出质量的显著批次间差异。当前方法进行了进一步改进,以确?...
讨论
用于创建 LTE 的协议在过去十年中已经发布7 ,并经历了定期更新25,34。然而,该技术的新手经常会遇到陡峭的学习曲线,导致延迟实现高质量和高产量的蛋白质表达。其他研究小组也报告了类似的挑战 LTE35,特别是关于显著的批次间变化。基于视频的协议格式可能会提供额外的、不太明显的?...
披露声明
不存在相互竞争的经济利益。
致谢
作者要感谢在过去 10 年中为 LTE 系统开发做出贡献的许多 Alexandrov 实验室成员,特别是开创该系统并开发 SITS 核糖体进入位点的 Sergey Mureev。 图 1 由 Biorender.com 创建,经许可复制。
材料
| Name | Company | Catalog Number | Comments |
| PD-10 SuperDex 25 Columns | Cytiva | 17085101 | Gel filtration columns |
| Nitrogen Cavitation cell disrupter | Parr Industries | 4635 or 4639 | Cell Disrupter |
| Bovine derived Hemin | Sigma-Aldrich | H5533 | Culture additive |
| Penicillin/Streptomycin 10000U/ml | Thermo-Fisher | 15140122 | Antibiotic mix |
| Optiplate 384 | Perkin-Elmer | 6007290 | Multiwell plate for 10ul expressions |
| Oligonucleotide | IDT synthesis | Oligo with sequence CAATAAAGTACAGAAACTGATAC TTATATAGCGTT | |
| Creatine Phosphokinase | Sigma-Aldrich | 9001-15-4 | Enzyme |
| Tecan Spark | Tecan | or similar Multimode Platereader | |
| Chemidoc MP Imager | Biorad | or similar SDS-PAGE gel Imager | |
| 4-12% Bis-Tris Gels | Invitrogen | NW04125 | SDS-PAGE gels |
| Biophotometer | Eppendorf | or similar Cuvette Specrophotometer | |
| Nanodrop One | Thermofisher | Nanodrop spectrophotometer | |
| Avanti JXN-26 centrifuge | Beckman Coulter | or similar centrifuge, with rotors/tubes rated 10K and 50K g | |
| 5424R microcentrifuge | Eppendorf | or similar microcentrifuge, with 1.5ml microcentrifuge tubes | |
| Flask Incubator Inova S44i | Eppendorf | or similar flask incubator shaker suitable for 5L Flasks | |
| 5L glass culture flasks | Baffled glass flasks for culture growth | ||
| Bactotryptone | BD | 211705 | Growth medium |
| Yeast Extract | Merck | VM930053 | Growth medium |
| Glycerol | Any analytical grade | ||
| Glucose | Any analytical grade | ||
| KH2PO4 | Any analytical grade | ||
| K2HPO4 | Any analytical grade | ||
| UltraPure water | Invitrogen | 10977-015 | Or output from any MilliQ-type water dispenser |
参考文献
- Nirenberg, M. W., Matthaei, J. H. The dependence of cell-free protein synthesis in E.coli upon naturally occurring or synthetic polyribonucleotides. Proc Natl Acad Sci USA. 47 (10), 1588-1602 (1961).
- Caschera, F., Noireaux, V. Synthesis of 2.3 mg/ml of protein with an all Escherichia coli cell-free transcription-translation system. Biochimie. 99, 162-168 (2014).
- Kelwick, R., Webb, A. J., MacDonald, J. T., Freemont, P. S. Development of a Bacillus subtilis cell-free transcription-translation system for prototyping regulatory elements. Metab Eng. 38, 370-381 (2016).
- Ezure, T., et al. Cell-free protein synthesis system prepared from insect cells by freeze-thawing. Biotechnol Prog. 22 (6), 1570-1577 (2006).
- Harbers, M. Wheat germ systems for cell-free protein expression. FEBS Letters. 588 (17), 2762-2773 (2014).
- Kobs, G. Selecting the cell-free protein expression system that meets your experimental goals. Promega Corporation. 21, 6-9 (2008).
- Kovtun, O., et al. Leishmania cell-free protein expression system. Methods. 55 (1), 58-64 (2011).
- Basile, G., Peticca, M. Recombinant protein expression in Leishmania tarentolae. Mol Biotechnol. 43 (3), 273-278 (2009).
- Mureev, S., Kovtun, O., Nguyen, U. T., Alexandrov, K. Species-independent translational leaders facilitate cell-free expression. Nat Biotechnol. 27 (8), 747-752 (2009).
- Gambin, Y., et al. Single-molecule fluorescence reveals the oligomerization and folding steps driving the prion-like behavior of ASC. J Mol Biol. 430 (4), 491-508 (2018).
- Sierecki, E., et al. Rapid mapping of interactions between human SNX-BAR proteins measured in vitro by AlphaScreen and single-molecule spectroscopy. Mol Cell Proteomics. 13 (9), 2233-2245 (2014).
- Sierecki, E., et al. Nanomolar oligomerization and selective co-aggregation of alpha-synuclein pathogenic mutants revealed by single-molecule fluorescence. Sci Rep. 6, 37630 (2016).
- Leitao, A., Bhumkar, A., Hunter, D. J. B., Gambin, Y., Sierecki, E. Unveiling a selective mechanism for the inhibition of alpha-synuclein aggregation by beta-synuclein. Int J Mol Sci. 19 (2), 334 (2018).
- Gambin, Y., et al. Single-molecule analysis reveals self assembly and nanoscale segregation of two distinct cavin subcomplexes on caveolae. Elife. 3, e01434 (2013).
- Ve, T., et al. Structural basis of TIR-domain-assembly formation in MAL- and MyD88-dependent TLR4 signaling. Nat Struct Mol Biol. 24 (9), 743-751 (2017).
- Guo, Z., et al. Subunit organisation of in vitro reconstituted HOPS and CORVET multisubunit membrane tethering complexes. PLoS One. 8 (12), e81534 (2013).
- Ruehrer, S., Michel, H. Exploiting Leishmania tarentolae cell-free extracts for the synthesis of human solute carriers. Mol Membr Biol. 30 (4), 288-302 (2013).
- Varasteh Moradi, S., et al. Mapping Interactions among cell-free expressed Zika virus proteins. J Proteome Res. 19 (4), 1522-1532 (2020).
- Gagoski, D., et al. Cell-free pipeline for discovery of thermotolerant xylanases and endo-1,4-beta-glucanases. J Biotechnol. 259, 191-198 (2017).
- Ergun Ayva, C., et al. Exploring performance parameters of artificial allosteric protein switches. J Mol Biol. 434 (17), 167678 (2022).
- Lau, D., et al. Fluorescence biosensor for real-time interaction dynamics of host proteins with HIV-1 capsid tubes. ACS Appl Mater Interfaces. 11 (38), 34586-34594 (2019).
- Ryan, S. M., et al. Novel antiinflammatory biologics shaped by parasite-host coevolution. Proc Natl Acad Sci USA. 119 (36), e2202795119 (2022).
- McMahon, K. A., et al. Identification of intracellular cavin target proteins reveals cavin-PP1alpha interactions regulate apoptosis. Nat Commun. 10 (1), 3279 (2019).
- Sierecki, E., et al. A cell-free approach to accelerate the study of protein-protein interactions in vitro. Interface Focus. 3 (5), 20230018 (2013).
- Johnston, W. A., Moradi, S. V., Alexandrov, K. Adaption of the Leishmania cell-free expression system to high-throughput analysis of protein interactions. Methods Mol Biol. 2025, 403-421 (2019).
- Jung, W., et al. Cell-free formation and interactome analysis of caveolae. J Cell Biol. 217 (6), 2141-2165 (2018).
- Fontaine, F. R., et al. Functional domain analysis of SOX18 transcription factor using a single-chain variable fragment-based approach. MAbs. 10 (4), 596-606 (2018).
- Overman, J., et al. Pharmacological targeting of the transcription factor SOX18 delays breast cancer in mice. Elife. 6, e21221 (2017).
- Kubala, M. H., et al. Mammalian farnesyltransferase alpha subunit regulates vacuolar protein sorting-associated protein 4A (Vps4A)--dependent intracellular trafficking through recycling endosomes. Biochem Biophys Res Commun. 468 (4), 580-586 (2015).
- Han, S. P., et al. Cortactin scaffolds Arp2/3 and WAVE2 at the epithelial zonula adherens. J Biol Chem. 289 (11), 7764-7775 (2014).
- Das Gupta, K., et al. Class IIa histone deacetylases drive toll-like receptor-inducible glycolysis and macrophage inflammatory responses via pyruvate kinase M2. Cell Rep. 30 (8), 2712-2728.e8 (2020).
- Leitão, A. D. G., et al. Selectivity of protein interactions along the aggregation pathway of α-synuclein. BioRxiv. , (2021).
- Gagoski, D., et al. Performance benchmarking of four cell-free protein expression systems. Biotechnol Bioeng. 113 (2), 292-300 (2016).
- Johnston, W. A., Alexandrov, K. Production of eukaryotic cell-free lysate from Leishmania tarentolae. Methods Mol Biol. 1118, 1-15 (2014).
- Hunter, D. J. B., Bhumkar, A., Giles, N., Sierecki, E., Gambin, Y. Unexpected instabilities explain batch-to-batch variability in cell-free protein expression systems. Biotechnol Bioeng. 115 (8), 1904-1914 (2018).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。