このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
リーシュマニアタレントラエ由来の無細胞タンパク質発現システムであるLTEの作製と最適化
要約
Leishmania Translational Extract(LTE)は、単細胞寄生虫である Leishmania tarentolaeに由来する真核生物の無細胞タンパク質発現系です。この最適化されたプロトコルにより、LTEは製造が簡単で費用対効果に優れています。これは、複雑な真核生物タンパク質とその相互作用のマルチパラレル発現と研究に焦点を当てたさまざまなアプリケーションに適しています。
要約
このプロトコルでは、Leishmania Translational ExtractまたはLTEと呼ばれる単細胞鞭毛 Leishmania tarentolaeに由来する真核生物の無細胞タンパク質発現システム(CFPS)の作製と最適化について概説しています。この生物はもともとヤモリの寄生虫として進化しましたが、フラスコやバイオリアクターで簡単かつ安価に培養できます。 リーシュマニアメジャーとは異なり、ヒトに対して非病原性であり、特別な実験室での予防措置は必要ありません。CFPSにリーシュマニアを使用するもう一つの利点は、すべてのタンパク質コードRNAの5'末端に保存されたスプライスリーダー配列を標的とする単一のアンチセンスオリゴヌクレオチドをCFPSに追加することで、内因性タンパク質の発現を抑制できることです。細胞破壊とライセート処理の手順を提供しており、以前のバージョンと比較して簡素化および改善されています。これらの手順は、単純なフラスコ培養から始まります。さらに、種に依存しない翻訳開始部位(SITS)を含むベクターを使用して遺伝情報を導入する方法や、一貫したタンパク質発現品質を確保するための簡単なバッチ最適化と品質管理を行う方法についても説明します。
概要
1960年代には、無細胞タンパク質発現系が遺伝暗号1の解明に重要な役割を果たしました。しかし、主に大腸菌に基づく原核生物の無細胞タンパク質発現系は、現在、実験室および商業アプリケーションの両方を支配しています。大腸菌ベースのシステムは、費用対効果、スケーラビリティ、高発現収率などの利点を提供する一方で、活性型のマルチドメインタンパク質を産生し、タンパク質複合体の組み立てを容易にする際に課題に直面します2,3。現在、真核生物の無細胞タンパク質合成(CFPS)の一般的に使用される形態には、小麦胚芽抽出物(WGE)、ウサギ網状赤血球溶解物(RRL)、および昆虫細胞溶解物(ICL)4,5,6が含まれます。この研究では、単細胞の鞭毛虫であるLeishmania tarentolaeに基づく、簡単でスケーラブルな代替の真核生物無細胞システムを紹介します。
リーシュマニアタレントラエ は、費用対効果の高い培地を使用してフラスコで簡単に培養でき、バイオリアクターでスケールアップして細胞密度を高めることもできます。細胞ライセート中の内因性mRNAの存在は、導入されたメッセージと競合する可能性がありますが、保存されたリーシュマニアmRNAスプライスリーダー配列7を標的とするアンチセンスオリゴヌクレオチドを使用して中和できます。ヒトの病気を引き起こす近縁種の リーシュマニアメジャーとは異なり、 L. tarentolae はムーアヤモリ(Tarentolae mauritanica)に感染するため、特別な予防措置を必要とせずにPC2実験室環境での培養に適しています。これまで、in vivoタンパク質発現のためのトランスジェニック生物として使用されてきました8。
無細胞系におけるテンプレートプライミングを容易にするために、翻訳開始を増強する高分子RNA構造に基づくユニバーサル配列が設計されています9。これらの種に依存しない翻訳配列(SITS)は、原核生物と真核生物の両方の無細胞システムに適用でき、LTEに遺伝情報を導入するのに適しています。このプロトコールでは、LTE無細胞タンパク質発現のためのベクター構築について詳細な説明は提供していませんが、最適化と品質管理には、SITSサイトの下流に目的の目的のタンパク質のフルオロフォア融合体を含む適切なベクターが必要です。この目的のために、Gatewayクローニングサイトを使用して目的のタンパク質へのN末端eGFP融合をコードするpCellFree_G03ベクターなどの適切なLTEベクターがAddgene遺伝子リポジトリに寄託されています。
LTEは、タンパク質の自己組織化10,16の解析、ヒト内在性膜タンパク質の産生17、抗ウイルス薬候補の研究18、バイオテクノロジーに有用な酵素の開発19、タンパク質バイオセンサーのプロトタイピング20,21、鉤虫からの生物製剤の研究など、タンパク質発現を必要とする幅広いアプリケーションでその価値を証明しています.LTEは、ウイルス学および細胞構造21,32の分野におけるタンパク質-タンパク質相互作用ネットワークのマッピングにも役立っています。LTEは、他の真核生物の無細胞系と同様に、完全長、単分散、および非凝集タンパク質を発現する性能を発揮することがベンチマークされており33、同時に、より費用対効果が高くスケーラブルな生産を提供します。
このプロトコルは、宿主生物の培養と破壊、ライセートの調製、および結合転写/翻訳タンパク質発現のための給餌液(FS)の補給のための技術を提供します。さらに、生産バッチを最適化するためのプロトコルが含まれています。リーシュマニア無細胞システムの初期バージョンでは、発現レベル、完全長タンパク質の画分、およびタンパク質凝集体の存在に望ましくないバッチ間の変動が観察され、バッチ34の廃棄につながりました。その後、この問題に対処するためにプロトコルの改善が行われました25。現在のプロトコールは、これらの改善に基づいて構築されており、個々のバッチをピークタンパク質の発現とサイズに最適化することができます。これは、細胞かく乱物質の負荷を厳密に制御することによって達成されます(600 nmの光学密度として測定されます。OD600nm)で溶解し、得られたライセート出力を280 nm(Abs280 nm)の吸光度を使用して正規化します。さらに、製造時にライセートにrNTPとマグネシウムを部分的に補給する方法を組み込んでおり、その後、試験発現中にこれらの供給溶液成分を最適化します。この最適化はプロトコルのオプションとして提示されていますが、著者によって強く推奨されています。
プロトコル
このプロトコールには、培養、遠心分離、マルチモードプレートリーダーを使用したGFP蛍光の測定、培養OD600nmの測定、ライセートAbs280nmの評価を含む詳細な培地レシピとステップが含まれています。また、SDS-PAGEタンパク質ゲルのセットアップとイメージングについても説明します。このプロトコールに必要な材料または推奨される材料は、材料スプレッドシートに記載されています。メディアコンポーネント、遠心分離機、チューブ、分光光度計、ゲル電気泳動セットアップなどの一般的なラボリソースは、特に指定がない限り、同じ意味で使用できる可能性が高いことに注意することが重要です。 図 1 に、LTE 製造プロセスの概要を示します。
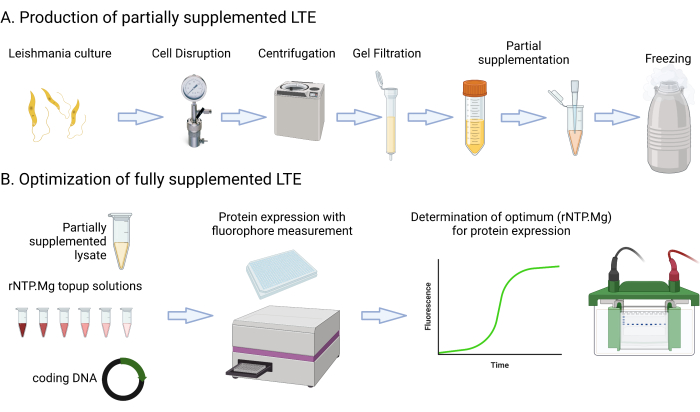
図1:LTE製造プロトコルの概要。 この漫画は、LTE 製造プロトコルの簡潔な要約を提供します。 この図の拡大版を表示するには、ここをクリックしてください。
1. リーシュマニア・タレントラエの培養物の成長
- 少なくとも3 LのTBGG増殖培地を調製します(バクトトリプトン12 g / L、酵母抽出物24 g / L、グリセロール8 mL / L、グルコース1 g / L、KH2PO4 2.3 g / L、K2HPO4 2.5 g / L、 材料表を参照)。0.22 μmフィルターを使用して、真空または同様のセットアップで培地を滅菌します。
- 培地を室温(RT)で保存し、L. tarentolaeを接種する直前に最終添加物(ヘミン、抗生物質)を添加します。ヘミン(50% トリエタノールアミン中 0.25% v/v)を 0.2% v/v で添加し、ペニシリン(10,000 units/mL)とストレプトマイシン(10,000 μg/mL)を 0.5% v/v で混合します。
注:このプロトコルの開始点は、野生型 L. tarentolaeの2 x 10 mL培養物を維持することです。維持培養物は、標準的な50 mL組織培養フラスコで27°Cで低振とう(75 rpm)で増殖します。このような10mLの培養物は、2〜3日ごとに無菌(TBGG+ヘミン、ペニシリン、ストレプトマイシン)で~1/20希釈して無期限に維持することができます。PC2ラボの標準的なバイオセーフティキャビネットをお勧めします。ただし、細菌汚染は追加された抗生物質によって防止される傾向がありますが、真菌汚染は一般に L.tarentolaeによって成長します。 - 2日間かけて、 L. tarentolae 維持培養物を200 mL(1日目)に拡大し、次に2 L(2日目)に1:10希釈 して 、TBGG +ヘミン/抗生物質の量を毎日増やします。オートクレーブ滅菌したバッフル5Lガラスフラスコ(最大1Lまで充填)で両方の希釈を行います。2回目の希釈は、翌日の午前8時から11時の間にライセートの生産を開始することを意図して、午後3時から6時の午後の間に行う必要があります。
注: このプロトコルでは、LTE プロダクションの最小開始ボリューム (2 x 1 L カルチャ) を使用します。また、追加の拡張ステップを組み込むことにより、LTE生産用の培養液を10 Lまで拡張することも可能である(例:Day 1:100 mL;2日目:1リットル;3日目:10 L)。このプロトコルでは、バッフルフラスコ( 材料の表を参照)を使用して L. tarentolaeを増殖させますが、攪拌速度が100 rpm未満に保たれていれば、オプションで、Rushtonインペラを使用して細菌増殖用に設計された従来のバイオリアクターを使用できます。バイオリアクターにおける曝気とpH制御の改善は、一般に L. tarentolae 培養物の対数期成長を延長し、ステップ1.4でより高い収穫OD600nm の10を使用できるようにします。 - TBGGで1:10希釈することにより、培養物のOD600nmを分光光度計キュベットに直接記録します。ライセートの作製に適した開始範囲は、OD600nm = 4.0-8.0です。
- OD600nmが4.0の場合は、追加のインキュベーション時間<してください。OD600nm > 8の培養は使用可能であり、無細胞発現ライセートの量が多くなりますが、遅延ログ増殖期の開始により品質が低下します。培養フラスコを氷の上に置き、次のステップを待ちます。
注:最終培養OD600nmの正確な測定は、分裂前の濃縮細胞の最終容量を計算するために使用されるため、非常に重要です。この計算は、プロトコルを簡素化するために、LTE製造の以前のバージョンで使用されていたペレット計量方法に取って代わるもので、ディスラプション34の前に細胞濃度を較正する。OD600nmの測定にはTBGGで希釈してください、そうしないと浸透圧ショックによって細胞の形状が変化し、測定誤差が発生します。L. tarentolae細胞が独特の曇った外観で急速に沈降するため、分光光度の読み取りを行う数秒前に、OD600nm測定用の1:10希釈液を(キュベットで直接)ピペットで混合します。発現培養物の最終容量が概算であると考えられる場合、収穫時にフラスコを計量し(適切な空のフラスコ風袋を使用)、より良い推定容量(1 g = 1 mL)を得ることも推奨されます。バッフルフラスコでのL. tarentolaeの成長から可能な最大外径600nmは15〜20ですが、これは固定相に達するため、ライセート製造には不適切です。
2. L. tarentolae の培養物の集中
- リーシュマニア細胞は、破壊する前に約60倍洗浄し、濃縮する必要があります。最終濃縮液のOD600nm = 300に基づいて細胞濃度の目標体積を計算します。式は、V = 収穫量 (mL) x (収穫 OD600nm/300) です。例えば、収穫量がOD 600 = 5 の 2 L 培養物を使用すると、目標容量は 33 mL になります。
注:300のOD600nm ターゲットは変更できます。以前の LTE 生産では、150 から 350 の範囲の値が使用されていました。細胞の濃度が高いほど、最終的な無細胞発現反応が起こり、タンパク質の収量は高くなりますが、脆弱なタンパク質が凝集する傾向が強くなります。OD600nm = 300 は、LTE 生産に適したデフォルトのターゲットを表します。 - 回収した培養物を適切な遠心分離ボトルに移し、2500 x g で4°Cで10分間遠心します。 上清を培養廃棄物に慎重にデカントします。
注:廃棄された上清への細胞の損失を最小限に抑えることが重要です。これは、破壊負荷の計算に影響を与えるためです。以前のLTE生産プロトコルでは、破壊のためのL. tarentolae細胞の濃度は、試験微量遠心チューブ内で細胞濃縮物をスピンダウンし、ペレット重量対総重量34を測定することによって較正された。この簡略化されたプロトコルは、代わりに、測定された収穫OD600nmに基づいて、濃縮物の理論的なOD600nmターゲットを使用し、細胞濃縮および洗浄中の細胞損失が少ないことを前提としています。 - 細胞ペレットをSEBバッファー(45 mM HEPES-KOH pH 7.6、250 mMスクロース、100 mM KOAc、3 mM Mg(OAc)2、氷上に保持)で3回洗浄し、毎回2500 x g で4°Cで10分間遠心分離します。 最初の洗浄では、ペレット化した培養物1 Lを100 mLのSEBバッファーに再懸濁し、1つの遠心フラスコに混ぜ合わせます。2回目の洗浄では、元の培養物1 Lごとに100 mLのSEBも使用します。
注:最終的なペレット再懸濁液については、SEBバッファーを最終目標再懸濁量の50%に添加します(ステップ2.1)。これにより、プールされた濃縮物をステップ2.4の最終目標量まで慎重に補充することができます。各再懸濁液は、 L. tarentolaeの早期溶解を避けるために、例えば、デカントされたペレットの周りで添加されたSEBを穏やかに渦巻かせたり、遠心分離管壁に付着したペレットにSEBをピペッティングしたりして、できるだけ穏やかに行う必要があります。最終ステップでは、上清を小さな遠心分離管に移す方が便利な場合があります。 - 再懸濁した濃縮物を適切な洗浄ガラス容量シリンダーに注ぎ、次に追加の冷たいSEBを使用して容量を目標容量まで補充し(ステップ2.1)、穏やかに混合します。
3. L. tarentolae濃縮物の溶解
- 細胞濃縮物を4°Cに予冷した窒素キャビテーション装置( 材料表を参照)に移し、窒素を70バールまで加圧し、氷上で45分間インキュベートします。
注:窒素キャビテーション撹乱器は一般的な実験室用品ではありませんが、LTE生産には推奨されます。細胞凍結融解やフレンチプレスタイプの撹乱器などの代替方法が試みられています。しかし、タンパク質の発現活性は窒素キャビテーション法と比較して<50%でした。窒素キャビテーション装置は、このステップ以降に細胞溶解物と接触するすべての再利用容器(レシーバーフラスコなど)と同様に、使用前および実行間で徹底的に洗浄する必要があります。適切な洗浄法には、実験用洗剤で洗浄した後、脱イオン水で徹底的にすすぐことが含まれます。 - 窒素キャビテーション装置のベントを開き、得られたライセートを氷上の真空レシーバーフラスコなどの適切に堅牢な容器に排出します。レシーバーフラスコを傾けて、得られたすべてのライセートが沈殿し、新しい遠心分離チューブまたは同様の容器にピペットで移せるようにします。
注意:窒素キャビテーションディスラプターは、70バールの窒素から周囲圧力への細胞濃縮物の突然の移行に依存しており、これは最初に液体が強く流れ、次にデバイスの出口バルブを通る窒素の強い流れによって達成されます。通気は、化学安全フード内の適切な個人用保護具(PPE)を使用して行う必要があります。目的地の容器が壊れてライセートが失われるリスクがあるため、一般的なフラスコではなく頑丈な真空レシーバーを使用しています。デバイスの出口バルブがチューブの場合は、ベントポイントでの過度の圧力の蓄積を防ぐために、チューブをレシーバー内に直接置かないでください。
4. 細胞溶解物の遠心分離
- ライセートを適切な g 力定格遠心チューブに移し、10,000 x g で4°Cで15分間遠心分離します。 上清を新鮮で類似の遠心分離チューブに移します。
- ライセートを30,000 x g で4°Cで15分間遠心分離し、最終的な上清を新しい遠心チューブまたは氷上に置いた同様の容器に移します。合計ボリュームを見積もります。
5. 細胞溶解物のゲルろ過
注:ゲルろ過は、SEBバッファーに含まれるスクロースを除去するために使用されます。スクロースは、細胞破壊中に細胞機構を安定化するのに役立ちますが、タンパク質発現反応で保持されると収量が減少します。
- 十分な数の PD-10 重力供給ゲルろ過カラム( 材料表を参照)をラック形式で設置し、その下の収集トレイまたは同様の容器に滴下に滴静注できるようにして、カラムあたり 2.5 mL で全ライセート量をろ過できるようにします。10 mLの4 °C EBバッファー(45 mM HEPES-KOH pH 7.6、100 mM KOAc、3 mM Mg(OAc)2)を事前に通過させて、カラムを事前に平衡化します。
注:この時点からのすべてのステップは、4°Cの冷蔵室で実施することでメリットが得られます。ただし、すべてのライセートと試薬をベンチトップの製氷皿に保管するのも適しています。例外の1つはゲルろ過ステップで、著者らは4°Cの冷蔵庫内にカラムのラックを置きながら再バッファリングします。このプロトコルの元のバージョンでは、新しいゲルろ過カラムは、最初にライセートをバッファーし、最初の出力を廃棄することで「ブロック」されていました。これは必須ではないと考えられていますが、カラムはEBバッファーでフラッシュし、ライセートバッチ間で4°Cで保存する必要があります。最初に出力されたライセートは、新しいカラム上のライセート成分のバックグラウンド保持により、その後の出力よりもタンパク質発現活性が低くなることがあります。 - 各カラムに2.5 mLのライセートを加え、カラムに入るまで待ちます。さらに 0.5 mL の EB を添加して、溶出液を廃棄しながらライセートをカラムに沈殿させます。
- ゲルろ過したライセートを溶出するには、各カラムにさらに 2.5 mL の EB を添加し、新鮮で清潔なトレイまたは別のレセプタクルをカラムの下に置いて出力を収集します。
6. 細胞ライセートの補給
- ナノドロップ分光光度計( 材料の表を参照)を使用して、ゲルろ過されたライセートのAbs280nm を測定します。60を超える場合は、追加の4°CEBバッファーを使用してAbs280nm = 60になるように希釈します。
注:OD600nm を使用して破壊への細胞密度入力を制御すると、ライセートの生産量がおよそ決定されますが、ライセートの破壊と処理後にAbs280nm を正規化すると、ライセートのバッチ一貫性がさらに向上します。ライセートAbs280nm は上下に調整でき、タンパク質の発現収率と凝集に影響を及ぼします(「ディスカッション」のセクションを参照)。未添加のライセートがAbs280nm <60を示している場合、ディスラクションステップにより多くのリーシュマニアバイオマスを含める必要があるかもしれません、すなわち、ステップ2.1で細胞かく乱物質の負荷量をOD600nm >300に増やす必要があります。 - 5x Feeding Solution(5x FS、 表1)をライセートに2:5の比率で加え、十分にボルテックスミックスします。適切な容器(1.5 mLマイクロチューブなど)に分注し、液体窒素で急速冷凍します。以下の LTE 式の最適化に関するオプションの手順 7.1 から 7.3 に従っている場合は、デフォルトの 5x FS ではなく、 表 1 の縮小 rNTP.Mg された 5x FS を使用します。最適化実験で使用するための凍結用の5 x 100 μLアリコートを含めます。
注:デフォルトの 5x FS を 2:5 の比率で凍結すると、7 μL/10 μL の発現で使用される、発現可能な補充 LTE が作成されます (したがって、5x FS は最終反応で 1x FS になります)。ただし、著者らは、デフォルトの0.6倍のrNTPとマグネシウムの量が5x FSで提供されている場合、さらにオプションの手順に従うことを推奨しています。これに続いて、2つの等モル混合物(rNTP.Mg と呼ばれる)を添加して、最適化された値への試験反応を補充する最適化ステップが続きます。パーシャル5x FSには、内因性mRNA発現をシャットダウンするオリゴヌクレオチドも含まれています(「はじめに」セクションを参照)。オリゴヌクレオチドの配列はCAATAAAGTACAGAAACTGATACTTATATAGCGTTです。
7. 最終追加LTEのQCと最適化
注:還元されたrNTPおよびマグネシウム添加ライセートへの rNTP.Mg の適切な「追加」追加を決定するために必要な最小限のステップには、融合パートナーなしでeGFPまたは同様の蛍光色素(sfGFPなど)を発現することが含まれます。反応に増大する濃度の rNTP.Mg を添加して、発現レベル(マルチモードプレートリーダー による eGFP RFUとして測定)が最適化されるポイントを決定します。蛍光性ではないeGFPの早期終結は、高すぎる rNTP.Mg 濃度でeGFP RFUを減少させることで明らかになります。しかし、LTEの短産物の誤動作は、大きく発現したタンパク質(>50 kDa)でより頻繁に発生します。したがって、eGFPよりも大きなテンプレートを使用してこの最適化を実行することが可能であり、特に適切な発現ベクターで利用可能なテンプレートがある場合、特定のアプリケーションまたは研究のためにLTEによって生成されることが望ましい蛍光色素融合を提供します(代表的な結果のセクションを参照)。
- 100 μLのアリコートを解凍し、ステップ6.2で部分的に添加したライセート7 μL、 表2の補充溶液1 μL、および反応液の最終濃度50 ng/μLを達成するのに十分なDNA制御テンプレートを含む超純水2 μLからなる6つの10 μL発現反応を設定します。
- 反応液を25°Cで2時間インキュベートし、マルチモードプレートリーダーを使用してGFP蛍光の増加をモニターします。
注:GFPの適切な設定値は、485 nm(帯域幅5 nm)での励起、516 nm(帯域幅5 nm)での発光、および読み取り間隔1分、2時間です。 - 最終的な発現値をランク付けして、最高のeGFP RFUに対応する rNTP.Mg 濃度を決定します。動態データが利用可能な場合、2時間の発現期間中のeGFP RFUの二相性増加によって、過剰な rNTP.Mg も示されます(代表的な結果のセクションを参照)。
- 最適化されたトップアップ rNTP.Mg 濃度が決定されたら、前のステップで部分的に補充したLTEのバッチを使用して、それをすべてのさらなるタンパク質発現に加えます。
注:ステップ6.2の分注が固定容量で慎重に行われる場合、補充は解凍せずに各分注に遡及的に追加できます(たとえば、分注をドライアイスの上に置くなど)。これらのアリコートは、解凍して使用するために混合すると、それぞれに正しい rNTP.Mg 補充が混合されるため、現在では完全に補充されています。
結果
無細胞タンパク質発現の目的は、幅広いアプリケーションに適した折り畳まれた活性型で完全長タンパク質を作製することです。LTE(Leishmania tarentolae extract)は、以前に他の原核生物および真核生物の無細胞発現系と比較されており、特に 大腸菌ベースの無細胞発現と比較して、最適に動作する際に切断および凝集を回避する高い能力を示しています
ディスカッション
LTEを作成するためのプロトコルは、過去10年間で公開されており7、定期的な更新を受けています25,34。しかし、この技術を初めて使用すると、学習曲線が急になることが多く、その結果、高品質で高収率のタンパク質発現を達成するのが遅れます。LTE35を扱う他の研究グループでも同様の...
開示事項
競合する金銭的利益は存在しません。
謝辞
著者らは、過去10年間にLTEシステムの開発に貢献した多くのAlexandrov研究室のメンバー、特にシステムの先駆者であり、SITSリボソームエントリーサイトを開発したSergey Mureevに感謝したいと思います。 図1 は Biorender.com によって作成され、ライセンスに基づいて複製されました。
資料
| Name | Company | Catalog Number | Comments |
| PD-10 SuperDex 25 Columns | Cytiva | 17085101 | Gel filtration columns |
| Nitrogen Cavitation cell disrupter | Parr Industries | 4635 or 4639 | Cell Disrupter |
| Bovine derived Hemin | Sigma-Aldrich | H5533 | Culture additive |
| Penicillin/Streptomycin 10000U/ml | Thermo-Fisher | 15140122 | Antibiotic mix |
| Optiplate 384 | Perkin-Elmer | 6007290 | Multiwell plate for 10ul expressions |
| Oligonucleotide | IDT synthesis | Oligo with sequence CAATAAAGTACAGAAACTGATAC TTATATAGCGTT | |
| Creatine Phosphokinase | Sigma-Aldrich | 9001-15-4 | Enzyme |
| Tecan Spark | Tecan | or similar Multimode Platereader | |
| Chemidoc MP Imager | Biorad | or similar SDS-PAGE gel Imager | |
| 4-12% Bis-Tris Gels | Invitrogen | NW04125 | SDS-PAGE gels |
| Biophotometer | Eppendorf | or similar Cuvette Specrophotometer | |
| Nanodrop One | Thermofisher | Nanodrop spectrophotometer | |
| Avanti JXN-26 centrifuge | Beckman Coulter | or similar centrifuge, with rotors/tubes rated 10K and 50K g | |
| 5424R microcentrifuge | Eppendorf | or similar microcentrifuge, with 1.5ml microcentrifuge tubes | |
| Flask Incubator Inova S44i | Eppendorf | or similar flask incubator shaker suitable for 5L Flasks | |
| 5L glass culture flasks | Baffled glass flasks for culture growth | ||
| Bactotryptone | BD | 211705 | Growth medium |
| Yeast Extract | Merck | VM930053 | Growth medium |
| Glycerol | Any analytical grade | ||
| Glucose | Any analytical grade | ||
| KH2PO4 | Any analytical grade | ||
| K2HPO4 | Any analytical grade | ||
| UltraPure water | Invitrogen | 10977-015 | Or output from any MilliQ-type water dispenser |
参考文献
- Nirenberg, M. W., Matthaei, J. H. The dependence of cell-free protein synthesis in E.coli upon naturally occurring or synthetic polyribonucleotides. Proc Natl Acad Sci USA. 47 (10), 1588-1602 (1961).
- Caschera, F., Noireaux, V. Synthesis of 2.3 mg/ml of protein with an all Escherichia coli cell-free transcription-translation system. Biochimie. 99, 162-168 (2014).
- Kelwick, R., Webb, A. J., MacDonald, J. T., Freemont, P. S. Development of a Bacillus subtilis cell-free transcription-translation system for prototyping regulatory elements. Metab Eng. 38, 370-381 (2016).
- Ezure, T., et al. Cell-free protein synthesis system prepared from insect cells by freeze-thawing. Biotechnol Prog. 22 (6), 1570-1577 (2006).
- Harbers, M. Wheat germ systems for cell-free protein expression. FEBS Letters. 588 (17), 2762-2773 (2014).
- Kobs, G. Selecting the cell-free protein expression system that meets your experimental goals. Promega Corporation. 21, 6-9 (2008).
- Kovtun, O., et al. Leishmania cell-free protein expression system. Methods. 55 (1), 58-64 (2011).
- Basile, G., Peticca, M. Recombinant protein expression in Leishmania tarentolae. Mol Biotechnol. 43 (3), 273-278 (2009).
- Mureev, S., Kovtun, O., Nguyen, U. T., Alexandrov, K. Species-independent translational leaders facilitate cell-free expression. Nat Biotechnol. 27 (8), 747-752 (2009).
- Gambin, Y., et al. Single-molecule fluorescence reveals the oligomerization and folding steps driving the prion-like behavior of ASC. J Mol Biol. 430 (4), 491-508 (2018).
- Sierecki, E., et al. Rapid mapping of interactions between human SNX-BAR proteins measured in vitro by AlphaScreen and single-molecule spectroscopy. Mol Cell Proteomics. 13 (9), 2233-2245 (2014).
- Sierecki, E., et al. Nanomolar oligomerization and selective co-aggregation of alpha-synuclein pathogenic mutants revealed by single-molecule fluorescence. Sci Rep. 6, 37630 (2016).
- Leitao, A., Bhumkar, A., Hunter, D. J. B., Gambin, Y., Sierecki, E. Unveiling a selective mechanism for the inhibition of alpha-synuclein aggregation by beta-synuclein. Int J Mol Sci. 19 (2), 334 (2018).
- Gambin, Y., et al. Single-molecule analysis reveals self assembly and nanoscale segregation of two distinct cavin subcomplexes on caveolae. Elife. 3, e01434 (2013).
- Ve, T., et al. Structural basis of TIR-domain-assembly formation in MAL- and MyD88-dependent TLR4 signaling. Nat Struct Mol Biol. 24 (9), 743-751 (2017).
- Guo, Z., et al. Subunit organisation of in vitro reconstituted HOPS and CORVET multisubunit membrane tethering complexes. PLoS One. 8 (12), e81534 (2013).
- Ruehrer, S., Michel, H. Exploiting Leishmania tarentolae cell-free extracts for the synthesis of human solute carriers. Mol Membr Biol. 30 (4), 288-302 (2013).
- Varasteh Moradi, S., et al. Mapping Interactions among cell-free expressed Zika virus proteins. J Proteome Res. 19 (4), 1522-1532 (2020).
- Gagoski, D., et al. Cell-free pipeline for discovery of thermotolerant xylanases and endo-1,4-beta-glucanases. J Biotechnol. 259, 191-198 (2017).
- Ergun Ayva, C., et al. Exploring performance parameters of artificial allosteric protein switches. J Mol Biol. 434 (17), 167678 (2022).
- Lau, D., et al. Fluorescence biosensor for real-time interaction dynamics of host proteins with HIV-1 capsid tubes. ACS Appl Mater Interfaces. 11 (38), 34586-34594 (2019).
- Ryan, S. M., et al. Novel antiinflammatory biologics shaped by parasite-host coevolution. Proc Natl Acad Sci USA. 119 (36), e2202795119 (2022).
- McMahon, K. A., et al. Identification of intracellular cavin target proteins reveals cavin-PP1alpha interactions regulate apoptosis. Nat Commun. 10 (1), 3279 (2019).
- Sierecki, E., et al. A cell-free approach to accelerate the study of protein-protein interactions in vitro. Interface Focus. 3 (5), 20230018 (2013).
- Johnston, W. A., Moradi, S. V., Alexandrov, K. Adaption of the Leishmania cell-free expression system to high-throughput analysis of protein interactions. Methods Mol Biol. 2025, 403-421 (2019).
- Jung, W., et al. Cell-free formation and interactome analysis of caveolae. J Cell Biol. 217 (6), 2141-2165 (2018).
- Fontaine, F. R., et al. Functional domain analysis of SOX18 transcription factor using a single-chain variable fragment-based approach. MAbs. 10 (4), 596-606 (2018).
- Overman, J., et al. Pharmacological targeting of the transcription factor SOX18 delays breast cancer in mice. Elife. 6, e21221 (2017).
- Kubala, M. H., et al. Mammalian farnesyltransferase alpha subunit regulates vacuolar protein sorting-associated protein 4A (Vps4A)--dependent intracellular trafficking through recycling endosomes. Biochem Biophys Res Commun. 468 (4), 580-586 (2015).
- Han, S. P., et al. Cortactin scaffolds Arp2/3 and WAVE2 at the epithelial zonula adherens. J Biol Chem. 289 (11), 7764-7775 (2014).
- Das Gupta, K., et al. Class IIa histone deacetylases drive toll-like receptor-inducible glycolysis and macrophage inflammatory responses via pyruvate kinase M2. Cell Rep. 30 (8), 2712-2728.e8 (2020).
- Leitão, A. D. G., et al. Selectivity of protein interactions along the aggregation pathway of α-synuclein. BioRxiv. , (2021).
- Gagoski, D., et al. Performance benchmarking of four cell-free protein expression systems. Biotechnol Bioeng. 113 (2), 292-300 (2016).
- Johnston, W. A., Alexandrov, K. Production of eukaryotic cell-free lysate from Leishmania tarentolae. Methods Mol Biol. 1118, 1-15 (2014).
- Hunter, D. J. B., Bhumkar, A., Giles, N., Sierecki, E., Gambin, Y. Unexpected instabilities explain batch-to-batch variability in cell-free protein expression systems. Biotechnol Bioeng. 115 (8), 1904-1914 (2018).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved