JoVE 비디오를 활용하시려면 도서관을 통한 기관 구독이 필요합니다. 전체 비디오를 보시려면 로그인하거나 무료 트라이얼을 시작하세요.
Method Article
재조합 단백질 생산을 위한 Leishmania tarentolae 유래 Cell-Free 단백질 발현 시스템인 LTE의 생산 및 최적화
요약
Leishmania Translational Extract(LTE)는 단세포 기생충인 Leishmania tarentolae에서 유래한 진핵 세포 없는 단백질 발현 시스템입니다. 이 최적화된 프로토콜은 LTE를 간단하고 비용 효율적으로 제조할 수 있도록 합니다. 복잡한 진핵생물 단백질의 다병렬 발현 및 연구와 그 상호 작용에 초점을 맞춘 다양한 응용 분야에 적합합니다.
초록
이 프로토콜은 Leishmania Translational Extract 또는 LTE라고 하는 단세포 편모 Leishmania tarentolae에서 유래한 진핵생물 Cell-Free Protein Expression System(CFPS)의 생산 및 최적화를 간략하게 설명합니다. 이 유기체는 원래 도마뱀붙이의 기생충으로 진화했지만 플라스크나 생물 반응기에서 쉽고 저렴하게 배양할 수 있습니다. Leishmania major와 달리 인간에게 비병원성이며 특별한 실험실 예방 조치가 필요하지 않습니다. CFPS에 Leishmania를 사용하는 또 다른 이점은 모든 단백질 코딩 RNA의 5'-말단에서 보존된 스플라이스 리더 서열을 표적으로 하는 CFPS에 단일 안티센스 올리고뉴클레오티드를 추가하면 내인성 단백질 발현을 억제할 수 있다는 것입니다. 당사는 세포 파괴 및 용해물 처리를 위한 절차를 제공하며, 이는 이전 버전에 비해 단순화되고 개선되었습니다. 이러한 절차는 간단한 플라스크 배양으로 시작합니다. 또한 종 독립적 번역 시작 사이트(SITS)를 포함하는 벡터를 사용하여 유전 정보를 도입하는 방법과 일관된 단백질 발현 품질을 보장하기 위해 간단한 배치 최적화 및 품질 관리를 수행하는 방법을 설명합니다.
서문
1960년대에는 cell-free 단백질 발현 시스템이 유전 코드1을 밝히는 데 중추적인 역할을 했습니다. 그러나 주로 대장균을 기반으로 하는 원핵 세포 무료 단백질 발현 시스템은 현재 실험실 및 상업 응용 분야를 지배하고 있습니다. 대장균 기반 시스템은 비용 효율성, 확장성 및 높은 발현 수율과 같은 이점을 제공하지만, 활성 형태의 다중 영역 단백질을 생산하고 단백질 복합체 2,3의 조립을 촉진할 때 문제에 직면합니다. 오늘날 일반적으로 사용되는 진핵생물 무세포 단백질 합성(CFPS) 형태에는 밀 배아 추출물(WGE), 토끼 망상적혈구 용해물(RRL) 및 곤충 세포 용해물(ICL)이 포함됩니다.4,5,6. 이 연구는 단세포 편모충 기생충 Leishmania tarentolae를 기반으로 하는 간단하고 확장 가능한 대체 진핵 세포 없는 시스템을 소개합니다.
Leishmania tarentolae 는 비용 효율적인 배지를 사용하여 플라스크에서 쉽게 배양할 수 있으며 더 높은 세포 밀도를 달성하기 위해 생물 반응기에서 확장할 수도 있습니다. 세포 용해물에서 내인성 mRNA의 존재는 그렇지 않으면 도입된 메시지와 경쟁할 수 있으며, 보존된 Leishmania mRNA 스플라이스 리더 서열7을 표적으로 하는 안티센스 올리고뉴클레오티드를 사용하여 중화할 수 있습니다. 인간의 질병을 일으키는 가까운 친척 Leishmania major와 달리 L. tarentolae 는 무어 도마뱀붙이 (Tarentolae mauritanica)를 감염시켜 특별한 예방 조치없이 PC2 실험실 환경에서 배양하기에 적합합니다. 이전에는 in vivo 단백질 발현을 위한 형질전환 유기체로 사용되었다8.
cell-free 시스템에서 template priming을 용이하게 하기 위해, universal sequences는 translational initiation을 향상시키는 polymeric RNA structure를 기반으로 설계되었습니다9. 이러한 종 독립적 번역 서열(SITS)은 원핵 및 진핵 cell-free 시스템 모두에 적용할 수 있으며 유전 정보를 LTE에 도입하는 데 적합합니다. 이 프로토콜은 LTE cell-free 단백질 발현을 위한 벡터 구성에 대한 자세한 설명을 제공하지 않지만, 최적화 및 품질 관리를 위해서는 SITS site의 다운스트림에서 원하는 관심 단백질의 형광단 융합을 포함하는 적절한 벡터가 필요합니다. 이를 위해 적절한 LTE 벡터가 pCellFree_G03 벡터와 같은 Addgene 유전자 저장소에 기탁되었으며, 이 벡터는 Gateway 클로닝 사이트를 사용하여 N-말단 eGFP 융합을 원하는 단백질로 인코딩합니다.
LTE는 단백질 자기 조립(10,16) 분석, 인간 통합 막 단백질(human integral membrane proteins)17 생성, 항바이러스 약물 후보 물질 연구(18), 생명공학적으로 유용한 효소(19) 개발, 단백질 바이오센서 프로토타이핑(20,21) 및 십이지장충(22)의 생물학적 제제 연구를 포함하여 단백질 발현이 필요한 광범위한 응용 분야에서 그 가치를 입증했습니다. LTE는 또한 바이러스학 및 세포 구조 분야에서 단백질-단백질 상호 작용 네트워크를 매핑하는 데 중요한 역할을 했습니다21,32. LTE는 전장, 단분산 및 비응집된 단백질33을 발현하는 동시에 보다 비용 효율적이고 확장 가능한 생산을 제공하는 동시에 다른 진핵 세포 없는 시스템과 유사한 성능을 발휘하는 것으로 벤치마킹되었습니다.
이 프로토콜은 숙주 유기체를 배양 및 파괴하고, 용해물을 준비하고, 결합 전사/번역 단백질 발현을 위한 공급 용액(FS)을 보충하는 기술을 제공합니다. 또한 생산 배치를 최적화하기 위한 프로토콜이 포함되어 있습니다. 리슈마니아 cell-free 시스템의 초기 버전에서는 발현 수준, 전장 단백질의 분율 및 단백질 응집체의 존재에서 원치 않는 배치 간 변이가 관찰되어 배치34의 폐기로 이어졌습니다. 이 문제를 해결하기 위해 후속 프로토콜 개선이 이루어졌습니다25. 현재 프로토콜은 이러한 개선 사항을 기반으로 하여 개별 배치를 최대 단백질 발현 및 크기에 맞게 최적화할 수 있습니다. 이는 cell-disrupter loading(600nm에서 optical density로 측정됨)을 면밀히 제어함으로써 이를 달성합니다. OD600nm) 및 280nm(Abs280nm)에서 흡광도를 사용하여 생성된 용해물 출력을 정규화합니다. 또한, 제조 과정에서 rNTP 및 마그네슘으로 용해물을 부분적으로 보충하는 방법을 통합하고, 테스트 발현 중에 이러한 공급 용액 구성 요소를 후속 최적화합니다. 이 최적화는 프로토콜에서 옵션으로 제공되지만 작성자가 강력히 권장합니다.
프로토콜
이 프로토콜에는 배양, 원심분리, 멀티모드 플레이트리더를 사용한 GFP 형광 측정, 배양 OD600nm 측정 및 용해물 Abs280nm 평가와 관련된 자세한 배지 레시피 및 단계가 포함되어 있습니다. 또한 SDS-PAGE 단백질 겔의 설정 및 이미징도 다룹니다. 이 프로토콜에 필요하거나 제안된 재료는 재료 스프레드시트에 나열되어 있습니다. 매체 구성 요소, 원심분리기, 튜브, 분광 광도계 및 겔 전기영동 설정과 같은 일반적인 실험실 리소스는 달리 명시되지 않는 한 상호 교환하여 사용할 수 있다는 점에 유의하는 것이 중요합니다. 그림 1 은 LTE 제조 공정을 요약한 것입니다.
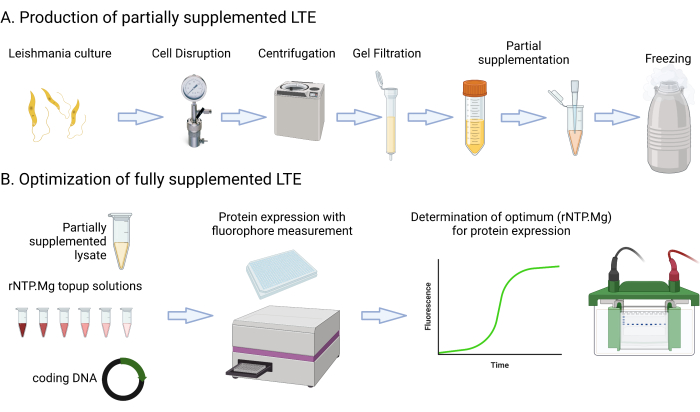
그림 1: LTE 제조 프로토콜 개요. 이 만화는 LTE 제조 프로토콜에 대한 간결한 요약을 제공합니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
1. 리슈마니아 타렌톨라에 문화의 성장
- 최소 3L의 TBGG 성장 배지(박토트립톤 12g/L, 효모 추출물 24g/L, 글리세롤 8mL/L, 포도당 1g/L, KH2PO4 2.3g/L, K2HPO4 2.5g/L, 재료 표 참조)를 준비합니다. 진공 상태에서 0.22μm 필터 또는 유사한 설정을 사용하여 매체를 멸균합니다.
- 배지를 실온(RT)에서 보관하고 L. tarentolae를 접종하기 직전에 최종 첨가물(Hemin, Antibiotics)을 첨가합니다. 헤민(50% 트리에탄올아민에서 0.25% v/v)을 0.2% v/v, 페니실린(10,000단위/mL)과 스트렙토마이신(10,000μg/mL) 혼합물을 0.5% v/v로 첨가합니다.
참고: 이 프로토콜의 시작점은 야생형 L. tarentolae의 유지된 2 x 10mL 배양입니다. 유지 배양액은 표준 50mL 조직 배양 플라스크에서 27°C에서 낮은 흔들림(75rpm)으로 성장합니다. 이러한 10mL 배양액은 2-3일마다 멸균(TBGG + 헤민, 페니실린, 스트렙토마이신)에서 ~1/20 희석으로 무기한 유지할 수 있습니다. PC2 실험실의 표준 생물 안전 작업대가 권장됩니다. 그러나 박테리아 오염은 항생제를 첨가하면 예방되는 경향이 있는 반면 곰팡이 오염은 일반적으로 L. tarentolae에 의해 증식되지 않습니다. - 2일에 걸쳐 L. tarentolae 유지 배양균을 200mL(1일)로 확장한 다음 매일 TBGG + 헤민/항생제의 양을 늘리고 1:10 희석을 통해 2L(2일)로 확장합니다. 오토클레이브 멸균된 배플 5L 유리 플라스크(최대 1L까지 채워짐)에서 두 희석을 모두 수행합니다. 두 번째 희석은 오후 3-6시 사이에 발생해야 하며, 다음날 오전 8-11시 사이에 용해물 생산을 시작하려는 의도로 이루어져야 합니다.
참고: 이 프로토콜은 LTE 생산을 위한 최소 시작 볼륨(2 x 1 L culture)을 사용합니다. 또한 추가 확장 단계를 통합하여 LTE 생산을 위해 배양액을 최대 10L까지 확장할 수 있습니다(예: Day 1: 100 mL; 2일차: 1리터; 3일차: 10 L). 이 프로토콜은 L. tarentolae를 성장시키기 위해 배플 플라스크(재료 표 참조)를 사용하지만, 교반 속도가 100rpm 미만으로 유지되는 경우 Rushton 임펠러를 사용하여 박테리아 성장을 위해 설계된 기존 바이오리액터를 선택적으로 사용할 수 있습니다. 바이오리액터에서 개선된 폭기 및 pH 제어는 일반적으로 L. tarentolae 배양의 로그 단계 성장을 확장하여 1.4단계에서 더 높은 수확 OD600nm of 10을 사용할 수 있도록 합니다. - TBGG에서 1:10 희석을 통해 배양의 OD600nm를 분광광도계 큐벳에서 직접 삼중으로 기록합니다. 용해물을 만들기에 적합한 시작 범위는 OD600nm = 4.0-8.0입니다.
- OD600nm < 4.0인 경우 추가 배양 시간을 제공합니다. OD600nm > 8을 사용한 배양은 사용할 수 있으며 더 많은 양의 cell-free expression lysate를 얻을 수 있지만 후기 로그 성장 단계가 시작되기 때문에 품질이 떨어집니다. 배양 플라스크를 얼음 위에 놓고 다음 단계를 기다립니다.
참고: 최종 배양 OD600nm의 정확한 측정은 파쇄 전에 농축된 세포의 최종 부피를 계산하는 데 사용되므로 매우 중요합니다. 이 계산은 프로토콜을 단순화하기 위해 중단(34) 이전의 세포 농도를 교정하기 위해 LTE 제조의 이전 버전에서 사용된 펠릿 칭량 방법을 대체합니다. OD600nm 측정을 위해 TBGG로 희석해야 하며, 그렇지 않으면 삼투압 충격으로 세포 모양이 변경되어 측정 오류가 발생합니다. 피펫은 L. tarentolae 세포가 특유의 탁한 모습으로 빠르게 가라앉기 때문에 분광광도계 판독을 하기 몇 초 전에 OD600nm 측정을 위해 1:10 희석액을 혼합(큐벳에서 직접). 발현 배양물의 최종 부피가 근사치로 간주되는 경우, 더 나은 추정 부피(1 g = 1 mL)를 얻기 위해 수확 시 플라스크를 칭량(적절한 빈 플라스크 용기 용기 포함)하는 것도 권장됩니다. 당황한 플라스크에서 L. tarentolae 성장으로 가능한 최대 OD600nm는 15-20이지만, 이는 고정상에 도달하기 때문에 용해물 제조에는 적합하지 않습니다.
2. L. tarentolae 배양의 농도
- 리슈마니아 세포는 파괴되기 전에 약 60배 세척하고 농축해야 합니다. 최종 농축액에 대한 OD600nm = 300을 기준으로 세포 농도의 목표 부피를 계산합니다. 방정식은 V = 수확량(mL) x (수확 OD600nm/300)입니다. 예를 들어, 수확 OD600 = 5인 2L 배양액을 사용하면 목표 부피는 33mL입니다.
참고: OD600nm 타겟 300은 수정할 수 있습니다. 이전 LTE 프로덕션은 150-350 범위의 값을 사용했습니다. 더 높은 농도의 세포가 파괴 상태에 들어가면 더 높은 단백질 수율로 최종 무세포 발현 반응이 발생하는 경향이 있지만 취약한 단백질이 응집되는 경향이 증가합니다. OD600nm = 300은 LTE 생산에 적합한 기본 대상을 나타냅니다. - 채취한 배양액을 적절한 원심분리기 병으로 옮기고 2500 x g 에서 4°C에서 10분 동안 회전시킵니다. 상층액을 배양 폐기물로 조심스럽게 디캔팅합니다.
참고: 폐기된 상층액으로의 세포 손실을 최소화하는 것이 중요한데, 이는 파괴 하중 계산에 영향을 미치기 때문입니다. 이전의 LTE 생산 프로토콜에서, 파괴를 위한 L. tarentolae 세포의 농도는 시험용 미세원심분리기에서 세포 농축액을 회전시키고 총 중량34 대 펠릿 중량을 측정하여 보정하였다. 이 간소화된 프로토콜은 대신 측정된 수확 OD600nm 를 기반으로 농축액에 대한 이론적인 OD600nm 목표를 사용하며 세포 농축 및 세척 중 세포 손실이 낮다고 가정합니다. - 세포 펠릿을 SEB 완충액(45mM HEPES-KOH pH 7.6, 250mM 자당, 100mM KOAc, 3mM Mg(OAc)2, 얼음에 보관)으로 매번 2500 x g 에서 4°C에서 10분 동안 원심분리하여 세 번 세척합니다. 첫 번째 세척의 경우 100mL의 SEB 완충액에 각 1L의 펠릿 배양액을 재현탁한 다음 단일 원심분리기 플라스크에 결합합니다. 두 번째 세척의 경우에도 원래 배양액 1L당 100mL의 SEB를 사용합니다.
참고: 최종 펠릿 재현탁의 경우 최종 목표 재현탁 부피의 50%에 SEB 버퍼를 추가합니다(2.1단계). 이를 통해 풀링된 농축액을 2.4단계에서 최종 목표 부피까지 정확하게 조심스럽게 보충할 수 있습니다. 각 재현탁액은 L. tarentolae의 조기 용해를 방지하기 위해 가능한 한 부드러워야 합니다(예: 디캔팅된 펠릿 주위로 첨가된 SEB를 부드럽게 소용돌이치거나 원심분리기 튜브 벽에 부착된 펠릿 위에 SEB를 피펫팅하는 방법). 최종 단계를 위해 상층액을 더 작은 원심분리기 튜브로 옮기는 것이 더 편리할 수 있습니다. - 재현탁 농축액을 적절한 세척 유리 부피 실린더에 붓고 추가 Cold SEB를 사용하여 목표 부피(단계 2.1)까지 부피를 채우고 부드럽게 혼합합니다.
3. L. tarentolae 농축액의 용해
- 세포 농축액을 4°C로 사전 냉각된 질소 캐비테이션 장치( 재료 표 참조)로 옮기고 70bar의 질소로 가압한 다음 얼음에서 45분 동안 배양합니다.
참고: 질소 캐비테이션 교란기는 일반적인 실험실 품목은 아니지만 LTE 생산에 권장됩니다. 세포 동결-해동 및 프렌치 프레스 유형 방해자와 같은 대체 방법이 시도되었습니다. 그러나 단백질 발현 활성은 질소 캐비테이션 방법을 사용하는 것에 비해 <50%였습니다. 질소 캐비테이션 장치는 이 단계부터 세포 용해물과 접촉하는 모든 재사용 용기(예: 리시버 플라스크)와 유사하게 사용 전과 실행 사이에 철저히 청소해야 합니다. 적절한 세척 요법에는 실험실 세제로 세척한 다음 탈이온수로 철저히 헹구는 것이 포함됩니다. - 질소 캐비테이션 장치의 통풍구를 열고 생성된 용해물을 얼음 위의 진공 리시버 플라스크와 같은 적절하게 견고한 용기로 배출합니다. 리시버 플라스크를 기울여 생성된 모든 용해물이 가라앉고 새 원심분리기 튜브 또는 유사한 용기에 피펫팅할 수 있는지 확인합니다.
주의: 질소 캐비테이션 교란기는 세포 농축액이 70bar의 질소에서 주변 압력으로 갑작스럽게 전환되는 것에 의존하며, 이는 첫 번째 액체의 강한 흐름을 통해 달성된 다음 장치의 출구 밸브를 통한 질소를 통해 달성됩니다. 환기는 화학 안전 후드에 적절한 개인 보호 장비(PPE)를 사용하여 수행해야 합니다. 목적지 용기가 파손되어 용해물이 손실될 위험이 있기 때문에 일반 플라스크 대신 견고한 진공 리시버를 사용합니다. 장치의 출구 밸브가 튜브인 경우 환기 지점에서 과도한 압력 상승을 방지하기 위해 튜브를 수신기 직접 내부에 배치하지 마십시오.
4. 세포 용해물의 원심분리
- 용해물을 적절한 중력 정격 원심분리기 튜브로 옮기고 4°C에서 15분 동안 10,000 x g 의 원심분리기를 사용합니다. 상층액을 신선하고 유사한 원심분리기 튜브로 제거합니다.
- 용해물을 30,000 x g 에서 4 °C에서 15 분 동안 원심 분리 한 다음 최종 상층액을 새 원심 분리기 튜브 또는 얼음 위에 놓인 유사한 용기에 제거합니다. 총 볼륨을 추정합니다.
5. 세포 용해물의 겔 여과
참고: 겔 여과는 SEB 완충액에 포함된 자당을 제거하는 데 사용됩니다. 슈크로스는 세포 파괴 동안 세포 기계를 안정화하는 데 도움이 되지만, 단백질 발현 반응에서 유지되는 경우 수율을 감소시킵니다.
- 충분한 수의 PD-10 중력 공급 겔 여과 컬럼( 재료 표 참조)을 랙 형식으로 설정하여 수집 트레이 또는 아래의 유사한 용기에 떨어뜨릴 수 있도록 하여 컬럼당 2.5mL로 전체 용해물 부피를 여과할 수 있도록 합니다. 사전에 10mL의 4°C EB 완충액(45mM HEPES-KOH pH 7.6, 100mM KOAc, 3mM Mg(OAc)2)을 컬럼에 통과시켜 컬럼을 사전 평형화합니다.
알림: 이 시점부터의 모든 단계는 4°C 냉장실에서 수행하는 것이 좋습니다. 그러나 모든 용해물과 시약을 탁상용 얼음 트레이에 보관하는 것도 적합합니다. 한 가지 예외는 겔 여과 단계로, 저자는 재버퍼링하는 동안 4°C 냉장고 내부에 컬럼 랙을 배치합니다. 이 프로토콜의 원래 버전에서는 새로운 겔 여과 컬럼이 초기에 용해물을 완충하고 첫 번째 출력물을 폐기하여 '차단'되었습니다. 이는 필수는 아니지만 컬럼은 EB 버퍼로 플러시하고 용해물 배치 사이에 4°C에서 보관해야 합니다. 첫 번째 output lysate는 새 컬럼에서 lysate 성분의 일부 배경 머무름으로 인해 후속 outputs보다 단백질 발현 활성이 낮을 수 있습니다. - 각 컬럼에 2.5mL의 용해물을 추가하고 컬럼으로 들어갈 때까지 기다립니다. 0.5mL의 EB를 추가하여 용리액을 버리고 용해물을 컬럼에 가라앉힙니다.
- 각 컬럼에 2.5mL의 EB를 추가하여 겔 여과된 용해물을 용리하고 컬럼 아래에 신선하고 깨끗한 트레이 또는 다른 용기를 놓아 출력물을 수집합니다.
6. 세포 용해물의 보충
- 나노드롭 분광광도계( 재료 표 참조)를 사용하여 겔 여과된 용해물의Abs 280nm 를 측정합니다. 60을 초과하면 추가 4°C EB 버퍼를 사용하여Abs 280nm = 60에 도달하도록 희석합니다.
참고: OD600nm 를 사용하여 붕괴에 대한 세포 밀도 입력을 제어하면 용해물 출력 강도가 대략적으로 결정되지만, 용해물 파괴 및 처리 후 Abs280nm 를 정규화하면 용해물 성능의 배치 일관성이 더욱 향상됩니다. Lysate Abs280nm 는 단백질 발현 수율 및 응집에 영향을 미치면서 상향 및 하향 조정할 수 있습니다(토론 섹션 참조). 보충되지 않은 용해물이 Abs280nm < 60을 나타내는 경우, 파괴 단계에서 더 많은 리슈마니아 바이오매스를 포함해야 할 수 있습니다(즉, 2.1단계에서 세포 분열체 로딩을 OD600nm > 300으로 증가). - 5x 공급 용액(5x FS, 표 1)을 용해물에 2:5 비율로 추가하고 와류가 완전히 혼합됩니다. 적절한 용기(예: 1.5mL 마이크로프리지 튜브)에 분취하고 액체 질소로 스냅 얼립니다. 아래의 LTE 표현 최적화를 위한 선택적 단계 7.1-7.3을 따르는 경우 기본 5x FS 대신 표 1 에서 감소된 rNTP.Mg 5x FS를 사용합니다. 최적화 실험에 사용하기 위해 동결용 5 x 100 μL 부분 표본을 포함합니다.
참고: 2:5 비율로 기본 5x FS로 동결하면 7 μL/10 μL 발현에서 사용되는 발현 준비 보충 LTE가 생성됩니다(따라서 5x FS는 최종 반응에서 1x FS가 됩니다). 그러나 저자는 5x FS에 기본 rNTP 및 마그네슘 양의 0.6배가 제공되는 추가 선택적 단계를 따를 것을 권장합니다. 그 다음에는 두 가지 등몰 혼합물(rNTP.Mg 이라고 함)을 추가하여 최적화된 값으로 테스트 반응을 보충하는 최적화 단계가 이어집니다. 또한 partial 5x FS에는 내인성 mRNA 발현을 차단하는 올리고뉴클레오티드가 포함되어 있습니다(소개 섹션 참조). 올리고뉴클레오티드의 서열은 CAATAAAGTACAGAAACTGATACTTATATAGCGTT입니다.
7. 최종 보충 LTE의 QC 및 최적화
참고: 환원된 rNTP 및 마그네슘 보충 용해물에 rNTP.Mg 의 적절한 '보충' 첨가를 결정하기 위해 필요한 최소 단계에는 융합 파트너 없이 eGFP 또는 유사한 형광단(예: sfGFP)을 발현하는 것이 포함됩니다. 반응에 rNTP.Mg 농도를 증가시켜 발현 수준(다중 모드 플레이트 리더를 통해 eGFP RFU로 측정)이 최적화되는 지점을 결정합니다. 형광이 아닌 eGFP의 조기 종료는 너무 높은 rNTP.Mg 농도에서 eGFP RFU를 감소시키면 분명해집니다. 그러나 LTE의 짧은 산물 오작동은 더 크게 발현된 단백질(>50kDa)에서 더 자주 발생합니다. 따라서, eGFP보다 큰 템플릿을 사용하여 이러한 최적화를 수행할 수 있으며, 특히 적절한 발현 벡터에서 사용 가능한 경우, 특정 응용 분야 또는 연구를 위해 LTE에 의해 생성되기를 원하는 형광단 융합을 제공할 수 있습니다(대표 결과 섹션 참조).
- 100 μL 부분 표본을 해동하고 6 개의 10 μL 발현 반응을 설정하며, 각 반응은 6.2 단계에서 부분적으로 보충 된 7 μL의 용해물, 표 2에 따른 1 μL의 보충 용액 및 반응에서 50 ng / μL의 최종 농도를 달성하기 위해 충분한 DNA 제어 템플릿을 포함하는 2 μL의 초순수로 구성됩니다.
- 25°C에서 2시간 동안 반응을 배양하고 다중 모드 플레이트 리더를 사용하여 GFP 형광의 증가를 모니터링합니다.
참고: GFP에 적합한 구성 값은 485nm(대역폭 5nm)에서의 여기, 516nm(대역폭 5nm)에서의 방출이며, 판독 간격은 2시간 동안 1분입니다. - 가장 높은 eGFP RFU에 해당하는 rNTP.Mg 농도를 결정하기 위해 최종 발현 값의 순위를 매깁니다. 키네틱 데이터를 이용할 수 있는 경우, rNTP.Mg 의 초과는 2시간 발현 기간 동안 eGFP RFU의 바이페이직 증가로도 표시됩니다(대표 결과 섹션 참조).
- 최적화된 보충 rNTP.Mg 농도가 결정되면 이전 단계에서 부분적으로 보충된 LTE 배치를 사용하여 모든 추가 단백질 발현에 추가합니다.
참고: 6.2단계의 부분 표본이 고정 부피로 조심스럽게 수행되는 경우, 예를 들어 부분 표본을 드라이아이스에 놓는 경우와 같이 해동하지 않고 각 부분 표본에 보충을 소급하여 추가할 수 있습니다. 이러한 부분 표본은 이제 완전히 보충되며, 사용을 위해 해동하고 혼합할 때 올바른 rNTP.Mg 보충이 각각을 통해 혼합됩니다.
결과
cell-free 단백질 발현의 목적은 광범위한 응용 분야에 적합한 접힌 활성 형태로 전장 단백질을 생산하는 것입니다. LTE(Leishmania tarentolae 추출물)는 이전에 다른 원핵생물 및 진핵생물 cell-free 발현 시스템과 비교되었으며, 특히 E. coli 기반 cell-free 발현과 비교하여 최적으로 작동할 때 절단 및 응집을 피할 수 있는 높은 용량을 입증했습니다33. ?...
토론
LTE를 생성하기 위한 프로토콜은 지난 10년 동안 발표되었으며7 주기적인 업데이트를 거쳤습니다25,34. 그러나 이 기술을 처음 접하는 사람들은 종종 가파른 학습 곡선에 직면하여 고품질 및 고수율 단백질 발현을 달성하는 데 지연이 발생합니다. LTE35를 사용하는 다른 연구 그룹에서도 유사한 ...
공개
경쟁하는 재정적 이해관계가 존재하지 않습니다.
감사의 말
저자들은 지난 10년 동안 LTE 시스템 개발에 기여한 많은 알렉산드로프 연구소 구성원들, 특히 이 시스템을 개척하고 SITS 리보솜 진입 부위를 개발한 세르게이 무레예프(Sergey Mureev)에게 감사를 표하고자 합니다. 그림 1 은 Biorender.com 에 의해 만들어졌으며 라이선스 하에 복제되었습니다.
자료
| Name | Company | Catalog Number | Comments |
| PD-10 SuperDex 25 Columns | Cytiva | 17085101 | Gel filtration columns |
| Nitrogen Cavitation cell disrupter | Parr Industries | 4635 or 4639 | Cell Disrupter |
| Bovine derived Hemin | Sigma-Aldrich | H5533 | Culture additive |
| Penicillin/Streptomycin 10000U/ml | Thermo-Fisher | 15140122 | Antibiotic mix |
| Optiplate 384 | Perkin-Elmer | 6007290 | Multiwell plate for 10ul expressions |
| Oligonucleotide | IDT synthesis | Oligo with sequence CAATAAAGTACAGAAACTGATAC TTATATAGCGTT | |
| Creatine Phosphokinase | Sigma-Aldrich | 9001-15-4 | Enzyme |
| Tecan Spark | Tecan | or similar Multimode Platereader | |
| Chemidoc MP Imager | Biorad | or similar SDS-PAGE gel Imager | |
| 4-12% Bis-Tris Gels | Invitrogen | NW04125 | SDS-PAGE gels |
| Biophotometer | Eppendorf | or similar Cuvette Specrophotometer | |
| Nanodrop One | Thermofisher | Nanodrop spectrophotometer | |
| Avanti JXN-26 centrifuge | Beckman Coulter | or similar centrifuge, with rotors/tubes rated 10K and 50K g | |
| 5424R microcentrifuge | Eppendorf | or similar microcentrifuge, with 1.5ml microcentrifuge tubes | |
| Flask Incubator Inova S44i | Eppendorf | or similar flask incubator shaker suitable for 5L Flasks | |
| 5L glass culture flasks | Baffled glass flasks for culture growth | ||
| Bactotryptone | BD | 211705 | Growth medium |
| Yeast Extract | Merck | VM930053 | Growth medium |
| Glycerol | Any analytical grade | ||
| Glucose | Any analytical grade | ||
| KH2PO4 | Any analytical grade | ||
| K2HPO4 | Any analytical grade | ||
| UltraPure water | Invitrogen | 10977-015 | Or output from any MilliQ-type water dispenser |
참고문헌
- Nirenberg, M. W., Matthaei, J. H. The dependence of cell-free protein synthesis in E.coli upon naturally occurring or synthetic polyribonucleotides. Proc Natl Acad Sci USA. 47 (10), 1588-1602 (1961).
- Caschera, F., Noireaux, V. Synthesis of 2.3 mg/ml of protein with an all Escherichia coli cell-free transcription-translation system. Biochimie. 99, 162-168 (2014).
- Kelwick, R., Webb, A. J., MacDonald, J. T., Freemont, P. S. Development of a Bacillus subtilis cell-free transcription-translation system for prototyping regulatory elements. Metab Eng. 38, 370-381 (2016).
- Ezure, T., et al. Cell-free protein synthesis system prepared from insect cells by freeze-thawing. Biotechnol Prog. 22 (6), 1570-1577 (2006).
- Harbers, M. Wheat germ systems for cell-free protein expression. FEBS Letters. 588 (17), 2762-2773 (2014).
- Kobs, G. Selecting the cell-free protein expression system that meets your experimental goals. Promega Corporation. 21, 6-9 (2008).
- Kovtun, O., et al. Leishmania cell-free protein expression system. Methods. 55 (1), 58-64 (2011).
- Basile, G., Peticca, M. Recombinant protein expression in Leishmania tarentolae. Mol Biotechnol. 43 (3), 273-278 (2009).
- Mureev, S., Kovtun, O., Nguyen, U. T., Alexandrov, K. Species-independent translational leaders facilitate cell-free expression. Nat Biotechnol. 27 (8), 747-752 (2009).
- Gambin, Y., et al. Single-molecule fluorescence reveals the oligomerization and folding steps driving the prion-like behavior of ASC. J Mol Biol. 430 (4), 491-508 (2018).
- Sierecki, E., et al. Rapid mapping of interactions between human SNX-BAR proteins measured in vitro by AlphaScreen and single-molecule spectroscopy. Mol Cell Proteomics. 13 (9), 2233-2245 (2014).
- Sierecki, E., et al. Nanomolar oligomerization and selective co-aggregation of alpha-synuclein pathogenic mutants revealed by single-molecule fluorescence. Sci Rep. 6, 37630 (2016).
- Leitao, A., Bhumkar, A., Hunter, D. J. B., Gambin, Y., Sierecki, E. Unveiling a selective mechanism for the inhibition of alpha-synuclein aggregation by beta-synuclein. Int J Mol Sci. 19 (2), 334 (2018).
- Gambin, Y., et al. Single-molecule analysis reveals self assembly and nanoscale segregation of two distinct cavin subcomplexes on caveolae. Elife. 3, e01434 (2013).
- Ve, T., et al. Structural basis of TIR-domain-assembly formation in MAL- and MyD88-dependent TLR4 signaling. Nat Struct Mol Biol. 24 (9), 743-751 (2017).
- Guo, Z., et al. Subunit organisation of in vitro reconstituted HOPS and CORVET multisubunit membrane tethering complexes. PLoS One. 8 (12), e81534 (2013).
- Ruehrer, S., Michel, H. Exploiting Leishmania tarentolae cell-free extracts for the synthesis of human solute carriers. Mol Membr Biol. 30 (4), 288-302 (2013).
- Varasteh Moradi, S., et al. Mapping Interactions among cell-free expressed Zika virus proteins. J Proteome Res. 19 (4), 1522-1532 (2020).
- Gagoski, D., et al. Cell-free pipeline for discovery of thermotolerant xylanases and endo-1,4-beta-glucanases. J Biotechnol. 259, 191-198 (2017).
- Ergun Ayva, C., et al. Exploring performance parameters of artificial allosteric protein switches. J Mol Biol. 434 (17), 167678 (2022).
- Lau, D., et al. Fluorescence biosensor for real-time interaction dynamics of host proteins with HIV-1 capsid tubes. ACS Appl Mater Interfaces. 11 (38), 34586-34594 (2019).
- Ryan, S. M., et al. Novel antiinflammatory biologics shaped by parasite-host coevolution. Proc Natl Acad Sci USA. 119 (36), e2202795119 (2022).
- McMahon, K. A., et al. Identification of intracellular cavin target proteins reveals cavin-PP1alpha interactions regulate apoptosis. Nat Commun. 10 (1), 3279 (2019).
- Sierecki, E., et al. A cell-free approach to accelerate the study of protein-protein interactions in vitro. Interface Focus. 3 (5), 20230018 (2013).
- Johnston, W. A., Moradi, S. V., Alexandrov, K. Adaption of the Leishmania cell-free expression system to high-throughput analysis of protein interactions. Methods Mol Biol. 2025, 403-421 (2019).
- Jung, W., et al. Cell-free formation and interactome analysis of caveolae. J Cell Biol. 217 (6), 2141-2165 (2018).
- Fontaine, F. R., et al. Functional domain analysis of SOX18 transcription factor using a single-chain variable fragment-based approach. MAbs. 10 (4), 596-606 (2018).
- Overman, J., et al. Pharmacological targeting of the transcription factor SOX18 delays breast cancer in mice. Elife. 6, e21221 (2017).
- Kubala, M. H., et al. Mammalian farnesyltransferase alpha subunit regulates vacuolar protein sorting-associated protein 4A (Vps4A)--dependent intracellular trafficking through recycling endosomes. Biochem Biophys Res Commun. 468 (4), 580-586 (2015).
- Han, S. P., et al. Cortactin scaffolds Arp2/3 and WAVE2 at the epithelial zonula adherens. J Biol Chem. 289 (11), 7764-7775 (2014).
- Das Gupta, K., et al. Class IIa histone deacetylases drive toll-like receptor-inducible glycolysis and macrophage inflammatory responses via pyruvate kinase M2. Cell Rep. 30 (8), 2712-2728.e8 (2020).
- Leitão, A. D. G., et al. Selectivity of protein interactions along the aggregation pathway of α-synuclein. BioRxiv. , (2021).
- Gagoski, D., et al. Performance benchmarking of four cell-free protein expression systems. Biotechnol Bioeng. 113 (2), 292-300 (2016).
- Johnston, W. A., Alexandrov, K. Production of eukaryotic cell-free lysate from Leishmania tarentolae. Methods Mol Biol. 1118, 1-15 (2014).
- Hunter, D. J. B., Bhumkar, A., Giles, N., Sierecki, E., Gambin, Y. Unexpected instabilities explain batch-to-batch variability in cell-free protein expression systems. Biotechnol Bioeng. 115 (8), 1904-1914 (2018).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유