Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Producción y optimización de LTE, un sistema de expresión de proteínas libres de células derivadas de Leishmania tarentolae para la producción de proteínas recombinantes
En este artículo
Resumen
El extracto traslacional de Leishmania (LTE) es un sistema de expresión de proteínas eucariotas libres de células derivado del parásito unicelular, Leishmania tarentolae. Este protocolo optimizado hace que la fabricación de LTE sea sencilla y rentable. Es adecuado para diversas aplicaciones centradas en la expresión y el estudio multiparalelo de proteínas eucariotas complejas y sus interacciones.
Resumen
Este protocolo describe la producción y optimización de un Sistema de Expresión de Proteínas Libres de Células (CFPS) eucariotas derivado del flagelado unicelular Leishmania tarentolae, denominado Extracto Traslacional de Leishmania o LTE. Aunque este organismo evolucionó originalmente como un parásito de los geckos, se puede cultivar de manera fácil y económica en frascos o biorreactores. A diferencia de la Leishmania major, no es patógena para los seres humanos y no requiere precauciones especiales de laboratorio. Otra ventaja del uso de Leishmania para el CFPS es que la adición de un solo oligonucleótido antisentido al CFPS, dirigido a una secuencia líder de empalme conservada en el extremo 5' de todos los ARN codificantes de proteínas, puede suprimir la expresión de proteínas endógenas. Proporcionamos procedimientos para la disrupción celular y el procesamiento de lisados, que se han simplificado y mejorado en comparación con las versiones anteriores. Estos procedimientos comienzan con cultivos de matraces simples. Además, explicamos cómo introducir información genética utilizando vectores que contienen sitios de iniciación de traducción independientes de la especie (SITS) y cómo realizar una optimización sencilla de los lotes y un control de calidad para garantizar una calidad constante de la expresión de proteínas.
Introducción
En la década de 1960, los sistemas de expresión de proteínas libres de células desempeñaron un papel fundamental en el descubrimientodel código genético. Sin embargo, los sistemas de expresión de proteínas libres de células procariotas, basados principalmente en E. coli, dominan actualmente tanto las aplicaciones de laboratorio como las comerciales. Si bien los sistemas basados en E. coli ofrecen ventajas como rentabilidad, escalabilidad y altos rendimientos de expresión, enfrentan desafíos cuando producen proteínas multidominio en sus formas activas y facilitan el ensamblaje de complejos proteicos 2,3. En la actualidad, las formas comúnmente utilizadas de síntesis de proteínas eucariotas libres de células (CFPS) incluyen extracto de germen de trigo (WGE), lisado de reticulocitos de conejo (RRL) y lisado de células de insectos (ICL)4,5,6. Este trabajo presenta un sistema alternativo sin células eucariotas, sencillo y escalable, basado en el parásito flagelado unicelular Leishmania tarentolae.
La Leishmania tarentolae puede cultivarse fácilmente en matraces utilizando medios rentables y también puede ampliarse en biorreactores para lograr una mayor densidad celular. La presencia de ARNm endógenos en el lisado celular, que de otro modo podrían competir con los mensajes introducidos, puede neutralizarse utilizando oligonucleótidos antisentido dirigidos a la secuencia líder de empalme de ARNm de Leishmania conservada7. A diferencia de su pariente cercano Leishmania major, que causa enfermedades humanas, L. tarentolae infecta al gecko moruno (Tarentolae mauritanica), por lo que es adecuado para el cultivo en ambientes de laboratorio PC2 sin necesidad de precauciones especiales. Anteriormente se ha utilizado como organismo transgénico para la expresión de proteínas in vivo8.
Para facilitar el cebado de plantillas en sistemas libres de células, se han diseñado secuencias universales basadas en estructuras poliméricas de ARN que mejoran la iniciación traduccional9. Estas secuencias de traducción independientes de la especie (SITS) son aplicables tanto a los sistemas libres de células procariotas como a los eucariotas y son adecuadas para introducir información genética en LTE. Si bien este protocolo no proporciona una explicación detallada de la construcción de vectores para la expresión de proteínas libres de células LTE, la optimización y el control de calidad requieren vectores adecuados que contengan fusiones de fluoróforos de las proteínas de interés deseadas aguas abajo del sitio SITS. Para ello, se han depositado vectores LTE apropiados en el repositorio de genes Addgene, como el vector pCellFree_G03, que codifica una fusión N-terminal de eGFP a la proteína deseada de interés utilizando sitios de clonación Gateway.
LTE ha demostrado su valor en una amplia gama de aplicaciones que requieren expresión de proteínas, incluido el análisis del autoensamblaje de proteínas10,16, la producción de proteínas de membrana integral humana17, la investigación de candidatos a fármacos antivirales18, el desarrollo de enzimas biotecnológicamente útiles19, la creación de prototipos de biosensores de proteínas20,21 y el estudio de productos biológicos a partir de anquilostomas22. LTE también ha sido fundamental en el mapeo de redes de interacción proteína-proteína en los campos de la virología y las estructuras celulares21,32. Se ha evaluado el rendimiento de LTE de manera similar a otros sistemas eucariotas libres de células en la expresión de proteínas de longitud completa, monodispersas y no agregadas33, al tiempo que ofrece una producción más rentable y escalable.
Este protocolo proporciona técnicas para cultivar y alterar el organismo huésped, preparar lisado y complementar una solución de alimentación (FS) para la expresión de proteínas de transcripción/traducción acopladas. Además, incluye un protocolo para optimizar los lotes de producción. En la versión inicial del sistema libre de células de Leishmania, se observó una variación no deseada de un lote a otro en los niveles de expresión, la fracción de proteínas de longitud completa y la presencia de agregados de proteínas, lo que llevó a la eliminación de los lotes34. Posteriormente, se realizaron mejoras en el protocolo para abordar este problema25. El protocolo actual se basa en estas mejoras, lo que permite optimizar los lotes individuales para la expresión y el tamaño máximos de la proteína. Esto se logra controlando de cerca la carga del disruptor celular (medida como densidad óptica a 600 nm; OD600 nm) y normalizando la salida de lisado resultante utilizando absorbancia a 280 nm (Abs280 nm). Además, incorpora un método para suplementar parcialmente el lisado con rNTP y magnesio durante la fabricación, con la posterior optimización de estos componentes de la solución alimenticia durante las expresiones de prueba. Aunque esta optimización se presenta como una opción en el protocolo, los autores la recomiendan encarecidamente.
Protocolo
Este protocolo incluye recetas detalladas de medios y pasos que implican cultivo, centrifugación, medición de la fluorescencia GFP mediante un lector de placas multimodo, medición de cultivo OD600 nm y evaluación de lisado Abs280 nm. También cubre la configuración y la obtención de imágenes de los geles de proteínas SDS-PAGE. Los materiales necesarios o sugeridos para este protocolo se enumeran en la hoja de cálculo Materiales. Es importante tener en cuenta que los recursos típicos de laboratorio, como los componentes de los medios, las centrífugas, los tubos, los espectrofotómetros y las configuraciones de electroforesis en gel, probablemente se puedan utilizar indistintamente, a menos que se especifique lo contrario. La Figura 1 proporciona un resumen del proceso de fabricación LTE.
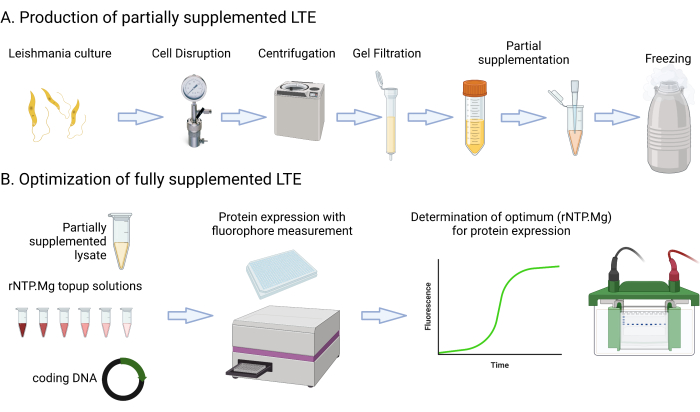
Figura 1: Descripción general del protocolo de fabricación LTE. Esta caricatura proporciona un resumen conciso del protocolo de fabricación LTE. Haga clic aquí para ver una versión más grande de esta figura.
1. Crecimiento de los cultivos de Leishmania tarentolae
- Prepare al menos 3 L de medio de crecimiento TBGG (Bactotriptona 12 g/L, Extracto de levadura 24 g/L, glicerol 8 mL/L, glucosa 1 g/L, KH2PO4 2,3 g/L, K2HPO4 2,5 g/L, ver Tabla de Materiales). Esterilice el medio utilizando un filtro de 0,22 μm al vacío o una configuración similar.
- Almacene los medios a temperatura ambiente (RT), con las adiciones finales (hemina, antibióticos) añadidas justo antes de la inoculación con L. tarentolae. Se añade hemina (0,25% v/v en 50% trietanolamina) al 0,2% v/v, penicilina (10.000 unidades/mL) más estreptomicina (10.000 μg/mL) a 0,5% v/v.
NOTA: El punto de partida de este protocolo es un cultivo mantenido de 2 x 10 mL de L . tarentolae de tipo silvestre. Los cultivos de mantenimiento se cultivan a 27 °C en matraces de cultivo de tejidos estándar de 50 mL con baja agitación (75 rpm). Estos cultivos de 10 mL se pueden mantener indefinidamente con diluciones de ~ 1/20 en estériles (TBGG + hemina, penicilina, estreptomicina) cada 2-3 días. Se recomienda una cabina de bioseguridad estándar en un laboratorio PC2; sin embargo, las contaminaciones bacterianas tienden a prevenirse con los antibióticos agregados, mientras que las contaminaciones fúngicas generalmente son superadas por L. tarentolae. - Durante dos días, amplíe los cultivos de mantenimiento de L. tarentolae a 200 mL (día 1) y luego a 2 L (día 2) mediante diluciones 1:10 con un volumen creciente de TBGG + hemina/antibióticos cada día. Realizar ambas diluciones en matraces de vidrio de 5 L esterilizados en autoclave (llenos hasta un máximo de 1 L). La segunda dilución debe ocurrir por la tarde entre las 3 y las 6 de la tarde, con la intención de comenzar la producción de lisado al día siguiente entre las 8 y las 11 de la mañana.
NOTA: Este protocolo utiliza el volumen inicial mínimo para la producción de LTE (2 cultivos de 1 L). También es posible ampliar el cultivo hasta 10 L para la producción de LTE incorporando un paso de expansión adicional (por ejemplo, Día 1: 100 mL; Día 2: 1 L; Día 3: 10 L). Aunque este protocolo utiliza matraces deflectores (ver Tabla de Materiales) para cultivar L. tarentolae, opcionalmente, se pueden utilizar biorreactores convencionales diseñados para el crecimiento bacteriano con impulsores Rushton, siempre que la tasa de agitación se mantenga por debajo de 100 rpm. La mejora de la aireación y el control del pH en los biorreactores generalmente extienden el crecimiento de la fase logarítmica de los cultivos de L. tarentolae , lo que permite una cosecha más alta de600 nm OD de 10 que se utilizará en el paso 1.4. - Registre el diámetro exterior de600 nm del cultivo por triplicado mediante una dilución 1:10 en TBGG directamente en la cubeta del espectrofotómetro. Un rango de inicio adecuado para hacer lisado es OD600nm = 4.0-8.0.
- Proporcione tiempo de incubación adicional si OD600 nm < 4.0. Un cultivo con DOde 600 nm > 8 es utilizable y dará como resultado un mayor volumen de lisado de expresión libre de células, pero con menor calidad debido al inicio de la fase de crecimiento logarítmico tardío. Coloque los matraces de cultivo en hielo, a la espera de los pasos siguientes.
NOTA: La medición precisa del diámetro exterior del cultivo final de600 nm es fundamental, ya que se utiliza para calcular el volumen final de las células concentradas antes de la interrupción. Este cálculo sustituye a un método de pesaje de pellets utilizado en versiones anteriores de la fabricación de LTE para calibrar la concentración de células antes de la interrupción34, con el fin de simplificar el protocolo. Asegúrese de diluir en TBGG para la medición de OD600 nm , ya que de lo contrario, el choque osmótico cambia la forma de la celda, causando un error de medición. Mezcle con pipeta la dilución 1:10 para la medición de OD600 nm (directamente en la cubeta) segundos antes de tomar la lectura espectrofotométrica, ya que las células de L. tarentolae se asientan rápidamente con una apariencia turbia distintiva. Si el volumen final del cultivo de expresión se considera aproximado, también se recomienda pesar los matraces en el momento de la cosecha (con la tara de los matraces vacíos adecuados) para obtener un volumen estimado mejor (a 1 g = 1 mL). El diámetro exterior máximo de600 nm posible del crecimiento de L. tarentolas en matraces deflectores es de 15-20, aunque esto es inapropiado para la fabricación de lisado debido a que alcanza la fase estacionaria.
2. Concentración de cultivos de L. tarentolae
- Las células de Leishmania deben lavarse y concentrarse aproximadamente 60 veces antes de la interrupción. Calcule el volumen objetivo para la concentración de celdas en función de OD600 nm = 300 para el concentrado final. La ecuación es V = volumen de cosecha (mL) x (DO de cosecha600nm/300). Por ejemplo, si se utiliza un cultivo de 2 L con un diámetro exterior de cosechade 600 = 5, el volumen objetivo es de 33 mL.
NOTA: El objetivo OD600nm de 300 se puede modificar; La producción anterior de LTE ha utilizado valores en el rango de 150-350. Las concentraciones más altas de células que entran en disrupción tenderán a producir reacciones finales de expresión libre de células con mayores rendimientos de proteínas, pero con una mayor tendencia a que las proteínas vulnerables se agreguen. OD600nm = 300 representa un objetivo predeterminado adecuado para la producción LTE. - Transfiera los cultivos cosechados a botellas centrífugas adecuadas y gírelas a 2500 x g durante 10 min a 4 °C. Decantar cuidadosamente el sobrenadante en los residuos de cultivo.
NOTA: Es importante minimizar la pérdida de células en el sobrenadante desechado, ya que afecta el cálculo de la carga de interrupción. En los protocolos de producción de LTE anteriores, la concentración de células de L. tarentolae para la disrupción se calibraba girando el concentrado de células en un tubo de microcentrífuga de prueba y midiendo el peso de los pellets en comparación con el peso total34. En cambio, este protocolo simplificado utiliza un objetivo teórico deDE 600 nm para el concentrado, basado en el DE de cosecha medido de600 nm, y supone una baja pérdida de células durante la concentración de células y el lavado. - Lavar el pellet de celda en tampón SEB (45 mM HEPES-KOH pH 7,6, 250 mM de sacarosa, 100 mM KOAc, 3 mM Mg(OAc)2, mantenido en hielo) tres veces, cada vez centrifugando a 2500 x g durante 10 min a 4 °C. Para el primer lavado, vuelva a suspender cada 1 L de cultivo granulado en 100 mL de tampón SEB, luego combínelos en un solo matraz de centrífuga. Para el segundo lavado, use también 100 mL de SEB por cada 1 L del cultivo original.
NOTA: Para la resuspensión final de pellets, agregue tampón SEB hasta el 50% del volumen de resuspensión objetivo final (paso 2.1). Esto permite que el concentrado agrupado se rellene cuidadosamente hasta exactamente el volumen objetivo final en el paso 2.4. Cada resuspensión debe ser lo más suave posible para evitar la lisis prematura de L. tarentolae, por ejemplo, girando suavemente el SEB añadido alrededor del pellet decantado o pipeteando el SEB sobre el pellet adherido a la pared del tubo de centrífuga. Puede ser más conveniente transferir los sobrenadantes a tubos de centrífuga más pequeños para el paso final. - Vierta el concentrado resuspendido en un cilindro volumétrico de vidrio lavado adecuado, luego rellene el volumen hasta el volumen objetivo (paso 2.1) con SEB frío adicional y mezcle suavemente.
3. Lisis del concentrado de L. tarentolae
- Transfiera el concentrado celular al dispositivo de cavitación de nitrógeno (consulte la tabla de materiales) preenfriado a 4 ° C, presurícelo a 70 bares de nitrógeno e incube durante 45 minutos en hielo.
NOTA: Si bien los disruptores de cavitación de nitrógeno no son elementos comunes de laboratorio, se recomiendan para la producción de LTE. Se han ensayado métodos alternativos como la congelación-descongelación celular y los disruptores de tipo prensa francesa; sin embargo, la actividad de expresión de proteínas fue <50% en comparación con el uso del método de cavitación de nitrógeno. El dispositivo de cavitación de nitrógeno debe limpiarse a fondo antes de su uso y entre pasadas, de forma similar a todos los recipientes reutilizados que entran en contacto con el lisado celular a partir de esta etapa (como el matraz receptor). Un régimen de limpieza adecuado consiste en lavar con detergentes de laboratorio seguido de un enjuague a fondo con agua desionizada. - Abra el respiradero del dispositivo de cavitación de nitrógeno y expulse el lisado resultante a un recipiente adecuadamente robusto, como un matraz receptor de vacío en hielo. Incline el matraz receptor para asegurarse de que todo el lisado resultante se asiente y pueda pipetearse en un tubo de centrífuga nuevo o en un recipiente similar.
PRECAUCIÓN: Los disruptores de cavitación de nitrógeno se basan en la transición abrupta del concentrado celular de 70 bares de nitrógeno a la presión ambiente, lograda a través de un fuerte flujo de líquido primero y luego de nitrógeno a través de la válvula de salida del dispositivo. La ventilación debe realizarse con el equipo de protección personal (EPP) adecuado en una capucha de seguridad química. Existe el riesgo de romper el recipiente de destino y perder el lisado, por lo que utilizamos un receptor de vacío resistente en lugar de un matraz genérico. Si la válvula de salida del dispositivo es un tubo, evite colocar el tubo directamente dentro del receptor para evitar la acumulación excesiva de presión en el punto de ventilación.
4. Centrifugación del lisado celular
- Transfiera el lisado a tubos de centrífuga con fuerza nominal g adecuada y centrifugar a 10.000 x g durante 15 min a 4 °C. Retire el sobrenadante y colóquelo en tubos de centrífuga nuevos y similares.
- Centrifugar el lisado a 30.000 x g durante 15 min a 4 °C y, a continuación, retirar el sobrenadante final a un tubo de centrífuga nuevo o a un recipiente similar colocado sobre hielo. Estimar el volumen total.
5. Filtración en gel de lisado celular
NOTA: La filtración en gel se utiliza para eliminar la sacarosa incluida en el tampón SEB. Si bien la sacarosa ayuda a estabilizar la maquinaria celular durante la disrupción celular, disminuye el rendimiento si se retiene en las reacciones de expresión de proteínas.
- Instale un número suficiente de columnas de filtración de gel PD-10 alimentadas por gravedad (consulte la Tabla de materiales) en un formato de rejilla que les permita gotear en una bandeja de recolección o en un recipiente similar que se encuentre debajo, asegurando que puedan filtrar todo el volumen de lisado a 2,5 mL por columna. Preequilibrar las columnas pasando previamente 10 mL de tampón EB a 4 °C (45 mM HEPES-KOH pH 7,6, 100 mM KOAc, 3 mM Mg(OAc)2) a través de ellas.
NOTA: Todos los pasos a partir de este punto se benefician de ser realizados en una cámara frigorífica a 4 °C. Sin embargo, también es adecuado mantener todos los lisados y reactivos en una bandeja de hielo de sobremesa. Una excepción es la etapa de filtración de gel, en la que los autores colocan un estante de columnas dentro de un refrigerador a 4 °C mientras se vuelve a almacenar. En las versiones originales de este protocolo, las nuevas columnas de filtración de gel se "bloqueaban" almacenando inicialmente el lisado y descartando la primera salida. Aunque esto no se considera necesario, las columnas deben lavarse con tampón EB y almacenarse a 4 °C entre lotes de lisado. El primer lisado de salida puede tener una actividad de expresión de proteínas más baja que las salidas posteriores debido a cierta retención de fondo de los componentes del lisado en la nueva columna. - Agregue 2,5 mL de lisado a cada columna y espere hasta que pase a la columna. Agregue 0,5 mL adicionales de EB para asentar el lisado en la columna mientras se desecha el eluido.
- Eluir el lisado filtrado en gel añadiendo 2,5 mL adicionales de EB a cada columna, recogiendo la salida colocando una bandeja fresca y limpia u otro receptáculo debajo de las columnas.
6. Suplementación con lisado celular
- Utilice el espectrofotómetro de nanogotas (consulte la tabla de materiales) para medir Abs280 nm del lisado filtrado en gel. Si supera los 60, diluirlo hasta alcanzar un Absde 280 nm = 60 utilizando un tampón EB adicional de 4 °C.
NOTA: Si bien el control de la entrada de densidad celular en la interrupción con OD600 nm determina aproximadamente la fuerza de salida del lisado, la normalización del Abs280 nm después de la interrupción y el procesamiento del lisado mejora aún más la consistencia del lote del rendimiento del lisado. El lisado Abs de280 nm se puede ajustar hacia arriba y hacia abajo, con consecuencias para el rendimiento y la agregación de la expresión de proteínas (consulte la sección de discusión). Si el lisado no suplementado indica un Absde 280 nm < 60, puede ser necesario incluir más biomasa de Leishmania en la etapa de interrupción, es decir, aumentar la carga del disruptor celular aDE 600 nm > 300 en la etapa 2.1. - Agregue 5 veces la solución de alimentación (5 veces FS, Tabla 1) al lisado en una proporción de 2:5 y mezcle bien el vórtice. Coloque alícuotas en recipientes adecuados (por ejemplo, tubos de microfuga de 1,5 ml) y congélelo en nitrógeno líquido. Si se siguen los pasos opcionales 7.1-7.3 para la optimización de expresiones LTE que se indican a continuación, utilice el FS reducido rNTP.Mg 5x de la Tabla 1 en lugar del FS 5x predeterminado. Incluya 5 alícuotas de 100 μL para congelar para su uso en los experimentos de optimización.
NOTA: La congelación con el FS predeterminado 5x en una proporción de 2:5 crea LTE suplementado listo para la expresión, utilizado a una expresión de 7 μL/10 μL (por lo tanto, el FS 5x se convierte en 1x FS en la reacción final). Sin embargo, los autores recomiendan seguir los pasos opcionales adicionales en los que se proporciona 0,6 veces la cantidad predeterminada de rNTP y magnesio en el FS 5x. A esto le sigue un paso de optimización en el que se añade una mezcla equimolar de los dos (denominada rNTP.Mg) para completar las reacciones de prueba a un valor optimizado. El FS parcial 5x también contiene un oligonucleótido que bloquea la expresión endógena del ARNm (ver sección de Introducción). La secuencia del oligonucleótido es CAATAAAGTACAGAAACTGATACTTATATAGCGTT.
7. Control de calidad y optimización de LTE final complementado
NOTA: Los pasos mínimos necesarios para determinar la adición "de recarga" adecuada de rNTP.Mg al rNTP reducido y al lisado suplementado con magnesio implican la expresión de eGFP o un fluoróforo similar (por ejemplo, sfGFP) sin un socio de fusión. Se añaden concentraciones crecientes de rNTP.Mg a las reacciones para determinar el punto en el que se optimiza el nivel de expresión (medido como RFU eGFP a través de un lector de placas multimodo). Las terminaciones prematuras de eGFP, que no son fluorescentes, se hacen evidentes al disminuir la RFU de eGFP a concentraciones de rNTP.Mg demasiado altas. Sin embargo, el mal funcionamiento de LTE de productos cortos ocurre con mayor frecuencia en proteínas expresadas más grandes (>50 kDa). Por lo tanto, es posible realizar esta optimización utilizando una plantilla más grande que eGFP, especialmente si hay una disponible en un vector de expresión adecuado, proporcionando una fusión de fluoróforos que se desea producir mediante LTE para una aplicación o estudio en particular (consulte la sección Resultados representativos).
- Descongele una alícuota de 100 μL y configure seis reacciones de expresión de 10 μL, cada una compuesta por 7 μL de lisado parcialmente suplementado del paso 6.2, 1 μL de solución de recarga según la Tabla 2 y 2 μL de agua ultrapura que contenga suficiente plantilla de control de ADN para lograr una concentración final de 50 ng/μL en la reacción.
- Incubar las reacciones durante 2 h a 25 °C y monitorizar el aumento de la fluorescencia de GFP mediante un lector de placas multimodo.
NOTA: Los valores de configuración adecuados para GFP son excitación a 485 nm (ancho de banda de 5 nm), emisión a 516 nm (ancho de banda de 5 nm), con un intervalo de lectura de 1 min durante 2 h. - Clasifique los valores de expresión final para determinar la concentración de rNTP.Mg correspondiente a la RFU de eGFP más alta. Si se dispone de datos cinéticos, un exceso de rNTP.Mg también se indicará mediante un aumento bifásico de la RFU de eGFP durante el período de expresión de 2 h (véase la sección Resultados representativos).
- Una vez que se determine la concentración de rNTP.Mg de recarga optimizada, agréguela a todas las expresiones de proteínas adicionales utilizando el lote de LTE que se suplementó parcialmente en los pasos anteriores.
NOTA: Si la alícuota en el paso 6.2 se realiza con cuidado con volúmenes fijos, la recarga se puede agregar retrospectivamente a cada alícuota sin descongelar, por ejemplo, con las alícuotas colocadas en hielo seco. Estas alícuotas ahora se complementan por completo, ya que la recarga de rNTP.Mg correcta se mezclará a través de cada una de ellas cuando se descongelen y se mezclen para su uso.
Resultados
El propósito de la expresión de proteínas libres de células es producir proteínas de longitud completa en una forma activa plegada adecuada para una amplia gama de aplicaciones. El LTE (extracto de Leishmania tarentoleae ) se ha comparado previamente con otros sistemas de expresión libre de células procariotas y eucariotas, demostrando una alta capacidad para evitar el truncamiento y la agregación cuando funciona de manera óptima, particularmente en comparación con la ...
Discusión
Los protocolos para la creación de LTE se han publicado en la última década7 y han sido objeto de actualizaciones periódicas25,34. Sin embargo, los recién llegados a la técnica a menudo se encuentran con una curva de aprendizaje empinada, lo que resulta en retrasos para lograr la expresión de proteínas de alta calidad y alto rendimiento. Otros grupos de investigación que trabajan con LTE
Divulgaciones
No hay intereses financieros contrapuestos.
Agradecimientos
Los autores desean agradecer a los muchos miembros del laboratorio Alexandrov que han contribuido al desarrollo de los sistemas LTE en los últimos 10 años, en particular a Sergey Mureev, quien fue pionero en el sistema y desarrolló el sitio de entrada de ribosomas SITS. La Figura 1 fue creada por Biorender.com y reproducida bajo licencia.
Materiales
| Name | Company | Catalog Number | Comments |
| PD-10 SuperDex 25 Columns | Cytiva | 17085101 | Gel filtration columns |
| Nitrogen Cavitation cell disrupter | Parr Industries | 4635 or 4639 | Cell Disrupter |
| Bovine derived Hemin | Sigma-Aldrich | H5533 | Culture additive |
| Penicillin/Streptomycin 10000U/ml | Thermo-Fisher | 15140122 | Antibiotic mix |
| Optiplate 384 | Perkin-Elmer | 6007290 | Multiwell plate for 10ul expressions |
| Oligonucleotide | IDT synthesis | Oligo with sequence CAATAAAGTACAGAAACTGATAC TTATATAGCGTT | |
| Creatine Phosphokinase | Sigma-Aldrich | 9001-15-4 | Enzyme |
| Tecan Spark | Tecan | or similar Multimode Platereader | |
| Chemidoc MP Imager | Biorad | or similar SDS-PAGE gel Imager | |
| 4-12% Bis-Tris Gels | Invitrogen | NW04125 | SDS-PAGE gels |
| Biophotometer | Eppendorf | or similar Cuvette Specrophotometer | |
| Nanodrop One | Thermofisher | Nanodrop spectrophotometer | |
| Avanti JXN-26 centrifuge | Beckman Coulter | or similar centrifuge, with rotors/tubes rated 10K and 50K g | |
| 5424R microcentrifuge | Eppendorf | or similar microcentrifuge, with 1.5ml microcentrifuge tubes | |
| Flask Incubator Inova S44i | Eppendorf | or similar flask incubator shaker suitable for 5L Flasks | |
| 5L glass culture flasks | Baffled glass flasks for culture growth | ||
| Bactotryptone | BD | 211705 | Growth medium |
| Yeast Extract | Merck | VM930053 | Growth medium |
| Glycerol | Any analytical grade | ||
| Glucose | Any analytical grade | ||
| KH2PO4 | Any analytical grade | ||
| K2HPO4 | Any analytical grade | ||
| UltraPure water | Invitrogen | 10977-015 | Or output from any MilliQ-type water dispenser |
Referencias
- Nirenberg, M. W., Matthaei, J. H. The dependence of cell-free protein synthesis in E.coli upon naturally occurring or synthetic polyribonucleotides. Proc Natl Acad Sci USA. 47 (10), 1588-1602 (1961).
- Caschera, F., Noireaux, V. Synthesis of 2.3 mg/ml of protein with an all Escherichia coli cell-free transcription-translation system. Biochimie. 99, 162-168 (2014).
- Kelwick, R., Webb, A. J., MacDonald, J. T., Freemont, P. S. Development of a Bacillus subtilis cell-free transcription-translation system for prototyping regulatory elements. Metab Eng. 38, 370-381 (2016).
- Ezure, T., et al. Cell-free protein synthesis system prepared from insect cells by freeze-thawing. Biotechnol Prog. 22 (6), 1570-1577 (2006).
- Harbers, M. Wheat germ systems for cell-free protein expression. FEBS Letters. 588 (17), 2762-2773 (2014).
- Kobs, G. Selecting the cell-free protein expression system that meets your experimental goals. Promega Corporation. 21, 6-9 (2008).
- Kovtun, O., et al. Leishmania cell-free protein expression system. Methods. 55 (1), 58-64 (2011).
- Basile, G., Peticca, M. Recombinant protein expression in Leishmania tarentolae. Mol Biotechnol. 43 (3), 273-278 (2009).
- Mureev, S., Kovtun, O., Nguyen, U. T., Alexandrov, K. Species-independent translational leaders facilitate cell-free expression. Nat Biotechnol. 27 (8), 747-752 (2009).
- Gambin, Y., et al. Single-molecule fluorescence reveals the oligomerization and folding steps driving the prion-like behavior of ASC. J Mol Biol. 430 (4), 491-508 (2018).
- Sierecki, E., et al. Rapid mapping of interactions between human SNX-BAR proteins measured in vitro by AlphaScreen and single-molecule spectroscopy. Mol Cell Proteomics. 13 (9), 2233-2245 (2014).
- Sierecki, E., et al. Nanomolar oligomerization and selective co-aggregation of alpha-synuclein pathogenic mutants revealed by single-molecule fluorescence. Sci Rep. 6, 37630 (2016).
- Leitao, A., Bhumkar, A., Hunter, D. J. B., Gambin, Y., Sierecki, E. Unveiling a selective mechanism for the inhibition of alpha-synuclein aggregation by beta-synuclein. Int J Mol Sci. 19 (2), 334 (2018).
- Gambin, Y., et al. Single-molecule analysis reveals self assembly and nanoscale segregation of two distinct cavin subcomplexes on caveolae. Elife. 3, e01434 (2013).
- Ve, T., et al. Structural basis of TIR-domain-assembly formation in MAL- and MyD88-dependent TLR4 signaling. Nat Struct Mol Biol. 24 (9), 743-751 (2017).
- Guo, Z., et al. Subunit organisation of in vitro reconstituted HOPS and CORVET multisubunit membrane tethering complexes. PLoS One. 8 (12), e81534 (2013).
- Ruehrer, S., Michel, H. Exploiting Leishmania tarentolae cell-free extracts for the synthesis of human solute carriers. Mol Membr Biol. 30 (4), 288-302 (2013).
- Varasteh Moradi, S., et al. Mapping Interactions among cell-free expressed Zika virus proteins. J Proteome Res. 19 (4), 1522-1532 (2020).
- Gagoski, D., et al. Cell-free pipeline for discovery of thermotolerant xylanases and endo-1,4-beta-glucanases. J Biotechnol. 259, 191-198 (2017).
- Ergun Ayva, C., et al. Exploring performance parameters of artificial allosteric protein switches. J Mol Biol. 434 (17), 167678 (2022).
- Lau, D., et al. Fluorescence biosensor for real-time interaction dynamics of host proteins with HIV-1 capsid tubes. ACS Appl Mater Interfaces. 11 (38), 34586-34594 (2019).
- Ryan, S. M., et al. Novel antiinflammatory biologics shaped by parasite-host coevolution. Proc Natl Acad Sci USA. 119 (36), e2202795119 (2022).
- McMahon, K. A., et al. Identification of intracellular cavin target proteins reveals cavin-PP1alpha interactions regulate apoptosis. Nat Commun. 10 (1), 3279 (2019).
- Sierecki, E., et al. A cell-free approach to accelerate the study of protein-protein interactions in vitro. Interface Focus. 3 (5), 20230018 (2013).
- Johnston, W. A., Moradi, S. V., Alexandrov, K. Adaption of the Leishmania cell-free expression system to high-throughput analysis of protein interactions. Methods Mol Biol. 2025, 403-421 (2019).
- Jung, W., et al. Cell-free formation and interactome analysis of caveolae. J Cell Biol. 217 (6), 2141-2165 (2018).
- Fontaine, F. R., et al. Functional domain analysis of SOX18 transcription factor using a single-chain variable fragment-based approach. MAbs. 10 (4), 596-606 (2018).
- Overman, J., et al. Pharmacological targeting of the transcription factor SOX18 delays breast cancer in mice. Elife. 6, e21221 (2017).
- Kubala, M. H., et al. Mammalian farnesyltransferase alpha subunit regulates vacuolar protein sorting-associated protein 4A (Vps4A)--dependent intracellular trafficking through recycling endosomes. Biochem Biophys Res Commun. 468 (4), 580-586 (2015).
- Han, S. P., et al. Cortactin scaffolds Arp2/3 and WAVE2 at the epithelial zonula adherens. J Biol Chem. 289 (11), 7764-7775 (2014).
- Das Gupta, K., et al. Class IIa histone deacetylases drive toll-like receptor-inducible glycolysis and macrophage inflammatory responses via pyruvate kinase M2. Cell Rep. 30 (8), 2712-2728.e8 (2020).
- Leitão, A. D. G., et al. Selectivity of protein interactions along the aggregation pathway of α-synuclein. BioRxiv. , (2021).
- Gagoski, D., et al. Performance benchmarking of four cell-free protein expression systems. Biotechnol Bioeng. 113 (2), 292-300 (2016).
- Johnston, W. A., Alexandrov, K. Production of eukaryotic cell-free lysate from Leishmania tarentolae. Methods Mol Biol. 1118, 1-15 (2014).
- Hunter, D. J. B., Bhumkar, A., Giles, N., Sierecki, E., Gambin, Y. Unexpected instabilities explain batch-to-batch variability in cell-free protein expression systems. Biotechnol Bioeng. 115 (8), 1904-1914 (2018).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados