需要订阅 JoVE 才能查看此. 登录或开始免费试用。
Method Article
新旧嗜坏死果蝇样品的 DNA 提取和比较
* 这些作者具有相同的贡献
Erratum Notice
摘要
本文介绍了使用改良的常用 DNA 提取试剂盒提取新鲜和旧嗜坏死果蝇 DNA 的方案。
摘要
总共选择了 5 个大 头金鸡 孢菌样品 - 3 个新鲜样品,1 个样品在酒精中储存 2 年,1 个样品在干燥密封储存中避光保存 2 年 - 以研究血液 DNA 提取试剂盒是否可以从嗜坏蝇中提取 DNA,并确定酒精是否可以延长嗜坏蝇 DNA 的保存时间。首先,使用血液 DNA 提取试剂盒从他们的胸部组织中提取 DNA。然后,使用酶标仪和荧光计检查 DNA 纯度和浓度。最后,使用位于线粒体 CO I 基因序列中的嗜坏苍蝇特异性引物对提取的 DNA 进行 PCR 扩增和电泳。结果表明,所有样品的 DNA 纯度均大于 2.0。观察到 DNA 浓度按以下顺序排列:新鲜样品>酒精保存的旧样品>未经处理的旧样品。PCR 扩增后所有样品均具有特异性电泳条带。综上所述,血液 DNA 提取试剂盒可用于成功提取嗜坏死果蝇的 DNA,且鲜果蝇样本的 DNA 浓度大于老果蝇样本。苍蝇可以长时间储存在酒精中。
引言
死亡时间的推论一直是司法实践中需要解决的关键和难点问题之一。在法医学实践中,对于刑事案件,确定验尸间隔对于推断犯罪时间、锁定嫌疑人、缩小侦查范围起着至关重要的作用。传统的推断死亡时间的方法主要基于早期的死后现象,通常只能用于推断 24 小时内的死亡时间。然而,死亡时间的长短不能由死后现象来确定。现在,法医科学界普遍认为,法医昆虫学有可能成为推断较长死亡时间的有效方法。
Necrophagous 昆虫以其以尸体为食的幼虫而得名。其中,每当有尸体出现时,成年嗜尸蝇就会第一个到达尸体并产卵或幼虫。幼虫以尸体组织为食,成熟为蛹,蛹长成成虫。嗜尸蝇由于其有规律的生命周期和地理分布,在法医学实践中发挥着巨大的作用,例如,扣除死亡时间并确定死亡地点 1,2。然而,在法医实践中使用嗜尸蝇的障碍是物种鉴定。形态学方法仍被用作嗜坏死果蝇物种鉴定的权威方法。然而,形态学物种鉴定需要高度的昆虫完整性。如果果蝇处于不同的发育阶段,或者由于环境选择引起的形态变化,这些都会使形态学检查更加困难。特别是,最常见于犯罪现场的蛹和蛹壳的形态几乎无法辨认。这一点,再加上形态学专家的稀缺,使物种的形态学鉴定变得非常困难。因此,出现了分子生物学方法对果蝇物种鉴定的应用,它降低了形态学鉴定的难度,并且独立于发育阶段 3,4,5,6,7。
基于分子生物学方法对嗜坏死果蝇进行物种鉴定的第一步是高效提取 DNA,而目前不常用用于昆虫 DNA 提取的专用试剂盒。如何使用普通血液试剂盒提取嗜坏死果蝇的 DNA 变得更具法医意义。在犯罪现场发现的嗜尸蝇通常被肢解或经历腐烂和 DNA 降解。果蝇样品在采集后不能立即用于 DNA 提取,或者由于证据保存要求,如果可能,需要保留完整的样品。基于上述要求,本研究采用基于蛋白酶K消化的血液DNA试剂盒,选取3个新鲜 的大头金丝菌(双翅目、鳞翅目) 样本(图1),1个大 头念珠 菌老样本在酒精中放置两年,1个巨 头念珠 菌样本在避光保存中放置两年,对5个样本各称重, 选择了昆虫胸部组织进行 DNA 提取。比较从新旧样品中提取的 DNA 的质量和纯度,使用位于线粒体 CO 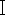 基因序列中的嗜坏苍蝇特异性引物对提取的 DNA 进行 PCR 扩增和电泳。
基因序列中的嗜坏苍蝇特异性引物对提取的 DNA 进行 PCR 扩增和电泳。
研究方案
注意:本方案共使用了 5 个 C. megacephala 样品 - 3 个新鲜样品(新鲜 1、新鲜 2 和新鲜 3),1 个样品在酒精中储存 2 年(Old 1),1 个样品在干燥密封储存中保存 2 年仅避光(Old 2)。样品必须根据实验要求进行标记。
1. 一般样品储存和制备
- 样品选择和制备
注意:使用乙酸乙酯处决坏死蝇时,请务必在通风橱中工作。称重前,从果蝇表面去除液体。- 新鲜样品:用乙酸乙酯杀死后,将收集的三个苍蝇样品放入气瓶中。称量样品并将其命名为 Fresh 1、Fresh 2 和 Fresh 3,并立即在实验中使用。
注:该方案中的新鲜果蝇样品质量为 56.8 mg、49.3 mg 和 52.1 mg。 - 旧样品 1:取出被乙酸乙酯杀死的果蝇样品,并在充满酒精的离心管中密闭保存 2 年,在室温下避光保存。用滤纸吸附表面液体后称取样品;称之为 Old 1。
注意:在该方案中,Old 1 的质量为 42.3 mg。 - 旧样品 2:取出被乙酸乙酯杀死的果蝇样品,并在离心管中密闭保存 2 年,在室温下避光保存。称重并称其为 Old 2。
- 新鲜样品:用乙酸乙酯杀死后,将收集的三个苍蝇样品放入气瓶中。称量样品并将其命名为 Fresh 1、Fresh 2 和 Fresh 3,并立即在实验中使用。
- 提取前的准备
- 加入少量液氮冷却不锈钢容器;等到所有液氮都蒸发完毕。
- 将苍蝇样本放入容器中;缓慢倒入液氮,以防止液氮飞溅并影响昆虫样品。
- 将样品冷冻在液氮中。液氮蒸发后取出样品。
- 单独分离胸部,放入 2 mL 离心管中,切成尽可能小的切片。
注:将样品切成小块,使其可以完全裂解,并且可以大量释放 DNA。 - 对每只果蝇重复上述步骤,单独冷冻,然后转移到新的离心管中,以防止 DNA 交叉污染。
2. DNA 提取
注:所有离心步骤必须在室温 (15-25 °C) 下在微量离心机中进行。所有步骤都必须严格遵守无菌原则并避免交叉污染。
- 组织裂解
- 将 180 μL 组织裂解缓冲液添加到含有夹住样品的 2 mL 离心管中。
- 加入 20 μL 蛋白酶 K。以 3 000 rpm 涡旋 10 秒充分混合,并在 56 °C 下孵育直至昆虫组织完全裂解。
注意:如果组织未完全消化,则应添加更多蛋白酶 K。裂解通常在 1-3 小时内完成,也可延长裂解时间过夜。
- DNA 沉淀
- 以 3,000 rpm 的速度涡旋 15 秒。向样品中加入 200 μL 裂解缓冲液,并以 3,000 rpm 涡旋 15 秒充分混合。
- 加入 200 μL 乙醇 (96-100%),并以 3,000 rpm 涡旋 15 秒,再次充分混合。
注:裂解缓冲液和乙醇可以一步预添加在一起,以节省时间。加入裂解缓冲液和乙醇后可能会形成白色沉淀。
- 将混合物移液到放置在 2 mL 收集管中的 DNA 吸附柱中。以 6,000 × g 离心 1 分钟。丢弃流通管和收集管。
- 将上一步的 DNA 吸附柱放入新的 2 mL 收集管中,加入 500 μL 蛋白质去除缓冲液,并以 6,000 × g 离心 1 分钟。丢弃流通管和收集管。
- 将 DNA 吸附柱转移到新的 2 mL 收集管中,加入 500 μL 脱盐缓冲液,并以 20,000 × g 离心 3 分钟以干燥 DNA 吸附柱膜。丢弃流通管和收集管。
注:此离心步骤可确保在下一次洗脱之前去除任何残留的乙醇。如果有任何乙醇残留,再次以 20,000 × g 离心 1 分钟。 - 将 DNA 吸附柱置于干净的 2 mL 微量离心管中,直接将 100 μL DNA 洗脱缓冲液吸至柱膜上,室温孵育 1 分钟,然后以 6,000 × g 离心 1 分钟洗脱。
注:用 100 μL(而不是 200 μL)洗脱会增加洗脱液中的最终 DNA 浓度,但也会降低 DNA 总产量。为了最大限度地提高 DNA 产量,请按照步骤 2.8 中的说明重复洗脱一次。 - 将 DNA 样品储存在 4 °C(长达 1 周)或 -80 °C 下进行长期储存。
3. DNA 浓度检测
- 通过酶标仪确定 OD 260 和 OD 280 的值,以比较 DNA 纯度和核酸浓度。对每个样品重复 3 次。
- 使用其配套试剂盒通过荧光计测定提取的 DNA 的浓度,每个样品重复 3 次。
- 取 190 μL 工作溶液放入两个 0.5 mL 离心管中,取标准 #1 和标准 #2 各 10 μL,加入工作溶液中,避光放置不少于 15 分钟。
注:标准品 #1 和标准品 #2 针对一定浓度的 DNA 进行了精确校准。应准确配置标准溶液,以确保形成准确的标准曲线。标准 #1 是 0.2 ng/μL 双链 DNA (dsDNA) 样品;标准 #2 为 2,000 ng/μL。工作溶液是一种与 dsDNA 特异性结合的染料。 - 取 2 μL 检测 DNA,在 0.5 mL 离心管中与 198 μL 工作溶液混合,避光不少于 15 分钟。
注:如果 DNA 浓度低,则添加 5 μL 测试 DNA 和 195 μL 工作溶液。 - 15 分钟后,打开仪器并选择 dsDNA 浓度测量 |长距离。首先,测试标准 #1,然后测试标准 #2 以获得标准曲线。
注意:标准溶液必须在配置后的同一天使用;放置时间过长会导致标准曲线出现较大的误差。 - 将待测样品逐一放入检测孔中,将 DNA 加标体积调节至 2 μL,重复测量 3 次,取平均值,记录。
- 取 190 μL 工作溶液放入两个 0.5 mL 离心管中,取标准 #1 和标准 #2 各 10 μL,加入工作溶液中,避光放置不少于 15 分钟。
4. PCR 和电泳检测
注:取长度为 278 bp 的线粒体 CO 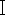 基因序列,用正向引物 C1-J-2495:CAGCTACTTTATGAGCTTTAGG,反向引物 C1-N-2800:CATTTCAAGCTGTGTAAGCATC8 进行 PCR 扩增,然后进行 2% 琼脂糖凝胶电泳,验证扩增效果和扩增产物的长度。
基因序列,用正向引物 C1-J-2495:CAGCTACTTTATGAGCTTTAGG,反向引物 C1-N-2800:CATTTCAAGCTGTGTAAGCATC8 进行 PCR 扩增,然后进行 2% 琼脂糖凝胶电泳,验证扩增效果和扩增产物的长度。
- 通过添加 10 μL 2x Taq 混合物、1 μL 每种引物、5 μL ddH2O 和 3 μL 模板 DNA,构建 20 μL PCR 扩增系统;在离心机中充分混合 10 秒。
- 使用以下PCR循环参数:预变性95°C,10分钟;变性 95 °C 持续 30 秒,退火 48 °C 持续 30 秒,延伸 72 °C 持续 45 秒,共 40 个循环;最终延伸 72 °C 10 分钟。
- 将 PCR 后的产物进行 2% 琼脂糖凝胶电泳,电泳后,将它们放在凝胶成像系统上进行光分析。
结果
我们使用酶标仪测量提取的 DNA 溶液的 OD260 和 OD280 值,然后获得 OD260/OD280 值以评估 DNA 的纯度。所有新鲜样品和旧样品 1 的 OD260/OD280 均大于 2。旧样品 2 的 OD260/OD280 的最大值为 2.187,尽管平均值为 1.753,并且由于其 OD280 和 OD280 的值都较小 (≤0.01),其三次测量波动很大(表 1)。根据获得的 OD260 值,我们估计了 DNA 的浓度。三种样品的 DNA 浓度依次为:新鲜样品>旧样品 1 >旧样品 2...
讨论
DNA 提取是嗜坏死果蝇分子鉴定中最关键的环节,其提取方法和提取的 DNA 质量直接影响后续检测。在本实验中,我们使用普通的血液试剂盒成功地从嗜坏死的果蝇中提取了 DNA,而无需购买特殊的昆虫 DNA 提取试剂盒,这使得昆虫 DNA 提取更容易完成。
果蝇表面富含几丁质,尤其是在幼虫和蛹期,表皮几丁质可达 60-70%,这使得嗜坏死果蝇的 DNA 提取变得困难9?...
披露声明
作者声明他们没有相互竞争的经济利益。
致谢
这项工作得到了中国国家自然科学基金 (82060341,81560304) 和海南省院士创新平台科学研究项目 (YSPTZX202134) 的支持。
材料
| Name | Company | Catalog Number | Comments |
| Buffer AE | Qiagen | 172026832 | DNA elution buffer |
| Buffer AL | Qiagen | 172028374 | lysis buffer |
| Buffer ATL | Qiagen | 172028162 | tissue lysis buffer |
| Buffer AW1 | Qiagen | 172028760 | protein removal buffer |
| Buffer AW2 | Qiagen | 57203108 | desalination buffer |
| C1-J-2495 | Taihe Biotechnology | TW21109216 | forword primer |
| C1-N-2800 | Taihe Biotechnology | TW21109217 | reverse primer |
| D2000 DNA ladder | Real-Times(Beijing) Biotechnology | RTM415 | Measure the position of electrophoretic bands |
| DNeasy Mini spin column | Qiagen | 166050343 | DNA adsorption column |
| Dry Bath Incubator | Miulab | DKT200-2D | used for heating |
| MIX-30S Mini Mixer | Miulab | MUC881206 | oscillatory action |
| Proteinase K | Qiagen | 172026218 | Inactivation of intracellular nucleases and other proteins |
| Qubit 3.0 Fluorometer | Thermo Fisher Scientific | 2321611188 | |
| Speed Micro-Centrifuge | Scilogex | 9013001121 | centrifuge |
| Standard#1 | Thermo Fisher Scientific | 2342797 | |
| Standard#2 | Thermo Fisher Scientific | 2342797 | |
| Tanon 3500R Gel Imager | Tanon | 16T5553R-455 | gel imaging |
| Taq Mix Pro | Monad | 00007808-140534 | PCR Mix |
| Taq Mix Pro | Monad | 00007808-140534 | PCR Mix |
| Thermo Cycler | Zhuhai Hema | VRB020A | ordinary PCR |
| Working Solution | Thermo Fisher Scientific | 2342797 |
参考文献
- Amendt, J., Richards, C. S., Campobasso, C. P., Zehner, R., Hall, M. J. Forensic entomology: applications and limitations. Forensic Sci Med Pathol. 7 (4), 379-392 (2011).
- Wydra, J., Matuszewski, S. The optimal post-eclosion interval while estimating the post-mortem interval based on an empty puparium. Forensic Sci Med Pathol. 17 (2), 192-198 (2021).
- Mazzanti, M., Alessandrini, F., Tagliabracci, A., Wells, J. D., Campobasso, C. P. DNA degradation and genetic analysis of empty puparia: genetic identification limits in forensic entomology. Forensic Sci Int. 195 (1-3), 99-102 (2010).
- Odeniran, P. O., et al. molecular identification and distribution of Trypanosome-transmitting Dipterans from cattle settlements in Southwest Nigeria. Acta Parasitol. 66 (1), 116-128 (2021).
- Wells, J. D., Stevens, J. R. Application of DNA-based methods in forensic entomology. Annu Rev Entomol. 53, 103-120 (2008).
- Kotrba, M., et al. DNA barcoding in forensic entomology-establishing a DNA reference library of potentially forensic relevant arthropod species. J Forensic Sci. 64 (2), 593-601 (2019).
- Weedon, M. N., et al. Use of SNP chips to detect rare pathogenic variants: Retrospective, population based diagnostic evaluation. BMJ. 372, 214 (2021).
- Cai, J. -. F., et al. The availability of mitochondrial DNA cytochrome oxidasegene for the distinction of forensically important flies in China. Acta Entomologica Sinica. 48 (3), 380-385 (2005).
- Khayrova, A., Lopatin, S., Varlamov, V. Obtaining chitin, chitosan and their melanin complexes from insects. Int J Biol Macromol. 167, 1319-1328 (2021).
- Guo, Y. Q., et al. Comparison of three DNA extracting methods from different parts of common necrophagous flies. J Henan Univ Sci Tech (Med Sci). 36 (02), 130-133 (2018).
- Du, H., Xie, H., Ma, M., Igarashi, Y., Luo, F. Modified methods obtain high-quality DNA and RNA from anaerobic activated sludge at a wide range of temperatures. J Microbiol Methods. 199, 106532 (2022).
- Sukumaran, S. Concentration determination of nucleic acids and proteins using the micro-volume BioSpec-nano-spectrophotometer. J Vis Exp. (48), e2699 (2011).
- Masago, K., et al. Comparison between fluorimetry (Qubit) and spectrophotometry (NanoDrop) in the quantification of DNA and RNA extracted from frozen and FFPE tissues from lung cancer patients: A real-world use of genomic tests. Medicina (Kaunas). 57 (12), 1375 (2021).
- Ponti, G., et al. The value of fluorimetry (Qubit) and spectrophotometry (NanoDrop) in the quantification of cell-free DNA (cfDNA) in malignant melanoma and prostate cancer patients. Clin Chim Acta. 479, 14-19 (2018).
- Adams, Z. J., Hall, M. J. Methods used for the killing and preservation of blowfly larvae, and their effect on post-mortem larval length. Forensic Sci Int. 138 (1-3), 50-61 (2003).
- Fliegerova, K., et al. Effect of DNA extraction and sample preservation method on rumen bacterial population. Anaerobe. 29, 80-84 (2014).
- Rodrigues, B. L., Galati, E. A. B. Molecular taxonomy of phlebotomine sand flies (Diptera, Psychodidae) with emphasis on DNA barcoding: A review. Acta Trop. 238, 106778 (2023).
Erratum
Formal Correction: Erratum: DNA Extraction and Comparison Between Old and Fresh Necrophilic Fly Samples
Posted by JoVE Editors on 8/12/2024. Citeable Link.
This corrects the article 10.3791/66737
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。