É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Extração de DNA e comparação entre amostras de moscas necrófilas antigas e frescas
* Estes autores contribuíram igualmente
Neste Artigo
Erratum Notice
Resumo
Este artigo descreve um protocolo para extração de DNA de mosca necrófila fresca e velha usando um kit de extração de DNA comum modificado.
Resumo
Um total de cinco amostras de amostras de Chrysomya megacephala - três amostras frescas, uma amostra armazenada em álcool por 2 anos e uma amostra armazenada em armazenamento selado a seco por 2 anos protegida apenas da luz - foram selecionadas para investigar se um kit de extração de DNA sanguíneo poderia extrair DNA de moscas necrófilas e para determinar se o álcool poderia prolongar a preservação do DNA das moscas necrófilas. Primeiro, o kit de extração de DNA do sangue foi usado para extrair DNA de seus tecidos do tórax. Em seguida, a pureza e a concentração do DNA foram examinadas usando um leitor de microplacas e um fluorômetro. Finalmente, a amplificação por PCR e a eletroforese do DNA extraído foram feitas com primers necrófilos específicos para moscas localizados na sequência mitocondrial do gene CO I. Os resultados mostraram que a pureza do DNA de todas as amostras foi superior a 2,0. Observou-se que a concentração de DNA era da seguinte ordem: amostras frescas > amostras antigas preservadas em álcool > amostras antigas não tratadas. Todas as amostras apresentaram bandas eletroforéticas específicas após a amplificação por PCR. Em conclusão, um kit de extração de DNA sanguíneo pode ser usado para extrair DNA de moscas necrófilas com sucesso, e a concentração de DNA de amostras de moscas frescas é maior do que a de amostras de moscas antigas. As moscas podem ser armazenadas em álcool por muito tempo.
Introdução
A inferência da hora da morte sempre foi uma das questões-chave e difíceis de serem resolvidas na prática judicial. Na prática da ciência forense, para um caso criminal, determinar o intervalo post-mortem desempenha um papel crucial na dedução da hora do crime, prendendo o suspeito e estreitando o escopo da investigação. Os métodos tradicionais de inferir a hora da morte são baseados principalmente em fenômenos post-mortem precoces, que geralmente só podem ser usados para inferir a hora da morte em 24 horas. No entanto, o longo tempo de morte não pode ser determinado por fenômenos post-mortem. Agora é geralmente reconhecido na comunidade de ciências forenses que a entomologia forense tem o potencial de ser um método eficaz de inferir um tempo mais longo de morte.
Os insetos necrófagos são nomeados por suas larvas que se alimentam de cadáveres. Entre eles, sempre que um cadáver estiver presente, as moscas necrófilas adultas serão as primeiras a chegar ao cadáver e colocar seus ovos ou larvas. As larvas se alimentam de tecido do cadáver e amadurecem em pupas, que se transformam em adultos. As moscas necrófilas desempenham um papel importante na prática da ciência forense devido ao seu ciclo de vida regular e distribuição geográfica, por exemplo, deduzindo a hora da morte e determinando o local da morte 1,2. No entanto, a barreira para o uso de moscas necrófilas na prática forense é a identificação de espécies. Os métodos morfológicos ainda são usados como método oficial para a identificação de espécies de moscas necrófilas. No entanto, a identificação morfológica das espécies requer um alto grau de integridade do inseto. Se as moscas estavam em diferentes estágios de desenvolvimento, ou devido a mudanças morfológicas causadas por escolhas ambientais, tudo isso dificulta o exame morfológico. Em particular, a morfologia das pupas e conchas de pupas, que são mais frequentemente encontradas na cena do crime, é quase irreconhecível. Isso, juntamente com a escassez de especialistas morfológicos, dificulta enormemente a identificação morfológica das espécies. Portanto, surgiu a aplicação de métodos de biologia molecular para identificação de espécies de moscas, o que reduz a dificuldade de identificação morfológica e é independente do estágio de desenvolvimento 3,4,5,6,7.
O primeiro passo na identificação de espécies de moscas necrófilas com base em métodos de biologia molecular é a extração eficiente de DNA, enquanto kits especializados para extração de DNA de insetos não estão comumente disponíveis atualmente. E como usar kits de sangue comuns para extração de DNA de moscas necrófilas torna-se mais relevante forense. As moscas necrófilas encontradas em cenas de crime são frequentemente mutiladas ou sofreram decomposição e degradação do DNA. As amostras de moscas não estão imediatamente disponíveis para extração de DNA após a aquisição ou, se possível, amostras completas precisam ser retidas devido aos requisitos de preservação de evidências. Com base nos requisitos acima, neste estudo, aplicamos o kit de DNA sanguíneo baseado na digestão da proteinase K e selecionamos três amostras frescas de Chrysomya megacephala (Diptera, Lepidoptera) (Figura 1), uma amostra antiga de C. megacephala colocada em álcool por dois anos e uma amostra de C. megacephala colocada em armazenamento protegido contra luz por dois anos, pesando cada uma das cinco amostras, elegeu o tecido do tórax do inseto para extração de DNA. Comparando a qualidade e pureza do DNA extraído das amostras antigas e novas, a amplificação por PCR e a eletroforese do DNA extraído foram realizadas com primers necrófilos específicos para moscas localizados na sequência do gene CO 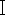 mitocondrial.
mitocondrial.
Protocolo
NOTA: Um total de cinco amostras de amostras de C. megacephala foram usadas neste protocolo - três amostras frescas (Fresh 1, Fresh 2 e Fresh 3), uma amostra armazenada em álcool por 2 anos (Old 1) e uma amostra armazenada em armazenamento selado a seco por 2 anos protegida apenas da luz (Old 2). As amostras devem ser rotuladas de acordo com os requisitos experimentais.
1. Armazenamento e preparações gerais de amostras
- Seleção e preparação de amostras
NOTA: Certifique-se de trabalhar na hotte ao aplicar acetato de etila para executar moscas necrófagas. Remova o líquido da superfície da mosca antes de pesar.- Amostras frescas: Coloque três amostras de moscas coletadas em frascos de gás depois de matá-las com acetato de etila. Pese e nomeie as amostras como Fresh 1, Fresh 2 e Fresh 3 e use-as imediatamente em experimentos.
NOTA: As amostras de moscas frescas neste protocolo tinham massas de 56,8 mg, 49,3 mg e 52,1 mg. - Amostra antiga 1: Retirar amostras de moscas mortas por acetato de etilo e armazenadas em tubos de centrífuga cheios de álcool durante 2 anos em contenção hermética, protegidas da luz à temperatura ambiente. Pesar a amostra após adsorver o líquido da superfície com papel de filtro; chame-o de Velho 1.
NOTA: A massa de Old 1 foi de 42,3 mg neste protocolo. - Amostra antiga 2: Retirar a amostra de mosca morta por acetato de etilo e armazenada num tubo de centrifugação durante 2 anos em contenção hermética, protegida da luz à temperatura ambiente. Pese e chame de Velho 2.
- Amostras frescas: Coloque três amostras de moscas coletadas em frascos de gás depois de matá-las com acetato de etila. Pese e nomeie as amostras como Fresh 1, Fresh 2 e Fresh 3 e use-as imediatamente em experimentos.
- Preparação antes da extração
- Adicione uma pequena quantidade de nitrogênio líquido para resfriar um recipiente de aço inoxidável; Espere até que todo o nitrogênio líquido tenha evaporado.
- Coloque uma amostra de mosca no recipiente; Despeje o nitrogênio líquido lentamente para evitar que o nitrogênio líquido respingue e impacte a amostra de inseto.
- Congele as amostras em nitrogênio líquido. Remova as amostras após a evaporação do nitrogênio líquido.
- Separe o tórax individualmente, coloque em um tubo de centrífuga de 2 mL e corte nas menores fatias possíveis.
NOTA: Corte a amostra em pedaços pequenos para que possa ser totalmente clivada e o DNA possa ser liberado em grandes quantidades. - Repita as etapas acima para cada mosca, congele individualmente e remova para um novo tubo de centrífuga para evitar a contaminação cruzada do DNA.
2. Extração de DNA
NOTA: Todas as etapas de centrifugação devem ser realizadas à temperatura ambiente (15-25 °C) em uma microcentrífuga. Todas as etapas devem ser executadas em estrita conformidade com os princípios da assepsia e para evitar contaminação cruzada.
- Lise tecidual
- Adicione 180 μL de tampão de lise tecidual a um tubo de centrífuga de 2 mL contendo uma amostra cortada.
- Adicionar 20 μl de proteinase K. Homogeneizar por vórtice a 3 000 rpm durante 10 s e incubar a 56 °C até que os tecidos dos insectos estejam completamente lisados.
NOTA: Adicione mais Proteinase K se o tecido não for totalmente digerido. A lise geralmente é concluída em 1-3h, também pode estender o tempo de lise durante a noite.
- Precipitação de DNA
- Vórtice a 3.000 rpm por 15 s. Adicione 200 μL de tampão de lise à amostra e misture bem por vórtice a 3.000 rpm por 15 s.
- Adicione 200 μL de etanol (96-100%) e misture novamente bem por vórtice a 3.000 rpm por 15 s.
NOTA: O tampão de lise e o etanol podem ser pré-adicionados juntos em uma única etapa para economizar tempo. Um precipitado branco pode se formar com a adição de tampão de lise e etanol.
- Pipetar a mistura para a coluna de adsorção de ADN colocada num tubo de colheita de 2 ml. Centrifugue a 6.000 × g por 1 min. Descarte o fluxo e o tubo de coleta.
- Coloque a coluna de adsorção de DNA da etapa anterior em um novo tubo de coleta de 2 mL, adicione 500 μL de tampão de remoção de proteína e centrifugue a 6.000 × g por 1 min. Descarte o fluxo e o tubo de coleta.
- Transfira a coluna de adsorção de DNA para um novo tubo de coleta de 2 mL, adicione 500 μL de tampão de dessalinização e centrifugue a 20.000 × g por 3 min para secar a membrana da coluna de adsorção de DNA. Descarte o fluxo e o tubo de coleta.
NOTA: Esta etapa de centrifugação garante a remoção de qualquer etanol residual antes da eluição seguinte. Se houver algum transporte de etanol, centrifugue novamente a 20.000 × g por 1 min. - Coloque a coluna de adsorção de DNA em um tubo de microcentrífuga limpo de 2 mL, pipete diretamente 100 μL de tampão de eluição de DNA na membrana da coluna, incube em temperatura ambiente por 1 min e, em seguida, centrifugue a 6.000 × g por 1 min para eluir.
NOTA: A eluição com 100 μL (em vez de 200 μL) aumenta a concentração final de DNA no eluato, mas também diminui o rendimento geral do DNA. Para maximizar o rendimento do DNA, repita a eluição uma vez, conforme descrito na etapa 2.8. - Armazene as amostras de DNA a 4 °C (até 1 semana) ou -80 °C para armazenamento de longo prazo.
3. Detecção de concentração de DNA
- Determine os valores de OD 260 e OD 280 para comparar a pureza do DNA e a concentração de ácido nucleico pelo leitor de microplacas. Repita três vezes em cada amostra.
- Determinar a concentração do ADN extraído por fluorómetro utilizando o seu kit complementar, com três repetições para cada amostra.
- Pegue 190 μL de solução de trabalho em dois tubos de centrífuga de 0,5 mL, pegue 10 μL de cada Padrão # 1 e Padrão # 2, adicione-os às soluções de trabalho e mantenha-os longe da luz por pelo menos 15 min.
NOTA: O padrão # 1 e o padrão # 2 são calibrados com precisão para uma determinada concentração de DNA. As soluções padrão devem ser configuradas com precisão para garantir a formação de uma curva padrão precisa. O padrão # 1 é uma amostra de DNA de fita dupla (dsDNA) de 0,2 ng/μL; o padrão #2 é de 2.000 ng/μL. A solução de trabalho é um corante que se liga especificamente ao dsDNA. - Pegue 2 μL de DNA de teste e misture com 198 μL de solução de trabalho em um tubo de centrífuga de 0,5 mL e mantenha longe da luz por pelo menos 15 min.
NOTA: Se a concentração de DNA for baixa, adicione 5 μL de DNA de teste com 195 μL de solução de trabalho. - Após 15 min, ligue o instrumento e selecione Medição de concentração de dsDNA | Longo alcance. Primeiro, teste o Padrão #1 e, em seguida, o Padrão #2 para obter a curva padrão.
NOTA: A solução padrão deve ser usada no mesmo dia após sua configuração; deixá-lo por muito tempo resultará em um grande erro na curva padrão. - Coloque as amostras a serem testadas nos poços de ensaio uma a uma, ajuste o volume de pico de DNA para 2 μL, repita a medição três vezes, pegue o valor médio e registre.
- Pegue 190 μL de solução de trabalho em dois tubos de centrífuga de 0,5 mL, pegue 10 μL de cada Padrão # 1 e Padrão # 2, adicione-os às soluções de trabalho e mantenha-os longe da luz por pelo menos 15 min.
4. PCR e detecção eletroforética
NOTA: A sequência do gene CO 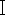 mitocondrial com comprimento de 278 pb foi obtida para amplificação por PCR com primer direto C1-J-2495: CAGCTACTTTATGAGCTTTAGG e primer reverso C1-N-2800: CATTTCAAGCTGTGTAAGCATC8 e, em seguida, eletroforese em gel de agarose a 2% feita para verificar o efeito de amplificação e o comprimento dos produtos de amplificação foi verificado.
mitocondrial com comprimento de 278 pb foi obtida para amplificação por PCR com primer direto C1-J-2495: CAGCTACTTTATGAGCTTTAGG e primer reverso C1-N-2800: CATTTCAAGCTGTGTAAGCATC8 e, em seguida, eletroforese em gel de agarose a 2% feita para verificar o efeito de amplificação e o comprimento dos produtos de amplificação foi verificado.
- Configure 20 μL do sistema de amplificação de PCR adicionando 10 μL de mistura 2x Taq, 1 μL de cada primer, 5 μL de ddH2O e 3 μL de DNA molde; Misture bem por 10 s em uma centrífuga.
- Use os seguintes parâmetros de ciclagem de PCR: pré-desnaturação 95 °C por 10 min; desnaturação a 95 °C durante 30 s, recozimento a 48 °C durante 30 s, extensão a 72 °C durante 45 s num total de 40 ciclos; e extensão final 72 °C por 10 min.
- Submeta os produtos após a PCR à eletroforese em gel de agarose a 2% e, após a eletroforese, coloque-os em um sistema de imagem em gel para análise fotográfica.
Resultados
Utilizamos um leitor de microplacas para medir os valores de OD260 e OD280 da solução de DNA extraída e, em seguida, obtivemos os valores de OD260/OD280 para avaliar a pureza do DNA. O OD260/OD280 de todas as amostras frescas e da amostra antiga 1 foi maior que 2. O OD260/OD280 da antiga amostra 2 teve um valor máximo de 2,187, embora a média tenha sido de 1,753, e suas três medidas flutuaram muito devido aos pequenos valores (≤0,01) de OD280 e OD280 (Tabela 1). Com base no valor de OD260 obtido,...
Discussão
A extração de DNA é o elo mais crítico na identificação molecular de moscas necrófilas, e seu método de extração e a qualidade do DNA extraído afetam diretamente a detecção subsequente. Neste experimento, extraímos com sucesso o DNA de moscas necrófilas usando um kit de sangue comum, sem a necessidade de comprar um kit especial de extração de DNA de inseto, o que facilita a extração de DNA de inseto.
A superfície das moscas é rica em quitina, principalmente nos estágios l...
Divulgações
Os autores declaram que não têm interesses financeiros concorrentes.
Agradecimentos
Este trabalho é apoiado pela Fundação Nacional de Ciências Naturais da China (82060341,81560304) e pelo Projeto de Pesquisa Científica da Plataforma de Inovação Acadêmica da Província de Hainan (YSPTZX202134).
Materiais
| Name | Company | Catalog Number | Comments |
| Buffer AE | Qiagen | 172026832 | DNA elution buffer |
| Buffer AL | Qiagen | 172028374 | lysis buffer |
| Buffer ATL | Qiagen | 172028162 | tissue lysis buffer |
| Buffer AW1 | Qiagen | 172028760 | protein removal buffer |
| Buffer AW2 | Qiagen | 57203108 | desalination buffer |
| C1-J-2495 | Taihe Biotechnology | TW21109216 | forword primer |
| C1-N-2800 | Taihe Biotechnology | TW21109217 | reverse primer |
| D2000 DNA ladder | Real-Times(Beijing) Biotechnology | RTM415 | Measure the position of electrophoretic bands |
| DNeasy Mini spin column | Qiagen | 166050343 | DNA adsorption column |
| Dry Bath Incubator | Miulab | DKT200-2D | used for heating |
| MIX-30S Mini Mixer | Miulab | MUC881206 | oscillatory action |
| Proteinase K | Qiagen | 172026218 | Inactivation of intracellular nucleases and other proteins |
| Qubit 3.0 Fluorometer | Thermo Fisher Scientific | 2321611188 | |
| Speed Micro-Centrifuge | Scilogex | 9013001121 | centrifuge |
| Standard#1 | Thermo Fisher Scientific | 2342797 | |
| Standard#2 | Thermo Fisher Scientific | 2342797 | |
| Tanon 3500R Gel Imager | Tanon | 16T5553R-455 | gel imaging |
| Taq Mix Pro | Monad | 00007808-140534 | PCR Mix |
| Taq Mix Pro | Monad | 00007808-140534 | PCR Mix |
| Thermo Cycler | Zhuhai Hema | VRB020A | ordinary PCR |
| Working Solution | Thermo Fisher Scientific | 2342797 |
Referências
- Amendt, J., Richards, C. S., Campobasso, C. P., Zehner, R., Hall, M. J. Forensic entomology: applications and limitations. Forensic Sci Med Pathol. 7 (4), 379-392 (2011).
- Wydra, J., Matuszewski, S. The optimal post-eclosion interval while estimating the post-mortem interval based on an empty puparium. Forensic Sci Med Pathol. 17 (2), 192-198 (2021).
- Mazzanti, M., Alessandrini, F., Tagliabracci, A., Wells, J. D., Campobasso, C. P. DNA degradation and genetic analysis of empty puparia: genetic identification limits in forensic entomology. Forensic Sci Int. 195 (1-3), 99-102 (2010).
- Odeniran, P. O., et al. molecular identification and distribution of Trypanosome-transmitting Dipterans from cattle settlements in Southwest Nigeria. Acta Parasitol. 66 (1), 116-128 (2021).
- Wells, J. D., Stevens, J. R. Application of DNA-based methods in forensic entomology. Annu Rev Entomol. 53, 103-120 (2008).
- Kotrba, M., et al. DNA barcoding in forensic entomology-establishing a DNA reference library of potentially forensic relevant arthropod species. J Forensic Sci. 64 (2), 593-601 (2019).
- Weedon, M. N., et al. Use of SNP chips to detect rare pathogenic variants: Retrospective, population based diagnostic evaluation. BMJ. 372, 214 (2021).
- Cai, J. -. F., et al. The availability of mitochondrial DNA cytochrome oxidasegene for the distinction of forensically important flies in China. Acta Entomologica Sinica. 48 (3), 380-385 (2005).
- Khayrova, A., Lopatin, S., Varlamov, V. Obtaining chitin, chitosan and their melanin complexes from insects. Int J Biol Macromol. 167, 1319-1328 (2021).
- Guo, Y. Q., et al. Comparison of three DNA extracting methods from different parts of common necrophagous flies. J Henan Univ Sci Tech (Med Sci). 36 (02), 130-133 (2018).
- Du, H., Xie, H., Ma, M., Igarashi, Y., Luo, F. Modified methods obtain high-quality DNA and RNA from anaerobic activated sludge at a wide range of temperatures. J Microbiol Methods. 199, 106532 (2022).
- Sukumaran, S. Concentration determination of nucleic acids and proteins using the micro-volume BioSpec-nano-spectrophotometer. J Vis Exp. (48), e2699 (2011).
- Masago, K., et al. Comparison between fluorimetry (Qubit) and spectrophotometry (NanoDrop) in the quantification of DNA and RNA extracted from frozen and FFPE tissues from lung cancer patients: A real-world use of genomic tests. Medicina (Kaunas). 57 (12), 1375 (2021).
- Ponti, G., et al. The value of fluorimetry (Qubit) and spectrophotometry (NanoDrop) in the quantification of cell-free DNA (cfDNA) in malignant melanoma and prostate cancer patients. Clin Chim Acta. 479, 14-19 (2018).
- Adams, Z. J., Hall, M. J. Methods used for the killing and preservation of blowfly larvae, and their effect on post-mortem larval length. Forensic Sci Int. 138 (1-3), 50-61 (2003).
- Fliegerova, K., et al. Effect of DNA extraction and sample preservation method on rumen bacterial population. Anaerobe. 29, 80-84 (2014).
- Rodrigues, B. L., Galati, E. A. B. Molecular taxonomy of phlebotomine sand flies (Diptera, Psychodidae) with emphasis on DNA barcoding: A review. Acta Trop. 238, 106778 (2023).
Erratum
Formal Correction: Erratum: DNA Extraction and Comparison Between Old and Fresh Necrophilic Fly Samples
Posted by JoVE Editors on 8/12/2024. Citeable Link.
This corrects the article 10.3791/66737
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados