このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
DNA抽出と古い屍好性ハエサンプルと新鮮なハエサンプルの比較
* これらの著者は同等に貢献しました
Erratum Notice
要約
この記事では、改変された一般的なDNA抽出キットを使用した、新鮮で古い屍好性ハエDNA抽出のプロトコルについて説明します。
要約
Chrysomya megacephalaのサンプルの合計5つのサンプル(新鮮なサンプル3つ、アルコールで2年間保存されたサンプル、および光のみから保護された乾燥密閉されたストレージで2年間保存されたサンプル)が選択されました。まず、血液DNA抽出キットを使用して、胸部組織からDNAを抽出しました。次に、DNAの純度と濃度をマイクロプレートリーダーと蛍光光度計を用いて調べました。最後に、抽出されたDNAのPCR増幅および電気泳動を、ミトコンドリアのCO I遺伝子配列に位置する屍好性のハエ特異的プライマーを用いて行った。その結果、すべてのサンプルのDNA純度が2.0より大きいことが示されました。DNA濃度は、新しいサンプル>アルコール保存された古いサンプル、>未処理の古いサンプルの順であることが観察されました。すべてのサンプルは、PCR増幅後に特定の電気泳動バンドを持っていました。結論として、血液DNA抽出キットを使用して屍親死性のハエからDNAを成功裏に抽出でき、新鮮なハエサンプルのDNA濃度は古いハエサンプルのDNA濃度よりも高くなります。ハエはアルコールに長期間保存できます。
概要
死亡時刻の推定は、常に司法実務において解決すべき重要かつ困難な問題の1つでした。法医学の実践では、刑事事件の場合、死後の間隔を決定することは、犯罪の時間を推測し、容疑者を固定し、捜査の範囲を狭める上で重要な役割を果たします。死亡時刻を推定する従来の方法は、主に死後早期の現象に基づいており、一般的には24時間以内の死亡時刻を推測するためにのみ使用できます。しかし、死の長い時間は死後の現象によって決定することはできません。現在、法医学界では、法医学昆虫学が死亡のより長い時間を推測する効果的な方法になる可能性があることが一般的に認識されています。
屍食性昆虫は、死体を食べる幼虫にちなんで名付けられました。その中で、死体が存在するときはいつでも、成虫の屍屍ハエが最初に死体に到達し、卵や幼虫を産みます。幼虫は死体組織を食べて成熟し、蛹になり、蛹は成虫に巣立ちます。屍親死性のハエは、その定期的なライフサイクルと地理的分布、たとえば死亡時刻を差し引いて死亡場所を決定するため、法医学の実践において大きな役割を果たします1,2。しかし、法医学の実践における屍姦ハエの使用に対する障壁は、種の識別です。形態学的方法は、屍姦ハエの種同定のための権威ある方法として今でも使用されています。しかし、形態学的種の同定には、高度な昆虫の完全性が必要です。ハエが異なる発達段階にあったり、環境の選択によって引き起こされた形態学的変化によるものであったりすると、それらすべてが形態学的調査をより困難にします。特に、犯罪現場で最も頻繁に見られる蛹と蛹の貝殻の形態は、ほとんど認識できません。これは、形態学の専門家の不足と相まって、形態学的に種を特定することを非常に困難にしています。したがって、ハエの種同定のための分子生物学的方法の適用が出現し、形態学的同定の困難さを軽減し、発生段階3,4,5,6,7とは無関係です。
分子生物学的手法に基づく屍屑性ハエの種同定の最初のステップは、DNAの効率的な抽出ですが、昆虫のDNA抽出のための専用キットは現在一般的に利用できません。また、屍親死性ハエのDNA抽出に一般的な血液キットをどのように使用するかは、法医学的に重要になります。犯罪現場で発見された屍姦性のハエは、しばしば切断されたり、腐敗やDNAの劣化を受けたりしています。ハエのサンプルは、取得後すぐにDNA抽出に利用できるわけではなく、証拠保存の要件により、可能であれば完全なサンプルを保持する必要があります。以上の要件に基づき、本研究では、プロテイナーゼK消化に基づく血液DNAキットを適用し、3つの新鮮な Chrysomya megacephala(双翅目、鱗翅目) サンプル(図1)、1つの古い C.megacephala サンプルをアルコールに2年間保存し、1つの C.megacephala サンプルを2年間光保護保存し、5つのサンプルのそれぞれを秤量しました。 昆虫の胸部組織をDNA抽出のために選出しました。新旧のサンプルから抽出したDNAの品質と純度を比較し、抽出したDNAのPCR増幅と電気泳動を、ミトコンドリアのCO 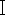 遺伝子配列に位置するハエ特異的なネクロフィリックプライマーを用いて行いました。
遺伝子配列に位置するハエ特異的なネクロフィリックプライマーを用いて行いました。
プロトコル
注:このプロトコルでは、 C. megacephala サンプルの合計5つのサンプルを使用しました-3つの新鮮なサンプル(フレッシュ1、フレッシュ2、フレッシュ3)、1つのサンプルをアルコールに2年間保存(古い1)、および1つのサンプルを光からのみ保護された2年間の乾燥密封保存(古い2)で保存しました。サンプルは、実験要件に従ってラベル付けする必要があります。
1. 一般的なサンプルの保管と調製
- サンプルの選択と調製
注:酢酸エチルを適用してネクロファガスフライを実行するときは、必ずヒュームフードで作業してください。計量する前に、フライの表面から液体を取り除きます。- 新鮮なサンプル:採取した3つのハエのサンプルを酢酸エチルで殺した後、ガスバイアルに入れます。サンプルの重量を量り、Fresh 1、Fresh 2、Fresh 3 と名前を付けて、すぐに実験に使用します。
注:このプロトコルの新鮮なハエサンプルの質量は、56.8 mg、49.3 mg、および52.1 mgでした。 - 古いサンプル1:酢酸エチルによって死滅したハエのサンプルを取り出し、アルコールを充填した遠心分離管に2年間保管し、室温で光から保護された密閉容器で保管します。表面の液体を濾紙で吸着した後、サンプルを計量します。それを古い1と呼んでください。
注:このプロトコルでは、Old 1の質量は42.3 mgでした。. - 古いサンプル2:酢酸エチルによって死滅したハエのサンプルを取り出し、室温で光から保護された密閉容器で2年間遠心分離管に保管します。重さを量り、それを古い2と呼びます。
- 新鮮なサンプル:採取した3つのハエのサンプルを酢酸エチルで殺した後、ガスバイアルに入れます。サンプルの重量を量り、Fresh 1、Fresh 2、Fresh 3 と名前を付けて、すぐに実験に使用します。
- 抽出前の準備
- 少量の液体窒素を加えて、ステンレス鋼の容器を冷却します。すべての液体窒素が蒸発するまで待ちます。
- ハエのサンプルを容器に入れます。液体窒素が飛散して昆虫サンプルに影響を与えるのを防ぐために、液体窒素をゆっくりと注ぎます。
- サンプルを液体窒素で凍結します。液体窒素が蒸発した後、サンプルを取り出します。
- 胸部を個別に分離し、2mLの遠心分離チューブに入れ、可能な限り小さなスライスにカットします。
注:サンプルを完全に切断し、DNAを大量に放出できるように、サンプルを細かく切ります。 - 各フライについて上記の手順を繰り返し、個別に凍結してから、DNAの交差汚染を防ぐために新しい遠心分離チューブに移します。
2. DNA抽出
注:すべての遠心分離ステップは、マイクロ遠心分離機で室温(15〜25°C)で実行する必要があります。すべてのステップは、アセプシスの原則を厳守し、相互汚染を避けるために実行する必要があります。
- 組織溶解
- 180 μLの組織溶解バッファーを、クリップしたサンプルが入った2 mLの遠心チューブに加えます。
- 20 μLのプロテイナーゼKを加え、3,000 rpmで10秒間ボルテックスして十分に混合し、昆虫組織が完全に溶解するまで56°Cでインキュベートします。
注:組織が完全に消化されていない場合は、プロテイナーゼKを追加する必要があります。溶解は通常1〜3時間で完了し、一晩で溶解時間を延長することもできます。
- DNA沈殿物
- 3,000 rpmで15秒間渦を巻きます。200 μLの溶解バッファーをサンプルに加え、3,000 rpmで15秒間ボルテックスして十分に混合します。
- 200 μLのエタノール(96-100%)を加え、3,000 rpmで15秒間ボルテックスして再度十分に混合します。
注:溶解バッファーとエタノールは、時間を節約するために1つのステップで一緒に事前に添加できます。溶解緩衝液とエタノールを添加すると、白色の沈殿物が形成されることがあります。
- 混合物を2 mLの収集チューブに入れたDNA吸着カラムにピペットで移します。6,000 × g で1分間遠心分離します。フロースルーと収集チューブを廃棄します。
- 前のステップで得たDNA吸着カラムを新しい2 mLコレクションチューブに入れ、500 μLのタンパク質除去バッファーを加え、6,000 × g で1分間遠心分離します。フロースルーと収集チューブを廃棄します。
- DNA吸着カラムを新しい2 mLコレクションチューブに移し、500 μLの淡水化緩衝液を加え、20,000 × g で3分間遠心分離してDNA吸着カラムメンブレムを乾燥させます。フロースルーと収集チューブを廃棄します。
注:この遠心分離ステップにより、次の溶出前に残留エタノールが確実に除去されます。エタノールのキャリーオーバーがある場合は、再度20,000 × g で1分間遠心分離します。 - DNA吸着カラムを清潔な2 mLの微量遠心チューブに入れ、100 μLのDNA溶出バッファーをカラムメンブレンに直接ピペットで移し、室温で1分間インキュベートした後、6,000 g× g で1分間遠心分離して溶出させます。
注:100 μL (200 μL ではなく) で溶出すると、溶出液中の最終 DNA 濃度は増加しますが、全体的な DNA 収量も減少します。DNA収量を最大化するには、ステップ2.8で説明したように溶出を1回繰り返します。 - DNAサンプルは4°C(最大1週間)または-80°Cで長期保存してください。
3. DNA濃度検出
- OD 260 と OD 280 の値を決定し、マイクロプレートリーダーで DNA 純度と核酸濃度を比較します。各サンプルで3回繰り返します。
- 抽出したDNAの濃度は、蛍光光度計のコンパニオンキットを使用し、サンプルごとに3回繰り返して測定します。
- 190 μLの作業溶液を2本の0.5 mL遠心分離チューブに取り、標準#1と標準#2をそれぞれ10 μL取り、それらを作業溶液に加えて、15分以上光から遠ざけます。
注:標準#1および標準#2は、特定の濃度のDNAに対して正確に較正されています。標準溶液は、正確な標準曲線の形成を確実にするために、正確に構成する必要があります。標準品#1は、0.2 ng/μLの二本鎖DNA(dsDNA)サンプルです。標準#2は2,000 ng/μLです。ワーキングソリューションは、dsDNAに特異的に結合する色素です。 - 2 μLの試験DNAを採取し、0.5 mLの遠心チューブで198 μLの作業溶液と混合し、15分以上光を避けてください。
注:DNA濃度が低い場合は、5 μLの試験DNAと195 μLの作業溶液を加えます。 - 15分後、装置の電源を入れ、[ dsDNA Concentration Measurement |ロングレンジ。まず、標準#1をテストし、次に標準#2をテストして、標準曲線を取得します。
注:標準ソリューションは、構成後と同じ日に使用する必要があります。長時間放置すると、標準曲線に大きな誤差が生じます。 - 試験するサンプルを1つずつアッセイウェルに入れ、DNAのスパイク量を2μLに調整し、測定を3回繰り返して平均値を取り、記録します。
- 190 μLの作業溶液を2本の0.5 mL遠心分離チューブに取り、標準#1と標準#2をそれぞれ10 μL取り、それらを作業溶液に加えて、15分以上光から遠ざけます。
4. PCRおよび電気泳動検出
注:278 bpの長さのミトコンドリアCO 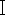 遺伝子配列を、フォワードプライマーC1-J-2495:CAGCTACTTTATGAGCTTTAGGおよびリバースプライマーC1-N-2800:CATTTCAAGCTGTAAGCATC8でPCR増幅するために採取し、その後、増幅効果と増幅製品の長さを確認するために2%アガロースゲル電気泳動を行いました。
遺伝子配列を、フォワードプライマーC1-J-2495:CAGCTACTTTATGAGCTTTAGGおよびリバースプライマーC1-N-2800:CATTTCAAGCTGTAAGCATC8でPCR増幅するために採取し、その後、増幅効果と増幅製品の長さを確認するために2%アガロースゲル電気泳動を行いました。
- 20 μL の 2x Taq ミックス 10 μL、各プライマー 1 μL、5 μL の ddH2O、3 μL のテンプレート DNA を添加して、20 μL の PCR 増幅システムをセットアップします。遠心分離機で10秒間よく混合します。
- 以下のPCRサイクリングパラメータを使用してください:predenationを95°Cで10分間;変性95°Cで30秒、アニーリング48°Cで30秒、伸長72°Cで45秒、合計40サイクル。最終延長は72°Cで10分間です。
- PCR後の産物を2%アガロースゲル電気泳動にかけ、電気泳動後、ゲルイメージングシステム上に置き、光分析を行います。
結果
抽出したDNA溶液のOD260とOD280の値をマイクロプレートリーダーで測定し、OD260/OD280の値を求めてDNAの純度を評価しました。すべての新しいサンプルと古いサンプル1のOD260/OD280は2より大きかった。旧サンプル2のOD260/OD280は、平均値が1.753であるのに対し、最大値は2.187であり、OD280とOD280ともに≤0.01という小さ(表1)により、3つの測定値が大きく変動しました。得られたOD260の値をもと?...
ディスカッション
DNA抽出は、屠姦バエの分子同定における最も重要なリンクであり、その抽出方法と抽出されたDNAの品質は、その後の検出に直接影響します。この実験では、昆虫のDNA抽出キットを特別な購入することなく、一般的な血液キットを使用して屍妄のハエからDNAを抽出することに成功しました。
ハエの表面はキチン質が豊富で、特に幼虫と蛹の段階では表皮のキチンが60〜70%に...
開示事項
著者らは、競合する金銭的利益がないことを宣言します。
謝辞
この研究は、中国国家自然科学基金会(82060341,81560304)および海南省のアカデミシャンイノベーションプラットフォーム科学研究プロジェクト(YSPTZX202134)の支援を受けています。
資料
| Name | Company | Catalog Number | Comments |
| Buffer AE | Qiagen | 172026832 | DNA elution buffer |
| Buffer AL | Qiagen | 172028374 | lysis buffer |
| Buffer ATL | Qiagen | 172028162 | tissue lysis buffer |
| Buffer AW1 | Qiagen | 172028760 | protein removal buffer |
| Buffer AW2 | Qiagen | 57203108 | desalination buffer |
| C1-J-2495 | Taihe Biotechnology | TW21109216 | forword primer |
| C1-N-2800 | Taihe Biotechnology | TW21109217 | reverse primer |
| D2000 DNA ladder | Real-Times(Beijing) Biotechnology | RTM415 | Measure the position of electrophoretic bands |
| DNeasy Mini spin column | Qiagen | 166050343 | DNA adsorption column |
| Dry Bath Incubator | Miulab | DKT200-2D | used for heating |
| MIX-30S Mini Mixer | Miulab | MUC881206 | oscillatory action |
| Proteinase K | Qiagen | 172026218 | Inactivation of intracellular nucleases and other proteins |
| Qubit 3.0 Fluorometer | Thermo Fisher Scientific | 2321611188 | |
| Speed Micro-Centrifuge | Scilogex | 9013001121 | centrifuge |
| Standard#1 | Thermo Fisher Scientific | 2342797 | |
| Standard#2 | Thermo Fisher Scientific | 2342797 | |
| Tanon 3500R Gel Imager | Tanon | 16T5553R-455 | gel imaging |
| Taq Mix Pro | Monad | 00007808-140534 | PCR Mix |
| Taq Mix Pro | Monad | 00007808-140534 | PCR Mix |
| Thermo Cycler | Zhuhai Hema | VRB020A | ordinary PCR |
| Working Solution | Thermo Fisher Scientific | 2342797 |
参考文献
- Amendt, J., Richards, C. S., Campobasso, C. P., Zehner, R., Hall, M. J. Forensic entomology: applications and limitations. Forensic Sci Med Pathol. 7 (4), 379-392 (2011).
- Wydra, J., Matuszewski, S. The optimal post-eclosion interval while estimating the post-mortem interval based on an empty puparium. Forensic Sci Med Pathol. 17 (2), 192-198 (2021).
- Mazzanti, M., Alessandrini, F., Tagliabracci, A., Wells, J. D., Campobasso, C. P. DNA degradation and genetic analysis of empty puparia: genetic identification limits in forensic entomology. Forensic Sci Int. 195 (1-3), 99-102 (2010).
- Odeniran, P. O., et al. molecular identification and distribution of Trypanosome-transmitting Dipterans from cattle settlements in Southwest Nigeria. Acta Parasitol. 66 (1), 116-128 (2021).
- Wells, J. D., Stevens, J. R. Application of DNA-based methods in forensic entomology. Annu Rev Entomol. 53, 103-120 (2008).
- Kotrba, M., et al. DNA barcoding in forensic entomology-establishing a DNA reference library of potentially forensic relevant arthropod species. J Forensic Sci. 64 (2), 593-601 (2019).
- Weedon, M. N., et al. Use of SNP chips to detect rare pathogenic variants: Retrospective, population based diagnostic evaluation. BMJ. 372, 214 (2021).
- Cai, J. -. F., et al. The availability of mitochondrial DNA cytochrome oxidasegene for the distinction of forensically important flies in China. Acta Entomologica Sinica. 48 (3), 380-385 (2005).
- Khayrova, A., Lopatin, S., Varlamov, V. Obtaining chitin, chitosan and their melanin complexes from insects. Int J Biol Macromol. 167, 1319-1328 (2021).
- Guo, Y. Q., et al. Comparison of three DNA extracting methods from different parts of common necrophagous flies. J Henan Univ Sci Tech (Med Sci). 36 (02), 130-133 (2018).
- Du, H., Xie, H., Ma, M., Igarashi, Y., Luo, F. Modified methods obtain high-quality DNA and RNA from anaerobic activated sludge at a wide range of temperatures. J Microbiol Methods. 199, 106532 (2022).
- Sukumaran, S. Concentration determination of nucleic acids and proteins using the micro-volume BioSpec-nano-spectrophotometer. J Vis Exp. (48), e2699 (2011).
- Masago, K., et al. Comparison between fluorimetry (Qubit) and spectrophotometry (NanoDrop) in the quantification of DNA and RNA extracted from frozen and FFPE tissues from lung cancer patients: A real-world use of genomic tests. Medicina (Kaunas). 57 (12), 1375 (2021).
- Ponti, G., et al. The value of fluorimetry (Qubit) and spectrophotometry (NanoDrop) in the quantification of cell-free DNA (cfDNA) in malignant melanoma and prostate cancer patients. Clin Chim Acta. 479, 14-19 (2018).
- Adams, Z. J., Hall, M. J. Methods used for the killing and preservation of blowfly larvae, and their effect on post-mortem larval length. Forensic Sci Int. 138 (1-3), 50-61 (2003).
- Fliegerova, K., et al. Effect of DNA extraction and sample preservation method on rumen bacterial population. Anaerobe. 29, 80-84 (2014).
- Rodrigues, B. L., Galati, E. A. B. Molecular taxonomy of phlebotomine sand flies (Diptera, Psychodidae) with emphasis on DNA barcoding: A review. Acta Trop. 238, 106778 (2023).
Erratum
Formal Correction: Erratum: DNA Extraction and Comparison Between Old and Fresh Necrophilic Fly Samples
Posted by JoVE Editors on 8/12/2024. Citeable Link.
This corrects the article 10.3791/66737
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved