Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Extraction d’ADN et comparaison entre des échantillons de mouches nécrophiles anciennes et fraîches
* Ces auteurs ont contribué à parts égales
Dans cet article
Erratum Notice
Résumé
Cet article décrit un protocole pour l’extraction de l’ADN de mouches nécrophiles fraîches et anciennes à l’aide d’un kit d’extraction d’ADN commun modifié.
Résumé
Au total, cinq échantillons d’échantillons de Chrysomya megacephala - trois échantillons frais, un échantillon stocké dans de l’alcool pendant 2 ans et un échantillon stocké dans un stockage scellé à sec pendant 2 ans à l’abri de la lumière seulement - ont été sélectionnés pour déterminer si un kit d’extraction d’ADN sanguin pouvait extraire de l’ADN de mouches nécrophiles et pour déterminer si l’alcool pouvait prolonger la conservation de l’ADN des mouches nécrophiles. Tout d’abord, le kit d’extraction d’ADN sanguin a été utilisé pour extraire l’ADN des tissus de leur thorax. Ensuite, la pureté et la concentration de l’ADN ont été examinées à l’aide d’un lecteur de microplaques et d’un fluorimètre. Enfin, l’amplification par PCR et l’électrophorèse de l’ADN extrait ont été réalisées avec des amorces spécifiques à la mouche nécrophile situées dans la séquence du gène mitochondrial CO I. Les résultats ont montré que la pureté de l’ADN de tous les échantillons était supérieure à 2,0. On a observé que la concentration d’ADN était de l’ordre suivant : des échantillons frais > des échantillons anciens conservés dans de l’alcool > des échantillons anciens non traités. Tous les échantillons présentaient des bandes électrophorétiques spécifiques après amplification par PCR. En conclusion, un kit d’extraction d’ADN sanguin peut être utilisé pour extraire avec succès l’ADN des mouches nécrophiles, et la concentration d’ADN des échantillons de mouches fraîches est supérieure à celle des anciens échantillons de mouches. Les mouches peuvent être stockées dans l’alcool pendant une longue période.
Introduction
L’inférence de l’heure du décès a toujours été l’une des questions clés et difficiles à résoudre dans la pratique judiciaire. Dans la pratique de la science médico-légale, pour une affaire criminelle, la détermination de l’intervalle post-mortem joue un rôle crucial dans la déduction de l’heure du crime, l’enfermement du suspect et la réduction de la portée de l’enquête. Les méthodes traditionnelles d’inférence de l’heure du décès sont basées principalement sur des phénomènes d’autopsie précoces, qui ne peuvent généralement être utilisés que pour déduire l’heure du décès dans les 24 heures. Cependant, la longue durée de la mort ne peut pas être déterminée par des phénomènes post-mortem. Il est maintenant généralement reconnu dans la communauté des sciences judiciaires que l’entomologie médico-légale a le potentiel d’être une méthode efficace pour déduire une durée de décès plus longue.
Les insectes nécrophages doivent leur nom à leurs larves qui se nourrissent de cadavres. Parmi elles, chaque fois qu’un cadavre est présent, les mouches nécrophiles adultes seront les premières à atteindre le cadavre et à pondre leurs œufs ou larves. Les larves se nourrissent de tissus cadavres et deviennent des nymphes, qui deviennent des adultes. Les mouches nécrophiles jouent un rôle énorme dans la pratique de la science médico-légale en raison de leur cycle de vie régulier et de leur distribution géographique, par exemple, en déduisant l’heure du décès et en déterminant le lieu du décès 1,2. Cependant, l’obstacle à l’utilisation de mouches nécrophiles dans la pratique médico-légale est l’identification des espèces. Les méthodes morphologiques sont toujours utilisées comme méthode faisant autorité pour l’identification des espèces de mouches nécrophiles. Cependant, l’identification morphologique des espèces nécessite un haut degré d’intégrité des insectes. Si les mouches étaient à des stades de développement différents, ou en raison de changements morphologiques causés par des choix environnementaux, tout cela rend l’examen morphologique plus difficile. En particulier, la morphologie des pupes et des coquilles de nymphes, que l’on trouve le plus souvent sur les lieux du crime, est à peine reconnaissable. Ceci, associé à la rareté des experts en morphologie, rend extrêmement difficile l’identification morphologique des espèces. Par conséquent, l’application de méthodes de biologie moléculaire pour l’identification des espèces de mouches a émergé, ce qui réduit la difficulté de l’identification morphologique et est indépendant des stadesde développement 3, 4, 5, 6, 7.
La première étape de l’identification des espèces de mouches nécrophiles basée sur des méthodes de biologie moléculaire est l’extraction efficace de l’ADN, tandis que des kits spécialisés pour l’extraction de l’ADN des insectes ne sont pas couramment disponibles actuellement. Et la façon d’utiliser les kits de sang courants pour l’extraction de l’ADN des mouches nécrophiles devient plus pertinente sur le plan médico-légal. Les mouches nécrophiles trouvées sur les scènes de crime sont souvent mutilées ou ont subi une décomposition et une dégradation de l’ADN. Les échantillons de mouches ne sont pas immédiatement disponibles pour l’extraction de l’ADN après l’acquisition ou, si possible, des échantillons complets doivent être conservés en raison des exigences de préservation des preuves. Sur la base des exigences ci-dessus, dans cette étude, nous avons appliqué le kit d’ADN sanguin basé sur la digestion à la protéinase K, et sélectionné trois échantillons frais de Chrysomya megacephala (Diptères, Lépidoptères) (Figure 1), un échantillon ancien de C. megacephala placé dans de l’alcool pendant deux ans, et un échantillon de C. megacephala placé dans un stockage à l’abri de la lumière pendant deux ans, pesé chacun des cinq échantillons, choisi le tissu du thorax d’insecte pour l’extraction de l’ADN. En comparant la qualité et la pureté de l’ADN extrait de l’ancien et du nouvel échantillon, l’amplification par PCR et l’électrophorèse de l’ADN extrait ont été réalisées avec des amorces spécifiques à la mouche nécrophile situées dans la séquence du gène CO 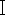 mitochondrial.
mitochondrial.
Protocole
REMARQUE : Au total, cinq échantillons de C. megacephala ont été utilisés dans le cadre de ce protocole : trois échantillons frais (frais 1, frais 2 et frais 3), un échantillon conservé dans de l’alcool pendant 2 ans (ancien 1) et un échantillon stocké dans un endroit sec scellé pendant 2 ans, à l’abri de la lumière seulement (ancien 2). Les échantillons doivent être étiquetés conformément aux exigences expérimentales.
1. Stockage et préparations généraux des échantillons
- Sélection et préparation des échantillons
REMARQUE : Assurez-vous de travailler dans la hotte lorsque vous appliquez de l’acétate d’éthyle pour exécuter les mouches nécrophages. Retirez le liquide de la surface de la mouche avant de la peser.- Échantillons frais : Placez trois échantillons de mouches collectés dans des flacons de gaz après les avoir tués avec de l’acétate d’éthyle. Pesez et nommez les échantillons comme Fresh 1, Fresh 2 et Fresh 3 et utilisez-les immédiatement dans les expériences.
REMARQUE : Les échantillons de mouches fraîches de ce protocole présentaient des masses de 56,8 mg, 49,3 mg et 52,1 mg. - Ancien échantillon 1 : Retirer les échantillons de mouches tués par l’acétate d’éthyle et stockés dans des tubes à centrifuger remplis d’alcool pendant 2 ans dans un confinement hermétique, à l’abri de la lumière à température ambiante. Peser l’échantillon après avoir adsorbé le liquide de surface avec du papier filtre ; appelez-le Vieux 1.
REMARQUE : La masse de Old 1 était de 42,3 mg dans ce protocole. - Ancien échantillon 2 : Retirer l’échantillon de mouches tué par l’acétate d’éthyle et stocké dans un tube à centrifuger pendant 2 ans dans un confinement hermétique, à l’abri de la lumière à température ambiante. Pesez et appelez-le Old 2.
- Échantillons frais : Placez trois échantillons de mouches collectés dans des flacons de gaz après les avoir tués avec de l’acétate d’éthyle. Pesez et nommez les échantillons comme Fresh 1, Fresh 2 et Fresh 3 et utilisez-les immédiatement dans les expériences.
- Préparation avant l’extraction
- Ajouter une petite quantité d’azote liquide pour refroidir un récipient en acier inoxydable ; Attendez que tout l’azote liquide se soit évaporé.
- Mettez un échantillon de mouches dans le récipient ; Versez lentement l’azote liquide pour éviter que l’azote liquide n’éclabousse et n’impacte l’échantillon d’insecte.
- Congelez les échantillons dans de l’azote liquide. Prélever les échantillons après l’évaporation de l’azote liquide.
- Séparez le thorax individuellement, placez-le dans un tube à centrifuger de 2 ml et coupez-le en tranches les plus petites possibles.
REMARQUE : Coupez l’échantillon en petits morceaux afin qu’il puisse être entièrement clivé et que l’ADN puisse être libéré en grande quantité. - Répétez les étapes ci-dessus pour chaque mouche, congelez-la individuellement, puis retirez-la dans un nouveau tube à centrifuger pour éviter la contamination croisée de l’ADN.
2. Extraction de l’ADN
REMARQUE : Toutes les étapes de centrifugation doivent être effectuées à température ambiante (15-25 °C) dans une microcentrifugeuse. Toutes les étapes doivent être effectuées dans le strict respect des principes de l’asepsie et pour éviter la contamination croisée.
- Lyse tissulaire
- Ajouter 180 μL de tampon de lyse tissulaire dans un tube à centrifuger de 2 mL contenant un échantillon coupé.
- Ajouter 20 μL de protéinase K. Bien mélanger par vortex à 3 000 tr/min pendant 10 s et incuber à 56 °C jusqu’à ce que les tissus de l’insecte soient complètement lysés.
REMARQUE : Ajouter plus de protéinase K devrait être si le tissu n’est pas complètement digéré. La lyse est généralement terminée en 1 à 3 heures, ce qui peut également prolonger le temps de lyse pendant la nuit.
- Précipitation de l’ADN
- Vortex à 3 000 tr/min pendant 15 s. Ajoutez un tampon de lyse de 200 μL à l’échantillon et mélangez soigneusement par vortex à 3 000 tr/min pendant 15 s.
- Ajouter 200 μL d’éthanol (96-100 %) et bien mélanger à nouveau par vortex à 3 000 tr/min pendant 15 s.
REMARQUE : Le tampon de lyse et l’éthanol peuvent être préajoutés ensemble en une seule étape pour gagner du temps. Un précipité blanc peut se former lors de l’ajout d’un tampon de lyse et d’éthanol.
- Pipeter le mélange dans la colonne d’adsorption d’ADN placée dans un tube de prélèvement de 2 ml. Centrifugeuse à 6 000 × g pendant 1 min. Jeter le tube d’écoulement et le tube de collecte.
- Placez la colonne d’adsorption d’ADN de l’étape précédente dans un nouveau tube de collecte de 2 ml, ajoutez 500 μL de tampon d’élimination des protéines et centrifugez à 6 000 × g pendant 1 min. Jeter le tube d’écoulement et le tube de collecte.
- Transférez la colonne d’adsorption de l’ADN dans un nouveau tube de collecte de 2 ml, ajoutez 500 μL de tampon de dessalement et centrifugez à 20 000 × g pendant 3 minutes pour sécher la membrane de la colonne d’adsorption de l’ADN. Jeter le tube d’écoulement et le tube de collecte.
REMARQUE : Cette étape de centrifugation assure l’élimination de tout résidu d’éthanol avant l’élution suivante. S’il y a un transfert d’éthanol, centrifuger à nouveau à 20 000 × g pendant 1 min. - Placez la colonne d’adsorption d’ADN dans un tube de microcentrifugation propre de 2 ml, pipetez directement 100 μL de tampon d’élution d’ADN sur la membrane de la colonne, incubez à température ambiante pendant 1 min, puis centrifugez à 6 000 × g pendant 1 min pour éluer.
REMARQUE : L’élution avec 100 μL (au lieu de 200 μL) augmente la concentration finale d’ADN dans l’éluat, mais diminue également le rendement global en ADN. Pour maximiser le rendement en ADN, répétez l’élution une fois comme décrit à l’étape 2.8. - Conservez les échantillons d’ADN à 4 °C (jusqu’à 1 semaine) ou à -80 °C pour un stockage à long terme.
3. Détection de la concentration d’ADN
- Déterminer les valeurs de OD 260 et OD 280 pour comparer la pureté de l’ADN et la concentration en acides nucléiques par lecteur de microplaques. Répétez l’opération trois fois sur chaque échantillon.
- Déterminez la concentration de l’ADN extrait à l’aide d’un fluoromètre à l’aide de son kit compagnon, avec trois répétitions pour chaque échantillon.
- Prenez 190 μL de solution de travail dans deux tubes à centrifuger de 0,5 mL, prenez 10 μL chacun de l’Étalon #1 et de l’Étalon #2, ajoutez-les aux solutions de travail et conservez-les à l’abri de la lumière pendant au moins 15 min.
REMARQUE : L’étalon #1 et l’étalon #2 sont étalonnés avec précision pour une certaine concentration d’ADN. Les solutions standard doivent être configurées avec précision pour assurer la formation d’une courbe standard précise. L’étalon #1 est un échantillon d’ADN double brin (ADNdb) de 0,2 ng/μL ; la norme #2 est de 2 000 ng/μL. La solution de travail est un colorant qui se lie spécifiquement à l’ADNdb. - Prélever 2 μL d’ADN d’essai et les mélanger avec 198 μL de solution de travail dans un tube à centrifuger de 0,5 mL, et les tenir à l’abri de la lumière pendant au moins 15 min.
REMARQUE : Si la concentration d’ADN est faible, ajouter 5 μL d’ADN d’essai avec 195 μL de solution de travail. - Après 15 min, allumez l’instrument et sélectionnez Mesure de la concentration d’ADNdb | Longue portée. Tout d’abord, testez la norme #1, puis la norme #2 pour obtenir la courbe standard.
REMARQUE : La solution standard doit être utilisée le jour même de sa configuration ; Le laisser trop longtemps entraînera une erreur importante dans la courbe standard. - Placez les échantillons à tester dans les puits de dosage un par un, ajustez le volume de dopage d’ADN à 2 μL, répétez la mesure trois fois, prenez la valeur moyenne et enregistrez.
- Prenez 190 μL de solution de travail dans deux tubes à centrifuger de 0,5 mL, prenez 10 μL chacun de l’Étalon #1 et de l’Étalon #2, ajoutez-les aux solutions de travail et conservez-les à l’abri de la lumière pendant au moins 15 min.
4. Détection PCR et électrophorétique
REMARQUE : La séquence du gène CO 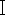 mitochondrial d’une longueur de 278 pb a été prélevée pour l’amplification par PCR avec l’amorce directe C1-J-2495 : CAGCTACTTTATGAGCTTTAGG, et l’amorce inverse C1-N-2800 : CATTTCAAGCTGTGTAAGCATC8, puis l’électrophorèse sur gel d’agarose à 2 % a été effectuée pour vérifier l’effet d’amplification et la longueur des produits d’amplification a été vérifiée.
mitochondrial d’une longueur de 278 pb a été prélevée pour l’amplification par PCR avec l’amorce directe C1-J-2495 : CAGCTACTTTATGAGCTTTAGG, et l’amorce inverse C1-N-2800 : CATTTCAAGCTGTGTAAGCATC8, puis l’électrophorèse sur gel d’agarose à 2 % a été effectuée pour vérifier l’effet d’amplification et la longueur des produits d’amplification a été vérifiée.
- Mettre en place 20 μL du système d’amplification PCR en ajoutant 10 μL de mélange 2x Taq, 1 μL de chaque amorce, 5 μL de ddH2O et 3 μL d’ADN matrice ; Bien mélanger pendant 10 s dans une centrifugeuse.
- Utilisez les paramètres de cycle PCR suivants : prédénaturation 95 °C pendant 10 min ; dénaturation 95 °C pendant 30 s, recuit 48 °C pendant 30 s, extension 72 °C pendant 45 s pour un total de 40 cycles ; et extension finale à 72 °C pendant 10 min.
- Soumettre les produits après PCR à une électrophorèse sur gel d’agarose à 2 %, et après électrophorèse, les placer sur un système d’imagerie sur gel pour l’analyse photo.
Résultats
Nous avons utilisé un lecteur de microplaques pour mesurer les valeurs de OD260 et OD280 de la solution d’ADN extraite, puis nous avons obtenu les valeurs OD260/OD280 pour évaluer la pureté de l’ADN. La DO 260/OD280 de tous les échantillons frais et de l’ancien échantillon 1 était supérieure à 2. Les OD260/OD280 de l’ancien échantillon 2 avaient une valeur maximale de 2,187 bien que la moyenne soit de 1,753, et ses trois mesures ont beaucoup fluctué en raison des petites valeurs (≤0,01) de ses OD280 e...
Discussion
L’extraction de l’ADN est le maillon le plus critique dans l’identification moléculaire des mouches nécrophiles, et sa méthode d’extraction et la qualité de l’ADN extrait affectent directement la détection ultérieure. Dans cette expérience, nous avons réussi à extraire de l’ADN de mouches nécrophiles en utilisant un kit de sang commun, sans avoir besoin d’acheter un kit spécial d’extraction d’ADN d’insecte, ce qui rend l’extraction de l’ADN d’insecte plus facile à réaliser.
Déclarations de divulgation
Les auteurs déclarent qu’ils n’ont pas d’intérêts financiers concurrents.
Remerciements
Ce travail est soutenu par la Fondation nationale des sciences naturelles de Chine (82060341,81560304) et par le projet de recherche scientifique de la plate-forme d’innovation académique de la province de Hainan (YSPTZX202134).
matériels
| Name | Company | Catalog Number | Comments |
| Buffer AE | Qiagen | 172026832 | DNA elution buffer |
| Buffer AL | Qiagen | 172028374 | lysis buffer |
| Buffer ATL | Qiagen | 172028162 | tissue lysis buffer |
| Buffer AW1 | Qiagen | 172028760 | protein removal buffer |
| Buffer AW2 | Qiagen | 57203108 | desalination buffer |
| C1-J-2495 | Taihe Biotechnology | TW21109216 | forword primer |
| C1-N-2800 | Taihe Biotechnology | TW21109217 | reverse primer |
| D2000 DNA ladder | Real-Times(Beijing) Biotechnology | RTM415 | Measure the position of electrophoretic bands |
| DNeasy Mini spin column | Qiagen | 166050343 | DNA adsorption column |
| Dry Bath Incubator | Miulab | DKT200-2D | used for heating |
| MIX-30S Mini Mixer | Miulab | MUC881206 | oscillatory action |
| Proteinase K | Qiagen | 172026218 | Inactivation of intracellular nucleases and other proteins |
| Qubit 3.0 Fluorometer | Thermo Fisher Scientific | 2321611188 | |
| Speed Micro-Centrifuge | Scilogex | 9013001121 | centrifuge |
| Standard#1 | Thermo Fisher Scientific | 2342797 | |
| Standard#2 | Thermo Fisher Scientific | 2342797 | |
| Tanon 3500R Gel Imager | Tanon | 16T5553R-455 | gel imaging |
| Taq Mix Pro | Monad | 00007808-140534 | PCR Mix |
| Taq Mix Pro | Monad | 00007808-140534 | PCR Mix |
| Thermo Cycler | Zhuhai Hema | VRB020A | ordinary PCR |
| Working Solution | Thermo Fisher Scientific | 2342797 |
Références
- Amendt, J., Richards, C. S., Campobasso, C. P., Zehner, R., Hall, M. J. Forensic entomology: applications and limitations. Forensic Sci Med Pathol. 7 (4), 379-392 (2011).
- Wydra, J., Matuszewski, S. The optimal post-eclosion interval while estimating the post-mortem interval based on an empty puparium. Forensic Sci Med Pathol. 17 (2), 192-198 (2021).
- Mazzanti, M., Alessandrini, F., Tagliabracci, A., Wells, J. D., Campobasso, C. P. DNA degradation and genetic analysis of empty puparia: genetic identification limits in forensic entomology. Forensic Sci Int. 195 (1-3), 99-102 (2010).
- Odeniran, P. O., et al. molecular identification and distribution of Trypanosome-transmitting Dipterans from cattle settlements in Southwest Nigeria. Acta Parasitol. 66 (1), 116-128 (2021).
- Wells, J. D., Stevens, J. R. Application of DNA-based methods in forensic entomology. Annu Rev Entomol. 53, 103-120 (2008).
- Kotrba, M., et al. DNA barcoding in forensic entomology-establishing a DNA reference library of potentially forensic relevant arthropod species. J Forensic Sci. 64 (2), 593-601 (2019).
- Weedon, M. N., et al. Use of SNP chips to detect rare pathogenic variants: Retrospective, population based diagnostic evaluation. BMJ. 372, 214 (2021).
- Cai, J. -. F., et al. The availability of mitochondrial DNA cytochrome oxidasegene for the distinction of forensically important flies in China. Acta Entomologica Sinica. 48 (3), 380-385 (2005).
- Khayrova, A., Lopatin, S., Varlamov, V. Obtaining chitin, chitosan and their melanin complexes from insects. Int J Biol Macromol. 167, 1319-1328 (2021).
- Guo, Y. Q., et al. Comparison of three DNA extracting methods from different parts of common necrophagous flies. J Henan Univ Sci Tech (Med Sci). 36 (02), 130-133 (2018).
- Du, H., Xie, H., Ma, M., Igarashi, Y., Luo, F. Modified methods obtain high-quality DNA and RNA from anaerobic activated sludge at a wide range of temperatures. J Microbiol Methods. 199, 106532 (2022).
- Sukumaran, S. Concentration determination of nucleic acids and proteins using the micro-volume BioSpec-nano-spectrophotometer. J Vis Exp. (48), e2699 (2011).
- Masago, K., et al. Comparison between fluorimetry (Qubit) and spectrophotometry (NanoDrop) in the quantification of DNA and RNA extracted from frozen and FFPE tissues from lung cancer patients: A real-world use of genomic tests. Medicina (Kaunas). 57 (12), 1375 (2021).
- Ponti, G., et al. The value of fluorimetry (Qubit) and spectrophotometry (NanoDrop) in the quantification of cell-free DNA (cfDNA) in malignant melanoma and prostate cancer patients. Clin Chim Acta. 479, 14-19 (2018).
- Adams, Z. J., Hall, M. J. Methods used for the killing and preservation of blowfly larvae, and their effect on post-mortem larval length. Forensic Sci Int. 138 (1-3), 50-61 (2003).
- Fliegerova, K., et al. Effect of DNA extraction and sample preservation method on rumen bacterial population. Anaerobe. 29, 80-84 (2014).
- Rodrigues, B. L., Galati, E. A. B. Molecular taxonomy of phlebotomine sand flies (Diptera, Psychodidae) with emphasis on DNA barcoding: A review. Acta Trop. 238, 106778 (2023).
Erratum
Formal Correction: Erratum: DNA Extraction and Comparison Between Old and Fresh Necrophilic Fly Samples
Posted by JoVE Editors on 8/12/2024. Citeable Link.
This corrects the article 10.3791/66737
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon