JoVE 비디오를 활용하시려면 도서관을 통한 기관 구독이 필요합니다. 전체 비디오를 보시려면 로그인하거나 무료 트라이얼을 시작하세요.
Method Article
DNA 추출 및 오래된 네크로필릭 플라이 샘플과 신선한 네크로필릭 플라이 샘플 간의 비교
* 이 저자들은 동등하게 기여했습니다
Erratum Notice
요약
이 기사에서는 수정된 일반 DNA 추출 키트를 사용하여 신선하고 오래된 네크로필릭 파리 DNA 추출을 위한 프로토콜에 대해 설명합니다.
초록
Chrysomya megacephala 검체 총 5개 검체(신선한 검체 3개, 알코올 속에 2년 동안 보관된 검체 1개, 빛만 차단된 건조 밀봉 보관 2년 보관 검체 1개)를 선정하여 혈액 DNA 추출 키트가 괴사파리의 DNA를 추출할 수 있는지 여부를 조사하고 알코올이 괴사파리의 DNA 보존을 연장할 수 있는지 여부를 확인했습니다. 먼저 혈액 DNA 추출 키트를 사용하여 흉부 조직에서 DNA를 추출했습니다. 그런 다음 마이크로플레이트 리더와 형광계를 사용하여 DNA 순도와 농도를 검사했습니다. 마지막으로, 추출된 DNA의 PCR 증폭 및 전기영동은 미토콘드리아 CO I 유전자 서열에 위치한 네크로필릭 플라이 특이적 프라이머로 수행되었습니다. 그 결과 모든 샘플의 DNA 순도가 2.0 이상인 것으로 나타났습니다. DNA 농도는 다음과 같은 순서로 관찰되었습니다 : 신선한 샘플 > 알코올로 보존 된 오래된 샘플 > 처리되지 않은 오래된 샘플. 모든 샘플은 PCR 증폭 후 특정 전기영동 밴드를 가졌습니다. 결론적으로, 혈액 DNA 추출 키트를 사용하여 괴사 파리에서 DNA를 성공적으로 추출할 수 있으며, 신선한 파리 샘플의 DNA 농도는 오래된 파리 샘플의 DNA 농도보다 높습니다. 파리는 알코올에 오랫동안 보관할 수 있습니다.
서문
사망 시각을 추정하는 것은 항상 사법 실무에서 해결해야 할 핵심적이고 어려운 문제 중 하나였습니다. 법의학 실무에서 형사 사건의 경우 사후 검시 간격을 결정하는 것은 범죄 시간을 추론하고 용의자를 확보하며 조사 범위를 좁히는 데 중요한 역할을 합니다. 사망 시간을 추론하는 전통적인 방법은 주로 초기 사후 현상에 기반을 두고 있으며, 일반적으로 24시간 이내의 사망 시간을 추론하는 데만 사용할 수 있습니다. 그러나 죽음의 긴 시간은 사후 현상에 의해 결정될 수 없다. 이제 법의학계에서는 법곤충학이 더 긴 사망 시간을 추론하는 효과적인 방법이 될 수 있는 잠재력을 가지고 있다는 것이 일반적으로 인정되고 있습니다.
Necrophagous 곤충은 시체를 먹는 유충의 이름을 따서 명명되었습니다. 그 중 사체가 있을 때마다 성충 괴사파리가 가장 먼저 사체에 도달하여 알이나 유충을 낳습니다. 유충은 시체 조직을 먹고 번성하여 번데기로 성장한 후 성충으로 번식합니다. 네크로필릭 파리는 규칙적인 생활 주기와 지리적 분포(예: 사망 시간 공제 및 사망 장소 결정) 때문에 법의학 실습에서 큰 역할을 합니다 1,2. 그러나 법의학 실습에서 괴사 파리를 사용하는 데 장벽이 되는 것은 종 식별입니다. 형태학적 방법은 여전히 괴사파리의 종 식별을 위한 권위 있는 방법으로 사용되고 있습니다. 그러나 형태학적 종 식별에는 높은 수준의 곤충 무결성이 필요합니다. 만약 파리들이 다른 발달 단계에 있었거나, 환경적 선택에 의한 형태학적 변화로 인해, 이 모든 것들이 형태학적 검사를 더 어렵게 만든다. 특히 범죄 현장에서 가장 많이 발견되는 번데기와 번데기 껍질의 형태는 거의 알아볼 수 없습니다. 이것은 형태 학적 전문가의 부족과 함께 형태 학적으로 종을 식별하는 데 큰 어려움을 초래합니다. 따라서 파리의 종 식별을 위한 분자 생물학 방법의 적용이 등장하여 형태학적 식별의 어려움을 줄이고 발달 단계 3,4,5,6,7과 무관합니다.
분자생물학 방법을 기반으로 한 괴사파리의 종 식별의 첫 번째 단계는 DNA의 효율적인 추출이며, 곤충 DNA 추출을 위한 특수 키트는 현재 일반적으로 구할 수 없습니다. 그리고 네크로필릭 파리의 DNA 추출을 위해 일반적인 혈액 키트를 사용하는 방법은 법의학적으로 더욱 중요해집니다. 범죄 현장에서 발견되는 괴사파리는 종종 훼손되거나 부패 및 DNA 분해를 겪었습니다. 플라이 샘플은 획득 후 DNA 추출에 즉시 사용할 수 없으며, 가능한 경우 증거 보존 요구 사항으로 인해 전체 샘플을 보관해야 합니다. 위의 요구 사항을 바탕으로 본 연구에서는 proteinase K 분해에 기반한 혈액 DNA 키트를 적용하고 3개의 신선한 Chrysomya megacephala (Diptera, Lepidoptera) 샘플(그림 1), 2년 동안 알코올에 넣은 오래된 C. megacephala 샘플 1개, 2년 동안 차광 보관에 넣은 C. megacephala 샘플 1개를 선택하여 5개 샘플의 무게를 각각 측정했습니다. DNA 추출을 위한 곤충 흉부 조직을 선출했습니다. 기존 샘플과 새 샘플에서 추출한 DNA의 품질과 순도를 비교하고, 추출된 DNA의 PCR 증폭 및 전기영동을 미토콘드리아 CO 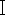 유전자 서열에 위치한 네크로필릭 플라이 특이적 프라이머로 수행했습니다.
유전자 서열에 위치한 네크로필릭 플라이 특이적 프라이머로 수행했습니다.
프로토콜
참고: 이 프로토콜에는 총 5개의 C. megacephala 샘플 샘플이 사용되었으며, 3개의 신선한 샘플(Fresh 1, Fresh 2 및 Fresh 3), 1개의 샘플은 2년 동안 알코올에 보관(Old 1), 1개의 샘플은 빛으로부터 보호되는 2년 동안 건조 밀봉 보관(Old 2)되었습니다. 샘플은 실험 요구 사항에 따라 라벨을 부착해야 합니다.
1. 일반 시료 보관 및 준비
- 시료 선택 및 준비
알림: 괴사파리를 처형하기 위해 에틸 아세테이트를 적용할 때 흄 후드에서 작업해야 합니다. 무게를 측정하기 전에 플라이 표면에서 액체를 제거하십시오.- 신선한 샘플: 수집된 3개의 플라이 샘플을 에틸 아세테이트로 죽인 후 가스 바이알에 넣습니다. 샘플을 계량하고 이름을 Fresh 1, Fresh 2 및 Fresh 3으로 지정하고 실험에 즉시 사용합니다.
참고: 이 프로토콜의 신선한 파리 샘플의 질량은 56.8mg, 49.3mg 및 52.1mg이었습니다. - 오래된 샘플 1: 에틸 아세테이트에 의해 사멸된 파리 샘플을 꺼내 알코올이 채워진 원심분리 튜브에 2년 동안 밀폐 밀봉하고 실온에서 빛으로부터 보호합니다. 여과지로 표면 액체를 흡착 한 후 샘플의 무게를 잰다. 올드 1이라고 부릅니다.
참고: 이 프로토콜에서 Old 1의 질량은 42.3mg이었습니다. - 오래된 샘플 2: 에틸 아세테이트에 의해 사멸된 파리 샘플을 꺼내 실온에서 빛으로부터 보호되는 밀폐 밀봉 상태로 원심분리기 튜브에 2년 동안 보관합니다. 무게를 잰 후 늙었다고 부르세요 2.
- 신선한 샘플: 수집된 3개의 플라이 샘플을 에틸 아세테이트로 죽인 후 가스 바이알에 넣습니다. 샘플을 계량하고 이름을 Fresh 1, Fresh 2 및 Fresh 3으로 지정하고 실험에 즉시 사용합니다.
- 추출 전 준비
- 스테인리스 스틸 용기를 냉각시키기 위해 소량의 액체 질소를 첨가하십시오. 모든 액체 질소가 증발할 때까지 기다리십시오.
- 컨테이너에 플라이 샘플을 넣으십시오. 액체 질소가 튀어 곤충 샘플에 충격을 가하는 것을 방지하기 위해 액체 질소를 천천히 붓습니다.
- 액체 질소로 샘플을 얼립니다. 액체 질소가 증발한 후 샘플을 제거합니다.
- 흉부를 개별적으로 분리하고 2mL 원심분리기 튜브에 넣고 가능한 가장 작은 조각으로 자릅니다.
참고: 샘플을 작은 조각으로 잘라 완전히 절단할 수 있도록 하면 DNA가 대량으로 방출될 수 있습니다. - 각 파리에 대해 위의 단계를 반복하고 개별적으로 얼린 다음 DNA 교차 오염을 방지하기 위해 새 원심분리기 튜브로 제거합니다.
2. DNA 추출
알림: 모든 원심분리 단계는 미세 원심분리기에서 실온(15-25°C)에서 수행해야 합니다. 모든 단계는 무균 원칙을 엄격히 준수하고 교차 오염을 방지하기 위해 수행해야 합니다.
- 조직 용해
- 클리핑된 샘플이 들어 있는 2mL 원심분리 튜브에 180μL의 조직 용해 완충액을 추가합니다.
- 20 μL의 proteinase K를 추가합니다. 3,000 rpm에서 10 초 동안 볼텍싱하여 철저히 혼합하고 곤충 조직이 완전히 용해될 때까지 56 °C에서 배양합니다.
참고: 조직이 완전히 소화되지 않은 경우 Proteinase K를 더 추가해야 합니다. 용해는 일반적으로 1-3시간 내에 완료되며 하룻밤 사이에 용해 시간을 연장할 수도 있습니다.
- DNA 침전
- 3,000rpm에서 15초 동안 소용돌이. 샘플에 200μL 용해 완충액을 추가하고 15초 동안 3,000rpm에서 볼텍싱하여 철저히 혼합합니다.
- 200μL의 에탄올(96-100%)을 넣고 3,000rpm에서 15초 동안 볼텍싱하여 다시 철저히 섞습니다.
참고: 용해 완충액과 에탄올을 한 번에 함께 사전 첨가하여 시간을 절약할 수 있습니다. 용해 완충액과 에탄올의 첨가시 백색 침전물이 형성될 수 있습니다.
- 혼합물을 2mL 수집 튜브에 배치된 DNA 흡착 컬럼에 피펫으로 넣습니다. 6,000× g 에서 1분 동안 원심분리기 플로우스루와 수집 튜브를 폐기하십시오.
- 이전 단계의 DNA 흡착 컬럼을 새 2mL 수집 튜브에 넣고 500μL의 단백질 제거 완충액을 추가한 다음 6,000× g 에서 1분 동안 원심분리합니다. 플로우스루와 수집 튜브를 폐기하십시오.
- DNA 흡착 컬럼을 새로운 2mL 수집 튜브로 옮기고 500μL의 담수화 완충액을 추가한 다음 20,000× g 에서 3분 동안 원심분리하여 DNA 흡착 컬럼 멤브레인을 건조시킵니다. 플로우스루와 수집 튜브를 폐기하십시오.
알림: 이 원심분리 단계는 다음 용출 전에 잔류 에탄올을 제거합니다. 에탄올의 캐리오버가 있는 경우 20,000× g 에서 1분 동안 다시 원심분리합니다. - DNA 흡착 컬럼을 깨끗한 2mL 미세 원심분리기 튜브에 넣고 100μL의 DNA 용출 버퍼를 컬럼 멤브레인에 직접 피펫하고 실온에서 1분 동안 배양한 다음 6,000× g 에서 1분 동안 원심분리하여 용리합니다.
참고: 200 μL 대신 100 μL로 용리하면 용리액의 최종 DNA 농도가 증가하지만 전체 DNA 수율도 감소합니다. DNA 수율을 최대화하려면 2.8단계에 설명된 대로 용리를 한 번 반복합니다. - DNA 샘플은 4°C(최대 1주) 또는 장기 보관을 위해 -80°C에서 보관합니다.
3. DNA 농도 검출
- OD 260 및 OD 280의 값을 측정하여 마이크로플레이트 리더로 DNA 순도와 핵산 농도를 비교합니다. 각 샘플에 대해 세 번 반복합니다.
- 각 샘플에 대해 3번의 반복이 있는 동반 키트를 사용하여 형광측정기로 추출된 DNA의 농도를 측정합니다.
- 190 μL의 작업 용액을 두 개의 0.5 mL 원심분리 튜브에 넣고 표준 #1 및 표준 #2 각각에 10 μL를 취하여 작업 용액에 첨가하고 15분 이상 빛으로부터 멀리 두십시오.
참고: 표준 #1 및 표준 #2는 특정 농도의 DNA에 대해 정확하게 보정됩니다. 표준 용액은 정확한 표준 곡선의 형성을 보장하도록 정확하게 구성되어야 합니다. 표준 #1은 0.2ng/μL 이중 가닥 DNA(dsDNA) 샘플입니다. 표준 #2는 2,000ng/μL입니다. 작용 용액은 dsDNA에 특이적으로 결합하는 염료입니다. - 2 μL의 테스트 DNA를 0.5 mL 원심 분리 튜브에 넣고 198 μL의 작업 용액과 혼합하고 15 분 이상 빛을 멀리하십시오.
참고: DNA 농도가 낮으면 5μL의 작업 용액과 195μL의 테스트 DNA를 추가합니다. - 15분 후 기기를 켜고 dsDNA 농도 측정 | 장거리. 먼저 표준 #1을 테스트한 다음 표준 #2를 테스트하여 표준 곡선을 얻습니다.
참고: 표준 솔루션은 구성된 후 같은 날에 사용해야 합니다. 너무 오래 두면 표준 곡선에 큰 오류가 발생합니다. - 검사할 샘플을 분석 웰에 하나씩 넣고 DNA 스파이킹 부피를 2μL로 조정한 후 측정을 세 번 반복하고 평균값을 취하여 기록합니다.
- 190 μL의 작업 용액을 두 개의 0.5 mL 원심분리 튜브에 넣고 표준 #1 및 표준 #2 각각에 10 μL를 취하여 작업 용액에 첨가하고 15분 이상 빛으로부터 멀리 두십시오.
4. PCR와 전기영동 검출
참고: 길이가 278bp인 미토콘드리아 CO 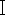 유전자 서열을 전방 프라이머 C1-J-2495: CAGCTACTTTATGAGCTTTTAG, 역방향 프라이머 C1-N-2800: CATTTCAAGCTGTTAAGCATC8로 PCR 증폭을 위해 취한 다음 증폭 효과 및 증폭 산물의 길이를 확인하기 위해 2% 아가로스 겔 전기영동을 수행했습니다.
유전자 서열을 전방 프라이머 C1-J-2495: CAGCTACTTTATGAGCTTTTAG, 역방향 프라이머 C1-N-2800: CATTTCAAGCTGTTAAGCATC8로 PCR 증폭을 위해 취한 다음 증폭 효과 및 증폭 산물의 길이를 확인하기 위해 2% 아가로스 겔 전기영동을 수행했습니다.
- 10 μL의 2x Taq 믹스, 각 프라이머의 1 μL, 5 μL의 ddH2O 및 3 μL의 템플릿 DNA를 추가하여 PCR 증폭 시스템의 20 μL를 설정합니다. 원심분리기에서 10초 동안 잘 섞습니다.
- 다음 PCR 순환 매개변수를 사용하십시오: 10분 동안 95°C의 사전 변성; 변성 95 ° C 30 초 동안, 어닐링 48 ° C 30 초, 확장 72 ° C 동안 45 초 동안 총 40 사이클; 최종 연장 72 ° C에서 10 분 동안.
- PCR 후 제품을 2% 아가로스 겔 전기영동에 적용하고 전기영동 후 사진 분석을 위해 겔 이미징 시스템에 놓습니다.
결과
마이크로플레이트 리더를 사용하여 추출된 DNA 용액의 OD260 및 OD280 값을 측정한 다음 OD260/OD280 값을 얻어 DNA의 순도를 평가했습니다. 모든 신선한 샘플과 오래된 샘플 1의 OD260/OD280은 2보다 컸습니다. 이전 샘플 2의 OD260/OD280은 평균이 1.753이지만 최대값은 2.187이었고, OD280과 OD280의 값이 작은(≤0.01) 때문에 세 번의 측정치가 크게 변동했습니다(표 1). 얻어진 OD260의 값을 기반으로 DNA의 농?...
토론
DNA 추출은 괴사파리의 분자 식별에 가장 중요한 연결 고리이며, 추출 방법과 추출된 DNA의 품질은 후속 검출에 직접적인 영향을 미칩니다. 이 실험에서는 곤충 DNA 추출이 더 쉬워지는 특별한 곤충 DNA 추출 키트를 구입할 필요 없이 일반 혈액 키트를 사용하여 괴사파리에서 DNA를 추출하는 데 성공했습니다.
파리의 표면은 키틴질이 풍부하며, 특히 표피 키틴이 60-70%에 달하는 유?...
공개
저자는 경쟁하는 재정적 이익이 없다고 선언합니다.
감사의 말
이 연구는 중국 국립자연과학재단(National Natural Science Foundation of China, 82060341,81560304)과 하이난성 학술 혁신 플랫폼 과학 연구 프로젝트(YSPTZX202134)의 지원을 받습니다.
자료
| Name | Company | Catalog Number | Comments |
| Buffer AE | Qiagen | 172026832 | DNA elution buffer |
| Buffer AL | Qiagen | 172028374 | lysis buffer |
| Buffer ATL | Qiagen | 172028162 | tissue lysis buffer |
| Buffer AW1 | Qiagen | 172028760 | protein removal buffer |
| Buffer AW2 | Qiagen | 57203108 | desalination buffer |
| C1-J-2495 | Taihe Biotechnology | TW21109216 | forword primer |
| C1-N-2800 | Taihe Biotechnology | TW21109217 | reverse primer |
| D2000 DNA ladder | Real-Times(Beijing) Biotechnology | RTM415 | Measure the position of electrophoretic bands |
| DNeasy Mini spin column | Qiagen | 166050343 | DNA adsorption column |
| Dry Bath Incubator | Miulab | DKT200-2D | used for heating |
| MIX-30S Mini Mixer | Miulab | MUC881206 | oscillatory action |
| Proteinase K | Qiagen | 172026218 | Inactivation of intracellular nucleases and other proteins |
| Qubit 3.0 Fluorometer | Thermo Fisher Scientific | 2321611188 | |
| Speed Micro-Centrifuge | Scilogex | 9013001121 | centrifuge |
| Standard#1 | Thermo Fisher Scientific | 2342797 | |
| Standard#2 | Thermo Fisher Scientific | 2342797 | |
| Tanon 3500R Gel Imager | Tanon | 16T5553R-455 | gel imaging |
| Taq Mix Pro | Monad | 00007808-140534 | PCR Mix |
| Taq Mix Pro | Monad | 00007808-140534 | PCR Mix |
| Thermo Cycler | Zhuhai Hema | VRB020A | ordinary PCR |
| Working Solution | Thermo Fisher Scientific | 2342797 |
참고문헌
- Amendt, J., Richards, C. S., Campobasso, C. P., Zehner, R., Hall, M. J. Forensic entomology: applications and limitations. Forensic Sci Med Pathol. 7 (4), 379-392 (2011).
- Wydra, J., Matuszewski, S. The optimal post-eclosion interval while estimating the post-mortem interval based on an empty puparium. Forensic Sci Med Pathol. 17 (2), 192-198 (2021).
- Mazzanti, M., Alessandrini, F., Tagliabracci, A., Wells, J. D., Campobasso, C. P. DNA degradation and genetic analysis of empty puparia: genetic identification limits in forensic entomology. Forensic Sci Int. 195 (1-3), 99-102 (2010).
- Odeniran, P. O., et al. molecular identification and distribution of Trypanosome-transmitting Dipterans from cattle settlements in Southwest Nigeria. Acta Parasitol. 66 (1), 116-128 (2021).
- Wells, J. D., Stevens, J. R. Application of DNA-based methods in forensic entomology. Annu Rev Entomol. 53, 103-120 (2008).
- Kotrba, M., et al. DNA barcoding in forensic entomology-establishing a DNA reference library of potentially forensic relevant arthropod species. J Forensic Sci. 64 (2), 593-601 (2019).
- Weedon, M. N., et al. Use of SNP chips to detect rare pathogenic variants: Retrospective, population based diagnostic evaluation. BMJ. 372, 214 (2021).
- Cai, J. -. F., et al. The availability of mitochondrial DNA cytochrome oxidasegene for the distinction of forensically important flies in China. Acta Entomologica Sinica. 48 (3), 380-385 (2005).
- Khayrova, A., Lopatin, S., Varlamov, V. Obtaining chitin, chitosan and their melanin complexes from insects. Int J Biol Macromol. 167, 1319-1328 (2021).
- Guo, Y. Q., et al. Comparison of three DNA extracting methods from different parts of common necrophagous flies. J Henan Univ Sci Tech (Med Sci). 36 (02), 130-133 (2018).
- Du, H., Xie, H., Ma, M., Igarashi, Y., Luo, F. Modified methods obtain high-quality DNA and RNA from anaerobic activated sludge at a wide range of temperatures. J Microbiol Methods. 199, 106532 (2022).
- Sukumaran, S. Concentration determination of nucleic acids and proteins using the micro-volume BioSpec-nano-spectrophotometer. J Vis Exp. (48), e2699 (2011).
- Masago, K., et al. Comparison between fluorimetry (Qubit) and spectrophotometry (NanoDrop) in the quantification of DNA and RNA extracted from frozen and FFPE tissues from lung cancer patients: A real-world use of genomic tests. Medicina (Kaunas). 57 (12), 1375 (2021).
- Ponti, G., et al. The value of fluorimetry (Qubit) and spectrophotometry (NanoDrop) in the quantification of cell-free DNA (cfDNA) in malignant melanoma and prostate cancer patients. Clin Chim Acta. 479, 14-19 (2018).
- Adams, Z. J., Hall, M. J. Methods used for the killing and preservation of blowfly larvae, and their effect on post-mortem larval length. Forensic Sci Int. 138 (1-3), 50-61 (2003).
- Fliegerova, K., et al. Effect of DNA extraction and sample preservation method on rumen bacterial population. Anaerobe. 29, 80-84 (2014).
- Rodrigues, B. L., Galati, E. A. B. Molecular taxonomy of phlebotomine sand flies (Diptera, Psychodidae) with emphasis on DNA barcoding: A review. Acta Trop. 238, 106778 (2023).
Erratum
Formal Correction: Erratum: DNA Extraction and Comparison Between Old and Fresh Necrophilic Fly Samples
Posted by JoVE Editors on 8/12/2024. Citeable Link.
This corrects the article 10.3791/66737
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유