需要订阅 JoVE 才能查看此. 登录或开始免费试用。
Method Article
使用互补金属氧化物半导体高密度微电极阵列系统从急性脑切片中获得高质量的癫痫样活性
* 这些作者具有相同的贡献
摘要
在这里,我们概述了一种使用互补金属氧化物半导体高密度微电极阵列系统 (CMOS-HD-MEA) 记录 离体 脑切片的癫痫样活动的协议。
摘要
互补金属氧化物半导体高密度微电极阵列 (CMOS-HD-MEA) 系统可以以前所未有的电生理学细节记录细胞培养物和 离体 脑切片的神经生理活动。CMOS-HD-MEA 最初被优化为记录来自细胞培养物的高质量神经元单位活性,但也已被证明可以从急性视网膜和小脑切片中产生高质量的数据。研究人员最近使用 CMOS-HD-MEA 记录了急性皮质啮齿动物脑切片的局部场电位 (LFP)。一种值得关注的 LFP 是癫痫样活动。虽然许多用户使用 CMOS-HD-MEA 产生了短暂的自发性癫痫样放电,但很少有用户能够可靠地产生高质量的癫痫样活动。许多因素可能导致这种困难,包括电噪声、使用浸没式记录室时产生癫痫样活动的不一致性质,以及 2D CMOS-MEA 芯片只能从脑切片表面记录的限制。本协议中详述的技术应使用户能够使用 CMOS-HD-MEA 系统从急性脑切片中持续诱导和记录高质量的癫痫样活动。此外,该协议概述了 CMOS-HD-MEA 芯片的正确处理、实验期间溶液和脑切片的管理以及设备维护。
引言
市售的高密度微电极阵列 (HD-MEA) 系统,包括具有数千个记录点的 MEA 芯片 1,2 和用于放大和数字化数据的 MEA 平台,是电生理学研究的新兴工具。这些 HD-MEA 系统使用互补金属氧化物半导体 (CMOS) 技术,以高灵敏度记录细胞培养物和离体脑切片制备的电生理数据。这些 MEA 系统通过高电极密度和高质量的信噪比为神经生理学研究提供了前所未有的空间和时间分辨率3。该技术主要用于研究细胞外动作电位,但它也可以从各种神经元脑切片制备物中捕获高质量的局部场电位 (LFP) 4,5,6,7,8,9,10,11,12,13,14,15 .由于 CMOS-HD-MEA 系统的上述高分辨率记录能力,用户可以以极高的空间精度跟踪电生理活动 16,17,18。此功能与跟踪网络 LFP5、12、15、19、20、21 的传播模式特别相关。因此,CMOS-HD-MEA 系统可以对各种细胞培养物和脑切片制备物的生理和病理活动的传播模式提供前所未有的理解。特别值得注意的是,CMOS-HD-MEA 系统的这些功能可以让研究人员同时对比不同大脑区域的癫痫发作模式,并分析各种抗癫痫化合物如何影响这些模式。通过这样做,它为研究发作发生和发作传播以及理解药理学如何破坏病理网络活动提供了一种创新方法 7,10,14。因此,CMOS-HD-MEA 系统的这些新能力可以为神经系统疾病的研究做出重大贡献,并有助于药物发现研究 5,7,11,22。我们旨在提供有关使用 CMOS-HD-MEA 系统研究癫痫样活动的详细信息。
当使用 CMOS-HD-MEA 系统研究 LFP 时,例如急性脑切片中的癫痫样活动,用户可能会面临许多挑战,包括降低电噪声、在实验期间保持切片健康,以及检测来自二维 (2D) CMOS-MEA 芯片的质量信号,该芯片仅从脑切片表面记录。该协议描述了将 MEA 平台和实验中使用的其他设备正确接地的基本步骤,这是一个关键步骤,可能需要对每个实验室设置进行单独定制。此外,我们还讨论了在与 CMOS-HD-MEA 系统一起使用的浸没室中进行长时间记录期间帮助保持脑切片健康的步骤 23,24,25。此外,与从脑切片深处记录的更常见的电生理记录方法相比,大多数 CMOS-HD-MEA 系统使用不会穿透到切片中的 2D 芯片。因此,这些系统需要一个健康的神经元外层来产生大部分记录的 LFP 信号。其他挑战包括可视化数千个电极生成的大量数据。为了克服这些挑战,我们推荐一种简单但有效的方案,以增加实现在脑切片中传播的高质量网络癫痫样活动的可能性。我们还简要描述了我们开发的公开可用的图形用户界面 (GUI) 以及相关资源,以帮助数据可视化10。
以前的出版物提供了使用 MEA 记录系统的相关协议 26,27,28,29。然而,这项工作旨在帮助使用带有 2D 芯片的 CMOS-HD-MEA 系统的实验者,特别是那些寻求从脑切片中研究高质量癫痫样活动的实验者。此外,我们比较了两种最常见的诱导癫痫发作样活动的溶液操作,即 0 Mg2+ 和 4-AP 范式,以帮助用户确定最适合其特定应用的惊厥培养基。尽管该方案主要集中在癫痫发作样活动的产生上,但可以对其进行修改以使用脑切片探索其他电生理现象。
Access restricted. Please log in or start a trial to view this content.
研究方案
涉及小鼠的程序由杨百翰大学的机构动物护理和使用委员会 (IACUC) 批准。在以下实验中使用年龄至少为 P21 的雄性和雌性 (n = 8) C57BL/6 小鼠。
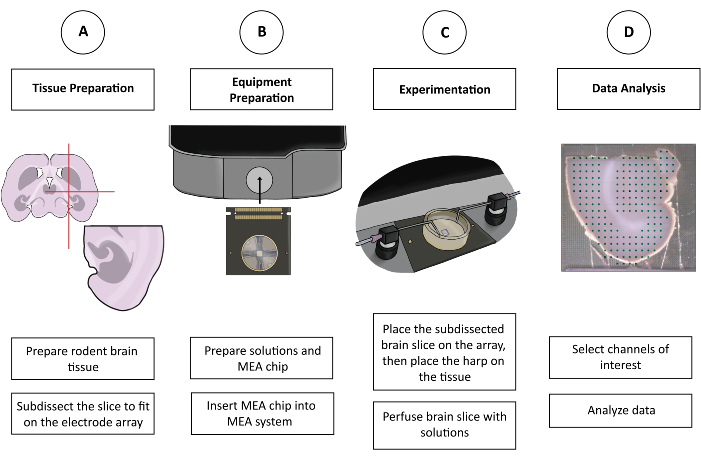
图 1:CMOS-HD-MEA 实验示意图。 (A) 通过自己喜欢的切割方法制备脑切片,并进行二次解剖以适合 MEA。(B) 制备溶液和 CMOS-HD-MEA 芯片。(C) 将子解剖的脑切片放在电极阵列上,并浸泡在适当的溶液中。(D) 从收集的数据中选择相关渠道。然后准备数据,以便在用户的首选程序中进行分析。 请单击此处查看此图的较大版本。
| 名字 | 浓度 (mM) | 克/升 | ||
| 氯化钠 (NaCl) | 126 | 7.36 | ||
| 氯化钾 (KCl) | 3.5 | 0.261 | ||
| 磷酸二氢钠 (NaH2PO4) | 1.26 | 0.151 | ||
| 碳酸氢钠 (NaHCO3) | 26 | 2.18 | ||
| 葡萄糖 (C6H12O6) | 10 | 1.80 | ||
| 氯化镁 (MgCl2) | 1 (来自 1 M 库存) | 1 毫升 | ||
| 氯化钙 (CaCl2) | 2 (来自 1 M 库存) | 2 毫升 | ||
表 1:aCSF 解决方案。
1. 准备解决方案
- 准备实验溶液。
- 准备 1 L 人工脑脊液 (aCSF)(详见 表 1 )。
- 准备 1 L 促惊厥溶液。
注:在该方案中用于产生癫痫样活性的溶液使用含 100 μM 4-氨基吡啶 (4-AP) 的 aCSF 或不含镁离子的 aCSF。 - 使用多孔结石将所有溶液碳化至少 10 分钟。
- 获得用于丢弃溶液的烧杯。
- 将 1 L aCSF、1 L 促惊厥溶液和丢弃烧杯放在靠近灌注系统的表面上。
- 准备切割室和保温室解决方案
- 制备 0.5 L aCSF,省略 CaCl2 并添加 3 mM MgCl2 (而不是 1 mM)以制备独特的切割溶液。将这种溶液放在急性脑切片准备区域附近,保留用于啮齿动物脑切片制备。
- 准备 0.5 L aCSF 并将其放入组织保存室中,该组织容纳室将用于保留切片以用于实验。将多孔结石插入组织保持室中,并将溶液碳化至少 10 分钟,然后将子解剖的切片放入腔室中。
2. 准备啮齿动物脑切片
- 如上所述准备所有解决方案(参见第 1 节)。确保在制备过程中所有溶液都正确碳化。
注意:建议使用切割溶液(参见第 1.2 节),但可以使用其他切割溶液来获得高质量的脑切片,例如蔗糖切割溶液30。 - 使用振动切片机制作 350 μm 啮齿动物脑切片。使用这些仪器的方案列在参考文献30、31、32、33 中。
- 准备啮齿动物脑切片以适合 MEA 芯片的记录电极区域(参见 图 2)。使用 10 号手术刀片对切片进行二次解剖, 如图 1A 和 图 2A 所示,通过在脑切片上轻轻地来回滚动刀片。在振动切片机切割室中进行子解剖。小心不要损坏子解剖的脑切片。
- 将子解剖的切片放入充满 aCSF 的组织保持室中。在添加子解剖切片之前,确保腔室已碳化至少 10 分钟。
3. 设备准备
- 准备 MEA 芯片和系统(图 2 和 图 3)
注意:塑料和金属等硬质材料如果强行接触芯片底部,很容易损坏 MEA 芯片。使用移液器将溶液移入或移出芯片时,不要将芯片底部与移液器接触,尤其是芯片井侧面的参比电极和芯片井中心的记录电极(图 2C)。相反,通过将鼠标悬停在芯片井的底部或通过接触或接近芯片的塑料边缘来添加溶液。为了轻松拉出所有溶液,请稍微倾斜芯片,让溶液充分聚集到芯片的一侧,然后将其从混合溶液的顶部取出,或使用防静电擦拭剂轻轻擦拭仍有溶液的区域。不要通过握住芯片或芯片引脚来运输芯片(图 2C)。芯片孔可容纳约 4 mL 溶液。对于后续步骤,如果未特别说明,则用大约 2 mL 的溶液填充芯片孔。- 在开始芯片制备之前,为各种任务指定移液管。为一个转移移液器标记乙醇,一个用于废液,另一个用于 aCSF,其他移液器用于任何剩余溶液,以防止意外混合。
- 用 190 级乙醇填充 MEA 芯片的孔,使芯片孔的底部完全覆盖(图 3)。让乙醇静置 30-60 秒,然后用弃置移液管将其取出。
- 用 aCSF 填充 MEA 芯片的孔,并用丢弃移液管将其取出,以冲洗掉芯片孔中残留的乙醇。使用先前指定的废液和 aCSF 移液器,从芯片孔中加入和取出 aCSF 三次。将芯片清洗 3 次后,加入 aCSF 并静置至少 30 秒。
注:用乙醇和 aCSF 冲洗后,当 aCSF 在腔室中保留至少 45 分钟时,记录电极因疏水相互作用而产生的噪音最少。 - 在对接 MEA 芯片之前,用 190 级乙醇润湿防静电抹布,然后用它来擦拭芯片的引脚(图 2C)。
- 轻轻地将 MEA 芯片滑入 MEA 平台,并接合对接机构以将芯片锁定到位。
- 检查记录电极和参比电极是否有气泡(图 2C)。如果存在气泡,请用干净的画笔轻轻扫过电极以去除它们。
- 使用 CMOS-HD-MEA 软件34 检查芯片的噪声,并目视扫描伪彩色图,以查找由电气干扰引起的气泡、非生物振荡或尖峰。将 MEA 系统适当接地以消除遇到的任何噪音。
注意: 接地设置将取决于录制环境。对于该方案中的实验,MEA 平台和灌注系统接地。
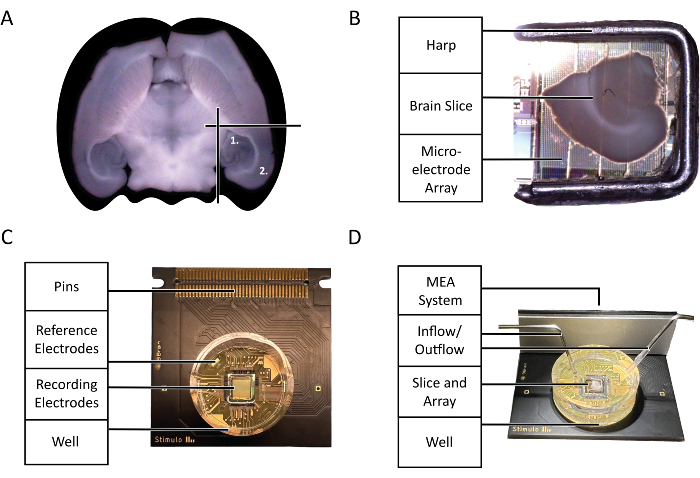
图 2:配置和技术图。 (A) 该协议突出显示的实验中使用的急性小鼠脑切片的选择图。(1) 海马区 (2) 新皮层区。(B) 在微电极阵列 (MEA) 上正确放置急性小鼠脑切片和竖琴。(C) 3Brain Accura CMOS-HD-MEA 芯片的解剖结构。(D) 灌注入口和出口的正确配置。输入应位于芯片井的深处,而输出应位于芯片井顶部入口的另一侧,以保证新鲜、含氧的 aCSF 的恒定流动。 请单击此处查看此图的较大版本。
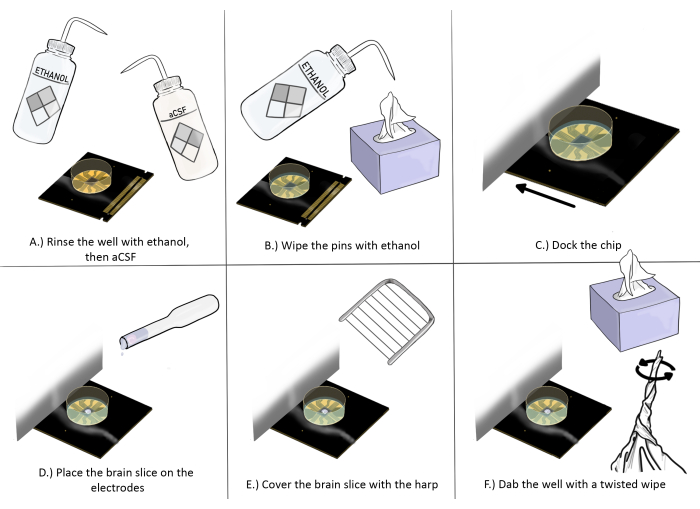
图 3:脑切片实验的芯片制备和放置示意图。 (A) 用乙醇冲洗芯片一次,然后用 aCSF 冲洗 3 次。(B) 使用防静电抹布蘸乙醇擦拭针脚。(C) 对接芯片。(D) 将脑切片放在电极上。(E) 将竖琴放在脑切片上(参见 图 2 了解正确的放置指南)。(F) 用扭曲的防静电擦洗剂轻拍靠近脑切片的记录电极井的角落。 请单击此处查看此图的较大版本。
4. 实验
- 放置脑切片
- 将铂金竖琴放在 MEA 平台附近的称重船(或其他干净的表面)中。用大约 3mL 的 aCSF 覆盖竖琴,以减少其疏水倾向。
- 使用剪刀去除移液管的薄尖端部分。切掉三分之一,约 1.5 英寸的移液器吸头。
注意:这种改进的移液器在收集和运输过程中不会收缩或损坏脑切片。 - 用改良的移液器从切片保持室中收集脑切片。轻轻地将脑切片和移液管中的任何溶液分配到芯片中。为了正确定位切片,请从移液管中轻轻释放更多的 aCSF 以操纵脑切片的位置,或使用软画笔在溶液中产生电流,将脑切片推到记录电极上。限制与记录电极或脑切片的接触,以尽量减少损伤。
- 使用镊子轻轻地将竖琴放在脑切片上,螺纹向下,将切片压在记录电极上。避免将电极阵列与竖琴接触。调整竖琴的方向,使没有框架的一侧面向流入针,并且竖琴的框架不接触任何记录电极(图 2C、D)。
- 取弃管并去除多余的 aCSF。取防静电擦拭布,扭动一个角以形成一个尖端,然后用它来吸收记录电极周围剩余的 aCSF,而无需接触记录电极、脑切片或竖琴(图 2)。
- 使用指定的 aCSF 移液器,快速加入足够的碳化 aCSF 以覆盖脑切片,约 2 mL。
- 重复步骤 4.1.5。和 4.1.6.再来两次。
- 用碳化的 aCSF 填充孔,直到孔大约满 3/4,约 3 mL。
- 用显微镜或相机拍摄 MEA 芯片上的脑切片照片。确保照片具有足够高的分辨率,可以看到记录电极阵列的边界和脑切片的解剖结构。
- 运行实验
- 操作灌注系统
- 将流入管和流出管放入装有 aCSF 的烧杯中,将流入和流出针放入芯片井中。将流入针放在靠近芯片底部的位置,就在记录电极外侧。将流出针靠近芯片顶部朝向边缘,使液体几乎上升到芯片井的边缘,约 4 mL,并且在整个实验过程中将切片浸没(图 2D)。
- 将灌注流入设置为 5 mL/min,将灌注流出设置为 7 mL/min。
注意:建议流出速率超过流入速率,以避免溶液从芯片孔中溢出,并在脑切片上产生溶液电流。 - 打开流入和流出。从芯片中取出流入针,直到针开始输出溶液而不是空气。然后,按照步骤 4.2.1.1 中的说明将针放回芯片内的位置。
- 使用溶液加热器将溶液保持在或接近生理温度,约 34-37 °C。
- 让 aCSF 灌注在脑切片上 10 分钟。这将允许切片适应录制环境。
- 10 分钟后,将流出管移至丢弃烧杯中。然后,将流入管移至装有促惊厥溶液的烧杯中。将非惊厥性 aCSF 从灌注系统中冲洗到丢弃烧杯中 10 分钟。
- 将流出管转移到装有促惊厥溶液的烧杯中。
- 让促惊厥溶液循环直到实验结束。
- 如果切片记录持续时间超过 2 小时,请考虑准备另一种促惊厥溶液来抵消活性脑切片的葡萄糖消耗。
- 交换脑切片
- 关闭流入。之后 10-15 秒关闭流出。
- 用镊子取下竖琴并将其放在称重船或其他表面上。
- 使用改良的移液器提取并丢弃脑切片。不要触摸参比电极或记录电极阵列(图 2C)。
- 将流入管放入装有非惊厥性 aCSF 溶液的烧杯中。将流出管放入丢弃烧杯中。将流入针和流出针留在切屑中。运行灌注系统 10 分钟,以去除芯片孔和灌注系统中残留的所有促惊厥溶液。使用新的脑切片再次开始试验过程(从步骤 4.1 开始)。
- 操作灌注系统
- 完成实验
- 清洁钻机
- 分别从孔中取出竖琴、脑切片和溶液。
- 拆下 MEA 芯片,将其放在干净的表面上,然后用去离子水填充芯片。
- 丢弃去离子水以清除 aCSF 溶液中残留的任何盐分。
- 使用移液管,用去污剂溶液充分填充芯片。将移液管的尖端悬停在芯片槽中,并将其指向记录电极。快速反复地将洗涤剂挤压并释放到移液管中,以在记录电极上用力洗涤剂 1 分钟。让洗涤剂静置 5-10 分钟。
- 去除去污剂,然后用去离子水将芯片冲洗 4-6 次,以去除去污剂溶液和任何残留的溶质。
- 去除芯片井中的所有水,然后在 MEA 芯片上涂上防静电擦布,放置过夜,让芯片井干燥。
- 将称重船或其他防水板放在 MEA 记录平台上,MEA 芯片之前所在的位置。将竖琴放在板的中央,并将流入和流出的针放在竖琴的两侧。
- 将 50 mL 水、50 mL 140 标准乙醇和 200 mL 水分别通过管道送入丢弃烧杯,清洁灌注系统和竖琴。
注:此时,可以提高灌注速度以减少清洁时间,但流出速度必须始终超过流入速度,以避免溢流。 - 将碳化石放入盛有蒸馏水的烧杯中,然后将碳化物通过管道 5 分钟,以清洁碳化石。将它们放在干燥、干净的表面上过夜,并盖上盖子以避免灰尘积聚。
- 清洁钻机
5. 数据分析
注:有多种分析包用于分析 CMOS-HD-MEA 生成的电生理数据,包括来自 3Brain 的 BrainWave、Yet Another Spike Sorter (YASS) 和自定义 Python 工具34、35、36、37。我们从 BrainWave 数据文件格式中提取数据,用于 Xenon LFP 分析平台,以生成图 4 和图 5 所示的数据。使用自定义 Matlab 代码来分析图 6 中的数据。Xenon LFP 分析平台的实验方案已公开发布10.以下协议步骤特定于使用 Brainwave 438 进行的录音;对于其他系统,请参阅与这些系统相关的支持文档 34,35,36,37。下面概述了使用此协议生成数据所采取的分析步骤。有关如何导出、可视化和分析数据的完整详细信息,包括教程视频和所有相关代码文件,请参阅39。
- 导出录制文件以进行分析。
- 为将进行缩减采样的数据记录文件创建一个文件夹。
- 裁剪在步骤 4.1.9 中拍摄的图像,以仅包含带有脑切片的录制数组。
- 打开 ExportToHDF5-ChannelSelection.py 文件(此代码文件可以在 GitHub 上的 xenon-lfp-analysis/code-files/3Brain-processing40 下访问),然后将窗口顶部列出的 URL 复制并粘贴到 Internet 浏览器中。
- 输入数据记录文件的文件路径,并上传与记录关联的阵列的裁剪图像。
- 在 Select Channels for Export 下,使用套索选择工具选择阵列裁剪图像上显示的海马和新皮质区域。
- 将要跳过的行数和要跳过的列数设置为 2。
注意:录制文件通常很大;如果适合所需的分析,则可以对导出的数据进行缩减采样。存在空间下采样(通过跳过记录电极的行和列)和时间上(通过下采样到较低采样频率)的选项。在分析中,每 2 行和 2 列跳过一次;这允许对数据进行空间下采样,而不会偏向于某些通道。每个通道之间的距离足够远,以至于它们之间的活动差异可能很大;因此,不执行通道平均。当跳过电极的行和列时,导出输出文件时不包括来自未选择通道的数据。 - 将 downsampling frequency 设置为 300 Hz。
- 单击 Export Channels 按钮以生成通道选择文件。
注意:软件会自动将通道选择文件保存到保存数据记录文件的文件夹中。 - 打开 ExportToHDF5-ExtractDownsample.py 文件(也可以在 GitHub 上找到 xenon-lfp-analysis/code-files/3brain-processing40 下),粘贴包含通道选择文件和数据记录文件的文件夹的文件路径,按键盘上的 Enter 键生成数据记录文件的缩减采样版本。
- 使用分析平台
- 在计算机的命令提示符下,键入 run_lfp_analysis 并按 Enter 键以打开分析平台。
- 输入图片和缩减采样记录文件的文件路径,然后单击 提交。
- 在 Select a Time Range for Analysis (选择用于分析的时间范围) 下,选择整个记录。
- 在 Select channels for Lots 下,将海马体突出显示为 Group-1,将新皮层突出显示为 Group-2。
- 在 Channel Raster 下,将 Threshold 设置为 0.06 mV,将 Time Duration 设置为 0.02 s。
- 单击 Apply Settings 和 Generate Raster。
- 使用栅格图可探索显示感兴趣活动模式的不同通道。
注意:参见图 4 中高质量癫痫样活动(图 4A-D)与次优活动(图 4E-F)的示例。
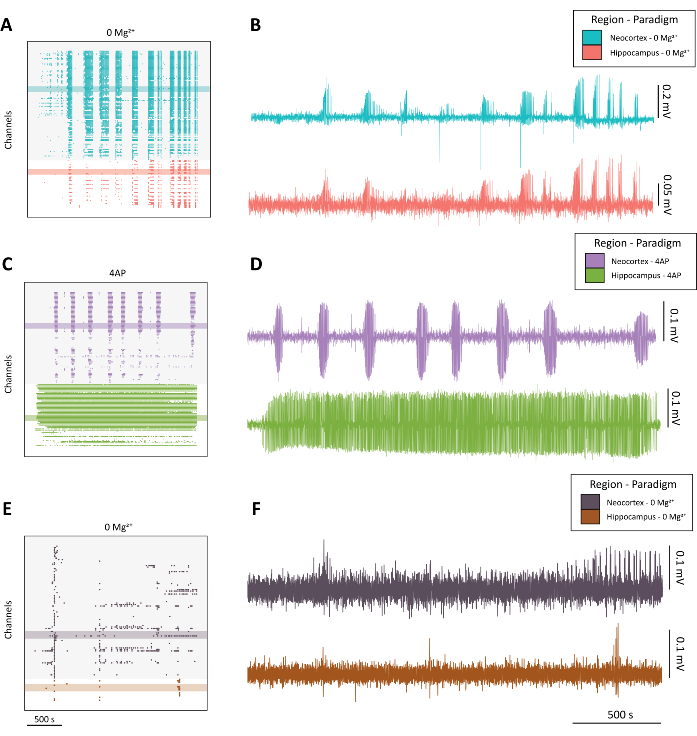
图 4:从 0 Mg2+ 和 4-AP 范式演变癫痫样活动的示例。 (A) 在大约 40 分钟内应用含 0 Mg2+ 的 aCSF 的示例光栅图。(B) 从新皮层(蓝色)和海马体(红色)采集的示例电生理学轨迹,显示 0 Mg2+ 范式的癫痫样活动。(C) 在大约 40 分钟内应用 100 mM 4-AP 的示例栅格图。(D) 从新皮层(紫色)和海马体(绿色)采集的示例电生理学迹线,显示应用 4-AP 产生的癫痫样活动。(E) 在大约 40 分钟内应用含 0 Mg2+ 的 aCSF 的示例栅格图,显示爆发活动,而不是在其他代表性迹线中发现的癫痫样活动。(F) 从新皮层(深紫色)和海马体(铁锈色)中提取的示例电生理学痕迹,证明 0 Mg2+ 范式的次优活性,旨在与 B 和 D 中发现的质量癫痫样活动 进行比较。
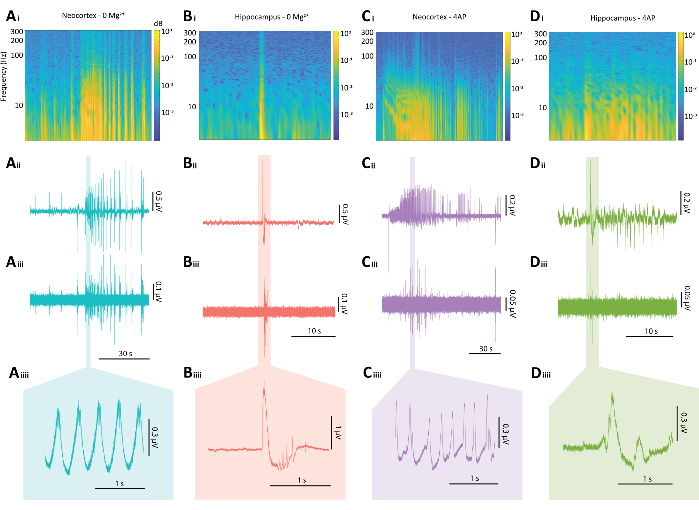
图 5:0 Mg2+ 和 4-AP 范式的癫痫样放电的代表性结果。 (A) 由 0 Mg2+ 范式诱导的典型新皮层癫痫发作样事件的示例图,包括 (Ai) 来自癫痫发作样事件的频谱图,(Aii) 相关的电生理学轨迹,(Aiii) 应用于来自 Aii 的轨迹的 80 Hz 高通滤波器,(Aiiii) 和来自 Aii 的轨迹的放大部分。(B) 由 0 Mg2+ 范式诱导的典型海马癫痫样爆发的示例图,包括 (Bi) 癫痫样爆发的频谱图,(Bii) 相关的电生理轨迹,(Biii) 应用于 Bii 轨迹的 80 Hz 高通滤波器,(Biiii) 和 Bii 轨迹的放大部分 (C) 由 4-AP 范式诱导的典型新皮质癫痫发作样事件的示例图,包括 (Ci) 癫痫样活动的频谱图,(Cii) 相关的电生理轨迹,(Ciii) 应用于 Cii 轨迹的 80 Hz 高通滤波器,(Ciiii) 和 Cii 轨迹的放大部分 (D) 4-AP 范式下海马癫痫样爆发的示例图,包括 (Di) 癫痫样活动的频谱图,(Dii) 相关的电生理轨迹, (Diii) 应用于 Dii 迹线的 80 Hz 高通滤波器 (Diiii) 和来自 Dii 的迹线的放大部分。 请单击此处查看此图的较大版本。
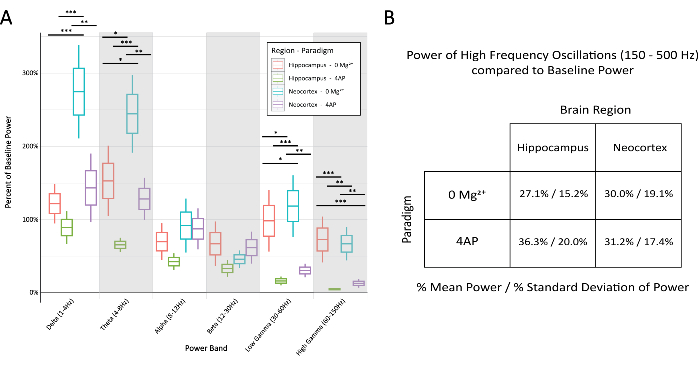
图 6:在刻板印象癫痫样放电期间跨范式和大脑区域的各个频段中基线功率百分比的比较。 (A) 对于大多数频段,癫痫样放电期间的功率在范式和大脑区域之间显着差异(带 Tukey 检验的 2 向方差分析,*P < 0.05,**P < 0.001,***P < 0.0001)。每个框的中间线代表平均值,框的边界±平均值的标准误差 (SEM) 和最外层的线 ±2 SEM。(B) 范式和大脑区域在与 150 Hz 以上的高频活动相关的频段中都表现出有限的功率。 请单击此处查看此图的较大版本。
Access restricted. Please log in or start a trial to view this content.
结果
作为可视化来自多个通道1、4、5、10 的活动的标准,我们发现首先生成我们使用 CMOS-HD-MEA 获取的数据的光栅图是有益的(图 4A、C、E)。该图可以通过在 y 轴上显示每个通道,在 x 轴上显示时间,在每个脑切片的所有记录通道?...
Access restricted. Please log in or start a trial to view this content.
讨论
该协议包括与急性脑切片管理相关的具体指南,以解决 CMOS-HD-MEA 用户面临的常见问题,即脑切片下的噪声产生和维持脑切片的健康环境。当切片未正确粘附到数组时,切片下方会产生噪声;如果脑切片没有充分粘附,切片下方会形成气穴,从而导致噪音。这将导致无法获取数据。为了缓解这些挑战,该协议强调了两个关键步骤:首先,确保在放置过程中正确的竖琴方向,?...
Access restricted. Please log in or start a trial to view this content.
披露声明
作者声明,本研究不存在利益冲突。
致谢
作者感谢前任和现任 Parrish 实验室成员对本手稿的编辑。我们还要感谢 3Brain 的 Alessandro Maccione 对这项工作的反馈。这项工作由 AES/EF 初级研究员奖和杨百翰大学生命科学学院和物理与数学科学学院资助。
Access restricted. Please log in or start a trial to view this content.
材料
| Name | Company | Catalog Number | Comments |
| 2D Workbench | Cloudray | LM04CLLD26B | |
| 4-Aminopyridine | Sigma-Aldrich | 275875 | |
| Accura Chip | 3Brain | Accura HD-MEA | CMOS-HD-MEA chip |
| Agarose | Thermo Fisher Scientific | BP160-100 | |
| Vibration isolation table | Kinetic Systems | 91010124 | |
| Beaker for the slice holding chamber, 270 mL | VWR | 10754-772 | |
| BioCam | 3Brain | BioCAM DupleX | CMOS-HD-MEA platform |
| Brainwave Software | 3Brain | Version 4 | CMOS-HD-MEA software |
| Calcium Chloride | Thermo Fisher Scientific | BP510-500 | |
| Carbogen | Airgas | X02OX95C2003102 | |
| Carbogen | Airgas | 12005 | |
| Carbogen Stones | Supelco | 59277 | |
| Compresstome | Precissionary | VF-300-0Z | |
| Computer | Dell | Precission3650 | |
| Crocodile Clip Grounding Cables | JWQIDI | B06WGZG17W | |
| Detergent | Metrex | 10-4100-0000 | |
| D-Glucose | Macron Fine Chemicals | 4912-12 | |
| Dihydrogen Sodium Phosphate | Thermo Fisher Scientific | BP329-500 | |
| DinoCam | Dino-Lite | AM73915MZTL | |
| Ethanol | Thermo Fisher Scientific | A407P-4 | |
| Forceps | Fine Science Tools | 11980-13 | |
| Hot plate | Thermo Fisher Scientific | SP88857200 | |
| Ice Machine | Hoshizaki | F801MWH | |
| Inflow and outflow needles | Jensen Global | JG 18-3.0X | |
| Inline Solution Heater | Warner Instruments | SH-27B | |
| Isofluorine | Dechra | 08PB-STE22002-0122 | |
| Kim Wipes | Thermo Fisher Scientific | 06-666 | |
| Magnesium Chloride | Thermo Fisher Scientific | FLM33500 | |
| Micropipets | Gilson | F144069 | |
| Mili-Q Water Filter | Mili-Q | ZR0Q008WW | |
| Paintbrush | Daler Rowney | AF85 Round: 0 | |
| Paper Filter | Whatman | EW-06648-24 | |
| Parafilm | American National Can | PM996 | |
| Perfusion System | Multi Channel System | PPS2 | |
| Pipetor | Thermo Fisher Scientific | FB14955202 | |
| Platinum Harp | 3Brain | 3Brain | |
| Potassium Chloride | Thermo Fisher Scientific | P330-3 | |
| Razor blade | Personna | BP9020 | |
| Scale | Metter Toledo | AB204 | |
| Scissors | Solingen | 92008 | |
| Slice Holding Chamber | Custom | Custom | Custom 3D Printer Design, available upon request |
| Sodium Bicarbonate | Macron Fine Chemicals | 7412-06 | |
| Sodium Chloride | Thermo Fisher Scientific | S271-3 | |
| Temperature Control Box | Warner Instruments | TC344B | |
| Transfer Pipettes | Genesee Scientific | 30-200 | |
| Tubing | Tygon | B-44-3 TPE | |
| Vibratome VZ-300 | Precissionary | VF-00-VM-NC | |
| Weigh Boat | Electron Microscopy Sciences | 70040 |
参考文献
- Obien, M. E. J., Frey, U. Large-scale, high-resolution microelectrode arrays for interrogation of neurons and networks. Adv Neurobiol. 22, 83-123 (2019).
- Schroter, M., et al. Functional imaging of brain organoids using high-density microelectrode arrays. MRS Bull. 47 (6), 530-544 (2022).
- Miccoli, B., et al. High-density electrical recording and impedance imaging with a multi-modal CMOS multi-electrode array chip. Front Neurosci. 13, 641(2019).
- Emery, B. A., Hu, X., Khanzada, S., Kempermann, G., Amin, H. High-resolution CMOS-based biosensor for assessing hippocampal circuit dynamics in experience-dependent plasticity. Biosens Bioelectron. 237, 115471(2023).
- Ferrea, E., et al. high-resolution electrophysiological imaging of field potentials in brain slices with microelectronic multielectrode arrays. Front Neural Circuits. 6, 80(2012).
- Gagliano, G., et al. Non-linear frequency dependence of neurovascular coupling in the cerebellar cortex implies vasodilation-vasoconstriction competition. Cells. 11 (6), 1047(2022).
- Goodchild, S. J., et al. Molecular pharmacology of selective Na(V)1.6 and dual Na(V)1.6/Na(V)1.2 channel inhibitors that suppress excitatory neuronal activity ex vivo. ACS Chem Neurosci. 15 (6), 1169-1184 (2024).
- Hu, X., Khanzada, S., Klutsch, D., Calegari, F., Amin, H. Implementation of biohybrid olfactory bulb on a high-density CMOS-chip to reveal large-scale spatiotemporal circuit information. Biosens Bioelectron. 198, 113834(2022).
- Kim, S., et al. Alteration of neural network and hippocampal slice activation through exosomes derived from 5XFAD nasal lavage fluid. Int J Mol Sci. 24 (18), 14064(2023).
- Mahadevan, A., Codadu, N. K., Parrish, R. R. Xenon LFP analysis platform is a novel graphical user interface for analysis of local field potential from large-scale MEA recordings. Front Neurosci. 16, 904931(2022).
- Medrihan, L., Ferrea, E., Greco, B., Baldelli, P., Benfenati, F. Asynchronous GABA release is a key determinant of tonic inhibition and controls neuronal excitability: A study in the synapsin II-/- mouse. Cereb Cortex. 25 (10), 3356-3368 (2015).
- Monteverdi, A., Di Domenico, D., D'Angelo, E., Mapelli, L. Anisotropy and frequency dependence of signal propagation in the cerebellar circuit revealed by high-density multielectrode array recordings. Biomedicines. 11 (5), 1475(2023).
- Obien, M. E. J., Hierlemann, A., Frey, U. Accurate signal-source localization in brain slices by means of high-density microelectrode arrays. Sci Rep. 9 (1), 788(2019).
- Thouta, S., et al. Pharmacological determination of the fractional block of Nav channels required to impair neuronal excitability and ex vivo seizures. Front Cell Neurosci. 16, 964691(2022).
- Tognolina, M., Monteverdi, A., D'Angelo, E. Discovering microcircuit secrets with multi-spot imaging and electrophysiological recordings: The example of cerebellar network dynamics. Front Cell Neurosci. 16, 805670(2022).
- Hierlemann, A., Frey, U., Hafizovic, S., Heer, F. Growing cells atop microelectronic chips: Interfacing electrogenic cells in vitro with CMOS-based microelectrode arrays. Proceedings of the IEEE. 99 (2), 252-284 (2011).
- Maccione, A., et al. Experimental investigation on spontaneously active hippocampal cultures recorded by means of high-density MEAs: Analysis of the spatial resolution effects. Front Neuroeng. 3, 4(2010).
- van Vliet, E., et al. Electrophysiological recording of re-aggregating brain cell cultures on multi-electrode arrays to detect acute neurotoxic effects. Neurotoxicology. 28 (6), 1136-1146 (2007).
- Emery, B. A., et al. Large-scale multimodal recordings on a high-density neurochip: Olfactory bulb and hippocampal networks. Annu Int Conf IEEE Eng Med Biol Soc. 2022, 3111-3114 (2022).
- Veleanu, M., et al. Modified climbing fiber/Purkinje cell synaptic connectivity in the cerebellum of the neonatal phencyclidine model of schizophrenia. Proc Natl Acad Sci U S A. 119 (21), e2122544119(2022).
- Giansante, G., et al. Neuronal network activity and connectivity are impaired in a conditional knockout mouse model with PCDH19 mosaic expression. Mol Psychiatry. , (2023).
- Dossi, E., Blauwblomme, T., Nabbout, R., Huberfeld, G., Rouach, N. Multi-electrode array recordings of human epileptic postoperative cortical tissue. J Vis Exp. (92), e51870(2014).
- Hajos, N., et al. Maintaining network activity in submerged hippocampal slices: importance of oxygen supply. Eur J Neurosci. 29 (2), 319-327 (2009).
- Hill, M. R., Greenfield, S. A. The membrane chamber: a new type of in vitro recording chamber. J Neurosci Methods. 195 (1), 15-23 (2011).
- Raimondo, J. V., et al. Methodological standards for in vitro models of epilepsy and epileptic seizures. A TASK1-WG4 report of the AES/ILAE Translational Task Force of the ILAE. Epilepsia. 58 (Suppl 4), 40-52 (2017).
- Hales, C. M., Rolston, J. D., Potter, S. M. How to culture, record and stimulate neuronal networks on micro-electrode arrays (MEAs). J Vis Exp. (39), 2056(2010).
- Lin, C. H., Lee, J. K., LaBarge, M. A. Fabrication and use of microenvironment microarrays (MEArrays). J Vis Exp. (68), 4152(2012).
- Panuccio, G., Colombi, I., Chiappalone, M. Recording and modulation of epileptiform activity in rodent brain slices coupled to microelectrode arrays. J Vis Exp. 135, 57548(2018).
- Patel, C., Muthuswamy, J. High efficiency, site-specific transfection of adherent cells with siRNA using microelectrode arrays (MEA). J Vis Exp. 67, e4415(2012).
- Ting, J. T., Daigle, T. L., Chen, Q., Feng, G. Acute brain slice methods for adult and aging animals: application of targeted patch clamp analysis and optogenetics. Methods Mol Biol. 1183, 221-242 (2014).
- Papouin, T., Haydon, P. G. Obtaining acute brain slices. Bio Protoc. 8 (2), e2699(2018).
- Ting, J. T., et al. Preparation of acute brain slices using an optimized N-Methyl-D-glucamine protective recovery method. J Vis Exp. 132, 53825(2018).
- Van Hoeymissen, E., Philippaert, K., Vennekens, R., Vriens, J., Held, K. Horizontal hippocampal slices of the mouse brain. J Vis Exp. (163), 61753(2020).
- 3Brain. , Available from: https://www.3brain.com/ (2022).
- Bridges, D. C., Tovar, K. R., Wu, B., Hansma, P. K., Kosik, K. S. MEA Viewer: A high-performance interactive application for visualizing electrophysiological data. PLoS One. 13 (2), e0192477(2018).
- Hawrylycz, M., et al. Inferring cortical function in the mouse visual system through large-scale systems neuroscience. Proc Natl Acad Sci U S A. 113 (27), 7337-7344 (2016).
- Maccione, A., et al. Microelectronics, bioinformatics and neurocomputation for massive neuronal recordings in brain circuits with large scale multielectrode array probes. Brain Res Bull. 119 (Pt B), 118-126 (2015).
- 3Brain. , BrainWave 4 https://www.3brain.com/products/software/brainwave4 (2022).
- Mahadevan, A. Xenon LFP Analysis. , https://xenon-lfp-analysis.readthedocs.io/en/latest/Installation.html (2022).
- Mahadevan, A. xenon-lfp-analysis github. , https://github.com/MicroBrew09/xenon-lfp-analysis (2022).
- Codadu, N. K., et al. Divergent paths to seizure-like events. Physiol Rep. 7 (19), e14226(2019).
- Kirsch, G. E., Drewe, J. A. Gating-dependent mechanism of 4-aminopyridine block in two related potassium channels. J Gen Physiol. 102 (5), 797-816 (1993).
- Levesque, M., Salami, P., Behr, C., Avoli, M. Temporal lobe epileptiform activity following systemic administration of 4-aminopyridine in rats. Epilepsia. 54 (4), 596-604 (2013).
- Myers, T. L., Gonzalez, O. C., Stein, J. B., Bazhenov, M. Characterizing concentration-dependent neural dynamics of 4-Aminopyridine-induced epileptiform activity. Epilepsy J. 4 (2), 128(2018).
- Perreault, P., Avoli, M. Physiology and pharmacology of epileptiform activity induced by 4-aminopyridine in rat hippocampal slices. J Neurophysiol. 65 (4), 771-785 (1991).
- Rutecki, P. A., Lebeda, F. J., Johnston, D. 4-Aminopyridine produces epileptiform activity in hippocampus and enhances synaptic excitation and inhibition. J Neurophysiol. 57 (6), 1911-1924 (1987).
- Chen, Y., Chad, J. E., Cannon, R. C., Wheal, H. V. Reduced Mg2+ blockade of synaptically activated N-methyl-D-aspartate receptor-channels in CA1 pyramidal neurons in kainic acid-lesioned rat hippocampus. Neuroscience. 88 (3), 727-739 (1999).
- Fujiwara-Tsukamoto, Y., Isomura, Y., Takada, M. Comparable GABAergic mechanisms of hippocampal seizure-like activity in posttetanic and low-Mg2+ conditions. J Neurophysiol. 95 (3), 2013-2019 (2006).
- Swartzwelder, H. S., Anderson, W. W., Wilson, W. A. Mechanism of electrographic seizure generation in the hippocampal slice in Mg2+-free medium: the role of GABAa inhibition. Epilepsy Res. 2 (4), 239-245 (1988).
- Trevelyan, A. J., Graham, R. T., Parrish, R. R., Codadu, N. K. Synergistic positive feedback mechanisms underlying seizure initiation. Epilepsy Curr. 23 (1), 38-43 (2023).
- Croning, M. D., Haddad, G. G. Comparison of brain slice chamber designs for investigations of oxygen deprivation in vitro. J Neurosci Methods. 81 (1-2), 103-111 (1998).
- Hajos, N., Mody, I. Establishing a physiological environment for visualized in vitro brain slice recordings by increasing oxygen supply and modifying aCSF content. J Neurosci Methods. 183 (2), 107-113 (2009).
- Huang, Y., Williams, J. C., Johnson, S. M. Brain slice on a chip: opportunities and challenges of applying microfluidic technology to intact tissues. Lab Chip. 12 (12), 2103-2117 (2012).
- Andrew, R. D., et al. The critical role of spreading depolarizations in early brain injury: Consensus and contention. Neurocrit Care. 37 (Suppl 1), 83-101 (2022).
- Devonshire, I. M., Dommett, E. J., Grandy, T. H., Halliday, A. C., Greenfield, S. A. Environmental enrichment differentially modifies specific components of sensory-evoked activity in rat barrel cortex as revealed by simultaneous electrophysiological recordings and optical imaging in vivo. Neuroscience. 170 (2), 662-669 (2010).
- Parrish, R. R., Codadu, N. K., Mackenzie-Gray Scott, C., Trevelyan, A. J. Feedforward inhibition ahead of ictal wavefronts is provided by both parvalbumin- and somatostatin-expressing interneurons. J Physiol. 597 (8), 2297-2314 (2019).
- Wang, H., Jing, M., Li, Y. Lighting up the brain: genetically encoded fluorescent sensors for imaging neurotransmitters and neuromodulators. Curr Opin Neurobiol. 50, 171-178 (2018).
- Yaksi, E., Jamali, A., Diaz Verdugo, C., Jurisch-Yaksi, N. Past, present and future of zebrafish in epilepsy research. FEBS J. 288 (24), 7243-7255 (2021).
- He, M. F., et al. Ex vivo calcium imaging for drosophila model of epilepsy. J Vis Exp. 200, 65825(2023).
- Driscoll, N., et al. Multimodal in vivo recording using transparent graphene microelectrodes illuminates spatiotemporal seizure dynamics at the microscale. Commun Biol. 4 (1), 136(2021).
- Parrish, R. R., Grady, J., Codadu, N. K., Trevelyan, A. J., Racca, C. Simultaneous profiling of activity patterns in multiple neuronal subclasses. J Neurosci Methods. 303, 16-29 (2018).
- Valderhaug, V. D., et al. Criticality as a measure of developing proteinopathy in engineered human neural networks. bioRxiv. , (2020).
- Multi-electrode array (MEASs) to investigate pathogenetic disease mechanisms and pharmacological properties in iPSC-derived neurons modelling neuropsychiatric diseases. Carleo, G., Lee, Y. -S., Secondo, A., Miceli, F., Taglialatela, M. 2022 IEEE International Conference on Metrology for Extended Reality, Artificial Intelligence and Neural Engineering (MetroXRAINE), , Rome, Italy. 667-672 (2022).
- Ruz, I. D., Schultz, S. R. Localising and classifying neurons from high density MEA recordings. J Neurosci Methods. 233, 115-128 (2014).
- Franke, F., Natora, M., Boucsein, C., Munk, M. H. J., Obermayer, K. An online spike detection and spike classification algorithm capable of instantaneous resolution of overlapping spikes. J Comput Neurosci. 29 (1-2), 127-148 (2010).
- Vollgraf, R., Obermayer, K. Improved optimal linear filters for the discrimination of multichannel waveform templates for spike-sorting applications. IEEE Signal Processing Letters. 13 (3), 121-124 (2006).
- Muller, J., et al. High-resolution CMOS MEA platform to study neurons at subcellular, cellular, and network levels. Lab Chip. 15 (13), 2767-2780 (2015).
- Mapelli, L., et al. implementation, and functional validation of a new generation of microneedle 3D high-density CMOS multi-electrode array for brain tissue and spheroids. bioRxiv. , (2022).
- Reddy, D. S., Kuruba, R. Experimental models of status epilepticus and neuronal injury for evaluation of therapeutic interventions. Int J Mol Sci. 14 (9), 18284-18318 (2013).
- Parrish, R. R., Trevelyan, A. J. Stress-testing the brain to understand its breaking points. J Physiol. 596 (11), 2033-2034 (2018).
Access restricted. Please log in or start a trial to view this content.
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。