このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
相補型金属酸化物半導体高密度微小電極アレイシステムによる急性脳切片からの高品質発作様活性
* これらの著者は同等に貢献しました
要約
ここでは、相補型金属酸化膜半導体高密度微小電極アレイシステム(CMOS-HD-MEA)を使用して、 ex vivo 脳スライスからの発作様活性を記録するためのプロトコルを概説します。
要約
相補型金属酸化膜半導体高密度微小電極アレイ(CMOS-HD-MEA)システムは、細胞培養および ex vivo 脳切片からの神経生理学的活性をこれまでにないほど詳細に記録できます。CMOS-HD-MEAは、細胞培養から高品質のニューロンユニット活性を記録するために最初に最適化されましたが、急性網膜切片および小脳切片からも高品質のデータを生成することが示されています。研究者は最近、CMOS-HD-MEAを使用して、急性の皮質げっ歯類の脳スライスからの局所電界電位(LFP)を記録しています。注目すべきLFPの1つは、発作様活動です。多くのユーザーがCMOS-HD-MEAを使用して短時間の自然てんかん様分泌物を引き起こしていますが、質の高い発作様活動を確実に引き出すユーザーは少ないです。この困難には、電気ノイズ、水中の記録チャンバーを使用したときに発作のような活動を引き起こす一貫性のない性質、2D CMOS-MEAチップが脳スライスの表面からのみ記録するという制限など、多くの要因が寄与している可能性があります。このプロトコルで詳述されている技術により、ユーザーは CMOS-HD-MEA システムを使用して、急性脳切片から高品質の発作様活動を一貫して誘導および記録できるはずです。さらに、このプロトコルでは、CMOS-HD-MEAチップの適切な取り扱い、実験中の溶液と脳スライスの管理、および機器のメンテナンスについて概説しています。
概要
市販の高密度微小電極アレイ(HD-MEA)システムには、数千の記録ポイント1,2を備えたMEAチップと、データを増幅およびデジタル化するためのMEAプラットフォームが含まれており、電気生理学的研究のための新しいツールです。これらのHD-MEAシステムは、相補型金属酸化膜半導体(CMOS)技術を使用して、細胞培養およびex vivo脳スライス調製物からの電気生理学的データを高感度で記録します。これらのMEAシステムは、高い電極密度と高品質の信号対雑音比3により、神経生理学的研究に前例のない空間的および時間的分解能を提供します。この技術は、主に細胞外活動電位の研究に使用されてきましたが、さまざまなニューロン脳スライス調製物4,5,6,7,8,9,10,11,12,13,14,15から高品質の局所電位(LFP)を捕捉することもできます.CMOS-HD-MEAシステムの上記の高解像度記録機能により、ユーザーは高い空間精度16,17,18で電気生理学的活動を追跡することができる。この機能は、ネットワーク LFP5、12、15、19、20、21 の伝搬パターンの追跡に特に関連します。したがって、CMOS-HD-MEAシステムは、さまざまな細胞培養および脳スライス調製物からの生理学的および病理学的活性の伝播パターンについて、これまでにない理解を提供することができます。特に注目すべきは、CMOS-HD-MEAシステムのこれらの機能により、研究者は異なる脳領域の発作パターンを同時に対比し、さまざまな抗てんかん化合物がこれらのパターンにどのように影響するかを分析できることです。これにより、発作発生と発作増殖を研究し、薬理学が病理学的ネットワーク活動をどのように混乱させるかを理解するための革新的な方法を提供します7,10,14。したがって、CMOS-HD-MEAシステムのこれらの新しい能力は、神経疾患の研究に大きく貢献するだけでなく、創薬研究にも役立つ可能性があります5,7,11,22。CMOS-HD-MEAシステムを使用して発作様活動を研究する方法について詳しく説明します。
CMOS-HD-MEAシステムを使用して急性脳切片のてんかん様活動などのLFPを研究する場合、ユーザーは、電気ノイズの衰弱、実験中のスライスの健康維持、脳スライスの表面からのみ記録する2次元(2D)CMOS-MEAチップからの高品質信号の検出など、多くの課題に直面する可能性があります。このプロトコルは、実験で使用されるMEAプラットフォームやその他の機器を適切に接地するための基本的な手順を説明しています。これは、ラボのセットアップごとに個別にカスタマイズする必要がある重要なステップです。さらに、CMOS-HD-MEAシステム23,24,25で使用される水中チャンバーでの長時間の録音中に脳スライスを健康に保つのを助けるための手順について説明します。さらに、脳スライスの深部から記録する一般的な電気生理学的記録方法とは対照的に、ほとんどのCMOS-HD-MEAシステムは、スライスに浸透しない2Dチップを使用します。したがって、これらのシステムでは、記録されたLFP信号の大部分を生成するために、健康なニューロンの外層が必要です。その他の課題には、数千の電極によって生成される大量のデータを視覚化することが含まれます。これらの課題を克服するために、脳スライス全体に伝播する高品質のネットワークてんかん様活動を達成する可能性を高める、シンプルかつ効果的なプロトコルをお勧めします。また、データの視覚化を支援するために開発した公開グラフィックユーザーインターフェイス(GUI)と関連リソースの簡単な説明も含まれています10。
以前の出版物は、MEA記録システムの使用に関する関連プロトコルを提供してきた26、27、28、29。ただし、この研究は、2Dチップを備えたCMOS-HD-MEAシステムを使用する実験者、特に脳スライスからの高品質のてんかん様活動を研究しようとしている実験者を支援することを目的としています。さらに、発作様活動の誘導のための最も一般的な溶液操作の 2 つ、すなわち 0 Mg2+ および 4-AP パラダイムを比較し、ユーザーが特定のアプリケーションに最も適したけいれん培地を特定できるようにします。このプロトコルは主に発作様活動の生成に焦点を当てていますが、脳スライスを使用して他の電気生理学的現象を探索するように変更することができます。
プロトコル
マウスを用いた手技は、ブリガム・ヤング大学の動物管理・使用委員会(IACUC)によって承認された。以下の実験では、少なくともP21に年齢を重ねた雄と雌(n = 8)のC57BL/6マウスを用いた。
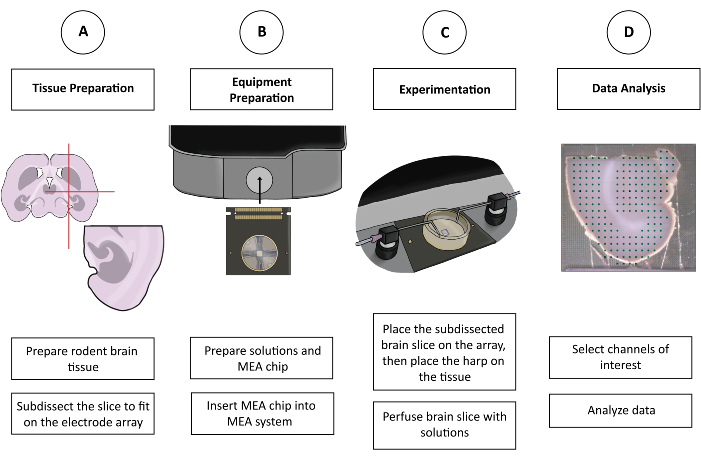
図1:CMOS-HD-MEA実験の概略図(A)脳スライスは、好みの切断方法で調製され、MEAに収まるようにサブディスセクションされます。(B)溶液とCMOS-HD-MEAチップを準備します。(C)サブディスセクションされた脳スライスを電極アレイ上に配置し、適切な溶液に浸します。(D)収集されたデータから関連するチャネルが選択されます。その後、データはユーザーの好みのプログラムで分析するために準備されます。この図の拡大版を表示するには、ここをクリックしてください。
| 名前 | 濃度(mM) | g/L | ||
| 塩化ナトリウム(NaCl) | 126 | 7.36 | ||
| 塩化カリウム(KCl) | 3.5 | 0.261 | ||
| リン酸二水素ナトリウム(NaH2PO4) | 1.26 | 0.151 | ||
| 重炭酸ナトリウム(NaHCO3) | 26 | 2.18 | ||
| グルコース(C6H12O6) | 10 | 1.80 | ||
| 塩化マグネシウム(MgCl2) | 1 (1 M ストックから) | 1 mLの | ||
| 塩化カルシウム(CaCl2) | 2 (1 Mストックから) | 2ミリリットル | ||
表1:aCSFソリューション。
1. 溶液の準備
- 実験溶液を準備します。
- 人工脳脊髄液(aCSF)1Lを調製します(詳細は 表1 を参照)。
- 1Lの抗けいれん薬溶液を調製します。
注:このプロトコルで発作様活性を生成するために使用された溶液は、100μM 4-アミノピリジン(4-AP)を含むaCSFまたはマグネシウムイオンを含まないaCSFのいずれかを利用しました。 - 多孔質石を使用して、使用前にすべての溶液を少なくとも10分間炭水化物化します。
- 溶液を捨てるためのビーカーを入手してください。
- 1 LのaCSF、1 Lの抗けいれん薬溶液、および廃棄ビーカーを灌流システムに近い表面に置きます。.
- 切断および保持チャンバー溶液の準備
- CaCl2 を省略し、3 mM MgCl2 (1 mM ではなく) を添加して 0.5 L の aCSF を調製し、独自の切断液を作製します。この溶液は、げっ歯類の脳スライス調製で使用するために、急性脳スライス調製領域の近くに置くことにより保持します。
- 0.5 L の aCSF を調製し、実験で使用するスライスを保持するために使用する組織保持チャンバーに入れます。多孔質の石を組織保持チャンバーに挿入し、溶液を少なくとも10分間炭水化物化してから、サブ解剖したスライスをチャンバーに入れます。
2.げっ歯類の脳スライスの準備
- 上記のようにすべての溶液を準備します(セクション1を参照)。調製プロセス中にすべての溶液が適切にカルボゲン化されていることを確認してください。
注:切断溶液(セクション1.2を参照)が推奨されるが、ショ糖切断溶液30のような他の切断溶液を使用して、高品質の脳スライスを得ることができる。 - 振動するミクロトームを使用して、350 μmのげっ歯類の脳スライスを作成します。これらの機器を使用するためのプロトコルは、参考文献30、31、32、33に記載されています。
- げっ歯類の脳スライスをMEAチップの記録電極領域に収まるように準備します( 図2を参照)。 図1A および 図2A に示すように、サイズ10の外科用ブレードを使用して、ブレードを脳スライス上で前後に静かに転がすことにより、スライスをサブダイセクションします。振動するミクロトーム切断チャンバーでサブダイセクションを行います。解剖された脳スライスを傷つけないように注意してください。
- サブディスセッションしたスライスを、aCSFで満たされた組織保持チャンバーに入れます。サブ解剖スライスを追加する前に、チャンバーが少なくとも10分間カルボゲン化されていることを確認してください。
3. 機器の準備
- MEA チップとシステムの準備 (図 2 と 図 3)
注意: プラスチックや金属などの硬い材料は、チップの底部に強く接触すると、MEAチップを簡単に損傷する可能性があります。ピペットを使用して溶液をチップに出し入れするときは、チップウェルの底部、特にチップウェルの側面にある参照電極とチップウェルの中央にある記録電極をピペットに接触させないでください(図2C)。代わりに、チップウェルの底にカーソルを合わせるか、チップウェルのプラスチックエッジに接触または近似して、溶液を追加します。すべての溶液を簡単に引き出すには、チップを少し傾けて溶液がチップの片側に溜まるようにして、プールされた溶液の上部から取り除くか、帯電防止ワイプを使用して、まだ溶液が残っている領域を軽くたたきます。チップをしっかりと保持したり、チップピンを固定してチップを輸送したりしないでください(図2C)。チップウェルには約4mLの溶液を保持できます。その後のステップでは、特に指定がない場合は、チップウェルに約2 mLの溶液を充填します。- チップ調製を開始する前に、さまざまなタスク用のトランスファーピペットを指定してください。1つの転写ピペットにエタノール用、1つが廃棄物用、もう1つがaCSF用、残りの溶液にもう1つのラベルを付けて、意図しない混合を防ぎます。
- MEAチップのウェルに190プルーフエタノールを充填し、チップウェルの底が完全に覆われるようにします(図3)。エタノールを30〜60秒間放置してから、廃棄ピペットで取り出します。
- MEAチップのウェルにCSFを充填し、廃棄ピペットで取り出して、チップウェルから残留エタノールを洗い流します。aCSFをチップウェルに3回追加および除去します。これは、事前に指定した廃棄物とaCSFピペットを使用して行います。チップを3回よく洗浄した後、CSFを加えて30秒以上放置します。
注:記録電極は、エタノールとaCSFですすいだ後、aCSFがチャンバー内に少なくとも45分間留まると、疎水性相互作用によるノイズが最も少なくなります。 - MEAチップをドッキングする前に、190プルーフエタノールで帯電防止ワイプを濡らし、それを使用してチップのピンをワイプします(図2C)。
- MEAチップをMEAプラットフォームにそっとスライドさせ、ドッキングメカニズムをかみ合わせてチップを所定の位置にロックします。
- 記録電極と参照電極に気泡がないか確認します(図2C)。気泡がある場合は、きれいな絵筆を取り、電極を軽く掃いて取り除きます。
- CMOS-HD-MEAソフトウェア34 を使用してチップのノイズをチェックし、電気的干渉によって引き起こされる気泡、非生物学的振動、またはスパイクの偽カラーマップを視覚的にスキャンします。MEAシステムを適切に接地して、発生したノイズを打ち消します。
注意: 接地のセットアップは、記録環境によって異なります。このプロトコルの実験では、MEAプラットフォームと灌流システムを接地しました。
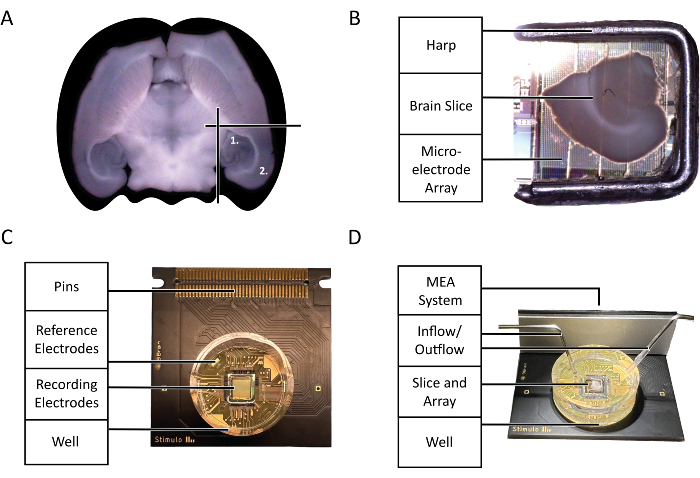
図2:構成図と技術図。 (A)このプロトコルで強調されている実験で使用された急性マウス脳スライスの選択の図。(1)海馬領域 (2)新皮質領域。(B)鋭敏なマウス脳スライスとハープの微小電極アレイ(MEA)上の適切な配置。(C)3Brain Accura CMOS-HD-MEAチップの構造。(D)灌流入口と出口の適切な構成。入力はチップウェルの奥深くにある必要がありますが、出力はチップウェルの上部にあるインレットの反対側にある必要があります。これにより、新鮮な酸素化されたaCSFが一定に流れるようにする必要があります。 この図の拡大版を表示するには、ここをクリックしてください。
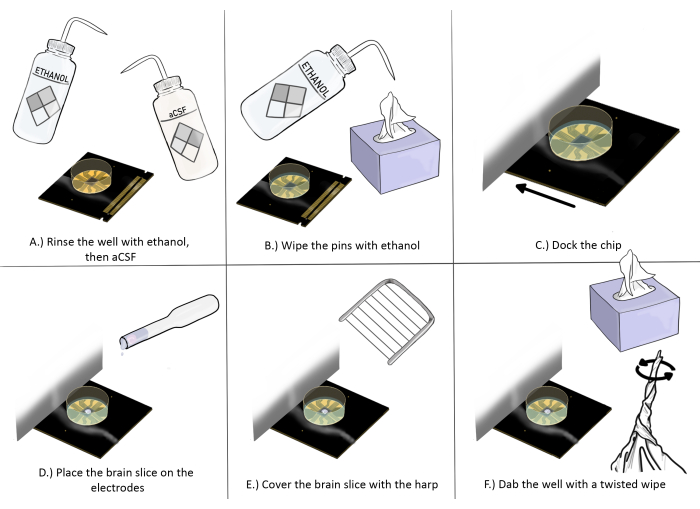
図3:脳スライス実験のためのチップ調製と配置の概略図(A)チップをエタノールで1回、次にCSFで3回よくすすいでください。(B)帯電防止ワイプを使用してピンをエタノールで拭きます。(C)チップをドッキングします。(D)脳スライスを電極に置きます。(E)ハープを脳スライスに置きます(適切な配置ガイドラインについては、図2を参照してください)。(F)ツイストした帯電防止ワイプで、記録電極のウェルの角を脳スライスの近くで軽くたたきます。この図の拡大版を表示するには、ここをクリックしてください。
4. 実験
- ブレインスライスの配置
- プラチナハープをMEAプラットフォームの近くの計量ボート(または別のきれいな表面)に置きます。ハープを約3mLのaCSFで覆い、疎水性の傾向を減らします。
- はさみを使用して、トランスファーピペットの細い先端部分を取り外します。ピペットチップの3分の1、約1.5インチを切り取ります。
注:この改造されたピペットは、収集および輸送中に脳スライスを収縮させたり損傷したりすることはありません。 - スライス保持チャンバーから、改良したピペットで脳スライスを採取します。ブレインスライスとピペット内の溶液をチップウェルに優しく分注します。スライスを適切に配置するには、トランスファーピペットからより多くのaCSFを静かに放出して脳スライスの位置を操作するか、柔らかいペイントブラシを使用して溶液に電流を生成し、脳スライスを記録電極に押し込みます。損傷を最小限に抑えるために、記録電極または脳スライスとの接触を制限します。
- 鉗子を使用して、ハープを脳のスライスの上にそっと置き、スレッドを下にしてスライスを記録電極に押し付けます。電極アレイをハープに接触させないでください。フレームのない側が流入針に向けられ、ハープのフレームが記録電極に接触しないように、ハープを向けます(図2C、D)。
- 廃棄ピペットを取り、余分なaCSFを取り除きます。帯電防止ワイプを取り、角をひねって先端を作り、それを使用して記録電極、ブレインスライス、またはハープに触れることなく、記録電極の周囲に残っているaCSFを吸収します(図2)。
- 指定のaCSFピペットを使用して、脳スライスを覆うのに十分なカルボゲン化aCSFを約2mLすばやく追加します。
- 手順4.1.5を繰り返します。および 4.1.6.さらに2回。
- ウェルが約3/4、約3mLになるまで、ウェルにカルボゲン化aCSFを充填します。
- MEAチップ上の脳スライスを顕微鏡またはカメラで撮影します。写真の解像度が十分に高く、記録電極アレイの境界と脳スライスの解剖学的構造が見えることを確認してください。
- 実験の実行
- 灌流システムの操作
- aCSFを充填したビーカーに流入管と流出管を入れ、流入針と流出針をチップウェルに入れます。インフローニードルをチップの底部近くに配置し、記録電極のすぐ外側に置きます。流出針をチップウェルの上部近く、端に向かって配置し、液体がチップウェルのほぼ縁(約4 mL)まで上昇し、スライスが実験全体を通して沈むようにします(図2D)。
- 灌流流入量を 5 mL/分に、灌流流流入量を 7 mL/分に設定します。
注:チップウェルからの溶液のオーバーフローを防ぎ、ブレインスライスに溶液電流を生成するために、流出速度が流入速度を超えることをお勧めします。 - 流入と流出をオンにします。ニードルが空気の代わりに溶液を出力し始めるまで、チップウェルからインフローニードルを取り外します。次に、手順4.2.1.1で説明したように、針をチップウェル内の位置に戻します。
- 溶液ヒーターを使用して、溶液を生理学的温度(約34〜37°C)に保ちます。
- aCSFを脳スライスに10分間灌流させます。これにより、スライスを記録環境に適合させることができます。
- 10分が経過したら、アウトフローチューブを廃棄ビーカーに移動します。次に、流入チューブをプロけいれん液が入ったビーカーに移動します。.非けいれん性 aCSF を Perflusant システムから Discard Beaker に 10 分間洗い流します。
- アウトフローチューブを、けいれん誘発性溶液を含むビーカーに移します。.
- 実験が終了するまで、痙攣誘発性の溶液を循環させます。
- スライスの記録時間が2時間を超える場合は、活動的な脳スライスのグルコース消費を相殺するために、別の抗けいれん薬溶液を準備することを検討してください。.
- ブレインスライスの交換
- 流入を止めます。その後、10〜15秒後に流出をオフにします。
- 鉗子を使用してハープを取り外し、計量ボートまたはその他の表面に置きます。
- 改造したピペットを使用して、脳のスライスを抽出して廃棄します。参照電極や記録電極アレイには触れないでください(図2C)。
- 流入チューブを非けいれん性のaCSF溶液が入ったビーカーに入れます。.アウトフローチューブを廃棄ビーカーに入れます。インフローとアウトフローの針はチップウェルに残します。灌流システムを10分間実行して、チップウェルと灌流システムからすべての残留抗痙攣促進溶液を除去します。新しい脳のスライスで実験プロセスを再開します(ステップ4.1から開始)。
- 灌流システムの操作
- 実験を終える
- リグのクリーニング
- ハープ、ブレインスライス、溶液をそれぞれウェルから取り出します。
- MEAチップをドッキング解除し、清潔な表面に置き、チップを脱イオン水で十分に満たします。
- 脱イオン水を廃棄して、aCSF溶液から残った塩分を取り除きます。
- トランスファーピペットを使用して、チップに洗剤溶液をよく充填します。トランスファーピペットの先端をチップウェルにホバリングし、記録電極に向けます。洗剤を移送ピペットにすばやく繰り返し絞って放出し、記録電極上で洗剤を1分間激しく洗い流します。洗剤を5〜10分間休ませます。
- 洗剤を取り除き、脱イオン水でチップを4〜6回よくすすいで、洗剤溶液と残っている溶質を取り除きます。
- チップウェルからすべての水を取り除き、MEAチップの上に帯電防止ワイプを置き、チップがよく乾くまで一晩放置します。
- 計量ボートまたは別の防水プレートを、MEAチップが以前に置かれていたMEA記録プラットフォームに置きます。ハープをプレートの中央に置き、ハープの両側に流入針と流出針を置きます。
- 灌流システムとハープを洗浄するには、それぞれ50 mLの水、50 mLの140プルーフエタノール、および200 mLの水をチューブを介して廃棄ビーカーに送ります。.
注意: このとき、灌流速度を上げて洗浄時間を短縮できますが、洪水を避けるために、流出速度は常に流入速度を超える必要があります。 - カルボゲンストーンを蒸留水のビーカーに入れ、カルボゲンをチューブに5分間送って洗浄します。それらを乾燥した清潔な表面に一晩置いて、ほこりの蓄積を避けるためにそれらを覆います。
- リグのクリーニング
5. データ分析
注:CMOS-HD-MEAによって生成された電気生理学的データの分析に使用されるさまざまな分析パッケージがあり、これには3BrainのBrainWave、Yet Another Spike Sorter(YASS)、カスタムPythonツール34、35、36、37が含まれます。図 4 と 図5に示すデータを生成するために、Xenon LFP Analysisプラットフォームで使用するために、BrainWaveデータファイル形式からデータを抽出しました。カスタムMatlabコードを使用して、 図6のデータを解析しました。Xenon LFP Analysisプラットフォームのプロトコルは公開されています10。次のプロトコル手順は、Brainwave 438で作成された録音に固有のものです。その他のシステムについては、それらのシステムに関連するサポート資料34、35、36、37 を参照してください。このプロトコルでデータを生成するために実行される分析手順の概要を以下に示します。チュートリアルビデオや関連するすべてのコードファイルなど、データをエクスポート、視覚化、および分析する方法の詳細については、39を参照してください。
- 分析のために記録ファイルをエクスポートします。
- ダウンサンプリングするデータ記録ファイル用のフォルダを作成します。
- 手順 4.1.9 で撮影した画像をトリミングして、ブレイン スライスを含む記録配列のみを含めます。
- ExportToHDF5-ChannelSelection.py ファイル(このコードファイルはGitHubのxenon-lfp-analysis/code-files/3Brain-processing40からアクセスできます)を開き、ウィンドウの上部にリストされているURLをコピーしてインターネットブラウザに貼り付けます。
- データ記録ファイルのファイルパスを入力し、記録に関連付けられたアレイのトリミングされた画像をアップロードします。
- [ エクスポートするチャネルを選択] で、なげなわ選択ツールを使用して、配列のトリミングされた画像に示されている海馬領域と新皮質領域を選択します。
- スキップする行数とスキップする列数の両方を 2 に設定します。
注:録音ファイルは多くの場合、大きくなります。目的の分析に適している場合、エクスポートされたデータをダウンサンプリングできます。空間的にダウンサンプリングする(記録電極の行と列をスキップする)方法と時間的にダウンサンプリングする(より低いサンプリング周波数にダウンサンプリングする)オプションがあります。分析では、2 行と 2 列ごとにスキップされます。これにより、特定のチャネルに偏ることなく、データを空間的にダウンサンプリングできます。各チャネルは十分に離れているため、アクティビティの違いがそれら間で大きくなる可能性があります。したがって、チャネル平均化は実行されません。電極の行と列をスキップすると、出力ファイルのエクスポート時に選択されていないチャンネルのデータは含まれません。 - ダウンサンプリング周波数を 300 Hz に設定します。
- 「 Export Channels」 ボタンをクリックして、チャンネル選択ファイルを生成します。
注意: ソフトウェアは、チャンネル選択を自動的に保存します file データ記録が保存されているフォルダに file. - ExportToHDF5-ExtractDownsample.py ファイル(GitHubのxenon-lfp-analysis/code-files/3brain-processing40にもあります)を開き、チャネル選択ファイルを含むフォルダのファイルパスとデータ記録ファイルを貼り付け、キーボードのEnterキーを押して、データ記録ファイルのダウンサンプリングバージョンを生成します。
- 分析プラットフォームの使用
- コンピューターのコマンドプロンプトで、「 run_lfp_analysis 」と入力し、Enterキーを押して分析プラットフォームを開きます。
- 画像とダウンサンプリングされた録音ファイルのファイルパスを入力し、[送信]をクリックします。
- [ 分析の時間範囲を選択] で、記録全体を選択します。
- Select Channels for Plotsで、海馬をGroup-1、新皮質をGroup-2としてハイライトします。
- [チャネル ラスター] で、[閾値] を 0.06 mV に、[時間継続時間] を 0.02 秒に設定します。
- [ 設定の適用] と [ラスターの生成] をクリックします。
- ラスター プロットを使用して、関心のあるアクティビティ パターンを示すさまざまなチャネルを探索します。
注:高品質の発作様活動(図4A-D)と最適でない活動(図4E-F)の例については、図4を参照してください。
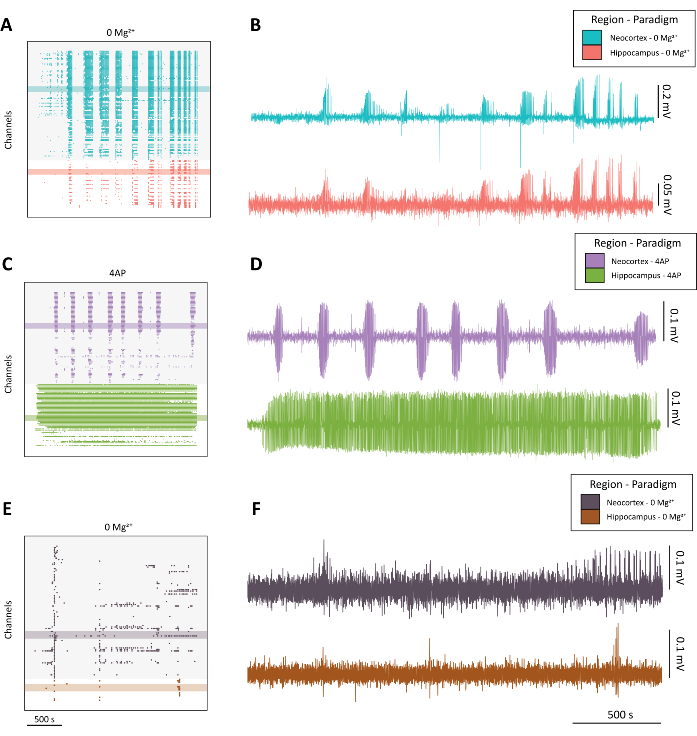
図4:0 Mg2+ および4-APパラダイムからの進化するてんかん様活性の例。 (A)0 Mg2+ のaCSFを約40分間適用した際のラスタープロットの例。(B)新皮質(青)と海馬(赤)から採取した電気生理学的トレースの例は、0 Mg2+ パラダイムからのてんかん様活動を示しています。(C)100 mM 4-APを約40分間にわたって適用したラスタプロットの例。(D)新皮質(紫色)と海馬(緑)から採取した電気生理学的トレースの例は、4-APの適用によるてんかん様活動を示しています。(E)0 Mg2+ を約40分間にわたってaCSFに適用した結果、他の代表的なトレースに見られる発作様活動とは対照的に、バースト活動を示すラスタプロットの例。(F)新皮質(濃い紫色)と海馬(錆)から採取された電気生理学の痕跡の例は、BとDで見つかった質の高い発作様活動と比較することを意図した0 Mg2+ パラダイムからの最適以下の活動を示しています 。
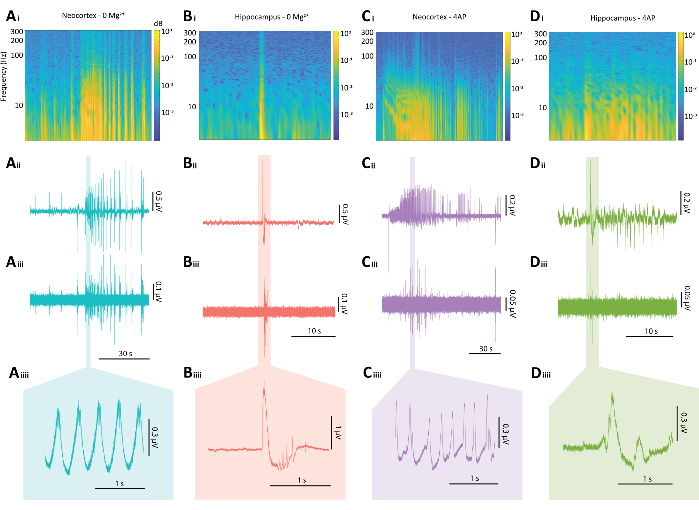
図5:0 Mg2+ および4-APパラダイムの両方からのてんかん様放電の代表的な結果。 (A)0 Mg2+ パラダイムによって誘発される典型的な新皮質発作様事象の例プロットには、(Ai)発作様事象からのスペクトログラム、(Aii)関連する電気生理学的トレース、(Aiii)Aiiからのトレースに適用された80Hzハイパスフィルタ、(Aiiii)およびAiiからのトレースの拡大セクションが含まれます。(B)0 Mg2+ パラダイムによって誘発された典型的な海馬てんかん様バーストの例プロットには、(Bi)てんかん様バーストのスペクトログラム、(Bii)関連する電気生理学的トレース、(Biii)Biiからのトレースに適用された80Hzハイパスフィルタ、(Biiii)およびBiiからのトレースの拡大セクション、(C)を含む4-APパラダイムによって誘発された典型的な新皮質発作様イベントのプロットの例(Ci)てんかん様活動のスペクトログラム、(Cii)関連する電気生理学的トレース、(Ciii)Ciiからのトレースに適用された80Hzハイパスフィルタ、(Ciiii)およびCiiからのトレースの拡大セクション、(D)4-APパラダイムの下で破裂した海馬てんかん様の例のプロット、(Di)てんかん様活動のスペクトログラム、(Dii)関連する電気生理学的トレース、 (Diii)Diiからのトレースに適用される80Hzのハイパスフィルター、(Diiii)およびDiiからのトレースの拡大セクション。 この図の拡大版を表示するには、ここをクリックしてください。
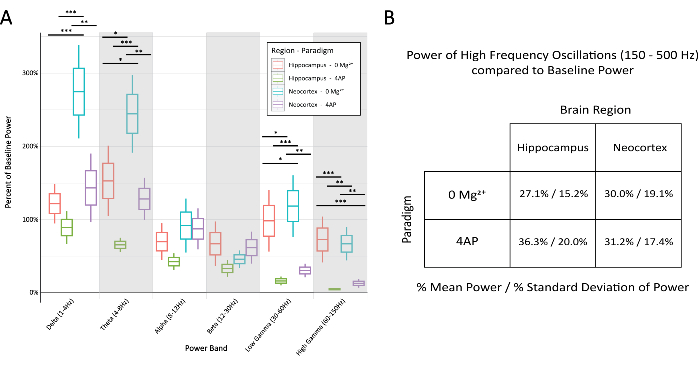
図6:常型てんかん様放電中のパラダイムと脳領域全体のさまざまな帯域におけるベースラインパワーの割合の比較(A)てんかん様放電中のパワーは、ほとんどの周波数帯域でパラダイムと脳領域の間で有意に異なっていました(テューキー検定による2元配置ANOVA、*P < 0.05、**P < 0.001、***P < 0.0001)。各ボックスの中央の線は平均を表し、ボックスの境界±1 平均の標準誤差 (SEM)、および最も外側の線 ±2 SEM. (B) パラダイムと脳領域の両方が、150 Hz を超える高周波活動に関連する帯域で限られたパワーを示しました。この図の拡大版を表示するには、ここをクリックしてください。
結果
多くのチャネル1、4、5、10からのアクティビティを視覚化する場合に標準的であるため、最初にCMOS-HD-MEA(図4A、C、E)で取得したデータのラスタープロットを生成することが有益であることがわかります。このプロットでは、?...
ディスカッション
このプロトコルには、CMOS-HD-MEAユーザーが直面する一般的な問題、すなわち脳スライス下のノイズ発生と脳スライスの健全な環境の維持に対処する、急性脳スライス管理に関連する特定のガイドラインが含まれています。スライスの下のノイズ発生は、スライスがアレイに適切に接着されていない場合に発生します。脳スライスが十分に接着されていないと、スライ?...
開示事項
著者らは、この調査研究に関連する利益相反がないことを宣言します。
謝辞
著者らは、この原稿の編集について、元および現在のParrish研究室のメンバーに感謝します。また、この作業に対するフィードバックを提供してくださった3BrainのAlessandro Maccione氏にも感謝します。この研究は、AES/EF Junior Investigator AwardとBrigham Young University Colleges of Life Sciences、of Physical and Mathematical Sciencesから資金提供を受けました。
資料
| Name | Company | Catalog Number | Comments |
| 2D Workbench | Cloudray | LM04CLLD26B | |
| 4-Aminopyridine | Sigma-Aldrich | 275875 | |
| Accura Chip | 3Brain | Accura HD-MEA | CMOS-HD-MEA chip |
| Agarose | Thermo Fisher Scientific | BP160-100 | |
| Vibration isolation table | Kinetic Systems | 91010124 | |
| Beaker for the slice holding chamber, 270 mL | VWR | 10754-772 | |
| BioCam | 3Brain | BioCAM DupleX | CMOS-HD-MEA platform |
| Brainwave Software | 3Brain | Version 4 | CMOS-HD-MEA software |
| Calcium Chloride | Thermo Fisher Scientific | BP510-500 | |
| Carbogen | Airgas | X02OX95C2003102 | |
| Carbogen | Airgas | 12005 | |
| Carbogen Stones | Supelco | 59277 | |
| Compresstome | Precissionary | VF-300-0Z | |
| Computer | Dell | Precission3650 | |
| Crocodile Clip Grounding Cables | JWQIDI | B06WGZG17W | |
| Detergent | Metrex | 10-4100-0000 | |
| D-Glucose | Macron Fine Chemicals | 4912-12 | |
| Dihydrogen Sodium Phosphate | Thermo Fisher Scientific | BP329-500 | |
| DinoCam | Dino-Lite | AM73915MZTL | |
| Ethanol | Thermo Fisher Scientific | A407P-4 | |
| Forceps | Fine Science Tools | 11980-13 | |
| Hot plate | Thermo Fisher Scientific | SP88857200 | |
| Ice Machine | Hoshizaki | F801MWH | |
| Inflow and outflow needles | Jensen Global | JG 18-3.0X | |
| Inline Solution Heater | Warner Instruments | SH-27B | |
| Isofluorine | Dechra | 08PB-STE22002-0122 | |
| Kim Wipes | Thermo Fisher Scientific | 06-666 | |
| Magnesium Chloride | Thermo Fisher Scientific | FLM33500 | |
| Micropipets | Gilson | F144069 | |
| Mili-Q Water Filter | Mili-Q | ZR0Q008WW | |
| Paintbrush | Daler Rowney | AF85 Round: 0 | |
| Paper Filter | Whatman | EW-06648-24 | |
| Parafilm | American National Can | PM996 | |
| Perfusion System | Multi Channel System | PPS2 | |
| Pipetor | Thermo Fisher Scientific | FB14955202 | |
| Platinum Harp | 3Brain | 3Brain | |
| Potassium Chloride | Thermo Fisher Scientific | P330-3 | |
| Razor blade | Personna | BP9020 | |
| Scale | Metter Toledo | AB204 | |
| Scissors | Solingen | 92008 | |
| Slice Holding Chamber | Custom | Custom | Custom 3D Printer Design, available upon request |
| Sodium Bicarbonate | Macron Fine Chemicals | 7412-06 | |
| Sodium Chloride | Thermo Fisher Scientific | S271-3 | |
| Temperature Control Box | Warner Instruments | TC344B | |
| Transfer Pipettes | Genesee Scientific | 30-200 | |
| Tubing | Tygon | B-44-3 TPE | |
| Vibratome VZ-300 | Precissionary | VF-00-VM-NC | |
| Weigh Boat | Electron Microscopy Sciences | 70040 |
参考文献
- Obien, M. E. J., Frey, U. Large-scale, high-resolution microelectrode arrays for interrogation of neurons and networks. Adv Neurobiol. 22, 83-123 (2019).
- Schroter, M., et al. Functional imaging of brain organoids using high-density microelectrode arrays. MRS Bull. 47 (6), 530-544 (2022).
- Miccoli, B., et al. High-density electrical recording and impedance imaging with a multi-modal CMOS multi-electrode array chip. Front Neurosci. 13, 641 (2019).
- Emery, B. A., Hu, X., Khanzada, S., Kempermann, G., Amin, H. High-resolution CMOS-based biosensor for assessing hippocampal circuit dynamics in experience-dependent plasticity. Biosens Bioelectron. 237, 115471 (2023).
- Ferrea, E., et al. high-resolution electrophysiological imaging of field potentials in brain slices with microelectronic multielectrode arrays. Front Neural Circuits. 6, 80 (2012).
- Gagliano, G., et al. Non-linear frequency dependence of neurovascular coupling in the cerebellar cortex implies vasodilation-vasoconstriction competition. Cells. 11 (6), 1047 (2022).
- Goodchild, S. J., et al. Molecular pharmacology of selective Na(V)1.6 and dual Na(V)1.6/Na(V)1.2 channel inhibitors that suppress excitatory neuronal activity ex vivo. ACS Chem Neurosci. 15 (6), 1169-1184 (2024).
- Hu, X., Khanzada, S., Klutsch, D., Calegari, F., Amin, H. Implementation of biohybrid olfactory bulb on a high-density CMOS-chip to reveal large-scale spatiotemporal circuit information. Biosens Bioelectron. 198, 113834 (2022).
- Kim, S., et al. Alteration of neural network and hippocampal slice activation through exosomes derived from 5XFAD nasal lavage fluid. Int J Mol Sci. 24 (18), 14064 (2023).
- Mahadevan, A., Codadu, N. K., Parrish, R. R. Xenon LFP analysis platform is a novel graphical user interface for analysis of local field potential from large-scale MEA recordings. Front Neurosci. 16, 904931 (2022).
- Medrihan, L., Ferrea, E., Greco, B., Baldelli, P., Benfenati, F. Asynchronous GABA release is a key determinant of tonic inhibition and controls neuronal excitability: A study in the synapsin II-/- mouse. Cereb Cortex. 25 (10), 3356-3368 (2015).
- Monteverdi, A., Di Domenico, D., D'Angelo, E., Mapelli, L. Anisotropy and frequency dependence of signal propagation in the cerebellar circuit revealed by high-density multielectrode array recordings. Biomedicines. 11 (5), 1475 (2023).
- Obien, M. E. J., Hierlemann, A., Frey, U. Accurate signal-source localization in brain slices by means of high-density microelectrode arrays. Sci Rep. 9 (1), 788 (2019).
- Thouta, S., et al. Pharmacological determination of the fractional block of Nav channels required to impair neuronal excitability and ex vivo seizures. Front Cell Neurosci. 16, 964691 (2022).
- Tognolina, M., Monteverdi, A., D'Angelo, E. Discovering microcircuit secrets with multi-spot imaging and electrophysiological recordings: The example of cerebellar network dynamics. Front Cell Neurosci. 16, 805670 (2022).
- Hierlemann, A., Frey, U., Hafizovic, S., Heer, F. Growing cells atop microelectronic chips: Interfacing electrogenic cells in vitro with CMOS-based microelectrode arrays. Proceedings of the IEEE. 99 (2), 252-284 (2011).
- Maccione, A., et al. Experimental investigation on spontaneously active hippocampal cultures recorded by means of high-density MEAs: Analysis of the spatial resolution effects. Front Neuroeng. 3, 4 (2010).
- van Vliet, E., et al. Electrophysiological recording of re-aggregating brain cell cultures on multi-electrode arrays to detect acute neurotoxic effects. Neurotoxicology. 28 (6), 1136-1146 (2007).
- Emery, B. A., et al. Large-scale multimodal recordings on a high-density neurochip: Olfactory bulb and hippocampal networks. Annu Int Conf IEEE Eng Med Biol Soc. 2022, 3111-3114 (2022).
- Veleanu, M., et al. Modified climbing fiber/Purkinje cell synaptic connectivity in the cerebellum of the neonatal phencyclidine model of schizophrenia. Proc Natl Acad Sci U S A. 119 (21), e2122544119 (2022).
- Giansante, G., et al. Neuronal network activity and connectivity are impaired in a conditional knockout mouse model with PCDH19 mosaic expression. Mol Psychiatry. , (2023).
- Dossi, E., Blauwblomme, T., Nabbout, R., Huberfeld, G., Rouach, N. Multi-electrode array recordings of human epileptic postoperative cortical tissue. J Vis Exp. (92), e51870 (2014).
- Hajos, N., et al. Maintaining network activity in submerged hippocampal slices: importance of oxygen supply. Eur J Neurosci. 29 (2), 319-327 (2009).
- Hill, M. R., Greenfield, S. A. The membrane chamber: a new type of in vitro recording chamber. J Neurosci Methods. 195 (1), 15-23 (2011).
- Raimondo, J. V., et al. Methodological standards for in vitro models of epilepsy and epileptic seizures. A TASK1-WG4 report of the AES/ILAE Translational Task Force of the ILAE. Epilepsia. 58 (Suppl 4), 40-52 (2017).
- Hales, C. M., Rolston, J. D., Potter, S. M. How to culture, record and stimulate neuronal networks on micro-electrode arrays (MEAs). J Vis Exp. (39), 2056 (2010).
- Lin, C. H., Lee, J. K., LaBarge, M. A. Fabrication and use of microenvironment microarrays (MEArrays). J Vis Exp. (68), 4152 (2012).
- Panuccio, G., Colombi, I., Chiappalone, M. Recording and modulation of epileptiform activity in rodent brain slices coupled to microelectrode arrays. J Vis Exp. 135, 57548 (2018).
- Patel, C., Muthuswamy, J. High efficiency, site-specific transfection of adherent cells with siRNA using microelectrode arrays (MEA). J Vis Exp. 67, e4415 (2012).
- Ting, J. T., Daigle, T. L., Chen, Q., Feng, G. Acute brain slice methods for adult and aging animals: application of targeted patch clamp analysis and optogenetics. Methods Mol Biol. 1183, 221-242 (2014).
- Papouin, T., Haydon, P. G. Obtaining acute brain slices. Bio Protoc. 8 (2), e2699 (2018).
- Ting, J. T., et al. Preparation of acute brain slices using an optimized N-Methyl-D-glucamine protective recovery method. J Vis Exp. 132, 53825 (2018).
- Van Hoeymissen, E., Philippaert, K., Vennekens, R., Vriens, J., Held, K. Horizontal hippocampal slices of the mouse brain. J Vis Exp. (163), 61753 (2020).
- . 3Brain Available from: https://www.3brain.com/ (2022)
- Bridges, D. C., Tovar, K. R., Wu, B., Hansma, P. K., Kosik, K. S. MEA Viewer: A high-performance interactive application for visualizing electrophysiological data. PLoS One. 13 (2), e0192477 (2018).
- Hawrylycz, M., et al. Inferring cortical function in the mouse visual system through large-scale systems neuroscience. Proc Natl Acad Sci U S A. 113 (27), 7337-7344 (2016).
- Maccione, A., et al. Microelectronics, bioinformatics and neurocomputation for massive neuronal recordings in brain circuits with large scale multielectrode array probes. Brain Res Bull. 119 (Pt B), 118-126 (2015).
- . 3Brain Available from: https://www.3brain.com/products/software/brainwave4 (2022)
- Mahadevan, A. . Xenon LFP Analysis. , (2022).
- Mahadevan, A. . xenon-lfp-analysis github. , (2022).
- Codadu, N. K., et al. Divergent paths to seizure-like events. Physiol Rep. 7 (19), e14226 (2019).
- Kirsch, G. E., Drewe, J. A. Gating-dependent mechanism of 4-aminopyridine block in two related potassium channels. J Gen Physiol. 102 (5), 797-816 (1993).
- Levesque, M., Salami, P., Behr, C., Avoli, M. Temporal lobe epileptiform activity following systemic administration of 4-aminopyridine in rats. Epilepsia. 54 (4), 596-604 (2013).
- Myers, T. L., Gonzalez, O. C., Stein, J. B., Bazhenov, M. Characterizing concentration-dependent neural dynamics of 4-Aminopyridine-induced epileptiform activity. Epilepsy J. 4 (2), 128 (2018).
- Perreault, P., Avoli, M. Physiology and pharmacology of epileptiform activity induced by 4-aminopyridine in rat hippocampal slices. J Neurophysiol. 65 (4), 771-785 (1991).
- Rutecki, P. A., Lebeda, F. J., Johnston, D. 4-Aminopyridine produces epileptiform activity in hippocampus and enhances synaptic excitation and inhibition. J Neurophysiol. 57 (6), 1911-1924 (1987).
- Chen, Y., Chad, J. E., Cannon, R. C., Wheal, H. V. Reduced Mg2+ blockade of synaptically activated N-methyl-D-aspartate receptor-channels in CA1 pyramidal neurons in kainic acid-lesioned rat hippocampus. Neuroscience. 88 (3), 727-739 (1999).
- Fujiwara-Tsukamoto, Y., Isomura, Y., Takada, M. Comparable GABAergic mechanisms of hippocampal seizure-like activity in posttetanic and low-Mg2+ conditions. J Neurophysiol. 95 (3), 2013-2019 (2006).
- Swartzwelder, H. S., Anderson, W. W., Wilson, W. A. Mechanism of electrographic seizure generation in the hippocampal slice in Mg2+-free medium: the role of GABAa inhibition. Epilepsy Res. 2 (4), 239-245 (1988).
- Trevelyan, A. J., Graham, R. T., Parrish, R. R., Codadu, N. K. Synergistic positive feedback mechanisms underlying seizure initiation. Epilepsy Curr. 23 (1), 38-43 (2023).
- Croning, M. D., Haddad, G. G. Comparison of brain slice chamber designs for investigations of oxygen deprivation in vitro. J Neurosci Methods. 81 (1-2), 103-111 (1998).
- Hajos, N., Mody, I. Establishing a physiological environment for visualized in vitro brain slice recordings by increasing oxygen supply and modifying aCSF content. J Neurosci Methods. 183 (2), 107-113 (2009).
- Huang, Y., Williams, J. C., Johnson, S. M. Brain slice on a chip: opportunities and challenges of applying microfluidic technology to intact tissues. Lab Chip. 12 (12), 2103-2117 (2012).
- Andrew, R. D., et al. The critical role of spreading depolarizations in early brain injury: Consensus and contention. Neurocrit Care. 37 (Suppl 1), 83-101 (2022).
- Devonshire, I. M., Dommett, E. J., Grandy, T. H., Halliday, A. C., Greenfield, S. A. Environmental enrichment differentially modifies specific components of sensory-evoked activity in rat barrel cortex as revealed by simultaneous electrophysiological recordings and optical imaging in vivo. Neuroscience. 170 (2), 662-669 (2010).
- Parrish, R. R., Codadu, N. K., Mackenzie-Gray Scott, C., Trevelyan, A. J. Feedforward inhibition ahead of ictal wavefronts is provided by both parvalbumin- and somatostatin-expressing interneurons. J Physiol. 597 (8), 2297-2314 (2019).
- Wang, H., Jing, M., Li, Y. Lighting up the brain: genetically encoded fluorescent sensors for imaging neurotransmitters and neuromodulators. Curr Opin Neurobiol. 50, 171-178 (2018).
- Yaksi, E., Jamali, A., Diaz Verdugo, C., Jurisch-Yaksi, N. Past, present and future of zebrafish in epilepsy research. FEBS J. 288 (24), 7243-7255 (2021).
- He, M. F., et al. Ex vivo calcium imaging for drosophila model of epilepsy. J Vis Exp. 200, 65825 (2023).
- Driscoll, N., et al. Multimodal in vivo recording using transparent graphene microelectrodes illuminates spatiotemporal seizure dynamics at the microscale. Commun Biol. 4 (1), 136 (2021).
- Parrish, R. R., Grady, J., Codadu, N. K., Trevelyan, A. J., Racca, C. Simultaneous profiling of activity patterns in multiple neuronal subclasses. J Neurosci Methods. 303, 16-29 (2018).
- Valderhaug, V. D., et al. Criticality as a measure of developing proteinopathy in engineered human neural networks. bioRxiv. , (2020).
- Carleo, G., Lee, Y. -. S., Secondo, A., Miceli, F., Taglialatela, M. Multi-electrode array (MEASs) to investigate pathogenetic disease mechanisms and pharmacological properties in iPSC-derived neurons modelling neuropsychiatric diseases. , 667-672 (2022).
- Ruz, I. D., Schultz, S. R. Localising and classifying neurons from high density MEA recordings. J Neurosci Methods. 233, 115-128 (2014).
- Franke, F., Natora, M., Boucsein, C., Munk, M. H. J., Obermayer, K. An online spike detection and spike classification algorithm capable of instantaneous resolution of overlapping spikes. J Comput Neurosci. 29 (1-2), 127-148 (2010).
- Vollgraf, R., Obermayer, K. Improved optimal linear filters for the discrimination of multichannel waveform templates for spike-sorting applications. IEEE Signal Processing Letters. 13 (3), 121-124 (2006).
- Muller, J., et al. High-resolution CMOS MEA platform to study neurons at subcellular, cellular, and network levels. Lab Chip. 15 (13), 2767-2780 (2015).
- Mapelli, L., et al. implementation, and functional validation of a new generation of microneedle 3D high-density CMOS multi-electrode array for brain tissue and spheroids. bioRxiv. , (2022).
- Reddy, D. S., Kuruba, R. Experimental models of status epilepticus and neuronal injury for evaluation of therapeutic interventions. Int J Mol Sci. 14 (9), 18284-18318 (2013).
- Parrish, R. R., Trevelyan, A. J. Stress-testing the brain to understand its breaking points. J Physiol. 596 (11), 2033-2034 (2018).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved